郑豆豆#, 罗素亚#, 杜穆花, 何广正, 徐书景, 鞠建松


河北师范大学生命科学学院, 河北 石家庄 050024
收稿日期:2020-01-15;修回日期:2020-04-07;网络出版日期:2020-08-18
基金项目:国家自然科学基金(31971204);河北师范大学科研基金(L2017K07,L2019B30);河北师范大学研究生创新资助项目
*通信作者:鞠建松, Tel:+86-311-80787573;Fax:+86-311-80789794;E-mail:jujiansong@126.com.
#并列第一作者
摘要:[目的] 通过易错PCR技术提高鼠伤寒沙门氏菌中丙氨酸消旋酶的催化活性。[方法] 利用易错PCR技术构建丙氨酸消旋酶基因alrSt的突变体文库,采用缺陷菌株UT5028筛选突变体基因,以D-氨基酸氧化酶偶联法检测各突变蛋白的活性,通过凝胶过滤层析法分析酶蛋白寡聚化状态,并采用HPLC检测酶蛋白的动力学参数。[结果] 经过易错PCR及定点突变技术最终获得了3个催化活性有所提高的突变体A3V、Y343H和A3VY343H,酶学特性分析发现,与野生型蛋白StAlr相比,突变体Y343H仅对底物L/D-丝氨酸的催化效率略有提高,kcat/Km值分别是StAlr的2.01和3.68倍;而突变体A3V则对底物L/D-丙氨酸或L/D-丝氨酸的Km、kcat和kcat/Km值均有较大幅度的改变,其kcat/Km值分别是StAlr的105.51、97.36、4.63和10.73倍。凝胶过滤层析结果显示,突变体A3V在蛋白含量极低时就呈现出单体和二聚体共存状态,且随着蛋白含量的增加,其向二聚体状态迁移的速率最为明显。[结论] 丙氨酸消旋酶StAlr的第3位点是影响其催化活性和低聚合状态的关键位点。
关键词:鼠伤寒沙门氏菌丙氨酸消旋酶易错PCR低聚化催化活性
Improving catalytic activity of alanine racemase from Salmonella typhimurium by error-prone PCR
Doudou Zheng#, Suya Luo#, Muhua Du, Guangzheng He, Shujing Xu, Jiansong Ju


College of Life Sciences, Hebei Normal University, Shijiazhuang 050024, Hebei Province, China
Received: 15 January 2020; Revised: 7 April 2020; Published online: 18 August 2020
*Corresponding author: Jiansong Ju, Tel:+86-311-80787573;Fax:+86-311-80789794;E-mail:jujiansong@126.com.
Foundation item: Supported by the National Natural Science Foundation of China (31971204), by the Foundation of Hebei Normal University (L2017K07, L2019B30) and by the Innovation Funding Program for Graduate Students of Hebei Normal University
#These authors contributed equally to this work
Abstract: [Objective] To enhance the catalytic activity of alanine racemase from Salmonella typhimurium by error-prone PCR. [Methods] A mutant library of alanine racemase from S. typhimurium was constructed by error-prone PCR using plasmid pTrc99A-StAlr or pTrc99A-Y343H as template, and DNA recombination with improved catalytic activity were screened by serine auxotroph strain UT5028. Racemase activities for converting both L-alanine to D-alanine and L-serine to D-serine were calculated based on the absorbance at 550 nm using Epoch Microplate Spectrophotometer. The cell lysate was separated by gel filtration chromatography, and each fraction was detected for the alanine racemase activity to analyze the oligomerization states. Kinetic parameters of StAlr and mutants were determined by measuring the total amount of L/D-alanine or L/D-serine by high performance liquid chromatography with a spectrofluorometer. [Results] Three mutants Y343H, A3VY343H and A3V with improved catalytic activities were obtained by two rounds of error-prone PCR and site-directed mutagenesis, separately. Based on the kinetic parameters, the mutant Y343H only displayed a 2.01 and 3.68-fold improvement in catalytic efficiency (kcat/Km) towards L/D-serine compared to the wild type StAlr, while the mutant A3V showed a distinct reduction in Km value and dramatic increase in kcat and kcat/Km values towards L/D-alanine and L/D-serine. For substrate L-alanine and D-alanine, the kcat/Km values of A3V were 105.51 and 97.36-fold of that of wild type StAlr, whereas for L-serine and D-serine, the kcat/Km values of A3V were 4.63 and 10.73-fold of that of wild type StAlr. Gel filtration chromatography revealed that only the mutant A3V eluted as two distinct peaks at a very low amount of protein, which may correspond to the dimeric and monomeric form, respectively. As the amount of protein increased, the oligomerization states of all proteins were gradually shifted from monomeric to dimeric form. These indicated that the monomer-dimer transition of mutant A3V might be much faster than that of protein StAlr and A3VY343H. [Conclusion] The residue A3 located at N-terminus of StAlr might be a key residue for its catalytic activity and oligomerization state.
Keywords: Salmonella typhimuriumalanine racemaseerror-prone PCRoligomerizationcatalytic activity
丙氨酸消旋酶(alanine racemase,Alr,EC 5.1.1.1)是以Pyridoxal 5′-phosphate (PLP)为辅酶催化L-丙氨酸和D-丙氨酸旋光性转换的一类异构酶[1]。丙氨酸消旋酶广泛分布于原核生物,为细菌细胞壁肽聚糖层的合成提供D-丙氨酸。丙氨酸消旋酶基因在不同菌株中的数量不同,如假坚强芽孢杆菌(Bacillus pseudofirmus)和志贺氏菌(Shigella)等中只含有1个基因,而在大肠杆菌(Escherichia coli)和枯草芽孢杆菌(Bacillus subtilis)等中却存在2个[2]。研究表明,尽管同一菌株中的两个丙氨酸消旋酶具有相同的催化功能,但二者分别属于同化作用型(Alr)和异化作用型(DadX/B)[3-4]。
丙氨酸消旋酶除了作为抗菌药物筛选的靶标广受关注外,它还是生物合成D-氨基酸的关键酶之一。Galkin等曾提出利用甲酸脱氢酶、L-丙氨酸脱氢酶、丙氨酸消旋酶和D-氨基酸转氨酶等共同作用生产D-氨基酸的方法[5]。此方法以甲酸脱氢酶实现NADH循环再生,降低了生产成本;不过,受微生物酶蛋白催化活性低和稳定性差等方面的影响,该方法未能进一步推广和应用。因此,筛选或改造获得催化活性高、稳定性好的酶蛋白是生产非天然D-氨基酸的重要前提。
当前开展酶分子改造策略主要分为两大类,即理性设计和非理性设计[6]。理性设计则是基于序列和结构信息,通过计算机预测目标突变体,从而实现高效改造酶蛋白的目的[7-8]。国内外有多个科研团队通过理性设计的方法成功筛选获得了理想的突变体,然而,受蛋白空间结构、计算量庞大和成功率低等因素的影响,理性设计技术的推广受到一定程度的限制[9]。酶的非理性设计又称为定向进化(directed evolution),包括定点突变、易错PCR和DNA重组等技术,主要通过随机突变或片段重组来改造蛋白。尽管这类方法的突变效率相对较低,但由于其操作简便,且无需了解蛋白的序列及结构,通过多轮突变亦能获得显著提高的突变体[6]。Tang等通过多次交替使用易错PCR、DNA改组和定点突变技术,使黑曲霉来源的植酸酶在酶活性和热稳定性方面都得到了大幅提升[10]。因此,非理性设计在酶蛋白的定向进化仍然占有重要地位。
本研究通过易错PCR技术,以鼠伤寒沙门氏菌(Salmonella typhimurium)的丙氨酸消旋酶基因(alrSt)为目的基因,构建突变体文库,通过两轮筛选最终获得了酶学特性有所提高的突变体A3V/Y343H,并通过定点突变技术确定了影响酶蛋白催化活性的关键位点,为丙氨酸消旋酶的进一步改造和应用提供实验依据。
1 材料和方法 1.1 菌株、质粒和试剂 大肠杆菌MB2795 (alr::frt,dadX::frt)为D-丙氨酸营养缺陷型菌株[11],菌株UT5028 (serB::frt)为丝氨酸营养缺陷型菌株[12],大肠杆菌(E. coli) DH12S用于基因克隆,E. coli BL21(DE3)用于蛋白表达;M9基础培养基用于缺陷菌株的培养[12];质粒pET-22b(+)和pTrc99A用于蛋白表达,含有丙氨酸消旋酶基因(alrSt)的质粒pTrc99A-StAlr由高知大学大西浩平教授惠赠,基因alrSt两端分别含有唯一的限制内切酶Hind Ⅲ和Xba Ⅰ识别位点[12]。
Taq DNA polymerase、T4 DNA Ligase、限制性内切酶等购自大连宝生物公司,Ni-NTA agarose购自QIAGEN公司,Low molecular weight standards购自生工生物工程(上海)股份有限公司,4-Aminoantipyrine、N-ethyl-N-(2-hydroxy-3- sulfopropyl)-3-methylaniline sodium salt (TOOS)、Peroxidase和D-amino acid oxidase购自Sigma公司;其他常用试剂均为分析纯。
1.2 易错PCR法构建突变文库 以表达载体pTrc99A-StAlr为模板进行易错PCR,引物为Rv-M (5′-GAGCGGATAACAATTTC ACACAGG-3′)和Trc-B (5′-TCTGTTTTATCAGA CCGCTTC-3′)。PCR反应程序为:85 ℃ 5 min;95 ℃ 45 s,54 ℃ 1 min,72 ℃ 2 min,50个循环;72 ℃ 2 min。
易错PCR产物经0.8%琼脂糖凝胶电泳分离、纯化,以限制性内切酶Hind Ⅲ和Xba Ⅰ双酶切处理,与经相同酶切处理的质粒pTrc99A连接;通过电转化法将连接产物转入感受态细胞DH12S,加入1 mL SOC培养液于37 ℃恒温培养1 h,涂布在含有Amp的LB固体培养基上,37 ℃培养过夜;收集所有菌落,提取质粒DNA混合物,采用乙醇沉淀法浓缩质粒DNA。
以第一轮筛选所得最佳突变体为模板,重复上述步骤,进行第二轮易错PCR。
1.3 突变体文库的筛选 采用电转化法将浓缩质粒DNA转入感受态细胞UT5028中,经SOC培养液预培养后,分别涂布在含Amp和D-丝氨酸(5.0 mmol/L)的M9基础培养基上,37 ℃下培养40 h;将所有克隆子分别培养于含Amp的LB液体培养基中,37 ℃过夜培养后离心收集菌体,提取其质粒DNA;随后通过化学转化法将质粒DNA再次转入UT5028中,经37 ℃过夜培养后,挑取克隆子划线培养于含Amp的M9培养基上,继续培养以剔除其中的假阳性突变体。
将筛选获得的突变体质粒及野生型pTrc99A- StAlr各取3.0 μL,通过化学转化法转入感受态细胞MB2795中,涂布于含Amp的LB固体培养基上,于37 ℃过夜培养;挑取单一克隆子接种于LB+Amp培养液中振荡培养,次日移取培养物100 μL转入5 mL含有Amp和IPTG (0.1 mmol/L)的LB培养液中培养7 h,离心收集菌体。
将菌体重悬于0.5 mL含有10 μmol/L 5′-磷酸吡哆醛(5′-phosphopyridoxal,PLP)和0.01% 2-巯基乙醇(2-mercaptoethanol,2-ME)的磷酸钾缓冲液(50 mmol/L,pH 7.4)中,通过超声破碎法裂解细胞,4 ℃下15000 r/min离心10 min除去细胞碎片,采用Protein Assay Reagent (Bio-Rad)检测裂解液的蛋白浓度。
1.4 构建单点突变体 分析发现,基因alrSt 861–866处和空载体pTrc99A的1150–1155处分别存在限制内切酶Sca Ⅰ的唯一识别位点(AGTACT)。为了分析单一位点突变(A3V)对酶蛋白的影响,采用限制内切酶ScaⅠ单酶切处理载体pTrc99A-StAlr和pTrc99A- A3VY343H,通过0.8%琼脂糖凝胶电泳分离获得不含突变位点Y343H、片段大小约为1 kb的DNA片段,以及含有突变位点A3V、片段大小约为4.2 kb的DNA片段,将二者混合通过T4 DNA连接酶相连接,构建含有单一位点突变的载体pTrc99A-A3V。
1.5 构建表达载体 以质粒pTrc99A-StAlr和筛选获得的突变体质粒DNA为模板,正向引物为StalrA2 (5′-CATATGCAAGCGGCAACAGTCGTC-3′)或Ep3A1 (5′-CATATGCAAGTGGCAACAGTC-3′,仅用于构建pET-A3V),反向引物为StalrB2 (5′- CTCGAGATCAATATACTTCATCGCCAC-3′),下划线分别为限制内切酶Nde Ⅰ和Xho Ⅰ识别位点。通过PCR扩增丙氨酸消旋酶基因,反应程序为:85 ℃ 5 min;95 ℃ 45 s,54 ℃ 1 min,72 ℃ 2 min,25个循环;72 ℃ 2 min。PCR产物经0.8%琼脂糖凝胶电泳分离、纯化后采用Nde Ⅰ和Xho Ⅰ双酶切,酶切产物与经相同酶切处理的载体pET-22b(+)连接,构建表达载体pET-StAlr、pET-A3V、pET-Y343H和pET-A3VY343H。
1.6 酶蛋白的纯化 将上述4个表达载体分别转入BL21(DE3)感受态细胞中,于LB培养液(Amp+)中培养至OD600为0.6,加入诱导剂IPTG (终浓度为1.0 mmol/L),30 ℃诱导5 h后,离心收集菌体。
加入细胞裂解液(50 mmol/L NaH2PO4 pH 8.0,300 mmol/L NaCl,10 mmol/L Imidazole)悬浮菌体,以超声破碎法裂解菌体,4 ℃、10000 r/min离心10 min,采用Ni-NTA亲和层析法纯化蛋白,以透析法除去咪唑和NaCl,保存于?80 ℃。以12.5% SDS-PAGE检测蛋白表达量及纯度,利用BCA Protein Assay Reagent Kit (Pierce,USA)测定其浓度。
1.7 酶蛋白活性分析 按照文献所述方法依次进行丙氨酸消旋反应和D-氨基酸氧化反应[12-13],反应体系中裂解液为20 μL或适量酶液,磷酸钾缓冲液浓度为50 mmol/L,pH为7.4,反应温度为37 ℃,采用酶标仪(BioTek,USA)测定氧化反应产物的OD550值,根据标准曲线计算反应液中D-丙氨酸的含量,计算裂解液或酶蛋白的比活力。1 min内转化生成1 μmol D-丙氨酸所需的酶量即为一个酶活力单位(unit)。
1.8 酶动力学参数测定 采用HPLC测定丙氨酸消旋酶的动力学参数[14-15]:反应体系含有40 mmol/L磷酸钾缓冲液(pH 7.4),10 μmol/L PLP以及不同浓度的D-或L-丙氨酸,加入酶蛋白于30 ℃下反应10 min,以磷酸盐缓冲液作为阴性对照;反应终止后,以柱前衍生法处理反应液,采用荧光检测器(RF-20A,Shimadzu,Japan)检测反应液中D-或L-丙氨酸的含量,通过非线性回归分析法计算酶蛋白的动力学参数。
1.9 蛋白低聚合状态分析 取1.3所制备的裂解液上清(丙氨酸消旋酶含量为54 mU、1.4 U和12 U),以凝胶过滤层析法分离蛋白,层析柱为HiLoad 16/60 Superdex 200 prep grade (1.6×60 cm,Amersham Biosciences),流动相为含有150 mmol/L NaCl、0.5 mmol/L EDTA、20 μmol/L PLP和0.01% (V/V) 2-mercaptoethanol的磷酸钾缓冲液(20 mmol/L,pH 7.2),流速为1 mL/min,收集体积为1 mL/份,检测各收集样品的相对酶活,计算丙氨酸消旋酶的洗脱体积。
蛋白标准品为Cytochrome C (12.4 kDa),Carbonic anhydrase (29 kDa),Bovine serum albumin (66 kDa),Alcohol dehydrogenase (150 kDa) (Sigma,USA),根据各蛋白在层析柱中洗脱体积绘制标准曲线计算丙氨酸消旋酶StAlr及突变体的分子量。
1.10 序列分析及结构模拟 核酸序列由金唯智生物科技有限公司(苏州)测定,采用Genetyx软件进行序列分析和比对。丙氨酸消旋酶基因alrSt的核苷酸序列的GenBank登录号为WP_001147297.1。采用Swiss-Model在线软件构建野生型蛋白StAlr和突变体A3V的三维结构(https://swissmodel.expasy.org/interactive)[16]。
2 结果和分析 2.1 突变体筛选 经检测,当M9选择培养基中D-丝氨酸浓度 > 5 mmol/L时,含质粒pTrc99A-StAlr的缺陷菌株UT5028能够顺利生长,而当D-丝氨酸≤5 mmol/L时,菌株不能正常生长,因此,以含5 mmol/LD-丝氨酸的M9培养基作为选择培养基。
将易错PCR产物与载体pTrc99A连接后转入缺陷菌株UT5028,经过培养从含有5 mmol/L D-丝氨酸的M9培养基上获得47个克隆子。提取质粒DNA后,通过化学转化法逐一转入感受态细胞UT5028中,过夜培养后,挑取克隆子划线培养于含5 mmol/L D-丝氨酸的M9培养基上,最终获得7个能够弥补菌株UT5028缺失功能的突变体。
将上述7个质粒逐一转入缺陷菌株MB2795中,所获得的重组子均能在不含D-丙氨酸的LB琼脂培养基上顺利生长,说明重组子能够弥补缺陷菌株缺失的功能。DNA测序发现,所有质粒中目的基因的核苷酸序列完全一致,与野生型基因alrSt相比,核苷酸和氨基酸序列同源性分别为99.1%和99.7%,其343位点由酪氨酸突变为组氨酸(Y343H,TAC→CAC),将该突变体质粒命名为pTrc99A-Y343H。
将质粒pTrc99A-Y343H作为模板,进行第二轮易错PCR,PCR产物经过连接、转化感受态细胞及培养后,在含有5 mmol/L D-丝氨酸的M9培养基上获得18个克隆子,提取的质粒DNA后经过菌株UT5028及M9培养基的重复筛选,最终获得12个能够弥补UT5028缺失功能的突变体。
序列检测发现,12个质粒所含的目的基因完全一致,其与野生型alrSt的核苷酸及氨基酸序列同源性分别为99.8%和99.4%,即除了Y343H外,第3位点由丙氨酸突变为缬氨酸(A3V,GCG→GTG) (图 1),该双点突变体命名为pTrc99A-A3VY343H。
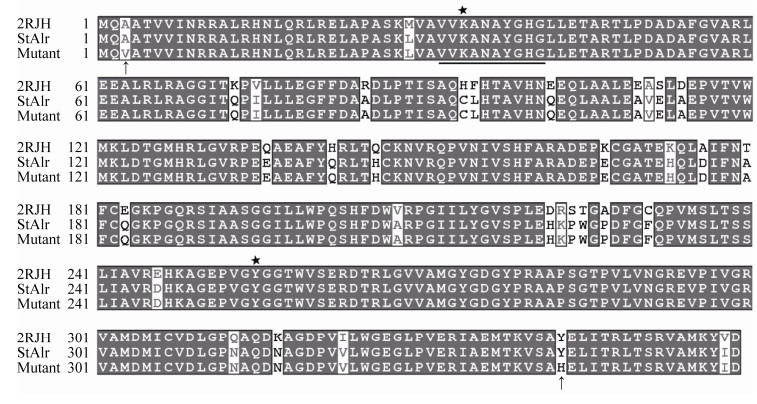 |
| 图 1 丙氨酸消旋酶的氨基酸序列比对图 Figure 1 Amino acid sequence comparison of alanine racemase. Deduced amino acid sequence of mutant was compared to alanine racemase from S. typhimurium (StAlr) and E. coli K12 (2RJH)[17]. Two missense mutations are shown by arrow (↑). Catalytic residues K34 and Y255 are marked with the star (★). |
| 图选项 |
为了明确上述双点突变体中各突变位点的具体作用,采用限制内切酶Sca Ⅰ分别处理载体pTrc99A-StAlr和pTrc99A-A3VY343H,目的片段通过T4 DNA连接酶连接构建获得单点突变体质粒pTrc99A-A3V。
2.2 酶蛋白的活性测定 将质粒pTrc99A-StAlr、pTrc99A-Y343H、pTrc99A-A3V和pTrc99A-A3VY343H分别转入感受态MB2795中,经过诱导、超声破碎、离心获得裂解液上清,根据所测裂解液中的蛋白含量及反应产物含量计算粗酶液的比活力。由表 1可见,以L-丙氨酸为底物,突变体蛋白Y343、A3VY343H和A3V的比活分别约为野生型的1.48、33.00和34.80倍;以L-丝氨酸为底物,突变体Y343H、A3VY343H和A3V的比活性分别约为野生型的1.33、31.24和28.54倍,以上结果可以说明第3位点由丙氨酸(A)突变为缬氨酸(V)是导致酶蛋白比活力大幅提升的关键。
表 1. 野生型及突变体蛋白的比活力 Table 1. Racemization of L-alanine and L-serine by wild type and mutants
| Substrate | StAlr | Y343H | A3VY343H | A3V |
| L-alanine/(units/mg) | 1.59±0.06 | 2.34±0.05 | 52.38±2.12 | 55.39±2.51 |
| Ratio | 1.00 | 1.48±0.02 | 33.00±1.92 | 34.80±0.93 |
| L-serine/(units/mg) | 0.10±0.00 | 0.13±0.01 | 3.11±0.02 | 2.84±0.02 |
| Ratio | 1.00 | 1.33±0.06 | 31.24±0.48 | 28.54±0.49 |
表选项
2.3 丙氨酸消旋酶诱导表达及纯化 将构建的表达载体pET-StAlr、pET-A3V、pET-Y343H和pET-A3VY343H逐一转入感受态细胞BL21(DE3)中,经过预培养、诱导、菌体收集、超声破碎及离心分离获得裂解液上清,采用镍亲和层析法纯化酶蛋白,并经透析脱盐后,获得目的蛋白StAlr、A3V、Y343H和A3VY343H。经SDS-PAGE (12.5%)检测,4个酶蛋白均在40 kDa附近有一条清晰的条带(图 2),与理论分子量39.9 kDa基本一致。
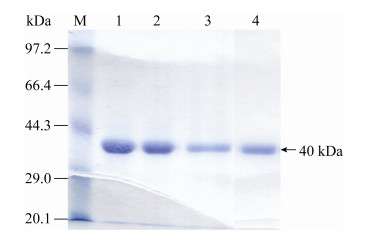 |
| 图 2 丙氨酸消旋酶及其突变体纯化SDS-PAGE检测图 Figure 2 SDS-PAGE identification for purification of alanine racemases and mutants. Lane 1: A3VY343H; lane 2: A3V; lane 3: StAlr; lane 4: Y343H; M: molecular mass standards. |
| 图选项 |
2.4 动力学参数分析 以不同浓度的L/D-丙氨酸和L/D-丝氨酸为底物,通过HPLC检测各蛋白催化反应后对映异构体的含量,从而计算StAlr和突变体蛋白的动力学参数(表 2)。由表 2可见,对于底物L-丙氨酸,突变体Y343H的亲和能力Km和反应常数kcat值均比野生型StAlr提高约10倍,导致其催化效率kcat/Km值仅为StAlr的1.10倍,而对于底物D-丙氨酸,突变体Y343H的kcat/Km值则与StAlr基本相当(0.97倍);但对于底物L-丝氨酸和D-丝氨酸,Y343H的kcat/Km值分别是StAlr的2.01和3.68倍,这些数据充分说明了含有突变质粒pTrc99A-Y343H的UT5028能够在M9基础培养基顺利生长的原因。
表 2. StAlr和突变体的动力学参数 Table 2. Kinetic parameters of proteins StAlr, Y343H, A3VY343H and A3V
| Protein | Substrate | Km (mmol/L) | kcat (s–1) | kcat/Km | Keq (L/D) |
| StAlr | L-Ala | 9.28±0.86 | 7.56±0.90 | 0.82±0.17 | 1.16±0.05 |
| D-Ala | 6.13±0.55 | 4.30±0.35 | 0.70±0.12 | ||
| L-Ser | 59.07±3.53 | 3.48±0.23 | 0.06±0.01 | 2.74±0.26 | |
| D-Ser | 5.62±0.43 | 0.12±0.02 | 0.02±0.00 | ||
| Y343H | L-Ala | 98.14±5.42 | 87.92±6.54 | 0.90±0.12 | 1.31±0.08 |
| D-Ala | 28.60±2.89 | 19.48±0.62 | 0.68±0.05 | ||
| L-Ser | 266.90±16.74 | 31.57±1.81 | 0.12±0.00 | 1.50±0.04 | |
| D-Ser | 129.10±8.24 | 10.23±0.90 | 0.08±0.00 | ||
| A3VY343H | L-Ala | 99.90±4.69 | 407.25±24.07 | 4.08±0.43 | 1.30±0.05 |
| D-Ala | 32.23±2.16 | 101.41±7.73 | 3.15±0.45 | ||
| L-Ser | 156.40±8.66 | 99.28±5.13 | 0.63±0.07 | 1.72±0.05 | |
| D-Ser | 87.45±4.81 | 32.45±2.55 | 0.37±0.05 | ||
| A3V | L-Ala | 0.93±0.08 | 79.72±5.40 | 85.81±1.58 | 1.25±0.01 |
| D-Ala | 0.84±0.06 | 57.59±3.07 | 68.33±1.59 | ||
| L-Ser | 20.70±1.47 | 5.64±0.38 | 0.27±0.00 | 1.18±0.00 | |
| D-Ser | 1.39±0.09 | 0.32±0.02 | 0.23±0.00 |
表选项
比较突变体Y343H、A3VY343H和A3V的动力学参数可以发现,第3位点由丙氨酸(A)突变为缬氨酸(V)是导致酶蛋白催化活性大幅提升的关键。当以L-丙氨酸为底物时,突变体A3V的Km、kcat和kcat/Km值分别是StAlr的0.10、10.54和105.51倍;以D-丙氨酸为底物时,A3V的相关参数分别是StAlr的0.14、13.38和97.36倍;以L-丝氨酸为底物时,A3V的Km、kcat和kcat/Km值分别是StAlr的0.35、1.62和4.63倍;以D-丝氨酸为底物时,其相关参数分别是StAlr的0.25、2.65和10.73倍。这些数据显示突变体A3V对底物L/D-丙氨酸或L/D-丝氨酸的亲和能力、反应常数、催化效率都有较大幅度的提升,说明该位点对于提高酶蛋白的催化活性有着非常重要的作用。
2.5 寡聚化状态和结构模拟分析 为了分析突变位点对酶蛋白催化活性的影响,取含不同酶量的裂解液上清,利用凝胶过滤色谱法分离、收集各馏分,并检测各馏分中酶蛋白的相对活性,如图 3所示,当丙氨酸消旋酶的含量较低时(约54 mU),蛋白StAlr和A3VY343H仅在接近单体状态(monomer,M)处有一个活性峰,而突变体A3V在靠近二聚体(dimer,D)处还有另一个较高的活性峰;当蛋白含量为1.4 U时,蛋白StAlr和A3VY343H仍然只有一个活性峰,但相对于StAlr来说,突变A3VY343H的活性峰逐渐开始向二聚体接近,而A3V在单体附近的活性峰则呈下降趋势,在二聚体附近的活性峰则呈现略微上升态势;当蛋白量继续增加,StAlr (12 U)、A3VY343H (15 U)和A3V (12 U)均呈现单一活性峰状态,其中StAlr和A3VY343H的活性峰呈现向二聚体状态继续偏移的态势,而A3V的活性峰则更为接近二聚体状态(图 3)。Ju等研究发现,丙氨酸消旋酶在单体和二聚体之间存在动态平衡,其低聚合平衡常数与酶蛋白的催化活性之间存在一定的联系,即催化活性低的酶蛋白其低聚合常数值也低,而高催化活性酶蛋白具有较高的聚合常数[11]。由此可以说明,第3位点由丙氨酸突变为缬氨酸(A3V)后促使突变体A3V较其他蛋白更易于形成二聚体状态,从而导致其对底物的亲和性及催化活性均有较大幅度的提升。
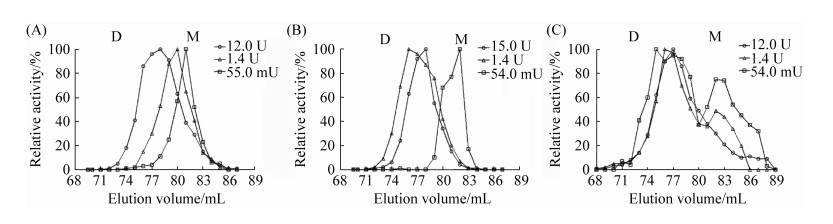 |
| 图 3 细胞裂解液的凝胶过滤色谱图 Figure 3 Gel filtration chromatography analysis of cell lysates. A: E. coli MB2795 containing pTrc99A-StAlr; B: E. coli MB2795 containing prc99A-A3VY343H; C: E. coli MB2795 containing prc99A-A3V. Letters D and M indicated the fractions corresponding to dimeric or monomeric form of protein. |
| 图选项 |
为了进一步分析突变位点的作用机制,以大肠杆菌K12中丙氨酸消旋酶EcAlr (PDB ID:2RJH,与StAlr同源率为91.36%)为模板[17],采用Swiss-Model在线软件构建丙氨酸消旋酶StAlr和突变体A3V的三维结构。由图 4-A可见,2个亚基首尾相连,活性中心K34和Y255′分别来自2个亚基,突变位点A3位于两个亚基的结合界面;当将氨基酸残基A3突变为缬氨酸(V)后,该位点与另一亚基中氨基酸残基E61′和F82′之间的最短距离分别由3.8 ?和4.8 ?缩短至3.3 ?和3.8 ? (图 4-B、C),说明该位点突变后使2个亚基间的距离有所接近,使突变体A3V比野生型更易于形成二聚体,从而提高了酶蛋白的催化活性。
 |
| 图 4 丙氨酸消旋酶StAlr和突变体A3V三维结构模拟图 Figure 4 Molecular structure modeling of StAlr and mutant A3V based on E. coli K12 EcAlr (PDB ID: 2RJH)[17]. A: Overall structure of wild type StAlr in dimer formation. B: Stereo-view of StAlr structure (partial). C: Stereo-view of A3V structure (partial). Dimer of StAlr and A3V were colored in green and cyan in one asymmetric unit. The residue in different color indicates from different subunit. |
| 图选项 |
3 讨论 易错PCR是较早开发的定向进化技术之一,其本质是通过改变PCR条件,增加目标基因扩增的错配率,从而导致其发生突变。易错PCR通常采取如下策略降低其保真性:选用非保真DNA聚合酶、降低退火温度、采取低浓度dNTP或使4种碱基的比例不均衡、提高Mg2+浓度使其超过正常值、加入Mn2+降低聚合酶对模板的特异性、增加反应循环数、采用多轮易错PCR等[18-19]。相对于DNA shuffling或理性进化技术来说,由于易错PCR技术具有操作简便、快速、成本低廉等优点,且无需了解蛋白结构信息和催化机制,使其成为当前运用最广泛的随机突变技术。本实验采取了最为简单的策略:普通的Taq DNA聚合酶、PCR反应循环数提高至50以及两轮易错PCR,将这3种策略组合后成功获得了酶学活性大幅提升的双点突变体A3VY343H。由此可见,即便在当前理性进化技术盛行之际,简单而快速的易错PCR技术在定向进化领域依然具有强大的生命力。
当然,影响定向进化策略效率的因素除了突变技术外,其筛选方法也至为关键。本实验中以营养缺陷菌株UT5028作为筛选菌株,UT5028缺失了serB基因[12, 20],该基因编码为磷酸丝氨酸磷酸酶(phosphoserine phosphatase,PSPH,EC 3.1.3.3),可逆催化丝氨酸磷酸化(O-phospho-L- serine+H2O=L-serine+phosphate),参与蛋白底物脱磷酸化作用[21]。基因serB缺失后,必须添加一定量的L-丝氨酸或转入具有相关功能的基因才能使缺陷菌株UT5028顺利生长于M9基础培养基上。Esaki等研究发现,来自鼠伤寒沙门菌丙氨酸消旋酶StAlr的第二底物为L-丝氨酸,其相对活性约为底物L-丙氨酸的15%[22]。根据这一特性,以缺陷菌株UT5028为宿主,提供一定量的D-丝氨酸,利用丙氨酸消旋酶的消旋反应将D-丝氨酸转化为L-丝氨酸,从而满足缺陷菌株生长的需要。实验前期通过筛选确定了M9基础培养基中添加D-丝氨酸的临界浓度为5 mmol/L,即在D-丝氨酸浓度 > 5 mmol/L时,携带野生型基因alrSt的菌株UT5028能正常生长,而在≤5 mmol/L时,该菌株的生长受到限制。换而言之,在含低浓度D-丝氨酸(≤5 mmol/L)的M9培养基上,凡能使缺陷菌株UT5028顺利生长的皆可能为催化活性有所增长的突变体,这为顺利筛选突变基因奠定了简便而高效的实验基础。至于营养缺陷菌株MB2795,该菌是丙氨酸消旋酶基因alr和dadX双缺失菌株[11],只要向该菌中转入具有丙氨酸消旋酶活性功能的基因后即可正常生长,因此,利用MB2795作为宿主可以发挥两方面的作用:一方面可以利用该菌筛选、剔除失去丙氨酸消旋酶活性的突变体;另一方面则可以避免宿主自身丙氨酸消旋酶对实验的干扰,确保了酶蛋白StAlr和突变体蛋白在寡聚化状态实验结果的可靠性。
目前,有关丙氨酸消旋酶的研究主要集中于酶蛋白抑制剂的筛选和设计方面,关于酶学特性改造方面的文献还比较少,且大多着眼于酶蛋白抑制剂的设计。Patrick等通过定点突变将来自Geobacillus stearothermophilus的丙氨酸消旋酶Y354突变为天冬酰胺(N)后,突变体Y354N对L-丙氨酸的催化效率kcat/Km较野生型蛋白降低了近5倍,但其对L-丝氨酸的催化效率则提高了近62倍,Parick等认为该位点可能在控制底物特异性方面发挥着一定的作用[23]。Wu等采用定点突变技术探究了丙氨酸消旋酶EcAlr中氨基酸位点E165和D164的作用机制,将二者分别突变为丙氨酸(A)或酪氨酸(K)后,突变体蛋白的反应常数kcat值均明显下降[17]。通过分析其三维结构发现,D164和E165分别位于酶蛋白底物通道的中间层和外层,二者与来自另一个亚基的氨基酸位点R280和R300相互协作,构成控制底物进入活性中心的关卡[16]。本研究采用易错PCR技术筛选所得可使酶蛋白催化活性大幅提高的氨基酸位点A3既不是酶蛋白的活性中心,也不属于底物通道的结构组成,而是位于酶蛋白的N-端区域,将该位点突变为缬氨酸(V)后,突变体蛋白的寡聚化状态和同源结构分析结果都显示出其比野生型StAlr更易于形成二聚体形式。这个结论与Ju等提出的“丙氨酸消旋酶催化活性的高低与低聚合状态相关:易于形成低聚合状态的,则其催化活性也较高”的结论相吻合[11]。因此,本文的发现既可以为酶蛋白抑制剂的设计提供思路,即通过抑制蛋白形成二聚体而抑制蛋白活性,也可为改造、提高酶蛋白催化活性,为生物合成D-氨基酸提供所需酶蛋白奠定实验基础。
References
| [1] | Milligan DL, Tran SL, Strych U, Cook GM, Krause KL. The alanine racemase of Mycobacterium smegmatis is essential for growth in the absence of D-alanine. Journal of Bacteriology, 2007, 189(22): 8381-8386. DOI:10.1128/JB.01201-07 |
| [2] | Strych U, Davlieva M, Longtin JP, Murphy EL, Im H, Benedik MJ, Krause KL. Purification and preliminary crystallization of alanine racemase from Streptococcus pneumoniae. BMC Microbiology, 2007, 7(1): 40. |
| [3] | Yoshimura T, Esak N. Amino acid racemases:functions and mechanisms. Journal of Bioscience and Bioengineering, 2003, 96(2): 103-109. DOI:10.1016/S1389-1723(03)90111-3 |
| [4] | Strych U, Benedik MJ. Mutant analysis shows that alanine racemases from Pseudomonas aeruginosa and Escherichia coli are dimeric. Journal of Bacteriology, 2002, 184(15): 4321-4325. DOI:10.1128/JB.184.15.4321-4325.2002 |
| [5] | Galkin A, Kulakova L, Yoshimura T, Soda K, Esaki N. Synthesis of optically active amino acids from alpha-keto acids with Escherichia coli cells expressing heterologous genes. Applied and Environmental Microbiology, 1997, 63(12): 4651-4656. DOI:10.1128/AEM.63.12.4651-4656.1997 |
| [6] | Qu G, Zhao J, Zheng P, Sun JB, Sun ZT. Recent advances in directed evolution. Chinese Journal of Biotechnology, 2018, 34(1): 1-11. (in Chinese) 曲戈, 赵晶, 郑平, 孙际宾, 孙周通. 定向进化技术的最新进展. 生物工程学报, 2018, 34(1): 1-11. |
| [7] | Wu B, Wijma HJ, Song L, Rozeboom HJ, Poloni C, Tian Y, Arif MI, Nuijens T, Quaedflieg PJLM, Szymanski W, Feringa BL, Janssen DB. Versatile peptide C-terminal functionalization via a computationally engineered peptide amidase. ACS Catalysis, 2016, 6(8): 5405-5414. DOI:10.1021/acscatal.6b01062 |
| [8] | Cui YL, Wu B. A brief overview of computational protein structure prediction and enzyme design. Guangxi Sciences, 2017, 24(1): 1-6. (in Chinese) 崔颖璐, 吴边. 计算机辅助蛋白结构预测及酶的计算设计研究进展. 广西科学, 2017, 24(1): 1-6. |
| [9] | Chen Y, Wang SZ, Chen YJ. Rational design of novel enzymes. Pharmaceutical Biotechnology, 2011, 18(6): 538-543. (in Chinese) 陈勇, 王淑珍, 陈依军. 酶的理性设计. 药物生物技术, 2011, 18(6): 538-543. |
| [10] | Tang ZZ, Jin WQ, Sun R, Liao Y, Zhen TR, Chen H, Wu Q, Gou L, Li CL. Improved thermostability and enzyme activity of a recombinant phyA mutant phytase from Aspergillus niger N25 by directed evolution and site-directed mutagenesis. Enzyme and Microbial Technology, 2018, 108: 74-81. DOI:10.1016/j.enzmictec.2017.09.010 |
| [11] | Ju JS, Xu SJ, Furukawa Y, Zhang Y, Misono H, Minamino T, Namba K, Zhao BH, Ohnishi K. Correlation between catalytic activity and monomer-dimer equilibrium of bacterial alanine racemases. The Journal of Biochemistry, 2011, 149(1): 83-89. |
| [12] | Ju J, Misono H, Ohnishi K. Directed evolution of bacterial alanine racemases with higher expression level. Journal of Bioscience and Bioengineering, 2005, 100(3): 246-254. DOI:10.1263/jbb.100.246 |
| [13] | He GZ, Han QQ, Xu SJ, Zhao BH, Ju JS. Function of amino acid sites in the substrate entryway of alanine racemase TtAlr from Thermoanaerobacter tengcongensis. Acta Microbiologica Sinica, 2018, 58(8): 1397-1406. (in Chinese) 何广正, 韩卿卿, 徐书景, 赵宝华, 鞠建松. 腾冲嗜热厌氧菌丙氨酸消旋酶底物通道氨基酸位点的功能. 微生物学报, 2018, 58(8): 1397-1406. |
| [14] | Hashimoto A, Nishikawa T, Oka T, Takahashi K, Hayashi T. Determination of free amino acid enantiomers in rat brain and serum by high-performance liquid chromatography after derivatization with N-tert.-butyloxycarbonyl-L-cysteine and o-phthaldialdehyde. Journal of Chromatography B:Biomedical Sciences and Applications, 1992, 582(1/2): 41-48. |
| [15] | Israr M, Lv GP, Xu SJ, Li YH, Ding ST, Zhao BH, Ju JS. Biochemical characterization and mutational analysis of alanine racemase from Clostridium perfringens. Journal of Bioscience and Bioengineering, 2019, 128(2): 149-155. DOI:10.1016/j.jbiosc.2019.02.003 |
| [16] | Biasini M, Bienert S, Waterhouse A, Arnold K, Studer G, Schmidt T, Kiefer F, Cassarino TG, Bertoni M, Bordoli L, Schwede T. SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information. Nucleic Acids Research, 2014, 42(W1): W252-W258. DOI:10.1093/nar/gku340 |
| [17] | Wu DL, Hu TC, Zhang L, Chen J, Du JM, Ding JP, Jiang HL, Shen X. Residues Asp164 and Glu165 at the substrate entryway function potently in substrate orientation of alanine racemase from E. coli:enzymatic characterization with crystal structure analysis. Protein Science, 2008, 17(6): 1066-1076. DOI:10.1110/ps.083495908 |
| [18] | Biles BD, Connolly BA. Low-fidelity Pyrococcus furiosus DNA polymerase mutants useful in error-prone PCR. Nucleic Acids Research, 2004, 32(22): e176. DOI:10.1093/nar/gnh174 |
| [19] | Li S, Zhang W, Zhao CM. Directed evolution of alginate lyase Alg-2 based on error prone PCR. Food Science, 2019, 40(4): 146-151. (in Chinese) 李树, 张伟, 赵春梅. 易错PCR技术定向进化褐藻胶裂解酶Alg-2的分析. 食品科学, 2019, 40(4): 146-151. |
| [20] | Andrews JC, Short SA. Genetic analysis of Escherichia coli oligopeptide transport mutants. Journal of Bacteriology, 1985, 161(2): 484-492. DOI:10.1128/JB.161.2.484-492.1985 |
| [21] | Kim K, Chiba Y, Kobayashi A, Arai H, Ishii M. Phosphoserine phosphatase is required for serine and one-carbon unit synthesis in Hydrogenobacter thermophilus. Journal of Bacteriology, 2017, 199(21): e00409-17. DOI:10.1128/JB.00409-17 |
| [22] | Esaki N, Walsh CT. Biosynthetic alanine racemase of Salmonella typhimurium:purification and characterization of the enzyme encoded by the alr gene. Biochemistry, 1986, 25(11): 3261-3267. DOI:10.1021/bi00359a027 |
| [23] | Patrick WM, Weisner J, Blackburn JM. Site-directed mutagenesis of Tyr354 in Geobacillus stearothermophilus alanine racemase identifies a role in controlling substrate specificity and a possible role in the evolution of antibiotic resistance. Chem Bio Chem, 2002, 3(8): 789-792. DOI:10.1002/1439-7633(20020802)3:8<789::AID-CBIC789>3.0.CO;2-D |
