王超1, 崔艳华1

 , 曲晓军2
, 曲晓军2 1. 哈尔滨工业大学化工与化学学院, 黑龙江 哈尔滨 150090;
2. 黑龙江省科学院微生物研究所, 黑龙江 哈尔滨 150010
收稿日期:2020-02-11;修回日期:2020-05-19;网络出版日期:2020-07-02
基金项目:国家自然科学基金(31371827,31471712)
*通信作者:崔艳华, Tel:+86-451-86282902;Fax:+86-451-86282906;E-mail:yhcui@hit.edu.cn.
摘要:[目的] 在乳酸乳球菌NZ9000中异源表达德氏乳杆菌保加利亚亚种中由双组分系统TCS1(JN675228/JN675229)调控的与酸适应相关基因,进而探究德氏乳杆菌保加利亚亚种应对酸胁迫的机制。[方法] 通过逆转录聚合酶链式反应和十二烷基硫酸钠-聚丙烯酰胺凝胶电泳验证由德氏乳杆菌保加利亚亚种TCS1调控的与酸适应相关基因中腺嘌呤磷酸核糖转移酶(aprt)、D-丙氨酸-D-丙氨酸连接酶(ddl)、寡肽ABC转运蛋白(oppDII)和延伸因子Ts(tsf)在乳酸乳球菌NZ9000中的表达情况。酸处理实验验证基因表达对宿主菌酸胁迫耐受能力的影响。并采用酵母双杂交验证双组分系统TCS1与表达的酸适应相关基因之间的互作关系及具体的互作部位。[结果] 结果表明,乳酸乳球菌NZ9000中成功表达了aprt、ddl、oppDII和tsf。aprt、ddl基因使重组菌对酸胁迫的抗性分别提高了75倍和114倍。oppDII和tsf基因的表达对重组菌株的耐酸能力没有明显影响。酵母双杂交实验表明TCS1中的组氨酸蛋白激酶HPK1与Ddl之间存在相互作用,且HPK1-C结构域是二者相互作用的关键区域。[结论] aprt和ddl过表达菌株酸刺激的适应能力显著高于对照菌株,该研究结果可为德氏乳杆菌保加利亚亚种及类似菌株耐酸性特性的获得策略提供参考。
关键词:德氏乳杆菌保加利亚亚种腺嘌呤磷酸核糖转移酶D-丙氨酸-D-丙氨酸连接酶耐酸性酵母双杂交
Expression of genes regulated by acid adaptation related TCS1 of Lactobacillus delbrueckii subsp. bulgaricus improved acid tolerance of Lactococcus lactis subsp. cremoris NZ9000
Chao Wang1, Yanhua Cui1

 , Xiaojun Qu2
, Xiaojun Qu2 1. School of Chemistry and Chemical Engineering, Harbin Institute of Technology, Harbin 150090, Heilongjiang Province, China;
2. Institute of Microbiology, Heilongjiang Academy of Sciences, Harbin 150010, Heilongjiang Province, China
Received: 11 February 2020; Revised: 19 May 2020; Published online: 2 July 2020
*Corresponding author: Yanhua Cui, Tel:+86-451-86282902;Fax:+86-451-86282906;E-mail:yhcui@hit.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31371827, 31471712)
Abstract: [Objective] We investigated the acid tolerance mechanisms of Lactobacillus delbrueckii subsp. bulgaricus by heterologous expression of genes related to acid adaptation regulated by two-component system TCS1 (JN675228/JN675229) of Lb. bulgaricus in Lactococcus lactis subsp. cremoris NZ9000. [Methods] We used reverse transcription polymerase chain reaction and sodium dodecyl sulfate-polyacrylamide gel electrophoresis to verify the expression of acid adaptation related genes, such as adenine phosphoribosyltransferase (aprt), D-alanine-D-alanine ligase (ddl), oligopeptide ABC transporter (oppDII) and elongation factor Ts (tsf), regulated by TCS1 of Lb. bulgaricus, in Lc. lactis NZ9000. We validated acid treatment experiment to verify the effect of gene expression on acid stress tolerance of host bacteria. We confirmed the interactions between TCS1 and the expressed genes related to acid adaptation by yeast two hybrid. [Results] Our results show that aprt, ddl, oppDII and tsf were successfully expressed in Lc. lactis NZ9000. The expression of aprt and ddl genes increased the resistance of recombinant strains to acid stress by 75 and 114 times. The expression of oppDII and tsf genes had no significant effect on the acid resistance of the recombinant strains. Yeast two hybrid system showed that there was interaction between histidine protein kinase (HPK1) of TCS1 and Ddl, and HPK1-C domain was the key region of the interaction. [Conclusion] The acid tolerance of strains expressing Aprt and Ddl was significantly higher than that of the control strain. The results of this study can provide references for the strategies of obtaining acid resistance of Lb. bulgaricus and other similar strains.
Keywords: Lactobacillus delbrueckii subsp. bulgaricusadenine phosphoribosyltransferaseD-alanine-D-alanine ligaseacid toleranceyeast two-hybrid
德氏乳杆菌保加利亚亚种(Lactobacillus delbrueckii subsp. bulgaricus,Lb. bulgaricus)广泛应用于食品和医药行业[1]。Lb. bulgaricus是一种乳酸菌,在其自身生长和应用过程中经常遇到各种酸性环境[2]。低pH环境产生酸胁迫,对细胞生理产生不利影响,包括膜损伤、抑制酶活性和运输系统等[3]。因此,保持对酸胁迫的耐受性对于该类菌株的生存和工业应用至关重要。
乳酸菌(lactic acid bacteria,LAB)中发现了多种耐酸机制,包括中和过程、质子泵、高分子保护、预适应和交叉保护作用等[4]。然而,通过对Lb. bulgaricus基因组序列的生物信息学分析表明,参与菌株调节pH稳定性的基因相对较少,探索菌株通过何种调节机制来维持自身在酸性环境的耐受性显得尤为重要[5]。
细菌中参与基因调节的双组分系统(two-component system,TCS)由组氨酸蛋白激酶(histidine protein kinase,HPK)和应答调节蛋白(response regulator protein,RR)组成[6]。本课题组前期研究表明,TCS1调节了由于突然暴露在酸性环境引起的酸耐受反应(acid tolerance reaction,ATR),参与了菌株的耐酸性和生长性能的调节[7]。分别构建了HPK1突变体CH3-H和RR1突变体CH3-R,并通过2-DE和RT-qPCR鉴定出了一些与耐酸相关的差异表达基因[8]。其中,腺嘌呤磷酸核糖转移酶(adenine phosphoribosyltransferase,Aprt)在嘌呤核苷酸补救合成途径中将激活的5-磷酸核糖-1-焦磷酸(5-phosphoribosyl 1-pyrophosphate,PRPP)添加到腺嘌呤上产生单磷酸腺苷(adenosine monophosphate,AMP),除了影响嘌呤核苷酸代谢外,还可能对能量代谢产生影响;D-丙氨酸-D-丙氨酸连接酶(D-alanine-D-alanine ligase,Ddl)参与了D-丙氨酸代谢和细胞壁中肽聚糖的生物合成过程,对细胞壁合成产生影响;寡肽ABC转运蛋白(oligopeptide ABC transporter,OppDII)影响代谢分泌物质的吸收过程;延伸因子Ts (elongation factor Ts,Tsf)有助于在蛋白质合成的延伸阶段将各种氨基酸加入到延伸的多肽链中,加快胞内蛋白的合成速率。
德氏乳杆菌保加利亚亚种缺乏有效的电转化策略和用于基因突变的分子手段,严重制约了对其基因功能的直接评价[9]。因此,本研究将Aprt、Ddl、OppDII和Tsf在乳酸乳球菌NZ9000中进行异源表达,探讨这4个关键差异基因的表达对宿主耐酸性的影响,并探讨TCS1调控的这4个关键差异基因在菌株ATR过程中的作用。翟远征发现,构建Lb. bulgaricus CAUH1电转化平台存在技术难点,采用Lc. lactis NZ9000表达可能参与Lb. bulgaricus CAUH1耐酸反应的丙酮酸激酶(pyruvate kinase,Pyk)和假定的转录因子(hypothetical transcription factors,Ldb0677),发现宿主菌的耐酸能力得到了明显提高[9]。类似地,干酪乳杆菌中的DNA修复蛋白recO (DNA repair protein,recO)基因在Lc. lactis NZ9000中表达,使得重组菌株在低pH环境下的耐酸性显著改善[10]。
本研究通过克隆关键耐酸调控相关基因aprt、ddl、oppDII和tsf,并在Lc. lactis NZ9000中进行异源表达,进一步确定这些差异表达基因是否参与了菌株的耐酸调控过程,并对这些基因的功能进行了验证和评价。利用酵母双杂交技术(yeast two hybrid,Y2H)研究了双组分系统中HPK1、RR1与耐酸调控蛋白Aprt、Ddl之间潜在的物理相互作用。本研究结果为aprt和ddl基因在酸适应中的功能研究及其在提高菌株耐酸性中的应用提供了依据。
1 材料和方法 1.1 菌株、质粒、引物、培养条件 本研究所用菌株、质粒和引物见表 1和表 2。本实验中所用的菌株为大肠杆菌DH5α、Lb. bulgaricus CH3、Lc. lactis NZ9000和酿酒酵母Y2HGold。实验中用到的质粒有大肠杆菌-乳酸乳球菌穿梭载体pMG36c[7]以及pGBKT7 DNA-BD、pGADT7 AD、pGBKT7-53、pGBKT7-Lam和pGADT7-T等用于酵母双杂交实验的质粒。实验中所用的引物用于扩增aprt、ddl、oppDII和tsf基因全长构建表达载体以及用于构建全长酵母双杂交载体和截短型酵母双杂交载体等(表 2)。
表 1. 本研究中使用的菌株和质粒 Table 1. Strains and plasmids used in this study
| Strains or plasmids | Characteristics | Sources |
| Strains | ||
| ??Lb. bulgaricus CH3 | Isolation and identification from traditional fermented milk in Chinese pastoral areas | Preserved in our laboratory |
| ??Lc. lactis NZ9000 | Host bacteria of the expression system of Lc. lactis | Preserved in our laboratory |
| ??Lc. lactis NZCK | Lc. lactis NZ9000 carrying pMG36c plasmid | This study |
| ??Lc. lactis NZAprt | Lc. lactis NZ9000 carrying pMG-aprt plasmid | This study |
| ??Lc. lactis NZDdl | Lc. lactis NZ9000 carrying pMG-ddl plasmid | This study |
| ??Lc. lactis NZOppDII | Lc. lactis NZ9000 carrying pMG-oppDII plasmid | This study |
| ??Lc. lactis NZTsf | Lc. lactis NZ9000 carrying pMG-tsf plasmid | This study |
| ??E. coli DH5α | Plasmid cloning host | Preserved in our laboratory |
| Y2HGold | Yeast two-hybrid expression strain | TaKaRa BIO INC. |
| Plasmids | ||
| ??pMG36c | Chloramphenicol resistance, expression system carrier | Preserved in our laboratory |
| ??pMG-aprt | pMG36c plasmid carrying aprt gene | This study |
| ??pMG-ddl | pMG36c plasmid carrying ddl gene | This study |
| ??pMG-oppDII | pMG36c plasmid carrying oppDII gene | This study |
| ??pMG-tsf | pMG36c plasmid carrying tsf gene | This study |
| ??pGBKT7 DNA-BD | Kanamycin resistance, bait plasmid for Y2H, BD | TaKaRa BIO INC. |
| ??pGADT7 AD | Ampicillin resistance, prey plasmid for Y2H, AD | TaKaRa BIO INC. |
| ??pGBKT7-53 | Recombinant plasmid as positive control for Y2H, BD-53 | TaKaRa BIO INC. |
| ??pGBKT7-Lam | Recombinant plasmid as negative control for Y2H, BD-Lam | TaKaRa BIO INC. |
| ??pGADT7-T | Recombinant plasmid as control for Y2H, AD-T | TaKaRa BIO INC. |
| ??pGBKT7-aprt | pGBKT7 carrying aprt gene, BD-APRT | This work |
| ??pGBKT7-ddl | pGBKT7 carrying ddl gene, BD-Ddl | This work |
| ??pGADT7-hpk1 | pGADT7 carrying hpk1 gene, AD-HPK1 | This work |
| ??pGADT7-rr1 | pGADT7 carrying rr1 gene, AD-RR1 | This work |
| ??pGADT7-A | pGADT7 carrying the A domain of hpk1 gene, AD-A | This work |
| ??pGADT7-B | pGADT7 carrying the B domain of hpk1 gene, AD-B | This work |
| ??pGADT7-C | pGADT7 carrying the C domain of hpk1 gene, AD-C | This work |
表选项
表 2. 本研究中使用的引物 Table 2. Primers used in this study
| Primers | Sequences (5′→3′) | Restriction sites | Targets |
| Aprt-F | TTCGAGCTCGCATGGAGGATTACAAAATGTCAATTG | Sac I | Full length of aprt for expression |
| Aprt-R | GACTCTAGATTAAGCGCCGGTGTATTGAAC | Xba I | |
| Ddl-F | TTCGAGCTCGCATGACGAAAAAAACACAAGTTGG | Sac I | Full length of ddl for expression |
| Ddl-R | GACTCTAGATTAGTCGTGCTTGTGCAGGATG | Xba I | |
| oppDII-F | TTCGAGCTCGCATGGCAAATAGAATTTTAGACGTTAAAAAC | Sac I | Full length of oppDII for expression |
| oppDII-R | GACTCTAGATTAATCCTCGTCAGCTGCTTG | Xba I | |
| Tsf-F | TTCGAGCTCGCATGGCAAACATTACTGCTAAGC | Sac I | Full length of tsf for expression |
| Tsf-R | GACTCTAGATTAGTTCTTCATTTGTGCAGCAAC | Xba I | |
| HPK1-EF | CCGGAATTCATGATCAACAGCCTGTTCAA | EcoR I | Full length of hpk1 for Y2H |
| HPK1-BR | CGCGGATCCCTATCCCTTCTGAATAACTA | BamH I | |
| RR1-BF | CGCGGATCCATGCTAGCCATCATCATTTT | BamH I | Full length of rr1 for Y2H |
| RR1-PR | GCCCTGCAGTTAAACAAGGTCATTTTTTG | Pst I | |
| RR1-XR | CCGCTCGAGTTAAACAAGGTCATTTTTTG | Xho I | |
| Aprt-EF | CCGGAATTCATGGAGGATTACAAAATGTC | EcoR I | Full length of aprt for Y2H |
| Aprt-BR | CGGATCCTTAAGCGCCGGTGTATTGAA | BamH I | |
| Ddl-BF | CGCGGATCCATGACGAAAAAAACACA | BamH I | Full length of ddl for Y2H |
| Ddl-XR | CCTCGAGTTAGTCGTGCTTGTGCAGGA | Xho I | |
| Ddl-SR | GCGTCGACTTAGTCGTGCTTGTGCAGGA | Sal I | |
| A-EF | CCGGAATTCCTGTTCAACAATCTAGCTTT | EcoR I | Truncated hpk 1 (hpk1-A) for Y2H |
| A-BR | CGGATCCTTATTCCTGGTTTTTTTGAATCG | BamH I | |
| B-EF | CCGGAATTCATTATGGCCACACGGATTCA | EcoR I | Truncated hpk 1 (hpk1-B) for Y2H |
| B-BR | CGCGGATCCTTAGTTGATCTCAACCTTGGTCT | BamH I | |
| C-EF | GGAATTCAAGCCAATTGCTACTCTGCC | EcoR I | Truncated hpk 1 (hpk1-C) for Y2H |
| C-BR | CGCGGATCCTTACTTCTGAATAACTAATTCAA | BamH I |
表选项
用于培养大肠杆菌DH5α的培养基是LB培养基。用于培养Lb. bulgaricusCH3的培养基是MRS培养基。Lc. lactis NZ9000在GM17培养基(添加0.5%葡萄糖的M17培养基)中进行培养。酵母菌株在YPDA培养基或SD培养基(Synthetic Drop-out Medium,SD培养基)中培养。
Lb. bulgaricus CH3在37 ℃静置培养。Lc. lactis NZ9000在30 ℃厌氧培养。GM17培养基和LB培养基中分别加入5 μg/mL和10 μg/mL氯霉素,筛选pMG36c阳性克隆。将Y2H重组质粒转化大肠杆菌DH5α,用含氨苄青霉素(100 μg/mL)或卡那霉素(50 μg/mL)的LB平板筛选阳性克隆。YPDA培养基、SD培养基购自TaKaRa BIO INC.公司。氯霉素、氨苄青霉素、卡那霉素、葡萄糖购自美国Sigma-Aldrich公司。配制LB培养基、M17培养基所用到的各种药品购自天津市百世化工有限公司。
1.2 重组菌株的构建 根据细菌DNA提取试剂盒(OMEGA bio tek,美国)说明书提取Lb. bulgaricus CH3基因组DNA。根据Lb. bulgaricus ATCC 11842全基因组(GenBank登录号CR954253.1)中aprt、ddl、oppDII和tsf基因序列设计引物[5]。以基因组DNA为模板,在上游引物加入SacⅠ位点,下游引物加入XbaⅠ位点,用PCR方法扩增aprt、ddl、oppDII和tsf基因片段。纯化后的PCR产物分别与Sac I-Xba I酶切后的pMG36c连接,获得重组质粒pMG-aprt、pMG-ddl、pMG-oppDII和pMG-tsf。将重组质粒pMG-aprt、pMG-ddl、pMG-oppDII和
pMG-tsf分别在2000 V、200 Ω和25 μF下电转化到Lc. lactis NZ9000感受态细胞中,构建重组菌株Lc. lactis NZAprt、Lc. lactis NZDdl、Lc. lactis NZOppDII和Lc. lactis NZTsf。将pMG36c转化到Lc. lactis NZ9000构建Lc. lactis NZCK对照菌株。
1.3 RT-PCR检测重组蛋白表达 采用逆转录PCR(reverse transcription PCR,RT-PCR)在转录水平确认差异基因的表达情况[11]。重组菌株Lc. lactis NZAprt、Lc. lactis NZDdl、Lc. lactis NZOppDII和Lc. lactis NZTsf以及对照菌株Lc. lactis NZCK在含有5 μg/mL氯霉素的GM17中,30 ℃下培养直到OD600=0.8。采用Trizol试剂提取细菌总RNA,去除基因组DNA[7]。采用TaKaRa BIO INC公司的PrimeScriptTMII第一链cDNA合成试剂盒合成cDNA,然后以cDNA为模板,用相应的引物分别扩增aprt、ddl、oppDII和tsf基因。
1.4 SDS-PAGE检测蛋白表达 菌株的培养方法与RT-PCR检测基因表达实验相同。细胞在4 ℃下离心(8000 r/min) 5 min,用预冷的PBS缓冲液洗涤2次,然后重新悬浮,冰上超声破碎30 min (75 W,工作10 s,间隔10 s)。4 ℃下离心(12000 r/min) 5 min去除细胞碎片,获得细胞提取液。将细胞提取液与2× SDS-PAGE上样缓冲液混合,煮沸5 min,用5% (W/V)浓缩胶和12% (W/V)分离胶对每个样品进行电泳,并用考马斯亮蓝(Coomassie brilliant blue,CBB) R-250染色。
1.5 菌株生长能力 将重组菌株Lc. lactis NZAprt、Lc. lactis NZDdl、Lc. lactis NZOppDII、Lc. lactis NZTsf和对照菌株Lc. lactis NZCK接种(2%,V/V)在GM17培养基中,并在30 ℃下培养。使用紫外分光光度计(UV-2100,Unicol Shanghai Co. LTD.)在600 nm处每隔2 h监测菌株的OD600,采用精密酸度计[PB-10,赛多利斯科学仪器(北京)有限公司]测定胞外pH (extracellular pH,pHex)。
1.6 菌株耐胁迫能力分析 所有菌株在30 ℃下培养于添加5 μg/mL氯霉素的GM17中。在OD600=0.8时收集细胞,将细胞平均分成2份,用PBS洗涤,一份重悬在含有5 μg/mL氯霉素的等体积GM17液体培养基(pH 2.5)中酸处理1 h,一份不做任何处理作为对照。将处理前后的细胞涂布于GM17平板上,30 ℃培养24–48 h后,对酸处理前后的细胞进行计数,存活率等于酸处理前后活细胞数的比值。在鉴定了异源表达基因对菌株耐酸性的影响后,评价了这些基因对菌株耐其他胁迫能力的影响,包括胆盐胁迫(1.25%牛胆汁,20 min)、热胁迫(50 ℃,30 min)、冷胁迫(10 ℃,12 h)和氧化胁迫(15 mmol/L H2O2,30 min)[12]。菌株耐其他胁迫实验中,菌株培养方法与耐酸性实验相同,仅处理方法按照此条件进行即可。
1.7 Y2H质粒构建 为确定双组分系统TCS1中的组氨酸蛋白激酶(HPK1)、应答调节蛋白(RR1)与耐酸调控差异蛋白Aprt、Ddl的相互作用及作用关键区域,进行了全长和截短型酵母双杂交试验。pGADT7(AD)、pGBKT7(BD)和其他对照质粒从TaKaRa BIO INC.购买。表 1所列用于Y2H分析的重组质粒是通过双酶切将需插入的DNA连接到穿梭载体上构建而成。用于构建融合蛋白的全长或截短部分是通过使用表 2中相应的引物从Lb. bulgaricus CH3的基因组DNA中进行PCR扩增获得。
1.8 酵母转化与斑点检测 利用醋酸锂(lithium acetate,LiAc)介导的方法,将编码杂交蛋白的AD和BD重组质粒共转化到Y2HGold菌株感受态细胞中[13]。在SD/-Trp-Leu培养基(SD培养基中缺少色氨酸、亮氨酸)上30 ℃孵育3 d后,选择带有诱饵质粒和猎物质粒的转化子,将适量的菌落转移到无菌水中,调节OD600约为1.0。将5 μL不同梯度稀释液(1:10、1:100和1:1000)用移液枪点到SD/-Trp-Leu-His-Ade选择性培养基(SD培养基中缺少色氨酸、亮氨酸、组氨酸和腺嘌呤)上,30 ℃静置培养2 d。
2 结果和分析 2.1 重组菌株验证 为了在Lc. lactis NZ9000中表达Lb. bulgaricus CH3中与菌株耐酸能力相关的Aprt、Ddl、OppDII和Tsf蛋白,分别扩增了aprt、ddl、oppDII和tsf基因片段,片段大小分别约为543 bp、1089 bp、1044 bp和1029 bp,分别连接到表达载体pMG36c上(图 1),经菌落PCR、双酶切鉴定和测序验证的阳性转化子分别命名为pMG-aprt、pMG-ddl、pMG-oppDII和pMG-tsf (数据未显示)。
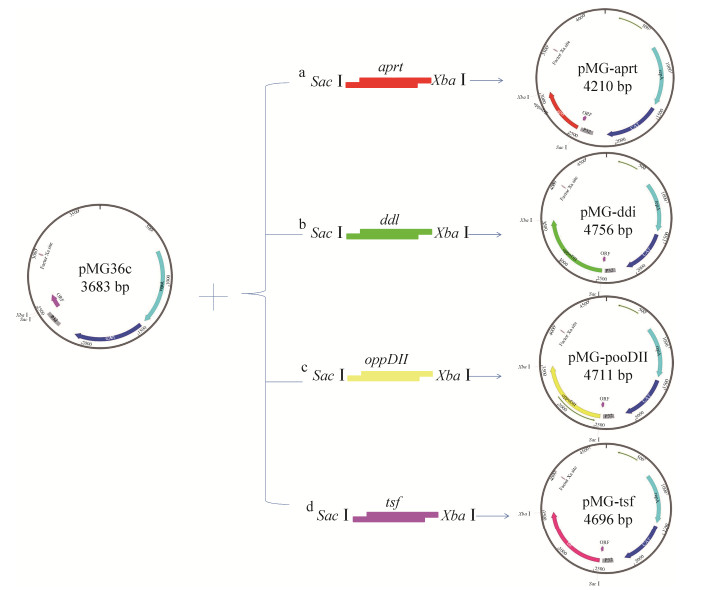 |
| 图 1 表达质粒的构建 Figure 1 Construction of the expression plasmids. a: pMG-aprt; b: pMG-ddl; c: pMG-oppDII; d: pMG-tsf. |
| 图选项 |
为研究aprt、ddl、oppDII和tsf在耐酸调节中的作用,将含有aprt、ddl、oppDII和tsf的质粒分别转化到Lc. lactis NZ9000感受态细胞中。重组质粒pMG-aprt、pMG-ddl、pMG-oppDII和pMG-tsf及对照质粒pMG36c经电穿孔分别转化到Lc. lactis NZ9000中,通过质粒提取并进行双酶切验证(图 2)。
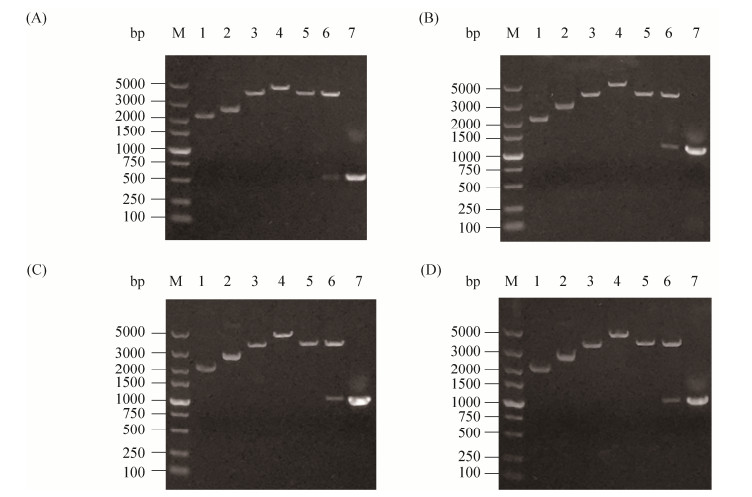 |
| 图 2 重组质粒酶切验证电泳图 Figure 2 Agarose electrophoresis of the recombinant plasmids verified by single and double enzyme digestion. M: DNA marker DL5000; 1: pMG36c; 2: recombinant plasmids; 3, 5: single enzyme digestion of pMG36c; 4, 6: single and double enzyme digestion of recombinant plasmids; 7: exogenous PCR products. A: pMG-aprt; B: pMG-ddl; C: pMG-oppDII; D: pMG-tsf. |
| 图选项 |
从图 2中可以看出,各个重组质粒条带大小均大于pMG36c空载体,出现不同程度的滞后现象。经Xba I单酶切后各个重组质粒条带大小明显大于酶切后线性的pMG36c空载体,条带滞后现象更加明显;经Sac I和Xba I双酶切后,有与酶切后线性的pMG36c空载体大小相同的条带以及外源连接的耐酸调控差异基因片段PCR产物大小一致的条带产生。说明重组质粒pMG-aprt、pMG-ddl、pMG-oppDII和pMG-tsf及对照质粒pMG36c经电穿孔成功转化到Lc. lactis NZ9000中,分别命名为乳酸乳球菌NZAprt、NZDdl、NZOppDII、NZTsf和NZCK。
2.2 RT-PCR和SDS-PAGE检测重组菌株表达情况 为了检测Lb. bulgaricus CH3中由双组分系统TCS1调控的与菌株耐酸能力相关差异蛋白Aprt、Ddl、OppDII和Tsf在Lc. lactis NZ9000中的表达情况,采用RT-PCR技术检测了这些基因在转录水平上的表达情况。以乳酸乳球菌NZAprt、NZDdl、NZOppDII、NZTsf和NZCK提取的总RNA反转录合成的单链cDNA为模板,用相应的引物进行RT-PCR,分别扩增aprt、ddl、oppDII和tsf基因,结果如图 3所示。
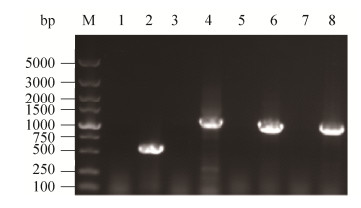 |
| 图 3 耐酸调控相关基因RT-PCR产物的电泳图 Figure 3 Agarose electrophoresis of RT-PCR product of acid-related genes. M: DNA marker DL5000; lane 1, 3, 5, 7: Negative control (NZCK); lane 2: aprt (NZAprt); lane 4: ddl(NZDdl); lane 6: oppDII(NZOppDII); lane 8: tsf (NZTsf). |
| 图选项 |
乳酸乳球菌NZ9000中也存在双组分信号转导系统TCSs,但经过将德氏乳杆菌保加利亚亚种中的TCS1与乳酸乳球菌NZ9000中的双组分信号转导系统TCSs进行比对发现同源性很低。且通过PCR扩增发现(图 3),乳酸乳球菌NZ9000中不含有本实验中异源表达的德氏乳杆菌保加利亚亚种的aprt、ddl、oppDII和tsf基因。因此,理论上乳酸乳球菌NZ9000中的双组分信号转导系统TCSs对异源表达的德氏乳杆菌保加利亚亚种这4个基因不存在调控作用。
从图 3中可以看出,Lb. bulgaricus CH3中由TCS1调控的耐酸性相关基因aprt、ddl、oppDII和tsf均可以从Lc. lactis NZ9000中扩增得到。在重组菌株中观察到相应条带,而在对照菌株NZCK中没有观察到条带。结果表明,与耐酸相关基因aprt、ddl、oppDII和tsf均在Lc. lactis NZ9000中成功表达。
采用SDS-PAGE检测乳酸乳球菌NZAprt、NZDdl、NZOppDII、NZTsf和NZCK的总蛋白表达情况。结果表明,Lb. bulgaricus CH3中的aprt、ddl、oppDII和tsf基因成功在Lc. lactis NZ9000中异源表达,但重组菌株表达重组蛋白后与对照菌株在总蛋白表达水平上无显著差异(图 4)。
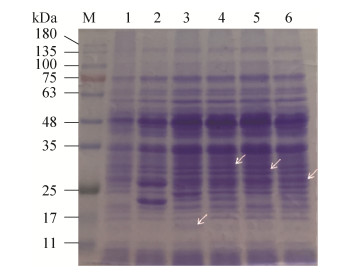 |
| 图 4 SDS-PAGE检测酸胁迫差异基因在Lc. lactis NZ9000中的表达情况 Figure 4 The expression of acid-related genes in Lc. lactis NZ9000 determined by SDS-PAGE. 1: NZ9000; 2: NZCK; 3: NZAprt; 4: NZDdl; 5: NZOppDII; 6: NZTsf. |
| 图选项 |
2.3 重组乳酸乳球菌NZ9000生长性能研究 在相同条件下,对乳酸乳球菌NZCK和重组乳酸乳球菌NZAprt、NZDdl、NZOppDII和NZTsf的生长性能进行了研究。重组菌株乳酸乳球菌NZAprt、NZDdl、NZOppDII和NZTsf的生长曲线与对照菌株乳酸乳球菌NZCK几乎没有差异(图 5-A)。所有菌株的延滞期大概持续2 h,2–10 h菌株处于指数生长期,之后所有菌株开始进入稳定期。在正常生长条件下检测了携带aprt、ddl、oppDII和tsf的乳酸乳球菌的胞外pH (图 5-B)。虽然重组菌株的OD600略有增加,胞外pH略有下降,但综合来看,表达外源蛋白对菌株生长和胞外pH没有显著差异,表明异源表达aprt、ddl、oppDII和tsf基因对细胞生长和细胞外pH没有显著影响,研究人员对其他基因的异源表达也得到了相似的结果[14]。
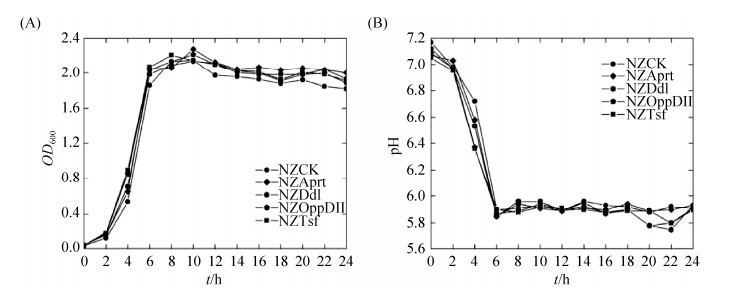 |
| 图 5 正常条件下NZCK、NZAprt、NZDdl、NZOppDII和NZTsf的生长曲线(A)和pHex(B) Figure 5 The growth curves (A) and pHex (B) of NZCK, NZAprt, NZDdl, NZOppDII and NZTsf under normal condition. |
| 图选项 |
2.4 重组乳酸乳球菌NZ9000耐酸性评价 为了阐明Aprt、Ddl、OppDII和Tsf的表达是否影响了Lc. lactis NZ9000的耐酸性,对乳酸乳球菌NZAprt、NZDdl、NZOppDII、NZTsf和NZCK在pH 2.5的GM17液体培养基中30 ℃静置培养1 h前后的活细胞进行了计数(图 6)。对照菌株乳酸乳球菌NZCK与重组菌株乳酸乳球菌NZAprt、NZDdl、NZOppDII和NZTsf在酸胁迫下的存活率有着明显的差异。对照菌株乳酸乳球菌NZCK和重组菌株乳酸乳球菌NZAprt、NZDdl、NZOppDII和NZTsf的存活率分别为5.0×10–6、3.8×10–4、5.7×10–4、3.2×10–6和5.4×10–6。
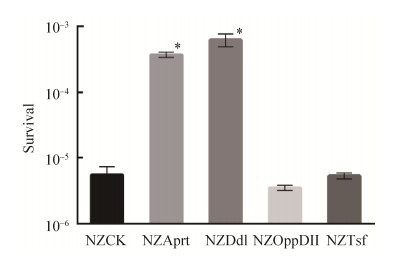 |
| 图 6 酸刺激后重组乳酸乳球菌NZ9000的存活情况 Figure 6 The survival of Lc. lactis NZ9000 derivatives after acid challenge. The survival rate was calculated equal to the number of living cells before and after the acid treatment. *: P < 0.05. |
| 图选项 |
结果表明,乳酸乳球菌NZAprt菌株的存活率是对照菌株NZCK的75倍,乳酸乳球菌NZDdl菌株的存活率是对照菌株NZCK的114倍。与对照菌株相比,乳酸乳球菌NZOppDII和NZTsf菌株的耐酸性没有显著提高。这些数据表明,NZAprt和NZDdl菌株在应对酸性环境时赋予了宿主菌更高的耐受能力,使宿主菌在各种酸性环境下可能表现更好。
2.5 Aprt和Ddl重组菌株对其他胁迫的耐受性 菌株在不同环境胁迫下的适应反应通常是相互联系的,不同的环境刺激,如冷、热、氧和胆盐,会产生相似的反应[15]。Aprt和Ddl在Lc. lactis NZ9000中的表达增强了菌株对酸胁迫的抗性,那么对酸胁迫以外的其他胁迫是否也具有抵抗力则需要进一步探讨。因此,本研究探讨了可能由Lb. bulgaricus CH3中TCS1调控的与菌株耐酸能力相关的Aprt和Ddl对宿主菌Lc. lactis NZ9000在其他胁迫下的影响,包括胆盐胁迫、低温胁迫、氧化胁迫和高温胁迫。
研究结果(图 7)显示,在不同的环境胁迫下,对照菌株乳酸乳球菌NZCK和重组菌株乳酸乳球菌NZAprt和NZDdl的存活率是不同的。Aprt的表达可使重组菌株NZAprt的胆盐耐受力比对照菌株高5倍,但对其他3种胁迫与对照菌株差异不显著。Ddl蛋白的表达可使NZDdl在胆盐胁迫下的耐受力比对照菌株高12倍,在低温胁迫下的耐受力比对照菌株高8倍,但对其他两种胁迫均无显著影响。结果表明,异源表达Aprt和Ddl为Lc. lactis NZ9000提供了对多种胁迫的耐受性。
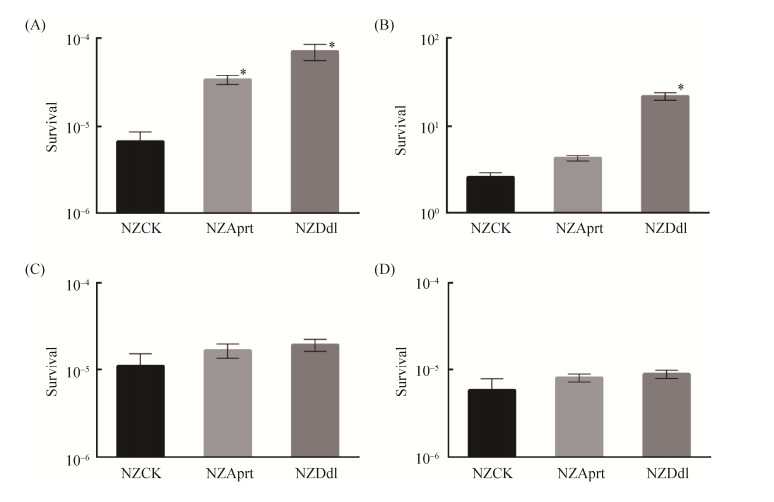 |
| 图 7 多种胁迫下重组菌株的存活情况 Figure 7 The survival of recombinant strains after various stresses. A: Bile salt stress: 1.25% W/V Ox-bile, 20 min; B: Cold stress: 10 ℃, 12 h; C: H2O2 stress: 15 mmol/L H2O2, 30 min; D: Heat stress: 50 ℃, 30 min. The survival rate was calculated equal to the number of living cells before and after the acid treatment. *: P < 0.05. |
| 图选项 |
2.6 Y2H分析 本课题组前期研究发现,自然发酵条件下,Lb. bulgaricus CH3菌株的组氨酸蛋白激酶HPK1突变体CH3-H和应答调节蛋白RR1突变体CH3-R中,Aprt分别提高2.87倍和下降2.01倍;酸适应条件下,D-丙氨酸-D-丙氨酸连接酶(D-alanine-D- alanine ligase,Ddl)在CH3-H和CH3-R中分别比CH3下降2.53倍和3.79倍。Aprt和Ddl的表达与野生型CH3相比有显著变化,且本研究显示Aprt和Ddl在Lc. lactis NZ9000中异源表达显著提高了宿主菌的酸耐受能力。为进一步确定双组分系统中的HPK1、RR1与Aprt、Ddl的潜在相互作用,验证二者之间是否存在蛋白质-蛋白质相互作用,构建了Y2H技术的诱饵质粒和猎物质粒(图 8)。
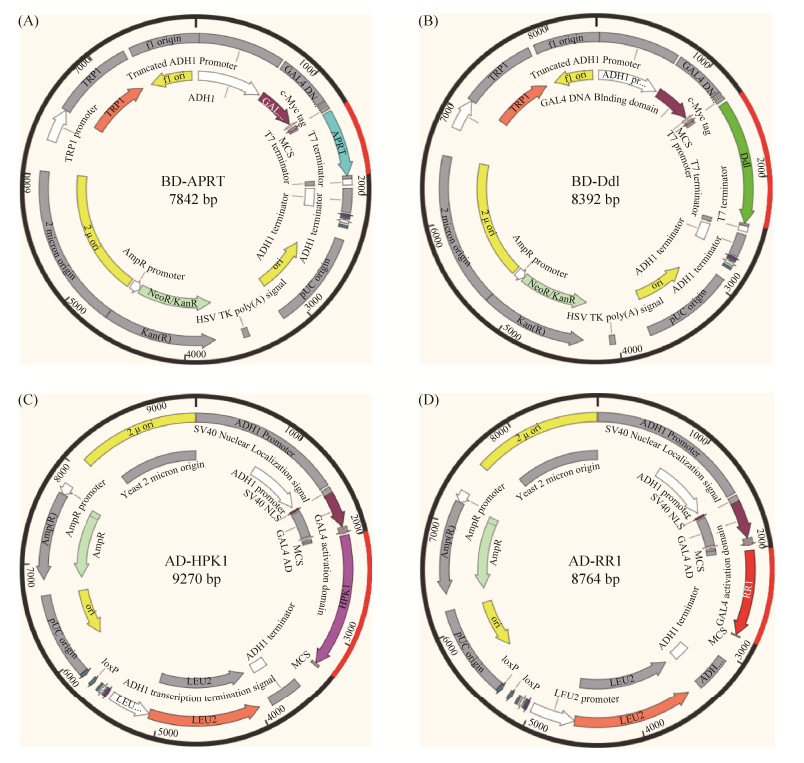 |
| 图 8 酵母双杂交实验中诱饵质粒和猎物质粒的图谱示意图 Figure 8 Sketch maps of bait plasmids and prey plasmids in yeast two hybrid experiments. A: BD-APRT; B: BD-Ddl; C: AD-HPK1; D: AD-RR1. A and B are bait plasmids, C and D are prey plasmids. |
| 图选项 |
本研究中,分别构建了含有转录激活蛋白GAL4 (galactose utilization enzyme,GAL4)激活域和GAL4-DNA结合域的融合表达载体(图 8),并通过DNA测序进行了验证。实验中选择BD-53和AD-T作为阳性对照,BD-Lam与AD、BD与AD-HPK1、BD-APRT与AD、BD-Ddl与AD、BD与AD-RR1作为阴性对照。BD空载体和猎物质粒AD-HPK1共转化子、AD空载体和诱饵质粒BD-Ddl共转化子均可以在营养缺陷型培养基SD/-Leu-Trp上生长,但均不能在营养缺陷型培养基SD/-Leu-Trp-Ade-His上生长,这些结果表明,单独的HPK1和Ddl本身均不能激活下游报告基因的表达。而诱饵质粒BD-Ddl和猎物质粒AD-HPK1共转化子可以在营养缺陷型培养基SD/-Leu-Trp-Ade-His上生长,说明诱饵质粒BD-Ddl和猎物质粒AD-HPK1在酵母Y2HGold中共同表达可以激活下游报告基因的表达,从而使酵母Y2HGold可以在不同的营养缺陷型培养基上正常生长,说明在酵母Y2HGold中HPK1可以与Ddl发生互作。同理,诱饵质粒BD-APRT和猎物质粒AD-HPK1共转化子无法在营养缺陷型培养基SD/-Leu-Trp-Ade-His上生长,说明在酵母Y2HGold中HPK1无法与APRT发生互作。以上Y2H实验结果表明,参与耐酸调节的蛋白质与由HPK1和Ddl组成的多聚体复合物有关(图 9)。
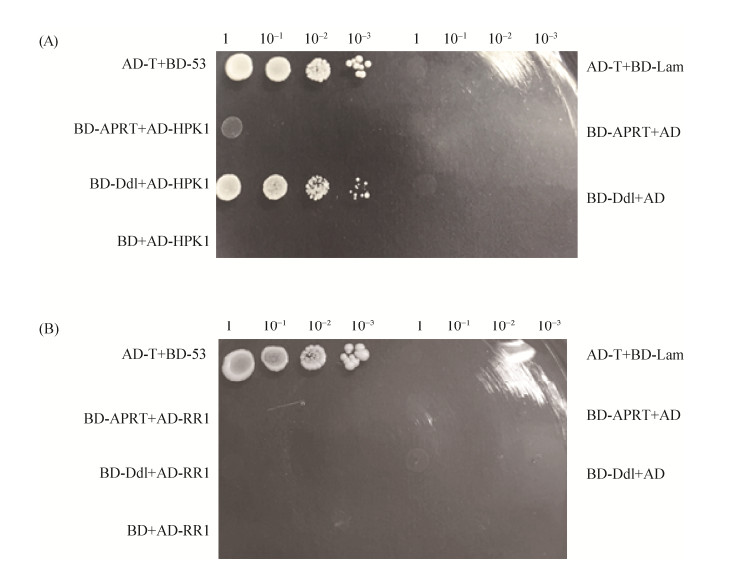 |
| 图 9 酵母Y2HGold中诱饵质粒与猎物质粒相互作用检测 Figure 9 Detection of interactions between bait plasmids and prey plasmids in yeast Y2HGold. A: AD-HPK1; B: AD-RR1. AD: pGADT7 AD cloning vector; AD-T: pGADT7-T; BD: pGBKT7 DNA-BD cloning vector; BD-53: pGBKT7-53; BD-Lam: pGBKT7-Lam; AD-T+BD-53: positive control; AD-T+BD-Lam, BD+AD-HPK1, BD-APRT+AD, BD-Ddl+AD, BD+AD-RR1: negative control; BD-APRT+AD-HPK1, BD-Ddl+AD-HPK1, BD-APRT+AD-RR1, BD-Ddl+AD-RR1: experimental groups to be verified. |
| 图选项 |
2.7 激活域分析 Y2H实验确定组氨酸蛋白激酶HPK1与D-丙氨酸-D-丙氨酸连接酶Ddl之间存在相互作用。根据HPK1蛋白的结构域构建Y2H截短型表达载体,确定Ddl与HPK1相互作用的关键结构域。通过SMART数据库(http://smart.embl.de/)对HPK1蛋白序列进行结构域预测,发现HPK1蛋白具有3类保守的结构域,如图 10所示。从图 10中可以看出,HPK1蛋白有6个跨膜区域(HPK1-A),一个螺旋区域(HPK1-B)和一个组氨酸激酶核心区域(HATPase_c,HPK1-C)。
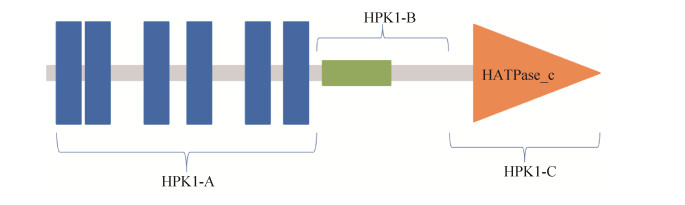 |
| 图 10 HPK1蛋白结构域分析 Figure 10 Domain analysis of HPK1. |
| 图选项 |
在进一步的实验中,将HPK1蛋白分为HPK1-A、HPK1-B和HPK1-C,并分别与GAL4转录激活结构域融合,分析这些结构域与Ddl的交互作用。诱饵质粒BD-Ddl和猎物质粒AD-A和AD-B在Y2HGold细胞中的共表达不能激活下游报告基因his3、ade2和mel1的表达,表明Ddl和HPK1-A、HPK1-B在Y2HGold细胞中不存在明显的互作作用。Ddl只能与HPK1蛋白的HPK1-C区域发生相互作用,导致酵母可以在选择培养基上生长,说明HPK1-C是与Ddl发生相互作用所必需的(图 11)。Y2H结果表明,HPK1与Ddl之间存在相互作用,即HPK1-C与Ddl发生相互作用。
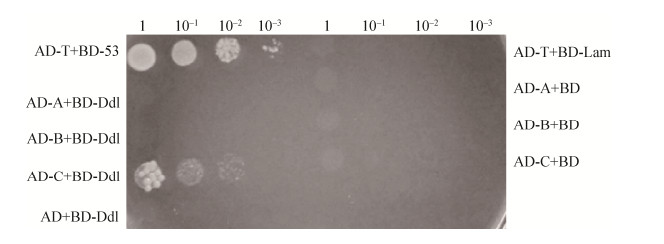 |
| 图 11 截短型诱饵质粒与猎物质粒相互作用检测 Figure 11 Detection of interactions between truncated bait plasmids and prey plasmids. AD: pGADT7 AD cloning vector; AD-T: pGADT7-T; BD: pGBKT7 DNA-BD cloning vector; BD-53: pGBKT7-53; BD-Lam: pGBKT7-Lam; AD-T+BD-53: Positive control; AD-T+BD-Lam, AD+BD-Ddl, AD-A+BD, AD-B+BD, AD-C+BD: Negative control; AD-A+BD-Ddl, AD-B+BD-Ddl, AD-C+BD-Ddl: experimental groups to be verified. |
| 图选项 |
3 讨论 德氏乳杆菌保加利亚亚种在食品工业中有着广泛的应用前景,研究者试图研究其耐酸机理以提高菌株活性和稳定性。本课题组前期研究发现,2-DE和RT-qPCR筛选出了自然发酵条件下和酸适应条件下Lb. bulgaricus CH3中大量由双组分系统TCS1调控的差异表达基因,其中组氨酸蛋白激酶HPK1突变体CH3-H和应答调节蛋白RR1突变体CH3-R中的腺嘌呤磷酸核糖转移酶(Aprt)、D-丙氨酸-D-丙氨酸连接酶(Ddl)、寡肽ABC转运蛋白(OppDII)和延伸因子Ts(Tsf)的表达水平与野生型CH3相比有显著变化。这些蛋白主要参与了嘌呤核苷酸代谢、细胞壁肽聚糖生物合成、氨基酸转运和蛋白质生物合成等过程[8]。
但是,仅仅通过双向电泳和实时定量PCR的结果并不能得出差异表达基因参与菌株耐酸反应的结论,因为并不是所有的差异表达基因都直接参与了菌体的酸耐受反应。因此,需要通过传统分子生物学手段(如基因突变或超量表达)来验证这些差异基因是否在菌体的酸耐受反应中直接发挥作用。由于Lb. bulgaricus的电转化方法不够成熟并且缺少基因突变或同源表达的载体系统,无法进行差异基因的功能分析,因此采用Lc. lactis NZ9000进行异源表达。本实验中,尽管Lc. lactis NZ9000菌株表达了Aprt、Ddl、OppDII和Tsf,但实验方案仍有待改进。在没有抗生素压力的情况下,自由复制的质粒可能是不稳定的。最后,表达系统的蛋白表达水平相对较低。
本研究通过表达Aprt和Ddl,观察到宿主菌耐酸能力的改善,表明了Aprt和Ddl对耐酸反应的贡献。由此可见,Aprt和Ddl是导致菌株在酸性环境中耐受性提高的原因之一,同时,菌株中的耐酸调控过程是涉及多种代谢途径的复杂调控过程,各种代谢过程协同发挥作用进而实现菌株的自我保护。表达Aprt、Ddl也能提高宿主菌的其他抗性能力,如表达Aprt能提高抗胆盐能力,表达Ddl还能提高宿主菌的抗低温能力。说明Aprt和Ddl在宿主菌中的表达在增强菌株抗多种胁迫中起重要作用。
腺嘌呤磷酸核糖转移酶(Aprt)由180个氨基酸组成,是一种在嘌呤补救合成途径中催化腺嘌呤与5-磷酸核糖-1-焦磷酸(PRPP)发生Mg依赖性缩合反应生成一磷酸腺苷(AMP)的酶,可以提高嘌呤碱的利用率,维持嘌呤碱在最佳水平。同时,AMP经过二磷酸腺苷(adenosine diphosphate,ADP)最终形成三磷酸腺苷(adenosine triphosphate,ATP),对菌体内的能量代谢过程产生影响。Aprt在乳酸菌中的具体功能研究较少,需要进一步的研究来阐明其在乳酸菌耐酸调节中的作用。
D-丙氨酸-D-丙氨酸连接酶(Ddl)由362个氨基酸组成,是一种参与细菌细胞壁肽聚糖生物合成过程的ATP依赖酶[16]。Ddl的结构与谷胱甘肽合酶相似,可能通过调节氨基酸代谢来调节菌株的耐酸性。大肠杆菌中,这两种酶可将ATP转化为ADP,并在反应过程中释放肽类物质[17]。酵母中谷胱甘肽合酶活性降低,会导致细胞内甘氨酸转运能力以及谷胱甘肽水平降低[18]。因此,推测该酶不仅参与了细胞壁肽聚糖的合成过程,而且还可能影响了菌株在酸性环境中的能量代谢过程及氨基酸代谢,进而影响了菌株的耐酸能力。
Ddl广泛的底物特异性可能是影响菌株耐酸能力的另一个原因。研究者研究了大肠杆菌K12、伊平屋桥大洋芽孢杆菌JCM 11309、蓝细菌PCC 6803和海栖热袍菌ATCC 43589中Ddl的底物特异性。伊平屋桥大洋芽孢杆菌JCM 11309中的Ddl对D-丙氨酸有很强的底物特异性,而其他3种菌中的Ddl具有广泛的底物特异性。除D-丙氨酸外,还可以使用D-丝氨酸、D-苏氨酸、D-半胱氨酸和甘氨酸作为底物[19]。海栖热袍菌ATCC 43589中Ddl热稳定性高,最适温度高达90 ℃,对15种D-氨基酸,特别是D-丙氨酸、D-半胱氨酸和D-丝氨酸具有底物特异性[20]。Ddl广泛的底物特异性导致菌株即使在酸性环境下,也能保持利用氨基酸高效合成细胞壁肽聚糖的能力,有助于保持细胞壁的物理屏障作用。
一般来说,菌株中的双组分系统可以感知周围环境的各种变化,并将这些信号传递给目标基因,以诱导代谢、生理和/或行为的适应性变化。本课题组前期研究发现,双组分系统TCS1中的HPK1或RR1突变导致菌株耐酸能力下降,Aprt、Ddl等表达量发生改变。本研究进一步研究证实,Aprt和Ddl表达的确提高了宿主的耐酸性,但HPK1、RR1和Aprt、Ddl之间经过怎样的信号转导过程来提高菌株的耐酸性尚有待进一步探讨。
Y2H是研究蛋白质相互作用的有效方法,具有时效性、通用性和适应性等特点。当一个蛋白融合到GAL4激活区,另一个蛋白融合到GAL4-DNA结合区时,GAL4基因的功能可以通过蛋白质的物理相互作用来重建。结构域是生物大分子的一种特殊结构和独立功能区,是蛋白质生理功能的结构基础,与蛋白质的功能密切相关。因此,根据本实验中酵母双杂交实验结果,我们认为HPK1通过HPK1-C和Ddl形成了介导菌株耐酸反应的复合物,来调控其他与耐酸相关基因的表达。本研究为HPK1和Ddl功能相互作用的深入研究奠定了基础,为研究HPK1/RR1双组分系统在耐酸调节中的作用提供了理论依据。
本研究克隆了德氏乳杆菌保加利亚亚种CH3的aprt、ddl、oppDII和tsf基因,并成功地将其导入到乳酸乳球菌NZ9000中。采用多种方法分析了重组菌株的特性。aprt和ddl基因在乳酸乳球菌NZ9000中的表达提高了其耐酸性,并改善了其耐胆盐和/或耐低温能力。因此,本研究所采用的方法是可行的,可以通过外源表达来提高耐酸性,为耐酸调控机制和菌株改良的研究提供了一条潜在的途径。进一步的实验仍需继续进行,表达其他与耐酸调节相关的基因,以评估它们在菌株耐酸调节中的特殊作用。此外,还应开展Aprt和Ddl在德氏乳杆菌保加利亚亚种CH3耐酸性调节中作用相关的研究。
References
| [1] | Makino S, Hemmi J, Kano H, Kashiwagi M, Hojo K, Asami Y. Anti-fatigue effects of yogurt fermented with Lactobacillus delbrueckii subsp. bulgaricus OLL1073R-1 in healthy people suffering from summer heat fatigue:a randomized, double-blind, placebo-controlled trial. Nutrients, 2018, 10(7): 798. DOI:10.3390/nu10070798 |
| [2] | Ai ZW, Lv XP, Huang S, Liu GF, Sun XM, Chen HY, Sun JH, Feng Z. The effect of controlled and uncontrolled pH cultures on the growth of Lactobacillus delbrueckii subsp. bulgaricus. LWT, 2017, 77: 269-275. DOI:10.1016/j.lwt.2016.11.057 |
| [3] | Liang SX, Gao DC, Liu HH, Wang C, Wen JP. Metabolomic and proteomic analysis of D-lactate-producing Lactobacillus delbrueckii under various fermentation conditions. Journal of Industrial Microbiology & Biotechnology, 2018, 45(8): 681-696. |
| [4] | Wang C, Cui YH, Qu XJ. Mechanisms and improvement of acid resistance in lactic acid bacteria. Archives of Microbiology, 2018, 200(2): 195-201. |
| [5] | Van De Guchte M, Penaud S, Grimaldi C, Barbe V, Bryson K, Nicolas P, Robert C, Oztas S, Mangenot S, Couloux A, Loux V, Dervyn R, Bossy R, Bolotin A, Batto JM, Walunas T, Gibrat JF, Bessières P, Weissenbach J, Ehrlich SD, Maguin E. The complete genome sequence of Lactobacillus bulgaricus reveals extensive and ongoing reductive evolution. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(24): 9274-9279. DOI:10.1073/pnas.0603024103 |
| [6] | Pang XY, Zhang SW, Lu J, Liu L, Ma CL, Yang Y, Ti PP, Gao WH, Lv JP. Identification and functional validation of autolysis-associated genes in Lactobacillus bulgaricus ATCC BAA-365. Frontiers in Microbiology, 2017, 8: 1367. DOI:10.3389/fmicb.2017.01367 |
| [7] | Cui YH, Liu W, Qu XJ, Chen ZT, Zhang X, Liu T, Zhang LW. A two component system is involved in acid adaptation of Lactobacillus delbrueckii subsp. bulgaricus. Microbiological Research, 2012, 167(5): 253-261. DOI:10.1016/j.micres.2011.11.003 |
| [8] | Wang C, Cui YH, Qu XJ. Identification of proteins regulated by acid adaptation related two component system HPK1/RR1 in Lactobacillus delbrueckii subsp. bulgaricus. Archives of Microbiology, 2018, 200(9): 1381-1393. DOI:10.1007/s00203-018-1552-9 |
| [9] | 翟征远.德氏乳杆菌保加利亚亚种CAUH1酸耐受机制的蛋白组学研究及抗酸胁迫基因Ldb0677和pyk的功能分析.中国农业大学博士学位论文, 2014. |
| [10] | Wu CD, Zhang J, Du GC, Chen J. Heterologous expression of Lactobacillus casei RecO improved the multiple-stress tolerance and lactic acid production in Lactococcus lactis NZ9000 during salt stress. Bioresource Technology, 2013, 143: 238-241. DOI:10.1016/j.biortech.2013.05.050 |
| [11] | Huang JF, Cheng LL, Li N, Si LF, Luo LX. Food-grade expression of β-glucosidase from Aspergillus aculeatus in Lactococcus lactis. Modern Food Science and Technology, 2014, 30(5): 33-37, 11. (in Chinese) 黄建飞, 成丽丽, 李娜, 司丽芳, 罗立新. 棘孢曲霉β-葡萄糖苷酶的乳酸乳球菌表达. 现代食品科技, 2014, 30(5): 33-37, 11. |
| [12] | Zhai ZY, Douillard FP, An HR, Wang GH, Guo XH, Luo YB, Hao YL. Proteomic characterization of the acid tolerance response in Lactobacillus delbrueckii subsp. bulgaricus CAUH1 and functional identification of a novel acid stress-related transcriptional regulator Ldb0677. Environmental Microbiology, 2014, 16(6): 1524-1537. DOI:10.1111/1462-2920.12280 |
| [13] | Tovpeko Y, Morrison DA. Competence for genetic transformation in Streptococcus pneumoniae:mutations in σA Bypass the comW requirement. Journal of Bacteriology, 2014, 196(21): 3724-3734. DOI:10.1128/JB.01933-14 |
| [14] | Tian HT, Tan JX, Zhang LF, Gu XX, Xu WT, Guo XH, Luo YB. Increase of stress resistance in Lactococcus lactis via a novel food-grade vector expressing a shsp gene from Streptococcus thermophilus. Brazilian Journal of Microbiology, 2012, 43(3): 1157-1164. DOI:10.1590/S1517-83822012000300043 |
| [15] | Wang YH, Li Y, Pei XL, Yu L, Feng Y. Genome-shuffling improved acid tolerance and L-lactic acid volumetric productivity in Lactobacillus rhamnosus. Journal of Biotechnology, 2007, 129(3): 510-515. |
| [16] | Zhang SW, Oh JH, M Alexander LM, ?z?am M, Van Pijkeren JP. D-Alanyl-D-Alanine ligase as a broad-host-range counterselection marker in vancomycin-resistant lactic acid bacteria. Journal of Bacteriology, 2018, 200(13): e00607-17. DOI:10.1128/JB.00607-17 |
| [17] | Fan C, Moews PC, Shi Y, Walsh CT, Knox JR. A common fold for peptide synthetases cleaving ATP to ADP:glutathione synthetase and D-alanine:d-alanine ligase of Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 1995, 92(4): 1172-1176. DOI:10.1073/pnas.92.4.1172 |
| [18] | Mooz ED. Association of glutathione synthetase deficiency and diminished amino acid transport in yeast. Biochemical and Biophysical Research Communications, 1979, 90(4): 1221-1228. DOI:10.1016/0006-291X(79)91167-7 |
| [19] | Sato M, Kirimura K, Kino K. D-amino acid dipeptide production utilizing D-alanine-D-alanine ligases with novel substrate specificity. Journal of Bioscience and Bioengineering, 2005, 99(6): 623-628. DOI:10.1263/jbb.99.623 |
| [20] | Sato M, Kirimura K, Kino K. Substrate specificity of thermostable D-alanine-D-alanine ligase from Thermotoga maritima ATCC 43589. Bioscience, Biotechnology, and Biochemistry, 2006, 70(11): 2790-2792. DOI:10.1271/bbb.60307 |
