柳朝阳1, 向利1, 王红梅1,2, 鲁小璐1


1. 中国地质大学(武汉)环境学院, 湖北武汉 430074;
2. 中国地质大学(武汉)生物地质与环境地质国家重点实验室, 湖北 武汉 430074
收稿日期:2020-02-14;修回日期:2020-04-12;网络出版日期:2020-05-21
基金项目:国家自然科学基金(41877320,41402300)
*通信作者:鲁小璐, E-mail:37113603@qq.com.
摘要:[目的] 对湖南省锡矿山地区的砷氧化菌株的种属进行初步鉴定,并对砷锑氧化菌株Bosea sp.AS-1(简称AS-1)进行全基因组测序和生物信息学分析。[方法] 分离砷氧化菌株,并利用16S rRNA基因测序进行菌种鉴定。在此基础上,对能够高效氧化砷的菌株AS-1进行全基因组测序,对测序数据进行基因组装和功能注释、COG、GO及KEGG聚类分析,以及次级代谢产物合成基因簇与代谢途径预测等。[结果] 湖南省锡矿山的砷氧化菌株主要分布在α-、β-、γ-变形菌纲以及厚壁菌门。菌株AS-1基因组的测序结果显示AS-1基因组包含一条大小为5.536 Mb环状染色体和两个大小分别为189.9 kb和112.1 kb的质粒。对AS-1基因组进一步分析发现该菌株的基因组中包含砷锑代谢相关基因,还有鞭毛形成、鞭毛运动及生物膜形成的基因,这些基因的存在可能与AS-1能高效耐受和氧化砷和锑的特性相关。此外,菌株AS-1中还存在部分碳固定基因和硫氧化基因,这暗示着AS-1能够进行自养生长并氧化环境中的硫元素。[结论] 菌株AS-1可以在自养条件下生长并且能够氧化Sb(Ⅲ)为Sb(V)。
关键词:Bosea sp.AS-1全基因组测序砷、锑代谢基因代谢通路
Screening and genome analysis of arsenite-oxidizing strain Bosea sp. AS-1
Chaoyang Liu1, Li Xiang1, Hongmei Wang1,2, Xiaolu Lu1


1. School of Environmental Studies, China University of Geosciences(Wuhan), Wuhan 430074, Hubei Province, China;
2. State Key Laboratory of Biogeology and Environmental Geology, China University of Geosciences(Wuhan), Wuhan 430074, Hubei Province, China
Received: 14 February 2020; Revised: 12 April 2020; Published online: 21 May 2020
*Corresponding author: Xiaolu Lu, E-mail: 37113603@qq.com.
Foundation item: Supported by the National Natural Science Foundation of China (41877320, 41402300)
Abstract: [Objective] In this study, we aimed to characterize the community structure of arsenite-oxidizing bacteria in Xikuangshan of Hunan Province, and the whole genome of an arsenite-antimonite-oxidizing strain Bosea sp. AS-1 (Abbreviation:AS-1). [Methods] Arsenite-oxidizing bacterial strains were isolated from the samples collected in Xikuangshan, and 16S rRNA genes were sequenced for phylogenic analysis. Whole genome of strain AS-1 was sequenced and analyzed using relevant software and databases for genome assembly, gene prediction and functional annotation. [Results] The arsenite-oxidizing bacteria in Xikuangshan were mainly distributed in α-, β-, γ-Proteobacteria and Firmicutes. AS-1's genome contained one circular chromosome with a size of 5.536 Mb and two plasmids of 189.9 kb and 112.1 kb, respectively. Further analysis on AS-1's genomic data reveal many genes related to arsenic and antimony metabolism, as well as flagella formation, flagellar movement, and biofilm formation. These genes may be involved in AS-1's resistance to high level of arsenic and antimony in environment. Besides, several carbon-fixation genes and sulfur-oxidizing genes were also found in the genome of AS-1, suggesting that AS-1 may grow autotrophically and oxidize sulfur. [Conclusion] Subsequent experiments confirmed autotrophic growth of AS-1.
Keywords: Bosea sp. AS-1whole genome sequencingarsenic and antimony metabolism genesmetabolic pathways
砷(As)和锑(Sb)是位于元素周期表中第V主族元素,它们具有相似的化学和毒理性质[1]。长期暴露于砷、锑污染的环境中会损害人们的肝脏、心脏和心血管系统等[2],对人类的健康造成严重危害,因此砷和锑及其化合物被美国环境保护署和欧盟视为主要污染物[3-4]。近年来由于人类活动导致砷和锑的大量释放,引发各种环境污染问题[5]。尽管砷和锑具有毒性,但是很多微生物可以在高砷和/或高锑的环境中存活,并且对As(Ⅲ)和/或Sb(Ⅲ)进行生物氧化,将它们由毒性较高的三价转换为五价,从而降低砷和锑在环境中的毒性。
目前已报到的砷氧化菌株种类很多,广泛分布于各种环境,如矿区尾矿、土壤、沉积物、酸性矿坑水、地下水以及热泉等。这些菌株主要隶属于变形菌门中的α-、β-、γ-变形菌纲、异常球菌门以及厚壁菌门[6-8]。目前对微生物参与砷代谢的机理已经研究得比较清楚:微生物对亚砷酸盐的摄入主要是通过细胞膜上的甘油通道蛋白GlpF,使亚砷酸盐进入微生物细胞内,此外甘油通道蛋白GlpF的同源蛋白FpslP也发挥相似的功能[9]。细胞中一部分未被氧化的As(Ⅲ)通过由ArsB形成的ATP依赖性亚砷酸盐转运体外排出[10],或通过另一种亚砷酸盐转运体ACR3将其外排出体外[11]。微生物对亚砷酸盐的氧化过程主要由砷氧化操纵子aio操纵子调控。该操纵子主要包括的基因有:亚砷酸盐氧化酶基因aioBA、c型细胞色素(cytC)、钼蛋白合成基因(chlE)[12]、感应器激酶基因aioS和响应器基因aioR。在含有亚砷酸盐的情况下,aio操纵子的各个基因共同控制微生物对As(Ⅲ)的氧化功能和耐受功能。另外,细菌编码砷抗性(ars)操纵子中除了基因arsC外,还包含arsH[13]、硫氧还原蛋白基因arsTX[14]以及乙酰胺转移酶基因arsN[15]等,这些基因共同构成了砷抗性系统,帮助微生物细胞耐受环境中高浓度的砷。
到目前为止已被鉴定的锑氧化菌株有70多株,它们分离于被锑污染的沉积物、矿井废水、工厂含锑排污水以及锑矿尾矿中,这些锑氧化菌株主要分布在α、β、γ-变形杆菌纲和放线杆菌门。在这些已报道的锑氧化菌株中只有两株分离自岩石矿物中的菌株Stibiobacter senarmontii[16]和IDSBO-4[17]能够在自养条件下氧化锑,其他菌株则是在异养条件下进行锑氧化。在大肠杆菌中的研究发现微生物对Sb(Ⅲ)的摄入可以通过甘油转运体GlpF进行[18]。锑氧化菌株对胞内的Sb(Ⅲ)的外排则主要是通过外排蛋白ArsB和Acr3作用[19]。2015年在菌株Agrobacterium tumefaciens 5A中的研究发现,砷氧化酶AioAB不仅可以氧化砷,还可以催化对Sb(Ⅲ)的氧化过程[20];此外,Li等在菌株GW4中鉴定出另一种Sb(Ⅲ)氧化酶AnoA[21],并且在后续的研究中发现在该菌株中的氧化应激反应产生的H2O2可以作为非生物因子催化从Sb(Ⅲ)到Sb(V)的氧化过程[22]。另外在对菌株Agrobacteriumtumefaciens GW4的研究中发现,基因phoB2对Sb(Ⅲ)的氧化具有正向调节作用,且该基因与菌株对Sb(Ⅲ)的抗性有关[23]。
中国湖南省锡矿山有世界上最大的锑矿,被誉为“世界锑都”。由于常年锑矿的开采和冶炼活动导致该地区土壤和地下水中砷、锑污染严重[24]。因此,该地区为研究微生物和砷、锑的相互作用以及微生物的砷、锑代谢研究提供了理想场所。在本研究中,我们从锡矿山采集并分离纯化砷氧化菌株,以了解该地区砷氧化菌的群落组成;另外对其中的一株能够高效氧化砷和锑的菌株AS-1进行了全基因组测序,通过分析该菌株的基因组组成与结构,尝试从基因组的层面理解该菌株能较好适应矿区污染环境的原因。
1 材料和方法 1.1 材料
1.1.1 实验材料: 本研究所用的样品采集于湖南省冷水江市锡矿山锑矿区,样品的种类主要有矿渣、尾矿和尾矿渗水等10个样品组。将所采集的样品封装在无菌的采样袋中并置于低温保温箱中带回实验室,立即进行富集、分离、纯化筛选菌株。
1.1.2 培养基: 实验中所用的培养基为改良后的CDM培养基[25]和MSM培养基[26]。CDM培养基配方为:0.975 g/L MgSO4,1.00 g/L NH4Cl,0.014 g/L K2HPO4,0.994 g/L Na2SO4,0.051 g/L CaCl2,0.1% (W/V) yeast extract,pH 7.2。MSM培养基配方为:7.9 g/L Na2HPO4,1.5 g/L KH2PO4,0.3 g/L NH4Cl,0.1 g/L MgSO4·7H2O,5 mL/L微量元素,10 mL/L维生素溶液,10 mmol/L的HCO3– (NaHCO3),pH 7.2。微量元素配方为:50 g/L EDTA,22 g/L ZnSO4·7H2O,5.54 g/L CaCl2,5.06 g/L MnCl2·4H2O,4.99 g/L FeSO4·7H2O,1.1 g/L (NH4)6Mo7O24·4H2O,1.57 g/L CuSO4·7H2O,1.61 g/L CoCl2。维生素溶液配方:0.002 g/L生物素(Biotin),0.002 g/L叶酸,0.01 g/L盐酸吡哆醇,0.005 g/L烟酸,0.005 g/L泛酸,0.0001 g/L维生素B12,0.005 g/L对氨基苯甲酸,0.005 g/L硫辛酸。
1.2 砷氧化菌株的富集与分离纯化 向100 mL含As(Ⅲ) (终浓度1 mmol/L)的CDM培养基加入1 g (水样为1 mL)采集的样品,将其置于恒温振荡摇床(37 ℃,150 r/min)中培养72–84 h,然后取200 μL涂平板,于恒温培养箱中培养48 h后,挑取不同形态的菌株利用平板划线法进行进一步的分离、纯化。
1.3 菌株的鉴定与系统发育树分析 将实验纯化得到的单菌株接种到含有2 mmol/L As(Ⅲ)的CDM液体培养基中培养72–96 h,取菌液,离心取上清,然后用高锰酸钾法对菌株的砷氧化能力进行检测[27]。记录具有砷氧化能力的菌株并对他们的16S rRNA基因进行PCR扩增,PCR扩增过程所需的引物27F (5′-AGAGTTTGATCCPTGGCTCAG-3′)和1492R (5′-GGTTACCTTGTTACGACTT-3′)。PCR扩增的体系为:Ex Taq (12.5 μL)、27F (1 μL)、1492R (1 μL)、DNA (2 μL)、ddH2O (8.5 μL);扩增的程序为94 ℃ 5 min,30个循环(94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min),72 ℃ 10 min。将扩增成功的PCR产物送测序公司测序,利用NCBI对测序成功的序列进行BLAST比对,并和各个菌株同属的砷氧化菌株的16S rRNA序列用MEGA7.0软件利用邻接法建进化发育树。
1.4 菌株AS-1基因组DNA的提取与全基因组测序 将纯化后的菌株AS-1接种到CDM液体培养中于30 ℃、150 r/min条件下培养至稳定期,后用细菌基因组DNA提取试剂盒提取菌株AS-1的基因组DNA,送到上海美吉生物医药科技有限公司进行基因组测序。
1.5 菌株AS-1的全基因组测序与基因注释 菌株AS-1全基因组测序与基因注释过程如下。(1)基因组测序主要分为两部分,一部分是利用CovarsM22仪器将基因组DNA片段化处理,然后构建小片段PE文库并对片段进行Illumina Hiseq测序。另一部分利用G-tubes方法将基因组DNA处理成片段并将片段两端分别连接成环状单链,然后对单链片段进行三代单分子测序并单分子测序质量评估。(2)对原始测序数据进行质控和对原始数据质量剪切。(3)利用SOAPdenovo (v2.04)初步组装Illumina测序数据,利用blasR比对Pacbio的测序数据,根据比对结果对单分子测序数据进行矫正与纠错;然后基于PacBio校正后的原始测序数据,利用HGAP组装软件对原始测序数据进行组装,最终拼接得到0 GAP的全基因组序列。(4)利用Glimmer 3.02软件进行菌株AS-1的基因预测。用Barmao7.0和tRNA-SE v1.3.1软件预测基因组中的rRNA和tRNA;利用NR数据库、GOC数据库、CO数据库、KEGG数据库等对菌株AS-1基因组中的基因进行预测和注释。
1.6 GenBank登录号 菌株AS-1的基因组序列已提交GenBank,登录号为CP022372。
1.7 菌株AS-1的自养生长及其对Sb(Ⅲ)的氧化 将活化后的AS-1的菌液接种到液体CDM培养基中于35 ℃、150 r/min振荡培养至OD600值约为0.8,按照实验MSM培养基体积的1% (W/V),取菌液于无菌的离心管中离心(3000 r/min,5 min)、弃上清,然后用无菌的生理盐水洗涤菌株沉淀,重复3次,以除去其中的CDM培养基的成分。最后将含有菌株AS-1的悬浊液接种到含1 mmol/L Sb(Ⅲ)的MSM液体培养基中于35 ℃、150 r/min恒温振荡培养。每隔12 h取1次样并用紫外分光光度计测定OD600值,将所取的样品进行离心(12000 r/min,3 min),取上清保存,待取样结束之后用高效液相色谱仪(LC-20AT,日本岛津公司,日本)与原子荧光光谱仪(SA-10,北京吉天仪器有限公司,中国)联用测定样品中不同价态Sb的浓度。
2 结果和讨论 2.1 菌株的鉴定及筛选 实验总共分离出17株不同的砷氧化菌株。通过利用邻接法将这17株菌株的16S rRNA序列和18株已发表的砷氧化菌菌株的16S rRNA序列构建系统进化发育树(图 1),发现本研究分离得到17株菌株主要分布在变形菌门中的无色杆菌属(Achromobacter)、农杆菌属(Agrobacterium)、申氏杆菌属(Shinella)、剑菌属(Ensifer)、芽孢杆菌属(Bacillus)、包西氏属(Bosea)、假单胞菌属(Pseudomonas)、苍白杆菌属(Ochrobactrum)和厚壁菌门中。这些菌属中均报道过砷氧化菌株的存在,与我们的结果一致。Yamamura等在2014年利用已报道的砷氧化菌株的16S rRNA基因构建进化发育树后发现,砷氧化菌株主要分布现在α-、β-、γ-变形菌纲、厚壁菌门和异常球菌门中[28]。本研究分离的砷氧化菌株的分类和Yamamura等的研究结果相吻合。
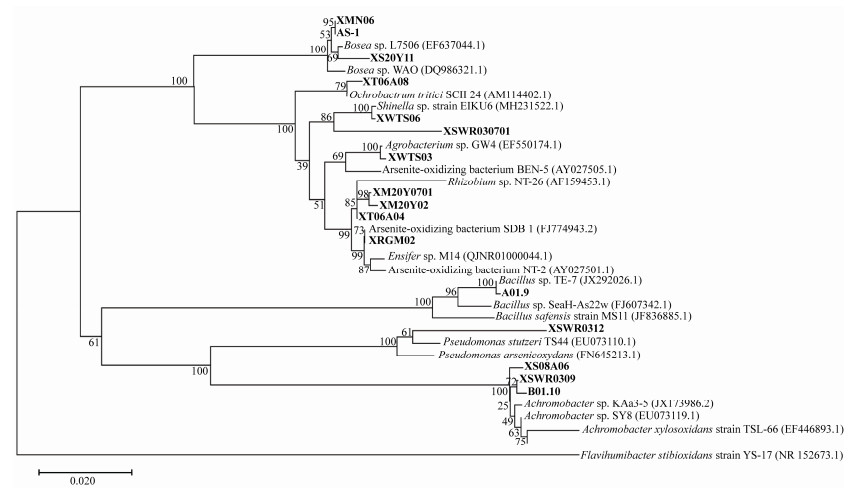 |
| 图 1 基于16S rRNA和NJ法构建的湖南锡矿山锑矿区砷氧化菌的系统发育进化树 Figure 1 The phylogenetic tree based on the 16S rRNA gene of arsenate-oxidizing bacteria at Xikuangshan mining area of Hunan province via N-J method with bootstrap values of 1000 resampling. The numbers at each node that indicate the percentage of bootstrap supporting. The numbers in the brackets followed each bacterial name are 16S rRNA sequence accession numbers in NCBI. |
| 图选项 |
2.2 锡矿山可培养砷氧化菌的特点 分离自湖南锡矿山的砷氧化菌株主要分为以下几个属(图 1):Bosea属主要包含菌株XMN06、AS-1和XS20Y11;Ochrobacterum属包含XT06A08;Shinella属包含XWTS06和XSWE030701;Agrobacterium属包含XWTS03;Rhizobium属包含菌株XM20Y0701、XM20Y02和XT03A04;Ensifer属包含XRGM02;Bacillus属包含A01.9;Pseudomonas属包含XSWR0312;Achromobacter属包含菌株XS08A06、XSWR0309、B01.10及XM0Y04。Bosea属的3株菌株中AS-1的16S rRNA基因与XMN06在进化距离上更为接近,而且这3株菌株的16S rRNA基因与分离自高砷地下水中的好氧自养型砷氧化菌株Bosea sp. L7506[29]的16S rRNA基因的相似度最高。分离的砷氧化菌株XWTS06属于Shinella属,该菌株和分离于含砷铀矿的能够耐受高浓度砷的砷氧化菌株Shinella sp. EIKU6相似,研究结果表明菌株Shinella sp. EIKU6不仅能够在不同的培养条件下进行砷氧化,而且对镉、铬、钴、锌等重金属也具有一定的抗性[30]。XRGM02隶属于Ensifer属,在进化树中和分离自韩国Sangdong矿区尾矿中的砷氧化菌株SDB1最为接近,SDB1既可以在异养条件下高效氧化砷,又可以在自养条件下对砷进行氧化[31]。在Agrobacterium属中和分离的菌株XWTS03相似的菌株是分离于山西省山阴市地下水沉积物中的砷锑氧化菌Agrobacterium tumefaciens GW4,研究结果表明菌株GW4既可以氧化砷又可以氧化锑[32]。和XM20Y04同属的菌株Achromobacter sp. SY8与菌株XSWR0312同属的砷氧化菌株Pseudomonasstutzeri TS44都分离自湖北省黄石市高砷污染环境[33],与菌株XT06A08同属的砷氧化菌株是分离自农田土壤的砷氧化菌株Ochrobactrum tritici SCII24T,实验结果显示该菌株不仅能够耐受50 mmol/L的亚砷酸盐和200 mmol/L的砷酸盐,而且该菌株也能耐受10 mmol/L的亚锑酸盐[34]。与菌株A01.9同属的砷氧化菌株有3株,由进化树可以看出,菌株A01.9和分离自印度马哈拉施特拉邦制革厂污水的砷氧化菌株Bacillus sp. TE-7进化距离比较近,实验表明TE-7能够在12 h内完全氧化2 mmol/L的As(Ⅲ),另外研究表明该菌株不仅能够氧化As(Ⅲ),还能够还原Cr(VI)[35]。综上所述,上述已发表的砷氧化菌株主要分离自含砷矿物、高砷或砷污染的水体以及砷污染的土壤和沉积物等环境中。这与本文筛选的砷氧化菌株的分离环境具有相似之处,如矿渣和尾矿、受污染的水体——尾矿渗水以及受污染的土壤中。由此推测这些菌株之间的砷氧化特性可能会相似,但是具体的砷锑氧化特性需要进一步的研究。
2.3 菌株AS-1基因组基本信息 我们前期的实验研究发现菌株Bosea sp. AS-1是一株高效的砷锑氧化菌株,且能耐受高达120 mmol/L的As(Ⅲ)和50 mmol/L的Sb(Ⅲ)[36]。因此我们对菌株AS-1进行了全基因组测序进一步探究该菌株耐受砷锑的分子机制,测序结果显示本次测序获得的reads的长度大小主要集中分布在0–20000 bp,测序质量良好,测序深度为296X,最终拼接得到0 GAP的全基因组序列。经过对测序结果分析我们发现菌株AS-1的基因组中包括一条大小为5.53 Mb环状的染色体,以及2个大小分别为189990 bp和112144 bp的质粒(图 2),其GC含量分别为65.86%、63.25%、66.89% (表 1);通过基因注释,在菌株AS-1染色体上共发现5514个CDS (coding sequences),两个质粒中的CDS分别为260 (质粒1)和152个(质粒2)。AS-1的基因组中含有50个tRNA和6个rRNA。
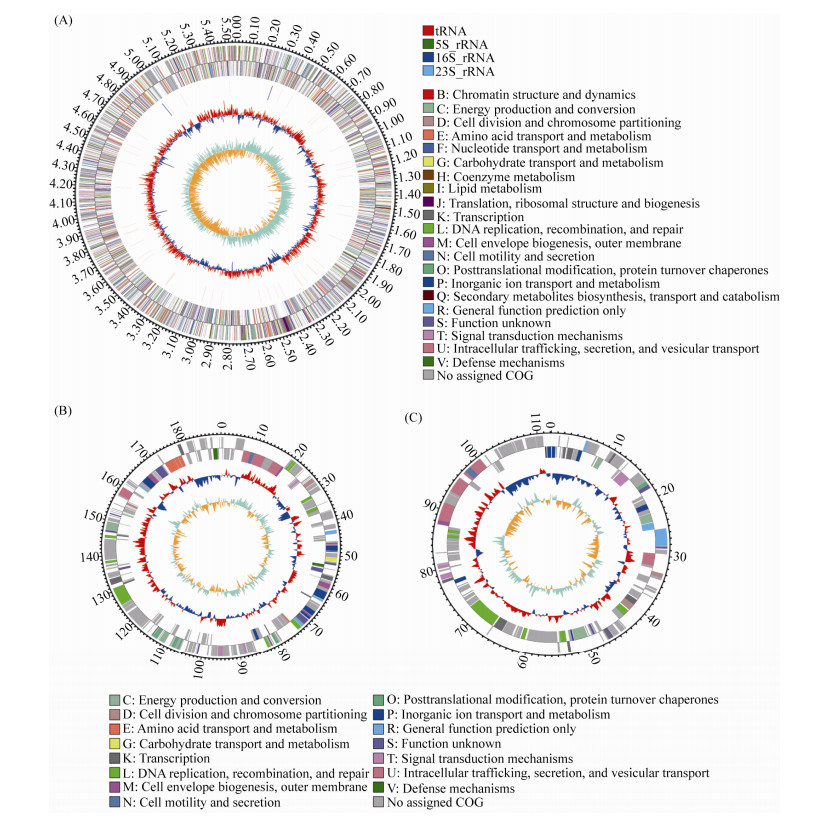 |
| 图 2 菌株Bosea sp. AS-1基因组基因圈图 Figure 2 The genomic map of chromosome (A), plasmid 1 (B) and plasmid 2 (C) of Bosea sp. AS-1. The outermost circle of the circle diagram is the genome-sized logo with 0.1 Mb between each scale value. The second and third circles are CDS on the positive and negative chains, and the different colors indicate the functional classification of different COGs of the CDS. The fourth circle is rRNA and tRNA; the fifth circle is GC content (Figure B, C does not have this circle). The outward red portion indicates that the GC content in the region is higher than the whole genome average GC content, and the inward blue portion indicates that the GC content in the region is lower than the whole genome average GC content. The innermost circle is the GC skew value 100 bp. The specific algorithm is G-C/G+C. In the biological sense, the positive value is more inclined to transcribe CDS, and the negative chain is more inclined to transcription CDS. |
| 图选项 |
表 1. 菌株Bosea sp. AS-1全基因组基本信息表 Table 1. The basic genome information of the Bosea sp. AS-1
| Features | Chromosome | Plasmid 1 | Plasmid 2 |
| Gene num (#) | 5514 | 260 | 152 |
| Gene total length/bp | 5536255 | 189990 | 112144 |
| Gene average length/bp | 905.27 | 640.68 | 653.76 |
| GC% | 65.86 | 63.25 | 66.89 |
| Gene density/(number/kb) | 1 | 1.37 | 1.36 |
表选项
将菌株AS-1的全基因组信息利用COG数据库进行功能注释(图 3-A),我们发现AS-1的基因可分为25个功能分类,其中注释基因数量最多的4个组别为氨基酸转运和代谢(16.6%)、无机盐离子转运和代谢(9.79%)、能量产生和转换(7.57%)以及碳水化合物的转运和代谢(9.87%)。
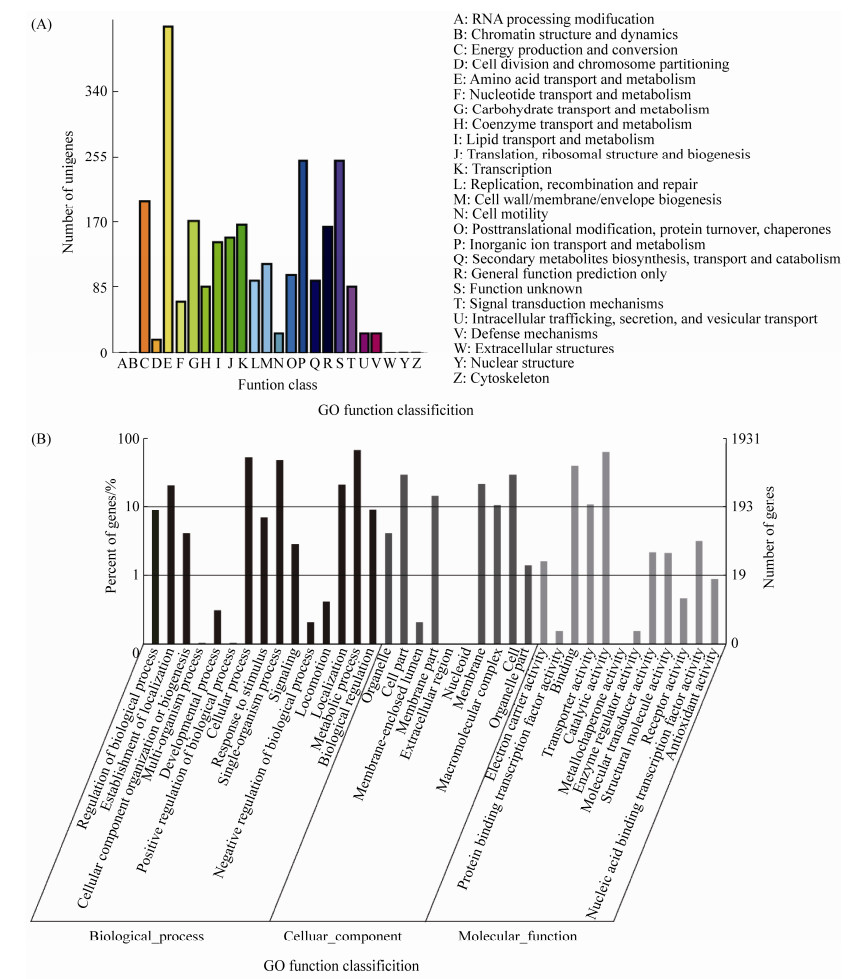 |
| 图 3 菌株Bosea sp. AS-1基因组中COG数据库(A)和GO (B)数据库的功能注释 Figure 3 Functional annotations of Bosea sp. AS-1 genome against the COG database (A) and GO database (B). |
| 图选项 |
利用GO (gene ontology)数据库对AS-1的基因组进行功能注释(图 3-B)后发现,AS-1的基因总共可分为37类,其中和代谢基因相关的基因数量最多,为1323,参与细胞过程的基因有1041个,参与单一生物过程中的基因为925个。
通过KEGG (Kyoto Encyclopedia of Genes and Genomes)注释(图 4),结果显示AS-1基因组中共注释到了2795个基因(50.69%)分布于197个代谢途径中。从图中可以看出,注释基因中和代谢相关的基因最多,包括碳水化合物代谢、氨基酸代谢、能量代谢以及脂类代谢等。
 |
| 图 4 菌株Bosea sp. AS-1基因组中KEGG代谢通路注释图 Figure 4 Annotation map of KEGG metabolic pathways in the genome of Bosea sp. AS-1. |
| 图选项 |
将AS-1的基因组数据和同属中已完成全基因组测序的其他菌株的基因组数据进行比较(表 3),发现Bosea属基因组大小为5.5–7.0 Mb,GC含量在65%左右。基因数量在5100到6500之间。本研究中的菌株AS-1的基因组大小、GC含量以及基因数量和CDS数量接近于平均值。此外,将菌株AS-1的基因组信息和Alexandra等研究中与菌株WAO比较的Bosea属中其他10株菌株的基因组信息进行比较[37],发现菌株AS-1的基因大小、功能基因数以及COG等均高于这10株菌株基因组的平均值。
表 3. 菌株Bosea sp. AS-1菌株与Bosea属其他菌株的全基因基本信息比较表 Table 3. Comparison of complete gene information among Bosea sp. AS-1 and other strains in the genus of Bosea
| #Organism name | Size/Mb | GC/% | Scaffolds | Genes | Proteins | Level | Replicons |
| Bosea sp. F3-2 | 7.044 | 65.4 | 3 | 6594 | 6305 | Complete genome | Chromosome: NZ_CP042331.1/CP042331.1; plasmid pB32-1: NZ_CP042332.1/CP042332.1; plasmid pB32-2: NZ_CP042333.1/CP042333.1 |
| Bosea sp. Tri-49 | 6.45 | 65.9 | 2 | 6242 | 6096 | Complete genome | Chromosome: NZ_CP017946.1/CP017946.1; plasmid unnamed1: NZ_CP017947.1/CP017947.1 |
| Bosea sp. AS-1 | 5.53 | 63.25 | 3 | 5667 | 5501 | Complete genome | Chromosome: NZ_CP022372.1/CP022372.1; plasmid unnamed1: NZ_CP022370.1/CP022370.1; plasmid unnamed2: NZ_CP022371.1/CP022371.1 |
| Bosea sp. PAMC 26642 | 5.51 | 65.2 | 2 | 5294 | 5153 | Complete genome | Chromosome: NZ_CP014301.1/CP014301.1; plasmid unnamed: NZ_CP014302.1/CP014302.1 |
| Bosea sp. RAC05 | 5.62 | 67.6 | 2 | 5412 | 5274 | Complete genome | Chromosome: NZ_CP016464.1/CP016464.1; plasmid pBSY19_1: NZ_CP016463.1/CP016463.1 |
表选项
2.4 菌株AS-1中砷锑代谢相关基因 对菌株AS-1基因组的进一步分析发现,菌株AS-1的基因组中含有许多与砷代谢相关基因:其染色体上有完整的aio操纵子,包含aioBA、cytc、moeA、aioS和aioR基因;负责细胞内As(Ⅲ)和As(V)转运的相关基因,如亚砷酸盐载体基因家族中的acr3、磷酸盐转运蛋白Pit、Pst以及砷抗性相关基因arsC、arsH和arsR (图 5)。有研究报道,aioR可能通过调节Ars释放细胞质中的As(Ⅲ),和/或调节anmK参与细胞壁/膜的代谢,以阻止As(Ⅲ)的进入,从而避免了As(Ⅲ)对细胞的毒害[38]。实际上,本课题组前期的研究表明,AS-1可以在24 h内完全氧化2 mmol/L的As(Ⅲ)[36]。因此,这些砷代谢基因的存在可能帮助AS-1氧化和耐受砷,从而更好地生存于高砷环境中。
 |
| 图 5 菌株Bosea sp. AS-1中砷、锑代谢相关基因 Figure 5 Possible arsenic and antimony metabolism genes in strain Bosea sp. AS-1. The solid line represents the metabolic process verified in other arsenite- or antimonite-oxidizing bacteria, while? indicates that the metabolic mechanism is unknown. |
| 图选项 |
通过比较菌株AS-1和Bosea属中其他几个已测序菌株(Bosea sp. 117、Bosea sp. WAO、Bosea sp. 67-29、Bosea sp. DSM18164及Bosea vavilaviae SD260)的砷代谢相关基因簇(图 6),笔者发现这几个菌株中均含有与aio操纵子相关的基因和与砷抗性相关的基因arsC、arsH以及arsR等,且这些菌株中的砷代谢相关的基因在染色体上的排列顺序基本是一致的(图 6)。由此推测菌株AS-1中砷代谢基因的进化和同属的菌株可能是一致的。其中菌株AS-1的aio操纵子和菌株Bosea sp. WAO和Bosea sp. 117的aio操纵子中各个基因的排列相似度最高,而且这3株菌株都具有砷氧化能力,这说明这AS-1的砷氧化机制可能与菌株WAO和117相似。但AS-1和其他5株菌株的砷代谢基因簇表现出差异(图 6)。其中,117菌株中缺失了moeA和cytC基因,而菌株B. vavilaviae SD260的aio操纵子中只含有aioB基因;菌株B. sp. DSM18164的砷代谢基因簇在染色体上的顺序与菌株AS-1的基因顺序相反。发生这种变化的原因可能是菌株在进化过程中发生了基因的水平转移,使得这些菌株中虽然含有和砷代谢相关的基因簇,但是各菌株中基因簇中的基因具有差异。
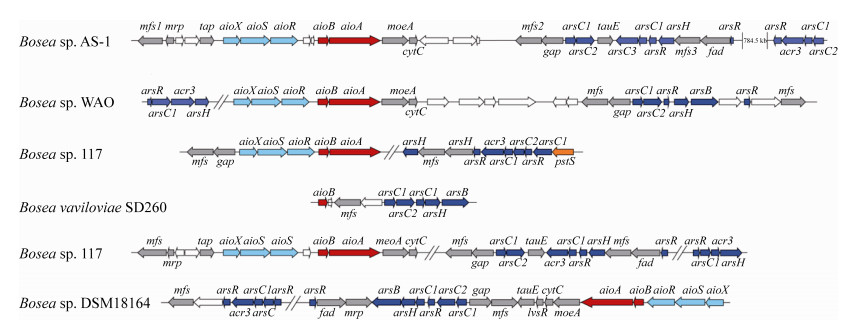 |
| 图 6 Bosea属菌株中砷代谢基因簇 Figure 6 Arsenic metabolism gene cluster in Bosea. The white arrow indicates the unknown functional protein, and the double slash indicates that the genes on both sides belong to different scaffolds. |
| 图选项 |
另外,在AS-1的基因组中发现了一些已报道的和锑代谢相关的基因,包括能够进行锑氧化的基因aioBA,能够将细胞内Sb(Ⅲ)排出的acr3,将Sb(V)运输出细胞外的ABC转运子,以及能够转运Sb(V)的磷酸盐通道蛋白Pit和Pst等。课题组前期研究表明,AS-1具有很高的锑氧化和锑耐受能力[36],AS-1基因组中这些锑代谢相关基因的存在表明AS-1可能通过这些基因的表达,增强菌株对锑的耐受性,减少Sb(Ⅲ)对细胞的毒害和胁迫作用,从而在含锑的环境中更好地生长。
此外,笔者还发现部分和砷锑代谢相关的基因在AS-1染色体和质粒上都存在,而且一些基因在染色体和质粒上存在着多拷贝,如aioA、arsC、arsR、GHS、arc3、cytC以及磷酸转运通道Pst相关基因等(图 5)。这些基因分布在染色体和质粒上,以及它们多拷贝特点可能和菌株AS-1能够耐受高浓度的砷、锑以及AS-1高效的砷锑氧化能力有关。进一步分析发现,分布在基因组上不同位置的这些砷锑代谢基因的氨基酸序列所注释到的源菌株不同,如AS-1染色体上的aioA基因注释的源菌株为Xanthobacter autotrophicus[39],质粒上的aioA基因的源菌株是一株分离自美国黄石公园热泉中的嗜热砷氧化菌株Thermocrinis ruber DSM 12173[40]。这说明AS-1的染色体和质粒上的aioA基因可能有不同的来源,发生这种现象的原因可能是细菌在进化的过程中发生了基因的水平转移。
前人研究表明,菌株A. tumefaciens 5-A可以利用自身的砷氧化酶AioBA对Sb(Ⅲ)进行氧化[20]。我们的前期实验发现,AS-1在不同碳源条件下对砷锑表现出不同的氧化特性,在以酵母提取物为碳源时,菌株AS-1对砷的氧化能力强于锑,但是在以乳酸钠为碳源的条件下菌株对锑的氧化效率优于砷[36],这说明菌株AS-1对As(Ⅲ)、Sb(Ⅲ)的氧化可能是由不同的代谢途径所控制,暗示AS-1中可能存在AioBA之外的酶负责对锑的氧化。目前已发现的另一个细菌锑氧化酶是anoA,但在AS-1的基因组中并没有发现anoA基因,因此AS-1中可能存在新的锑氧化酶基因。
另外,基因组分析发现菌株AS-1中缺少可介导As(Ⅲ)和Sb(Ⅲ)外排的转运蛋白基因arsB,但是AS-1中含有一种三价类金属或H+逆向转运基因acr3。研究表明基因acr3可与arsC进行共转录,并代替arsB发挥功能使得细胞内的As(Ⅲ)和Sb(Ⅲ)的外排至体外[40]。在AS-1的基因组中并未发现编码吸收As(Ⅲ)和Sb(Ⅲ)的通道蛋白甘油通道蛋白的基因glpF,AS-1能够耐受高浓度的Sb(Ⅲ)并具有高效的Sb(Ⅲ)氧化效率[35],这说明菌株AS-1中可能存在非GlpF依赖的其他的Sb(Ⅲ)吸收途径。
2.5 重金属抗性的基因 AS-1基因组中还包含许多与重金属抗性相关的基因,如铜抗性的基因,铜转运P型ATP酶(可能涉及铜、铅、镉、锌和汞的转运),钴、锌和镉的抗性基因czcA等,暗示着AS-1可能具有这些重金属抗性,这与课题组前期的实验结果菌株AS-1可以在含有Zn(Ⅱ)、Pb(Ⅱ)、Cu(Ⅱ)的环境中生长相一致[36]。这也和菌株AS-1的分离地环境相符合,锑矿在生成时会和一些其他的重金属共生成矿,因此分离地含有多种重金属污染。AS-1的这些重金属抗性基因可以增加自身对生存环境的适应能力。
2.6 AS-1基因组中其他的代谢途径 碳代谢途径:在AS-1基因组中含有与碳代谢以及碳固定相关的基因大约200个。目前已发现的微生物碳固定的途径有6条,分别是卡尔文循环、还原乙酰辅酶A途径、还原柠檬酸途径、3-羟基丙酸双循环、3-羟基丙酸/4-羟基丁酸循环、二羧酸/4-羟基丁酸循环[41-42]。AS-1基因组中含有3-羟基丙酸/4-羟基丁酸循环和还原乙酰辅酶A途径中的一些基因。AS-1基因组中含有3-羟基丙酸/4-羟基丁酸循环中的关键酶基因——乙酰辅酶A羧化酶基因。而且据研究该固碳途径可以存在于严格的厌氧菌株如Stygiolobus azoricus,也可在微厌氧微生物存在[43]。而且在一些Acidanus spp.和Sulfolobus spp.种中也检测到3-羟基丙酸/4-羟基丁酸循环途径中的关键酶基因[43-44]。另外在AS-1基因组中还发现了还原乙酰辅酶A途径中的关键酶基因——一氧化碳脱氢酶和乙酰辅酶A合成酶。因此我们推测菌株AS-1中可能进行自养生长。此外菌株中还有完整的三羧酸循环、戊糖磷酸途径D-核糖利用、脱氧核糖和脱氧核苷酸分解代谢以及乳酸利用等生理代谢途径。
为验证AS-1是否能进行自养生长,我们将AS-1于含Sb(Ⅲ)的MSM培养基中培养,结果发现在培养第11天时OD600达到0.048且菌株AS-1对Sb(Ⅲ)的氧化效率可达到80%左右,对照组无明显生长和氧化现象,这说明菌株AS-1能够在自养条件下缓慢生长并且对Sb(Ⅲ)进行氧化(图 7)。MSM培养基中包含多种的电子供体(如Sb3+、Fe2+、Mn2+等),但是除了Sb3+之外,其他的电子供体在溶液中的浓度非常低,而且实验结果显示,随着实验时间的增加,溶液中的Sb(Ⅲ)浓度减少,Sb(V)浓度显著增加。因此我们推测菌株AS-1通过对亚锑酸盐[Sb(Ⅲ)]的氧化以获取自养生长所需的能源,促进其在自养条件下的生长。
 |
| 图 7 自养条件下菌株Bosea sp. AS-1对Sb(Ⅲ)的氧化 Figure 7 The Sb(Ⅲ) oxidation curve of Bosea sp. AS-1 under autotrophic conditions. The standard deviation of the mean value and the measured value indicates the error bar. |
| 图选项 |
硫代谢途径:基因组分析表明菌株AS-1中含有完整的sox基因簇和硫代谢相关的一些基因,sox基因簇由7个基因soxXYZABCD组成,编码将硫化物氧化成硫酸盐所需的蛋白质[44]。在与AS-1同属的菌株Bosea sp. WAO、B. thiooxidans CGMCC 9174 V5_1、Bosea sp. 117、Bosea sp. LC85和B. lupini中也都发现了完整的sox系统。经过对菌株AS-1的基因组中sox系统分析发现soxX、soxA基因位于染色体的负链,soxY、soxB、soxC、soxD基因位于AS-1染色体正链上。对比Bosea属中其他菌株基因组中的sox系统分析发现,菌株Bosea sp. 117和Bosea sp. LC85中sox系统的基因在其染色体上的分布与AS-1相同,而菌株Bosea sp. WAO和B. lupini中的sox系统染色体的分布与AS-1相反,soxX、soxA基因位于染色体的正链,soxY、soxB、soxC、soxD基因位于染色体负链上[37]。通过比较菌株AS-1和菌株Bosea sp. 117、Bosea sp. LC85以及Bosea sp. WAO中的关键硫氧化酶基因soxB的氨基酸序列发现,AS-1的soxB基因和菌株Bosea sp. WAO最相似,相似度为85.66%,前人研究发现菌株Bosea sp. WAO在自养生长条件下能够将硫化物氧化为硫酸盐。由于AS-1分离于锑矿尾矿中,并且其基因组中具有完整的sox系统,由此我们推测菌株AS-1可能具有能够将硫化物氧化成硫酸盐的能力。
菌株AS-1基因组中除了硫代谢相关的基因外,还具有和氮代谢相关的基因,主要包括编码硝酸盐还原、亚硝酸盐还原和一氧化氮还原酶基因:napEFABC、nirK以及norCBQD。而且据研究Nap系统对于菌株的好氧反硝化作用极为重要[45]。
2.7 鞭毛基因分析 菌株AS-1具有鞭毛[36]。通过对菌株AS-1中的鞭毛基因进行分析发现,AS-1总共有35个和鞭毛相关的基因,鞭毛M蛋白编码基因fliF,鞭毛运动旋转蛋白编码基因motA、motB,参与鞭毛和细菌运动调节的基因qseBC、fihA、flhB、flhCD、fihB2、flgABCDEFGHIK、fliEFGHILMNPQR、noeJ和kdsA等基因。Jérémy等对砷氧化菌株Rhizobium sp. NT-26研究发现,其对亚砷酸盐的氧化会影响鞭毛形成与菌株运动,在亚砷酸盐存在的情况下NT-26基因组中和鞭毛相关的基因表达上调[46],而当把NT-26的aioA和aioR基因突变后,菌株的运动能力下降,作者认为这可能是由于aioA和aioR的缺失使得菌株无法代谢亚砷酸盐,从而导致了细菌能量的减少[46-47]。另外,在菌株NT-26中发现编码甘露糖-1-磷酸鸟苷基转移酶基因noeJ和编码2-脱氢-3-脱氧磷辛酸醛缩酶基因(kdsA)在有As(Ⅲ)的条件下会诱导菌株胞外多糖基质合成基因的表达,这说明As(Ⅲ)与菌株NT-26的鞭毛合成(即运动型)和生物膜的形成有关系[46, 48-49]。另外有研究报道As(Ⅲ)存在的条件下,基因qseBC通过群体感应机制参与鞭毛和细菌运动的调节,通过qseB与fihDC操纵子上的启动子直接结合,从而增强qseBC诱导菌株鞭毛基因发生转录[50]。菌株AS-1中的这些鞭毛相关基因可能发挥类似的功能,在As(Ⅲ)存在的情况下,使得菌株AS-1的耐受性与运动性增强,从而在含有高浓度的As(Ⅲ)的环境中更好地生存。
3 结论 本文通过纯化分离砷氧化菌株,利用16S rRNA基因测序进行菌种鉴定,发现采自湖南省锡矿山锑矿区不同样品中的砷氧化菌株主要分布在α-、β-、γ-变形菌纲以及厚壁菌门。通过全基因组测序的方法,获得了菌株Bosea sp. AS-1全基因组序列。通过对菌株AS-1基因组进一步分析我们发现,AS-1基因组中含有砷锑氧化以及和砷锑抗性相关的基因和重金属抗性基因,这与AS-1能高效氧化砷锑、耐受砷锑和多种重金属的特性相符;菌株AS-1基因组中含有一些碳固定基因以及完整的sox系统;暗示菌株AS-1可能具有自养生长和将硫化物氧化为硫酸盐的能力,进一步实验证明菌株AS-1能够在自养条件下生长。此外,AS-1中含有的众多鞭毛相关基因,可能帮助菌株提高对砷的耐受性及增强运动性,从而在高砷环境中更好地生存。
References
| [1] | Ali H, Khan E, Sajad MA. Phytoremediation of heavy metals-concepts and applications. Chemosphere, 2013, 91: 869-881. DOI:10.1016/j.chemosphere.2013.01.075 |
| [2] | Feng RW, Wei CY, Tu SX, Ding YZ, Wang RG, Guo JK. The uptake and detoxification of antimony by plants:a review. Environmental and Experimental Botany, 2013, 96: 28-34. DOI:10.1016/j.envexpbot.2013.08.006 |
| [3] | Filella M, Belzile N, Chen YW. Antimony in the environment:a review focused on natural waters:I. Occurrence. Earth-Science Reviews, 2002, 57(1/2): 125-176. |
| [4] | Wang N, Lu XL, Wu MXJ, Wang M, Wang HM. Progress in microbial oxidation of As(Ⅲ) and Sb(Ⅲ). Microbiology China, 2017, 44(3): 689-700. (in Chinese) 王年, 鲁小璐, 邬梦晓俊, 王猛, 王红梅. 微生物氧化As(Ⅲ)和Sb(Ⅲ)的研究进展. 微生物学通报, 2017, 44(3): 689-700. |
| [5] | Reimann C, Matschullat J, Birke M, Salminen R. Antimony in the environment:lessons from geochemical mapping. Applied Geochemistry, 2010, 25(2): 175-198. |
| [6] | Oremland RS, Stolz JF, Hollibaugh JT. The microbial arsenic cycle in Mono Lake, California. FEMS Microbiology Ecology, 2004, 48(1): 15-27. |
| [7] | Quéméneur M, Heinrich-Salmeron A, Muller D, Lièvremont D, Jauzein M, Bertin PN, Garrido F, Joulian C. Diversity surveys and evolutionary relationships of aoxB genes in aerobic arsenite-oxidizing bacteria. Applied and Environmental Microbiology, 2008, 74(14): 4567-4573. DOI:10.1128/AEM.02851-07 |
| [8] | Fan H, Su C, Wang Y, Yao J, Zhao K, Wang Y, Wang G. Sedimentary arsenite-oxidizing and arsenate-reducing bacteria associated with high arsenic groundwater from Shanyin, Northwestern China. Journal of Applied Microbiology, 2008, 105(2): 529-539. DOI:10.1111/j.1365-2672.2008.03790.x |
| [9] | Wysocki R, Chéry CC, Wawrzycka D, Van-Hulle M, Cornelis R, Thevelein JM, Tamás MJ. The glycerol channel Fps1p mediates the uptake of arsenite and antimonite in Saccharomyces cerevisiae. Molecular Microbiology, 2001, 40(6): 1391-1401. DOI:10.1046/j.1365-2958.2001.02485.x |
| [10] | Martin P, DeMel S, Shi J, Gladysheva T, Gatti DL, Rosen BP, Edwards BFP. Insights into the structure, solvation, and mechanism of ArsC arsenate reductase, a novel arsenic detoxification enzyme. Structure, 2001, 9(11): 1071-1081. DOI:10.1016/S0969-2126(01)00672-4 |
| [11] | Nakajima T, Hayashi K, Nagatomi R, Matsubara K, Moore JE, Millar BC, Matsuda M. Molecular identification of an arsenic four-gene operon in Campylobacter lari. Folia Microbiologica, 2013, 58(3): 253-260. DOI:10.1007/s12223-012-0207-5 |
| [12] | Kashyap DR, Botero LM, Franck WL, Hassett DJ, McDermott TR. Complex regulation of arsenite oxidation in Agrobacterium tumefaciens. Journal of Bacteriology, 2006, 188(3): 1081-1088. DOI:10.1128/JB.188.3.1081-1088.2006 |
| [13] | Muller D, Médigue C, Koechler S, Barbe V, Barakat M, Talla E, Bonnefoy V, Krin E, Arsène-Ploetze F, Carapito C, Chandler M, Cournoyer B, Cruveiller S, Dossat C, Duval S, Heymann M, Leize E, Lieutaud A, Lièvremont D, Makita Y, Mangenot S, Nitschke W, Ortet P, Perdrial N, Schoepp B, Siguier P, Simeonova DD, Rouy Z, Segurens B, Turlin E, Vallenet D, van Dorsselaer A, Weiss S, Weissenbach J, Lett MC, Danchin A, Bertin PN. A tale of two oxidation states:bacterial colonization of arsenic-rich environments. PLoS Genetics, 2007, 3(4): e53. DOI:10.1371/journal.pgen.0030053 |
| [14] | Achour AR, Bauda P, Billard P. Diversity of arsenite transporter genes from arsenic-resistant soil bacteria. Research in Microbiology, 2007, 158(2): 128-137. DOI:10.1016/j.resmic.2006.11.006 |
| [15] | Chauhan NS, Ranjan R, Purohit HJ, Kalia VC, Sharma R. Identification of genes conferring arsenic resistance to Escherichia coli from an effluent treatment plant sludge metagenomic library. FEMS Microbiology Ecology, 2009, 67(1): 130-139. |
| [16] | Lialikova NN. Stibiobacter senarmontii-a new microorganism oxidizing antimony. Mikrobiologiia, 1974, 43(6): 941-943. |
| [17] | Terry LR, Kulp TR, Wiatrowski H, Miller LG, Oremland SR. Microbiological oxidation of antimony(Ⅲ) with oxygen or nitrate by bacteria isolated from contaminated mine sediments. Applied and Environmental Microbiology, 2015, 81(24): 8478-8488. DOI:10.1128/AEM.01970-15 |
| [18] | Gourbal B, Sonuc N, Bhattacharjee H, Legare D, Sundar S, Ouellette M, Rosen BP, Mukhopadhyay R. Drug uptake and modulation of drug resistance in Leishmania by an aquaglyceroporin. The Journal of Biological Chemistry, 2004, 279(30): 31010-31017. DOI:10.1074/jbc.M403959200 |
| [19] | Meng YL, Liu ZJ, Rosen BP. As(Ⅲ) and Sb(Ⅲ) uptake by GlpF and efflux by ArsB in Escherichia coli. The Journal of Biological Chemistry, 2004, 279(18): 18334-18341. DOI:10.1074/jbc.M400037200 |
| [20] | Lehr CR, Kashyap DR, McDermott TR. New insights into microbial oxidation of antimony and arsenic. Applied and Environmental Microbiology, 2007, 73(7): 2386-2389. DOI:10.1128/AEM.02789-06 |
| [21] | Li JX, Wang Q, Li MS, Yang BR, Shi MM, Guo W, McDermott TR, Rensing C, Wang GJ. Proteomics and genetics for identification of a bacterial antimonite oxidase in Agrobacterium tumefaciens. Environmental Science & Technology, 2015, 49(10): 5980-5989. |
| [22] | Li JX, Yang BR, Shi MM, Yuan K, Guo W, Wang Q, Wang GJ. Abiotic and biotic factors responsible for antimonite oxidation in Agrobacterium tumefaciens GW4. Scientific Reports, 2017, 7: 43225. DOI:10.1038/srep43225 |
| [23] | Li JX, Qiao ZX, Shi MM, Zhang YX, Wang GJ. Regulation of antimonite oxidation and resistance by the phosphate regulator PhoB in Agrobacterium tumefaciens GW4. Microbiological Research, 2019, 226: 10-18. DOI:10.1016/j.micres.2019.04.008 |
| [24] | Zhou JW, Nyirenda MT, Xie LN, Li Y, Zhou BL, Zhu Y, Liu HL. Mine waste acidic potential and distribution of antimony and arsenic in waters of the Xikuangshan mine, China. Applied Geochemistry, 2017, 77: 52-61. DOI:10.1016/j.apgeochem.2016.04.010 |
| [25] | Weeger W, Lièvremont D, Perret M, Lagarde F, Hubert JC, Leroy M, Lett MC. Oxidation of arsenite to arsenate by a bacterium isolated from an aquatic environment. Biometals, 1999, 12(2): 141-149. DOI:10.1023/A:1009255012328 |
| [26] | Rhine ED, Onesios KM, Serfes ME, Reinfelder JR, Young LY. Arsenic transformation and mobilization from minerals by the arsenite oxidizing strain WAO. Environmental Science & Technology, 2008, 42(5): 1423-1429. |
| [27] | Salmassi TM, Venkateswaren K, Satomi M, Newman DK, Hering JG. Oxidation of arsenite by Agrobacterium albertimagni, AOL15, sp. nov., isolated from Hot Creek, California. Geomicrobiology Journal, 2002, 19(1): 53-66. DOI:10.1080/014904502317246165 |
| [28] | Yamamura S, Amachi S. Microbiology of inorganic arsenic:from metabolism to bioremediation. Journal of Bioscience and Bioengineering, 2014, 118(1): 1-9. DOI:10.1016/j.jbiosc.2013.12.011 |
| [29] | Liao VHC, Chu YJ, Su YC, Hsiao SY, Wei CC, Liu C, Liu CW, Liao CM, Shen WC, Chang FJ. Arsenite-oxidizing and arsenate-reducing bacteria associated with arsenic-rich groundwater in Taiwan. Journal of Contaminant Hydrology, 2011, 123(1/2): 20-29. |
| [30] | Bhakat K, Chakraborty A, Islam E. Characterization of arsenic oxidation and uranium bioremediation potential of arsenic resistant bacteria isolated from uranium ore. Environmental Science and Pollution Research, 2019, 26(13): 12907-12919. DOI:10.1007/s11356-019-04827-6 |
| [31] | Lugtu RT, Choi SC, Oh YS. Arsenite oxidation by a facultative chemolithotrophic bacterium SDB1 isolated from mine tailing. The Journal of Microbiology, 2009, 47(6): 686-692. DOI:10.1007/s12275-009-0279-3 |
| [32] | Wang Q, Qin D, Zhang SZ, Wang L, Li JX, Rensing C, McDermott TR, Wang GJ. Fate of arsenate following arsenite oxidation in Agrobacterium tumefaciens GW4. Environmental Microbiology, 2015, 17(6): 1926-1940. DOI:10.1111/1462-2920.12465 |
| [33] | Cai L, Rensing C, Li XY, Wang GJ. Novel gene clusters involved in arsenite oxidation and resistance in two arsenite oxidizers:Achromobacter sp. SY8 and Pseudomonas sp. TS44. Applied Microbiology and Biotechnology, 2009, 83(4): 715-725. DOI:10.1007/s00253-009-1929-4 |
| [34] | Branco R, Chung AP, Morais PV. Sequencing and expression of two arsenic resistance operons with different functions in the highly arsenic-resistant strain Ochrobactrum tritici SCII24T. BMC Microbiology, 2008, 8: 95. DOI:10.1186/1471-2180-8-95 |
| [35] | Bagade A, Nandre VS, Ghosh S, Battu S, Haram S, Giri A, Kodam K. Rapid and efficient sequestration of arsenic from contaminated water using hypertolerant Bacillus L-148 sp.:a two-step process. Green Chemistry, 2019, 21(9): 2245-2251. DOI:10.1039/C9GC00878K |
| [36] | Lu XL, Zhang YN, Liu CY, Wu MXJ, Wang HM. Characterization of the antimonite-and arsenite-oxidizing bacterium Bosea sp. AS-1 and its potential application in arsenic removal. Journal of Hazardous Materials, 2018, 359: 527-534. DOI:10.1016/j.jhazmat.2018.07.112 |
| [37] | Walczak AB, Yee N, Young LY. Draft genome sequence of Bosea sp. WAO an arsenite and sulfide oxidizer isolated from a pyrite rock outcrop in New Jersey. Standards in Genomic Sciences, 2018, 13: 6. DOI:10.1186/s40793-018-0312-4 |
| [38] | Shi KX, Wang Q, Fan X, Wang GJ. Proteomics and genetic analyses reveal the effects of arsenite oxidation on metabolic pathways and the roles of AioR in Agrobacterium tumefaciens GW4. Environmental Pollution, 2018, 235: 700-709. DOI:10.1016/j.envpol.2018.01.006 |
| [39] | Stolz JF, Oremland RS. Microbial metal and metalloid metabolism: advances and applications. Washington: ASM, 2011. |
| [40] | H?rtig C, Lohmayer R, Kolb S, Horn MA, Inskeep WP, Planer-Friedrich B. Chemolithotrophic growth of the aerobic hyperthermophilic bacterium Thermocrinis ruber OC 14/7/2 on monothioarsenate and arsenite. FEMS Microbiology Ecology, 2014, 90(3): 747-760. |
| [41] | Yuan HZ, Qin HL, Liu SL, Nie SA, Wei WX, Wu JS. Advances in research of molecular ecology of carbon fixation microorganism. Scientia Agricultura Sinica, 2011, 44(14): 2951-2958. (in Chinese) 袁红朝, 秦红灵, 刘守龙, 聂三安, 魏文学, 吴金水. 固碳微生物分子生态学研究. 中国农业科学, 2011, 44(14): 2951-2958. |
| [42] | Berg IA, Ramos-Vera WH, Petri A, Huber H, Fuchs G. Study of the distribution of autotrophic CO2 fixation cycles in Crenarchaeota. Microbiology, 2010, 156(1): 256-269. |
| [43] | Ishii M, Miyake T, Satoh T, Sugiyama H, Oshima Y, Kodama T, Igarashi Y. Autotrophic carbon dioxide fixation in Acidianus brierleyi. Archives of Microbiology, 1996, 166(6): 368-371. DOI:10.1007/BF01682981 |
| [44] | Friedrich CG, Rother D, Bardischewsky F, Quentmeier A, Fischer J. Oxidation of reduced inorganic sulfur compounds by bacteria:emergence of a common mechanism?. Applied and Environmental Microbiology, 2001, 67(7): 2873-2882. DOI:10.1128/AEM.67.7.2873-2882.2001 |
| [45] | Bedmar EJ, Robles EF, Delgado MJ. The complete denitrification pathway of the symbiotic, nitrogen-fixing bacterium Bradyrhizobium japonicum. Biochemical Society Transactions, 2005, 33(1): 141-144. |
| [46] | Andres J, Arsène-Ploetze F, Barbe V, Brochier-Armanet C, Cleiss-Arnold J, Coppée JY, Dillies MA, Geist L, Joublin A, Koechler S, Lassalle F, Marchal M, Médigue C, Muller D, Nesme X, Plewniak F, Proux C, Ramírez-Bahena MH, Schenowitz C, Sismeiro O, Vallenet D, Santini JM, Bertin PN. Life in an arsenic-containing gold mine:genome and physiology of the autotrophic arsenite-oxidizing bacterium Rhizobium sp. NT-26. Genome Biology and Evolution, 2013, 5(5): 934-953. DOI:10.1093/gbe/evt061 |
| [47] | Santini JM, Sly LI, Schnagl RD, Macy JM. A new chemolithoautotrophic arsenite-oxidizing bacterium isolated from a gold mine:phylogenetic, physiological, and preliminary biochemical studies. Applied and Environmental Microbiology, 2000, 66(1): 92-97. |
| [48] | Muller D, Médigue C, Koechler S, Barbe V, Barakat M, Talla E, Bonnefoy V, Krin E, Arsène-Ploetze F, Carapito C, Chandler M, Cournoyer B, Cruveiller S, Dossat C, Duval S, Heymann M, Leize E, Lieutaud A, Lièvremont D, Makita Y, Mangenot S, Nitschke W, Ortet P, Perdrial N, Schoepp B, Siguier P, Simeonova DD, Rouy Z, Segurens B, Turlin E, Vallenet D, van Dorsselaer A, Weiss S, Weissenbach J, Lett MC, Danchin A, Bertin PN. A tale of two oxidation states:bacterial colonization of arsenic-rich environments. PLoS Genetics, 2007, 3(4): e53. DOI:10.1371/journal.pgen.0030053 |
| [49] | Marchal M, Briandet R, Koechler S, Kammerer B, Bertin PN. Effect of arsenite on swimming motility delays surface colonization in Herminiimonas arsenicoxydans. Microbiology, 2010, 156(8): 2336-2342. DOI:10.1099/mic.0.039313-0 |
| [50] | Clarke MB, Sperandio V. Transcriptional regulation of flhDC by QseBC and σ?28 (FliA) in enterohaemorrhagic Escherichia coli. Molecular Microbiology, 2005, 57(6): 1734-1749. DOI:10.1111/j.1365-2958.2005.04792.x |
