蒲语涵1,2, 刘欢2, 陈国参3, 郭莉霞1

 , 尹文兵2
, 尹文兵2

1. 重庆工商大学, 天然药物研究重庆高校市级重点实验室, 重庆 400067;
2. 中国科学院微生物研究所真菌学国家重点实验室, 北京 100101;
3. 河南省科学院生物研究所, 河南省微生物工程重点实验室, 河南 郑州 450008
收稿日期:2019-12-30;修回日期:2020-03-02;网络出版日期:2020-06-18
基金项目:国家重点研发计划(2018YFA0901901);国家自然科学基金(21778075)
*通信作者:郭莉霞, Tel/Fax:+86-10-64806170;E-mail:80074241@qq.com;
尹文兵, Tel/Fax:+86-10-64806170;E-mail:yinwb@im.ac.cn.
摘要:[目的] 木霉属真菌是应用最为广泛和潜力最大的生防真菌,其产生的典型化合物哌珀霉素(peptaibols)类抗生素在生物防治中发挥重要作用。本研究采用基因组挖掘技术(genome mining)发现炭团木霉(Trichoderma hypoxylon)的潜在哌珀霉素生物合成基因簇及对病原菌的防治作用。[方法] 生物信息学分析预测合成哌珀霉素的基因簇,利用Quick-change技术构建基因骨架敲除盒,通过PEG介导的原生质体转化方法获得敲除突变株,通过平板对峙法和菌丝生长毒力实验验证该基因簇对炭团木霉生物活性的影响。[结果] 基因挖掘鉴定一个非核糖体多肽合成酶(nonribosomal peptide synthetases,NRPS)可能合成哌珀霉素类抗生素,命名为NRPS1,对该基因进行部分敲除,成功获得3株NRPS1缺失突变株。对峙实验表明,突变株对寄生曲霉(Aspergillus parasiticus)、尖孢镰刀菌(Fusarium oxysporum)、黑白轮枝菌(Verticillium alboatrum)等9株植物病原真菌的抑制作用与野生株相比显著下降,且突变株的粗提物的抑菌活性明显弱于野生型。[结论] NRPS1是一个潜在的哌珀霉素合成基因,该基因在宿主与病原真菌对抗过程中起关键作用,该研究为炭团木霉哌珀霉素结构解析及生物防治机理研究奠定了基础。
关键词:炭团木霉次级代谢产物哌珀霉素生物防治
Identification and functional study of NRPS1 in Trichoderma hypoxylon
Yuhan Pu1,2, Huan Liu2, Guocan Chen3, Lixia Guo1

 , Wenbing Yin2
, Wenbing Yin2

1. Key Laboratory of Natural Medicine Research of Chongqing Education Commission, Chongqing Technology and Business University, Chongqing 400067, China;
2. State Key Laboratory of Mycology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. The Key Laboratory of Microbial Engineering of Henan Province, Institute of Biology, Henan Academy of Sciences Co. Ltd, Zhengzhou 450008, Henan Province, China
Received: 30 December 2019; Revised: 2 March 2020; Published online: 18 June 2020
*Corresponding author: Lixia Guo, Tel/Fax: +86-10-64806170; E-mail:80074241@qq.com;
Wenbing Yin, Tel/Fax: +86-10-64806170; E-mail:yinwb@im.ac.cn.
Foundation item: Supported by the National Key Research and Development Program (2018YFA0901901) and by the National Natural Science Foundation of China (21778075)
Abstract: [Objective] Trichoderma is a genus of common filamentous fungi used for biological control, by producing a variety of secondary metabolites that play an important role in biological control. In this study, a putative nonribosomal peptide synthetase (NRPS) NRPS1 was identified in Trichoderma hypoxylon by genome mining approach and the function of this gene's function was studied by using NRPS1 mutant against the pathogens. [Methods] Bioinformatics analysis reveals a putative NRPS containing biosynthetic gene cluster that is potential for the biosynthesis of peptaibol. The deletion cassette containing the hygromycin resistance gene was constructed by Quick-change method. The partial gene was deleted by using PEG mediated transformation in T. hypoxylon. NRPS1's function was identified by the biological activity assay against pathogenic fungi. [Results] We identified a putative peptaibol synthesis gene NRPS1 and obtained three deletion mutants. Deletion of NRPS1 significantly decreased the ability to resistance the pathogenic fungi, such as Aspergillus parasiticus, Fusarium oxysporum, and Verticillium alboatrum. [Conclusion] NRPS1 is a potential peptaibol synthesis gene in T. hypoxylon and plays important roles in the resistance to pathogenic fungi. This work lays the foundation for the elucidation of peptaibol biosynthesis and its application for the biological control in agriculture.
Keywords: Trichoderma hypoxylonsecondary metabolitespeptaibolsbiological control
木霉属(Trichoderma spp.)真菌,属于半知菌亚门,丛梗孢目,广泛存在于土壤、腐木、植物根系等[1]。作为最重要的有益真菌之一,早在19世纪20年代就被作为生防制剂,用于对抗植物病原菌[2-3]。其生物防治的机制复杂且多元化,其中主要机制之一是通过产生大量有活性的次级代谢产物来发挥生防作用[4-5]。目前从木霉属中发现的次级代谢产物多达180种,包括肽类(peptides)、聚酮类(polyketides)、萜烯类(terpenes)、吡喃酮类(pyrones)、铁载体(siderophores)和非挥发性萜(nonvolatile terpenes)等[6-8]。这些次级代谢产物具有多种生物活性如抗真菌、抗细菌、促进植物生长活性等[9-11]。例如,Lynch等发现哈茨木霉(T. harzianum)能够产生易挥发的抗生素六戊烷基吡喃及戊烯基吡喃,对立枯丝核菌(Rhizoctonia solani)有良好抑制活性[12];Meyer等在绿色木霉(T. viride)中发现一个由20个氨基酸残基构成的哌珀霉素类(peptaibols)化合物——丙甲菌素(alamethicin) (图 1),对革兰氏阳性菌有抑制作用[13]。
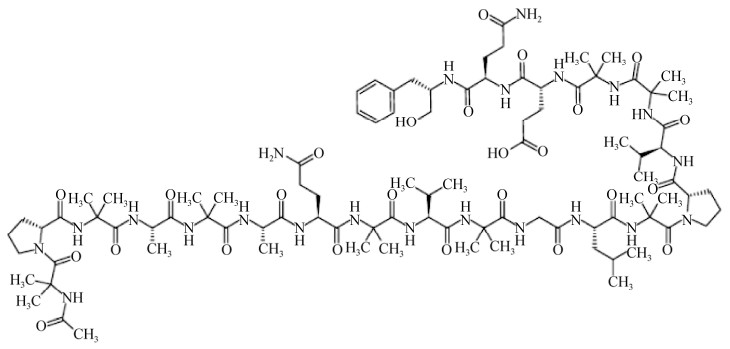 |
| 图 1 哌珀霉素类化合物丙甲菌素的结构 Figure 1 The structure of alamethicin. |
| 图选项 |
哌珀霉素是一类主要由木霉属真菌产生的典型活性化合物,其化学结构具有典型的特征,即N端富含非天然氨基酸α-氨基异丁酸(α-aminoisobutyric acid,Aib),C末端会常常被羟基化成氨基醇(alcohol)的肽(peptide)类抗生素[14-16]。哌珀霉素的主要长度在5–20个氨基酸之间,多数含有15–20个氨基酸残基[17-18]。迄今为止,已鉴定的哌珀霉素类化合物超过300个[19],随着越来越多的哌珀霉素被发现,这类特殊化合物的生物合成和功能研究也逐渐成为焦点。2002年,Wiest等从绿木霉(T. virens)中鉴定并克隆了第一个哌珀霉素合成酶基因tex1,该基因由18个完整的最小模块(A-T-C,adenylation-thiolation-condensation,腺苷酰化结构域-巯基化结构域-缩合结构域)组成,每个模块序列之间的同源性达到35%–58%[20]。其中腺苷酰化结构域(adenylation,A-domain)的差异决定了对底物的选择性不同,进而导致化合物结构多样性,也决定了该类化合物对革兰氏阳性菌、阴性菌、真菌、螺原体和病毒等活性的不同[13-14]。哌珀霉素抗菌的作用机制是复杂的,目前的研究主要集中于其穿膜时三维构象的差异,其作用机制主要有“桶-板”模型(barrel-stave model)、“毯式”模型(carpet model)、“虫孔”模型(toroidal pore model)等。最广为接受的机制是Ehrenstein等在1977年提出的“桶-板”模型,认为哌珀霉素以多聚体的形式插入细胞膜双分子层,形成跨膜的离子通道,破坏细胞膜的完整性,干扰细胞正常的代谢,从而导致细胞的死亡而达到杀菌的目的[15, 21]。哌珀霉素区别于其他抗生素的作用机制使其具有广谱的抗微生物活性,且不易使病原微生物产生抗药性。
本研究中使用的炭团木霉菌株是2016年从Hypoxylon anthochroum中分离得到的[6],在前期的工作中已建立了该菌高效的遗传操作体系[22]。鉴于其丰富的次级代谢产物,对其生物学功能的研究具有重要理论意义。通过基因挖掘策略,对炭团木霉基因组进行生物信息学分析,发现了一个含有19个A-T-C模块的NRPS1,且该序列在不同木霉中均保守。通过与已知哌珀霉素的生物合成基因进行序列比对,预测NRPS1可能是合成哌珀霉素类化合物的核心基因。通过PEG介导的原生质体转化的方法获得NRPS1缺失株,并研究了炭团木霉野生型与突变株的活性差异。证明了该基因在炭团木霉生防过程中发挥重要作用,为该菌的后续研究提供了方向,为其生防应用奠定了基础。
1 材料和方法 1.1 供试菌株 本研究中所用的炭团木霉、炭团菌(Hypoxylon sp.)和植物病原菌均由中国科学院微生物研究所真菌学国家重点实验室保藏。供试病原菌:寄生曲霉Aspergillus parasiticus (CGMCC3.0124)、赤霉菌Gibberella zeae (CGMCC3.2873)、黑白轮枝菌Verticillium alboatrum (CGMCC3.4306)、灰霉菌Botrytis cinerea (CGMCC3.4584)、雪腐镰刀菌Fusarium nivale (CGMCC3.4600)、尖孢镰刀菌Fusarium oxysporum (CGMCC3.2830)、Pochonia chlamlosporia(CGMCC3.0829)、Cladosporium cladosporioides和Mucor corcinelloides。
1.2 培养基及试剂 PDA培养基:39 g/L Potato Dextrose Agar,高压灭菌,用于菌株活化和对峙培养。
PDB培养基:24 g/L Potato Dextrose Broth,高压灭菌,用于菌丝培养和基因组的提取。
TG培养基:10 g/L Tryptone,100 g/L Glucose,高压灭菌,用于菌丝培养。
SPDA培养基:218.6 g/L D-sorbitol,39 g/L Potato Dextrose Agar,高压灭菌,用于遗传转化。
TOP SPDA培养基:218.6 g/L D-sorbitol,24 g/L Potato Dextrose Broth,8 g/L Agar,高压灭菌,用于遗传转化。
大米培养基:600 g/L大米,高压灭菌,用于菌株发酵提取次级代谢产物。
N-M溶液:17.532 g/L NaCl,73.941 g/L MgSO4·7H2O,10 mmol/L Tris-HCl (pH=7.5),高压灭菌,用于酶解原生质体。
STC缓冲液:218.6 g/L D-sorbitol,1.47 g/L CaCl2·2H2O,10 mmol/L Tris-HCl (pH=7.5),高压灭菌,用于遗传转化。
PEG溶液:600 g/L聚乙二醇6000,7.35 g/L CaCl2·2H2O,10 mmol/L Tris-HCl (pH=7.5),高压灭菌,用于遗传转化。
1.3 引物和质粒 本研究所用引物和质粒见表 1及表 2。
表 1. 本研究中使用的引物 Table 1. PCR primer sets used in this study
| Primers | Sequences (5′→3′) | Uses |
| NRPS1 5f-for (P1) | CTATAGGGCGAATTGGAGCTCCACCGCGCTGGCTACCGCGTTGATAG | Up flanks’ amplification |
| NRPS1 5f-rev (P2) | GATCCACTAGTTCTAGAGCGGCCGCCACCCTGAACTGCGGCAGCCATC | |
| NRPS1 3f-for (P3) | GAGCCGGAAGCATAAAGTGTAAAGCCTGCGTGCTGCTAACCGACTAGC | Down flanks’ amplification |
| NRPS1 3f-rev (P4) | GTGAGTTAGCTCACTCATTAGGCACCCCTGCAGGCCTTGCGCCATTG | |
| NRPS1 5f scr-for (P5) | CATTTATCTCTGACGCGGCG | Transformant up screening |
| HYG5f scr-rev (P6) | GCCTATGCCTACAGCATCC | |
| HYG3f scr-for (P7) | CGTGGTCGAGCTACAAAGC | Transformant down screening |
| NRPS1 3f scr-rev (P8) | GCTCAGCAGTCATTGACAC | |
| NRPS1RT-for (P9) | CGAGCCTCTAGTGCAGGAAG | Transformant target screening |
| NRPS1RT-rev (P10) | GATGGCCGAAGTCTGTGCAG |
表选项
表 2. 本研究中使用的质粒 Table 2. Plasmids used in this study
| Plasmid | Description |
| pUCH2-8 | Hygromycin B[23] |
| pYHL12 | NRPS1 (1-4526 bp) deletion cassette in pUCH2-8 |
表选项
1.4 生物信息学分析 在NCBI数据库中获得哌珀霉素合成基因的蛋白序列,用已报道的绿木霉(T. virens GV29-8)哌珀霉素合成骨架基因tex1(XP_013953110.1)为探针,对炭团木霉基因组进行本地比对分析,确认其基因组中的哌珀霉素合成基因。并通过antiSMASH (antibiotics & secondary metabolite analysis shell)软件对该基因所在的基因簇进行在线分析[24]。
1.5 NRPS1骨架基因敲除盒的构建 炭团木霉哌珀霉素核心基因NRPS1共含有66642个碱基对(base pair,bp),采用敲除完整基因策略较困难,因此,本实验目标性地对NRPS1基因5′端4526 bp进行敲除。敲除载体的构建采用Quick-change方法[25],引物、质粒信息如表 1表 2所示,其中P1–4引物为长引物,设计时将目的基因两侧分别加上和质粒插入位点两侧同源序列。首先,以炭团木霉基因组DNA为模板,用引物P1/P2、P3/P4分别扩增得到1197 bp上游同源臂和1201 bp下游同源臂;其次,利用PCR将模板质粒pUCH2-8与目的基因片段连接,然后使用Dpn I内切酶消化甲基化的模板质粒;最后,将酶切产物进行大肠杆菌转化,筛选验证即得到阳性质粒。
1.6 炭团木霉NRPS1缺失菌株构建 炭团木霉的转化采用PEG介导的原生质体法,原生质体的制备、抗生素浓度与筛选策略均参考实验室发表的方法[22]。原生质体的制备方法如下:首先,在玻璃纸上接种活化后的炭团木霉菌株,生长3 d后经过无菌破碎转移至PDB培养基,于28 ℃、200 r/min培养24 h;其次,取菌体再经过无菌破碎转移至TG培养基,于28 ℃、200 r/min培养12 h;最后,过滤收集菌丝转移至10 mL的酶解液(20 mg/mL lysing enzymes,Sigma),于28 ℃、100 r/min酶解8 h,收集原生质体,溶于适量STC溶液中。突变株的筛选策略如下:本实验采用的抗生素为潮霉素,转化中SPDA培养基的抗性浓度为60 μg/mL,TOP SPDA培养基的抗性浓度为30 μg/mL。转化的平板于26 ℃生长5–7 d后,将转化子转接至含60 μg/mL潮霉素的PDA平板上,重复转接操作使其传代2次,再挑取生长状态良好的菌落接种至PDB培养基,培养24 h后提取基因组DNA经3对筛选引物(P5/P6、P7/P8和P9/P10)进行PCR验证(表 1),从而获得NRPS1基因完整性被破坏的正确突变株。
1.7 炭团木霉次级代谢产物的提取与活性测定 将活化后的炭团木霉野生型(wild type,WT)和NRPS1缺失株(TYHL3)接种于大米培养基中,于26 ℃静置培养10 d。利用乙酸乙酯萃取,旋转蒸发仪去除有机试剂,再用1 mL的甲醇溶解样品,0.22 μm微孔滤膜过滤后,溶解于二甲亚砜(DMSO),配制为100 mg/mL母液备用。按照20 μg/mL的浓度将粗提物加到10 mL PDA培养基中,以加入等量DMSO的PDA为空白对照,于第7天测量菌落直径来表征菌株生长速度。按生长速率法测定WT和TYHL3的粗提物对炭团菌生长的抑制作用,设置3个生物学平行[26]。
1.8 炭团木霉菌株抗真菌活性测定 采用平板对峙法对A. parasiticus等9株病原菌进行炭团木霉抗真菌的活性测定。首先在PDA平板上活化菌株,然后在直径为60 mm的PDA平板两侧固定位置分别接种直径为3 mm活化后的炭团木霉菌饼(WT或TYHL3)与植物病原菌,以单独接种的病原菌为对照,各设3个生物学平行。26 ℃培养7 d,观察菌落形态变化,并测量病原菌菌落直径,按公式(1)计算抑制率。其中D1为对照组菌落直径(mm),D2为木霉对峙组菌落直径(mm)[27-29]。
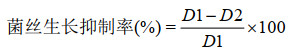 | 公式(1) |
2 结果和分析 2.1 炭团木霉中哌珀霉素生物合成基因簇预测 以绿木霉T. virens GV29-8中哌珀霉素合成骨架基因tex1(XP_013953110.1)蛋白序列为探针,通过本地比对确定了炭团木霉中tex1的同源基因,获得假定的哌珀霉素生物合成基因NRPS1。然后利用antiSMASH预测分析炭团木霉NRPS1基因所在的基因簇[24],结果如图 2-A所示,该簇包括非核糖体肽合酶NRPS1、黄曲霉毒素B1醛还原酶(aflatoxin b1 aldehyde reductase)、MFS转运蛋白(MFS general substrate transporter)、转录调控蛋白(retrograde regulation protein)、细胞色素P450单氧化酶(cytochrome P450 monooxygenase)、UbiA异戊烯基转移酶(UbiA prenyltransferase)等。其骨架基因NRPS1的结构域分析如图 2-B所示,由19个A-T-C模块组成,5′端为KS-AT结构域,3′端为NAD结构域,含有3个内含子和4个外显子,开放阅读框为66156 bp,编码蛋白为22052个氨基酸。且在木霉属中高度保守,与T. virens (XP_013953110.1)、T. parareesei(OTA01063.1)和T. citrinoviride (XP_024748081.1)相似性分别为74%、74%和76%。
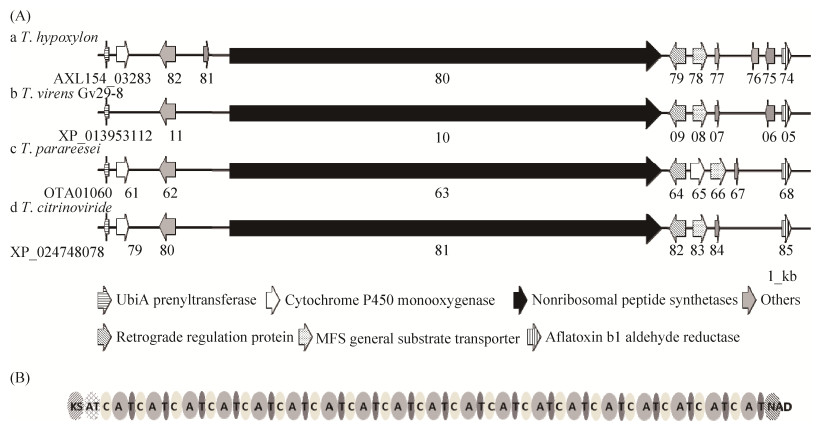 |
| 图 2 炭团木霉中NRPS1基因簇生物信息学分析 Figure 2 The NRPS1 gene cluster is conserved in Trichoderma species. A: Orthologous gene analysis of the NRPS1 gene cluster. a: T. hypoxylon NRPS1; b: T. virens GV29-8 XP_013953110.1 (tex1); c: T. parareesei OTA01063.1; d: T. citrinoviride XP_024748081.1. B: The domain structure of NRPS1. |
| 图选项 |
由于NRPS1具有特殊结构与高度保守性,因此,推测NRPS1是炭团木霉哌珀霉素类化合物的生物合成基因。
2.2 炭团木霉NRPS1缺失菌株的构建及分析 生物信息学分析表明,炭团木霉哌珀霉素生物合成基因NRPS1基因长度为66642 bp。利用Quick-Change技术构建敲除盒,split-marker策略和PEG介导的原生质体转化法对靶基因NRPS1 5′端4526 bp DNA片段进行敲除。验证结果如图 3-A、B所示,NRPS1突变株可以扩增得到片段长度为1517 bp (P5/P6)和1462 bp (P7/P8)的条带,而野生型菌株不能扩增出该条带,与预期结果符合。进而使用NRPS1基因内部引物(P9/P10)扩增814 bp片段,野生型菌株能扩增出目的条带,NRPS1突变株由于目标基因片段被潮霉素抗性基因替换而不能扩增出该条带,说明炭团木霉NRPS1基因5′端4526 bp DNA片段在染色体水平已被定点敲除,NRPS1基因被破坏。通过PCR验证成功获得了3株能稳定遗传的NRPS1基因缺失株,验证结果如图 3-B所示。突变株的形态和生长速度与野生型相比均无明显差异,命名为TYHL3.1、3.2、3.3。
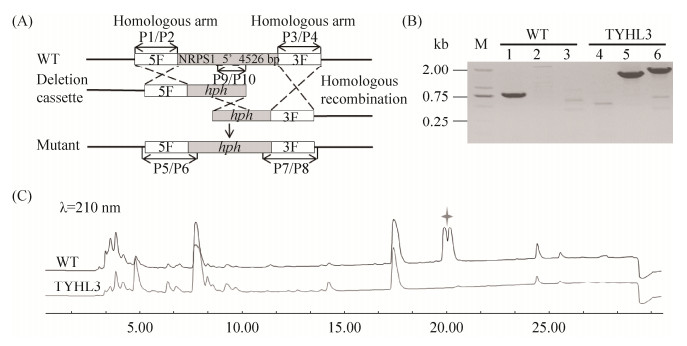 |
| 图 3 炭团木霉NRPS1基因缺失菌株的构建及验证分析 Figure 3 Schematic illustration of deletion and confirmation of T. hypoxylon NRPS1. A: Schematic representation of the deletion of NRPS1. B: Confirmation of NRPS1 deletion by genomic DNA PCR amplification. TYHL3 with primer pairs 9/10 (lanes 1 and 4), primer pairs 5/6 (lanes 2 and 5) and primer pairs 7/8 (lanes 3 and 6). M: D2000 ladder DNA marker. C: The HPLC analysis the mutant of deletion NRPS1. |
| 图选项 |
将炭团木霉WT和TYHL3在PDA培养基上培养7 d,用乙酸乙酯萃取菌丝及培养基,利用高效液相色谱法(HPLC)分析对次级代谢产物进行分析,分析结果如图 3-C所示。在210 nm波长下,野生型中保留时间为20.2 min的代谢产物峰在NRPS1基因缺失后消失,推测该峰为炭团木霉哌珀霉素类化合物的代谢产物峰,NRPS1基因为哌珀霉素类化合物的生物合成基因。
2.3 NRPS1对炭团木霉次级代谢产物活性的影响 采用炭团木霉的寄生菌株H. anthochroum同属的一株炭团菌作为试验菌株,以不同粗提物作用下的炭团菌的菌丝生长直径来表征菌丝生长速率,进一步探究NRPS1对炭团木霉次级代谢及其相关活性的影响。实验结果显示,在WT与TYHL3的粗提物作用下,炭团菌菌丝生长速度与对照菌株相比显著变慢,20 μg/mL浓度的WT粗提物能够完全抑制炭团菌菌丝的生长,而缺失基因NRPS1后该抑制作用显著下降(图 4-A,B)。因此,NRPS1基因的缺失导致具有抑菌活性的代谢产物减少,从而显著降低了炭团木霉对寄主菌的抑制作用,此结论与之前推测NRPS1是哌珀霉素的生物合成基因功能相符。
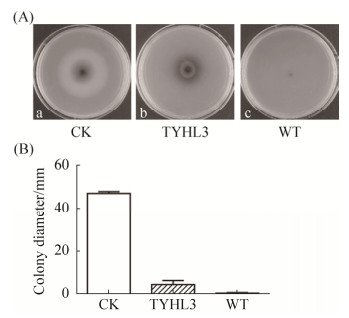 |
| 图 4 NRPS1缺失对炭团木霉活性的影响 Figure 4 Effect of NRPS1 deletion on the activity of T. hypoxylon. A: Mycelia growth of Hypoxylon sp. under crude extract of T. hypoxylon. The Hypoxylon sp. strain was cultured on PDA plate at 26 ℃ for 7 days with DMSO (a), crude extracts (b) of TYHL3 (△NRPS1), crude extracts of WT (c). B: The colony diameters of Hypoxylon sp. were measured. Three replicates are for each culture. Error bars represent the standard deviations. |
| 图选项 |
2.4 NRPS1对炭团木霉抗植物病原菌活性的影响 木霉属真菌是重要的生防真菌,对多种植物病原菌具有良好拮抗作用。因此,本研究对炭团木霉的活性与农业上应用前景作了初步探究。炭团木霉野生型与NRPS1缺失突变株分别与A. parasiticus等9株植物病原真菌进行平板对峙培养,以单独生长的植物病原真菌为对照。如图 5所示,野生型炭团木霉与病原真菌对峙培养时。炭团木霉对8株病原菌的生长均有明显的抑制作用,仅对M. cinelloides (图 5-I)的拮抗作用较弱。其中抑制效果最显著的菌株为P. Chlamlosporia (图 5-A),野生型炭团木霉菌落的生长能够完全包围P. Chlamlosporia的菌落。NRPS1基因缺失株TYHL3与病原真菌对峙培养时,相比于野生型,TYHL3对9株病原菌的生长抑制作用均下降。炭团木霉野生型的抑菌能力强于NRPS1基因缺失株,这说明了NRPS1基因的缺失降低了抑菌活性物质的产生。此结论与之前推测NRPS1是哌珀霉素的生物合成基因功能相符。
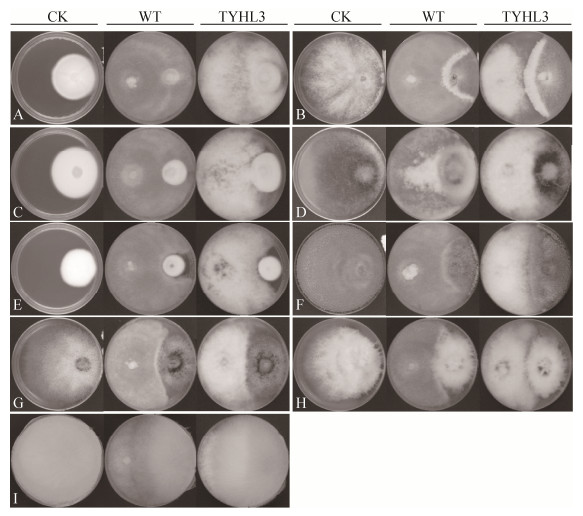 |
| 图 5 NRPS1突变株对炭团木霉抗病原菌活性的影响 Figure 5 Influences of NRPS1 mutant against pathogenic fungi. All strains were growth on PDA at 26 ℃ for 7 days; T. hypoxylon was plated on left side and pathogenic fungus was plated on right side; A: P. chlamlosporia; B: C. cladosporioides; C: A. parasiticus; D: G. zeae; E: F. nivale; F: B. cinerea; G: V. alboatrum; H: F. oxysporum; I: M. cinelloides. |
| 图选项 |
以生长7 d的实验组与对照组菌丝直径来表征菌丝生长抑制率,结果如表 3和图 6所示。其中,菌丝生长抑制率最高的3株病原菌为P. chlamlosporia、C. cladosporioides和A. parasiticus,炭团木霉WT对其抑制率分别为61.52%、58.67%、53.62%,而TYHL3对其抑制率分别仅为43.63%、31.00%、44.20%,这表明WT对病原菌菌丝生长的抑制效果较TYHL3更为显著。其中TYHL3对P. chlamlosporia的抑菌率较WT下降大于20%,其余菌株也下降5%–20%不等,分析可知NRPS1基因的缺失导致炭团木霉对植物病原菌的抑制能力显著下降。因此,NRPS1所合成的哌珀霉素类化合物具有良好的抑菌活性,进一步也说明了NRPS1基因在炭团木霉适应自然环境中与其他菌株竞争、拮抗过程中发挥重要的作用。
表 3. 炭团木霉对植物病原菌的抑制作用 Table 3. Inhibition of T. hypoxylon on plant pathogenic fungi
| No. | Strains | WT (7 d) | TYHL3 (7 d) |
| 1 | P. chlamlosporia | 61.52±0.05 | 43.63±0.03 |
| 2 | C. cladosporioides | 58.67±0.03 | 31.00±0.08 |
| 3 | A. parasiticus | 53.62±0.06 | 44.20±0.04 |
| 4 | G. zeae | 39.06±0.08 | 33.33±0.08 |
| 5 | F. nivale | 44.27±0.08 | 36.86±0.03 |
| 6 | B. cinerea | 44.67±0.11 | 34.67±0.07 |
| 7 | V. alboatrum | 42.38±0.04 | 32.33±0.04 |
| 8 | F. oxysporum | 39.06±0.08 | 33.33±0.08 |
| 9 | M. corcinelloides | 27.67±0.03 | 21.67±0.03 |
| Inhibition of growth is defined as (diameter of growth in the absence of the test strain-diameter of growth in the direction of the test strain)/(diameter of growth in the absence of the test strain)×100%. Data are shown with mean±SEM, n=3. | |||
表选项
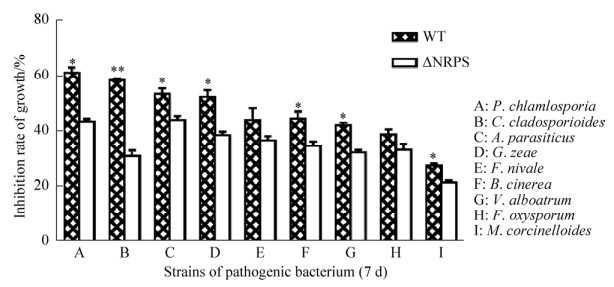 |
| 图 6 炭团木霉(野生型和突变株)对9株病原菌的抑制率 Figure 6 Inhibition rates of T. hypoxylon against pathogenic fungi. Data were presented as Mean±SD, values with capital letters and lowercase letters in the same column indicated significant difference at 0.01 and 0.05 levels, respectively; *: P < 0.05;**: P < 0.01. |
| 图选项 |
3 讨论 基因组测序和生物信息分析技术的迅速发展,表明微生物基因组中蕴藏着大量未知天然产物,是活性分子发现和开发的巨大宝库。但在传统的筛选办法和常规的实验室培养条件下,微生物失去了选择和竞争压力,导致越来越难发现新的活性化合物,目前已被鉴定和分离的化合物仅仅是冰山一角。Wang等完成了无花果拟盘多毛孢(Pestalotiopsis fici)的全基因组测序,生物信息学分析显示,其基因组中共包含76个次级代谢产物合成基因簇,远远大于已鉴定的化合物类型。转录组分析显示,超过86%基因簇未表达,证明该菌株中大量的基因簇仍处于“沉默”状态[31]。因此,从基因组入手的基因组挖掘技术已经成为发现新天然产物的重要策略之一[32]。该技术主要通过对基因组中特定酶及基因簇的保守区序列进行生物信息分析,预测酶的功能和基因簇编码的天然产物的结构,再通过遗传调控、异源表达等方法激活表达量低或沉默的基因与基因簇,从而特异性地发现新酶以寻找结构新颖的化合物[33-34]。例如,Zheng等通过对P. fici中COP9信号小体亚基PfcsnE基因的敲除,从PfcsnE敲除突变株中分离到了结构新颖的化合物,该蛋白对P. fici次级代谢起到重要的调控作用[35]。Udwary等利用基因组挖掘策略首次鉴定了来源于海洋放线菌Salinispora tropica CNB-400中的多烯大环内酰胺类化合物salinilactam A[36]。因此,该策略能够有效避免化合物的重复发现,有助于发现新化合物,打破以往的天然产物发现瓶颈,使微生物天然产物研究进入新时代[37]。
木霉作为重要的生防真菌,其中哌珀霉素是其特征性次级代谢产物,也是重要的活性物质。为了从新鉴定的炭团木霉中挖掘新活性物质,利用绿木霉中tex1基因为参考序列,搜索到一个与其同源性性高达74%的非核糖体肽合酶基因NRPS1,推测该基因可能参与哌珀霉素类化合物的合成。利用先前构建的遗传操作体系,成功敲除该基因并获得突变株,提取其粗提物进行了活性实验。研究炭团木霉和NRPS1缺失突变株TYHL3粗提物对寄生宿主炭团菌生长影响及对植物病原菌的拮抗作用。结果发现,与野生菌株相比,NRPS1基因的缺失菌株的粗提物对炭团菌的抑制作用显著降低(图 4),说明该基因的缺失影响了活性物质的产生,进而降低了对炭团菌的抑制。推测炭团木霉可能通过活性次级代谢产物的产生,达到对寄主菌株的抑制作用,这可能是两个真菌在进化过程中长期适应环境的结果。为了研究该基因在抗病原菌的作用,将炭团木霉野生菌株和突变株与A. parasiticus等9株病原菌进行共培养,发现NRPS1基因的缺失突变株对抗病原菌的能力大大减弱(图 5),证明该基因是合成活性物质的关键基因,进而表明该基因的防御功能。由于该基因与木霉属中哌珀霉素类化合物合成基因同源性很高,这些基因编码的均为结构特殊且高度保守的非核糖体多肽合成酶,比如与绿木霉中哌珀霉素tex1的同源性74%。因此,推测该基因可能是合成哌珀霉素类化合物,相关化合物结构的解析对其作用机制的研究将会更加清晰。本研究利用基因组挖掘策略成功鉴定一个木霉中有生防功能的基因,并发现其在对抗病原菌中起关键作用。该研究对木霉中活性物质的挖掘及其将来生防应用具有重要应用价值。
References
| [1] | Harman GE, Howell CR, Viterbo A, Chet I, Lorito M. Trichoderma species-opportunistic, avirulent plant symbionts. Nature Reviews Microbiology, 2004, 2(1): 43-56. DOI:10.1038/nrmicro797 |
| [2] | Harman GE. Overview of mechanisms and uses of Trichoderma spp. Phytopathology, 2006, 96(2): 190-194. DOI:10.1094/PHYTO-96-0190 |
| [3] | Howell CR. Mechanisms employed by Trichoderma species in the biological control of plant diseases:the history and evolution of current concepts. Plant Disease, 2003, 87(1): 4-10. DOI:10.1094/PDIS.2003.87.1.4 |
| [4] | Ghorbanpour M, Omidvari M, Abbaszadeh-Dahaji P, Omidvar R, Kariman K. Mechanisms underlying the protective effects of beneficial fungi against plant diseases. Biological Control, 2018, 117: 147-157. DOI:10.1016/j.biocontrol.2017.11.006 |
| [5] | Papavizas GC. Trichoderma and Gliocladium:biology, ecology, and potential for biocontrol. Annual Review of Phytopathology, 1985, 23: 23-54. DOI:10.1146/annurev.py.23.090185.000323 |
| [6] | Sun JZ, Pei YF, Li EW, Li W, Hyde KD, Yin WB, Liu XZ. A new species of Trichoderma hypoxylon harbours abundant secondary metabolites. Scientific Reports, 2016, 6: 37369. DOI:10.1038/srep37369 |
| [7] | Reino JL, Guerrero RF, Hernández-Galán R, Collado IG. Secondary metabolites from species of the biocontrol agent Trichoderma. Phytochemistry Reviews, 2007, 7(1): 89-123. DOI:10.1007/s11101-006-9032-2 |
| [8] | Contreras-Cornejo HA, Macías-Rodríguez L, Del-Val E, Larsen J. Ecological functions of Trichoderma spp. and their secondary metabolites in the rhizosphere:interactions with plants. FEMS Microbiology Ecology, 2016, 92(4): fiw036. DOI:10.1093/femsec/fiw036 |
| [9] | Inbar J, Abramsky M, Cohen D, Chet I. Plant growth enhancement and disease control by Trichoderma harzianum in vegetable seedlings grown under commercial conditions. European Journal of Plant Pathology, 1994, 100(5): 337-346. DOI:10.1007/BF01876444 |
| [10] | Zhu ZX, Zhuang WY. Current understanding of the genus Trichoderma (Hypocreales, Ascomycota). Mycosystema, 2014, 33(6): 1136-1153. (in Chinese) 朱兆香, 庄文颖. 木霉属研究概况. 菌物学报, 2014, 33(6): 1136-1153. |
| [11] | Song XY, Zhang YZ, Wang YX. Antimicrobial peptides peptaibols from Trichoderma-a review. Acta Microbiologica Sinica, 2011, 51(4): 438-444. (in Chinese) 宋晓妍, 张玉忠, 王元秀. 木霉peptaibols抗菌肽的研究进展. 微生物学报, 2011, 51(4): 438-444. |
| [12] | Lynch JM, Lumsden RD, Atkey PT, Ousley MA. Prospects for control of pythium damping-off of lettuce with Trichoderma, Gliocladium and Enterobacter spp. Biology and Fertility of Soils, 1991, 12(2): 95-99. DOI:10.1007/BF00341482 |
| [13] | Meyer CE, Reusser F. A polypeptide antibacterial agent isolated from Trichoderma viride. Experientia, 1967, 23(2): 85-86. DOI:10.1007/BF02135929 |
| [14] | Leitgeb B, Szekeres A, Manczinger L, Vágv?lgyi C, Kredics L. The history of alamethicin:a review of the most extensively studied peptaibol. Chemistry & Biodiversity, 2007, 4(6): 1027-1051. |
| [15] | Chugh JK, Wallace BA. Peptaibols:models for ion channels. Biochemical Society Transactions, 2001, 29(4): 565-570. |
| [16] | Degenkolb T, Kirschbaum J, Brückner H. New sequences, constituents, and producers of peptaibiotics:an updated review. Chemistry & Biodiversity, 2007, 4(6): 1052-1067. |
| [17] | Whitmore L, Wallace BA. The peptaibol database:a database for sequences and structures of naturally occurring peptaibols. Nucleic Acids Research, 2004, 32(S1): 593-594. |
| [18] | Whitmore L, Chugh JK, Snook CF, Wallace BA. The peptaibol database:a sequence and structure resource. Journal of Peptide Science, 2003, 9(11/12): 663-665. |
| [19] | Marik T, Tyagi C, Racic G, Rakk D, Szekeres A, Vagvolgyi C, Kredics L. New 19-residue peptaibols from Trichoderma clade viride. Microorganisms, 2018, 6(3): 85. DOI:10.3390/microorganisms6030085 |
| [20] | Wiest A, Grzegorski D, Xu BW, Goulard C, Rebuffat S, Ebbole DJ, Bodo B, Kenerley C. Identification of peptaibols from Trichoderma virens and cloning of a peptaibol synthetase. Journal of Biological Chemistry, 2002, 277(23): 20862-20868. DOI:10.1074/jbc.M201654200 |
| [21] | Ehrenstein G, Lecar H. Electrically gated ionic channels in lipid bilayers. Quarterly Reviews of Biophysics, 1977, 10(1): 1-34. |
| [22] | Liu H, Wang G, Li W, Liu XZ, Li EW, Yin WB. A highly efficient genetic system for the identification of a harzianum B biosynthetic gene cluster in Trichoderma hypoxylon. Microbiology, 2018, 164(5): 769-778. DOI:10.1099/mic.0.000649 |
| [23] | Alexander NJ, Hohn TM, McCormick SP. The TRI11 gene of Fusarium sporotrichioides encodes a cytochrome P-450 monooxygenase required for C-15 hydroxylation in trichothecene biosynthesis. Applied and Environmental Microbiology, 1998, 64: 221-225. DOI:10.1128/AEM.64.1.221-225.1998 |
| [24] | Blin K, Shaw S, Steinke K, Villebro R, Ziemert N, Lee SY, Medema MH, Weber T. AntiSMASH 5.0:updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research, 2019, 41(W1): 81-87. |
| [25] | Bok JW, Keller NP. Fast and easy method for construction of plasmid vectors using modified quick-change mutagenesis//Keller N, Turner G. Fungal Secondary Metabolism. Totowa: Humana Press, 2012: 163-174. |
| [26] | Fang ZD. Method of plant pathology research. 3rd ed. Beijing: Agricultural Press, 1998: 141. (in Chinese) 方中达. 植病研究方法. 第3版. 北京: 中国农业出版社, 1998: 141. |
| [27] | Marik T, Szekeres A, Várszegi C, Czifra D, Vágv?lgyi C, Kredics L. Rapid bioactivity-based pre-screening method for the detection of peptaibiotic-producing Trichoderma strains. Acta Biologica Szegediensis, 2013, 57(1): 1-7. |
| [28] | Limón MC, Chacón MR, Mejías R, Delgado-Jarana J, Rincón AM, Codón AC, Benítez T. Increased antifungal and chitinase specific activities of Trichoderma harzianum CECT 2413 by addition of a cellulose binding domain. Applied Microbiology and Biotechnology, 2004, 64(5): 675-685. DOI:10.1007/s00253-003-1538-6 |
| [29] | Tronsmo A, Dennis C. Effect of temperature on antagonistic properties of Trichoderma species. Transactions of the British Mycological Society, 1978, 71(3): 469-474. DOI:10.1016/S0007-1536(78)80075-8 |
| [30] | Yelton MM, Hamer JE, Timberlake WE. Transformation of Aspergillus nidulans by using a trpC plasmid. Proceedings of the National Academy of Sciences of the United States of America, 1984, 81(5): 1470-1474. DOI:10.1073/pnas.81.5.1470 |
| [31] | Wang XN, Zhang XL, Liu L, Xiang MC, Wang WZ, Sun X, Che YS, Guo LD, Liu G, Guo LY, Wang CS, Yin WB, Stadler M, Zhang XY, Liu XZ. Genomic and transcriptomic analysis of the endophytic fungus Pestalotiopsis fici reveals its lifestyle and high potential for synthesis of natural products. BMC Genomics, 2015, 16: 28. DOI:10.1186/s12864-014-1190-9 |
| [32] | Martinez D, Berka RM, Henrissat B, Saloheimo M, Arvas M, Baker SE, Chapman J, Chertkov O, Coutinho PM, Cullen D, Danchin EGJ, Grigoriev IV, Harris P, Jackson M, Kubicek CP, Han CS, Ho I, Larrondo LF, De Leon AL, Magnuson JK, Merino S, Misra M, Nelson B, Putnam N, Robbertse B, Salamov AA, Schmoll M, Terry A, Thayer N, Westerholm-Parvinen A, Schoch CL, Yao J, Barabote R, Nelson MA, Detter C, Bruce D, Kuske CR, Xie G, Richardson P, Rokhsar DS, Lucas SM, Rubin EM, Dunn-Coleman N, Ward M, Brettin TS. Genome sequencing and analysis of the biomass-degrading fungus Trichoderma reesei (syn. Hypocrea jecorina). Nature Biotechnology, 2008, 26(5): 553-560. DOI:10.1038/nbt1403 |
| [33] | Huang DW, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 |
| [34] | Li W, Wu GW, Yang YP, Yin WB. Discovery of fungal natural product in the post-genomic era. Mycosystema, 2015, 34(5): 914-926. (in Chinese) 李伟, 吴广畏, 杨玉萍, 尹文兵. 后基因组时代的真菌天然产物发现. 菌物学报, 2015, 34(5): 914-926. |
| [35] | Zheng YJ, Ma K, Lyu HN, Huang Y, Liu HW, Liu L, Che YS, Liu XZ, Zou HX, Yin WB. Genetic manipulation of the COP9 signalosome subunit PfCsnE leads to the discovery of pestaloficins in Pestalotiopsis fici. Organic Letters, 2017, 19(17): 4700-4703. DOI:10.1021/acs.orglett.7b02346 |
| [36] | Udwary DW, Zeigler L, Asolkar RN, Singan V, Lapidus A, Fenical W, Jensen PR, Moore BS. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(25): 10376-10381. DOI:10.1073/pnas.0700962104 |
| [37] | Cragg GM, Newman DJ. Natural products:a continuing source of novel drug leads. Biochimica et Biophysica Acta (BBA)-General Subjects, 2013, 1830(6): 3670-3695. DOI:10.1016/j.bbagen.2013.02.008 |
