张丹, 赵芹芹, 蒋明, 康前进, 白林泉


上海交通大学生命科学技术学院, 微生物代谢国家重点实验室, 上海 200240
收稿日期:2019-03-18;修回日期:2019-04-13;网络出版日期:2019-07-11
基金项目:国家自然科学基金(31830104)
*通信作者:白林泉, Tel/Fax:+86-21-34206722;E-mail:bailq@sjtu.edu.cn.
摘要:[目的] 解析Actinoplanes sp. SE50/110(简称SE50/110)中阿卡波糖脱氧氨基糖单元的生物合成机制。[方法] 经过BLASTp分析,推测了AcbA、AcbB和AcbV负责阿卡波糖脱氧氨基糖单元的生物合成。首先,本研究在SE50/110中分别构建了acbA、acbB和acbV的同框缺失和回补突变株。然后,利用大肠杆菌BL21(DE3)/pGro7分别对AcbA、AcbB和AcbV成功实现了可溶性表达。最后,以D-葡萄糖-1-磷酸为起始底物,通过体外催化反应,研究脱氧氨基糖单元的生物合成过程和相关蛋白的酶学性质。[结果] 在SE50/110中分别缺失acbA、acbB和acbV基因后,相应突变株均丧失了阿卡波糖的合成能力,将acbA、acbB和acbV基因分别回补后,各菌株又恢复了阿卡波糖的合成能力,证明了它们均为阿卡波糖生物合成的必需基因。在体外酶促反应中,D-葡萄糖-1-磷酸-胸腺嘧啶转移酶AcbA催化D-葡萄糖-1-磷酸和dTTP合成dTDP-D-葡萄糖,对D-葡萄糖-1-磷酸的Km值为(0.185±0.053)mmol/L,Vmax为(2.366±0.217)μmol/(min·mg);对dTTP的Km值为(4.964±1.089)mmol/L,Vmax为(60.310±5.419)μmol/(min·mg)。dTDP-D-葡萄糖-4,6-脱水酶AcbB催化dTDP-D-葡萄糖转化为dTDP-4-酮基-6-脱氧-D-葡萄糖,Km值和Vmax分别为(0.353±0.089)mmol/L和(306.401±28.740)μmol/(min·mg)。氨基转移酶AcbV催化dTDP-4-酮基-6-脱氧-D-葡萄糖生成dTDP-4-氨基-4,6-双脱氧-D-葡萄糖,Km值和Vmax分别为(1.411±0.293)mmol/L和(3.447±0.279)μmol/(min·mg)。[结论] 本研究阐明了阿卡波糖脱氧氨基糖单元的生物合成过程,为全面解析阿卡波糖生物合成途径奠定了基础。同时,测定了相关酶的动力学参数,为代谢工程改造SE50/110,提高阿卡波糖产量提供了重要的理论依据。
关键词:阿卡波糖脱氧氨基糖生物合成酶促反应
Biosynthetic pathway of deoxyaminosugar moiety in acarbose from Actinoplanes sp. SE50/110
Dan Zhang, Qinqin Zhao, Ming Jiang, Qianjin Kang, Linquan Bai


State Key Laboratory of Microbial Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China
Received: 18 March 2019; Revised: 13 April 2019; Published online: 11 July 2019
*Corresponding author: Linquan Bai, Tel/Fax: +86-21-34206722; E-mail: bailq@sjtu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31830104)
Abstract: [Objective] Elucidation of the biosynthetic mechanism of the deoxyaminosugar moiety in acarbose from Actinoplanes sp. SE50/110. [Methods] On the basis of BlastP analysis, AcbA, AcbB and AcbV were proposed to be associated with the biosynthesis of deoxyaminosugar moiety. Firstly, the in-frame deletion and trans-complementation of acbA, acbB and acbV were performed in SE50/110 to investigate their involvement in acarbose biosynthesis. Then, AcbA, AcbB and AcbV were heterologously expressed in E. coli BL21(DE3)/pGro7 and purified by Ni affinity chromatography. Finally, using D-glucose-1-phosphate as the starting substrate, the biosynthetic process of deoxyaminosugar moiety was elucidated by enzymatic assay. In addition, the properties of these involved enzymes were characterized. [Results] Deletion mutants of acbA, acbB and acbV in SE50/110 were named as ZD03, ZD04 and ZD05, respectively, all of which lost the productivity of acarbose. And then, the production of acarbose was recovered through trans-complementation of acbA, acbB and acbV in ZD03, ZD04 and ZD05, respectively. In vitro enzymatic analysis suggested that AcbA, a D-glucose-1-phosphate thymidylyltransferase, is responsible for the biosynthesis of dTDP-D-glucose from D-glucose-1-phosphate and dTTP. It showed a Km of (0.185±0.053) mmol/L and a Vmax of (2.366±0.217) μmol/(min·mg) with D-glucose-1-phosphate, as well as a Km of (4.964±1.089) mmol/L and a Vmax of (60.310±5.419) μmol/(min·mg) with dTTP. AcbB, a TDP-D-glucose-4, 6-dehydratase, catalyzed the dehydration of dTDP-D-glucose to dTDP-4-keto-6-deoxy-D-glucose. The Km and Vmax of AcbB are (0.353±0.089) mmol/L and (306.401±28.740) μmol/(min·mg), respectively. AcbV is a dTDP-4-keto-6-deoxy-D-glucose-aminotransferase and catalyzes the transamination of dTDP-4-keto-6-deoxy-D-glucose to dTDP-4-amino-4, 6-dideoxy-D-glucose using glutamic acid as amino donor. The Km and Vmax of AcbV are (1.411±0.293) mmol/L and (3.447±0.279) μmol/(min·mg), respectively. [Conclusion] This study elucidated the biosynthetic pathway of deoxyaminosugar moiety in acarbose, which paved a solide way for a full elucidation of acarbose biosynthesis. Meanwhile, the characterization of the involved enzymes provided important information for the metabolic engineering of SE50/110 to improve acarbose production.
Keywords: acarbosedeoxyaminosugarbiosynthesisenzymatic assay
阿卡波糖(acarbose)作为α-葡萄糖苷酶抑制剂,主要用于临床治疗2型糖尿病[1]。目前,阿卡波糖主要通过游动放线菌(Actinoplanes spp.)发酵生产[2]。近年来,全球糖尿病患病率激增,截止2017年,患病人数已高达4.51亿,预计2045年将超过6.93亿[3],其中,2型糖尿病约占所有成人病例的90%[4]。阿卡波糖作为重要的口服降糖药物,市场需求量不断增加。因此,如何提高阿卡波糖产生菌的生产能力与产品的质量是亟待解决的重要问题。
阿卡波糖的化学结构(图 1-A)分别由一分子的C7-环醇(C7-cyclitol)、脱氧氨基糖(deoxyaminosugar)和麦芽糖(maltose)组成[5]。其生物合成基因簇(acb cluster) (图 1-B)全长约30 kb,包括22个结构基因,负责该化合物的生物合成(acbAB和acbVUSRPIJQKLMNOC)、转运(acbWXY)及相关衍生物的形成(acbDEZ)。研究者们通过蛋白序列同源性比对、同位素标记喂养及酶催化反应等分析手段,揭示了C7-环醇单元的合成是以7-磷酸-景天庚酮糖为前体,经AcbC催化生成2-表-5-表-有效醇酮,再由AcbM和AcbO依次催化生成7-磷酸-5-表-有效醇酮。在此基础上,提出了阿卡波糖的生物合成是C7-环醇单元与脱氧氨基糖单元经糖基转移酶催化缩合,再与一分子麦芽糖缩合,最后被去磷酸化生成阿卡波糖[6-7]。但除了C7-环醇单元合成中负责前三步催化反应的蛋白功能被解析以外,其他的催化步骤均未得到证实。经过不断努力,已经成功建立了高效的游动放线菌遗传操作体系[8-11],为鉴定阿卡波糖的生物合成基因、解析其生物合成途径奠定了良好的基础。这些方面的研究也为通过代谢工程创制优产的工业菌株、提高阿卡波糖产量与品质提供了重要依据。
 |
| 图 1 阿卡波糖的化学结构式及生物合成基因簇 Figure 1 The chemical structure and biosynthetic gene cluster of acarbose. |
| 图选项 |
结构多样的脱氧糖单元广泛存在于糖脂和糖蛋白的寡糖模块及众多次级代谢产物中,并与重要的生物学活性有关。一般来说,这些寡糖或次级代谢产物结构中的糖基是由核苷酸活化的脱氧糖经过糖基转移酶的催化而加载上的。其中,dTDP-脱氧糖普遍以D-葡萄糖-1-磷酸为前体,经D-葡萄糖-1-磷酸-胸腺嘧啶转移酶和dTDP-D-葡萄糖-4, 6-脱水酶的催化生成中间体dTDP-4-酮基-6-脱氧-D-葡萄糖[12],然后,再经差向异构、转氨等多种修饰反应合成多样化的糖基结构单元,参与各种天然产物的合成,例如:广泛存在于植物和微生物中的dTDP-鼠李糖[13-15]、Shigella dysenteriae中的O-抗原[16]、Streptomyces GERI-155中的GERI-155[17-19]、S. argillaceus中的光神霉素[20]以及S. violaceoruber Tü22中的榴菌素[21]。这些研究工作为解析阿卡波糖脱氧糖单元的生物合成途径提供了借鉴。
鉴定与解析脱氧氨基糖生物合成中相关基因的功能是解析阿卡波糖生物合成机制的重要组成部分。因此,本研究在详细比对分析基因功能的基础上推测了acbA、acbB和acbV参与脱氧氨基糖单元的生物合成过程。然后,通过体内基因敲除与回补,确定了这些基因为阿卡波糖生物合成的必需基因。最后,通过体外催化反应,揭示了脱氧氨基糖单元的生物合成过程,并分别表征了相关酶的特性,为解析阿卡波糖生物合成途径和代谢工程改造奠定了基础。
1 材料和方法 1.1 菌株、质粒、引物 试验中所用菌株、质粒和引物见表 1。
表 1. 实验中所用菌株、质粒和引物 Table 1. Strains, plasmids and primers used in this study
| Strains/plasmids/primers | Related characters or sequences | Sources or references |
| Actinoplanes spp. | ||
| SE50/110 | Acarbose producer | ATCC |
| ZD03 | Actinoplanes sp. SE50/110 ΔacbA | This work |
| ZD04 | Actinoplanes sp. SE50/110 ΔacbB | This work |
| ZD05 | Actinoplanes sp. SE50/110 ΔacbV | This work |
| ZD03:: acbA | Actinoplanes sp. SE50/110 ΔacbA:: acbA | This work |
| ZD04:: acbB | Actinoplanes sp. SE50/110 ΔacbB:: acbB | This work |
| ZD05:: acbV | Actinoplanes sp. SE50/110 ΔacbV:: acbV | This work |
| Escerichia coli | ||
| DH10B | Cloning host | GIBCO-BRL |
| ET12567(pUZ8002) | recE, dam-, dcm-, hsdS, Cmr, Strr, Tetr, Kmr | [22] |
| BL21(DE3)/pGro7 | Protein expression host | Sangon Biotech |
| Plasmids | ||
| pLQ752 | pJTU1278-derived vector with the tsr gene replaced by aac(3)IV and inserted codA gene | [11] |
| pDR4-K* | aac(3)IV, xylE-neo, kasOp* | [23] |
| pSET152 | oriTRK2, aac(3)IV, ?31 int, attP | [24] |
| pET30a | oripBR322, Kmr, lacI, His-tag and S-tag coding genes | Novagen |
| pLQ1103 | pLQ752-based plasmid for acbA inactivation | This work |
| pLQ1104 | pLQ752-based plasmid for acbB inactivation | This work |
| pLQ1105 | pLQ752-based plasmid for acbV inactivation | This work |
| pLQ1106 | pSET152-based plasmid for overexpression of acbA | This work |
| pLQ1107 | pSET152-based plasmid for overexpression of acbB | This work |
| pLQ1108 | pSET152-based plasmid for overexpression of acbV | This work |
| pLQ1109 | pET30a-based plasmid for AcbA expression | This work |
| pLQ1110 | pET30a-based plasmid for AcbB expression | This work |
| pLQ1111 | pET30a-based plasmid for AcbV expression | This work |
| Primers | Sequences (5′→3′) | |
| acbA-L-F (Xba I) | GCTCTAGACGGATCTTCGCGGTGATGAC | |
| acbA-L-R (EcoR I) | CAGAATTCCTCGGTCTCGGTCGTGATGA | |
| acbA-R-F (EcoR I) | CAGAATTCGACAGTACCTGCTGGCTCTG | |
| acbA-R-R (Hind Ⅲ) | GCAAGCTTCACCTTCGACACCTACACCC | |
| acbA-C-F | GACGACCGTAACCTGCGTCA | |
| acbA-C-R | CACCGGAACTGTGGCAGTAC | |
| acbB-L-F (Xba I) | GCTCTAGACGGCGAAACTCGATCTGACC | |
| acbB-L-R (EcoR I) | CAGAATTCGTGTGGCAATGTCGCCACTG | |
| acbB-R-F (EcoR I) | CAGAATTCGAAACGGTCGCGTGGTATCG | |
| acbB-R-R (Hind Ⅲ) | GCAAGCTTCTGCTGGACACCTTCAACCC | |
| acbB-C-F | CGTGATGACCAGGATCTCCC | |
| acbB-C-R | CGGATCTTCGCGGTGATGAC | |
| acbV-L-F (Xba I) | GCTCTAGAAGGATGCGGAACGAGTCGAG | |
| acbV-L-R (EcoR I) | CAGAATTCCGAAGTCGTGGACGTTCCAC | |
| acbV-R-F (EcoR I) | CAGAATTCCGTTCGTCATCGACGAGACC | |
| acbV-R-R (Hind Ⅲ) | GCAAGCTTGTCTGGAGGACGTGTCGAAG | |
| acbV-C-F | CAGGAGTGACCCGTGATGTG | |
| acbV-C-R | GAAGACGATCAGATGGCCGG | |
| kasOp*-F (Xba I) | GCTCTAGATGTTCACATTCGAACGGTCTCTGCT | |
| kasOp*-A-R | ACCGGCATGACGTCACCTCTTCAAC | |
| acbA-F | GTGACGTCATGCCGGTCTATGACAAACCGATGA | |
| acbA-R (BamH I) | CTGGATCCTCACCCGGCGGCGGCGGTCAAGGCC | |
| kasOp*-B-R | ATTTTCATGACGTCACCTCTTCAAC | |
| acbB-F | GTGACGTCATGAAAATCTTGGTCACCGGCGGAG | |
| acbB-R (BamH I) | CTGGATCCTCAGGTCCACCAGGAACGGTTGGCG | |
| kasOp*-V-R | CTGCTCACGACGTCACCTCTTCAAC | |
| acbV-F | GTGACGTCGTGAGCAGGCAGGCCGACCTGCTCG | |
| acbV-R (BamH I) | CTGGATCCTCATGCCGTCACCCGCCCGGCCTCG | |
| AcbA-EXP-F (EcoR I) | GAGAATTCATGCCGGTCTATGACAAACCGATG | |
| AcbA-EXP-R (Hind Ⅲ) | GCAAGCTTTCACCCGGCGGCGGCGGTCA | |
| AcbB-EXP-F (EcoR I) | GAGAATTCATGAAAATCTTGGTCACCGG | |
| AcbB-EXP-R (Hind Ⅲ) | GCAAGCTTTCAGGTCCACCAGGAACGGT | |
| AcbV-EXP-F (EcoR I) | GAGAATTCGTGAGCAGGCAGGCCGACCT | |
| AcbV-EXP-R (Hind Ⅲ) | GCAAGCTTTCATGCCGTCACCCGCCCGG | |
表选项
1.2 培养基和培养方法 大肠杆菌培养基包括LB液体和固体培养基;游动放线菌培养基包括黄豆饼粉浸汁琼脂(SFM)培养基、STY培养基、SM培养基、TSB培养基和阿卡波糖发酵培养基。大肠杆菌和游动放线菌的培养基配制方法和培养方法均参照文献[11]。
1.3 工具酶及试剂 实验中所用的高保真DNA聚合酶和T4 DNA连接酶均购自大连宝生物工程有限公司;小量质粒DNA抽提试剂盒、细菌DNA基因组提取试剂盒均购自上海捷瑞生物工程有限公司;各种限制性内切酶购自Thermo Scientific公司;胶回收试剂盒购自Omega公司;焦磷酸酶购自Sigma公司;各种抗生素、dTTP、D-葡萄糖-1-磷酸、NAD+和PLP等试剂购自生工生物工程(上海)股份有限公司;培养基相关试剂均购自上海国药集团化学试剂有限公司;所用引物由上海擎科生物科技有限公司合成;dTDP-D-葡萄糖和dTDP-4-酮基-6-脱氧-D-葡萄糖标品购自卡博森斯化学科技(苏州)有限公司。
1.4 Actinoplanes sp.基因缺失的方法 为构建acbA基因缺失的突变株,首先以SE50/110基因组DNA为模板,分别以携带Xba I/ EcoR I酶切位点的acbA-L-F/R和携带EcoR I/ Hind Ⅲ酶切位点的acbA-R-F/R为引物,利用PCR扩增得到1.52 kb和1.62 kb的上下游同源片段,将测序正确的片段经酶切、酶连插入游离型载体pLQ752,获得重组质粒pLQ1103。将重组质粒转入大肠杆菌ET12567(pUZ8002)中,采用菌丝体接合转移的方式将质粒导入SE50/110中,经松弛和筛选,获得基因同框缺失菌株。抽提其基因组DNA作为模板,以acbA-C-F/R为验证引物进行PCR扩增,扩增得到0.62 kb的片段,而以SE50/110基因组DNA为模板,得到1.32 kb的片段,则表明突变株构建成功,命名为ZD03。在构建acbB基因缺失的突变株时,首先以SE50/110基因组DNA为模板,分别以acbB-L-F/R和acbB-R-F/R为引物,经PCR扩增得到1.54 kb和1.62 kb的上下游同源片段,并将测序正确的片段插入载体pLQ752中,获得重组质粒pLQ1104。将质粒导入大肠杆菌ET12567(pUZ8002)中,经接合转移、松弛和筛选后,以acbB-C-F/R为验证引物进行PCR验证,若以重组菌株基因组DNA为模板,扩增得到0.63 kb的片段,而以SE50/110基因组DNA为模板,得到1.50 kb的片段,则表明成功构建了突变株,命名为ZD04。按照上述的方法构建用于acbV敲除的重组质粒pLQ1105,然后,构建筛选得到相应的突变株,以acbV-C-F/R为引物进行PCR验证,若以突变株基因组DNA为模板,扩增获得0.69 kb的片段,而以SE50/110基因组DNA为模板获得1.67 kb的片段,则表明突变株构建正确,命名为ZD05。
1.5 Actinoplanes sp.基因回补的方法 为构建acbA基因回补的突变株,首先,以pDR4-K*质粒DNA为模板,以kasOp*-F/kasOp*- A-R为引物,通过PCR扩增得到0.13 kb的kasOp*启动子片段;以SE50/110基因组DNA为模板,以acbA-F/R为引物扩增得到0.85 kb的acbA基因片段。再以kasOp*-F/acbA-R为引物,经重叠PCR扩增得到长为0.98 kb的kasOp*-acbA片段。将测序正确的片段经酶切、酶连插入pSET152中,获得整合型质粒pLQ1106,并转入大肠杆菌ET12567(pUZ8002),经接合转移将质粒导入ZD03,利用引物kasOp*-F/acbA-R进行PCR验证。若以突变株DNA为模板获得长为0.96 kb的PCR片段,而以SE50/110基因组DNA为模板无扩增条带,则表明基因回补成功。按照同样的方法分别构建用于acbB和acbV回补的整合型质粒pLQ1107和pLQ1108,分别导入ZD04和ZD05中,筛选获取相应的突变株,分别以引物kasOp*-F/acbB-R和kasOp*-F/acbV-R进行PCR验证,若分别扩增获得1.09 kb和1.42 kb的PCR产物,则表明基因回补成功。
1.6 蛋白的表达菌株构建、表达及纯化
1.6.1 蛋白表达菌株的构建: 为获得AcbA蛋白表达质粒,首先以SE50/110基因组DNA为模板,以携带EcoR I/Hind Ⅲ酶切位点的AcbA-EXP-F/R为引物,利用PCR扩增得到0.85 kb的acbA基因片段。将测序正确的片段经酶切、酶连插入载体pET30a,获得质粒pLQ1109。以同样的方法构建分别用于AcbB和AcbV蛋白表达的质粒pLQ1110和pLQ1111。将上述质粒分别转化至大肠杆菌BL21(DE3)/pGro7中,获得蛋白表达菌株。
1.6.2 蛋白表达: 将蛋白表达菌株接种于含50 mg/L卡那霉素和25 mg/L氯霉素的LB培养基中过夜培养,而后转接至含有0.5 mg/mL L-阿拉伯糖、50 mg/L卡那霉素和25 mg/L氯霉素的LB培养基中,在37 ℃、220 r/min条件下培养至OD600达到0.6?0.8时,加入终浓度为0.8 mmol/L的IPTG进行诱导,在16 ℃条件下继续培养20?22 h。最后,将菌液以5000 r/min离心10 min,收集菌体,并放置于?80 ℃保存待用。
1.6.3 蛋白纯化: 用含25 mmol/L Tris-HCl、300 mmol/L NaCl的Buffer A (pH 8.0)重悬菌体,经超声破碎后,在4 ℃离心机中,以12000 r/min离心30 min,将上清液上载至镍亲和层析柱上,依次用Buffer A和25 mmol/L咪唑洗去杂蛋白,再用250 mmol/L咪唑洗脱目标蛋白,而后,经10 kDa的Millipore超滤管脱盐浓缩后,利用SDS-PAGE分析蛋白的纯化情况。
1.7 蛋白的体外功能分析及酶学性质表征
1.7.1 AcbA蛋白功能鉴定: 在20 μL反应体系中加入50 mmol/L Tris-HCl (pH 8.0)、24 mmol/L D-葡萄糖-1-磷酸、12 mmol/L dTTP、12 mmol/L MgCl2、0.12 U焦磷酸酶、20 μmol/L AcbA蛋白,置于30 ℃反应12 h后,用40 μL甲醇终止反应,而后利用LC-ESI-MS检测产物的生成情况。
1.7.2 AcbB蛋白功能鉴定: 在20 μL反应体系中加入50 mmol/L Tris-HCl (pH 8.0)、5 mmol/L dTDP-D-葡萄糖、1 mmol/L NAD+、20 μmol/L AcbB蛋白,置于30 ℃反应1 h后,用40 μL甲醇终止反应,然后利用LC-ESI-MS检测产物的产生情况。
1.7.3 AcbV蛋白功能鉴定: 在20 μL反应体系中加入50 mmol/L Tris-HCl (pH 8.0)、5 mmol/L dTDP-4-酮基-6-脱氧-D-葡萄糖、1 mmol/L PLP、8 mmol/L L-谷氨酸、20 μmol/L AcbV蛋白,置于30 ℃反应6 h后,用40 μL甲醇终止反应,再利用LC-ESI-MS检测产物的生成情况。
1.7.4 温度对酶活性的影响: 利用蛋白功能鉴定的反应体系,分别置于20、25、30、33、37、40、45和50 ℃进行反应,每个条件进行了3次生物学重复。其中,AcbA、AcbB和AcbV的反应时间分别为8、1和6 h。终止反应后,利用HPLC-QQQMS分别测定AcbA和AcbB反应后产物的生成量或AcbV反应后底物的消耗量,计算反应速率,确定酶的相对活性,进而确定最适温度。
1.7.5 pH对酶活性的影响: 在最适温度条件下,分别利用pH 5.0、6.0、6.5、7.0、7.5、8.0、8.5和9.0的缓冲体系进行反应,每个条件进行3次生物学重复。其中pH 5.0?7.0采用Na2HPO4-KH2PO4缓冲液,pH 7.5?9.0采用Tris-HCl缓冲液,AcbA、AcbB和AcbV的反应时间分别为8、1和6 h。终止反应后,利用HPLC- QQQMS分别测定AcbA和AcbB反应后产物的生成量及AcbV反应后底物的消耗量,计算反应速率,确定酶的相对活性,进而确定最适pH。
1.7.6 测定酶动力学参数: 为测定AcbA对底物D-葡萄糖-1-磷酸的酶动力学参数,在反应体系(20 μL)中加入50 mmol/L Tris-HCl (最适pH)、12 mmol/L dTTP、12 mmol/L MgCl2、0.12 U焦磷酸酶、20 μmol/L AcbA蛋白以及D-葡萄糖-1-磷酸(浓度分别为0.06、0.08、0.20、0.40、0.80、1.00 mmol/L),在最适温度下反应8 h后,用40 μL甲醇终止反应,随后,利用HPLC-QQQMS测定产物dTDP-D-葡萄糖的生成量来计算反应速率,每个条件进行了3次生物学重复。最终,利用软件GraphPad Prism 5绘制Michaelis-Menten拟合曲线并计算酶动力学参数。在测定AcbA对底物dTTP的酶动力学参数时,体系中D-葡萄糖-1-磷酸的含量为24 mmol/L,底物dTTP的浓度分别为0.4、0.6、2.0、4.0、10.0和12.0 mmol/L,其他参数保持不变,通过测定dTDP-D-葡萄糖生成量来计算反应速率,进而计算酶动力学参数。为测定AcbB对底物dTDP-D-葡萄糖的动力学参数,在20 μL反应体系中加入50 mmol/L Tris-HCl (最适pH)、1 mmol/L NAD+、20 μmol/L AcbB蛋白以及dTDP-D-葡萄糖(浓度分别为0.1、0.2、0.4、0.6、0.8和1.0 mmol/L),在最适温度下反应1 h后,用40 μL甲醇终止反应,各条件分别有3次生物学重复。通过测定产物dTDP-4-酮基-6-脱氧-D-葡萄糖的生成量来计算反应速率,进而拟合计算酶动力学参数。为测定AcbV对底物dTDP-4-酮基-6-脱氧-D-葡萄糖的酶动力学参数,在20 μL反应体系中加入50 mmol/L Tris-HCl (最适pH)、1 mmol/L PLP、8 mmol/L L-谷氨酸、20 μmol/L AcbV蛋白以及dTDP-4-酮基-6-脱氧-D-葡萄糖(浓度分别为0.4、0.6、0.8、2.0和8.0 mmol/L),在最适温度下反应6 h后,用40 μL甲醇终止反应,每个条件有3次重复。通过检测底物dTDP-4-酮基-6-脱氧-D-葡萄糖的消耗量来计算反应速率,并拟合计算酶动力学参数。
1.8 化合物的定性与定量分析方法
1.8.1 发酵液中阿卡波糖的定量测定: 吸取1 mL的发酵液,经12000 r/min离心后取上清液进行过滤,再利用HPLC (Agilent HPLC 1260)进行检测。色谱条件:色谱柱Agilent phenomenex NH2柱(4.6 mm×250 mm,5 μm);流动相:65%乙腈-35%磷酸盐(0.6 g/L KH2PO4、0.35 g/L Na2HPO4·2H2O);检测波长:210 nm,流速:1.0 mL/min,进样体积:10 μL,柱温:25 ℃。
1.8.2 脱氧氨基糖单元相关化合物的定性测定: 将终止后的反应混合物经12000 r/min离心10 min,上清液用UPLC-TOFMS (Agilent UPLC 1290-MS 6230)在负离子模式下检测。色谱条件:色谱柱Agilent ZORBAX SB-Aq柱(4.6 mm×250 mm,5 μm);流动相:0.5%甲醇-99.5%水;检测波长:254 nm;流速:0.4 mL/min;进样体积:10 μL;柱温:25 ℃。
1.8.3 脱氧氨基糖单元相关化合物的定量测定: 将终止后的反应混合物经12000 r/min离心10 min,上清液用HPLC-QQQMS (Agilent HPLC 1260- QQQMS 6470)检测。色谱条件与UPLC-TOFMS条件相同。dTDP-D-葡萄糖的母离子为563.0685,定量片段为320.80,dTDP-4-酮基-6-脱氧-D-葡萄糖的母离子为545.0579,定量片段为321.40。
2 结果和分析 2.1 鉴定负责阿卡波糖脱氧氨基糖单元合成的必需基因 采用BLASTp对阿卡波糖生物合成基因簇所编码的蛋白进行功能预测,发现AcbA蛋白与E. coli的RfbA和Pseudomonas aeruginosa的RmlA在氨基酸水平上分别具有53.61%和55.00%的一致性,为D-葡萄糖-1-磷酸胸腺嘧啶转移酶,负责催化D-葡萄糖-1-磷酸和dTTP生成dTDP-D-葡萄糖;AcbB蛋白分别与E. coli的RfbB和P. aeruginosa的RmlB有45.11%和45.75%的一致性,为dTDP-D-葡萄糖-4, 6-脱水酶,可催化dTDP-D-葡萄糖转化为dTDP-4-酮基-6-脱氧-D-葡萄糖;AcbV蛋白与转氨酶有较高的一致性,其可能为dTDP-4-酮基-6-脱氧-D-葡萄糖转氨酶,催化dTDP-4-酮基-6-脱氧-D-葡萄糖生成dTDP-4-氨基-4, 6-双脱氧-D-葡萄糖。因此,推测编码这3个蛋白的基因可能参与阿卡波糖脱氧氨基糖单元的合成。
为了对编码上述蛋白的基因进行体内功能验证,利用PCR扩增,获得了acbA、acbB和acbV的上下游同源臂,分别构建了用于基因同框缺失的质粒pLQ1103、pLQ1104和pLQ1105。然后,将质粒分别转化至大肠杆菌ET12567(pUZ8002)中,再利用接合转移的方法分别将质粒导入至SE50/110中。获得接合子后,经松弛、筛选和PCR验证得到缺失目标基因的突变株。其中,SE50/110ΔacbA、SE50/110ΔacbB和SE50/110ΔacbV分别被命名为ZD03、ZD04和ZD05 (图 2-A–C,ⅰ,ⅱ)。将突变株进行发酵,利用HPLC分析相关突变株的发酵产物。结果显示,在ZD03、ZD04和ZD05的发酵液中均无阿卡波糖积累(图 2-D),该结果表明acbA、acbB和acbV编码的蛋白参与了阿卡波糖的生物合成过程。
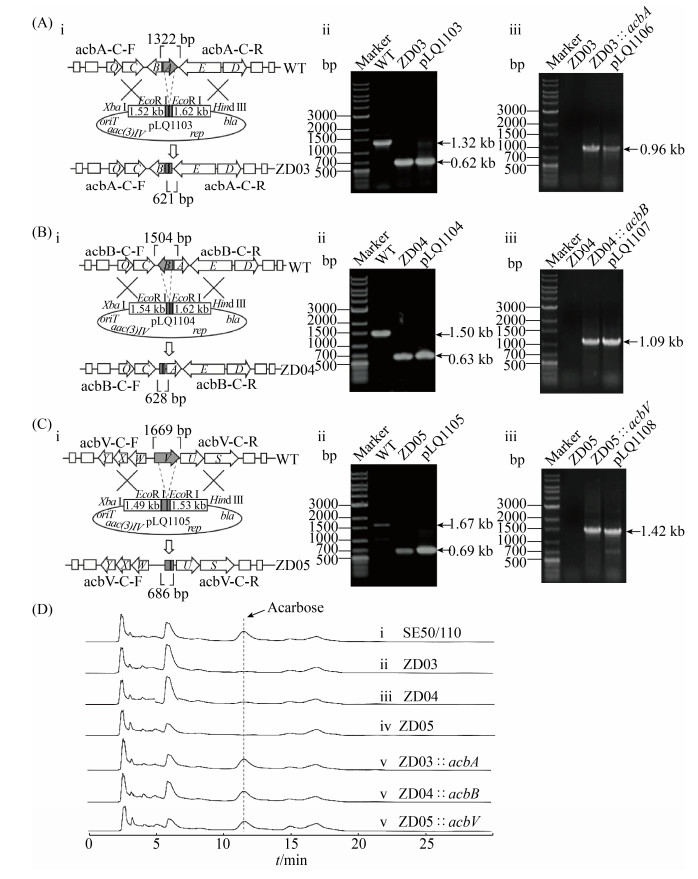 |
| 图 2 acbA、acbB和acbV的基因缺失与回补及突变株产生阿卡波糖的情况 Figure 2 Deletion and trans-complementation of genes acbA, acbB and acbV and productions of acarbose by mutants. A: i: Schematic diagram of acbA deletion; ii: Verification of ZD03 by PCR amplification; Ⅲ: Verification of ZD03::acbA by PCR amplification. B: i: Schematic diagram of acbB deletion; ii: Verification of ZD04 by PCR amplification; Ⅲ: Verification of ZD04::acbB by PCR amplification. C: i: Schematic diagram of acbV deletion; ii: Verification of ZD05 by PCR amplification; Ⅲ: Verification of ZD05::acbV by PCR amplification. D: Fermentation product analysis of mutants by HPLC. |
| 图选项 |
为了进一步验证上述突变株丧失阿卡波糖合成能力的确是由基因缺失所导致,又分别将acbA、acbB和acbV经PCR扩增后,构建至强启动子kasOp*的控制下,获得了分别携带基因盒kasOp*-acbA、kasOp*-acbB和kasOp*-acbV的整合型质粒pLQ1106、pLQ1107和pLQ1108,利用接合转移的方法将基因分别回补至ZD03、ZD04和ZD05,经PCR验证正确后,获得突变株ZD03::acbA、ZD04::acbB和ZD05::acbV(图 2-A-C,Ⅲ)。而后,将获得的突变株进行摇瓶发酵,利用HPLC检测相应的发酵产物,结果显示(图 2-D),突变株均恢复了阿卡波糖的合成能力。该结果表明,acbA、acbB和acbV的确是阿卡波糖生物合成的必需基因。
2.2 AcbA的功能分析及其酶学性质表征
2.2.1 AcbA蛋白的表达及其功能分析: AcbA蛋白属于D-葡萄糖-1-磷酸胸腺嘧啶转移酶,催化脱氧氨基糖单元生物合成的第一步。为获得该蛋白并验证其功能,首先,按照1.6中所述的方法构建了表达质粒pLQ1109,并将其导入至BL21(DE3)/pGro7中,经诱导表达、镍亲和层析柱纯化及SDS-PAGE分析,成功纯化得到了大小约为36 kDa的蛋白(图 3-A)。在蛋白表达过程中,由于没有引入分子伴侣,无可溶性目标蛋白表达,所以改用含有pGro7质粒的BL21(DE3)蛋白表达宿主,但由于分子伴侣GroES-GroEL表达量较大,且非特异性吸附至亲和层析填料上,所以即使经过了数次纯化方法的优化,在目标蛋白溶液中仍然残余部分GroES-GroEL蛋白。因此,利用GIS 1D分析软件计算了AcbA的含量为31.51%。
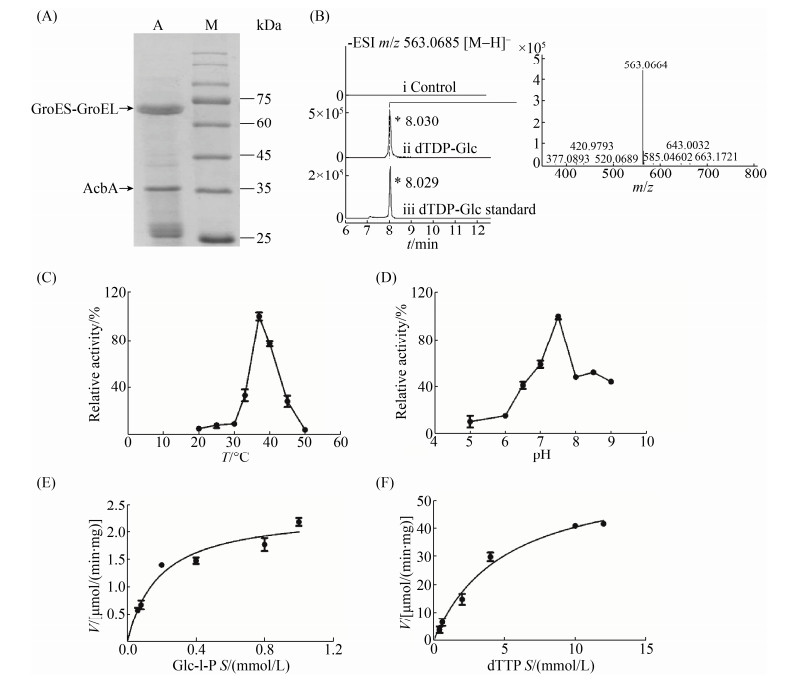 |
| 图 3 AcbA蛋白的功能及酶学性质分析 Figure 3 Analyses of enzymatic function and properties of AcbA. A: SDS-PAGE analysis; B: LC-ESI-MS analysis; C: Effects of different temperatures on enzyme activity; D: Effects of different pHs on enzyme activity; E: Fitted curve of reaction rate and substrate concentration of AcbA at varying D-glucose-1-phosphate concentrations; F: Fitted curve of reaction rate and substrate concentration of AcbA at varying dTTP concentrations. |
| 图选项 |
随后,参考D-葡萄糖-1-磷酸胸腺嘧啶转移酶的反应条件[17],按照1.7.1所述方法,以D-葡萄糖-1-磷酸(Glc-1-P)和dTTP为底物,利用Tris-HCl (pH 8.0)为缓冲液,以Mg2+为辅因子,建立了AcbA的反应体系。在30 ℃反应12 h后,利用2倍体积的甲醇终止反应。采用LC-ESI-MS分析,在8.0 min处出现了产物dTDP-D-葡萄糖(dTDP-Glc)的分子离子峰(C16H26O16N2P2),m/z=563.0664 [M?H]? (计算值m/z=563.0685 [M?H]?),与dTDP-Glc标准品的出峰时间和m/z吻合。而在失活蛋白的对照反应组中,没有出现dTDP-Glc的信号(图 3-B)。该研究结果表明,AcbA可催化D-葡萄糖-1-磷酸和dTTP反应生成dTDP-D-葡萄糖。
2.2.2 AcbA的酶学性质表征: 为系统了解AcbA的酶促反应特性,首先考察了不同温度和pH对其催化活性的影响。研究结果显示,AcbA的相对酶活在20?50 ℃呈现先上升后下降的趋势,在37 ℃达到最大值(图 3-C)。而后,测定了AcbA在pH 5.0?9.0的酶活,结果显示,随着pH的变化,酶活力在pH 7.5时达到最高(图 3-D)。因此,AcbA的最适反应温度和pH分别为37 ℃和7.5。
随后,在AcbA的最适反应条件下,测定了其反应动力学参数。反应体系中AcbA的浓度为20 μmol/L,改变底物D-葡萄糖-1-磷酸或dTTP的添加量进行反应,反应时间为8 h。结合HPLC-QQQMS定量分析,通过检测dTDP-D-葡萄糖的生成量,计算了不同底物添加量的反应速率。而后,利用软件GraphPad Prism 5绘制了Michaelis-Menten拟合曲线(图 3-E、3-F),计算了AcbA对底物D-葡萄糖-1-磷酸和dTTP的Km值分别为(0.185±0.053) mmol/L和(4.964±1.089) mmol/L,Vmax分别为(2.366±0.217)μmol/(min·mg)和(60.310±5.419) μmol/(min·mg) (表 2)。
表 2. AcbA、AcbB和AcbV的动力学参数 Table 2. The enzyme kinetic parameters of AcbA, AcbB and AcbV
| Protein | Substrate | Km/(mmol/L) | Vmax/[μmol/(min·mg)] |
| AcbA | D-glucose-1-phosphate | 0.185±0.053 | 2.366±0.217 |
| dTTP | 4.964±1.089 | 60.310±5.419 | |
| AcbB | dTDP-D-glucose | 0.353±0.089 | 306.401±28.740 |
| AcbV | dTDP-4-keto-6-deoxy-D-glucose | 1.411±0.293 | 3.447±0.279 |
表选项
2.3 AcbB的功能分析及其酶学性质表征
2.3.1 AcbB蛋白的表达及其功能分析: AcbB蛋白为dTDP-D-葡萄糖-4, 6-脱水酶,催化dTDP-D-葡萄糖发生分子内脱水反应。为了详细研究该蛋白的功能,首先进行了蛋白的表达和纯化,经SDS-PAGE分析,获得了大小约为41 kDa的蛋白(图 4-A),并计算了蛋白的含量为63.96%。
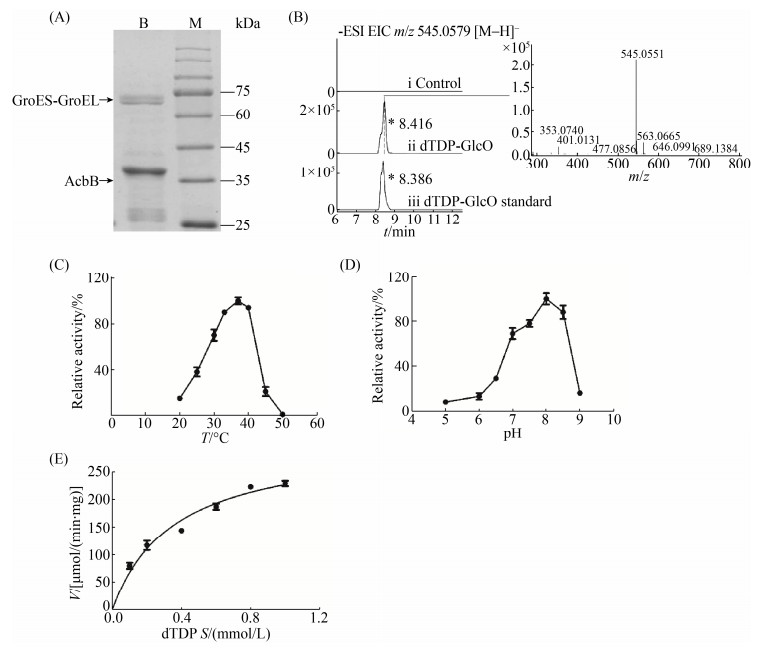 |
| 图 4 AcbB蛋白的功能及酶学性质分析 Figure 4 Analyses of enzymatic function and properties of AcbB. A: SDS-PAGE analysis; B: LC-ESI-MS analysis; C: Effects of different temperatures on enzyme activity; D: Effects of different pHs on enzyme activity; E: Fitted curve of reaction rate and substrate concentration of AcbB at varying dTDP-D-glucose concentrations. |
| 图选项 |
随后,参考dTDP-D-葡萄糖-4, 6-脱水酶的反应条件[25],按照1.7.2所述方法,以dTDP-D-葡萄糖为底物,Tris-HCl (pH 8.0)为缓冲液,加入1 mmol/L的NAD+辅因子和20 μmol/L AcbB,在30 ℃反应1 h后,利用2倍体积的甲醇终止反应。采用LC-ESI-MS检测反应产物的生成情况,发现在8.4 min左右抽提出目标产物dTDP-4-酮基-6-脱氧-D-葡萄糖(dTDP-GlcO)的分子离子峰(C16H24O15N2P2),m/z=545.0551 [M?H]?(计算值m/z=545.0579 [M?H]?),与dTDP-GlcO标准品的出峰时间和m/z一致。而在蛋白失活的对照组中无此产物积累(图 4-B)。该研究结果表明,AcbB作为dTDP-D-葡萄糖-4, 6-脱水酶,催化了dTDP-D-葡萄糖脱水生成dTDP-4-酮基-6-脱氧-D-葡萄糖,为脱氧氨基糖单元合成的第二步反应。
2.3.2 AcbB的酶学性质表征: 为了分析AcbB的酶催化反应特性,首先评估了不同温度和pH对其催化活性的影响。如图 4-C所示,AcbB的相对酶活在20?50 ℃呈现先上升后下降的趋势,在37 ℃时活性达到最大值。而后,测定了AcbB在pH 5.0?9.0的相对酶活,结果如图 4-D所示,随着pH的升高,酶活先不断升高,在pH 8.0时达到最高后呈下降趋势。因此,AcbB在温度和pH分别为37 ℃和8.0时,显示了最高的催化效率。
随后,在最适温度和pH条件下,测定了AcbB的酶动力学参数。在反应体系中分别添加了20 μmol/L的AcbB蛋白、1 mmol/L的辅因子NAD+以及不同浓度的底物dTDP-D-葡萄糖,浓度分别为0.1、0.2、0.4、0.6、0.8、1.0 mmol/L,反应1 h后终止反应。利用HPLC-QQQMS定量检测产物dTDP-4-酮基-6-脱氧-D-葡萄糖的生成量来计算反应速率,并拟合计算出AcbB蛋白的酶动力学参数(图 4-E和表 2),其Km值和Vmax分别为(0.353±0.089) mmol/L和(306.401±28.740)μmol/(min·mg)。
2.4 AcbV的功能分析及其酶学性质表征
2.4.1 AcbV蛋白的表达及其功能分析: AcbV蛋白是转氨酶,负责将来自谷氨酸的氨基转移到dTDP-4-酮基-6-脱氧-D-葡萄糖的C-4位的酮基上,生成dTDP-4-氨基-4, 6-双脱氧-D-葡萄糖。为了研究AcbV的催化功能,首先构建了AcbV的异源表达质粒pLQ1111,然后,将其转化至大肠杆菌BL21(DE3)/pGro7中,进行了蛋白表达条件的优化。经过镍柱亲和层析纯化后,得到了大小约为53 kDa的蛋白(图 5-A),利用GIS 1D软件定量分析其含量为71.99%。
 |
| 图 5 AcbV蛋白的功能及酶学性质分析 Figure 5 Analyses of enzymatic function and properties of AcbV. A: SDS-PAGE analysis; B: LC-ESI-MS analysis; C: Effects of different temperatures on enzyme activity; D: Effects of different pHs on enzyme activity; E: Fitted curve of reaction rate and substrate concentration of AcbV at varying dTDP-4-keto-6-deoxy-D-glucose concentrations. |
| 图选项 |
随后,参考dTDP-4-酮基-6-脱氧-D-葡萄糖转氨酶的反应条件[19],按照1.7.3所述方法,以dTDP-4-酮基-6-脱氧-D-葡萄糖为底物、谷氨酸为氨基供体、磷酸吡哆醛(pyridoxal phosphate,PLP)为辅因子,加入20 mmol/L的AcbV,并置于30 ℃反应6 h后终止反应。利用LC-ESI-MS检测反应产物,发现在9.3 min处出现目标产物dTDP-4-氨基-4, 6-双脱氧-D-葡萄糖(dTDP-Qui4N)的分子离子峰(C16H27N3O14P2),该化合物的质荷比为m/z=546.0878 [M?H]?(计算值为m/z=546.0895 [M?H]?),同时,对照组无此产物积累(图 5-B)。该研究结果表明,AcbV蛋白催化了dTDP-4-酮基- 6-脱氧-D-葡萄糖发生转氨反应,生成了dTDP-4-氨基-4, 6-双脱氧-D-葡萄糖,即阿卡波糖脱氧氨基糖单元合成途径的终产物。
2.4.2 AcbV的酶学性质表征: 为了深入探究AcbV的反应特性,首先考察了不同反应温度和溶液pH对其催化活性的影响。如图 5-C所示,AcbV的相对酶活在20?30 ℃时随温度升高不断提高至最大值,在30?50 ℃呈下降趋势。而后,测定了AcbV在pH 5.0?9.0范围内的相对酶活,结果如图 5-D所示,酶活力在pH 7.5时达到最大值。因此,AcbV的最适反应温度和pH分别为30 ℃和7.5。
随后,为计算AcbV蛋白的酶动力学参数,在最适酶反应条件下,分别添加1 mmol/L PLP、8 mmol/L L-谷氨酸、20 μmol/L AcbV蛋白和底物dTDP-4-酮基-6-脱氧-D-葡萄糖(浓度分别为0.4、0.6、0.8、2.0和8.0 mmol/L)进行反应,6 h后用两倍体积的甲醇终止反应。通过定量检测底物dTDP-4-酮基-6-脱氧-D-葡萄糖的消耗量,计算了AcbV的反应速率。最终,利用软件GraphPad Prism 5拟合计算出AcbV蛋白的Km=(1.411± 0.293) mmol/L,Vmax=(3.447±0.279)μmol/(min·mg) (图 5-E和表 2)。
3 讨论 通过解析天然产物的生物合成机制来定向指导代谢工程改造是天然产物工业化产量提高的重要手段之一。脱氧氨基糖作为阿卡波糖结构中的重要单元,研究其合成过程对于全面解析阿卡波糖的生物合成机制,指导代谢工程改造,从而提高阿卡波糖的产量具有重要意义。本研究通过生物信息学分析,推测了负责SE50/110中阿卡波糖脱氧氨基糖单元合成的相关基因。而后,利用体内基因突变与体外酶学分析相结合的研究策略,系统地解析了阿卡波糖脱氧氨基糖单元的生物合成机制。
通过BLASTp分析,对阿卡波糖生物合成基因簇中编码的蛋白进行了功能预测,发现编码AcbA、AcbB和AcbV三个蛋白的基因可能参与阿卡波糖脱氧氨基糖单元的生物合成。在体内研究中,采用了同源重组无痕敲除的方法,对acbA、acbB和acbV基因的功能进行验证,发现这3个基因均参与了阿卡波糖的生物合成,并结合回补实验确定了这3个基因均为阿卡波糖生物合成的必需基因。在体外实验中,经异源表达、纯化获得了AcbA、AcbB和AcbV的可溶性蛋白,并通过酶促反应鉴定了3个蛋白的功能及酶学特征,证实了阿卡波糖脱氧氨基糖单元的生物合成是以D-葡萄糖-1-磷酸为底物,依次经AcbA (D-葡萄糖-1-磷酸胸腺嘧啶转移酶)、AcbB (dTDP-D-葡萄糖-4, 6-脱水酶)和AcbV (dTDP-4-酮基-6-脱氧-D-葡萄糖转氨酶)催化,分别生成dTDP-D-葡萄糖、dTDP-4-酮基-6-脱氧-D-葡萄糖和dTDP-4-氨基-4, 6-双脱氧-D-葡萄糖,这也表明了在阿卡波糖合成过程中其核心部分组装的糖基底物为dTDP-4-氨基-4, 6-双脱氧-D-葡萄糖。同时,通过逐个摸索酶的最适反应条件,研究了相关蛋白的酶学性质。对于dTDP-D-葡萄糖底物,AcbA蛋白的kcat/Km值为0.355 L/(min·mmol),低于已报道的其他细菌中的同工酶。例如E. coli和Salmonella enterica LT2中的RfbA蛋白,它们的kcat/Km分别为173.960 L/(min·mmol)和16.041 L/(min·mmol)[14-15],说明AcbA的催化能力较低。此外,AcbB蛋白的kcat/Km值为21.390 L/(min·mmol),与S. enterica LT2中RfbB蛋白的kcat/Km 54.861 L/(min·mmol)[26]相近。AcbV蛋白的kcat/Km值为0.046 L/(min·mmol),与S. dysenteriae中VioA蛋白kcat/Km 0.013 L/(min·mmol)[27]相差不大。蛋白的动力学参数评估结果显示,在脱氧氨基糖单元合成的第一步中,D-葡萄糖-1-磷酸胸腺嘧啶转移酶(AcbA)的催化活性较低,这可能是阿卡波糖生物合成过程中的限速步骤。因此,提高AcbA蛋白的胞内表达量,或表达高活性的异源同工蛋白,可能有利于阿卡波糖脱氧氨基糖单元的合成,从而达到提高阿卡波糖产量的目的。
总之,本研究阐释了阿卡波糖脱氧氨基糖单元的生物合成过程,为全面解析阿卡波糖生物合成途径奠定了基础。同时,测定了相关酶的动力学参数,发现了阿卡波糖生物合成中可能存在的限速步骤,为代谢工程改造SE50/110来提高阿卡波糖产量提供了重要依据。
References
| [1] | Campbell LK, White JR, Campbell RK. Acarbose: its role in the treatment of diabetes mellitus. Annals of Pharmacotherapy, 1996, 30(11): 1255-1262. DOI:10.1177/106002809603001110 |
| [2] | Feng ZH, Wang YS, Zheng YG. Progress in biosynthesis pathway of acarbose. Biotechnology Bulletin, 2011(8): 60-67. (in Chinese) 冯志华, 王远山, 郑裕国. 阿卡波糖的生物合成途径研究进展. 生物技术通报, 2011(8): 60-67. |
| [3] | Cho NH, Shaw JE, Karuranga S, Huang Y, da Rocha Fernandes JD, Ohlrogge AW, Malanda B. IDF diabetes atlas: global estimates of diabetes prevalence for 2017 and projections for 2045. Diabetes Research and Clinical Practice, 2018, 138: 271-281. DOI:10.1016/j.diabres.2018.02.023 |
| [4] | Scully T. Diabetes in numbers. Nature, 2012, 485(7398): S2-S3. DOI:10.1038/485S2a |
| [5] | Mahmud T. The C7N aminocyclitol family of natural products. Natural Product Reports, 2003, 20(1): 137-166. DOI:10.1039/b205561a |
| [6] | Wehmeier UF, Piepersberg W. Biotechnology and molecular biology of the α-glucosidase inhibitor acarbose. Applied Microbiology and Biotechnology, 2004, 63(6): 613-625. DOI:10.1007/s00253-003-1477-2 |
| [7] | Wehmeier UF. The biosynthesis and metabolism of acarbose in Actinoplanes sp. SE 50/110: a progress report. Biocatalysis and Biotransformation, 2003, 21(4/5): 279-284. |
| [8] | Yu Z, Li MH, Li N, Zheng LH, Huang J. Effect of treY gene inactivation on reducing of acarbose component C in Actinoplanes 8-22. Microbiology China, 2015, 42(7): 1301-1306. (in Chinese) 余贞, 李美红, 李娜, 郑玲辉, 黄隽. 游动放线菌8-22中treY基因敲除对于降低阿卡波糖C组分的作用. 微生物学通报, 2015, 42(7): 1301-1306. |
| [9] | Gren T, Ortseifen V, Wibberg D, Schneiker-Bekel S, Bednarz H, Niehaus K, Zemke T, Persicke M, Pühler A, Kalinowski J. Genetic engineering in Actinoplanes sp. SE50/110 - development of an intergeneric conjugation system for the introduction of actinophage-based integrative vectors. Journal of Biotechnology, 2016, 232: 79-88. DOI:10.1016/j.jbiotec.2016.05.012 |
| [10] | Wolf T, Gren T, Thieme E, Wibberg D, Zemke T, Pühler A, Kalinowski J. Targeted genome editing in the rare actinomycete Actinoplanes sp. SE50/110 by using the CRISPR/Cas9 system. Journal of Biotechnology, 2016, 231: 122-128. DOI:10.1016/j.jbiotec.2016.05.039 |
| [11] | Zhao QQ, Xie HX, Peng Y, Wang XR, Bai LQ. Improving acarbose production and eliminating the by-product component C with an efficient genetic manipulation system of Actinoplanes sp. SE50/110. Synthetic and Systems Biotechnology, 2017, 2(4): 302-309. DOI:10.1016/j.synbio.2017.11.005 |
| [12] | Liu HW, Thorson JS. Pathways and mechanisms in the biogenesis of novel deoxysugars by bacteria. Annual Review of Microbiology, 1994, 48: 223-256. DOI:10.1146/annurev.mi.48.100194.001255 |
| [13] | Blankenfeldt W, Giraud MF, Leonard G, Rahim R, Creuzenet C, Lam JS, Naismith JH. The purification, crystallization and preliminary structural characterization of glucose-1-phosphate thymidylyltransferase (RmlA), the first enzyme of the dTDP-L-rhamnose synthesis pathway from Pseudomonas aeruginosa. Acta Crystallographica Section D, Biological Crystallography, 2000, 56(11): 1501-1504. DOI:10.1107/S0907444900010040 |
| [14] | Zuccotti S, Zanardi D, Rosano C, Sturla L, Tonetti M, Bolognesi M. Kinetic and crystallographic analyses support a sequential-ordered bi bi catalytic mechanism for Escherichia coli glucose-1-phosphate thymidylyltransferase. Journal of Molecular Biology, 2001, 313(4): 831-843. DOI:10.1006/jmbi.2001.5073 |
| [15] | Lindquist L, Kaiser R, Reeves PR, Lindberg AA. Purification, characterization and HPLC assay of Salmonella glucose-1-phosphate thymidylyltransferase from the cloned rfbA gene. European Journal of Biochemistry, 1993, 211(3): 763-770. DOI:10.1111/j.1432-1033.1993.tb17607.x |
| [16] | Feng L, Tao J, Guo HJ, Xu JG, Li YY, Rezwan F, Reeves P, Wang L. Structure of the Shigella dysenteriae 7 O antigen gene cluster and identification of its antigen specific genes. Microbial Pathogenesis, 2004, 36(2): 109-115. DOI:10.1016/j.micpath.2003.10.003 |
| [17] | Lee HC, Sohng JK, Kim HJ, Nam DH, Han JM, Cho SS, Choi JH, Yoo JC. Cloning and expression of the glucose-1- phosphate thymidylyltransferase gene (gerD) from Streptomyces sp. GERI-155. Molecules and Cells, 2004, 17(2): 274-280. |
| [18] | Sohng JK, Kim HJ, Nam DH, Lim DO, Han JM, Lee HJ, Yoo JC. Cloning, expression, and biological function of a dTDP-deoxyglucose epimerase (gerF) gene from Streptomyces sp. GERI-155. Biotechnology Letters, 2004, 26(3): 185-191. DOI:10.1023/B:BILE.0000013709.80691.97 |
| [19] | Chung YS, Kim DH, Seo WM, Lee HC, Liou K, Oh TJ, Sohng JK. Enzymatic synthesis of dTDP-4-amino-4, 6- dideoxy-D-glucose using GerB (dTDP-4-keto-6-deoxy-D- glucose aminotransferase). Carbohydrate Research, 2007, 342(11): 1412-1418. DOI:10.1016/j.carres.2007.04.007 |
| [20] | Lombó F, Siems K, Bra?a AF, Méndez C, Bindseil K, Salas JA. Cloning and insertional inactivation of Streptomyces argillaceus genes involved in the earliest steps of biosynthesis of the sugar moieties of the antitumor polyketide mithramycin. Journal of Bacteriology, 1997, 179(10): 3354-3357. DOI:10.1128/JB.179.10.3354-3357.1997 |
| [21] | Bechthold A, Sohng AK, Smith TM, Chu X, Floss HG. Identification of Streptomyces violaceoruber Tü22 genes involved in the biosynthesis of granaticin. Molecular and General Genetics, 1995, 248(5): 610-620. DOI:10.1007/BF02423457 |
| [22] | Paget MS, Chamberlin L, Atrih A, Foster SJ, Buttner MJ. Evidence that the extracytoplasmic function sigma factor σE is required for normal cell wall structure in Streptomyces coelicolor A3(2). Journal of Bacteriology, 1999, 181(1): 204-211. DOI:10.1128/JB.181.1.204-211.1999 |
| [23] | Wang WS, Li X, Wang J, Xiang SH, Feng XZ, Yang KQ. An engineered strong promoter for Streptomycetes. Applied and Environmental Microbiology, 2013, 79(14): 4484-4492. DOI:10.1128/AEM.00985-13 |
| [24] | Bierman M, Logan R, O'Brien K, Seno ET, Rao RN, Schoner BE. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp. Gene, 1992, 116(1): 43-49. DOI:10.1016/0378-1119(92)90627-2 |
| [25] | Oh J, Lee SG, Kim BG, Sohng JK, Liou K, Lee HC. One-pot enzymatic production of dTDP-4-keto-6-deoxy-D-glucose from dTMP and glucose-1-phosphate. Biotechnology and Bioengineering, 2003, 84(4): 452-458. DOI:10.1002/bit.10789 |
| [26] | Romana LK, Santiago FS, Reeves PR. High level expression and purification of dThymidine diphospho-D-glucose 4, 6- dehydratase (rfbB) from Salm onella serovar typhimurium LT2. Biochemical and Biophysical Research Communications, 1991, 174(2): 846-852. DOI:10.1016/0006-291X(91)91495-X |
| [27] | Wang L, Wang Y, Xu YL, Qi YY, Feng L. Method for synthesizing dTDP-4-amino-4, 6-dideoxy-D-glucose, preparation method and use thereof. CN: 200710127579.8. 2007-07-03. (in Chinese) 王磊, 王颖, 徐艳丽, 齐源远, 冯露.一种利用生物法合成dTDP-4-氨基-4, 6-双脱氧-D-葡萄糖、制备方法及其用途.中国: 200710127579.8. 2007-07-03. |
