童金蓉1, 张昭寰1,2

 , 黄振华1, 刘海泉1,3,4, 潘迎捷1,3,4, 赵勇1,3,4
, 黄振华1, 刘海泉1,3,4, 潘迎捷1,3,4, 赵勇1,3,4

1. 上海海洋大学食品学院, 上海 201306;
2. 上海海洋大学水产与生命学院, 上海 201306;
3. 农业农村部水产品贮藏保鲜质量安全风险评估实验室(上海), 上海 201306;
4. 上海水产品加工及贮藏工程技术研究中心, 上海 201306
收稿日期:2019-12-10;修回日期:2020-02-26;网络出版日期:2020-06-17
基金项目:国家自然科学基金(31972188,31671779);国家博士后创新人才支持计划(BX20190194);上海市“超级博士后”激励计划(2019348);上海市教育委员会科研创新计划(2017-01-07-00-10-E00056);国家重点研发计划(2018YFC1602205);上海市科技兴农项目(沪农科推字2017第4-4号);中国博士后科学基金(2019M661469)
*通信作者:张昭寰,Tel/Fax:+86-21-61900503,E-mail:gongziwuhen@126.com;
赵勇,Tel/Fax:+86-21-61900768,E-mail:yzhao@shou.edu.cn.
摘要:[目的] 副溶血性弧菌是一种重要的人畜共患病原菌,脂蛋白定位系统(Localization of lipoprotein system,Lol)负责该菌脂蛋白的转运与定位,与其致病力及耐药性密切相关,对Lol系统转运蛋白进行系统的生物信息学分析,有助于推动副溶血性弧菌致病与耐药机理的进一步研究。[方法] 本文通过生物信息学分析技术,结合ExPASy在线工具、SignalP 4.0 Server、TMHMM-2.0、STRING、SWISS-MODEL等软件,分析了副溶血性弧菌Lol系统转运蛋白LolA-E及LolCD2E的基本性质、蛋白互作关系及三级结构。[结果] LolA和LolB为酸性亲水蛋白,含信号肽位点,无跨膜区域。LolC和LolE为碱性疏水膜蛋白,LolCD2E为中性疏水膜蛋白,LolC-E及LolCD2E均无显著的信号肽位点。蛋白相互作用网络显示,LolA-E五个蛋白的编码基因均共表达,负责脂蛋白的合成与转运,并与BamA、Pal、MacB、CmeC等外膜蛋白具有密切的互作关系。三级结构同源建模发现,副溶血性弧菌与大肠杆菌拥有相似的LolA和LolB结构,LolC-E含有MacB蛋白的同源结构,赋予了该系统消耗ATP运输脂蛋白的重要功能。此外,本研究还首次发现了副溶血性弧菌LolC和LolE中存在一段保守的Hook结构,是LolCD2E复合物与LolA结合并转运脂蛋白的关键区域。[结论] 本研究为副溶血性弧菌Lol系统转运蛋白的表达纯化、结构与功能的研究提供了重要的数据基础,为后续抗菌药物的研发提供了新型作用靶点。
关键词:副溶血性弧菌脂蛋白定位系统生物信息学分析蛋白三级结构
Bioinformatics analysis for structure and function of localization of lipoprotein system transporters in Vibrio parahaemolyticus
Jinrong Tong1, Zhaohuan Zhang1,2

 , Zhenhua Huang1, Haiquan Liu1,3,4, Yingjie Pan1,3,4, Yong Zhao1,3,4
, Zhenhua Huang1, Haiquan Liu1,3,4, Yingjie Pan1,3,4, Yong Zhao1,3,4

1. College of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China;
2. College of Fisheries and Life Sciences, Shanghai Ocean University, Shanghai 201306, China;
3. Laboratory of Quality&Safety Risk Assessment for Aquatic Product on Storage and Preservation(Shanghai), Ministry of Agriculture and Rural Affairs, Shanghai 201306, China;
4. Shanghai Engineering Research Center of Aquatic-Product Processing&Preservation, Shanghai 201306, China
Received: 10 December 2019; Revised: 26 February 2020; Published online: 17 June 2020
*Corresponding author: Zhaohuan Zhang , Tel/Fax: +86-21-61900503, E-mail:gongziwuhen@126.com;
Yong Zhao , Tel/Fax: +86-21-61900768, E-mail:yzhao@shou.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31972188, 31671779), by the National Postdoctoral Program for Innovation Talent (BX20190194), by the Shanghai Post-doctoral Excellence Program (2019348), by the Innovation Program of Shanghai Municipal Education Commission (2017-01-07-00-10-E00056), by the National Key Research and Development Program (2018YFC1602205), by the Shanghai Agriculture Applied Technology Development Program (T20170404) and by the China Postdoctoral Science Foundation (2019M661469)
Abstract: [Objective] Vibrio parahaemolyticus is an important zoonotic pathogen. Its localization of lipoprotein (Lol) system is responsible for the transport and localization of lipoproteins in this bacterium, which is closely related to its pathogenicity and drug resistance. Systematic bioinformatics analysis of the Lol system transporters is helpful to promote further research on pathogenesis and resistance mechanism of V. parahaemolyticus. [Methods] The basic properties, protein interactions and tertiary structures of Lol system transporters LolA-E and LolCD2E of V. parahaemolyticus were investigated by bioinformatics analysis, combining with ExPASy online tools, SignalP 4.0 Server, TMHMM-2.0, STRING, SWISS-MODEL and other softwares. [Results] LolA and LolB were acidic hydrophilic proteins with signal peptide sites and no transmembrane region, while LolC and LolE were alkaline hydrophobic membrane proteins. LolCD2E was an neutral hydrophobic membrane protein, and LolC-E and LolCD2E had no significant signal peptide sites. Subsequent studies should add a signal peptide sequence in the recombinant expression vector of LolCD2E protein, and combine with detergent treatment to ensure the protein expression and purification. Protein interaction network showed that the coding genes of five proteins LolA-E can co-express, and be responsible for the synthesis and transport of lipoprotein. LolA-E had close interaction with other outer membrane proteins such as BamA, Pal, MacB and CmeC. Tertiary structural homology modeling showed that V. parahaemolyticus and Escherichia coli had the similar structures in LolA and LolB proteins. And LolC-E contained the homologous structure of MacB protein, which gave Lol system an important function for consuming ATP to transport lipoproteins. Furthermore, this study found a conserved Hook structure in LolC and LolE of V. parahaemolyticus for the first tim, which is a key region for LolCD2E complex to bind with LolA and transport lipoprotein. [Conclusion] This study provides an important data basis for the study of expression purification, structure and function of Lol system transporter in V. parahaemolyticus, and provides new targets for the subsequent research and development of new antibacterials.
Keywords: Vibrio parahaemolyticuslocalization of lipoprotein systembioinformatics analysistertiary structure of protein
副溶血性弧菌(Vibrio parahaemolyticus)是一种重要的人畜共患病原菌[1],广泛分布于水产食品及水产养殖环境中。该菌不仅能够引发腹泻、败血症等严重的食源性疾病[2],对人类健康及食品安全造成了极大的威胁,而且能够感染南美白对虾的幼虾,引起对虾急性肝胰腺坏死病(Acute hepatopancreatic necrosis disease,AHPND)[3],造成了严重的经济损失,制约着水产行业的可持续发展[3]。脂蛋白是副溶血性弧菌细胞外膜的重要组成部分,与其致病机制密切相关,在该菌细胞膜合成、黏附与侵袭、耐药性产生等重要生理过程中发挥着至关重要的作用[4]。
脂蛋白定位系统(Localization of lipoprotein system,Lol)由LolA–E五个蛋白构成[5],主要负责副溶血性弧菌细胞内脂蛋白的转运与定位。现有研究表明,Lol系统通过细胞内膜上的ABC转运体LolCD2E复合物,将脂蛋白转运至周质空间,由周质空间内的伴侣蛋白LolA将其运送至细胞外膜,再通过外膜受体蛋白LolB将其接收并正确定位于细胞外膜内侧[6-8],从而使脂蛋白发挥正常的生理功能。然而,现阶段Lol系统的三级结构还未完全明晰,且大部分研究常基于大肠杆菌[4, 9]、铜绿假单胞菌[10-11]、沙门氏菌[12]等细菌,对于副溶血性弧菌Lol的系统研究尚未展开,大大限制了人们对该菌致病及耐药机制的理解。
因此,本研究通过生物信息学技术,首次分析了副溶血性弧菌Lol系统转运蛋白的理化性质,并对其信号肽及跨膜区进行了预测,进一步基于STRING数据库探究了Lol系统相关蛋白之间的相互作用关系,并通过同源建模技术预测这些蛋白的三级结构,揭示副溶血性弧菌脂蛋白转运过程中关键结构,从生物信息学的角度展示副溶血性弧菌Lol系统转运脂蛋白的核心步骤。本研究可为后续副溶血性弧菌Lol系统转运蛋白的表达纯化、结构解析提供重要的数据基础,为副溶血性弧菌Lol系统功能的进一步揭示提供理论依据。
1 材料和方法 1.1 菌株 在NCBI的Protein数据库中,选取副溶血性弧菌菌株RIMD 2210633为研究对象,该菌株为世界上第一株被测序的副溶血性弧菌菌株,具有完整的基因组信息,广泛应用于揭示副溶血性弧菌的致病机理,被视为副溶血性弧菌相关研究中的模式菌株[13]。下载该菌脂蛋白转运系统Lol相关转运蛋白LolA、LolB、LolC、LolD以及LolE的氨基酸序列,并拼接LolCD2E蛋白复合物的氨基酸序列,用于后续的生物信息学分析。
1.2 Lol系统转运蛋白基本性质分析 使用ExPASy在线软件(http://web.expasy.org/ protparam/)分析了副溶血性弧菌Lol系统转运蛋白LolA、LolB、LolC、LolD、LolE和LolCD2E蛋白复合物的分子结构式、相对分子质量、平均亲水性、理论等电点、摩尔吸光系数等一系列理化性质[14]。
使用SignalP 4.0 Server在线软件(http://www. cbs.dtu.dk/services/SignalP-4.0/)对副溶血性弧菌Lol系统转运蛋白的信号肽进行预测分析[15]。
使用TMHMM-2.0 (http://www.cbs.dtu.dk/ services/TMHMM-2.0/)对副溶血性弧菌Lol系统转运蛋白是否含有跨膜区进行预测,并对跨膜区的个数及所在位置进行分析[16]。
1.3 蛋白互作关系分析 基于副溶血性弧菌Lol系统转运蛋白的氨基酸序列,通过STRING数据库(https://string-db.org/)绘制了蛋白质互作网络图(Protein Protein Interaction Network,PPI),分析了副溶血性弧菌LolA-E蛋白之间的相互作用关系,以及Lol系统相关蛋白之间的互作关系。通过以上蛋白的注释、结构及结构域、基因共表达、邻接、共生等信息,分析副溶血性弧菌Lol系统转运蛋白的功能[17]。
1.4 蛋白结构与功能分析 进一步运用SWISS-MODEL在线数据库(https://swissmodel.expasy.org/)对副溶血性弧菌Lol系统转运蛋白进行同源建模分析,预测并构建了LolA-E的三级结构[18],并着重分析了副溶血性弧菌LolA与LolC之间发挥脂蛋白转运功能的关键的Hook结构[6]。
2 结果和分析 2.1 副溶血性弧菌Lol系统转运蛋白基本性质
2.1.1 理化性质: 利用ExPASy软件分析了副溶血性弧菌Lol系统转运蛋白LolA、LolB、LolC、LolD、LolE以及LolCD2E复合物的理化性质,结果如表 1所示。LolA蛋白由208个氨基酸构成,分子式为C1055H1621N275O321S3,相对分子质量为23389.31 Da。LolB蛋白由212个氨基酸构成,分子式为C1084H1709N285O319S6,相对分子质量为24030.57 Da。LolC蛋白由405个氨基酸构成,分子式为C1966H3155N521O555S22,相对分子质量为43676.16 Da。LolD蛋白由235个氨基酸构成,分子式为C1121H1834N322O349S9,相对分子质量为25695.38 Da。LolE蛋白由414个氨基酸构成,分子式为C2042H3276N540O567S13,相对分子质量为44880.53 Da。LolCD2E蛋白由1289个氨基酸构成,分子式为C6250H10093N1705O1817S53,相对分子质量为139893.40 Da。
表 1. 副溶血性弧菌Lol系统转运蛋白的理化性质分析 Table 1. Physical and chemical properties of Lol system transporters in Vibrio parahaemolyticus
| Protein | Number of amino acids | Molecular formula | Relative molecular weight | The total number of atoms | Theoretical isoelectric point | Molar absorption coefficient | Mean hydrophilicity |
| LolA | 208 | C1055H1621N275O321S3 | 23389.31 | 3275 | 5.19 | 1.195 | –0.396 |
| LolB | 212 | C1084H1709N285O319S6 | 24030.57 | 3403 | 6.19 | 1.683 | –0.194 |
| LolC | 405 | C1966H3155N521O555S22 | 43676.16 | 6219 | 8.89 | 0.777 | 0.393 |
| LolD | 235 | C1121H1834N322O349S9 | 25695.38 | 3635 | 5.71 | 0.174 | –0.157 |
| LolE | 414 | C2042H3276N540O567S13 | 44880.53 | 6438 | 9.40 | 1.011 | 0.386 |
| LolCD2E | 1289 | C6250H10093N1705O1817S53 | 139893.40 | 19918 | 6.90 | 0.633 | 0.190 |
表选项
进一步分析显示:LolA、LolB和LolD蛋白呈现酸性,理论等电点(pI值)分别为5.19、6.19和5.71,LolCD2E蛋白复合物偏中性,pI值为6.90,LolC和LolE蛋白为碱性蛋白,pI值分别为8.89和9.40。LolA–E及LolCD2E的摩尔吸光系数分别为:1.195、1.683、0.777、0.174、1.011和0.633。明晰这些蛋白的等电点和摩尔吸光系数,有助于后续这些蛋白提取与纯化方法的构建与优化[19]。
此外,LolA、LolB和LolD蛋白平均亲水性 < 0,为亲水性蛋白,其余为疏水性蛋白。疏水性蛋白有可能含有跨膜结构[20],因此,本研究进一步分析了Lol系统转运蛋白的信号肽和跨膜结构。
2.1.2 信号肽分析: 号肽通常位于分泌蛋白和膜蛋白氨基酸序列的N端,由16到30个氨基酸残基组成[21]。信号肽的主要作用是促进蛋白分泌到细胞外,信号肽的有无直接决定着重组蛋白表达与纯化方式的选择[22]。因此,本研究通过在线数据库SignalP 4.0 Server,根据默认的神经网络模型(Neural networks)和隐马尔可夫模型(Hidden Markov models)对副溶血性弧菌Lol转运系统转运蛋白进行信号肽分析。结果如图 1所示,LolA和LolB蛋白含有信号肽切割位点,信号肽分值(S-score)的峰值分别位于第20位氨基酸和第25位氨基酸处。而LolC、LolD、LolE及LolCD2E均无显著的信号肽切割位点。
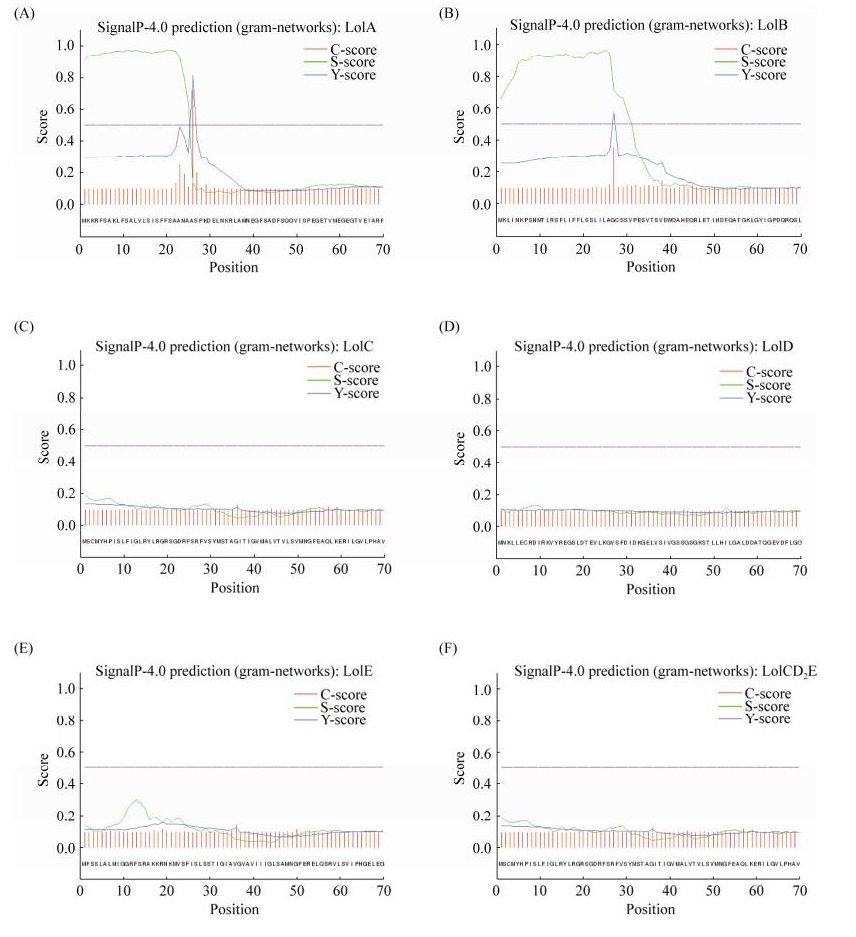 |
| 图 1 副溶血性弧菌Lol系统转运蛋白的信号肽预测 Figure 1 Signal peptide prediction of Lol system transporters in Vibrio parahaemolyticus. A: LolA; B: LolB; C: LolC; D: LolD; E: LolE; F: LolCD2E. |
| 图选项 |
2.1.3 跨膜区分析: 使用在线分析软件TMHMM-2.0对Lol系统转运蛋白的跨膜方式进行分析,结果如图 2所示,LolA、LolB和LolD蛋白结构中无明显的跨膜区域。LolC蛋白含有5个跨膜区,其跨膜螺旋区(TMhelix)预测位置分别在29–51、272–294、314–336、343–365、369–391的氨基酸处。LolE蛋白含有4个跨膜区,其跨膜螺旋区(TMhelix)预测位置分别在23–45、267–289、318–340、375–397的氨基酸处;LolCD2E蛋白含有9个跨膜区,其跨膜螺旋区(TMhelix)预测位置分别在29–51、272–294、314–336、343–365、369–391、898–920、1142–1164、1193–1215和1250–1272的氨基酸处。
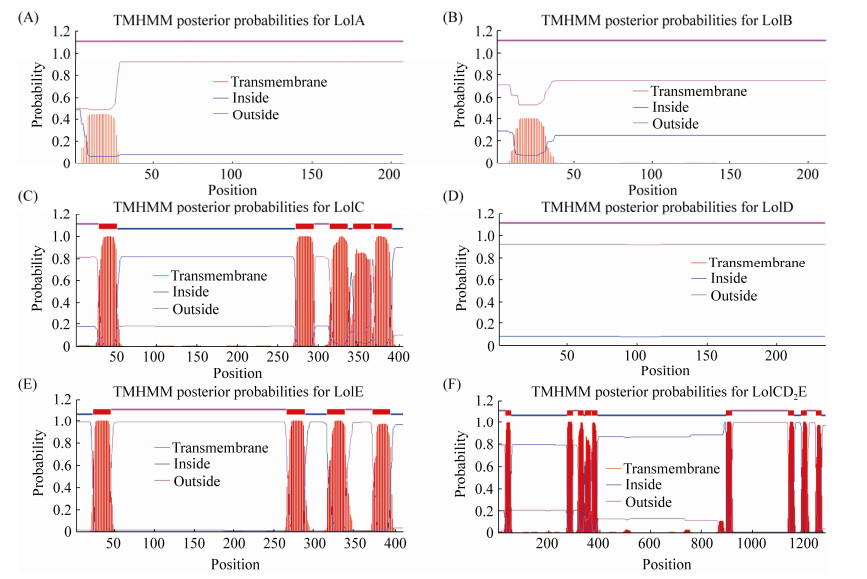 |
| 图 2 副溶血性弧菌Lol系统转运蛋白的跨膜区域预测 Figure 2 Prediction of transmembrance region of Lol system transporters in Vibrio parahaemolyticus. A: LolA; B: LolB; C: LolC; D: LolD; E: LolE; F: LolCD2E. |
| 图选项 |
2.2 互作网络分析 利用STRING数据库,对副溶血性弧菌Lol系统相关蛋白的相互作用网络进行分析,结果如图 3所示。每个圆形的节点代表一种蛋白,节点之间的连线表示两个蛋白之间存在相互作用关系,不同颜色的连线表示不同的相互作用途径,两种蛋白之间的连线越丰富,表示这两种蛋白间具有更为密切的相互作用关系[23]。对于Lol系统转运蛋白之间的相互作用分析可知,LolA–E五个蛋白的编码基因均共表达,负责脂蛋白的合成与转运。LolC、LolE和LolD位于副溶血性弧菌的细胞内膜上,共同组成LolCD2E蛋白复合物,其中LolD是脂蛋白转运系统ATP结合蛋白,通过消耗ATP的方式,将脂蛋白运输通过细胞内膜。LolA为伴侣蛋白,参与脂蛋白从细胞内膜到细胞外膜的转运。LolB是一种外膜脂蛋白,在脂蛋白被LolA蛋白释放后与外膜结合的过程中起关键作用。此外,STRING蛋白相互作用网络显示,Lol系统转运蛋白LolA-E与BamA、Pal、MacB、CmeC等蛋白均具有密切的互作关系。
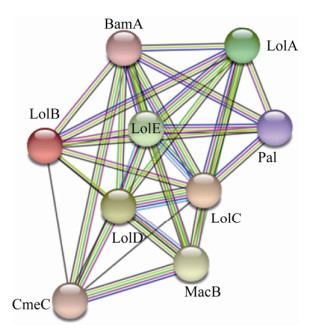 |
| 图 3 副溶血性弧菌Lol系统转运蛋白的蛋白互作网络 Figure 3 Protein interaction network of Lol system transporters in Vibrio parahaemolyticus. |
| 图选项 |
2.3 三级结构预测与分析 利用SWISS-MODEL在线数据库对副溶血性弧菌Lol系统转运蛋白进行同源建模,结果显示,副溶血性弧菌LolA蛋白与其同源蛋白大肠杆菌(Escherichia coli) LolA[6]相似度为43%,LolB与E. coli外膜脂蛋白受体LolB[24]序列相似度为35%。LolC、LolD与LolE均依据放线共生放线杆菌(Aggregatibacter actinomycetemcomitans) ATP结合蛋白MacB结构进行同源建模[25],相似度分别为29%、40%和29%。选取以上蛋白的结构作为模板,预测了副溶血性弧菌LolA–E三级结构预测模型,并结合上文对Lol系统的功能分析,模拟了副溶血性弧菌Lol系统转运蛋白在细胞膜上的位置分布,及运输脂蛋白的过程(图 4)。
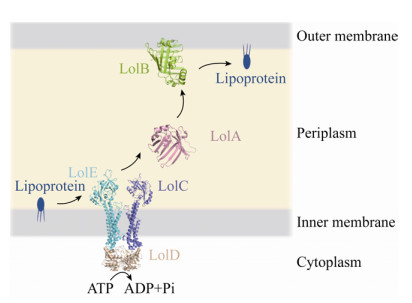 |
| 图 4 副溶血性弧菌Lol系统转运蛋白的三级结构预测模型在细胞膜上的位置分布 Figure 4 Location distribution of the prediction models of tertiary structure for Lol system transporters in Vibrio parahaemolyticus. It represents the process of transport lipoprotein by LolA–E transporters. |
| 图选项 |
在大肠杆菌、霍乱弧菌、沙门氏菌、鼠疫耶尔森菌、流感嗜血杆菌、铜绿假单胞菌等细菌的Lol系统中,LolC和LolE蛋白中存在一段特殊的Hook结构,是LolCD2E复合物与LolA结合并转运脂蛋白的关键区域[6],但尚未有研究揭示副溶血性弧菌是否存在类似的结构。本研究以其他革兰氏阴性菌Hook结构为基础,比对了副溶血性弧菌与其他革兰氏阴性菌LolC和LolE蛋白的氨基酸序列,结果如图 5-A所示,副溶血性弧菌LolC和LolE蛋白中均存在一段保守的Hook结构,位于2个β折叠的序列之间,LolC的Hook结构在第172位与第182位氨基酸之间,LolE的Hook结构在第172位与第181位氨基酸之间。并进一步结合副溶血性弧菌LolC与LolE蛋白的同源建模结果,绘制了Hook区域的三维结构,结果如图 5-B所示,该Hook区域呈现特有的β-发夹结构,使LolCD2E具有结合并转运脂蛋白的特殊功能。
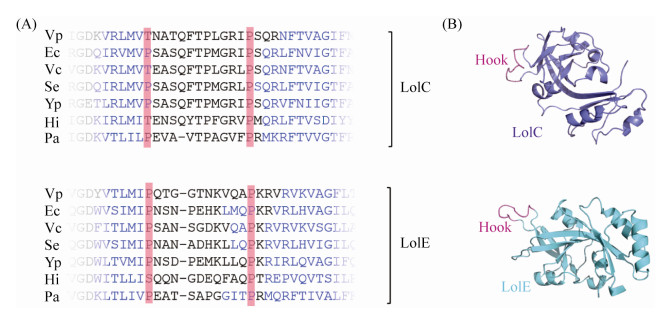 |
| 图 5 副溶血性弧菌LolC和LolE蛋白Hook区域氨基酸序列及三维结构 Figure 5 Bioinformation and tertiary structural information of LolC and LolE Hooks. A: Multiple sequence alignment of LolC and LolE Hooks. Proline residues flanking the Hook are highlighted in pink, and predicted β-sheets in blue. Vp: Vibrio parahaemolyticus; Ec: Escherichia coli; Vc: Vibrio cholerae; Se: Salmonella enterica serovar Typhimurium; Yp: Yersinia pestis; Hi: Haemophilus influenzae; Pa: Pseudomonas aeruginosa. B: Tertiary structural information of LolC and LolE. LolC and LolE are shown in purpleblue and cyans respectievely. LolC and LolE Hooks are both shown in magenta. |
| 图选项 |
3 讨论 本研究首次针对副溶血性弧菌Lol系统转运蛋白相关的氨基酸序列进行了系统的生物信息学分析。首先分析了副溶血性弧菌Lol系统转运蛋白的基本性质,LolA、LolB和LolD为酸性亲水蛋白,无跨膜区域,研究显示LolB是位于外膜上的脂蛋白受体蛋白,因此LolB可能是通过特定锚定方式与外膜结合[26-27]。LolC和LolE为碱性疏水蛋白,包含4–5个跨膜区,LolCD2E为中性疏水蛋白复合物,包含9个跨膜区。LolA和LolB蛋白含有信号肽切割位点,LolC、LolD、LolE及LolCD2E均无显著的信号肽切割位点。因此,在后续该菌LolA–E、LolCD2E的重组表达载体设计中,应根据实验的需要添加或删除信号肽区域,以保证蛋白的正常表达。并针对LolC、LolE、LolCD2E这3个具有跨膜区域的膜蛋白,结合去垢剂等物质进行处理,以确保此类蛋白正确地提取与纯化。
进一步通过蛋白质-蛋白质互作网络分析阐述了副溶血性弧菌Lol系统相关蛋白互作关系,为后续副溶血性弧菌Lol系统转运蛋白功能的研究提供理论参考。通过分析STRING数据库提供注释信息可知,LolA–E五个蛋白的编码基因均共表达,负责脂蛋白的合成与转运。LolC、LolE和LolD共同组成LolCD2E蛋白复合物,位于副溶血性弧菌的细胞内膜上,拥有类似于MacB的周质核心区域结构。此类结构的存在,赋予了副溶血性弧菌Lol系统消耗ATP运输脂蛋白的重要功能。LolA为周质空间内的伴侣蛋白,负责将脂蛋白从细胞内膜转运到细胞外膜上,再通过外膜受体蛋白LolB将其接收并正确定位于细胞外膜内侧。
此外,蛋白相互作用网络还显示,副溶血性弧菌Lol系统与BamA、Pal、MacB、CmeC等外膜蛋白均具有密切的互作关系。其中,BamA蛋白为外膜蛋白装配系统的重要组成部分[28],Pal蛋白参与细菌外膜肽聚糖的定位[29]。MacB蛋白含有特定ABC转运体结构域,能够与Lol系统共同表达[30]。CmeC为重要外膜外排泵蛋白,含有OEP家族特定结构域,能够形成三聚体通道,允许革兰氏阴性菌输出多种底物[31]。由此可见,副溶血性弧菌的Lol系统不仅负责细胞脂蛋白的合成与转运,还在细胞外膜结构的组装、细菌生命活动的调控中发挥着重要的作用。
最后,本研究预测并分析了副溶血性弧菌Lol系统转运蛋白的三级结构,发现其具有与大肠杆菌相似的LolA和LolB结构,推测其可能拥有相似的脂蛋白转运机制[32]。副溶血性弧菌的LolC、LolD和LolE蛋白均含有MacB的同源结构,与Wang等[33]以及Khwaja等[34]的结论相符,也与上述STRING数据库注释相一致,进一步证明了LolCD2E可能和MacB具有类似的通过消耗ATP转运物质的机制[35]。此外,先前研究表明,大肠杆菌、沙门氏菌等革兰氏阴性菌的LolC和LolE蛋白中包含一段特殊的Hook结构,被视为LolCD2E蛋白复合物与LolA蛋白识别并转运脂蛋白过程中的关键结构[6]。本研究Lol系统转运蛋白结构分析显示,副溶血性弧菌LolC和LolE蛋白中也存在这样一段保守的Hook结构,揭示了副溶血性弧菌Lol系统转运脂蛋白的关键区域。若将该Hook结构进行破坏或阻断,可终止副溶血性弧菌脂蛋白的转运,从而抑制副溶血性弧菌的生长与繁殖。后续研究可针对副溶血性弧菌LolC和LolE蛋白中的Hook结构进行开展,进一步探究其脂蛋白转运功能,并以此为靶标研发新型抗菌类药物。
References
| [1] | Wang RZ, Zhong YF, Gu XS, Yuan J, Saeed AF, Wang SH. The pathogenesis, detection, and prevention of Vibrio parahaemolyticus. Frontiers in Microbiology, 2015, 6: 144. |
| [2] | Hubbard TP, Chao MC, Abel S, Blondel CJ, Abel zur Wiesch P, Zhou XH, Davis BM, Waldor MK. Genetic analysis of Vibrio parahaemolyticus intestinal colonization. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(22): 6283-6288. DOI:10.1073/pnas.1601718113 |
| [3] | Xiao JZ, Liu LY, Ke YY, Li XF, Liu YF, Pan YJ, Yan SL, Wang YJ. Shrimp AHPND-causing plasmids encoding the PirAB toxins as mediated by pirAB-Tn903 are prevalent in various Vibrio species. Scientific Reports, 2017, 7(1): 42177. DOI:10.1038/srep42177 |
| [4] | Kovacs-Simon A, Titball RW, Michell SL. Lipoproteins of bacterial pathogens. Infection and Immunity, 2011, 79(2): 548-561. DOI:10.1128/IAI.00682-10 |
| [5] | Konovalova A, Silhavy TJ. Outer membrane lipoprotein biogenesis:Lol is not the end. Philosophical Transactions of the Royal Society B:Biological Sciences, 2015, 370(1679): 20150030. DOI:10.1098/rstb.2015.0030 |
| [6] | Kaplan E, Greene NP, Crow A, Koronakis V. Insights into bacterial lipoprotein trafficking from a structure of LolA bound to the LolC periplasmic domain. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(31): E7389-E7397. DOI:10.1073/pnas.1806822115 |
| [7] | Chen GZ, Zhang YJ, Chen SY. Functions of Lol system proteins and surface-exposed mechanisms of lipoproteins in Gram-negative bacteria. Acta Microbiologica Sinica, 2017, 57(12): 1769-1777. (in Chinese) 陈国忠, 张燕娇, 陈师勇. 革兰氏阴性菌脂蛋白Lol系统转运蛋白的功能及表面展示分泌机制. 微生物学报, 2017, 57(12): 1769-1777. |
| [8] | Okuda S, Tokuda H. Lipoprotein sorting in bacteria. Annual Review of Microbiology, 2011, 65: 239-259. DOI:10.1146/annurev-micro-090110-102859 |
| [9] | Nickerson NN, Jao CC, Xu YM, Quimn J, Skippington E, Alexander MK, Miu A, Skelton N, Hankins JV, Lopez MS, Koth CM, Rutherford S, Nishiyama M. A novel inhibitor of the LolCDE ABC transporter essential for lipoprotein trafficking in Gram-negative bacteria. Antimicrobial Agents and Chemotherapy, 2018, 62(4): e02151-17. |
| [10] | Lorenz C, Dougherty TJ, Lory S. Correct sorting of lipoproteins into the inner and outer membranes of Pseudomonas aeruginosa by the Escherichia coli LolCDE transport system. mBio, 2019, 10(2): e00194-19. |
| [11] | Tanaka SY, Narita SI, Tokuda H. Characterization of the Pseudomonas aeruginosa Lol system as a lipoprotein sorting mechanism. Journal of Biological Chemistry, 2007, 282(18): 13379-13384. DOI:10.1074/jbc.M611840200 |
| [12] | Naser IB, Hoque MM, Nahid MA, Tareq TM, Rocky MK, Faruque SM. Analysis of the CRISPR-Cas system in bacteriophages active on epidemic strains of Vibrio cholerae in Bangladesh. Scientific Reports, 2017, 7(1): 14880. DOI:10.1038/s41598-017-14839-2 |
| [13] | Makino K, Oshima K, Kurokawa K, Yokoyama K, Uda T, Tagomori K, Iijima Y, Najima M, Nakano M, Yamashita A, Kubota Y, Kimura S, Yasunaga T, Honda T, Shinagawa H, Hattori M, Iida T. Genome sequence of Vibrio parahaemolyticus: a pathogenic mechanism distinct from that of V. cholerae. The Lancet, 2003, 361(9359): 743-749. |
| [14] | Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E, Duvaud S, Flegel V, Fortier A, Gasteiger E, Grosdidier A, Hernandez C, Ioannidis V, Kuznetsov D, Liechti R, Moretti S, Mostaguir K, Redaschi N, Rossier G, Xenarios I, Stockinger H. ExPASy:SIB bioinformatics resource portal. Nucleic Acids Research, 2012, 40(W1): W597-W603. DOI:10.1093/nar/gks400 |
| [15] | Zamani M, Nezafat N, Negahdaripour M, Dabbagh F, Ghasemi Y. In silico evaluation of different signal peptides for the secretory production of human growth hormone in E. coli.. International Journal of Peptide Research and Therapeutics, 2015, 21(3): 261-268. DOI:10.1007/s10989-015-9454-z |
| [16] | Chen QM, Cheng DJ, Liu SP, Ma ZG, Tan X, Zhao P. Genome-wide identification and expression profiling of the fatty acid desaturase gene family in the silkworm, Bombyx mori. Genetics and Molecular Research, 2014, 13(2): 3747-3760. DOI:10.4238/2014.May.13.2 |
| [17] | Mei SY. In silico enhancing M. tuberculosis protein interaction networks in STRING to predict drug-resistance pathways and pharmacological risks.. Journal of Proteome Research, 2018, 17(5): 1749-1760. DOI:10.1021/acs.jproteome.7b00702 |
| [18] | Mazaheri S, Nafian DF. Modeling of membrane protein serpentin receptor with SWISS MODEL software. Clinical Biochemistry, 2011, 44(13): S321. |
| [19] | Kubicek J, Block H, Maertens B, Spriestersbach A, Labahn J. Expression and purification of membrane proteins. Methods in Enzymology, 2014, 541: 117-140. DOI:10.1016/B978-0-12-420119-4.00010-0 |
| [20] | Qiu JD, Rong LQ, Liang RP, Tan XC, Zou XY, Mo JY. Prediction of transmembrane protein structure based on hydrophobicity wavelet analysis. Journal of Instrumental Analysis, 2004, 23(6): 1-4, 8. (in Chinese) 邱建丁, 荣联清, 梁汝萍, 谭学才, 邹小勇, 莫金垣. 基于疏水性小波分析的膜蛋白结构预测. 分析测试学报, 2004, 23(6): 1-4, 8. |
| [21] | Owji H, Nezafat N, Negahdaripour M, Hajiebrahimi A, Ghasemi Y. A comprehensive review of signal peptides:Structure, roles, and applications. European Journal of Cell Biology, 2018, 97(6): 422-441. DOI:10.1016/j.ejcb.2018.06.003 |
| [22] | Zheng B, Zhan XM. Signal peptide sequence and its application to protein expression. Letters in Biotechnology, 2005, 16(3): 296-298. (in Chinese) 郑斌, 詹希美. 信号肽序列及其在蛋白质表达中的应用. 生物技术通讯, 2005, 16(3): 296-298. |
| [23] | Wang YG, Yang GR, Leng FF, Yang MJ, Wang MG, Chen K. Bioinformatic analysis of three membrane proteins in Escherichia coli MG1655. Genomics and Applied Biology, 2018, 37(11): 4763-4768. (in Chinese) 王永刚, 杨光瑞, 冷非凡, 杨明俊, 王鸣刚, 陈凯. 大肠杆菌MG1655三个膜蛋白的生物信息学分析. 基因组学与应用生物学, 2018, 37(11): 4763-4768. |
| [24] | Takeda K, Miyatake H, Yokota N, Matsuyama S, Tokuda H, Miki K. Crystal structures of bacterial lipoprotein localization factors, LolA and LolB. The EMBO Journal, 2003, 22(13): 3199-3209. DOI:10.1093/emboj/cdg324 |
| [25] | Crow A, Greene NP, Kaplan E, Koronakis V. Structure and mechanotransmission mechanism of the MacB ABC transporter superfamily. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(47): 12572-12577. DOI:10.1073/pnas.1712153114 |
| [26] | Tanaka K, Matsuyama SI, Tokuda H. Deletion of lolB, encoding an outer membrane lipoprotein, is lethal for Escherichia coli and causes accumulation of lipoprotein localization intermediates in the periplasm. Journal of Bacteriology, 2001, 183(22): 6538-6542. DOI:10.1128/JB.183.22.6538-6542.2001 |
| [27] | Grabowicz M, Silhavy TJ. Redefining the essential trafficking pathway for outer membrane lipoproteins. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(18): 4769-4774. DOI:10.1073/pnas.1702248114 |
| [28] | Doyle MT, Bernstein HD. Bacterial outer membrane proteins assemble via asymmetric interactions with the BamA β-barrel. Nature Communications, 2019, 10(1): 3358. DOI:10.1038/s41467-019-11230-9 |
| [29] | Bouveret E, Bénédetti H, Rigal A, Loret E, Lazdunski C. In vitro characterization of peptidoglycan-associated lipoprotein (PAL)-peptidoglycan and PAL-TolB interactions. Journal of Bacteriology, 1999, 181(20): 6306-6311. DOI:10.1128/JB.181.20.6306-6311.1999 |
| [30] | Xu YB, Sim SH, Nam KH, Jin XL, Kim HM, Hwang KY, Lee K, Ha NC. Crystal structure of the periplasmic region of MacB, a noncanonic ABC transporter. Biochemistry, 2009, 48(23): 5218-5225. DOI:10.1021/bi900415t |
| [31] | Johnson JM, Church GM. Alignment and structure prediction of divergent protein families:periplasmic and outer membrane proteins of bacterial efflux pumps. Journal of Molecular Biology, 1999, 287(3): 695-715. DOI:10.1006/jmbi.1999.2630 |
| [32] | Okuda S, Tokuda H. Model of mouth-to-mouth transfer of bacterial lipoproteins through inner membrane LolC, periplasmic LolA, and outer membrane LolB. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(14): 5877-5882. DOI:10.1073/pnas.0900896106 |
| [33] | Wang B, Dukarevich M, Sun EI, Yen MR, Saier MH Jr. Membrane porters of ATP-binding cassette transport systems are polyphyletic. Journal of Membrane Biology, 2009, 231(1): 1. |
| [34] | Khwaja M, Ma QH, Saier MH. Topological analysis of integral membrane constituents of prokaryotic ABC efflux systems. Research in Microbiology, 2005, 156(2): 270-277. DOI:10.1016/j.resmic.2004.07.010 |
| [35] | Yasuda M, Iguchi-Yokoyama A, Matsuyama SI, Tokuda H, Narita SI. Membrane topology and functional importance of the periplasmic region of ABC transporter LolCDE. Bioscience, Biotechnology, Biochemistry, 2009, 73(10): 2310-2316. DOI:10.1271/bbb.90451 |
