 ,1, 董志平
,1, 董志平 ,2
,2Associated Loci Detection and Elite Allelic Variations Analysis of Main Agronomic Traits in Foxtail Millet (Setaria italica L.) Based on SSR Markers
LI JianFeng1, ZHANG Bo1, QUAN JianZhang2, WANG YongFang2, ZHANG XiaoMei1, ZHAO Yuan1, YUAN XiLei1, JIA XiaoPing ,1, DONG ZhiPing
,1, DONG ZhiPing ,2
,2通讯作者:
责任编辑: 李莉
收稿日期:2019-05-28接受日期:2019-06-25网络出版日期:2019-12-16
| 基金资助: |
Received:2019-05-28Accepted:2019-06-25Online:2019-12-16
作者简介 About authors
李剑峰,E-mail:1299569468@qq.com。

摘要
关键词:
Abstract
Keywords:
PDF (3107KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李剑峰, 张博, 全建章, 王永芳, 张小梅, 赵渊, 袁玺垒, 贾小平, 董志平. 基于SSR标记的谷子主要农艺性状关联位点检测及等位变异分析[J]. 中国农业科学, 2019, 52(24): 4453-4469 doi:10.3864/j.issn.0578-1752.2019.24.002
LI JianFeng, ZHANG Bo, QUAN JianZhang, WANG YongFang, ZHANG XiaoMei, ZHAO Yuan, YUAN XiLei, JIA XiaoPing, DONG ZhiPing.
0 引言
【研究意义】谷子(Setaria italica Beauv.)属禾本科一年生草本植物,具有抗旱、耐贫瘠、高光合效率等特点,籽粒和谷草均有较高的营养价值,粮饲兼用,是中国北方干旱半干旱地区重要的杂粮作物[1]。然而作为一种短日照喜温作物,谷子对光温反应敏感,生产上跨区种植的广适应性品种较缺少,开展谷子广生态适应性相关QTL定位研究,可以揭示谷子生态适应性形成的遗传基础,为利用标记辅助选择育种技术选育广生态适应性品种奠定基础,具有重要理论价值和实践意义。【前人研究进展】目前,有关谷子生态适应性的报道极少,部分****对光周期和环境温度2个影响谷子生态适应性的主要因素开展了研究,如MARGARITA等[2]以开花期为指标性状开展了谷子光周期敏感性位点的QTL定位研究,鉴定了8个不同环境下控制开花期的一些QTL;DOUST等[3,4]先定位了控制谷子分蘖的4个QTL和控制分枝的4个QTL,后又通过QTL共聚焦定位了控制谷子开花的一些QTL,并得到候选基因包括开花位点T(FT)同源物;ZHANG等[5]在2个光周期条件下对14个农艺性状进行了QTL定位和遗传效应分析,长日照和短日照下的的14个农艺性状共鉴定出59个QTL;JIA等[6]对5个不同地理环境下的960个品种进行表型分析,通过全基因组关联分析确定了与47个农艺性状相关联的52个基因位点;NI等[7]发现控制抽穗期的3个QTL;谢丽莉[8]整合了94个长短日照环境下光周期敏感相关性状QTL,获得了8个一致性QTL。此外,有关谷子光温敏感性评价指标的研究近期也有报道,如贾小平等[9,10]利用建立光温敏感性指数进行回归分析和光温相对敏感度比较2种方法筛选谷子光温敏感性评价指标,筛选出抽穗期、穗长、穗码数、叶片数4个适合谷子光温敏感性评价的性状指标,为开展谷子光温敏感性遗传规律分析及QTL定位研究奠定了基础。上述QTL定位研究多数是基于2个亲本杂交产生的分离群体进行的,发掘的变异位点有限,且所用的SNP标记也存在成本高、不方便不同图谱间进行整合等缺点。此外定位所选择的性状主要局限于开花期,事实上除了开花期,其他的农艺性状对不同环境条件也存在不同程度的敏感性[9,10],因此,在不同地理环境下开展多个性状的QTL定位研究能够更充分揭示谷子生态适应性的遗传基础。SSR(simple sequence repeat)作为第二代分子标记,具有重复性好、多态性高、变异丰富、呈共显性且广泛分布于植物基因组等优点,已被广泛应用于高粱[11,12]、大麦[13]、小麦[14]、青稞[15]、水稻[16]、大豆[17,18]、甘蔗[19]和玉米[20]等作物遗传多样性分析和基因发掘研究。相比SNP标记,SSR标记的优势是成本低、试验技术简单,但也存在分布密度低、不能将目标基因精细定位的缺点,因此,可以利用SSR标记对基因进行初步定位,然后在定位区间开发新的SNP、Indel标记进行精细定位,充分利用各自的优势。目前,谷子全基因组序列已经测定并公布到公共数据库[21,22],利用这些序列信息已开发了大量SSR标记[23,24,25,26,27],这些标记已经广泛用于谷子遗传多样性研究[28,29,30,31,32]、基于杂交分离群体的农艺性状QTL定位[33,34,35,36,37,38]。基于连锁不平衡的关联分析法以自然群体为对象来定位目标性状,获得的等位变异较基于双亲杂交分离群体定位法更丰富,GUPTA等[39]首次开展了谷子SSR标记与农艺性状的关联分析,获得了与9个农艺性状显著相关的8个SSR标记。【本研究切入点】作物的生态适应性受光周期、环境温度、土壤水肥状况等多种因素影响,揭示作物生态适应性形成的遗传机制,是开展广生态适应性分子育种的前提和基础。谷子作为一个理想的C4作物模型,全基因组序列已经测定,海量SSR标记的开发为开展生态适应性相关QTL定位研究提供了保障,而对谷子生态适应性形成遗传基础的揭示对其他C4作物生态适应性形成机理的阐明也有重要的借鉴作用。虽然近些年谷子农艺性状定位的研究已经大量报道,但是多数研究选择的地理环境有限、环境间光周期、温度等环境条件差异不明显,此外这些研究主要基于双亲杂交产生的分离群体开展定位,检测到的等位变异数目有限,不能充分揭示谷子生态适应性相关的QTL位点。【拟解决的关键问题】本研究在海南省乐东县、河南省洛阳市、吉林省吉林市和公主岭市4个光周期、温度、土壤条件、水肥条件存在差异的环境连续2年调查102份谷子资源的10个农艺性状,用70对多态性SSR标记对谷子资源进行基因型鉴定,开展SSR标记与10个农艺性状关联分析,发掘不同地理环境与表型性状显著关联的SSR标记,揭示谷子生态适应性形成的遗传基础。1 材料与方法
1.1 谷子材料及种植方法
102份谷子资源包括95份来自河南、河北、山东、内蒙古、北京、陕西、黑龙江几个国内地区的材料以及7份来自朝鲜、法国、印度、日本、美国、德国的国外材料(电子附表1)。2015年5月中旬至10月中旬,2016年5月中旬至10月中旬将102份谷子种植于地理环境Ⅰ:河南科技大学试验田(洛阳市,34°37′N,112°26′E);2015年5月中旬至10月中旬、2016年5月中旬至10月中旬分别将102份谷子材料种植于吉林市农业科学院试验田(吉林市,42°31′N,125°40′E)、吉林省农业科学院试验田(公主岭市,43°11′N,124°02′E),吉林省的这两个试验点分别为地理环境Ⅱ和地理环境Ⅲ;2015年11月中旬至2016年2月中旬,2016年11月中旬至2017年2月中旬,将102份谷子种植于地理环境Ⅳ:海南乐东县九所镇(18°45′N,109°10′E)。附表1
附表1102份谷子材料表Attached table 1 102 cultivars of foxtail millet
| 品种编号 Variety number | 品种名 Variety name | 来源地 Origin area | 品种编号 Variety number | 品种名 Variety name | 来源地 Origin area |
|---|---|---|---|---|---|
| HN1 | 郑05-1 Zheng05-1 | 中国河南 Henan, China | HB21 | 104 104 | 未知 Unknown |
| HN2 | 郑05-2 Zheng05-2 | 中国河南 Henan, China | HB22 | 200152 200152 | 未知 Unknown |
| HN3 | 安04-4705 An04-4705 | 中国河南 Henan, China | HB23 | 8322-14 8322-14 | 未知 Unknown |
| HN4 | 安04-4852 An04-4852 | 中国河南 Henan, China | HB24 | 冀谷32 Jigu32 | 中国河北 Hebei, China |
| HN5 | 安04-5014 An04-5014 | 中国河南 Henan, China | HB25 | 冀谷30 Jigu30 | 中国河北 Hebei, China |
| HN6 | 豫谷1 Yugu1 | 中国河南 Henan, China | HB26 | 冀谷28 Jigu28 | 中国河北 Hebei, China |
| HN7 | 安09-8525 An09-8525 | 中国河南 Henan, China | HB27 | 冀谷27 Jigu27 | 中国河北 Hebei, China |
| HN8 | 安08-4125 An08-4125 | 中国河南 Henan, China | HB28 | 95307 95307 | 中国河北 Hebei, China |
| HN9 | 郑07-2 Zheng07-2 | 中国河南 Henan, China | HB29 | 谷丰2 Gufeng2 | 中国河北 Hebei, China |
| HN10 | 郑谷2 Zhenggu2 | 中国河南 Henan, China | HB30 | K325 K325 | 未知 Unknown |
| HN11 | 豫谷6 Yugu6 | 中国河南 Henan, China | HB31 | 白米1号 Baimi 1 hao | 中国河北 Hebei, China |
| HN12 | 安10-4240 An10-4240 | 中国河南 Henan, China | HB32 | 冀谷1 Jigu1 | 中国河北 Hebei, China |
| HN13 | 郑07-1 Zheng07-1 | 中国河南 Henan, China | HB33 | 冀特5 Jite5 | 中国河北 Hebei, China |
| HN14 | 安10-4172 An10-4172 | 中国河南 Henan, China | HB34 | 坝谷139 Bagu139 | 中国河北 Hebei, China |
| HN15 | 豫谷17 Yugu17 | 中国河南 Henan, China | HB35 | 坝91-0079 Ba91-0079 | 中国河北 Hebei, China |
| HN16 | 豫谷11 Yugu11 | 中国河南 Henan, China | HB36 | 毛谷2 Maogu2 | 中国河北 Hebei, China |
| HN17 | 豫谷8 Yugu8 | 中国河南 Henan, China | HB37 | 大白谷 Dabaigu | 中国河北 Hebei, China |
| HN18 | 郑06-6 Zheng06-6 | 中国河南 Henan, China | HB38 | 大九根齐 Dajiugenqi | 中国河北 Hebei, China |
| HN19 | 豫谷3 Yugu3 | 中国河南 Henan, China | HB39 | 坝谷6 Bagu6 | 中国河北 Hebei, China |
| HN20 | 安4117 An4117 | 中国河南 Henan, China | HB40 | 四留钱 Siliuqian | 中国河北 Hebei, China |
| HN21 | 安-4585 An-4585 | 中国河南 Henan, China | HB41 | 坝谷210 Bagu210 | 中国河北 Hebei, China |
| HN22 | 安5424 An5424 | 中国河南 Henan, China | SD1 | 鲁谷10 Lugu10 | 中国山东 Shandong, China |
| HN23 | 豫谷15 Yugu15 | 中国河南 Henan, China | SD2 | 早白糯 Zaobainuo | 中国山东 Shandong, China |
| HN24 | 豫谷13 Yugu13 | 中国河南 Henan, China | SD3 | 济大粒2 Jidali2 | 中国山东 Shandong, China |
| HN25 | 豫谷16 Yugu16 | 中国河南 Henan, China | SD4 | 济叶冲4 Jiyechong4 | 中国山东 Shandong, China |
| HN26 | 豫谷18 Yugu18 | 中国河南 Henan, China | SD5 | 济叶冲20 Jiyechong20 | 中国山东 Shandong, China |
| HN27 | 郑州12 Zhengzhou12 | 中国河南 Henan, China | SD6 | 济长穗8 Jichangsui8 | 中国山东 Shandong, China |
| HN28 | 郑315 Zheng315 | 中国河南 Henan, China | SD7 | 济丰30 Jifeng30 | 中国山东 Shandong, China |
| HN29 | 郑8041 Zheng8041 | 中国河南 Henan, China | SD8 | 金线子 Jinxianzi | 中国山东 Shandong, China |
| HN30 | 豫谷5 Yugu5 | 中国河南 Henan, China | SD9 | 鲁谷3 Lugu3 | 中国山东 Shandong, China |
| HN31 | 新农673 Xinnong673 | 中国河南 Henan, China | SD10 | 鲁谷7 Lugu7 | 中国山东 Shandong, China |
| HB1 | 谷丰1 Gufeng1 | 中国河北 Hebei, China | SD11 | 书香1号 Shuxiang1hao | 中国山东 Shandong, China |
| HB2 | 71杂30-2 71za30-2 | 未知 Unknown | SC1 | 11郄961 11 xi 961 | 未知 Unknown |
| HB3 | 张庙谷选变Zhangmiaoguxuanbian | 未知 Unknown | SC2 | 11郄1071 11 xi 1071 | 未知 Unknown |
| HB4 | 冀谷15 Jigu15 | 中国河北 Hebei, China | BJ1 | 06-766 06-766 | 中国北京 Beijing, China |
| HB5 | 冀谷17 Jigu17 | 中国河北 Hebei, China | BJ2 | 小早谷 Xiaozaogu | 中国北京 Beijing, China |
| HB6 | 衡谷9 Jigu9 | 中国河北 Hebei, China | NMG1 | 小香米 Xiaoxiangmi | 中国河北 Hebei, China |
| HB7 | 金谷1 Jingu1 | 中国河北 Hebei, China | NMG2 | 籼紫灰谷Shanzihuigu | 中国内蒙古 Neimenggu, China |
| HB8 | 沧372 Cang372 | 中国河北 Hebei, China | NMG3 | 二白谷 Erbaigu | 中国内蒙古 Neimenggu, China |
| HB9 | 沧369 Cang369 | 中国河北 Hebei, China | NMG4 | 黄玉3 Huangyu3 | 中国内蒙古 Neimenggu, China |
| HB10 | 冀谷29 Jigu29 | 中国河北 Hebei, China | SX1 | 呼和浩特大毛谷Huhehaotedamaogu | 中国陕西 Shanxi, China |
| HB11 | 冀谷26 Jigu26 | 中国河北 Hebei, China | SX2 | 延谷4 Yangu4 | 中国陕西 Shanxi, China |
| HB12 | 冀谷24 Jigu24 | 中国河北 Hebei, China | SX3 | 红杆谷 Honggangu | 中国陕西 Shanxi, China |
| HB13 | 冀谷22 Jigu22 | 中国河北 Hebei, China | MFGY1 | ISE430 ISE430 | 美国 America |
| HB14 | 冀谷19 Jigu19 | 中国河北 Hebei, China | MFGY2 | 法谷28-81 Fagu28-81 | 法国 French |
| HB15 | 冀谷18 Jigu18 | 中国河北 Hebei, China | MFGY3 | SET3/80 SET3/80 | 德国 Germany |
| HB16 | 冀谷31 Jigu31 | 中国河北 Hebei, China | MFGY4 | ISE775 ISE775 | 印度 India |
| HB17 | 复12 Fu12 | 中国河北 Hebei, China | HCCR1 | 龙谷26 Longgu26 | 中国黑龙江 Heilongjiang, China |
| HB18 | 冀创1 Jichuang1 | 中国河北 Hebei, China | HCCR2 | 朝鲜谷子 Chaoxianguzi | 朝鲜 Korea |
| HB19 | 2015 2015 | 中国河北 Hebei, China | HCCR3 | 谷绿早1 Gulvzao1 | 朝鲜 Korea |
| HB20 | 2013 2013 | 中国河北 Hebei, China | HCCR4 | 大王国 Dawangguo | 日本 Japan |
新窗口打开|下载CSV
种植方式为每品种(系)种植1行,行长2 m,行距为45 cm,株距为3—4 cm,地两头设2行保护行,4个试验点谷子材料均在雨季种植,随后整个生育期未进行灌溉,种植前施基肥量为600 kg·hm-2,随后整个生育期未进行施肥处理,最大程度保证不同环境间水肥管理一致性;所有试验点均在谷子长到3叶期间苗,4叶期定苗,定苗后严格控制杂草生长。所测定的10个表型性状包括抽穗期(heading stage,HS)、株高(plant height,PH)、叶片数(number of leaves,NL)、穗长(panicle length,PL)、穗粗(panicle diameter,PD)、穗码数(spikelet number,SN)、码粒数(grain number per branch,GN)、穗重(spike weight,SW)、穗粒重(grain weight per panicle,GW)和千粒重(1000-grain weight,1000-GW),其中抽穗期以每个品种(系)从出苗至超过50%植株抽穗的天数表示,其他各性状均选择每个品种(系)行中部10株进行测量,取均值作为该性状的最终测量值。
1.2 DNA提取
待各试验点田间谷子材料长至4叶期时分别提取洛阳、海南试验点每份谷子材料基因组DNA,提取吉林市、公主岭市2个试验点每份谷子材料基因组DNA,用于后续SSR标记扩增模板。采用改良的CTAB法提取谷子基因组DNA,用超微量分光光度计测量DNA浓度,用无菌ddH2O将每份谷子材料DNA稀释为试验所需浓度。1.3 SSR引物及分子标记
70对SSR多态性引物来于文献[23-24,27],引物信息见电子附表2。引物由北京鼎国昌盛生物技术有限责任公司合成。PCR扩增体系为20 μL,包含50 ng DNA、2×Es Taq MasterMix (Dye) 10 μL(康为世纪生物科技有限公司)、10 μmol·L-1正、反向引物各0.5 μL,最后用ddH2O补足到20 μL。PCR扩增条件为94℃ 5 min;94℃ 30 s;55—59℃ 30 s;72℃ 60 s,35个循环,72℃ 5 min。取8 μL PCR扩增产物于 8%的非变性聚丙烯酰胺凝胶中电泳,电压为120 v恒压,电泳时间2—3 h。电泳结束后银染、显影,最后观察并照相,人工观察确定每对引物扩增的等位基因数。附表2
附表270对SSR引物Attached table 2 70 pairs of SSR primers
| SSR标记 SSR markers | 引物序列 Primer sequence (5’-3’) | SSR标记 SSR markers | 引物序列 Primer sequence (5’-3’) |
|---|---|---|---|
| b115 | F:GGTAGCGACGGATCTACAGC R:GCTAGCAAATGCTGTCATGG | b202 | F:AGAGCCCACGTCAAACC R:AAACTGGACTAGAAGAAGCATAG |
| b116 | F:GCAAGCGTGATGTCAGATTAT R:ATAGGATGGTGGAAGCCCA | b101 | F:ATCTAGGTGCCGATGCGT R:TGTGGGAAGAAGCTAGGGAA |
| b122 | F:ACTTCTTCCTTCCTTGCGG R:TGTGGGATTAAGGTGCATCG | b106 | F:TGCTTTGCTCTCTTCTCTCA R:ACGGACGATGAGGAATTGT |
| b125 | F:GCCATGAAACAGGTACAAAAGG R:GCATCCCCTTAATTTGTCAATG | p59 | F:TAATTTTGTGGCGTGGGATG R:GCACTGGTTTTGTTGAATGG |
| p6 | F:AAGGATGGAATTTGCCACTG R:TTTCGACGATTTGCTTCAAC | p98 | F:ATTCATCAGTAGCACAGC R:TGGAACTAAGAACAGGAAAC |
| p5 | F:CTTCCCTCCCTCCCTGAC R:CTGAGCTGAGCTGCCTTTG | p56 | F:GATGTGTACGGGTTGCATTG R:TGGGTTTCAGGGCTCTCTC |
| p3 | F:GCAGAAAGCATGCCGTAGTC R:GCTTGGAGTCCACATGGATAG | p61 | F:CATCCGCGTCATCTGAATC R:ACCTGCTGCTATCCATCACC |
| p8 | F:CGATCGAATGATCGATGAAC R:CCCTTTGTCCGATCACGTC | p68 | F:CATGCGTTGATCGTTTGTG R:ACCACGCATTTACATGATCG |
| p10 | F:CAATCACATCCGAGCATTTC R:CACCCACCGTGTTGATCTG | p74 | F:CAACCTAGTTTGCCTCAGTTATTC R:AGTCACGTACATGGGTGCAG |
| p12x | F:ACGAGTCACAAATCACAGCAC R:ATGCCTGAGCGGAACGGAA | p80 | F:GCCGTTGGATTTGATTATGG R:TGTGGTTAGTTTATGTGGCTTG |
| p16 | F:TTTCTCCCTCTCTCGATTCC R:AAATTGGCGTGCTAACAACC | p87 | F:ACCTTTGACAAACGAGACACG R:GTTCGACTTGCATTGACTGG |
| b127 | F:CCTCAAGTCAGTGAGATGCAA R:CAGAGCTGTTTAATCCTTGTTCA | p89 | F:GCCTGTCTGAAAATTCTCAATG R:AGACGTGACATTAGCGCTTG |
| b129 | F:CACACTCTTCTCCCCTTTTCC R:ACGGTAACGGAGGATGGCTA | p88 | F:CAAGCCACCCAGTCTAGAGG R:TTCATCAGAACTGCGCAAAC |
| b142 | F:TGGTAAAACTCCCATATTGAGC R:GCCCCATCCTTGATAACAGA | p92 | F:TGGAATTGGAACCCTTTCG R:GCCATGCAAACAGTACCATC |
| b147 | F:CTACTGCCTTCTGGCCTCC R:GGGCATTCTTGCTTCAGTCA | p85 | F:GAATTAGGCCGATGCACAAC R:ATCCTAACTGCATGGCAAGG |
| b158 | F:GATGAGGAAAAGGTAGGTTGGA R:CTGCAACGTGCAGAACTACG | p100 | F:AGTTGACACCACACATAACAA R:AGAATACTCCTACCTGCCAC |
| b161 | F:GGCATAAAAGTAAAAACCAACCA R:ACCTGGCTTCTGTCAGTGAA | p95X | F:GTCTCTGATAGTGCTTGAGCG R:ACAGGGATGAAGGGCGATG |
| b163 | F:CTCGGAAGCTCAGATTCTCC R:CACTTCCTGCAGCTCTCACA | p29 | F:GATGAGCACACGTTGATTGG R:GGACTTCACCACCGAGATG |
| b165 | F:GCTTTGGTTTGGTTTGGTTGG R:CCATTAGTCTCTGCCCTTGTT | p44 | F:TTCCCGGAACAGACAAGAAC R:GCGTTGGAAGCCATGGAG |
| b166 | F:CGCCCATACTACCCAACAG R:ACCTCACCTTCCACTCCTC | p42 | F:GCGACTTTCCCCTTCCAATC R:TTCCTTTTGTTGGCTTCTCC |
| b194 | F:CTGGGTTCCGTCTACCGTA R:CACACCCGAAGAGGCAAAG | p91 | F:AGCTGTGCTCCTCTGATCTTG R:TAACGTGGGGATGCACTAGC |
| b200 | F:CATCGATCTCAACCTGTCCTT R:ATGAGCCGTCATGTCACAAA | sigms11655 | F:TCGTTTAAGCTGGAATTGGG R:AGAGTACCGTCGGCGTCTAA |
| b107 | F:AGAACGAGGTGGTGTGTGG R:GGGTCTCACGCTCTCATCA | P18 | F:TTCTCTCGTTGGAATTTTTGTG R:GGAACAGATATCCTTTTCACTCTT |
| b123 | F:GGTGTTCTCCTGTGTGC R:AGAGTTATTTCCAGCATTAGTG | p20 | F:GTGCCCGCTTAGCTTTAATC R:ATGCACGTGGGACCCATAC |
| b159 | F:GCCAGTCCGAGATGGTTAAG R:AGCTCTAGCAGTTGGGGACA | p17x | F:CGGACACCTGAAAGACGAA R:GTCACTTGTTGTTGTTGCG |
| b190 | F:GAAATTTCACAAGTGTTGGTG R:TGATCGGAGCAGAGTGTTGA | sigms9034 | F:TGCTGGTCGCAGTACTTGAT R:TCCTCTGCTCTGCTCTCCTC |
| b234 | F:GCCGCAACGAACAACCG R:CCTGTCCCTATCCCTGTCG | sigms10291 | F:TCGTCTCCGTCAGTAATCCC R:GCAGCAGAAGCAGATGAGG |
| b255 | F:GAGGACAGCGGCCATT R:CCTCCCTCCATTTACTTTGG | sigms11641 | F:CACCATGGCCACCATATGTA R:TGGTTGCTCAGCATAGCCTT |
| b242 | F:CACTACCACTGTTCCAGATCG R:CAGGGACCTTGCTTGCATAC | sigms11693 | F:CTAATTTTGCATGTGCCAGG R:CCACATACACCATGAGGTTCC |
| b185 | F:GCACGTGTGACTTTCCACAT R:GTGAATGGCACACGAAACTG | sigms11672 | F:TAGTAAACTTGGGCCCATCC R:ATAACTTCTCCCCCACCACC |
| b227 | F:TGATCTGGCAGAACGAACA R:CAATTCCTGGACCAATATGC | sigms10287 | F:GACCGATTCCTTCCAAACAA R:TGTCAGCTGGAGATGGTGTC |
| b233 | F:GCCACGCACACCAACTT R:CTCCCGCAGAACACGCA | MPGD46 | F:GATGGGTCGTCGTTAGAGTTT R:GGAAAGGGAAAGGAAGATAGC |
| b258 | F:GGGCCAATAATGGTTGCATA R:TTGCACATCCAAATCTTTCC | MPGD10 | F:CATCTGTTGCTGCTGATGCT R:AATTCTCAAAAGAGAGACCCACTG |
| b153 | F:ACCCAACACATTCTCCTGAA R:TGCTATCAAAATAGTGCTAGAAT | sigms11 | F:AGATCCTCGCAGACTTCGTC R:TGCAATCACCAGAGAGAAGC |
| b187 | F:TTGGACAAATGACGCTATGC R:CTGCATCAAATCAGGACCAC | MPGC13 | F:CTGAAGCATTCGATAAACTAGCTG R:GTAGTGAAAAATGAGCAAGGGTCT |
新窗口打开|下载CSV
1.4 遗传多样性分析及聚类分析
根据SSR引物扩增产生的基因型数据,用GenAlEx v6.5插件计算引物的多样性基础遗传参数,用MEGA 6.05软件中的UPGMA算法对102份谷子资源进行聚类分析。1.5 群体结构分析及LD分析
用Structure 2.3.4软件进行群体遗传结构分析,估计最佳群体组群数K,其取值范围为2—10,将参数iterations设为10 000,burn-in period设为100 000,每个K值重复运行10次,依据似然值最大原则选取合适的K值为群体数目,以此K值为亚群数,重新运行Structure 2.3.4软件计算Q参数。将数据转换为Tassel 2.1软件能够识别的格式,进行连锁不平衡(linkage disequilibrium,LD)分析。1.6 标记与性状间的关联分析
采用Tassel 5.0软件简单计算Kinship矩阵,用一般线性模型(general linear model,GLM)和混合线性模型(mixed linear model,MLM)2种程序进行关联分析。在GLM分析中,以群体结构分析Q值作为协变量,将SSR标记与102份谷子2年(2015和2016)4地(海南乐东县、河南洛阳市、吉林市、公主岭市)的抽穗期、株高、叶片数、穗长、穗粗、穗码数、码粒数、穗重、穗粒重和千粒重10个表型数据进行回归分析,寻找与之相关联的标记,并确定其对表型变异的贡献度;在MLM分析中,在群体结构分析的基础上采用Q+K方法,分别运用2年4地试验表型数据结合分子标记数据和群体结构分析数据、Kinship数据,进行标记-性状关联分析,确定关联位点(P<0.05),并计算标记对表型变异的贡献度。2 结果
2.1 10个农艺性状的相关性分析
102份谷子材料2年4地的抽穗期、株高、叶片数、穗长、穗粗、穗码数、码粒数、穗重、穗粒重和千粒重10个表型数据各自求平均值,利用SPSS 19.0软件进行相关性分析(表1)。在4个不同地理环境下,HS与NL、SN均呈极显著正相关;GW与PD、GN、SW呈极显著正相关;NL与HS、PD、SN、GN、SW均呈显著正相关;PL与SN、SW呈显著正相关;PD与GW、NL、GN、SW均呈极显著正相关;SN与HS、NL、PL呈显著正相关;GN与GW、NL、PD、SW均呈显著正相关;SW与GW、NL、PL、PD、GN均呈显著正相关;1000-GW与GN无显著相关性。Table 1
表1
表1不同地理环境谷子主要农艺性状相关性分析
Table 1
| 环境 Environments | 性状Trait | HS | GW | PH | NL | PL | PD | SN | GN | SW | 1000-GW |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 吉林市 Jilin | HS | 1 | |||||||||
| GW | -0.157 | 1 | |||||||||
| PH | 0.035 | 0.340** | 1 | ||||||||
| NL | 0.538** | 0.195 | 0.105 | 1 | |||||||
| PL | 0.04 | 0.464** | 0.530** | 0.109 | 1 | ||||||
| PD | 0.173 | 0.415** | 0.012 | 0.366** | 0.208 | 1 | |||||
| SN | 0.421** | 0.101 | 0.116 | 0.299* | 0.285* | 0.033 | 1 | ||||
| GN | -0.08 | 0.667** | 0.129 | 0.283* | 0.119 | 0.547** | -0.209 | 1 | |||
| SW | -0.09 | 0.957** | 0.361** | 0.254* | 0.547** | 0.478** | 0.122 | 0.694** | 1 | ||
| 1000-GW | -0.315** | 0.388** | 0.242* | -0.226 | 0.321** | 0.051 | -0.269* | -0.005 | 0.382** | 1 | |
| 公主岭市 Gongzhuling | HS | 1 | |||||||||
| GW | 0.367** | 1 | |||||||||
| PH | 0.420** | 0.343** | 1 | ||||||||
| NL | 0.756** | 0.552** | 0.485** | 1 | |||||||
| PL | 0.329* | 0.332** | 0.686** | 0.282* | 1 | ||||||
| PD | 0.571** | 0.480** | 0.173 | 0.520** | 0.260* | 1 | |||||
| SN | 0.363** | 0.206 | 0.380** | 0.342** | 0.302* | -0.115 | 1 | ||||
| GN | 0.323* | 0.657** | 0.064 | 0.401** | 0.077 | 0.594** | -0.170 | 1 | |||
| SW | 0.437** | 0.938** | 0.373** | 0.596** | 0.395** | 0.618** | 0.132 | 0.674** | 1 | ||
| 1000-GW | -0.018 | 0.307* | 0.383** | 0.068 | 0.427** | 0.092 | 0.202 | 0.012 | 0.384** | 1 | |
| 洛阳市 Luoyang | HS | 1 | |||||||||
| GW | 0.552** | 1 | |||||||||
| PH | 0.571** | 0.575** | 1 | ||||||||
| NL | 0.662** | 0.742** | 0.670** | 1 | |||||||
| PL | 0.338** | 0.205 | 0.716** | 0.333** | 1 | ||||||
| PD | 0.567** | 0.688** | 0.558** | 0.761** | 0.388** | 1 | |||||
| SN | 0.565** | 0.554** | 0.667** | 0.648** | 0.513** | 0.541** | 1 | ||||
| GN | 0.281* | 0.745** | 0.302** | 0.493** | -0.079 | 0.544** | 0.180 | 1 | |||
| SW | 0.619** | 0.951** | 0.672** | 0.794** | 0.363** | 0.766** | 0.614** | 0.687** | 1 | ||
| 1000-GW | 0.211 | 0.347** | 0.343** | 0.300** | 0.330** | 0.296** | 0.264* | 0.046 | 0.371** | 1 | |
| 乐东县Ledong | HS | 1 | |||||||||
| GW | 0.623** | 1 | |||||||||
| PH | 0.163 | 0.228* | 1 | ||||||||
| NL | 0.771** | 0.781** | 0.230* | 1 | |||||||
| PL | 0.607** | 0.754** | 0.189 | 0.609** | 1 | ||||||
| PD | 0.665** | 0.858** | 0.244** | 0.845** | 0.613** | 1 | |||||
| SN | 0.429** | 0.553** | 0.284* | 0.534** | 0.558** | 0.442** | 1 | ||||
| GN | 0.353** | 0.735** | 0.159 | 0.557** | 0.364** | 0.697** | 0.132 | 1 | |||
| SW | 0.622** | 0.808** | 0.292* | 0.708** | 0.672** | 0.775** | 0.557** | 0.503** | 1 | ||
| 1000-GW | -0.003 | 0.194 | -0.083 | 0.064 | 0.120 | 0.129 | 0.079 | -0.008 | 0.122 | 1 |
新窗口打开|下载CSV
2.2 SSR标记分析
70对SSR引物在102份谷子材料中均表现多态性,共检测到397个等位变异,单个引物检测到3—9个等位变异,平均每对引物检测出6个。sigms11641和P18标记检测出的等位变异最少(3个),p95X标记的等位变异最多(9个)。70对标记检测出有效等位基因数为1.519—3.092,平均有效等位基因数为2.24,期望杂合度范围为0.448—0.642,平均期望杂合度为0.4637。SSR标记的Shannon指数变幅为0.439—1.118,平均为0.7738(表2)。Table 2
表2
表270对SSR标记在102份谷子材料中的多态性
Table 2
| SSR标记 SSR markers | 等位基因数 Allele number | 有效等位基因数 Number of effective alleles | Shannon指数 Shannon index | 期望杂合度 Expected heterozygosity |
|---|---|---|---|---|
| b115 | 6 | 2.005 | 0.618 | 0.350 |
| b116 | 4 | 1.712 | 0.521 | 0.325 |
| b122 | 5 | 2.200 | 0.719 | 0.431 |
| b125 | 5 | 2.447 | 0.926 | 0.575 |
| p6 | 4 | 2.059 | 0.738 | 0.468 |
| p5 | 6 | 2.022 | 0.632 | 0.363 |
| p3 | 8 | 2.776 | 1.026 | 0.602 |
| p8 | 6 | 1.983 | 0.611 | 0.346 |
| p10 | 5 | 2.204 | 0.786 | 0.494 |
| p12x | 8 | 2.226 | 0.767 | 0.448 |
| p16 | 5 | 1.849 | 0.631 | 0.390 |
| b127 | 7 | 2.355 | 0.765 | 0.438 |
| b129 | 4 | 2.283 | 0.849 | 0.516 |
| b142 | 7 | 2.595 | 0.903 | 0.522 |
| b147 | 4 | 1.773 | 0.622 | 0.400 |
| b158 | 5 | 2.148 | 0.784 | 0.495 |
| b161 | 5 | 1.926 | 0.666 | 0.405 |
| b163 | 4 | 1.909 | 0.568 | 0.332 |
| b165 | 4 | 1.827 | 0.590 | 0.395 |
| b166 | 4 | 1.943 | 0.685 | 0.442 |
| b194 | 6 | 1.909 | 0.657 | 0.380 |
| b200 | 6 | 2.277 | 0.802 | 0.470 |
| b107 | 7 | 1.961 | 0.721 | 0.446 |
| b123 | 6 | 2.426 | 0.872 | 0.520 |
| b159 | 6 | 2.600 | 0.900 | 0.509 |
| b190 | 5 | 1.933 | 0.696 | 0.433 |
| b234 | 4 | 1.742 | 0.503 | 0.308 |
| b255 | 6 | 2.434 | 0.816 | 0.469 |
| b242 | 4 | 2.100 | 0.766 | 0.477 |
| b185 | 4 | 2.153 | 0.803 | 0.512 |
| b227 | 4 | 2.025 | 0.637 | 0.392 |
| b233 | 5 | 2.354 | 0.895 | 0.558 |
| b258 | 6 | 2.118 | 0.775 | 0.478 |
| b153 | 5 | 2.497 | 0.854 | 0.498 |
| b187 | 5 | 2.498 | 0.940 | 0.578 |
| b202 | 7 | 2.329 | 0.790 | 0.449 |
| b101 | 6 | 2.218 | 0.779 | 0.448 |
| b106 | 8 | 2.325 | 0.775 | 0.448 |
| p59 | 5 | 2.258 | 0.812 | 0.496 |
| p98 | 4 | 2.177 | 0.728 | 0.443 |
| p56 | 5 | 2.123 | 0.806 | 0.508 |
| p61 | 4 | 1.671 | 0.498 | 0.319 |
| p68 | 5 | 2.098 | 0.734 | 0.466 |
| p74 | 5 | 2.366 | 0.825 | 0.515 |
| p80 | 8 | 2.797 | 1.025 | 0.598 |
| p87 | 8 | 3.092 | 1.118 | 0.642 |
| p89 | 8 | 2.538 | 0.892 | 0.497 |
| p88 | 8 | 2.832 | 0.974 | 0.560 |
| p92 | 7 | 2.425 | 0.843 | 0.498 |
| p85 | 6 | 2.286 | 0.838 | 0.503 |
| p100 | 6 | 2.448 | 0.879 | 0.530 |
| p95X | 9 | 2.827 | 0.935 | 0.502 |
| p29 | 7 | 2.714 | 0.964 | 0.534 |
| p44 | 8 | 2.932 | 1.036 | 0.605 |
| p42 | 7 | 2.785 | 0.989 | 0.567 |
| p91 | 8 | 2.362 | 0.802 | 0.474 |
| sigms11655 | 6 | 1.788 | 0.490 | 0.295 |
| P18 | 3 | 1.519 | 0.439 | 0.279 |
| p20 | 7 | 2.519 | 0.937 | 0.570 |
| p17x | 6 | 2.335 | 0.818 | 0.495 |
| sigms9034 | 5 | 1.593 | 0.493 | 0.311 |
| sigms10291 | 4 | 2.174 | 0.728 | 0.451 |
| sigms11641 | 3 | 1.726 | 0.538 | 0.368 |
| sigms11693 | 6 | 2.463 | 0.946 | 0.572 |
| sigms11672 | 4 | 1.686 | 0.516 | 0.321 |
| sigms10287 | 7 | 2.723 | 0.913 | 0.498 |
| MPGD46 | 6 | 2.297 | 0.804 | 0.466 |
| MPGD10 | 5 | 2.649 | 0.974 | 0.588 |
| sigms11 | 6 | 2.390 | 0.883 | 0.522 |
| MPGC13 | 5 | 1.794 | 0.599 | 0.355 |
新窗口打开|下载CSV
2.3 聚类分析
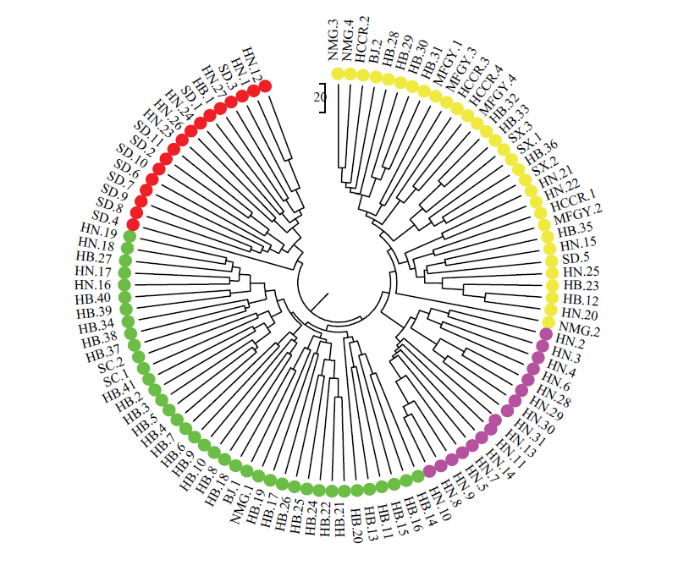
102份谷子材料按UPGMA法被聚为4组,第一组包含17份材料,多数来自山东省;第二组包含38份材料,多数来自河北省;第三组包含16份材料,全部来自河南省;第四组包含31份材料,成员来源复杂,包括了来自国内各地区的种质24份以及来自法国、美国、德国、印度、朝鲜和日本的种质7份(图1)。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1基于SSR标记的102份谷子材料的聚类图
Fig. 1Dendrogram of 102 foxtail millet materials based on SSR markers
2.4 群体遗传结构分析
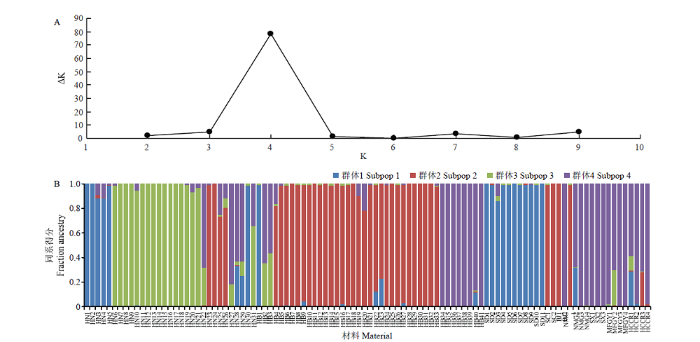
通过ΔK确定102份谷子的等位变异频率特征类型数K值,从图2-A可以看出,当K为4时ΔK出现峰值,因此,将102份谷子材料分为4个亚群(图2-B)。群体1包含18份材料,其中11份来自山东省,6份来自河南省,1份来自河北省;群体2包括38份材料,其中27份来自河北省,4份来自河南省,1份来自北京市,6份未知来源;群体3包含17份材料,全部来自河南省;群体4包含29份材料,其中8份来自河北省,4份来自河南省,3份来自内蒙古自治区,3份来自陕西省,1份来自北京市,2份未知来源,余下的8份材料来自中国黑龙江省、美国、法国、德国、印度、朝鲜和日本。因此,在进行关联分析时应将群体结构纳入计算。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2基于 SSR 标记的 102 份谷子材料群体遗传结构
A:ΔK随K值的变化趋势,峰值表明该自然群体可分为4个类群。B:表示102份材料的群体遗传结构
Fig. 2Population genetic structure of 102 millet materials based on SSR markers
A: The changing trend of ΔK with K value, the peak value indicated that the natural population used in this study could be divided into four groups; B: The population genetic structure of the 102 millet materials
2.5 连锁不平衡(LD)分析
关联分析的基础是连锁不平衡分析,LD是群体内同一条染色体上或不同染色体之间不同座位上等位基因非随机关联的结果,LD水平越高,则连锁越紧密,进行GWAS分析时需要标记的数目就越少[40]。对70个SSR标记位点进行连锁不平衡分析,发现存在显著连锁不平衡的成对位点有335对,仅占所有位点组合的13.87%(图3)。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图370对SSR标记间的连锁不平衡模式
上三角中给出了每对标记的平方相关系数(R2),下三角中给出了相应的P值
Fig. 3Linkage disequilibrium patterns among 70 SSR markers
The squared correlation coefficients (R2) for each pair of markers are presented in the upper triangle and their corresponding P values in the lower triangle
2.6 利用GLM、MLM 2种模型进行SSR标记与农艺性状的关联分析
利用群体结构Q矩阵(GLM模型)、群体结构Q矩阵和亲缘关系K矩阵(MLM模型)分别对吉林市、公主岭市、洛阳市和乐东县4个地理环境2年的谷子表型性状进行关联分析(表3),2种模型共检测到10个标记位点与7个农艺性状显著或极显著关联,其中,吉林市和公主岭市检测到的位点最多,2地2个年份稳定地检测到5个SSR标记:b115用GLM模型检测到同时与穗粒重、穗重显著关联,平均可解释的表型贡献率分别为为0.3406和0.3591,用MLM模型检测到与穗长显著关联,平均可解释的表型贡献率为0.1614;MPGC13用GLM模型检测到与叶片数极显著关联,平均可解释的表型贡献率为0.2778;b227用GLM、MLM模型均检测到与抽穗期显著或极显著关联,平均可解释的表型贡献率为0.1836;b194、p56用MLM模型均检测到与抽穗期显著关联,平均可解释的表型贡献率分别为0.1206和0.1069。洛阳市检测到2个SSR标记与穗长、码粒数关联:sigms9034用GLM模型检测到与穗长极显著关联,平均可解释的表型贡献率为0.2130;b125用GLM、MLM模型分别检测到与穗长和穗码数显著或极显著关联,平均可解释的表型贡献率分别为0.2563和0.1525。乐东县用MLM模型检测到2个SSR标记P18和p59,均与穗重显著关联,平均可解释的表型贡献率分别为0.0953和0.1033。还有1个标记p6在洛阳市、吉林市、公主岭市同时检测到,在洛阳市用MLM模型检测到与穗码数显著相关,平均可解释的表型贡献率为0.0987;在吉林市、公主岭市用GLM、MLM模型均检测到与穗长显著关联,平均可解释的表型贡献率为0.1374,用MLM模型检测到与株高、叶片数显著关联,平均可解释的表型贡献率分别为0.1146和0.0776。Table 3
表3
表34个地理环境GLM、MLM模型关联分析结果
Table 3
| 性状 Traits | 关联标记 Associated markers | 染色体 Chromosome | 显著性检验值(P) Significance test value (P) | 表型贡献率(R2) Phenotypic variation (R2) | 环境 Environments | 年份 Years | 模型 Model |
|---|---|---|---|---|---|---|---|
| GW SW PL | b115 | 2 | 9.99E-04 0.021 9.99E-04 0.013 0.0317 1.54E-04 | 0.4355 0.2456 0.4661 0.2521 0.0807 0.2421 | 吉林Jilin 公主岭Gongzhuling 吉林Jilin 公主岭Gongzhuling 吉林Jilin 公主岭Gongzhuling | 2015 2016 2015 2016 2015 2016 | GLM MLM |
| NL | MPGC13 | 7 | 9.99E-04 9.99E-04 | 0.24 0.3156 | 吉林Jilin 公主岭Gongzhuling | 2015 2016 | GLM |
| PL PH NL SN | p6 | 8 | 9.99E-04 0.025 0.0057 0.0118 0.0123 8.46E-04 0.033 0.0171 0.0127 0.015 | 0.1958 0.1712 0.0718 0.1109 0.0701 0.1591 0.0778 0.0774 0.0943 0.1031 | 吉林Jilin 公主岭Gongzhuling 吉林Jilin 公主岭Gongzhuling 吉林Jilin 公主岭Gongzhuling 吉林Jilin 公主岭Gongzhuling 洛阳Luoyang 洛阳Luoyang | 2015 2016 2015 2016 2015 2016 2015 2016 2015 2016 | GLM MLM |
| PL | sigms9034 | 6 | 9.99E-04 0.009 | 0.2361 0.1898 | 洛阳Luoyang 洛阳Luoyang | 2015 2016 | GLM |
| PL SN | b125 | 5 | 0.026 0.014 0.0094 0.0073 | 0.2717 0.2409 0.1405 0.1645 | 洛阳Luoyang 洛阳Luoyang 洛阳Luoyang 洛阳Luoyang | 2015 2016 2015 2016 | GLM MLM |
| HS | b227 | 1 | 9.99E-04 9.99E-04 0.0372 0.0308 | 0.2460 0.3075 0.0999 0.0811 | 吉林Jilin 公主岭Gongzhuling 吉林Jilin 公主岭Gongzhuling | 2015 2016 2015 2016 | GLM MLM |
| HS | b194 | 9 | 0.01 0.032 | 0.1537 0.0874 | 吉林Jilin 公主岭Gongzhuling | 2015 2016 | MLM |
| HS | p56 | 2 | 0.0215 0.0204 | 0.1202 0.0936 | 吉林Jilin 公主岭Gongzhuling | 2015 2016 | MLM |
| SW | P18 | 5 | 0.0117 0.0039 | 0.0861 0.1044 | 乐东Ledong 乐东Ledong | 2015 2016 | MLM |
| SW | p59 | 未知 Unknown | 0.0452 0.016 | 0.0894 0.1171 | 乐东Ledong 乐东Ledong | 2015 2016 | MLM |
新窗口打开|下载CSV
2.7 显著关联位点等位效应分析
通过对10个与农艺性状显著或极显著关联的位点进行等位效应分析,在吉林(吉林市、公主岭市)特异表达的位点b115,不同等位基因间穗重、穗长和穗粒重3个性状差异达到显著或极显著水平,其中,大小为226和232 bp的等位基因穗重、穗长和穗粒重显著或极显著高于其他等位基因类型,为优势等位基因,具有改善谷子穗部性状的潜力(图4-A—图4-C);MPGC13不同等位基因间叶片数差异达到显著水平,其中,大小为320 bp的等位基因为优势等位基因,可以增加谷子叶片数(图4-D);b227不同等位基因间抽穗期差异达到显著水平,其中,大小为156 bp的等位基因抽穗期最短,在吉林地区(吉林市、公主岭市)具有缩短谷子生育期的应用潜力(图4-I);b194不同等位基因间抽穗期差异达到显著或极显著水平,其中,大小为256 bp的等位基因抽穗期显著或极显著低于其他等位基因,同样可以在吉林地区(吉林市、公主岭市)用于缩短谷子生育期(图4-J);p56大小为244 bp的等位基因抽穗期极显著短于其他等位基因类型,效应明显,也是一个较理想的缩短谷子生育期的等位基因(图4-K)。在洛阳环境特异表达的位点sigms9034,大小为168 bp的等位基因穗长极显著高于其他等位基因类型,可以作为改良谷子穗部性状的有利等位基因(图4-G);b125不同等位基因间穗码数差异达到显著水平(图4-H)。在乐东环境特异表达的位点P18,大小为166 bp的等位基因穗重极显著高于其他两类等位基因,在海南地理环境具有改善谷子穗部性状的潜力(图4-L);p59不同等位基因之间穗重差异达到显著水平,其中,为201 bp的等位基因为有利等位基因(图4-M)。p6在吉林市、公主岭市、洛阳市同时检测到,在吉林关联到的叶片数和在洛阳关联到的穗码数在不同等位基因间存在显著或极显著差异,其中,大小为188 bp的等位基因对叶片数和穗码数都具有正向作用(图4-E和图4-F)。图4
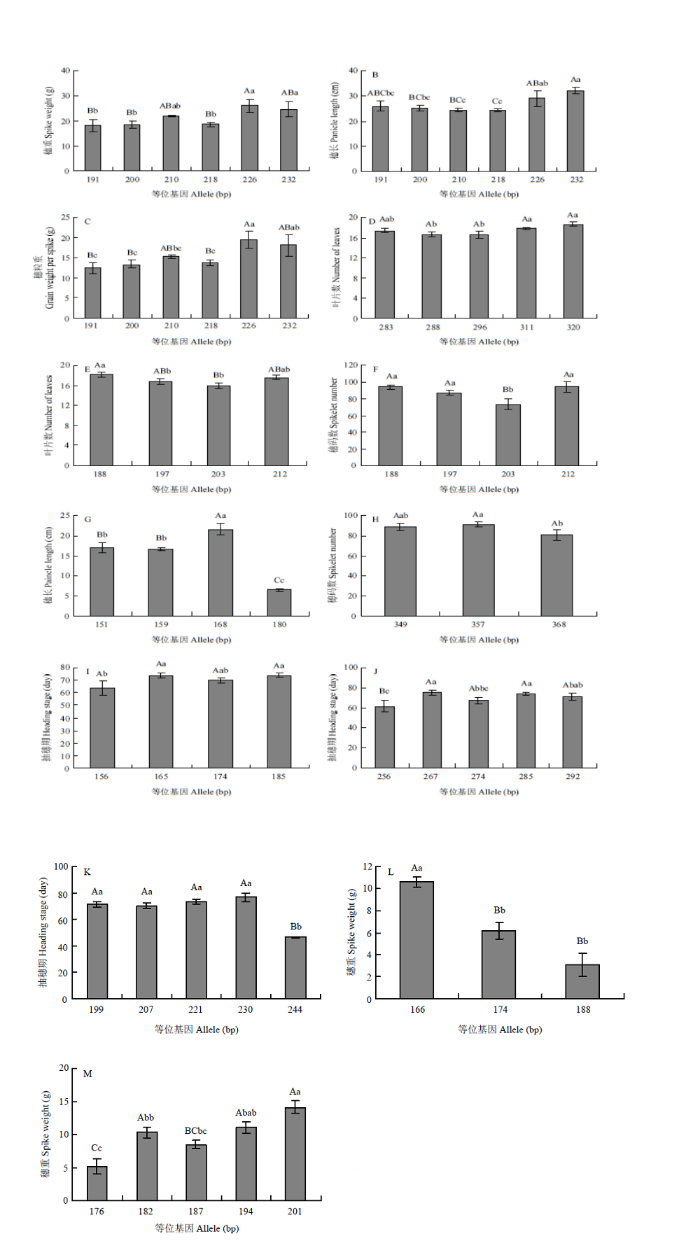 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4关联标记等位效应分析
A、B、C:b115;D:MPGC13;E、F:p6;G:sigms9034;H:b125;I:b227;J:b194;K:p56;L:P18;M:p59
Fig. 4Allele effect analysis of associated markers
3 讨论
3.1 不同地理环境谷子主要农艺性状间的相关性分析
通过4个不同地理环境性状间相关性分析,发现一些农艺性状间关系比较稳定,不受环境条件影响,如抽穗期与叶片数、穗码数之间,穗粒重与株高、叶片数、穗粗、码粒数、穗重之间,株高和叶片数、穗粗、穗码数、穗重之间,叶片数和穗长、穗粗、穗码数、码粒数、穗重之间,穗长与穗粗、穗码数、穗重之间,穗长与穗粗、穗码数、穗重之间,穗粗与码粒数、穗重之间,穗码数与穗重之间,码粒数和穗重之间,在乐东县、洛阳市、吉林市、公主岭市4个不同地理环境均稳定表现显著或极显著正相关,说明在实际育种中可以通过其中一个或几个性状改良使谷子综合表现得到提高。研究发现千粒重与其他各性状的关系在不同地理环境间存在差异,一些性状如穗粒重、株高、穗长、穗重与千粒重之间在吉林市、公主岭市和洛阳市均呈显著或极显著正相关,但在乐东县并未达到显著水平,这表明海南地理环境与洛阳市、吉林市、公主岭市3个地理环境间存在较大的差异,在海南地理环境,株高、穗长、穗重、穗粒重几个性状对千粒重没有明显的影响。3.2 SSR标记多样性与谷子群体遗传结构
本研究所用的70个SSR标记共在谷子群体中检测到397个等位基因,平均每个SSR标记扩增出6个等位基因,与JIA等[23]报道的6.16个等位基因/标记接近,低于朱学海等[28]报道的14.5个等位基因/标记以及WANG等[32]报道的20.9个等位基因/标记,但高于杨天育等[29]报道的2.71个等位基因/标记。谷子育成品种亲本来源较狭窄,因此遗传多样性水平较低。从图1中可以看出,总体上来源于同一地区的育成品种容易聚在一个亚群里,如第一个亚群主要是来自山东省的品种,还有少量来自河南省的品种;第二个亚群主要是来自河北省的品种,还有少数来自河南省的品种;第三个亚群主要是来自河南省的品种;第四个亚群成分比较复杂,包括了所有国外种质及来自全国各地区的品种。这种结果反映了同一地区选育谷子品种所用亲本材料的单一性。值得注意的是在4个亚群中都有河南的品种分布,说明来自河南省的谷子品种遗传多样性较丰富,可以作为亲本来源,拓宽谷子的遗传基础。3.3 分子标记与表型性状的关联分析
本研究共检测到10个SSR位点与农艺性状关联,其中5个标记b115、MPGC13、b227、b194、p56在吉林省的2个地区吉林市、公主岭市特异检测到,2个标记sigms9034、b125在洛阳市特异性检测到,2个标记P18和p59在海南乐东县特异性检测到,1个标记p6在洛阳市、吉林市、公主岭市3个地理环境均检测到。10个标记中有2个在前人的研究中被报道,如王晓宇等[34]将谷子株高性状定位到标记p56附近区间,而在本研究中p56与抽穗期关联,已有研究表明谷子抽穗期与株高之间呈显著正相关[5],推测标记p56与株高和抽穗期都相关,而受环境条件影响可能导致定位的性状有所不同,本研究在吉林关联到抽穗期,而王晓宇等[34]是在山西省关联到株高,两地环境的差异可能导致定位性状的不同。GUPTA等[39]通过SSR标记与谷子产量性状的关联分析发现标记p59与旗叶宽度显著关联,该标记在本研究中发现在海南乐东县环境与穗重显著关联。旗叶离穗部较近,其宽度影响光合效率,进而影响碳水化物向穗部的输送,可能会影响穗重。有研究表明旗叶宽度与穗重之间存在正相关[5],因此,标记p59可能和旗叶宽、穗重都有关联,只是在不同的环境标记位点对2个性状的效应有差异,导致不能被同时检测到。尽管在谷子上已经定位了许多控制抽穗期、株高、穗长、穗重、穗码数的QTL位点[5-7,34,39],一些控制谷子光周期敏感性的QTL位点也被定位于第3、4、9染色体[2,8],但是除了p56和p59,本研究获得的另外8个与穗长、穗重、穗码数、株高、抽穗期关联的位点均未见报道,属于新发现的位点,这些位点多数在特定地理环境表达,由于不同地理环境间除了光周期和环境温度不同,在降雨量、土壤特性等方面也存在差异,因此这些位点是否只受光周期和温度调控仍需进一步在人工控制环境下深入研究来确定。对10个关联位点进行等位效应分析,发现b115所关联到的穗重、穗长,MPGC13所关联到的叶片数,b227、b194、p56所关联到的抽穗期,sigms9034所关联到的穗长,b125所关联到的穗码数,P18、p59所关联到的穗重,p6所关联到的叶片数和穗码数在对应标记的不同等位基因间均达到显著或极显著差异(图4-A—图4-L)。其中在洛阳市地理环境特异表达的位点sigms9034,大小为168 bp的等位基因对穗长具有正向效应,且明显高于其他等位基因类型,因此可用于洛阳地区谷子穗长的标记辅助选择;在吉林2个环境特异表达的位点p56,大小为244 bp的等位基因对抽穗期具有负向效应,明显比其他等位基因类型抽穗期短。吉林省地区日照长,多数谷子品种从海南省乐东县或者河南省洛阳市转移到吉林市、公主岭市,抽穗期会推迟,甚至一些品种由于抽穗太晚而不能正常成熟影响产量,因此,可以在吉林地理环境利用244 bp的等位基因开展标记辅助选择缩短生育期,降低谷子的光周期敏感性;在海南省乐东县特异表达的位点P18,大小为166 bp的等位基因对穗重具有正向效应,且明显高于其他两类等位基因,海南省地理环境谷子品种普遍生育期短,穗小,产量潜能受到限制,利用166 bp的等位基因可以在海南开展谷子穗重的标记辅助选择,提高产量潜能。
在上述3个可用于标记辅助选择育种的标记附近区域寻找候选基因,发现sigms9034位于一个晚期胚胎发生丰富蛋白LEA_2基因内(Seita.6G003000)。在拟南芥中发现LEA_2蛋白基因的表达受干旱胁迫、冷胁迫、盐胁迫、和强光的诱导[41],说明该基因受光温调控,可能与谷子光温敏感性有关。P18位于一个E3泛素-蛋白连接酶RBBP6基因内(Seita.5G427100),在水稻和小麦中E3泛素-蛋白连接酶是控制粒宽和粒重的主效基因,这种酶蛋白可以调控糖信号转导途径、氮缺乏适应信号途径及光敏形态发生过程[42,43]。在p56标记一侧发现葡萄糖醛苷转移酶基因(Seita.2G324800),该基因是植物花青素合成途径中的一种关键基因,在植物果实颜色调控方面具有重要的作用,目前尚未有调控植物抽穗期的报道。因此对这3个候选基因需要进一步进行功能验证,才能确定其具体的作用。
4 结论
所调查的10个农艺性状除千粒重外多数表现一致的正相关,不受地理环境影响,千粒重随地理环境的改变与其他9个性状的关系也发生改变,在海南乐东县千粒重与其他9个性状没有明显相关性;关联分析共检测到10个标记位点,其中4个标记位点在单一环境表达,6个标记位点在多环境表达,10个标记位点分布在谷子的第1、2、5、6、7、8和9染色体上,表明谷子地区适应性的遗传基础较复杂,受多个基因位点调控;3个优势等位基因sigms9034(168 bp)、P18(166 bp)、p56(244 bp)可以显著增加穗重、穗长,缩短抽穗期。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 1]
[本文引用: 2]
[D].
[本文引用: 2]
[D].
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[D].
[本文引用: 1]
.[D].
[本文引用: 1]
[本文引用: 4]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
