 ,2, 陆平2
,2, 陆平2Genetic Diversity and Population Structure of Broomcorn Millet in China Based on Fluorescently Labeled SSR
KOU ShuJun1, HUO AHong1, FU GuoQing1, JI JunJian1, WANG Yao1, ZUO ZhenXing1, LIU MinXuan ,2, LU Ping2
,2, LU Ping2通讯作者:
收稿日期:2018-01-8接受日期:2019-03-5网络出版日期:2019-05-01
| 基金资助: |
Received:2018-01-8Accepted:2019-03-5Online:2019-05-01
作者简介 About authors
寇淑君,Tel:18503135993;E-mail:koushujun1986@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (1895KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
寇淑君, 霍阿红, 付国庆, 纪军建, 王瑶, 左振兴, 刘敏轩, 陆平. 利用荧光SSR分析中国糜子的遗传多样性和群体遗传结构[J]. 中国农业科学, 2019, 52(9): 1475-1477 doi:10.3864/j.issn.0578-1752.2019.09.001
KOU ShuJun, HUO AHong, FU GuoQing, JI JunJian, WANG Yao, ZUO ZhenXing, LIU MinXuan, LU Ping.
0 引言
【研究意义】糜子(Panicum miliaceum L.)属于禾本科黍属(Panicum),为一年生草本植物,有2n=4x=36条染色体。糜子起源于中国,是最古老的栽培作物,距今已有10 000余年的栽培历史[1],具有抗旱、耐盐碱、耐瘠,生育期短等特点[2],不仅为干旱半干旱地区的人们提供粮食保障,也是该地区重要的救灾备荒作物[3]。中国糜子种质资源非常丰富,目前,保存在国家种质库中的糜子资源超过8 800多份,如何准确快速评估所保存资源的遗传多样性和群体遗传结构,对于开展糜子抗逆相关基因的挖掘,促进糜子种质创新和品种改良等具有重要意义。【前人研究进展】分子标记是揭示不同品种间遗传多样性和亲缘关系的有效手段[4],RAPD、AFLP等分子标记曾被用于糜子的遗传多样性分析。M'RIBU等[5]应用RAPD技术分析了4类糜子的遗传多样性;KARAM等[6]用AFLP标记检测了12份来自美国和加拿大的糜子遗传多样性;LáGLER等[7]使用来自草莓属植物的9个ISSR标记在21份匈牙利糜子中共检测到15个等位基因,其中7个引物能扩增出DNA片段。SSR分子标记具有共显性、在基因组中含量丰富、多态性信息高、重复性好等优点[8],被广泛用于各种作物的遗传多样性研究中[9,10,11,12]。近年来,利用SSR分子标记对糜子的遗传多样性研究也取得了一些进展。HU等[2]用46对来源于水稻、小麦、燕麦和大麦的SSR标记分析了118份糜子资源的遗传多样性,PIC值为0.284—0.980。CHO等[13]基于糜子基因组DNA富集SSR文库开发了首批糜子SSR标记,利用25个标记在50份材料中检测到110个等位变异。HUNT等[14]用16个标记检测了98份来源于欧亚大陆的糜子地方品种的遗传多样性。连帅等[15]、LIU等[16]、薛延桃等[17]、王银月等[18]用高通量测序手段开发的SSR标记全面分析了中国糜子地方品种、育成品种、野生材料以及新引进国外资源的遗传多样性和群体遗传结构。与传统的聚丙烯酰胺凝胶电泳相比,毛细管电泳检测技术具有灵敏度高、分辨率高、重复性好、所需样品量少、分析通量大、可以有效减少错读等优点[19],在玉米[20]、小麦[21]、烟草[22]、油菜[23]等植物中广泛应用,但在糜子上相关报道较少[14,24]。【本研究切入点】已开展的遗传多样性研究结果显示,糜子虽然是异源四倍体作物,但是其遗传多样性水平总体不高,一方面可能是目前可用于糜子遗传多样性研究的SSR分子标记较少且多态性不高;另一方面多数研究采用的传统聚丙烯酰胺凝胶电泳检测技术分辨率较低,不能准确反映糜子不同材料间的遗传多样性。【拟解决的关键问题】本研究选用严格筛选后的22对SSR引物进行荧光标记,利用全自动基因分析仪检测技术分析来自中国不同生态区的72份糜子育成品种及59份当地主要农家种的遗传多样性和群体遗传结构,为糜子品种改良、种质创新和资源有效利用提供依据。1 材料与方法
1.1 试验材料
试验材料包括由国家种质资源库提供的72份育成品种(宁夏回族自治区11份、辽宁省2份、吉林省8份、黑龙江省10份、甘肃省8份、山西省16份、内蒙古自治区14份和陕西省3份)和59份农家种(宁夏回族自治区4份、辽宁省2份、吉林省2份、黑龙江省6份、甘肃省4份、山西省10份、内蒙古自治区5份、陕西省5份、青海省8份、河北省9份、新疆维吾尔自治区3份和山东省1份)(电子附表1)。根据糜子栽培生态区划[25],131份材料所属生态区包括东北春糜子区(30)、华北夏糜子区(3)、北方春糜子区(59)、黄土高原春夏糜子区(36)和西北春夏糜子区(3)。Table 1
表1
表122对SSR标记的遗传参数
Table 1
| 位点 Locus | 等位基因数目 Allele number | 主要等位基因 Major allele | 稀有基因数 Rare gene number | 基因多样性指数 H | 多态性信息含量 PIC | Shannon信息指数 I | |
|---|---|---|---|---|---|---|---|
| 片段大小 Size (bp) | 频率 Frequency | ||||||
| BM552 | 7 | 240 | 0.4427 | 5 | 0.7403 | 0.7124 | 1.4924 |
| BM637 | 5 | 167 | 0.3382 | 1 | 0.7373 | 0.6897 | 1.3527 |
| BM787 | 5 | 290 | 0.6908 | 1 | 0.4874 | 0.4530 | 0.8807 |
| BM2654 | 7 | 206 | 0.3015 | 1 | 0.7835 | 0.7525 | 1.6300 |
| BM2715 | 6 | 227 | 0.3893 | 4 | 0.7324 | 0.6914 | 1.3745 |
| BM2717 | 7 | 284 | 0.5916 | 5 | 0.6109 | 0.5825 | 1.2828 |
| BM2729 | 4 | 122 | 0.6107 | 4 | 0.5593 | 0.5064 | 0.9212 |
| BM2766 | 5 | 194 | 0.6450 | 1 | 0.5285 | 0.4817 | 1.0270 |
| BM2792 | 5 | 132 | 0.4084 | 1 | 0.6626 | 0.5991 | 1.1651 |
| BM6414 | 7 | 270 | 0.4618 | 5 | 0.7149 | 0.6814 | 1.3771 |
| BM6573 | 2 | 118 | 0.7672 | 2 | 0.3572 | 0.2934 | 0.5427 |
| BM6598 | 6 | 210 | 0.6450 | 1 | 0.5364 | 0.4964 | 1.0300 |
| LMX503 | 5 | 226 | 0.6336 | 1 | 0.5489 | 0.5081 | 1.0867 |
| LMX780 | 5 | 229 | 0.5076 | 1 | 0.6278 | 0.5659 | 1.1080 |
| LMX836 | 8 | 224 | 0.4275 | 1 | 0.7562 | 0.7310 | 1.6617 |
| LMX1553 | 6 | 192 | 0.7061 | 4 | 0.4788 | 0.4560 | 0.8776 |
| LMX1959 | 5 | 266 | 0.4847 | 3 | 0.6584 | 0.6043 | 1.1549 |
| LMX2019 | 8 | 250 | 0.3321 | 2 | 0.8132 | 0.7918 | 1.7681 |
| LMX2068 | 8 | 289 | 0.7405 | 1 | 0.4411 | 0.4294 | 0.9994 |
| LMX2281 | 5 | 248 | 0.6718 | 0 | 0.5140 | 0.4818 | 0.9819 |
| LMX2382 | 8 | 195 | 0.2710 | 6 | 0.8350 | 0.8150 | 1.7779 |
| LMX2734 | 4 | 210 | 0.4733 | 1 | 0.6826 | 0.6351 | 1.2678 |
| 合计Total | 128 | 51 | |||||
| 平均Average | 5.8 | 0.5189 | 2.3 | 0.6284 | 0.5874 | 1.2062 | |
新窗口打开|下载CSV
1.2 DNA提取与检测
每份材料随机选取10个单株,采集三叶一心期幼嫩叶片混合后液氮中研磨,用新型植物基因组DNA快速提取试剂盒(北京鼎国昌盛生物有限公司)提取基因组DNA,用1%琼脂糖凝胶电泳检测DNA完整性,再用NanoDropND-1000检测浓度并将扩增浓度调成50 ng·μL-1。1.3 SSR引物
根据中国农业科学院作物科学研究所小宗作物课题组通过高通量测序技术开发的糜子基因组SSR引物和文献中发表的引物信息,共选取202对SSR引物进行合成。其中22对来源于连帅等[16],4对来源于王银月等[18],178对来源于未发表的高通量测序开发的糜子SSR引物。对筛选出的SSR标记的正向引物5′末端用6-FAM、HEX、ROX和TAMRA 4种荧光染料中的一种进行标记。引物的合成和标记均由英潍捷基(上海)贸易有限公司完成。1.4 PCR扩增与标记检测
1.4.1 PCR扩增 PCR扩增体系包含11 μL的反应体积,其中PCR Mix(北京擎科生物技术有限公司)5.5 μL、正向引物和反向引物各0.6 μL、基因组DNA 1 μL和ddH2O 3.3 μL。首先通过温度梯度PCR扩增,确定引物的最适退火温度。PCR反应程序(以退火温度55℃为例)为98℃ 3 min;98℃ 10 s,55℃ 10 s,72℃ 10 s,30个循环;72℃ 3 min,4℃保存。1.4.2 变性聚丙烯酰胺凝胶电泳检测 6%的聚丙烯酰胺凝胶,恒功率65 W预电泳15—20 min,每个加样孔点入3 μL样品,65 W恒功率电泳1—1.5 h,银染检测扩增产物。
1.4.3 毛细管电泳荧光检测 将6-FAM和HEX荧光标记以及TAMRA和ROX荧光标记的PCR产物分别用超纯水稀释10—30倍和5—10倍,随后取等体积的上述4种稀释液混合,从中吸取1 μL加到DNA分析仪深孔板中,同时加入0.1 μL LIZ500分子量内标和8.9 μL去离子甲酰胺。将样品在PCR仪上95℃变性5 min,立即取出置于碎冰上冷却10 min后,放置到ABI 3730 XL型DNA分析仪上进行自动荧光检测。
1.5 数据统计与分析
用Genemapper 4.0软件自动生成每个位点的图谱文件,分析扩增片段峰值,读取扩增片段大小;用PowerMarker3.25[26]计算每对引物的等位基因数(allele number)、主要等位基因频率(major allele frequency)、基因多样性指数(gene diversity index,H)、多态性信息含量指数(polymorphism information content,PIC);用Popgen1.32[27]计算Shannon 信息指数(Shannon’s Information index,I);在MEGA 6.06[28]中构建遗传聚类图;用STRUCTURE2.2.3[29]软件进行群体结构分析。2 结果
2.1 引物筛选与验证
利用地理来源远、形态差异大的6份糜子材料对收集的202对SSR引物进行PCR扩增,并对扩增产物进行变性聚丙烯酰胺凝胶电泳检测,淘汰不具多态性或多态性差的引物,筛选出26对条带清晰、多态性较高、扩增稳定的SSR引物。对26对高多态性SSR引物进行毛细管荧光电泳检测,结果表明,其中有22对引物的峰图简单、多态性高、等位变异大小变化符合规律,可用于全部参试材料的遗传多样性分析(表1)。入选的22对引物既适用于传统聚丙烯酰胺凝胶电泳检测技术,也适用于荧光SSR标记-全自动分析检测技术。为了检验荧光标记毛细管电泳检测方法的可靠性,用变性聚丙烯酰胺凝胶电泳检测方法对毛细管电泳检测指纹数据进行了验证。以引物BM2654为例(图1),结果显示在BM2654标记位点上,6%变性聚丙烯酰胺凝胶电泳检测到6个等位基因,与图2荧光毛细管电泳检测到的大小分别是197、200、203、206、209和212 bp的多态性片段长度一一对应,说明荧光标记毛细管电泳检测方法结果可靠,能有效用于后续参试材料的遗传多样性研究。
图1
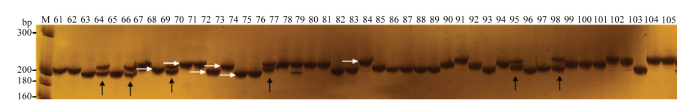 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图145份糜子材料在BM2654标记位点上的带型表现
M:DNA分子量标准;61—105为电子附
Fig. 1Alleles of 45 broomcorn millet accessions at marker locus BM2654
M: DNA molecular weight standard;Number 61-105 is the serial number of varieties listed in supplement table 1; The vertical arrow indicates heterozygous sites band patterns; The horizontal arrow indicates six different alleles
图2
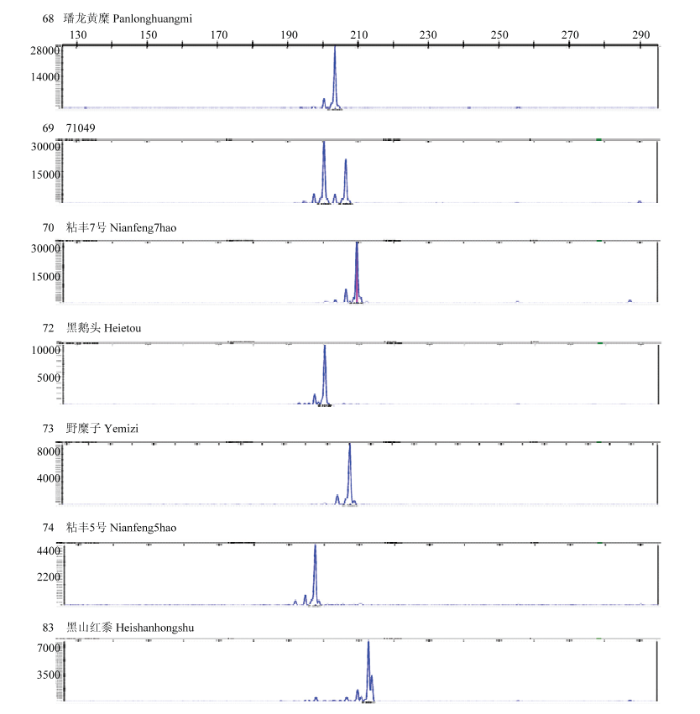 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图27份糜子材料在荧光标记BM2654位点上的等位变异峰图
品种71049在该位点检测到了2个等位变异。纵坐标表示荧光信号强度;横坐标表示6个不同的扩增片段大小(bp)
Fig. 2Peak patterns detected in seven broomcorn millet accessions at fluorescent marker locus BM2654
Two alleles were detected in variety 71049 because of heterozygosity at this locus. The Y axis indicates the intensity of fluorescent signals. The X axis indicates six different sizes of amplified fragment(bp)
2.2 中国糜子种质资源SSR位点多样性分析
利用22对引物在131份材料中共检测出128个主要等位变异,变幅为2—8个,平均每对SSR引物扩增出5.82个主要等位变异,其中引物LMX836、LMX2019、LMX2068、LMX2382扩增出的等位变异最多,为8个。主要等位基因频率为0.3015—0.7672,平均0.5241。多态性信息含量(PIC)的变幅介于0.2934—0.8150,平均0.5890,PIC最大的标记为LMX2382,最小的标记为BM6573。15个位点的PIC>0.5,为高度多态性位点,7个位点的PIC为0.25—0.5,为中度多态性位点。基因多样性指数的变化范围是0.3572—0.8132,平均0.6276,其中,BM6573最低,LMX2019最高。Shannon多样性指数为0.5427—1.7681,平均1.2062,最高为LMX2019,最低为BM6573。基因多样性指数,多态性信息含量,Shannon信息指数呈正相关,能够反映每对标记的鉴别能力。22个SSR位点的多态性丰富,可以检测到不同糜子材料间丰富的遗传多样性。同时利用这22对引物分析了不同生态区不同类型糜子资源的遗传多样性(表2),不同生态区育成品种的基因多样性指数、多态性信息含量、Shannon信息指数的变化范围分别是0.5474—0.5808、0.4953—0.5337、0.9848—1.1074,平均值为0.5608、0.5111和1.0433,其中黄土高原春夏糜子区育成品种的遗传参数最高,北方春糜子区次之。不同生态区农家种的基因多样性指数、多态性信息含量、Shannon信息指数的变化范围分别是0.3838—0.6221、0.3197—0.5725和0.4402—1.1114,平均值为0.5041、0.4525和0.7806,其中北方春糜子区农家种的遗传参数最高,黄土高原春夏糜子区次之。总体来讲,北方春糜子区各项遗传参数均最高,黄土高原春夏糜子区次之,华北夏糜子区各项遗传参数均最低,说明北方春糜子区和黄土高原春夏糜子区的遗传多样性比较丰富,北方春糜子区农家种的遗传多样性丰富,可以从中挖掘优良基因用于品种改良。
Table 2
表2
表2不同生态区糜子的遗传多样性分析
Table 2
| 生态区 Ecotype | 类型 Type | 资源数 Accessions | 等位基因数 Allele number | 基因多样性指数 H | 多态性信息含量 PIC | Shannon信息指数 I |
|---|---|---|---|---|---|---|
| 东北春糜子区 NES | 育成品种 Cultivars | 20 | 3.8636 | 0.5474 | 0.4953 | 0.9848 |
| 农家种 Landraces | 11 | 3.3636 | 0.5373 | 0.4901 | 0.9225 | |
| 材料数 Number | 31 | 4.3182 | 0.5732 | 0.5291 | 1.0562 | |
| 华北夏糜子区 NSU | 育成品种 Cultivars | 0 | ||||
| 农家种 Landraces | 3 | 2.0455 | 0.3838 | 0.3197 | 0.4402 | |
| 材料数 Number | 3 | 2.0455 | 0.3838 | 0.3197 | 0.4402 | |
| 北方春糜子区 NSP | 育成品种 Cultivars | 32 | 4.4091 | 0.5521 | 0.5042 | 1.1074 |
| 农家种 Landraces | 26 | 4.5455 | 0.6221 | 0.5725 | 1.1114 | |
| 材料数 Number | 58 | 5.0455 | 0.6083 | 0.5607 | 1.1283 | |
| 黄土高原春夏糜子区 LPSS | 育成品种 Cultivars | 20 | 4.3636 | 0.5808 | 0.5337 | 1.0379 |
| 农家种 Landraces | 16 | 4.0000 | 0.5250 | 0.4993 | 0.9290 | |
| 材料数 Number | 36 | 5.1364 | 0.6020 | 0.5576 | 1.1222 | |
| 西北春夏糜子区 NWSS | 育成品种 Cultivars | 0 | ||||
| 农家种 Landraces | 3 | 2.1818 | 0.4621 | 0.3811 | 0.5001 | |
| 材料数 Number | 3 | 2.1818 | 0.4621 | 0.3811 | 0.5001 |
新窗口打开|下载CSV
2.3 中国糜子种质资源的遗传相似性分析
利用Popgene1.32计算不同生态区糜子种质间的遗传相似性(表3),结果显示,遗传距离的变化范围为0.0764—0.7251,平均0.3121,遗传一致度的变化范围为0.4843—0.9265,平均0.7465。其中,北方春糜子区和黄土高原春夏糜子区的遗传距离最小(0.0764),遗传一致度最大(0.9265),这两个生态区的地理位置相邻,生态环境差异较小,亲缘关系较近。华北夏糜子区和西北春夏糜子区的遗传距离最大(0.7251),遗传一致度最小(0.4843),这两个生态区的地理分布较远,生态环境差异大,亲缘关系较远。说明遗传相似性与地理来源密切相关,地理来源越近,遗传距离越小,遗传一致度越高,亲缘关系越近。Table 3
表3
表3不同生态区糜子资源的遗传距离和遗传一致度
Table 3
| 生态区 Ecotype | 东北春糜子区 NES | 北方春糜子区 NSP | 华北夏糜子区 NSU | 西北春夏糜子区 NWSS | 黄土高原春夏糜子区 LPSS |
|---|---|---|---|---|---|
| 东北春糜子区 NES | — | 0.8548 | 0.6408 | 0.7078 | 0.8814 |
| 北方春糜子区 NSP | 0.1569 | — | 0.5993 | 0.8552 | 0.9265 |
| 华北夏糜子区 NSU | 0.4451 | 0.5120 | — | 0.4843 | 0.6463 |
| 西北春夏糜子区 NWSS | 0.3456 | 0.1564 | 0.7251 | — | 0.8684 |
| 黄土高原春夏糜子区 LPSS | 0.1263 | 0.0764 | 0.4364 | 0.1411 | — |
新窗口打开|下载CSV
2.4 基于遗传距离的中国糜子种质资源聚类分析
基于UPGMA对不同生态区糜子材料进行聚类(图3),在遗传距离0.1284处将5个生态区划分为4个组群(组群Ⅰ、Ⅱ、Ⅲ和Ⅳ)。组群Ⅰ将华北夏糜子区归为一类,材料来自于山东省、河北省。组群Ⅱ将西北春夏糜子区归为一类,材料来自于新疆维吾尔自治区。组群Ⅲ将东北春糜子区归为一类,材料来自于黑龙江省、吉林省、辽宁省。组群Ⅳ将北方春糜子区和黄土高原春夏糜子区归为一类,材料来自于宁夏回族自治区、山西省、陕西省、内蒙古自治区、甘肃省、青海省。聚类结果与遗传相似性分析结果一致。图3
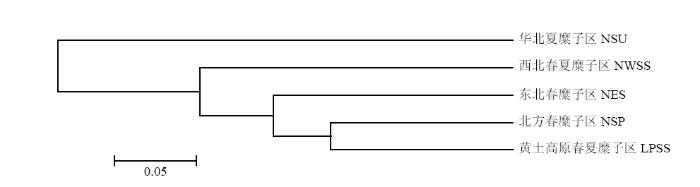 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3基于遗传距离的不同生态区糜子资源聚类图
NES:东北春糜子区Northeast spring-sowing ecotype;NSU:华北夏糜子区Northern summer-sowing ecotype;NSP:北方春糜子区Northern spring-sowing ecotype;LPSS:黄土高原春夏糜子区Loess Plateau spring&summer-sowing ecotype;NWSS:西北春夏糜子区Northwest spring&summer-sowing ecotype
Fig. 3Cluster diagram of broomcorn millet accessions with different ecotypes based on genetic distance
基于UPGMA对131份糜子材料进行聚类分析(图4),131份糜子材料聚为7个组群。组群A有30份材料,包括黑龙江省12份、吉林省4份、辽宁省1份、甘肃省2份、山西省3份、陕西省1份、宁夏回族自治区1份、青海省6份,大部分基因型属于东北春糜子区。组群B有7份材料,其中河北省2份、内蒙古自治区2份、宁夏回族自治区、山东省、陕西省各1份,主要属于华北夏糜子区,组群C有3份材料,包括来自山西省的品黍1号,辽宁省的本溪褐糜子和山西省的灰脸蛋糜,表明这3份材料遗传背景相似且与其他材料的遗传差异较大。组群D包括16份材料,其中内蒙古自治区5份、河北省7份、山西省3份、青海省1份,均来自北方春糜子区。组群E包含3份材料,其中内蒙古自治区2份,陕西省1份。组群F有21份材料,包括山西省5份、陕西省4份、甘肃省2份、新疆维吾尔自治区2份,宁夏回族自治区2份、辽宁省1份、吉林省5份,主要属于黄土高原春夏糜子区。组群G包含51份材料,包括山西省11份,内蒙古自治区11份,甘肃省8份,宁夏回族自治区11份、黑龙江省4份,新疆维吾尔自治区、青海省、陕西省、吉林省、辽宁省、河北省各1份,主要属于北方春糜子区和黄土高原春夏糜子区。
图4
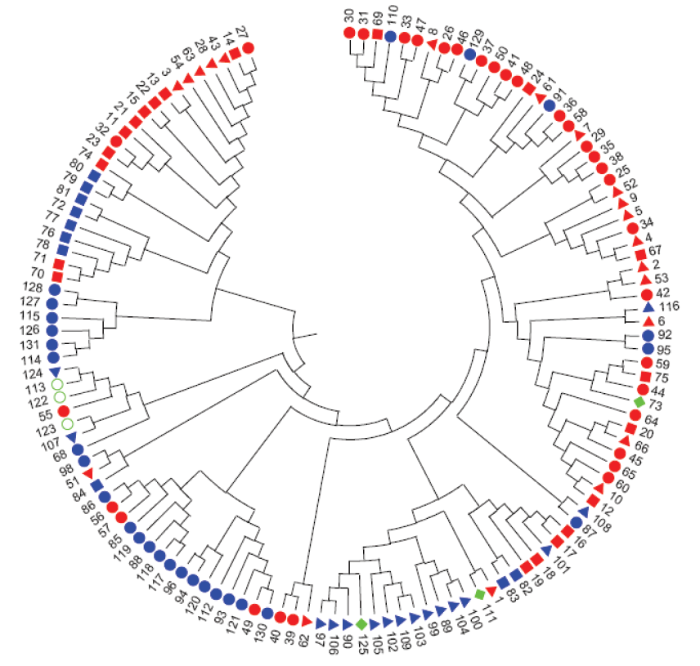 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4基于荧光SSR分子标记分析的131份糜子材料聚类图
北方春糜子区育成品种 Cultivars of NSP; 北方春糜子区农家种 Landraces of NSP; 黄土高原春夏糜子区育成品种 Cultivars of LPSS; 黄土高原春夏糜子区农家种 Landraces of LPSS; 东北春糜子区育成品种 Cultivars of NES; 东北春糜子区农家种 Landraces of NES; 华北夏糜子区农家种 Landraces of NSU; 西北春夏糜子区农家种 Landraces of NWSS
Fig. 4Cluster diagram of 131 broomcorn millet accessions based on data from 22 SSR markers
东北春糜子区的育成品种中,来自黑龙江省的材料主要聚在组群A中,来自吉林省的材料主要聚在组群F中,东北春糜子区的农家种与育成品种聚类结果一致。北方春糜子区的育成品种主要聚在组群G中,农家种主要聚在A、D中,在组群G中也零星出现,其中,来自青海省的农家种集中聚在组群A中。黄土高原春夏糜子区的育成品种聚在组群A和G中,农家种集中聚在组群F中。来自华北夏糜子区的3份材料全部聚在组群B中。来自西北春夏糜子区的3份材料,2份聚在组群F中,1份聚在组群G中。来自北方春糜子区和黄土高原春夏糜子区的育成品种多数聚在组群G中,说明这两个生态区的部分育成品种遗传背景相似。农家种在各组群中的分布与地理来源密切相关,部分育成品种的遗传背景可能包含其他生态区的基因型,在各组群中的分布没有明显的区域性。
2.5 中国糜子种质资源的群体遗传结构分析
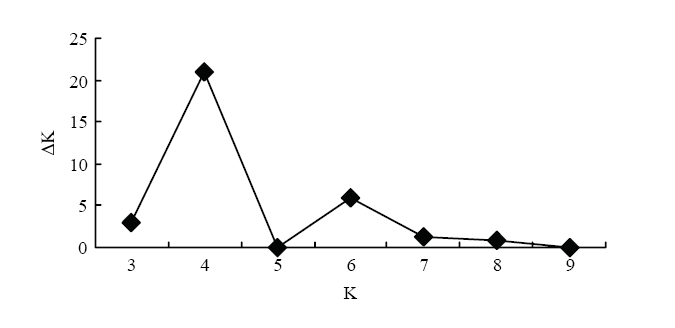
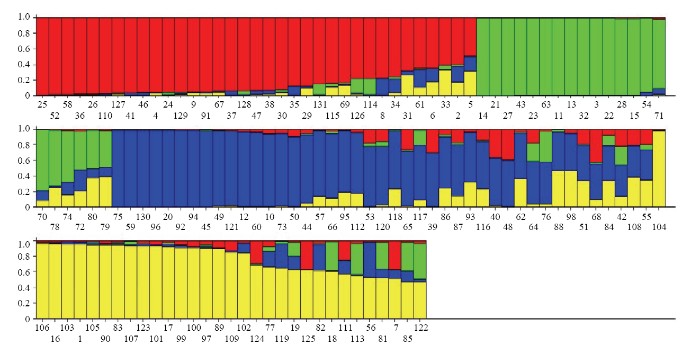
用STRUCTURE软件分析来自中国不同地区的131份糜子材料,将K设为2—10,绘制K与ΔK的关系图(图5),K=4时,ΔK最大,因此在K=4的模式下分析糜子品种的遗传结构[30],并计算出每个类群的最大Q值分布。不同的色块代表不同的类群(图6,表4),类群Ⅰ为红色部分(35份),代表北方春糜子区,主要包含甘肃省8份、青海省7份、内蒙古自治区7份、宁夏回族自治区6份、山西省3份、黑龙江省3份、陕西省1份。类群Ⅱ为绿色部分(21份),包括黑龙江省10份、吉林省3份、山西省3份、甘肃省2份、宁夏回族自治区、陕西省和辽宁省各1份,主要来自东北春糜子区。类群Ⅲ为蓝色部分(43份),包括山西省15份、内蒙古自治区9份、河北省6份、宁夏回族自治区4份、陕西省、黑龙江省、辽宁省各2份、吉林省、新疆维吾尔自治区、青海省各1份,主要来自北方春糜子区。群组Ⅳ为黄色部分(32份),包括山西省6份、陕西省5份、吉林省6份、宁夏回族自治区4份、河北省3份、甘肃省、新疆维吾尔自治区各2份、辽宁省、内蒙古自治区、黑龙江省、山东省各1份,主要来自黄土高原春夏糜子区。图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5131份糜子材料的K值与Δk值的关系
Fig. 5Graphical relationship between K and Δk for 131 broomcorn millet accessions
图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6131份糜子材料的群体遗传结构分析
Fig. 6Population genetic structure of 131 broomcorn millet accessions
Table 4
表4
表4131份糜子材料在各组群中的分布(K=4)
Table 4
| 省份 Province | 红色 Red | 绿色 Green | 蓝色 Blue | 黄色 Yellow |
|---|---|---|---|---|
| 山西省Shanxi | 3 | 3 | 15 | 6 |
| 甘肃省Gansu | 8 | 2 | 2 | |
| 内蒙古自治区Inner Mongolia | 7 | 9 | 1 | |
| 青海省Qinghai | 7 | 1 | ||
| 宁夏回族自治区Ningxia | 6 | 1 | 4 | 4 |
| 黑龙江省Heilongjiang | 3 | 10 | 2 | 1 |
| 陕西省Shaanxi | 1 | 1 | 2 | 5 |
| 辽宁省 Liaoning | 1 | 2 | 1 | |
| 吉林省 Jilin | 3 | 1 | 6 | |
| 河北省 Hebei | 6 | 3 | ||
| 新疆维吾尔自治区 Xinjiang | 1 | 2 | ||
| 山东省 Shandong | 1 | |||
| 合计 Total | 35 | 21 | 43 | 32 |
新窗口打开|下载CSV
黄土高原春夏糜子区的育成品种在组群Ⅰ、Ⅱ、Ⅲ中都有出现,农家种集中分布在组群Ⅳ中。北方春糜子区的育成品种和农家种主要分布在组群Ⅰ和组群Ⅲ中,其中来自青海省的农家种全部分布在组群Ⅰ中,来自河北省北部的农家种分布在组群Ⅲ中,东北春糜子区的育成品种和农家种主要分布在组群Ⅱ和Ⅳ中,其中来自黑龙江省的材料主要分布在组群Ⅱ中,来自吉林省的材料主要分布在组群Ⅳ中,华北夏糜子区的材料均分布在组群Ⅳ中。西北春夏糜子区的材料分布在组群Ⅲ和Ⅳ中。遗传结构分析结果与聚类结果基本一致。
STRUCTURE群体结构分析中,当某一材料在某类群中的Q≥0.6时,则认为该材料血缘关系相对比较单一,否则认为该材料血缘关系来源复杂[9]。131份糜子材料在各个群体中的Q值分布见表5,131份糜子材料中有107个品种Q>0.6,占所有供试材料的81.7%,Q>0.8和Q>0.9的品种分别占58%和48.1%,说明各群体中大部分品种亲缘关系比较单一,较少品种含有其他类群的基因成分。品黍1号在4个类群中的Q值分别为0.190、0.011、0.453和0.346;本溪褐糜子在4个类群中的Q值分别为0.074、0.134、0.443和0.349;灰脸蛋糜在4个类群中的Q值分别为0.047、0.004、0.478和0.471,它们的基因都主要来源于类群Ⅲ和类群Ⅳ中,聚类分析也被单独聚为一个小支,说明这三个品种的遗传背景比较相似,与聚类结果一致。
Table 5
表5
表5各群体Q值分布
Table 5
| 类群 Group | 各群体品种数量 Number of varieties in each group | 品种数量Number of varieties | |||
|---|---|---|---|---|---|
| Q<0.6 | Q≥0.6 | Q≥0.8 | Q≥0.9 | ||
| 1 | 35 | 1 | 34 | 26 | 21 |
| 2 | 21 | 3 | 18 | 15 | 14 |
| 3 | 43 | 12 | 31 | 18 | 14 |
| 4 | 32 | 7 | 25 | 18 | 15 |
| 合计 Total | 131 | 24 | 107 | 76 | 63 |
新窗口打开|下载CSV
3 讨论
3.1 荧光SSR标记分析技术在糜子上的应用
SSR标记的检测可采用琼脂糖凝胶电泳、非变性聚丙烯酰胺凝胶电泳、变性聚丙烯酰胺凝胶电泳和毛细管电泳荧光检测法,因方法不同,结果也会有所差别。基于全自动基因分析仪的SSR荧光标记技术是用6-FAM、HEX、ROX和TAMRA 4种荧光染料中的一种对引物进行标记,扩增的PCR产物和标准分子量样品(内标)在同一毛细管泳道中电泳,利用软件进行图像收集和扩增片段的大小分析,实现了SSR标记与高效、自动化技术的结合,比传统聚丙烯酰胺凝胶电泳检测技术准确性更高,重复性更好。本试验同时采用变性聚丙烯酰胺和毛细管电泳双向检测技术分析22对引物在131份糜子材料中的遗传多样性,检测结果基本一致。但传统的变性聚丙烯酰胺凝胶电泳检测技术在不同板之间数据整合比较困难,当片段差异较小时,读带也有一定的困难,荧光SSR分子标记技术有效解决了不同板,不同实验室数据整合问题。本研究所选用的22对标记中,有11对与连帅等[15]相同,这11对检测出的等位变异数平均为6.5个,而连帅用非变性聚丙烯酰胺凝胶电泳检测出的等位变异数平均为3个,这说明SSR荧光标记检测技术能检测到传统聚丙烯酰胺凝胶电泳发现不了的等位变异,结果更加准确可靠。
3.2 SSR标记在糜子种质资源遗传多样性研究中的应用
CHO等[13]基于糜子基因组DNA富集SSR文库开发了首批糜子的SSR标记,利用25个标记在50份材料中检测到110个等位变异,平均每个标记的等位变异数为4.4个,平均PIC为0.33。HUNT等[14]用其中的16个标记检测了98份欧亚大陆糜子地方品种的遗传多样性,平均每个位点检测到4.9个等位变异,平均基因多样性指数和多态性信息含量分别为0.391和0.360。连帅等[15]用63对引物分析了来自国内外的192份糜子品种的遗传多样性,平均每个SSR位点检测到2.56个等位变异,平均PIC值为0.4855。王瑞云等[24]利用15个糜子特异性SSR标记检测来自中国11个省(区)的132份糜子材料,平均基因多样性指数和多态性信息含量分别为0.5298和0.4864。本研究选用22对SSR引物对131份糜子材料进行分析,共检测出128个主要等位变异,平均每个5.82个,基因多样性指数为0.3572—0.8132,平均0.6284;多态性信息含量为0.2934—0.8150,平均0.5874;Shannon多样性指数为0.5427—1.7681,平均1.2062。本试验所用SSR标记的多态性均高于前人研究结果。一方面本试验的引物经过两次筛选,引物多态性高,同时参试材料数量多、来源广,遗传多样性丰富;另一方面所用的检测方法分辨率更高,准确性更好。这也表明对已开发的糜子SSR标记进行进一步筛选的必要性,筛选高多态性引物用于糜子遗传多样性研究和DNA指纹图谱的构建。此外,建立高分辨率且成本较低的分子标记检测方法对于促进糜子遗传多样性研究也具有非常重要的意义。3.3 中国糜子种质资源的遗传差异
中国糜子资源分布广泛,主要分布在七个生态区,分别为东北春糜子区、华北夏糜子区、北方春糜子区、黄土高原春夏糜子区、西北春夏糜子区、青藏高原春糜子区和南方秋冬糜子区。不同生态区,不同材料间的遗传多样性以及遗传关系成为近年来糜子研究的热点。王瑞云等[31]利用高基元微卫星标记分析了96份糜子材料的遗传多样性并提出黄土高原和北方春糜子区的遗传多样性最丰富。连帅等[15]采用63对高多态性SSR标记研究了来自于国内外的192份糜子地方品种和野生材料的遗传多样性,并发现内蒙古、东北、黄土高原地区种质资源遗传关系较其他地区更为复杂。薛延桃等[17]对近几年新收集引进的国外糜子地方品种和野生材料共计146份进行分析发现国内野生材料的遗传多样性高于国外地方品种,且河北群体的遗传多样性最为丰富。HU等[2]发现黄土高原地区的遗传多样性最丰富,可能是糜子的起源中心。董俊丽等[32]对糜子骨干种质进行遗传多样性分析发现遗传多样性与生态环境密切相关,且山西糜子资源的遗传多样性最丰富。本研究分析了来自5个生态区的131份糜子育成品种和农家种的遗传多样性和群体结构,发现北方春糜子区和黄土高原春夏糜子区的遗传多样性最丰富,与王瑞云等[31]结论一致。通过比较本研究与前人研究结果发现不同结果之间遗传差异存在较大差别,究其原因,可能有以下几个方面:一是各研究所采用的试验材料种类有所差异,如连帅等[15]和薛延桃等[17]文章中均包含了一定数量的国外品种以及国内野生资源,从而造成不同区域遗传多样性有所差异;二是不同研究中对同一生态区的试验材料选择数量的差别也会造成结果出现差异,如本研究选用北方春糜子区和黄土高原春夏糜子区的材料较多,西北春夏糜子区和南方秋冬糜子区的材料较少,为更加准确评估中国糜子资源的遗传多样性,选材需更加丰富。群体结构分析结果表明,甘肃省材料66.7%集中在群组Ⅰ中,青海省材料87.5%聚在群组Ⅰ中,黑龙江省材料66.7%聚在群组Ⅱ中,山西省的材料在各个组群中均有出现,与聚类结果基本一致。说明大部分糜子品种的遗传差异与地理来源相关,有些品种没有明显的区域特征,可能是不同区域间品种经过多次基因重组所致。
3.4 中国糜子育成品种和农家种的遗传多样性比较
国内外对糜子的遗传多样性和群体结构分析有很多[13,14,15,16,17,18],但有关中国糜子育成品种和农家种遗传差异的相关报道并不多见,本研究分析比较了来自中国不同生态区的72份糜子育成品种及当地主要农家种的遗传多样性和群体遗传结构,发现北方春糜子区农家种的遗传多样性高于育成品种,东北春糜子区和黄土高原春夏糜子区农家种的遗传多样性略低于育成品种。聚类分析和群体结构分析结果表明,东北春糜子区的育成品种和农家种并不独立成群,在各组群中的分布基本一致,这可能因为糜子育成品种主要以农家种为遗传背景选育而来,而黄土高原春夏糜子区的育成品种和农家种分布在不同的组群中,这说明黄土高原春夏糜子区在育种过程中引种资源广泛,有效利用优良种质用于育种创新。北方春糜子区的农家种具有更高的遗传多样性,可为糜子新品种选育过程中挖掘优良基因和拓宽遗传基础提供重要的利用价值。4 结论
北方春糜子区和黄土高原春夏糜子区的遗传多样性高于东北春糜子区,糜子的遗传差异与地理来源相关,东北春糜子区的育成品种主要以农家种为遗传背景选育而来,黄土高原春夏糜子区在育种过程中引种资源广泛,与其他生态区存在基因交流。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1073/pnas.0809960106URL [本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
DOI:10.3724/SP.J.1006.2010.01425URLMagsci [本文引用: 1]

近年来,随着基因组测序等多种技术实现突破,基因组学、表型组学等多门“组学”及生物信息学得到迅猛发展,作物育种理论和技术也发生了重大变革。以分子标记育种、转基因育种、分子设计育种为代表的现代作物分子育种技术逐渐成为了全世界作物育种的主流,在我国也正在成为作物遗传改良的重要手段。本文在界定分子育种的基础上,简要分析了中国作物分子育种研究现状和面临的问题,探讨了未来我国作物分子育种的发展策略。
DOI:10.3724/SP.J.1006.2010.01425URLMagsci [本文引用: 1]

近年来,随着基因组测序等多种技术实现突破,基因组学、表型组学等多门“组学”及生物信息学得到迅猛发展,作物育种理论和技术也发生了重大变革。以分子标记育种、转基因育种、分子设计育种为代表的现代作物分子育种技术逐渐成为了全世界作物育种的主流,在我国也正在成为作物遗传改良的重要手段。本文在界定分子育种的基础上,简要分析了中国作物分子育种研究现状和面临的问题,探讨了未来我国作物分子育种的发展策略。
DOI:10.1007/BF00223653URLPMID:24186027 [本文引用: 1]

The potential use of random amplified polymorphic DNA (RAPD) was evaluated as a source of genetic markers for studying variation among four species of Panicum and within the crop species P. miliaceum and P. sumatrense . Polymorphism in RAPD markers was observed across and within species. The four species were distinct in RAPD patterns and were separated at low correlation values even with small samples involving single genotypes per species. Accessions of P. miliaceum were grouped according to geographical regions of origin. The study demonstrated that unlike isozyme and protein electrophoresis patterns, RAPD markers can be applied to studying genetic diversity, defining gene pools, and identifying cultivars for this group of millets.
[本文引用: 1]
DOI:10.1007/s10681-005-5814-7URL [本文引用: 1]

Morphological characterization of 20 common millet ( Panicum miliaceum L., 2 n = 4 x = 36) cultivars and landraces revealed four distinct clusters which were apparently consistent with the grain colors of black, black and brown, red, yellow, and white. Seed remains of medieval millet, recovered from a 15th century layer (King's Palace, Budapest, Hungary), showed reddish yellow grain color after rehydrating on tissue culture medium that was close to grain color of modern cultivar Omszkoje . aDNA of medieval commom millet was extracted successfully, analyzed and compared to modern common millets by ISSR, SSR, CAP and mtDNA. Analyses of fragments and sequences revealed polymorphism at seven ISSR loci (15 alleles) and at the 5S-18S rDNA locus of mtDNA. CAP analysis of the 5S-18S rDNA fragment revealed no SNPs in the restriction sites of six endonucleases Taq I, Bsu RI, Hinf I, Mbo I, Alu I and Rsa I. Sequence alignments of the restriction fragments Rsa I also revealed consensus sequence in the medieval sample compared to a modern variety. An attempted phenotype reconstruction indicated that medieval common millet showed the closest morphological similarity to modern millet cultivar Omszkoje .
DOI:10.4236/ajps.2014.51023URL [本文引用: 1]
DOI:10.3864/j.issn.0578-1752.2016.14.011 [本文引用: 2]

【目的】枣原产中国,种质资源丰富。对来自中国22个省区不同用途的255个枣品种进行SSR分析,揭示这些不同产地来源的枣种质资源之间的亲缘关系和群体遗传结构,为枣种质资源的科学管理和分子标记辅助育种提供参考。【方法】利用改良CTAB提取供试枣种质的基因组DNA,以前期枣基因组测序挖掘出的SSR引物为基础,进行高效率引物筛选,并利用SSR分子标记技术对255份枣种质资源的基因组DNA进行PCR扩增,然后利用8%聚丙烯酰胺凝胶电泳分离,银染后显色。根据条带有无统计数据,计算出多态性位点百分率(PIC),用NTSYS软件进行UPGMA聚类分析;利用Structure软件分析群体遗传结构,计算出最适群体数目,构建遗传结构图。【结果】从64对SSR引物中筛选出23对高效率SSR引物,在供试材料中共检测出117个多态性位点,各引物扩增的多态性位点数为2—10条,每对引物平均扩增多态位点数为5.09个,PIC值变幅为0.359—0.727,平均为0.548,这些多态性引物可应用到其他枣种质资源的研究中;建立了只需1—2个标记就可鉴别出来的部分枣品种的SSR指纹,可用于这些品种的快速分子鉴定;255个枣品种的聚类分析将所有枣品种分为15个亚类,包括4个大类和11个小类,不同品种间的相似系数范围0.71—1.00,其中北京花生枣单独聚为一类,与其他枣品种关系较远;‘奉节鸡蛋枣’和‘溆浦鸡蛋枣’、‘陕西奶枣’和‘天津大马牙枣’的相似系数均为1.00;结合聚类图、供试品种的用途和原产地分析,不同枣品种间的亲缘关系与品种原产地有一定相关性,但和品种用途没有显著相关性。群体结构分析中,通过绘制K与ΔK的关系图,K=15时,ΔK最大,据此将255个枣品种也同样划分为15个群体,与聚类分析结果基本一致;进一步分析表明,各群体中大部分品种血缘关系比较单一,较少品种含有其他类群的遗传成分。总体看,山西或陕西的枣品种出现在绝大部分居群中,说明这两个省的资源在不同群体间的基因交流中发挥了重要作用;南方栽培区域中来自湖南的枣品种形成了相对独立的居群,可能是其起源相对单一,且在长期栽培过程中和其他产地枣品种间基因交流较少所致。上述结果表明,供试枣品种中与来源区域相关性明显的品种由相同地域内枣品种演化而来,而另一部分与来源产地相关性不明显的品种则是由不同区域间品种经过频繁的基因交流和重组选育而来,融合了不同区域品种的特点,从而没有了明显的区域特征。【结论】不同地理环境在枣品种的群体进化中发挥了较重要的作用,影响了不同产地间枣种质资源的遗传结构组成。
DOI:10.3864/j.issn.0578-1752.2016.14.011 [本文引用: 2]

【目的】枣原产中国,种质资源丰富。对来自中国22个省区不同用途的255个枣品种进行SSR分析,揭示这些不同产地来源的枣种质资源之间的亲缘关系和群体遗传结构,为枣种质资源的科学管理和分子标记辅助育种提供参考。【方法】利用改良CTAB提取供试枣种质的基因组DNA,以前期枣基因组测序挖掘出的SSR引物为基础,进行高效率引物筛选,并利用SSR分子标记技术对255份枣种质资源的基因组DNA进行PCR扩增,然后利用8%聚丙烯酰胺凝胶电泳分离,银染后显色。根据条带有无统计数据,计算出多态性位点百分率(PIC),用NTSYS软件进行UPGMA聚类分析;利用Structure软件分析群体遗传结构,计算出最适群体数目,构建遗传结构图。【结果】从64对SSR引物中筛选出23对高效率SSR引物,在供试材料中共检测出117个多态性位点,各引物扩增的多态性位点数为2—10条,每对引物平均扩增多态位点数为5.09个,PIC值变幅为0.359—0.727,平均为0.548,这些多态性引物可应用到其他枣种质资源的研究中;建立了只需1—2个标记就可鉴别出来的部分枣品种的SSR指纹,可用于这些品种的快速分子鉴定;255个枣品种的聚类分析将所有枣品种分为15个亚类,包括4个大类和11个小类,不同品种间的相似系数范围0.71—1.00,其中北京花生枣单独聚为一类,与其他枣品种关系较远;‘奉节鸡蛋枣’和‘溆浦鸡蛋枣’、‘陕西奶枣’和‘天津大马牙枣’的相似系数均为1.00;结合聚类图、供试品种的用途和原产地分析,不同枣品种间的亲缘关系与品种原产地有一定相关性,但和品种用途没有显著相关性。群体结构分析中,通过绘制K与ΔK的关系图,K=15时,ΔK最大,据此将255个枣品种也同样划分为15个群体,与聚类分析结果基本一致;进一步分析表明,各群体中大部分品种血缘关系比较单一,较少品种含有其他类群的遗传成分。总体看,山西或陕西的枣品种出现在绝大部分居群中,说明这两个省的资源在不同群体间的基因交流中发挥了重要作用;南方栽培区域中来自湖南的枣品种形成了相对独立的居群,可能是其起源相对单一,且在长期栽培过程中和其他产地枣品种间基因交流较少所致。上述结果表明,供试枣品种中与来源区域相关性明显的品种由相同地域内枣品种演化而来,而另一部分与来源产地相关性不明显的品种则是由不同区域间品种经过频繁的基因交流和重组选育而来,融合了不同区域品种的特点,从而没有了明显的区域特征。【结论】不同地理环境在枣品种的群体进化中发挥了较重要的作用,影响了不同产地间枣种质资源的遗传结构组成。
DOI:10.3724/SP.J.1006.2010.01820URLMagsci [本文引用: 1]

玉米自交系遗传关系和遗传结构的解析,对自交系类群划分和杂交组配具有重要的指导意义。本文选用玉米基因组的112个SSR标记对我国97个玉米自交系进行遗传关系和遗传结构分析,并评价了遗传距离聚类和模型聚类方法在玉米自交系遗传关系研究中的应用价值。结果表明,模型聚类方法更适于玉米自交系的遗传关系研究。解析自交系的遗传基础发现,各类群中均有大量自交系含有其他类群的遗传成分。根据模型聚类结果,97个自交系被划分为PB、Reid、塘四平头和旅大红骨4个类群。Reid群与旅大红骨群的遗传关系最近,与塘四平头群遗传关系最远。为了实现杂种优势模式的简化,4个类群可被简化为3大种质类群[A(旅大红骨群与Reid群)、B(PB群)、C(塘四平头群)],或2大种质类群[A(旅大红骨群、Reid群、PB群)、B(塘四平头群)]。研究结果为自交系的改良和利用及杂种优势模式确定提供了理论基础。
DOI:10.3724/SP.J.1006.2010.01820URLMagsci [本文引用: 1]

玉米自交系遗传关系和遗传结构的解析,对自交系类群划分和杂交组配具有重要的指导意义。本文选用玉米基因组的112个SSR标记对我国97个玉米自交系进行遗传关系和遗传结构分析,并评价了遗传距离聚类和模型聚类方法在玉米自交系遗传关系研究中的应用价值。结果表明,模型聚类方法更适于玉米自交系的遗传关系研究。解析自交系的遗传基础发现,各类群中均有大量自交系含有其他类群的遗传成分。根据模型聚类结果,97个自交系被划分为PB、Reid、塘四平头和旅大红骨4个类群。Reid群与旅大红骨群的遗传关系最近,与塘四平头群遗传关系最远。为了实现杂种优势模式的简化,4个类群可被简化为3大种质类群[A(旅大红骨群与Reid群)、B(PB群)、C(塘四平头群)],或2大种质类群[A(旅大红骨群、Reid群、PB群)、B(塘四平头群)]。研究结果为自交系的改良和利用及杂种优势模式确定提供了理论基础。
DOI:10.3864/j.issn.0578-1752.2012.06.002URLMagsci [本文引用: 1]

【目的】从分子水平优化并构建用于中国苦荞种质资源遗传多样性分析的SSR分子标记体系,为综合评价中国苦荞种质资源提供依据。【方法】以50份苦荞种质为试验材料,用正交设计法[L16(45)]筛选适用于苦荞SSR标记分析的PCR反应体系,浓度梯度检测最佳胶分离效果,并从250对不同科属作物SSR引物中筛选出19对引物进行苦荞遗传多样性分析。【结果】优化的苦荞SSR反应体系为DNA模板30 ng,Taq酶2.0 U•L-1,dNTP、引物和Mg2+终浓度分别为150 μmol•L-1、0.1 μmol•L-1、2.0 mmol•L-1,总体积为25 μL,6%聚丙烯酰胺凝胶电泳检测。SSR引物筛选率为7.6%,蓼科同属甜荞的SSR引物适用于苦荞SSR扩增。19对引物共检测到157个等位变异,每对SSR引物检测到的等位变异2—11个,平均等位变异(NA)7.42个,平均多态性信息量(PIC)0.888,平均鉴定力(DP)5.684,2对为SSR骨干引物。利用Popgen Ver.1.31软件,当遗传相似度(GS)为0.578时,50份苦荞材料被分为5个组群,聚类结果与苦荞地理分布相关性不大。四川苦荞资源组群各遗传多样性参数均最高,该区域苦荞种质资源多样性最丰富。利用骨干引物可鉴定部分近缘苦荞品种。【结论】构建的SSR分子标记体系适用于中国苦荞种质资源遗传多样性分析,甜荞SSR引物可用于苦荞SSR标记分析,TBP5和Fes2695为苦荞SSR骨干引物,50份苦荞材料遗传多样性丰富,可划分为5个组群。
DOI:10.3864/j.issn.0578-1752.2012.06.002URLMagsci [本文引用: 1]

【目的】从分子水平优化并构建用于中国苦荞种质资源遗传多样性分析的SSR分子标记体系,为综合评价中国苦荞种质资源提供依据。【方法】以50份苦荞种质为试验材料,用正交设计法[L16(45)]筛选适用于苦荞SSR标记分析的PCR反应体系,浓度梯度检测最佳胶分离效果,并从250对不同科属作物SSR引物中筛选出19对引物进行苦荞遗传多样性分析。【结果】优化的苦荞SSR反应体系为DNA模板30 ng,Taq酶2.0 U•L-1,dNTP、引物和Mg2+终浓度分别为150 μmol•L-1、0.1 μmol•L-1、2.0 mmol•L-1,总体积为25 μL,6%聚丙烯酰胺凝胶电泳检测。SSR引物筛选率为7.6%,蓼科同属甜荞的SSR引物适用于苦荞SSR扩增。19对引物共检测到157个等位变异,每对SSR引物检测到的等位变异2—11个,平均等位变异(NA)7.42个,平均多态性信息量(PIC)0.888,平均鉴定力(DP)5.684,2对为SSR骨干引物。利用Popgen Ver.1.31软件,当遗传相似度(GS)为0.578时,50份苦荞材料被分为5个组群,聚类结果与苦荞地理分布相关性不大。四川苦荞资源组群各遗传多样性参数均最高,该区域苦荞种质资源多样性最丰富。利用骨干引物可鉴定部分近缘苦荞品种。【结论】构建的SSR分子标记体系适用于中国苦荞种质资源遗传多样性分析,甜荞SSR引物可用于苦荞SSR标记分析,TBP5和Fes2695为苦荞SSR骨干引物,50份苦荞材料遗传多样性丰富,可划分为5个组群。
DOI:10.3864/j.issn.0578-1752.2013.20.003URLMagsci [本文引用: 1]

【目的】分析中国栽培绿豆种质资源的遗传多样性、亲缘关系和遗传分化,为资源的有效利用、新基因的挖掘和新品种选育奠定基础。【方法】利用40对SSR引物对18个不同地理来源(共272份种质)的绿豆群体进行遗传多样性分析。【结果】共检测到125个等位基因,平均等位基因数(NA)为3.1个,平均有效等位基因数(NE)为1.8个,平均Nei’s基因多样性(H)为0.4233,平均多态性信息含量(PIC)为0.3497,平均期望杂合度(He)为0.4241,平均Shannon信息指数(I)为0.6754,比较发现,河北、山东和安徽是绿豆资源遗传变异较为丰富的地区;平均观测杂合度(Ho)为0.1001,种群内总近交系数(Fis)为0.6759,表明中国绿豆种质间存在一定程度地近交现象;18个参试群体整体水平上的基因流(Nm)值为0.6936,种群间遗传分化系数(Fst)为0.2649,遗传变异水平较高;基于Popgene软件的聚类结果可将272份参试个体聚为2大类,将18个参试群体分为3大类,群体间地理来源越近,亲缘关系也越近。【结论】中国绿豆种质资源遗传多样性较高;地理生态条件等对绿豆种质资源的遗传变异影响很大;群体间遗传分化较大,但同时也存在一定程度地近交现象。
DOI:10.3864/j.issn.0578-1752.2013.20.003URLMagsci [本文引用: 1]

【目的】分析中国栽培绿豆种质资源的遗传多样性、亲缘关系和遗传分化,为资源的有效利用、新基因的挖掘和新品种选育奠定基础。【方法】利用40对SSR引物对18个不同地理来源(共272份种质)的绿豆群体进行遗传多样性分析。【结果】共检测到125个等位基因,平均等位基因数(NA)为3.1个,平均有效等位基因数(NE)为1.8个,平均Nei’s基因多样性(H)为0.4233,平均多态性信息含量(PIC)为0.3497,平均期望杂合度(He)为0.4241,平均Shannon信息指数(I)为0.6754,比较发现,河北、山东和安徽是绿豆资源遗传变异较为丰富的地区;平均观测杂合度(Ho)为0.1001,种群内总近交系数(Fis)为0.6759,表明中国绿豆种质间存在一定程度地近交现象;18个参试群体整体水平上的基因流(Nm)值为0.6936,种群间遗传分化系数(Fst)为0.2649,遗传变异水平较高;基于Popgene软件的聚类结果可将272份参试个体聚为2大类,将18个参试群体分为3大类,群体间地理来源越近,亲缘关系也越近。【结论】中国绿豆种质资源遗传多样性较高;地理生态条件等对绿豆种质资源的遗传变异影响很大;群体间遗传分化较大,但同时也存在一定程度地近交现象。
DOI:10.1007/s13258-010-0007-8URL [本文引用: 3]

Millets such as proso millet have excellent nutritional properties and could become a basic resource for crop breeding programs and food diversification. In this study, 25 polymorphic microsatellite markers were developed and characterized through construction of an SSR-enriched library from genomic DNA of proso millet ( Panicum miliaceum L.). In total, 110 alleles were detected, with an average of 4.4 alleles per locus. Values of major allele frequency ( M AF ) and expected heterozygosity ( H E ) ranged from 0.36 to 0.98 (mean = 0.73) and from 0.04 to 0.74 (mean = 0.37), respectively. The mean genetic similarity coefficient was 0.3711, indicating that among 50 accessions of proso millet there was wide genetic variation. The newly developed microsatellite markers should be useful tools for assessing genetic diversity, understanding population structure, and breeding of proso millet.
DOI:10.1111/j.1365-294X.2011.05318.xURLPMID:22004244 [本文引用: 4]

Broomcorn millet (Panicum miliaceumL.) is one of the world's oldest cultivated cereals, with several lines of recent evidence indicating that it was grown in northern China from at least 10 000 calbp. Additionally, a cluster of archaeobotanical records ofP. miliaceumdated to at least 7000 calbpexists in eastern Europe. These two centres of early records could either represent independent domestications or cross-continental movement of this cereal that would predate that of any other crop by some 2 millennia. Here, we analysed genetic diversity among 98 landrace accessions from across Eurasia using 16 microsatellite loci, to explore phylogeographic structure in the Old World range of this historically important crop. The major genetic split in the data divided the accessions into an eastern and a western grouping with an approximate boundary in northwestern China. A substantial number of accessions belonging to the estern genetic group were also found in northeastern China. Further resolution subdivided the western and eastern genepools into 2 and 4 clusters respectively, each showing clear geographic patterning. The genetic data are consistent with both the single and multiple domestication centre hypotheses and add specific detail to what these hypotheses would entail regarding the spread of broomcorn millet. Discrepancies exist between the predictions from the genetic data and the current archaeobotanical record, highlighting priorities for investigation into early farming in Central Asia.
DOI:10.3864/j.issn.0578-1752.2016.17.002 [本文引用: 6]

【目的】从分子水平研究国内外黍稷种质资源的遗传多样性差异,为黍稷种质资源的研究、保护和利用提供依据。【方法】用不同地理来源且性状差异显著的6份黍稷种质资源对来自高通量测序技术开发的黍稷基因组SSR引物进行筛选,从而获得条带清晰,稳定性好的63对SSR黍稷基因组引物,利用这63对SSR多态性引物对来自国内外的192份黍稷地方品种和野生种质进行遗传多样性分析。统计各试材在同一引物中的条带情况,并以此来分析试材的遗传多样性与所在群体间的亲缘关系。【结果】63对SSR引物共检测出161个等位变异位点,平均每个SSR位点2.56个;平均Shannon-Weaver指数(I)为0.6275,平均基因多样度(Nei)为0.3874,平均PIC值为0.4855。10个不同地理来源群体间表现出显著的遗传多样性差异,各群体的有效等位变异变化范围较窄,最小的是南方群体,为1.2407±0.4315;最大的是内蒙古高原群体,为1.8846±0.4892。国内群体Shannon-Weaver指数为内蒙古高原〉东北地区〉黄土高原〉西北地区〉南方地区,而国外Shannon-Weaver指数排序依次为前苏联〉欧洲〉蒙古〉印度〉美国。从Nei’s基因杂合度分析,观察杂合度(Ho)最小的是印度群体,为0.2372±0.2962,最大的是内蒙古高原群体,为0.3966±0.3250。期望杂合度(He)最小的是美国群体,为0.3114±0.2203;最大的是内蒙古高原群体,为0.4622±0.1862。从国外种、国内栽培种和国内野生种3个大群体来看,野生种质资源有效等位基因数(1.9285±0.5101)、Shannon-Weaver指数(0.6948±0.2852)、Nei基因多样性指数(0.4373±0.1773)远大于国外种和国内栽培种。而对国内外两大群体而言,国内资源的有效等位基因数(1.8145±0.4519)、Shannon-Weaver指数(0.6657±0.2413)和Nei基因多样性指数(0.412±0.1574)均大于国外资源(1.6862±0.4527、0.5897±0.2469、0.3652±0.1655)。UPGMA聚类分析结17
DOI:10.3864/j.issn.0578-1752.2016.17.002 [本文引用: 6]

【目的】从分子水平研究国内外黍稷种质资源的遗传多样性差异,为黍稷种质资源的研究、保护和利用提供依据。【方法】用不同地理来源且性状差异显著的6份黍稷种质资源对来自高通量测序技术开发的黍稷基因组SSR引物进行筛选,从而获得条带清晰,稳定性好的63对SSR黍稷基因组引物,利用这63对SSR多态性引物对来自国内外的192份黍稷地方品种和野生种质进行遗传多样性分析。统计各试材在同一引物中的条带情况,并以此来分析试材的遗传多样性与所在群体间的亲缘关系。【结果】63对SSR引物共检测出161个等位变异位点,平均每个SSR位点2.56个;平均Shannon-Weaver指数(I)为0.6275,平均基因多样度(Nei)为0.3874,平均PIC值为0.4855。10个不同地理来源群体间表现出显著的遗传多样性差异,各群体的有效等位变异变化范围较窄,最小的是南方群体,为1.2407±0.4315;最大的是内蒙古高原群体,为1.8846±0.4892。国内群体Shannon-Weaver指数为内蒙古高原〉东北地区〉黄土高原〉西北地区〉南方地区,而国外Shannon-Weaver指数排序依次为前苏联〉欧洲〉蒙古〉印度〉美国。从Nei’s基因杂合度分析,观察杂合度(Ho)最小的是印度群体,为0.2372±0.2962,最大的是内蒙古高原群体,为0.3966±0.3250。期望杂合度(He)最小的是美国群体,为0.3114±0.2203;最大的是内蒙古高原群体,为0.4622±0.1862。从国外种、国内栽培种和国内野生种3个大群体来看,野生种质资源有效等位基因数(1.9285±0.5101)、Shannon-Weaver指数(0.6948±0.2852)、Nei基因多样性指数(0.4373±0.1773)远大于国外种和国内栽培种。而对国内外两大群体而言,国内资源的有效等位基因数(1.8145±0.4519)、Shannon-Weaver指数(0.6657±0.2413)和Nei基因多样性指数(0.412±0.1574)均大于国外资源(1.6862±0.4527、0.5897±0.2469、0.3652±0.1655)。UPGMA聚类分析结17
DOI:10.3390/ijms17030370URLPMID:4813230 [本文引用: 3]

Broomcorn millet (Panicum miliaceumL.), one of the first domesticated crops, has been grown in Northern China for at least 10,000 years. The species is presently a minor crop, and evaluation of its genetic diversity has been very limited. In this study, we analyzed the genetic diversity of 88 accessions of broomcorn millet collected from various provinces of China. Amplification with 67 simple sequence repeat (SSR) primers revealed moderate levels of diversity in the investigated accessions. A total of 179 alleles were detected, with an average of 2.7 alleles per locus. Polymorphism information content and expected heterozygosity ranged from 0.043 to 0.729 (mean = 0.376) and 0.045 to 0.771 (mean = 0.445), respectively. Cluster analysis based on the unweighted pair group method of mathematical averages separated the 88 accessions into four groups at a genetic similarity level of 0.633. A genetic structure assay indicated a close correlation between geographical regions and genetic diversity. The uncovered information will be valuable for defining gene pools and developing breeding programs for broomcorn millet. Furthermore, the millet-specific SSR markers developed in this study should serve as useful tools for assessment of genetic diversity and elucidation of population structure in broomcorn millet.
URL [本文引用: 4]

【目的】利用SSR标记,分析黍稷种质资源(野生材料和地方品种)的遗传多样性水平,揭示不同来源黍稷种质资源的亲缘关系和遗传群体结构差异,为黍稷起源进化研究奠定基础。【方法】用6份地理差异显著的黍稷种质资源对137对小宗作物课题组开发的具有多态性的SSR引物进行初步筛选,最终筛选103对条带清晰、扩增良好且多态性稳定的SSR引物,利用这103对多态性SSR标记对146份黍稷材料进行PCR扩增,通过遗传参数、聚类、遗传结构等分析,评估不同个体间及不同群体间的遗传多样性,探讨遗传结构差异。【结果】103对SSR标记共检测出308个等位基因(Na),平均值为2.99,平均Shannon-Weaver指数(I)为0.8478,平均期望杂合度为0.3642,平均多态性信息含量指数(PIC)为0.5544。103对SSR标记的分布区间为0—1、1—2、2—3、3—4和4—5,分辨率范围为0.334—4.002,77.67%的标记分布于区间1—4,具有适度分辨力。国内资源的观测等位基因数(2.9126)、多样性指数(0.8302)、期望杂合度(0.5023)、多态性信息含量指数(0.5278)均高于国外资源,遗传多样性更丰富。12个群体的遗传距离的变化范围为0.0783—0.5762,均值为0.2938;遗传一致度变化范围为0.5620—0.9247,均值为0.75,遗传相似性与地理分布具有一定相关性,地理分布越近,遗传距离越小,遗传一致度越高。聚类分析在遗传距离为0.15处可以把12个群体分为4个组群,其中南美洲和山西资源各自独立分为一支,与其他资源亲缘关系较远。个体间聚类中,国内外资源划分非常显著,在遗传距离为0.63处,146份黍稷资源可分为3大组群,组群Ⅰ和组群Ⅱ为国外资源,组群Ⅲ为国内资源。组群Ⅱ在遗传距离为0.39处又分为3个亚群,组群Ⅲ在遗传距离为0.45处分为5个亚群,其中亚洲与欧洲资源、中国河北与中国山西、中国内蒙古资源的遗传关系较近。遗传结构分析结果显示国内外群体间存在明显的遗传分化,其中5个组群(组群2、组群5、组群6、组群7和组群9)为国内野生资源特有基因型,分布较为分散;2个组群(组群1和组群4)为国外资源特有基因型,分布较为集中。中国宁夏、南美洲资源的群体结构趋向单一化,中国河北、中国黑龙江、亚洲资源的群体结构趋向多元化。UPGMA聚类结果与遗传结构分析结果一致,且不同地区黍稷资源群体间遗传关系远近均与其地理分布相关。【结论】野生资源的遗传多样性高于国外资源,其中中国河北群体的遗传多样性最丰富,中国河北可能是黍稷的起源中心。
URL [本文引用: 4]

【目的】利用SSR标记,分析黍稷种质资源(野生材料和地方品种)的遗传多样性水平,揭示不同来源黍稷种质资源的亲缘关系和遗传群体结构差异,为黍稷起源进化研究奠定基础。【方法】用6份地理差异显著的黍稷种质资源对137对小宗作物课题组开发的具有多态性的SSR引物进行初步筛选,最终筛选103对条带清晰、扩增良好且多态性稳定的SSR引物,利用这103对多态性SSR标记对146份黍稷材料进行PCR扩增,通过遗传参数、聚类、遗传结构等分析,评估不同个体间及不同群体间的遗传多样性,探讨遗传结构差异。【结果】103对SSR标记共检测出308个等位基因(Na),平均值为2.99,平均Shannon-Weaver指数(I)为0.8478,平均期望杂合度为0.3642,平均多态性信息含量指数(PIC)为0.5544。103对SSR标记的分布区间为0—1、1—2、2—3、3—4和4—5,分辨率范围为0.334—4.002,77.67%的标记分布于区间1—4,具有适度分辨力。国内资源的观测等位基因数(2.9126)、多样性指数(0.8302)、期望杂合度(0.5023)、多态性信息含量指数(0.5278)均高于国外资源,遗传多样性更丰富。12个群体的遗传距离的变化范围为0.0783—0.5762,均值为0.2938;遗传一致度变化范围为0.5620—0.9247,均值为0.75,遗传相似性与地理分布具有一定相关性,地理分布越近,遗传距离越小,遗传一致度越高。聚类分析在遗传距离为0.15处可以把12个群体分为4个组群,其中南美洲和山西资源各自独立分为一支,与其他资源亲缘关系较远。个体间聚类中,国内外资源划分非常显著,在遗传距离为0.63处,146份黍稷资源可分为3大组群,组群Ⅰ和组群Ⅱ为国外资源,组群Ⅲ为国内资源。组群Ⅱ在遗传距离为0.39处又分为3个亚群,组群Ⅲ在遗传距离为0.45处分为5个亚群,其中亚洲与欧洲资源、中国河北与中国山西、中国内蒙古资源的遗传关系较近。遗传结构分析结果显示国内外群体间存在明显的遗传分化,其中5个组群(组群2、组群5、组群6、组群7和组群9)为国内野生资源特有基因型,分布较为分散;2个组群(组群1和组群4)为国外资源特有基因型,分布较为集中。中国宁夏、南美洲资源的群体结构趋向单一化,中国河北、中国黑龙江、亚洲资源的群体结构趋向多元化。UPGMA聚类结果与遗传结构分析结果一致,且不同地区黍稷资源群体间遗传关系远近均与其地理分布相关。【结论】野生资源的遗传多样性高于国外资源,其中中国河北群体的遗传多样性最丰富,中国河北可能是黍稷的起源中心。
URL [本文引用: 3]

采用SSR-PCR扩增技术,将实验室利用高通量测序与磁珠富集法结合开发得到的1210对黍稷SSR引物进行筛选,筛选出在会宁大黄糜、陇糜8号、太原55号和会宁野糜子4个黍稷品种之间表现出条带清晰、多态性明显、重复性较好的引物;并通过提取DNA以及PCR过程中退火温度的简单优化来建立比较适宜的黍稷SSR分子标记反应体系.结果表明,在DNA提取过程中将ddH2O的量减少到100μL,增加65℃的水浴时间并增加4%β-巯基乙醇和DNA浓度等可以获得较高质量的DNA.利用优化好的反应体系从1 210对SSR引物中筛选出1 16对多态性较好的引物,占总数的9.59%,其中,群体一的2个亲本所特有的引物有36对,群体二的2个亲本中特有的引物有97对,2个群体共有的多态性引物有19对;对引物碱基结构分析发现,选用的1 210对引物中有1 173个双碱基重复SSR和36个三碱基重复SSR;筛选到的多态性引物中双碱基重复SSR有102个.三碱基重复SSR有14个.筛选出的多态性SSR标记能用于黍稷的遗传多样性和群体遗传结构研究.
URL [本文引用: 3]

采用SSR-PCR扩增技术,将实验室利用高通量测序与磁珠富集法结合开发得到的1210对黍稷SSR引物进行筛选,筛选出在会宁大黄糜、陇糜8号、太原55号和会宁野糜子4个黍稷品种之间表现出条带清晰、多态性明显、重复性较好的引物;并通过提取DNA以及PCR过程中退火温度的简单优化来建立比较适宜的黍稷SSR分子标记反应体系.结果表明,在DNA提取过程中将ddH2O的量减少到100μL,增加65℃的水浴时间并增加4%β-巯基乙醇和DNA浓度等可以获得较高质量的DNA.利用优化好的反应体系从1 210对SSR引物中筛选出1 16对多态性较好的引物,占总数的9.59%,其中,群体一的2个亲本所特有的引物有36对,群体二的2个亲本中特有的引物有97对,2个群体共有的多态性引物有19对;对引物碱基结构分析发现,选用的1 210对引物中有1 173个双碱基重复SSR和36个三碱基重复SSR;筛选到的多态性引物中双碱基重复SSR有102个.三碱基重复SSR有14个.筛选出的多态性SSR标记能用于黍稷的遗传多样性和群体遗传结构研究.
DOI:10.1016/j.ygeno.2005.11.008URLPMID:16406726 [本文引用: 1]

We report the development of a heteroduplex-based mutation detection method using multicapillary automated sequencers, known as conformation-sensitive capillary electrophoresis (CSCE). Our optimized CSCE protocol detected 93 of 95 known base substitution sequence variants. Since the optimization of the method, we have analyzed 215 Mb of DNA and identified 3397 unique variants. An analysis of this data set indicates that the sensitivity of CSCE is above 95% in the central 56% of the average PCR product. To fully exploit the mutation detection capacity of this method, we have developed software, canplot, which automatically compares normal and test results to prioritize samples that are most likely to contain variants. Using multiple fluorescent dyes, CSCE has the capacity to screen over 2.2 Mb on one ABI3730 each day. Therefore this technique is suitable for projects where a rapid and sensitive DNA mutation detection system is required.
URL [本文引用: 1]

采取混合取样策略,利用荧光SSR技术对来源于我国7个省(自治区)的90份糯玉米地方品种和6个标准测验种自交系基因组的31个简单序列重复(SSR)位点进行了分析.在90个糯玉米地方品种中,共检测出216个等位变异,每个位点检测出2~12个,平均6.97个,多态性信息量(PIC,polymorphic information content)为0.67,标记指数(MI,marker index)为4.92.可初步将90份糯玉米地方品种划为3个类群,其中,广西和贵州的糯玉米地方品种的遗传多样性相对更丰富.
URL [本文引用: 1]

采取混合取样策略,利用荧光SSR技术对来源于我国7个省(自治区)的90份糯玉米地方品种和6个标准测验种自交系基因组的31个简单序列重复(SSR)位点进行了分析.在90个糯玉米地方品种中,共检测出216个等位变异,每个位点检测出2~12个,平均6.97个,多态性信息量(PIC,polymorphic information content)为0.67,标记指数(MI,marker index)为4.92.可初步将90份糯玉米地方品种划为3个类群,其中,广西和贵州的糯玉米地方品种的遗传多样性相对更丰富.
DOI:10.3864/j.issn.0578-1752.2014.19.001URLMagsci [本文引用: 1]

【目的】建立基于SSR荧光标记的小麦品种DNA指纹鉴定体系,为中国小麦育成品种鉴定提供高通量技术手段。【方法】收集已定位到小麦21条染色体上的SSR标记引物,通过PCR扩增和变性聚丙烯酰胺凝胶电泳检测技术,筛选在中国小麦育成品种中多态性高的标记,对筛选出的引物5′末端利用6-FAM、HEX、ROX和TAMRA 4种荧光染料之一进行标记,利用DNA分析仪对扩增产物的峰型进行评价并检测不同等位变异的扩增片段大小,选择峰型简单易读、多态性高、较均匀分布到21条染色体上的标记,确定不同位点等位变异的大小及相应的参照品种,建立基于荧光SSR标记的高通量小麦品种鉴定体系。【结果】利用2 438对SSR标记对8份植物学性状差异大的小麦育成品种进行初步筛选,共筛选出260对多态性较高、扩增稳定的SSR引物。利用上述260对引物对48份小麦品种继续进行复筛,选出130对扩增稳定、多态性高、带型好的SSR引物。对这些引物的正向5′末端分别标记6-FAM、HEX、ROX和TAMRA荧光后,利用DNA分析仪进行检测评价,最终选择了42个PCR扩增稳定、峰图简单、多态性高、连锁群分布均匀的SSR荧光标记。根据DNA分析仪检测到的每个标记的不同等位变异大小,为相应位点的不同等位变异进行了命名,并为每个等位变异选取了相应参照品种。根据每个引物标记的荧光和扩增的片段大小范围对42对引物进行合理搭配,在同一毛细管内对多个荧光标记进行检测,提高了DNA指纹数据采集效率,降低了检测成本。利用该体系对1 625份小麦育成品种进行DNA指纹数据采集,在42个位点中共检测到434个等位变异,每个位点的等位变异个数在3—23,平均10.3个;每个位点的PIC值范围为0.240—0.829,平均0.610。利用1 625份小麦育成品种DNA指纹数据,构建了中国小麦育成品种的DNA指纹数据库。【结论】筛选出42对SSR标记引物,建立了基于荧光SSR标记的小麦品种鉴定体系,可用于高通量小麦品种DNA指纹鉴定。
DOI:10.3864/j.issn.0578-1752.2014.19.001URLMagsci [本文引用: 1]

【目的】建立基于SSR荧光标记的小麦品种DNA指纹鉴定体系,为中国小麦育成品种鉴定提供高通量技术手段。【方法】收集已定位到小麦21条染色体上的SSR标记引物,通过PCR扩增和变性聚丙烯酰胺凝胶电泳检测技术,筛选在中国小麦育成品种中多态性高的标记,对筛选出的引物5′末端利用6-FAM、HEX、ROX和TAMRA 4种荧光染料之一进行标记,利用DNA分析仪对扩增产物的峰型进行评价并检测不同等位变异的扩增片段大小,选择峰型简单易读、多态性高、较均匀分布到21条染色体上的标记,确定不同位点等位变异的大小及相应的参照品种,建立基于荧光SSR标记的高通量小麦品种鉴定体系。【结果】利用2 438对SSR标记对8份植物学性状差异大的小麦育成品种进行初步筛选,共筛选出260对多态性较高、扩增稳定的SSR引物。利用上述260对引物对48份小麦品种继续进行复筛,选出130对扩增稳定、多态性高、带型好的SSR引物。对这些引物的正向5′末端分别标记6-FAM、HEX、ROX和TAMRA荧光后,利用DNA分析仪进行检测评价,最终选择了42个PCR扩增稳定、峰图简单、多态性高、连锁群分布均匀的SSR荧光标记。根据DNA分析仪检测到的每个标记的不同等位变异大小,为相应位点的不同等位变异进行了命名,并为每个等位变异选取了相应参照品种。根据每个引物标记的荧光和扩增的片段大小范围对42对引物进行合理搭配,在同一毛细管内对多个荧光标记进行检测,提高了DNA指纹数据采集效率,降低了检测成本。利用该体系对1 625份小麦育成品种进行DNA指纹数据采集,在42个位点中共检测到434个等位变异,每个位点的等位变异个数在3—23,平均10.3个;每个位点的PIC值范围为0.240—0.829,平均0.610。利用1 625份小麦育成品种DNA指纹数据,构建了中国小麦育成品种的DNA指纹数据库。【结论】筛选出42对SSR标记引物,建立了基于荧光SSR标记的小麦品种鉴定体系,可用于高通量小麦品种DNA指纹鉴定。
DOI:10.3969/j.issn.1000-6850.2012.10.018URL [本文引用: 1]

According to the situation of narrow genetic background in Chinese tobacco cultivars, the genetic diversity analysis of tobacco germplasm could provide important information for breeding and introducing cultivars, and germplasm resource protection. In this study, genetic diversity analysis for 50 tobacco cultivars collected from different countries/regions were performed by using fluorescence SSR technique with 20 SSR primer combinations, a total of 81 allelic variations among the collected tobacco germplasm were detected with an average of 4.1 alleles per locus of each SSR. The result showed that the SSR combination of PT20457 possessed the highest values of polymorphism information content (PIC) (0.944) and could be effective locus for identifying DNA variation among these tobacco cultivars. Based on the polymorphic data, fifty tobacco cultivars were classified into 3 groups by UPGMA method. Group I contains Yunluo-01 which was sun-cured tobacco and derived from Guangdong, group II contained Atnarello Maoliang and Brazil Shailiang which were burley, group III contains 47 tobacco cultivars which were derived from 4 different countries. By using principle coordinates analysis, these tobacco varieties were clustered into 4 groups and 8 subgroups. Both cluster analysis methods markers revealed the genetic diversity and genetic relationships among tobacco varieties, and could provide a scientific basis for genetic research and tobacco breeding.
DOI:10.3969/j.issn.1000-6850.2012.10.018URL [本文引用: 1]

According to the situation of narrow genetic background in Chinese tobacco cultivars, the genetic diversity analysis of tobacco germplasm could provide important information for breeding and introducing cultivars, and germplasm resource protection. In this study, genetic diversity analysis for 50 tobacco cultivars collected from different countries/regions were performed by using fluorescence SSR technique with 20 SSR primer combinations, a total of 81 allelic variations among the collected tobacco germplasm were detected with an average of 4.1 alleles per locus of each SSR. The result showed that the SSR combination of PT20457 possessed the highest values of polymorphism information content (PIC) (0.944) and could be effective locus for identifying DNA variation among these tobacco cultivars. Based on the polymorphic data, fifty tobacco cultivars were classified into 3 groups by UPGMA method. Group I contains Yunluo-01 which was sun-cured tobacco and derived from Guangdong, group II contained Atnarello Maoliang and Brazil Shailiang which were burley, group III contains 47 tobacco cultivars which were derived from 4 different countries. By using principle coordinates analysis, these tobacco varieties were clustered into 4 groups and 8 subgroups. Both cluster analysis methods markers revealed the genetic diversity and genetic relationships among tobacco varieties, and could provide a scientific basis for genetic research and tobacco breeding.
DOI:10.7505/j.issn.1007-9084.2014.02.003URLMagsci [本文引用: 1]

<p>为了提高油菜品种指纹检测的精确性及未来构建大规模油菜新品种指纹数据库的需求,应用SSR荧光标记毛细管电泳检测法构建国家冬油菜区试指纹鉴定平台。以40对荧光引物对2012-2013年度163份国家冬油菜区试参试品种(系)进行分析。结果共检测到42个等位位点和131个等位变异。其中A基因组(n1~n10)检测位点25个,C基因组(n11~n19)检测位点17个。每个位点等位变异数从2到6不等,平均为3.02。42个检测位点的PIC值变化范围在0.10~0.69之间,平均值为0.36。其中引物BRGMS171的杂合度、PIC值分别高达0.67、0.70,可考虑作为以后区试杂交种纯度鉴定的核心标记。SSR位点的纯合度分析得出本年度18份常规种平均纯合度为81.9%,145份杂交种为57.9%。以131个等位变异计算品种(系)间DICE相似系数,163份品种(系)间平均遗传相似系数变幅为0.607~0.765,变幅最大的为品种(系)FC03(0.438~0.879),变幅最小为品种(系)宜油21(0.611~0.806)。</p>
DOI:10.7505/j.issn.1007-9084.2014.02.003URLMagsci [本文引用: 1]

<p>为了提高油菜品种指纹检测的精确性及未来构建大规模油菜新品种指纹数据库的需求,应用SSR荧光标记毛细管电泳检测法构建国家冬油菜区试指纹鉴定平台。以40对荧光引物对2012-2013年度163份国家冬油菜区试参试品种(系)进行分析。结果共检测到42个等位位点和131个等位变异。其中A基因组(n1~n10)检测位点25个,C基因组(n11~n19)检测位点17个。每个位点等位变异数从2到6不等,平均为3.02。42个检测位点的PIC值变化范围在0.10~0.69之间,平均值为0.36。其中引物BRGMS171的杂合度、PIC值分别高达0.67、0.70,可考虑作为以后区试杂交种纯度鉴定的核心标记。SSR位点的纯合度分析得出本年度18份常规种平均纯合度为81.9%,145份杂交种为57.9%。以131个等位变异计算品种(系)间DICE相似系数,163份品种(系)间平均遗传相似系数变幅为0.607~0.765,变幅最大的为品种(系)FC03(0.438~0.879),变幅最小为品种(系)宜油21(0.611~0.806)。</p>
DOI:10.3724/SP.J.1006.2017.00530URL [本文引用: 2]

分析糜子种质资源的遗传多样性,有助于了解糜子起源与进化,可为糜子优异种质发掘及资源高效利用提供理论基础。本研究利用15个糜子特异性荧光SSR标记检测来源于中国11个省(区)的132份糜子种质资源,检测到107个等位变异,每个位点等位变异数为2~14个,平均7个;基因多样性指数为0.0936~0.8676,平均0.5298;多态性信息含量为0.0893~0.8538,平均0.4864。采用遗传距离的聚类将试验材料分为4类,类群I来自东北春糜子区,类群II来自黄土高原春、夏糜子区,类群III来自于北方春糜子区,类群IV来自北方春糜子区和黄土高原春、夏糜子区。分析模型的遗传结构表明,中国糜子资源来自4个(东北地区、黄土高原、北方地区和西北地区)基因库,与基于遗传距离的聚类结果基本一致,均与材料的地理起源相关。糜子遗传变异丰富,主要存在于糜子材料间。该结果从分子水平上准确揭示了中国糜子的遗传多样性。
DOI:10.3724/SP.J.1006.2017.00530URL [本文引用: 2]

分析糜子种质资源的遗传多样性,有助于了解糜子起源与进化,可为糜子优异种质发掘及资源高效利用提供理论基础。本研究利用15个糜子特异性荧光SSR标记检测来源于中国11个省(区)的132份糜子种质资源,检测到107个等位变异,每个位点等位变异数为2~14个,平均7个;基因多样性指数为0.0936~0.8676,平均0.5298;多态性信息含量为0.0893~0.8538,平均0.4864。采用遗传距离的聚类将试验材料分为4类,类群I来自东北春糜子区,类群II来自黄土高原春、夏糜子区,类群III来自于北方春糜子区,类群IV来自北方春糜子区和黄土高原春、夏糜子区。分析模型的遗传结构表明,中国糜子资源来自4个(东北地区、黄土高原、北方地区和西北地区)基因库,与基于遗传距离的聚类结果基本一致,均与材料的地理起源相关。糜子遗传变异丰富,主要存在于糜子材料间。该结果从分子水平上准确揭示了中国糜子的遗传多样性。
[本文引用: 1]
[本文引用: 1]
DOI:10.1093/bioinformatics/bti282URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.3410/f.1015548.197423URLPMID:12930761 [本文引用: 1]

Abstract We describe extensions to the method of Pritchard et al. for inferring population structure from multilocus genotype data. Most importantly, we develop methods that allow for linkage between loci. The new model accounts for the correlations between linked loci that arise in admixed populations ("admixture linkage disequilibium"). This modification has several advantages, allowing (1) detection of admixture events farther back into the past, (2) inference of the population of origin of chromosomal regions, and (3) more accurate estimates of statistical uncertainty when linked loci are used. It is also of potential use for admixture mapping. In addition, we describe a new prior model for the allele frequencies within each population, which allows identification of subtle population subdivisions that were not detectable using the existing method. We present results applying the new methods to study admixture in African-Americans, recombination in Helicobacter pylori, and drift in populations of Drosophila melanogaster. The methods are implemented in a program, structure, version 2.0, which is available at http://pritch.bsd.uchicago.edu.
DOI:10.1111/mec.2005.14.issue-8URL [本文引用: 1]
DOI:10.3864/j.issn.0578-1752.2017.20.002URL [本文引用: 2]

【目的】开发高基元(4—6)碱基重复微卫星标记,分析种质资源遗传多样性,为糜子遗传和进化研究提供理论基础。【方法】用隶属函数、主成分分析和聚类分析综合评价糜子资源表型多样性,用前期糜子转录组测序获得高基元SSR引物对地理来源差异大的糜子材料进行PCR扩增检测其多态性,用Power Marker 3.25计算遗传多样性参数,用Pop Gen 1.32计算Nei’s遗传距离,用MEGA 5.0进行聚类分析,用Structure 2.2鉴定遗传类群。【结果】96份糜子资源株高和穗长变异最丰富,多样性指数分别为2.08和1.91。PCR扩增发现,占56.29%的85对引物具多态性,其中四、五和六碱基重复引物分别为71对(83.53%)、10对(11.76%)和4对(4.7%)。85个标记扩增产物大小分布为100—450 bp,PIC值平均为0.51,Rp值为1.00—5.75,平均为3.15。四、五和六碱基重复SSR的平均Rp值分别为3.15、2.8和4.0。基于Rp值分析SSR的分布频次,发现85个标记分布区间为0—1、1—2、2—3、3—4、4—5和5—6,分别包含1(1.18%)、15(17.65%)、31(36.47%)、20(23.53%)、12(14.12%)和6(7.06%)个标记,60%(51个)的标记分布在区间2—3和3—4。用85个SSR扩增96份糜子资源,共检测到232个等位变异,每个位点检测到等位变异2—3个,平均2.7294个;62个位点产生3个变异,23个位点产生2个变异;多样性指数为0.2842—1.0633,平均为0.7708;PIC值为0.0400—0.7281,平均为0.4723。不同生态区糜子种质间的遗传距离为0.0093—0.5052(平均为0.1798),遗传一致度为0.6034—0.9907(平均为0.8485)。基于UPGMA将96个糜子基因型聚为4个群组,第一群组主要属于北方春糜子区;第二群主要属于东北春糜子区;第三群组主要属于华北夏糜子区;第四群组主要属于黄土高原春夏糜子区。遗传结构分析将96份试材划分为4个类群,分别代表黄土高原、华北、东北和北方基因库。UPGMA聚类分析和遗传结构分析结果基本一致,均与地理起源相关。【结论】在糜子中构建了85个四、五和六碱基重复微卫星标记,这些高基元SSR的引物分辨率(Rp)高,对不同基因型分辨能力强,PCR扩增多态性好;用其评估中国糜子资源的遗传差异发现,黄土高原春夏糜子区和北方春糜子区资源遗传多样性最丰富。
DOI:10.3864/j.issn.0578-1752.2017.20.002URL [本文引用: 2]

【目的】开发高基元(4—6)碱基重复微卫星标记,分析种质资源遗传多样性,为糜子遗传和进化研究提供理论基础。【方法】用隶属函数、主成分分析和聚类分析综合评价糜子资源表型多样性,用前期糜子转录组测序获得高基元SSR引物对地理来源差异大的糜子材料进行PCR扩增检测其多态性,用Power Marker 3.25计算遗传多样性参数,用Pop Gen 1.32计算Nei’s遗传距离,用MEGA 5.0进行聚类分析,用Structure 2.2鉴定遗传类群。【结果】96份糜子资源株高和穗长变异最丰富,多样性指数分别为2.08和1.91。PCR扩增发现,占56.29%的85对引物具多态性,其中四、五和六碱基重复引物分别为71对(83.53%)、10对(11.76%)和4对(4.7%)。85个标记扩增产物大小分布为100—450 bp,PIC值平均为0.51,Rp值为1.00—5.75,平均为3.15。四、五和六碱基重复SSR的平均Rp值分别为3.15、2.8和4.0。基于Rp值分析SSR的分布频次,发现85个标记分布区间为0—1、1—2、2—3、3—4、4—5和5—6,分别包含1(1.18%)、15(17.65%)、31(36.47%)、20(23.53%)、12(14.12%)和6(7.06%)个标记,60%(51个)的标记分布在区间2—3和3—4。用85个SSR扩增96份糜子资源,共检测到232个等位变异,每个位点检测到等位变异2—3个,平均2.7294个;62个位点产生3个变异,23个位点产生2个变异;多样性指数为0.2842—1.0633,平均为0.7708;PIC值为0.0400—0.7281,平均为0.4723。不同生态区糜子种质间的遗传距离为0.0093—0.5052(平均为0.1798),遗传一致度为0.6034—0.9907(平均为0.8485)。基于UPGMA将96个糜子基因型聚为4个群组,第一群组主要属于北方春糜子区;第二群主要属于东北春糜子区;第三群组主要属于华北夏糜子区;第四群组主要属于黄土高原春夏糜子区。遗传结构分析将96份试材划分为4个类群,分别代表黄土高原、华北、东北和北方基因库。UPGMA聚类分析和遗传结构分析结果基本一致,均与地理起源相关。【结论】在糜子中构建了85个四、五和六碱基重复微卫星标记,这些高基元SSR的引物分辨率(Rp)高,对不同基因型分辨能力强,PCR扩增多态性好;用其评估中国糜子资源的遗传差异发现,黄土高原春夏糜子区和北方春糜子区资源遗传多样性最丰富。
DOI:10.3864/j.issn.0578-1752.2015.16.003URL [本文引用: 1]

【目的】糜子生育期短、耐干旱、耐瘠薄、水分利用效率高,了解糜子资源的遗传多样性和遗传结构,为今后糜子杂交育种、种质创新、挖掘抗旱基因及资源的高效利用提供理论依据。【方法】采用表型鉴定和SSR分子标记对糜子资源进行遗传多样性检测。利用模糊隶属函数法分析糜子种质的株高、主穗长、叶片长、叶片宽、主茎节数、主茎粗、单株穗重、单株粒重和千粒重9个表型性状的分布情况。利用DPS7.05软件进行表型性状的遗传多样性分析、相关性分析和主成分分析,综合评价糜子种质资源的优劣。利用CTAB法提取糜子嫩叶基因组DNA,并利用SSR分子标记技术对不同地区的96份糜子种质资源的基因组DNA进行PCR扩增,后经8%聚丙烯酰胺凝胶电泳分离,银染后显色。利用Power Maker 3.25软件计算每对引物的等位基因数(A)、主要等位基因频率(M)、基因多样性指数(He)和多态性信息含量指数(PIC),并进行N-J遗传距离的统计分析;利用Structure 2.3.1分析群体遗传结构。【结果】糜子表型遗传多样性分析表明:9个表型性状分布集中,且绝大部分呈极显著相关;单株粒重和单株穗重遗传变异最丰富,不同省份的资源在表型性状上表现出不同的遗传多样性,山西资源表型遗传多样性最丰富。采用主成分分析法和综合评价法表明,内糜1号的综合性状表现最差,宁糜15号的综合性状表现最好。采用19对SSR引物对96份糜子种质资源进行遗传多样性分析,共检测出112个等位变异;每个位点的等位变异数为3—9个,平均5.9个;平均主要等位基因频率为0.7045;平均基因多样性指数为0.4097;平均多态性信息含量位点百分数为39.2%。不同地理来源糜子种质资源的遗传多样性分析表明,各省份间糜子资源的亲缘关系均较近;山西省资源的基因多样性指数及多态性信息含量百分数最高,分别为0.35717
DOI:10.3864/j.issn.0578-1752.2015.16.003URL [本文引用: 1]

【目的】糜子生育期短、耐干旱、耐瘠薄、水分利用效率高,了解糜子资源的遗传多样性和遗传结构,为今后糜子杂交育种、种质创新、挖掘抗旱基因及资源的高效利用提供理论依据。【方法】采用表型鉴定和SSR分子标记对糜子资源进行遗传多样性检测。利用模糊隶属函数法分析糜子种质的株高、主穗长、叶片长、叶片宽、主茎节数、主茎粗、单株穗重、单株粒重和千粒重9个表型性状的分布情况。利用DPS7.05软件进行表型性状的遗传多样性分析、相关性分析和主成分分析,综合评价糜子种质资源的优劣。利用CTAB法提取糜子嫩叶基因组DNA,并利用SSR分子标记技术对不同地区的96份糜子种质资源的基因组DNA进行PCR扩增,后经8%聚丙烯酰胺凝胶电泳分离,银染后显色。利用Power Maker 3.25软件计算每对引物的等位基因数(A)、主要等位基因频率(M)、基因多样性指数(He)和多态性信息含量指数(PIC),并进行N-J遗传距离的统计分析;利用Structure 2.3.1分析群体遗传结构。【结果】糜子表型遗传多样性分析表明:9个表型性状分布集中,且绝大部分呈极显著相关;单株粒重和单株穗重遗传变异最丰富,不同省份的资源在表型性状上表现出不同的遗传多样性,山西资源表型遗传多样性最丰富。采用主成分分析法和综合评价法表明,内糜1号的综合性状表现最差,宁糜15号的综合性状表现最好。采用19对SSR引物对96份糜子种质资源进行遗传多样性分析,共检测出112个等位变异;每个位点的等位变异数为3—9个,平均5.9个;平均主要等位基因频率为0.7045;平均基因多样性指数为0.4097;平均多态性信息含量位点百分数为39.2%。不同地理来源糜子种质资源的遗传多样性分析表明,各省份间糜子资源的亲缘关系均较近;山西省资源的基因多样性指数及多态性信息含量百分数最高,分别为0.35717
