
华中农业大学植物科学技术学院 / 作物遗传改良国家重点实验室 / 国家油菜工程技术研究中心, 湖北武汉 430070;
Homologous Cloning of BnGS3 and BnGhd7 Genes in Brassica napus and Their Relationship with Rapeseed Yield-related Traits
XUEZhi-Fei, WANGXia, LIFu-Peng, MAChao-Zhi
通讯作者:
收稿日期:2017-04-19
接受日期:2017-11-21
网络出版日期:2018-02-12
版权声明:2018作物学报编辑部作物学报编辑部
基金资助:
作者简介:
-->
展开
摘要
关键词:
Abstract
Keywords:
-->0
PDF (1668KB)元数据多维度评价相关文章收藏文章
本文引用格式导出EndNoteRisBibtex收藏本文-->
油菜是世界上最主要的油料作物之一, 提高产量始终是油菜育种的一个重要目标。研究产量相关性状、发展分子标记进行辅助选择将是提高产量的有效途径。现已完成甘蓝型冬油菜品种Tapidor全基因组测序, 预测有101 040个基因, 在测序植物中基因数目最多[1]。甘蓝型油菜(Brassica napus, AACC=38)是由白菜(Brassica rapa, AA=20)和甘蓝(Brassica oleracea, CC=18)自然杂交、加倍而成的异源四倍体[2], A/C基因组在进化过程中发生了大量的重组和突变, 增加了基因组结构的复杂性, 导致甘蓝型油菜功能基因组研究难度加大。油菜与水稻(Oryza sativa)基因组虽然差异大, 但仍然具有共线性[3]。作为高等模式生物, 水稻的大量功能基因已被克隆, 利用水稻基因序列克隆油菜同源基因, 很可能是研究油菜基因功能的有效手段。
无论水稻还是油菜, 生育期、千粒重和籽粒大小都是重要的产量相关性状, 控制这些性状的很多基因或QTL已在水稻中被克隆。其中, GS3是控制粒长与千粒重的主效QTL, 同时也是粒宽和粒厚相关的微效QTL[4]; Ghd7同时影响株高、穗粒数和抽穗期等多个性状, 是一个控制水稻产量的多效基因[5]。GS3全长5363 bp, 其编码蛋白包含4个保守结构域, 分别是磷脂酞乙醇胺结合蛋白区域(phosphatidylethanolamine-binding protein, PEBP-like)、跨膜区域、生长因子受体家族富半胱氨酸同源区域(tumour necrosis factor receptor, TNFR或nerve growth factor receptor, NGFR)和von Willebrand因子C型结构域(von Willebrand factor type C, VWFC)[6]。Ghd7编码257个氨基酸残基, 其蛋白C端具有一个明显的CCT (constans, constans-like, TOCl)结构域, 对控制植物花期的重要基因CO发挥核心功能, Ghd7缺少位于CO基因N端的另一个重要的B-BOX结构域[7]。甘蓝型油菜是否有GS3和Ghd7的同源基因, 其同源基因是否与生育期和千粒重等产量性状有关, 目前还未见相关的研究报道。
本研究根据水稻GS3和Ghd7基因序列, 通过比较基因组学方法, 克隆甘蓝型油菜的同源基因, 并对候选基因进行比较测序, 分析其与产量性状的相关性, 目的在于探究利用模式植物水稻功能基因信息研究油菜相关基因的可行性。
1 材料与方法
1.1 植物材料
选用58份甘蓝型油菜自交系和由Tapidor × Ningyou 7创建而来具有202个DH系(double haploid)的TN群体[8]。58份材料中, 自交不亲和系SI-1300用于同源基因克隆, 57份自交亲和系用于表型性状的相关分析, TN群体用于连锁图谱分析和基因定位。1.2 表型调查与分析
2009年10月上旬, 将57份自交亲和系分别在湖北武汉和黄冈两地种植, 均采用随机区组设计, 3行区, 行距0.27 m, 行长3.50 m, 小区面积2.84 m2, 定苗后每行20株、每小区60株。记录初花期(小区内50%植株开第1朵花)和终花期日期, 分别计算播种-初花期、初花期-终花期的天数。成熟后随机连续取10株, 考察株高、一次有效分枝数、单株角果总数、角果粒数、单株产量、千粒重和小区产量。利用SAS 9.4软件进行方差分析(ANOVA)以及表型与基因型的简单相关分析(simple correlation analysis)。
1.3 引物设计及同源基因克隆
使用天根公司植物基因组DNA提取试剂盒(OSR- M301)提取油菜苗期幼嫩叶片总DNA。使用软件Oligo v. 7设计同源克隆引物。根据水稻GS3的DNA序列(GenBank登录号为AB743898.1)设计引物bngs-1-1、bngs-1-2和bngs-1-3 (见附表1), 但是该3对引物在甘蓝型油菜中没有特异扩增, 因此利用GS3蛋白序列重新进行同源搜索, 获得同源的拟南芥NP_680175.2蛋白序列, 利用其对应的基因序列信息在NCBI中再次进行同源比对, 获得白菜型油菜的同源序列AC189411.2。根据拟南芥NP_680175.2基因序列设计引物gs3-1和gs3-4, 根据白菜型油菜AC189411.2的基因序列设计了3对引物, 分别是gs3-2、gs3-3和gs3-5 (见附表1)。
Supplementary table 1
附表1
附表1同源克隆BnGS3和BnGhd7的引物
Supplementary table 1Primers used in homologous clonging of BnGS3 and BnGhd7
| 引物 Primer | 正向序列 Forward sequence (5'-3') | 反向序列 Reverse sequence (5'-3') | 退火温度 Tm (°C) | |
|---|---|---|---|---|
| bngs-1-1 | TACTCATCCTCCTCCTCC | CGCTCATCATCGAGAACT | 55 | |
| bngs-1-2 | GCGTAATGCCTCAGCTAC | GACCCATCATCATCGAGA | 55 | |
| bngs-1-3 | GCATGATGCTTTCACCAC | CGATCATCATCGAGAACT | 55 | |
| gs3-1 | TGTTAGGGTTTCTGTTGGTGG | CCAGATGCTGCAAAGAGTAAG | 59 | |
| gs3-2 | GGGTTTCTGTTGGTGGGCTT | CCCATGAGGTATGTCAGCAT | 59 | |
| gs3-3 | TTCTAGACAATGAATGGCAGG | ACGAAAGAGTCGACGACACTG | 58 | |
| gs3-4 | TTCTAGACAATGAATGGCAG | TGCAACTGCAAGATCAAATG | 59 | |
| gs3-5 | CCGCAGCGGAAAAGGTATGT | GTCCAGATGCTGCAAAGAGT | 59 | |
| ghd7-2-4 | CCAAAAGCCAACGTCACCAT | GCGATCAGCGACCATTAAAG | 56 | |
| ghd7-2-5 | CCAAAAGCCAACGTCACCAT | GGAGCGACCGAAAACTACAT | 54 | |
| ghd7-2-6 | TCCCTCACCAACAACAAACC | ATCCTTGGTCTTTTCTCTGC | 55 | |
| ghd7-1 | CAAAAGCCAACGTCACCATC | CGACCATTAAAGAACAGGCT | 58 | |
| ghd7-2 | TTCGGATTCGGTTCTGGTTC | GCTCACATGATTGACAGACT | 59 | |
| ghd7-3 | AGCAGAGGCGGCTTCTTGGT | GCGATCAGCGACCATTAAAG | 59 | |
| ghd7-4 | CCAAAAGCCAACGTCACCAT | GCGATCAGCGACCATTAAAG | 58 | |
| ghd7-5 | CCAAAAGCCAACGTCACCAT | GGAGCGACCGAAAACTACAT | 59 | |
| ghd7-6 | TCCCTCACCAACAACAAACC | ATCCTTGGTCTTTTCTCTGC | 59 | |
| ghd7-7 | GGGTTGTTCCACTTCAGGTT | ACCCATGGAAAGTGGTAGAT | 59 | |
| ghd7-8 | CAAAAGCCAACGTCACCATC | CATCCGATATTTTTGTCTCC | 60 | |
| ghd7-9 | GATCCATCACGGGCCATAAC | GGGTTTGAAACTGTTGTCTC | 59 | |
新窗口打开
水稻Ghd7具有保守的CCT结构域, CCT结构域是CO基因家族编码蛋白的核心功能域。同源比对发现Ghd7与甘蓝型油菜AY280868.2序列和拟南芥CO基因具有相似的同源性。根据水稻Ghd7 (GenBank登录号为JF926542.1)、甘蓝型油菜AY280868.2的序列信息设计12对引物(见附表1)。
PCR体系使用1×扩增缓冲液配制, 加入2 mmol L-1的MgCl2、0.1 mmol L-1的dNTP、左右引物各0.2 μmol L-1、0.5 U Taq酶、50 ng模板DNA, ddH2O定容至20 μL。PCR反应程序为94°C运行2 min, 94°C运行1 min, 55°C运行30 s, 72°C运行45 s, 35个循环; 72°C运行5 min; 于4°C保存产物。
1.4 候选基因及其编码产物的序列分析
用SSCP (single-strand conformation polymorphism)技术可以有效检测InDel和SNP所造成的序列差异, 但PCR产物需要经高温变性后才能检测到单链折叠的构象差异, 已经被用来开发甘蓝型油菜分子标记[9]。考虑到SSCP对检测片段大小有严格要求, 在8%聚丙烯酰胺凝胶的基础上, 又配制了3.5%和6%非变性聚丙烯酰胺凝胶, 分别检测>800 bp和400~500 bp的片段。使用天根公司纯化回收试剂盒(DP214)回收PCR产物, 使用pMD18-T Vector (TaKaRa)进行T-A克隆, 挑选转化的大肠杆菌感受态(K12菌株制备)阳性单克隆送北京擎科新业生物技术公司测序, 使用软件SeqMan Pro (http://www.dnastar.com/)分析测序结果。通过数据库NCBI (https://www.ncbi.nlm.nih.gov/)分别对GS3、Ghd7的序列进行同源比对。用ClustalW完成多序列对位排列分析[10]; 利用在线软件Softberry (http:// www.softberry.com/) [11]和Genscan (http://genes.mit.edu/ GENSCAN.html)预测基因结构, 用在线软件InterProScan (http://www.ebi.ac.uk/interpro/search/sequence-search)[12,13]和Conserved Domain Database (https://www.ncbi.nlm. nih.gov/cdd/)预测同源蛋白结构域。
1.5 基因型分析
参照MapMaker/Exp 3.0[14,15]的方法记载分子标记的基因型。2 结果与分析
2.1 GS3在甘蓝型油菜中的同源克隆
2.1.1 GS3在甘蓝型油菜中的同源克隆 根据水稻GS3基因序列设计的引物bngs-1-1、bngs-1-2和bngs-1-3, 在甘蓝型油菜SI-1300中为非特异扩增(图1-A), 说明直接利用水稻序列设计引物不能在油菜上得到有效扩增。而根据白菜型油菜AC189411.2序列设计的3对引物则可检测到特异扩增产物(图1-B)。扩增片段的测序结果与白菜型油菜AC189411.2序列一致, 拼接产生3040 bp的DNA片段。预测基因BnGS3位于拼接片段的1271~2965 bp处, 有6个外显子, 开放阅读框(ORF)全长666 bp, 编码222个氨基酸(图2)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1根据同源序列设计引物的扩增结果
A: 由水稻GS3序列设计的引物在水稻(1~3泳道)和甘蓝型油菜(4~6泳道)中的扩增结果。M: DL2000 marker; 1和4泳道: 引物bngs-1-1; 2和5泳道: 引物bngs-1-2; 3和6泳道: 引物bngs-1-3。B: 由白菜型油菜AC189411.2基因序列设计的引物在甘蓝型油菜SI-1300中的扩增结果。M: DL2000 marker; 1: 引物gs3-2; 2: 引物gs3-3; 3: 引物gs3-5。
-->Fig. 1Amplification results of primers designed according to homologous sequences
A: amplification profile in rice (lanes 1-3) and B. napus (lanes 4-6) with primers designed from rice GS3 gene sequence. M: DL2000 marker; Lanes 1 and 4: primer bngs-1-1; Lanes 2 and 5: primer bngs-1-2; Lanes 3 and 6: primer bngs-1-3. B: amplification profile in B. napus SI-1300 with primers designed from B. rapa AC189411.2 gene sequence. M: DL2000 marker; 1: primer gs3-2; 2: primer gs3-3; 3: primer gs3-5.
-->
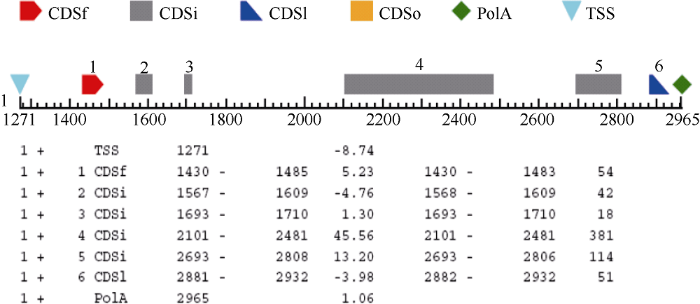 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2BnGS3基因结构预测
TSS: 转录起始位点; CDSf: 第1个外显子翻译起始位置; CDSi: 具有多个外显子; CDSo: 只有1个外显子; CDSI: 最后1个外显子翻译终止位置; Poly A: 转录终止位置; 数字表示碱基的位置。
-->Fig. 2Prediction of BnGS3 gene construction by FGENSH 2.0
TSS: transcription start site; CDSf: start coding site of the first exon; CDSi: inter exons of coding sequence; CDSo: only one exon of coding sequence; CDSI: last exon of coding sequence; Poly A: the site of termination of transcription; numbers indicate the site of base pair.
-->
根据BnGS3序列信息进行同源搜索, 发现与甘蓝型油菜EST序列DY004631同源性较高, 而BnGS3蛋白与GS3蛋白的序列同源性较低。借助预测蛋白的保守序列进行高级结构分析, 发现BnGS3蛋白缺少跨膜区域、TNFR/NGFR和PEBP-like的功能结构域, 只具有属于VWF结构域中的VWFA型的Iso_dh super family结构, 而GS3为VWFC型。甘蓝型油菜BnGS3与水稻GS3有一定的同源性, 但序列差异大。
2.1.2 Ghd7在甘蓝型油菜中的同源克隆 以甘蓝型油菜AY280868.2为模板设计的引物ghd7-2、ghd7-5、ghd7-6、ghd7-7和ghd7-9 (见附表1), 在甘蓝型油菜SI-1300中均有一条清晰扩增带(图3)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3引物ghd7-2、ghd7-5、ghd7-6、ghd7-7和ghd7-9在甘蓝型油菜SI-1300中的扩增
泳道M: DNA ladder DL2000; 泳道1: ghd7-2; 泳道2: ghd7-5; 泳道3: ghd7-6; 泳道4: ghd7-7; 泳道5: ghd7-9。
-->Fig. 3Amplification of primers ghd7-2, ghd7-5, ghd7-6, ghd7-7, and ghd7-9 in B. napus SI-1300
M: DNA ladder DL2000; 1: ghd7-2; 2: ghd7-5; 3: ghd7-6; 4: ghd7-7; 5: ghd7-9.
-->
扩增片段长度为2725 bp, 测序结果与甘蓝型油菜AY280868.2一致。通过结构预测发现在714~2700 bp处存在一个候选基因BnGhd7, 含有1个外显子, ORF全长1014 bp, 编码337个氨基酸(图4)。
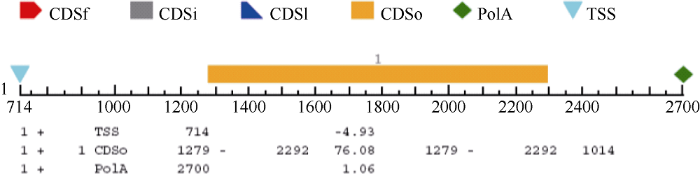 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4BnGhd7基因结构预测
TSS: 转录起始位点; CDSf: 第1个外显子翻译起始位置; CDSi: 具有多个外显子; CDSo: 只有1个外显子; CDSI: 最后1个外显子翻译终止位置; Poly A: 转录终止位置; 数字表示碱基的位置。
-->Fig. 4Prediction of BnGhd7 gene construction by FGENSH 2.0
TSS: transcription start site; CDSf: start coding site of the first exon; CDSi: inter exons of coding sequence; CDSo: only one exon of coding sequence; CDSI: last exon of coding sequence; Poly A: the site of termination of transcription; numbers indicate the site of base pair.
-->
根据BnGhd7的ORF序列进行同源搜索, 发现了多个高度同源的甘蓝型油菜EST序列。使用InterProScan软件对预测蛋白BnGhd7进行结构分析, 发现了2个重要的结构域, 其中N端存在1个B-Box结构域, C端存在1个CCT结构域, 与水稻Ghd7有高度同源性, 属于典型的CO基因家族。
2.2 甘蓝型油菜BnGS3和BnGhd7基因的变异位点检测
2.2.1 BnGS3基因的变异位点检测 在TN群体中随机选取15份材料, 通过引物gs3-2、gs3-3和gs3-5发现疑似变异位点, 但甘蓝型油菜作为异源四倍体存在基因多拷贝现象, 直接测序不能有效发现变异位点。根据测序结果重新设计了18对引物(编号brgs-1至brgs-18), 扩增范围介于400~1500 bp, 与引物gs3-2、gs3-3和gs3-5共同覆盖BnGS3的全长并形成有效重叠(见附表2)。通过SSCP技术在57份材料中发现多个突变位点, 筛选突变频率大于5的位点, 绘制BnGS3的比较测序结果(图5)。Supplementary table 2
附表2
附表2检测预测基因BnGS3、BnGhd7变异位点所用引物
Supplementary table 2Primers used in detecting BnGS3 and BnGhd7 polymorphic alleles
| 引物 Primer | 正向序列 Forward sequence (5'-3') | 反向序列 Reverse sequence (5'-3') | 退火温度 Tm (°C) |
|---|---|---|---|
| gs3-2 | GGGTTTCTGTTGGTGGGCTT | CCCATGAGGTATGTCAGCAT | 59 |
| gs3-5 | CCGCAGCGGAAAAGGTATGT | GTCCAGATGCTGCAAAGAGT | 59 |
| gs3-3 | TTCTAGACAATGAATGGCAGG | ACGAAAGAGTCGACGACACTG | 58 |
| brgs-1 | TAGTTTGGTGCTCACTCCTG | TCCTGCCATTCATTGTCTAG | 57 |
| brgs-2 | TAGACAATGAATGGCAGGAT | CTACCTTCGACCACAACCAG | 58 |
| brgs-3 | CGCCTCAACGAAGACTGGTT | ACGTCTGAGATGATCAAATG | 59 |
| brgs-4 | ATGTTGCCGTTGACCACGAT | CTGCAAGATCAAATGGTCAT | 60 |
| brgs-5 | TCGACTGCAACTGCGGAAAC | GGCGAGTTTCTGCTGTTGTT | 60 |
| brgs-6 | CACGCTCAAACACGGGAAAC | CCCAATGATACCTGCGTAAG | 61 |
| brgs-7 | TTGCCTCGCCGTCGGTTAGT | CCTCGCTTCGAATCAACACG | 59 |
| brgs-8 | CGAAGCCTCGTGGGTGTTAG | GGATCCAACTTGGTGATCAG | 60 |
| brgs-9 | GTTGGATCCGTACCGTACAT | AGCAATGATATCGGTTTGGT | 58 |
| brgs-10 | TCTCAAATTTGATGGGAAGC | CCCATGAGGTATGTCAGCAT | 59 |
| brgs-11 | GCTGACATACCTCATGGGAG | GTTGCTGTTGTTCTTGTCCG | 58 |
| brgs-12 | CTTTCGTTGTCTGCCCGGAT | GGACCATATACATGTTCACC | 58 |
| brgs-13 | TCTATGAAAGCAACATGACG | GCACCGTTGTGTTATGTTTG | 60 |
| 引物 Primer | 正向序列 Forward sequence (5'-3') | 反向序列 Reverse sequence (5'-3') | 退火温度 Tm (°C) |
| brgs-14 | TTCTAGACAATGAATGGCAG | AGAACACAAAAACGTACGTC | 60 |
| brgs-15 | TAAAGCTGAGAGCAGACTTG | GTCGACCATCATGTCTTAAT | 58 |
| brgs-16 | CAACAAAGTCGTAGCTTAGG | ATTCTGACCCAATGATACCT | 59 |
| brgs-17 | CTTTCGTTGTCTGCCCGGAT | GGTGTTAGTTCGTGCATGTG | 59 |
| brgs-18 | GTATATGGTCCTTTCAAACG | TTGCCCACTCAAATTAATTG | 58 |
| ghd7-2 | TTCGGATTCGGTTCTGGTTC | GCTCACATGATTGACAGACT | 59 |
| ghd7-5 | CCAAAAGCCAACGTCACCAT | GGAGCGACCGAAAACTACAT | 59 |
| ghd7-6 | TCCCTCACCAACAACAAACC | ATCCTTGGTCTTTTCTCTGC | 59 |
| ghd7-7 | GGGTTGTTCCACTTCAGGTT | ACCCATGGAAAGTGGTAGAT | 59 |
| ghd7-9 | GATCCATCACGGGCCATAAC | GGGTTTGAAACTGTTGTCTC | 59 |
| brghd-1 | TTATGTCGGGTTCGAATCGT | ATGGTTGGTCAAACGTGTAT | 60 |
| brghd-2 | TTGATGCAGAGGAGGCAGAC | TTCACCAAGAAAGCAATCAC | 60 |
| brghd-3 | TTATGTCGGGTTCGAATCGT | ATGGTTGGTCAAACGTGTAT | 58 |
| brghd-7 | ACTTTATGTTGCCGTTGACC | GCAACTGCAAGATCAAATGG | 60 |
| brghd-8 | CATGATGGTCGACTGCAACT | GCGAGTTTCTGCTGTTGTTG | 57 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5BnGS3比较测序结果示意图
*: 点突变; -: 小片段缺失; ^: 点突变密集区。
-->Fig. 5Schematic diagram of BnGS3 comparative sequencing
*: point mutation; -: small deletion; ^: point mutations concentrated area.
-->
引物brgs-16扩增片段长851 bp, 具有突变频率最高的多态性位点, 位于BnGS3第4个外显子区域的1010 bp到1050 bp处, 属于缺失突变位点(图6)。brgs-16扩增片段完全包含在引物gs3-5的扩增区域内, 且测序结果一致, 证明结果可靠。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6引物brgs-16扩增片段序列比较
-->Fig. 6Sequence alignment of amplified fragments in accessions by primer brgs-16
-->
2.2.2 BnGhd7基因的变异位点检测 设计可以有效覆盖目标区段的10对引物ghd7-2、ghd7-5~ghd7-7、ghd7-9、brghd-1~brghd-3、brghd-7和brghd-8 (见附表2), 在15份材料进行扩增。扩增片段测序, 仅有2个有效突变位点, 引物ghd7-7扩增片段在BnGhd7编码区下游有1个300 bp的缺失突变位点, 引物brghd-3扩增片段在BnGhd7起始位点上游700 bp处有1个突变位点(图7)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7BnGhd7比较测序结果示意图
*: 点突变; —: 长片段的缺失。
-->Fig. 7Schematic diagram of BnGhd7 comparative sequencing
*: point mutation; —: long fragment deletion.
-->
引物ghd7-7扩增片段长1578 bp, 该缺失位点介于BnGhd7下游非编码区的1488~1783 bp处。引物brghd-3位于BnGhd7起始位点上游700 bp处, 引物ghd7-2与brghd-3的重叠范围序列一致, 证明结果可靠(图8)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图8不同材料中brghd-3测序结果的比较
-->Fig. 8Alignment of the sequencing results of brghd-3
-->
2.3 变异位点与产量相关性状的相关性分析
在武汉和黄冈两地, 分别对57份甘蓝型油菜自交系进行产量相关性状的表型鉴定, 除武汉点播种至初花期天数外, 各性状的变异系数都超过5% (表1)。材料之间差异明显, 能够用来进行相关性分析。Table 1
表1
表157份甘蓝型油菜自交系9个产量相关性状的变异
Table 1Variations of nine yield-related traits in 57 B. napus inbred lines
| 性状 Trait | 平均数 Average | 标准差 SD | 变异区间 Variation interval | 变异系数 CV (%) |
|---|---|---|---|---|
| 武汉点 Wuhan location | ||||
| 株高 Plant height (cm) | 146.66 | 13.70 | 93.53-169.60 | 9.34 |
| 一次有效分枝 Number of primary branch | 6.19 | 1.08 | 4.40-9.67 | 17.37 |
| 单株角果数 Number of siliques per plant | 206.60 | 45.76 | 134.60-312.20 | 22.15 |
| 角果粒数 Number of seeds per silique | 19.51 | 3.89 | 10.30-28.11 | 19.91 |
| 单株产量 Yield per plant (g) | 10.91 | 2.34 | 5.25-16.36 | 21.42 |
| 小区产量 Plot yield (g) | 452.69 | 94.12 | 209.31-694.48 | 20.79 |
| 千粒重 Thousand-seed weight (g) | 3.62 | 0.58 | 2.28-5.07 | 16.08 |
| 播种-初花期天数 Days from sowing to initial bloom | 159.19 | 4.87 | 151.00-168.00 | 3.06 |
| 初花期-终花期天数 Days from initial bloom to end flowering | 17.19 | 4.53 | 9.00-26.00 | 26.37 |
| 黄冈点 Huanggang location | ||||
| 株高 Plant height (cm) | 162.26 | 14.60 | 89.73-191.53 | 9.00 |
| 一次有效分枝 Number of primary branch | 6.49 | 1.18 | 3.47-9.67 | 18.18 |
| 单株角果数 Number of siliques per plant | 206.25 | 39.89 | 116.60-308.93 | 19.34 |
| 角果粒数 Number of seeds per silique | 20.60 | 4.00 | 12.81-32.49 | 19.45 |
| 单株产量 Yield per plant (g) | 13.40 | 2.14 | 9.72-18.02 | 15.95 |
| 小区产量 Plot yield (g) | 558.95 | 102.46 | 311.37-842.05 | 18.33 |
| 千粒重 Thousand-seed weight (g) | 3.77 | 0.62 | 2.25-5.43 | 16.46 |
| 播种-初花期天数 Days from sowing to initial bloom | 158.13 | 8.88 | 135.00-171.67 | 5.61 |
| 初花期-终花期天数 Days from initial bloom to end flowering | 27.04 | 7.09 | 17.00-49.00 | 26.21 |
新窗口打开
分别将3个标记brgs-16、ghd7-7和brghd-3与黄冈测得的表型数据进行相关性分析。BnGS3的标记brgs-16与表型数据不存在相关性。BnGhd7的标记brghd-3与千粒重显著相关, ghd7-7与花期显著负相关, 与株高极显著相关(表2)。
Table 2
表2
表2标记与产量等相关性状之间的相关分析结果(黄冈)
Table 2Correlation coefficients between markers and traits (site: Huanggang)
| 性状 Trait | brgs-16 | brghd-3 | ghd7-7 |
|---|---|---|---|
| 株高 Plant height | 0.01 | -0.05 | 0.34** |
| 一次有效分枝 Number of first branches | 0.10 | 0.08 | 0.17 |
| 单株角果数 Number of siliques per plant | 0.06 | -0.01 | 0.12 |
| 角果粒数 Number of seeds per silique | -0.11 | -0.20 | -0.05 |
| 单株产量 Yield per plant | 0.00 | -0.13 | -0.06 |
| 小区产量 Plot yield | 0.18 | -0.05 | 0.21 |
| 千粒重 Thousand-seed weight | 0.12 | 0.30* | 0.14 |
| 播种-初花期天数 Days from sowing to initial bloom | -0.06 | -0.13 | 0.25 |
| 初花期-终花期天数 Days from initial bloom to end flowering | 0.10 | 0.16 | -0.32* |
新窗口打开
以TN群体作为定位群体, 引物brgs-15和引物ghd7-7在两亲本间具有多态性扩增产物。brgs-15是1个SSCP标记, 定位于A2连锁群(见附图1); ghd7-7是1个显性标记, 定位于A10连锁群(见附图2)。因而BnGS3和BnGhd7被分别定位于油菜A2和A10连锁群。
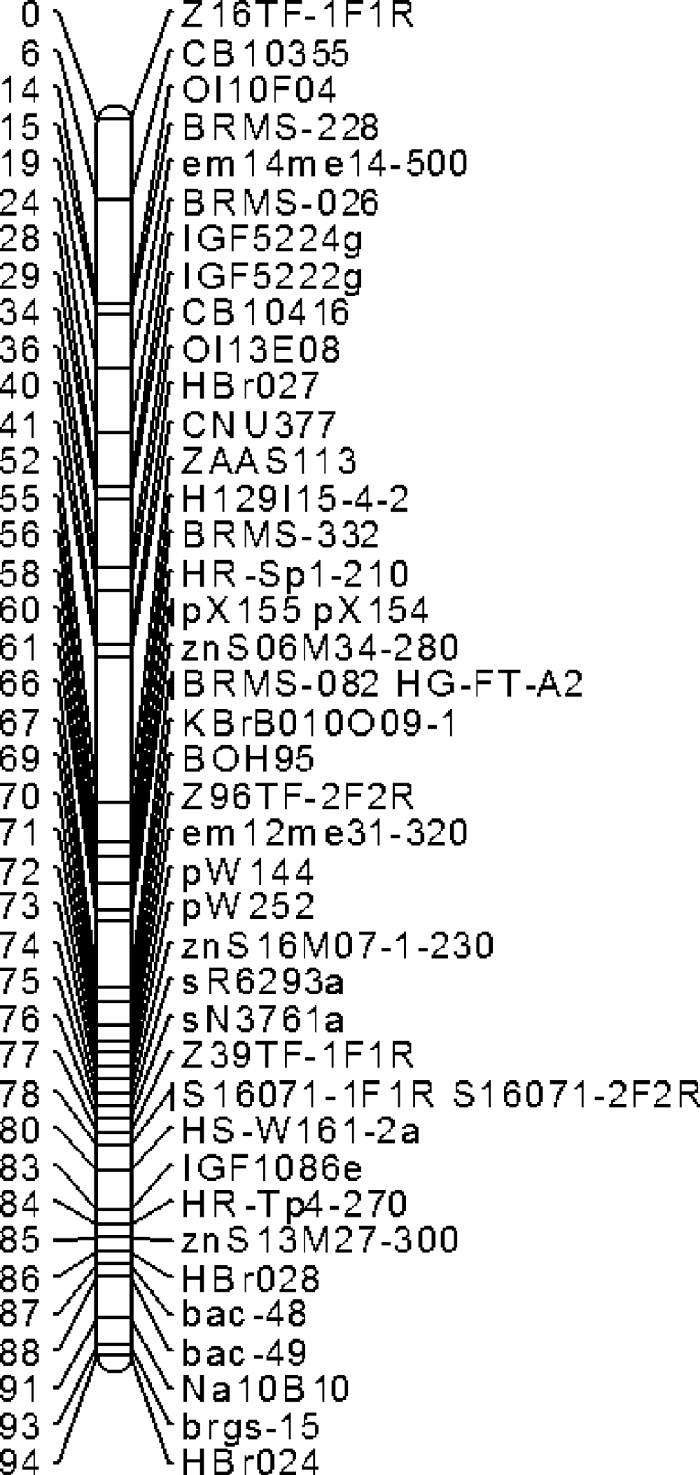 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT附图1brgs-15被定位在A2连锁群
-->Supplementary fig. 1brgs-15 located in linkage group A2
-->
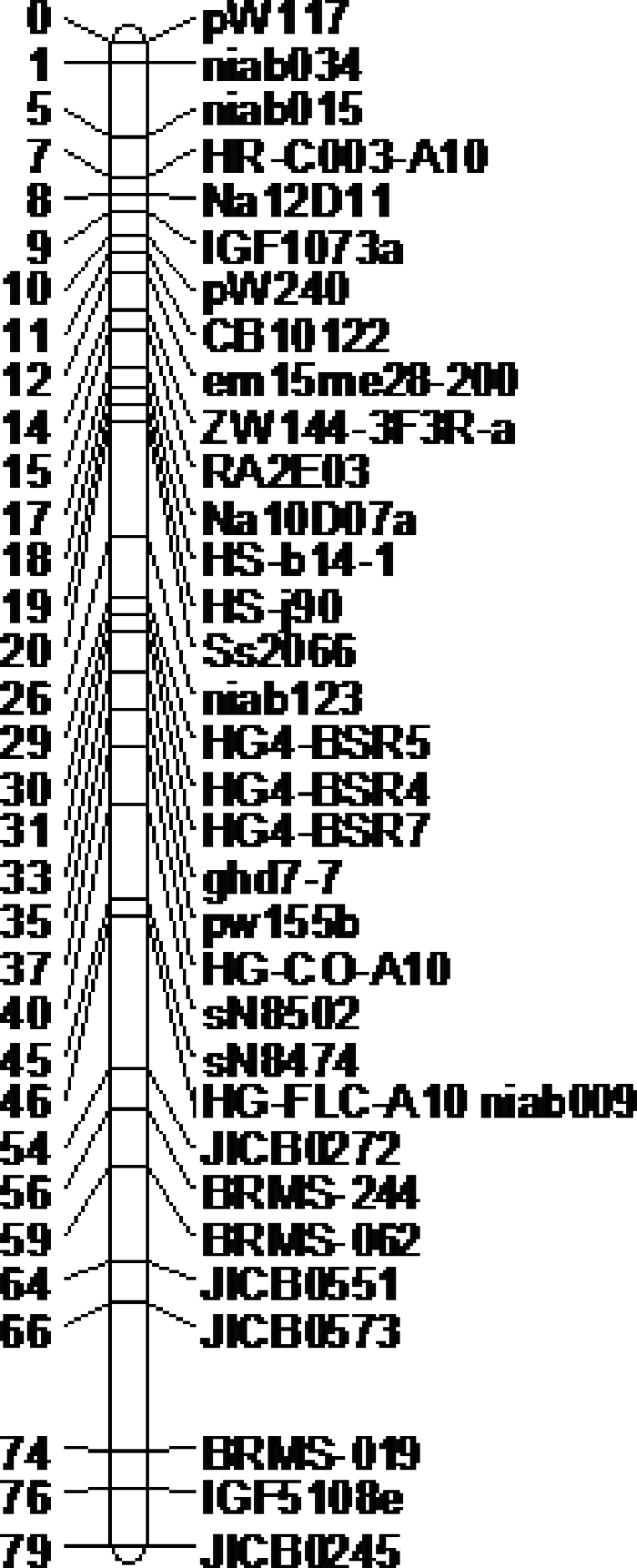 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT附图2ghd7-7被定位于A10连锁群
-->Supplementary fig. 2ghd7-7 located in linkage group A10
-->
3 讨论
作为第1个基因组被完整测序的植物, 拟南芥成为遗传学、分子生物学等领域的典型研究对象[16]。对模式生物拟南芥的深入解析曾加速了水稻等一些更高等植物的研究[17]。近年来水稻作为高等模式生物成为新的研究热点并在基因组和功能基因的研究中取得了巨大成果[18]。禾本科作物水稻和十字花科芸薹属甘蓝型油菜在进化上亲缘关系较远, 在基因组结构和组成上存在很大差异, 缺乏利用水稻基因信息在甘蓝型油菜中克隆同源基因的报道, 但比较基因组学研究结果表明, 两种作物基因组仍然具有共线性。本研究利用水稻GS3和Ghd7的序列设计引物扩增油菜基因组DNA, 没有扩增产物或无特异扩增。因而, 尝试将拟南芥作为同源克隆的媒介, 首先通过GS3的序列信息分别在拟南芥和白菜中发现了同源序列NP_ 680175.2和AC189411.2, 分别设计引物, 但是, 只有依据白菜AC189411.2设计引物gs3-2、gs3-3和gs3-5在甘蓝型油菜SI-1300中能有效扩增。同样地, 根据与Ghd7同源的甘蓝型油菜AY280868.2序列设计引物, 在SI-1300有特异扩增带。说明利用远缘物种如水稻的功能基因进行油菜基因功能研究时, 借助拟南芥为媒介, 搜索油菜或其两个祖先种甘蓝和白菜序列的同源片段, 设计引物进行同源克隆, 是一种有效的途径。本研究克隆到油菜基因BnGS3和BnGhd7, 比较基因变异位点得到BnGS3的多态性标记brgs-16、Bghd7的多态性标记brghd-3和brgs-16。BnGS3有6个外显子, ORF全长666 bp, 编码222个氨基酸。水稻GS3全长5363 bp, 包含4个内含子和5个外显子, 阅读框长699 bp。GS3编码的蛋白由232个氨基酸组成, 包含4个保守的结构域, 分别为磷脂酞乙醇胺结合蛋白区域(PEBP-like)、跨膜区域、生长因子受体家族富半胱氨酸同源区域(TNFR)/ (NGFR)和von Willebrand因子C型结构域(VWFC)[19,20]。BnGS3虽然只具有水稻GS3四个保守结构域中的von Willebrand factor(VWF)结构, 且属于A型(水稻为C型), 但该结构域已被发现存在于许多胞外蛋白中, 参与调节生长因子信号传导[21,22]。BnGS3的标记brgs-16与产量性状不存在相关性。因而甘蓝型油菜BnGS3与水稻GS3有一定的同源性, 但基因功能上的相似性需要进一步验证。
BnGhd7含有1个外显子, ORF全长1014 bp, 编码337个氨基酸。BnGhd7蛋白具有N端的B-Box和C端的CCT两个重要的结构域, 据此认为属于CO基因家族, 与水稻Ghd7有高度同源性, 植物中CO基因家族在调控开花期上发挥重要功能[23,24]。相关性分析发现BnGhd7不仅与花期相关, 也与株高和千粒重相关, BnGhd7很可能与水稻Ghd7具有相似的功能。
BnGS3和BnGhd7被分别定位在油菜A2连锁群、A10连锁群。A2连锁群存在大量产量及相关性状的QTL, 包括花期、株高、全株角果数、角果粒数、千粒重、种子产量等[25]。由于brgs-15标记位于连锁群末端, 与A2连锁群包含的产量性状相关性低, 因而BnGS3的标记brgs-16与产量性状不存在相关性。ghd7-7被定位到A10连锁群, 该区域同样存在大量产量及相关性状QTL, 包括产量、花期、成熟期、株高、全株角果数、千粒重和种子产量等性状[26,27]。A10连锁群上与花期相关的QTL区间与拟南芥第5染色体顶端的区间具有共线性, 在该区域具有拟南芥开花基因CO、FY和FLC等, 而ghd7-7也被定位于该区间内。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | . |
| [2] | . |
| [3] | . |
| [4] | . |
| [5] | . |
| [6] | . |
| [7] | . |
| [8] | . |
| [9] | . . |
| [10] | |
| [11] | . |
| [12] | . |
| [13] | . |
| [14] | . |
| [15] | . |
| [16] | . |
| [17] | . |
| [18] | . |
| [19] | . |
| [20] | . |
| [21] | . |
| [22] | . |
| [23] | . |
| [24] | . |
| [25] | . |
| [26] | . |
| [27] | . |
