 ,**, 左同鸿**, 胡燈科, 刘倩莹, 张以忠, 张贺翠, 曾文艺, 袁崇墨, 朱利泉
,**, 左同鸿**, 胡燈科, 刘倩莹, 张以忠, 张贺翠, 曾文艺, 袁崇墨, 朱利泉 ,*西南大学农学与生物科技学院, 重庆 400716
,*西南大学农学与生物科技学院, 重庆 400716Molecular cloning and expression analysis of BoPUB9 in self-incompatibility Brassica oleracea
XIE Qin-Qin ,**, ZUO Tong-Hong**, HU Deng-Ke, LIU Qian-Ying, ZHANG Yi-Zhong, ZHANG He-Cui, ZENG Wen-Yi, YUAN Chong-Mo, ZHU Li-Quan
,**, ZUO Tong-Hong**, HU Deng-Ke, LIU Qian-Ying, ZHANG Yi-Zhong, ZHANG He-Cui, ZENG Wen-Yi, YUAN Chong-Mo, ZHU Li-Quan ,*College of Agronomy and Biotechnology, Southwest University, Chongqing 400716, China
,*College of Agronomy and Biotechnology, Southwest University, Chongqing 400716, China通讯作者: *朱利泉, E-mail:zhuliquan@swu.edu.cn
第一联系人:
收稿日期:2020-12-16接受日期:2021-04-14网络出版日期:2021-06-09
| 基金资助: |
Corresponding authors: *E-mail:zhuliquan@swu.edu.cn
First author contact:
Received:2020-12-16Accepted:2021-04-14Published online:2021-06-09
| Fund supported: |
作者简介 About authors
E-mail:xieqq1995@163.com

摘要
自交不亲和性(self-incompatibility, SI)是指植株的雌蕊对自身花粉和异体花粉进行识别从而抑制自身花粉萌发的特性, PUB (Plant U-Box)蛋白在植物抗逆及信号转导过程中起着重要的作用。本研究通过分析甘蓝自花授粉0~60 min的柱头转录组数据, 筛选到1个受自花授粉诱导上调表达的PUB蛋白编码基因BoPUB9。BoPUB9 cDNA序列长度为1368 bp, gDNA序列全长1720 bp, 含有1个臂重复结构域和1个U-box结构域。荧光定量PCR结果显示, BoPUB9基因在甘蓝的不同组织中均有表达, 在萼片中的表达量最高, 花瓣、雄蕊、柱头表达量次之, 在花粉花蕾中表达量相对较低, 同GUS染色结果保持一致。BoPUB9基因在自花授粉0~30 min内快速上调表达, 15 min后为异花授粉后表达量的20多倍, 在30 min后达到差异最大, 自花授粉后的表达量为异花授粉的40多倍。亚细胞定位结果显示, BoPUB9蛋白在细胞核和细胞质中均有表达, 且BoPUB9蛋白在大肠杆菌中被成功诱导表达, 相对分子质量为51 kD, 与预测结果一致。酵母双杂交与poll-down结果显示, SRK胞内域与PUB9蛋白存在相互作用。推测BoPUB9可能是一种参与甘蓝自交不亲和反应过程的新基因, 这为甘蓝自交不亲和的进一步研究和利用提供了新内容。
关键词:
Abstract
Self-incompatibility (SI) refers to the characteristic that the pistil of a plant can recognize its own pollen and allogeneic pollen so as to inhibit the germination of its own pollen. PUB (Plant U-Box) proteins play an important role in plant stress resistance and signal transduction. In this study, through the analysis of stigma transcriptome data of Brassica oleracea self-pollination at 0-60 minute(s), a PUB protein coding gene BoPUB9 was screened and up-regulated by self-pollination. The BoPUB9 cDNA sequence was 1368 bp, gDNA of 1720 bp in length, containing one arm repeat domain and one U-box domain. The relative expression levels showed that BoPUB9 gene was expressed in different tissues of Brassica oleracea, and the relative expression level was the highest in sepals, followed by petals, stamens and stigmas, and relatively lower in pollen buds, which was consistent with the results of GUS staining. The relative expression level of BoPUB9 gene was up-regulated rapidly in self-pollination 0-30 minute(s), the expression level after 15 min was more than 20 times that after cross-pollination, and the difference was the biggest after 30 min, the expression level after self-pollination was more than 40 times that after cross-pollination. Subcellular localization revealed that BoPUB9 protein was expressed in both nucleus and cytoplasm, and BoPUB9 protein was successfully induced in E. coli, and the relative molecular weight of 51 kD was consistent with the predicted results. The results of yeast two-hybrid and poll-down indicated that the intracellular domain of SRK interacted with PUB9 protein. It was speculated that BoPUB9 might be a novel gene involved in the process of self-incompatibility in Brassica oleracea, which providing a new content for further research and utilization of self-incompatibility in Brassica oleracea.
Keywords:
PDF (7629KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
谢琴琴, 左同鸿, 胡燈科, 刘倩莹, 张以忠, 张贺翠, 曾文艺, 袁崇墨, 朱利泉. 甘蓝自交不亲和相关基因BoPUB9的克隆及表达分析. 作物学报, 2022, 48(1): 108-120 DOI:10.3724/SP.J.1006.2022.04276
XIE Qin-Qin, ZUO Tong-Hong, HU Deng-Ke, LIU Qian-Ying, ZHANG Yi-Zhong, ZHANG He-Cui, ZENG Wen-Yi, YUAN Chong-Mo, ZHU Li-Quan.
植物的自交不亲和性(self-incompatibility, SI)是指植株的雌蕊对自身花粉和异体花粉进行识别, 并能抑制自身花粉或同一品系内的异株花粉在自身的柱头上萌发或生长的特性, 此特性有利于促进杂交优势, 防止自交衰退[1,2]。甘蓝属于典型自交不亲和植物, 当前国内外研究其分子机制主要集中在SI信号传导元件协同作用抑制自花花粉萌发或花粉管生长上, 这一过程是通过磷酸化激活SI信号元件进行信号传导, 在SI信号传导下游再通过泛素化降解花粉亲和因子, 这其中S位点受体激酶(S-locus receptor kinase, SRK)、S位点糖蛋白(Slocus glyco- protein, SLG)、S位点富集半胱氨酸蛋白(Slocus cysteinrichprotein, SCR)是自交不亲和反应中已被鉴定出的关键因子[3,4]。Indriolo与Nasrallah 2个研究团队曾对E3连接酶ARC1 (arm repeat-containing)是否为SI信号途径中的关键因子产生2种不同的结论, 前者将SCR-SRK-ARC1基因同时转化到拟南芥的2个生态型中发现, 所有的转基因植株都表现出了强烈而稳定的自交不亲和表型, 因此认为ARC1是致使拟南芥重新恢复自交不亲和性的关键因子, 后者提出相反意见, 将SCR-SRK基因转入拟南芥中后也得到了自交不亲和表型, 猜测拟南芥表现出强的自交不亲和性并不需要ARC1基因的参与[5,6,7]。但仅通过转化SRK-SCR基因后植株就可以表现出强自交不亲和性, 并且在甘蓝型油菜中过表达ARC1下游底物Exo70A1只能部分打破自交不亲和性, 说明的确存在除ARC1之外的信号元件和信号通路参与SRK下游信号传导。因此, 探索其他参与调控SI信号途径的蛋白质元件的编码基因, 可为芸薹属植物的育性理论研究及育种措施提供新的参考策略。
ARC1作为典型植物U-Box基因在自交不亲和信号传导下游使底物蛋白泛素化, 通过发挥U-Box与C末端臂重复区(ARM)相结合的功能促使甘蓝自交不亲和信号成功传导, 有****指出在自花授粉的过程中, 柱头内泛素蛋白的活性急剧增加, 由此可能存在其他泛素蛋白与SRK发生反应, 继续向下游传递SI信号[8]。U-Box功能域最初是在酵母的UFDZ蛋白中发现的, 约由70个氨基酸残基组成, 是真核生物体内广泛存在的一种高度保守的基序, 发挥E3泛素连接酶作用, 在生物体内促进下游底物蛋白的泛素化降解[3,9]。与在酵母和人类基因组中鉴定的2个和21个U-box基因相比, 在各种植物基因组中预测到的包含U-box的基因数量大大增加, 已经有64个U-box基因在拟南芥基因组中的预测, 在水稻基因组中标注了77个Plant U-Box (PUB)基因, 大豆中有125个被注释[3,6,10-12]。基于对这些蛋白质中存在的其他结构域分析, PUB蛋白家族可分为5个不同的亚类, I类PUB蛋白、II类PUB蛋白、III类PUB蛋白、IV类PUB蛋白、V类PUB蛋白, 其中I类PUB蛋白为UFD2直系同源物[13]。PUB最先的功能研究是它们对多种压力的转录诱导, 通过ABA途径对干旱或渗透胁迫产生响应的细胞信号传导, 并且许多PUB参与了免疫应答和细胞死亡的调节, 在拟南芥中PUB22、PUB23和PUB24以及PUB12和PUB13的突变体均显示出增强的免疫应答和对病原体的抗性, 表明它们参与了免疫应答的机制[14,15,16,17,18,19]。PUB可通过不同水平参与蛋白酶解系统进行细胞器的降解, 如参与降解叶绿体过程等, PUB与囊泡运输相关的过程可直接联系起来, 大多数已鉴定的PUB底物和相互作用的蛋白是固有的膜蛋白, 是与膜运输相关的调节剂[20,21]。
本研究前期通过甘蓝自花和异花授粉后的柱头转录组数据, 首次筛选到一个受自花授粉诱导上调表达的新基因BoPUB9, 该基因与ARC1同属于U-Box家族成员, 进而通过基因克隆、生物信息学分析、组织特异性分析、启动子活性分析、亚细胞定位、原核表达以及酵母双杂交等深入探索, 以期证明该基因编码蛋白参与甘蓝自交不亲和反应, 为甘蓝SI的深入研究提供依据。
1 材料与方法
1.1 材料
本试验材料为由西南大学园艺园林学院十字花科蔬菜研究所隔离种植的甘蓝高代自交不亲和系A4与F1植株, 在2019年3月至4月对材料进行人工去雄, 选取长势一致的A4材料为母本分别以F1与A4为父本进行异花授粉与自花授粉, 授粉时间为0、15、30、60 min, 授粉处理后迅速除去柱头上的花粉, 立刻将柱头放入液氮速冻, -80℃保存备用。同时保存叶片、花药、花瓣、花蕾、柱头、萼片材料备用。后续启动子活性分析试验所用材料为哥伦比亚野生型拟南芥, 由本实验室保存。质粒小提试剂盒、各种引物合成及测序等均由上海生工生物工程有限公司完成, 所用大肠杆菌DH5α购自北京擎科新业生物技术有限公司, 农杆菌GV3101、酵母Y2Hgold和原核表达菌株E. coli BL21 (DE3)均购自上海唯地生物技术有限公司。
1.2 转录组测序及差异表达基因筛选与克隆
将差异授粉0、15、30、60 min材料送百迈客公司进行Illumina HiSeq高通量测序, 采用FPKM (Fragments Per Kilobase of transcript per Million fragments mapped)作为衡量转录本或基因表达水平的指标。在差异表达基因检测过程中, 将差异倍数Fold Change≥2且错误发现率(False Discovery Rate) FDR<0.01作为筛选标准, 筛选差异显著基因。根据上述转录组筛选获得的目的基因cDNA序列, 结合芸薹属数据库(http://brassicadb.org/brad/ geneFamily.php), 参考同源重组原理及酵母表达载体特征, 利用Primer premier 6.0软件设计引物BoPUB9-AD-F和BoPUB9-AD-R (表1), 分别以甘蓝叶片gDNA和柱头cDNA为模板, 利用PrimerSTAR Max DNA Polymerase (TaKaRa, 北京) 25 µL PCR反应体系扩增目的基因的编码序列与全长。反应体系包含模板1 µL (200 ng µL-1)、上下游引物各1 µL (10 pmol µL-1)、ddH2O 9.5 µL、2×PCR Mixster Mix混合酶12.5 µL。反应程序为94℃预变性3 min; 94℃ 30 s, 59℃ 30 s, 72℃延伸1 min 25 s, 40个循环; 72℃终延伸5 min。将目的片段与pGADT7载体连接后转入大肠杆菌感受态细胞DH5α, 经菌液PCR将筛选出的阳性克隆送上海生物工程股份有限公司测序。同时参考NCBI网站和SMART (http://smart.embl- heidelberg.de/)在线分析目的蛋白保守结构域, 根据分析结果, 按照上述方法设计引物SRKj-AD-F、SRKj-AD-R以扩增甘蓝SI关键因子SRK的激酶结构域序列(表1), 连接至酵母表达载体pGBKT7。
Table 1
表1
表1本试验所用引物
Table 1
| 引物名称 Primer name | 引物序列 Primer sequence (5°-3°) | 用途 Functions |
|---|---|---|
| BoPUB9-AD | F: GTACCAGATTACGCTCATATGATGGCAAAGACCGGATTTTTC | 酵母双杂交引物 |
| R: ATGCCCACCCGGGTGGAATTCGGCTGTGTGAGTAAGATTCATA | Yeast two-hybrid primers | |
| SRKj-BK | F: GTACCAGATTACGCTCATATGTCAGATCGTGAGGTTTTCAATTC | 酵母双杂交引物 |
| R: ATGCCCACCCGGGTGGAATTCGCCTTGTTATCGTTCCATGACTA | Yeast two-hybrid primers | |
| BoPUB9-PAN | F: AAGTCCGGAGCTAGCTCTAGAATGGCAAAGACCGGATTTTTC | 基因亚细胞定位 |
| R: GCCCTTGCTCACCATGGATCCGGCTGTGTGAGTAAGATTCATA | Subcellular location | |
| BoPUB9-PGEX | F: GATCTGGTTCCGCGTGGATCCATGGCAAAGACCGGATTTTTC | 基因的原核表达 |
| R: GATGCGGCCGCTCGAGTCGACGGCTGTGTGAGTAAGATTCATA | Prokaryotic expression | |
| BoPUB9-GUS | F: CAAGCTTGGCTGCAGGTCGACGTCTTCTTAACGACATGGCTTG | 基因的启动子活性分析 |
| R: GGTGGACTCCTCTTAGAATTCCTCCGTCGGATCCACCG | Promoter activity analysis | |
| dActin | F: GGCTGATGGTGAAGATATTCA | 内参引物 |
| R: CAAGCACAATACCAGTAGTAC | Internal reference primers | |
| qRT-PCR | F: TGGCTTCTGGTCAGACATACGA | 荧光定量PCR引物 |
| R: GTGACAGCTTTCTTGGACTCG | qRT-PCR primers |
新窗口打开|下载CSV
1.3 生物信息学分析
利用在线网站或软件对目的基因进行生物信息学分析, 具体内容如表2所示。Table 2
表2
表2生物信息学分析所需软件及在线网站
Table 2
| 软件或在线网站 Software and online websites | 功能 Functions |
|---|---|
| Bio-soft ( | 推导基因编码的氨基酸序列 |
| Bio-soft ( | Derive the amino acid sequence encoded by the gene |
| ExPASy-ProtParam tool ( | 分析蛋白质理化性质 |
| Analysis of physical and chemical properties of protein | |
| Signalp ( | 预测蛋白质的信号肽 |
| Signal peptides for predicting proteins | |
| TMHMM ( | 预测蛋白质的跨膜结构域 |
| Prediction of transmembrane domains of proteins | |
| Netphos ( | 预测磷酸化位点 |
| Predicted phosphorylation site | |
| NetNGlyc ( | 预测N-糖基化位点 |
| Prediction of N-glycosylation sites | |
| ProtScale ( | 预测疏水性/亲水性 |
| Prediction of hydrophobicity/hydrophilicity | |
| SMART( | 蛋白质的高级结构域 |
| Advanced domain of protein | |
| PROSITE ( | 蛋白质的功能位点 |
| Functional sites of proteins | |
| PlantCARE ( | 分析启动子的顺式作用元件 |
| Analysis of cis-acting elements of promoter | |
| MEGA7软件和NCBI ( | 构建进化树 |
| MEGA7 software and NCBI ( | Construct an evolutionary tree |
| BRAD ( | 分析基因启动子序列 |
| Analysis of gene promoter sequence |
新窗口打开|下载CSV
1.4 表达分析
按照RNAprep pure Plant Kit (天根生化科技有限公司)说明书提取甘蓝叶片、花药、花瓣、花蕾、柱头、萼片的RNA, 再反转录合成cDNA作为荧光定量PCR的模板, 分别用1.0%和1.5%琼脂糖凝胶电泳检测DNA和RNA的完整性, 内参Actin3引物序列及后续荧光定量PCR引物见表1。利用Bio- RadCFX-1000荧光定量PCR仪上对目的基因进行植物组织荧光定量PCR反应。荧光定量PCR的反应体系为20 µL, 包含2×SYBR Premix Ex Taq 10 μL、上下游引物各0.5 μL (10 pmol µL-1)、cDNA模板1 μL (80 ng µL-1)、ddH2O 8 μL。荧光定量PCR的反应程序为95℃预变性2 min; 95℃变性15 s, 60℃退火15 s, 72℃延伸20 s, 40个循环。每个反应3次生物学重复, 采用2-ΔΔCT法计算目的基因的相对表达水平, 同时利用不同授粉处理后各个时间段的柱头cDNA作为模板, 利用荧光定量PCR检测目的基因的表达模式, 反应体系及过程与组织表达荧光定量PCR一致。1.5 启动子活性分析
于NCBI上查找甘蓝基因组信息, 获得目的基因启动子上游2000 bp左右的序列作为目的片段, 根据同源重组的方法设计引物BoPUB9-GUSF/ BoPUB9-GUSR, 序列见表1, 以甘蓝A4柱头gDNA为模板扩增目的片段, 与pCAMBIA 1391载体构建融合表达载体, 将融合产物转入大肠杆菌感受态DH5α, 挑选出阳性克隆测序正确后提取质粒, 将质粒转化农杆菌GV3101感受态细胞涂布含Kana/Strep/Rif的YEB固体培养基, 挑斑检测阳性单菌落, 将阳性转化子按照1∶100扩大培养, 利用花序侵染法转化野生型拟南芥。将收获种子进行潮霉素抗性筛选, 经PCR鉴定为阳性的转基因植株单株收种, 继续抗性筛选直至得到纯合转基因株系。取转基因植株叶片、果荚、花和花蕾进行37℃ GUS染色, 分别用95%、75%的酒精依次脱色, 以野生型拟南芥作为对照, 在体式显微镜下观察并照相。1.6 亚细胞定位
根据目的基因编码序列与带有绿色荧光GFP标签的PAN580载体序列信息设计引物(表1), 以甘蓝A4柱头cDNA为模板扩增目的基因编码区序列, PCR体系含1 μL (10 pmol µL-1)模板、上下游引物各1 μL (200 ng µL-1)、12.5 μL Kodaq 2 × PCR MasterMix缓冲液, ddH2O补足体系至25 μL。反应程序为94℃ 3 min; 94℃ 30 s, 59℃ 30 s, 72℃ 1 min 30 s, 40个循环; 72℃延伸5 min。回收目的基因片段, 运用快速克隆技术将目的基因与载体连接, 将融合表达载体转入大肠杆菌感受态DH5α, 挑选出阳性克隆测序正确后提取质粒, 以生长4周龄的拟南芥叶片为材料按照Steffen Abel方法提取原生质体[22]。将重组质粒与空载质粒用PEG法转入拟南芥原生质体, 23℃黑暗培养12~16 h后在激光共聚焦显微镜下观察绿色荧光以研究目的蛋白的亚细胞定位情况。1.7 原核表达
根据目的基因编码序列与原核表达载体pGEX-4T-1序列设计引物(表1)扩增目的基因片段, 将目的基因与pGEX-4T-1载体连接, 将测序正确后的菌株扩大培养提取质粒, 将质粒转化E. coli BL21 (DE3), 进行PCR检测后将阳性转化子单克隆和pGEX-4T-1空载菌液按照1∶100扩大培养, 取100 μL菌液接种于100 mL含100 µg mL-1氨苄抗性LB液体培养基中, 置于摇床37℃ 200转 min-1振荡培养至OD600达到0.6左右, 加入IPTG至终浓度为1 mmol L-1, 16℃过夜诱导后, 收集菌体, 进行超声破碎, 破碎结束后离心收集上清液, 经海狸GST融合蛋白纯化磁珠纯化目的蛋白, 用SDS-PAGE电泳检测空载与目的基因蛋白的表达, 其中空载pGEX-4T-1作为对照。1.8 共转酵母感受态细胞及酵母双杂互作的鉴定
1.8.1 构建诱饵载体及自激活验证 以BoSRK激酶区域序列与诱饵载体pGBKT7的限制性酶切位点特征、以目的基因编码序列与pGADT7载体序列特征分别设计引物, 利用同源重组的方法连接目的片段转化大肠杆菌, 经测序正确后将阳性克隆提取质粒, 将重组质粒BoSRKj-pGBKT7与pGADT7空载质粒利用聚乙二醇/醋酸锂法(PEG/LiAc)共转化酵母Y2HGold感受态细胞, 转化后的菌液涂布在SD/-Trp、DDO (SD/-Leu/-Trp)、QDO (SD/-Leu/- Trp/-His/-Ade/)平板上, 并设立阴性对照(pGADT7- T×pGBDT7-Lam)及阳性对照(pGADT7-T×pGBDT7- 53), 在30℃培养环境倒置培养3~5 d, 验证BoSRKj-pGBKT7重组质粒是否存在自激活。1.8.2 BoSRKj-pGBKT7与BoPUB9-pGADT7酵母双杂鉴定 根据制造商酵母方案手册(Invitrogen), 使用Clontech双杂交系统筛选酵母双杂交, 按照聚乙二醇/醋酸锂法(PEG/LiAc)将BoSRKj-pGBKT7与BoPUB9-pGADT7质粒共转将共转化后的酵母感受态细胞涂布于营养缺陷型SD/-Leu/-Trp固体培养基上, 在30℃培养环境倒置培养3~5 d, 观察是否有菌斑生成, 若有菌斑生成, 将菌斑用无菌水稀释100倍点涂在营养缺陷型QDO固体培养基上, 观察其生长情况。
1.9 BoPUB9蛋白与BoSRKj相互作用的体外检测
分别从甘蓝中克隆BoSRKj以及BoPUB9基因连至载体pGEX-4T-1和pET43.1a。提取质粒BoPUB9-GST和BoSRKj-His分别测序无误后转入原核表达菌E. coli DE3中进进行大量诱导表达, 分别用Beaver Beads GSH磁珠和MagneHis Protein Purification System试剂盒进行纯化。诱导表达菌pGEX-BoSRKj经纯化后分别与pET43.1a-PUB9 融合蛋白的提取液进行孵育, 同时用pGEX-4T-1空载体纯化后分别与pET43.1a-PUB9蛋白提取液孵育作为对照, 孵育结束后用Beaver Beads GSH磁珠进行纯化, 最后洗脱产物、靶蛋白和诱饵蛋白的纯化产物分别同时进行SDS-PAGE电泳得到检测结果。2 结果与分析
2.1 BoPUB9基因的转录水平分析
根据甘蓝授粉后的柱头转录组数据, 筛选到1个自花授粉后表达量存在差异明显的基因, 该基因的相对表达量随着授粉时间变化如图1所示, 从自花授粉(self-pollination, SP) 0 min开始上调表达, 发现该基因在自花授粉30 min时相对表达量达到最高, 随后上升速度变缓, 但仍高于自花授粉0 min的表达量; 而在异花授粉(cross-pollination, CP)表现为先上调后下调的趋势, 在异花授粉60 min时远低于0 min的表达量。在授粉30 min内, 该基因在自花和异花授粉的表达变化趋势相同, 均为上调, 但是上调速度不同, 自花授粉表达量更高, 30~60 min内该基因在自花授粉后依然上调表达, 但在异花授粉后下调表达。根据甘蓝自交不亲和的特性, 该基因在整个SI信号传导过程中于30 min时表达量高度上调, 此时为SI相关基因表达的关键时刻, 表明该基因可能参与自交不亲和信号传导。对该基因序列进行Blast比对发现, 它与拟南芥中具有泛素蛋白连接酶活性的ARM超家族蛋白基因AtPUB9高度同源, 命名为BoPUB9。图1
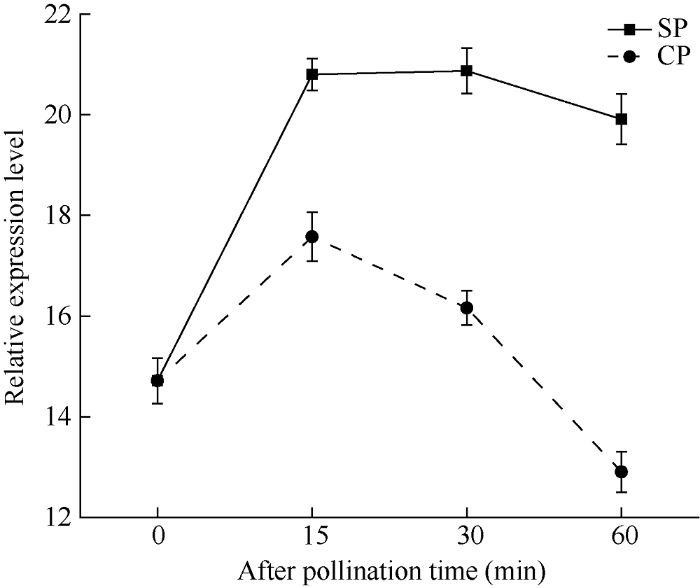 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1转录组数据分析BoPUB9自花授粉和异花授粉后的表达模式
SP: 自花授粉; CP: 异花授粉。
Fig. 1Relative expression patterns of BoPUB9 after self- pollination and cross-pollination based on transcriptome data
SP: self-pollination; CP: cross-pollination.
2.2 BoPUB9基因的克隆
以提取甘蓝A4柱头RNA经反转得到的cDNA为模板, 进行PCR扩增, 获得该基因全长及编码区PCR产物大小约1800 bp与1400 bp (图2)。经测序cDNA序列长度为1368 bp, 包含了其完整的编码框, gDNA序列全长1720 bp, 得到的测序结果与数据库获得的序列完全一致。图2
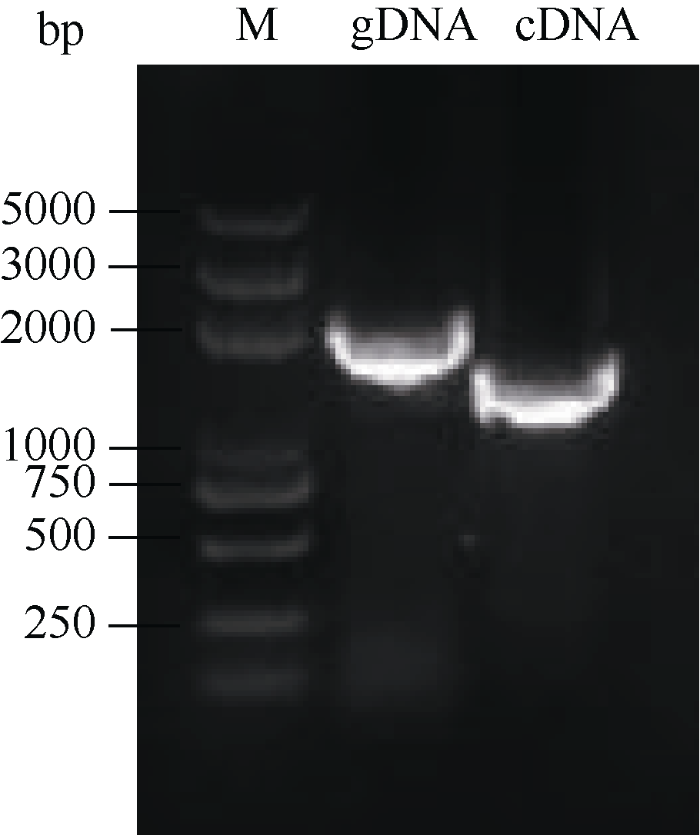 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2甘蓝BoPUB9基因cDNA与gDNA PCR扩增产物电泳图
M: DNA分子量标准。
Fig. 2Products of cDNA and gDNA PCR amplification of BoPUB9 genes in Brassica oleracea
M: marker.
2.3 BoPUB9的蛋白结构分析
序列分析表明(图3), BoPUB9基因编码含456个氨基酸残基的蛋白质, 含Ala (A)最多, 占28.9%, 不含Arg、Asn、Asp、Gln、Gly。理论等电点(pI)为5.03, 分子量为50.58224 kD; 该蛋白的不稳定指数(II)计算为42.86, 预测该蛋白属于不稳定性蛋白; 该蛋白带正电残基(Arg+Lys)总数为0, 带负电残基(Asp+Glu)总数为0, 其脂肪指数为28.87, 亲水性的平均值(GRAVY)为0.739, 预测该蛋白属于亲水性蛋白, 且在位于较靠后第1350左右氨基酸处达到最大值1.85, 位于第440氨基酸处最小值为-0.51。对蛋白质进行功能位点分析发现该蛋白不含信号肽, 说明其不是一种膜蛋白或分泌蛋白。为进一步了解BoPUB9基因编码蛋白是否有其他膜结合相关的结构域, 利用TMHMM (http://www.cbs.dtu.dk/services/ TMHMM/)在线分析预测发现该蛋白无跨膜螺旋区。利用SMART在线预测(http://smart.embl-heidelberg. de/)分析BoPUB9蛋白结构功能域, 表明BoPUB9蛋白具有1个臂重复结构域, 臂重复结构域位于284~323位氨基酸残基。另外它还具有1个U-box结构域, 位于79~142位氨基酸残基之间。通过SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/secpred_ sopma.pl)在线分析BoPUB9编码蛋白的结构发现, BoPUB9的二级结构主要由60.00%的α螺旋、6.81%的β转角、4.84%的延伸链和28.35%的无规则卷曲构成。通过NetPhos3.1在线网站分析发现BoPUB9蛋白有28个Ser、13个Thr, 2个Tyr可能成为蛋白激酶磷酸化位点。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3甘蓝BoPUB9 cDNA与其氨基酸序列
实线框为U-box结构域, 虚线框为臂重复结构域。
Fig. 3BoPUB9 cDNA and its amino acid sequence
The solid frame is the U-box domain, and the dashed frame is the repeating domain of the arm.
2.4 BoPUB9的蛋白进化分析
利用MEGA X软件将BoPUB9与其他物种氨基酸序列进行比对构建进化树发现, 甘蓝的BoPUB9与甘蓝型油菜BnPUB9亲缘关系最近, 与芜菁BrPUB9亲缘关系次之, 与AtPUB9亲缘关系最远(图4)。为更加明确各个蛋白之间的同源性, 将BoPUB9与亲缘关系较近的油菜BnPUB9、芜菁BrPUB9和萝卜RsPUB9利用DNAMAN软件进行多序列比较发现, 在这些物种之间的PUB9蛋白存在几个相对保守的片段, U-box结构域与臂重复结构域高度保守, 说明这些蛋白均具有功能一致或者相似的保守蛋白元件。图4
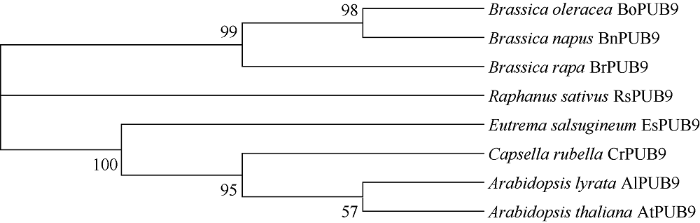 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4BoPUB9与其他物种PUB9蛋白的系统进化树
Fig. 4Phylogenetic tree of BoPUB9 and PUB9 protein from other species
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5甘蓝BoPUB9与其他物种的同源蛋白氨基酸序列比对
黑色: 氨基酸一致性为100%; 粉色: 氨基酸一致性为75%; 蓝色: 氨基酸一致性为50%。下画直线为U-box结构域, 下画虚线为臂重复区, 同时也代表该蛋白在这一区段保守性高。
Fig. 5Alignment of BoPUB9 of Brassica oleracea with homologous proteins of other species
Black: the amino acid identity is 100%; Pink: the amino acid identity is 75%; Blue: the amino acid identity is 50%. The underlined solid line denotes the U-box terminal domain, and the underlined dotted line denotes the repeating domain of the arm, indicating that the protein is highly conserved in this segment.
2.5 BoPUB9基因的表达分析
对不同授粉处理后柱头中BoPUB9基因的qRT-PCR分析(图6-A)表明, 在自花与异花授粉后该基因均呈现先上调后下调的表达趋势, BoPUB9基因在自花授粉0~30 min内快速上调表达, 15 min后为异花授粉后表达量的20多倍, 在30 min后达到差异最大, 自花授粉后的表达量为异花授粉的40多倍, 60 min后该基因的表达量均开始下降, 在0~30 min内该基因表达模式与转录组分析结果一致, 此时正是SI反应的关键时期, BoPUB9基因上调表达表明该基因可以响应甘蓝自花授粉。通过对甘蓝不同组织中的BoPUB9基因进行qRT-PCR结果分析表明, BoPUB9基因在甘蓝的不同组织中均有表达, 且它们间的表达量存在差异(图6-B), 在萼片中的表达量最高, 花瓣、雄蕊、柱头表达量次之; 在花粉花蕾中表达量相对较低。图6
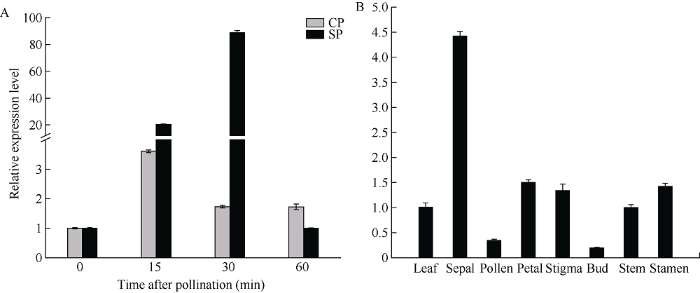 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6甘蓝BoPUB9授粉后表达分析(A)和组织表达分析(B)
SP: 自花授粉; CP: 异花授粉。
Fig. 6Relative expression patterns of BoPUB9 genes after pollination (A) and in tissues (B)
SP: self-pollination; CP: cross-pollination.
2.6 BoPUB9的启动子特异性分析
为更深入明确BoPUB9基因是否受自花授粉诱导上调, 对该基因起始密码子上游约2000 bp进行顺式作用元件分析发现该基因启动子区包括多个元件: 脱落酸响应元件、赤霉素响应元件、乙烯响应元件、生长素响应元件、光响应元件、低温响应元件、干旱响应元件和花粉特异性顺式作用元件等(表3)。表明该基因除具有授粉刺激响应外还可能通过其他信号响应多种生理刺激, 该基因也可能通过脱落酸、生长素、赤霉素响应元件以及花粉特异性作用元件参与SI反应。Table 3
表3
表3BoPUB9上游调控区顺式作用元件
Table 3
| 相关功能预测 Putative associated function | 启动子顺式作用元件 Cis-elements in the promoter region |
|---|---|
| 脱落酸应答 Abscisic acid (ABA) response | ABRE元件 ABRE |
| 赤霉素应答 Gibberellin (GA) response | GARE基序, P-box元件 GARE-motif, P-box |
| 生长素应答 Auxin (IAA) response | TGA元件 TGA-element |
| 乙烯应答 Ethylene-response | ERELEE4元件 ERELEE4 |
| 水杨酸反应 Salicylic acid (SA) response | TCA元件 TCA-element |
| 茉莉酸响应 MeJA-response | CGTCA基序, TGACG基序 CGTCA-motif, TGACG-motif |
| 花粉特异性顺式作用元件 Pollen-specific cis-acting element | POLLENLELAT52元件 POLLENLELAT52 |
| 光响应 Light response | G-Box元件, GT1基序, TCT基序 G-Box, GT1-motif, TCT-motif |
| 低温响应 Low-temperature response | LTR元件 LTR |
| 干旱诱导 Drought induced response | MBS元件 MBS |
| 压力与防御相关反应元件 Stress and defense related response elements | WBOXATNPR1元件 WBOXATNPR1 |
新窗口打开|下载CSV
为更加直观地观察该基因启动子活性, 成功构建BoPUB9-pCAMBIA1391双元表达载体后转化野生型拟南芥植株后进行潮霉素抗性筛选转基因植株(图7), 所使用潮霉素浓度为25 mg mL-1, 潮霉素与培养基配制比例为1∶1000, 利用β-葡糖醛酸糖苷酶(GUS)报告基因观察该基因表达情况。将筛选获得的T2代阳性植株的莲座期叶片、花瓣、花蕾和果荚等浸没到GUS染液中进行GUS染色。染色结果如图8所示, 在果荚中BoPUB9主要集中在顶部与基部表达, 其次果荚的果壳中也有表达(图8-a, b); 在叶片发育时期, 主要在叶脉中表达(图8-c, d); 在花中主要在萼片中高表达, 其次集中于柱头、花丝及雄蕊等部位表达, 在柱头中主要主要集中在柱头乳突细胞以下的基本组织中表达(图8-e, f)。在花蕾初现时期, 花蕾表达不明显, 随着花蕾向花朵生长过渡, 该基因高表达于最外侧的萼片中(图8-g, h)。
图7
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7BoPUB9-GUS转基因植株潮霉素筛选
Fig. 7Screening of BoPUB9-GUS transgenic plants with hygromycin
图8
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8GUS染色图
a: 果荚; c: 叶片; e: 花; g: 花蕾; b、d、f、h: 对照; 标尺为25 μm。
Fig. 8GUS staining detection
a: pods; c: leaves; e: flowers; g: buds; b, d, f, h: control (CK); Bar: 25 μm.
2.7 BoPUB9的亚细胞定位
构建BoPUB9-GFP融合表达载体, 利用PEG法成功转入拟南芥原生质体。结果发现GFP空载蛋白分布在细胞核、细胞质和细胞膜上, 而BoPUB9-GFP融合蛋白绿色荧光明显定位在细胞核与细胞质(图9), 亚细胞定位的结果表明, 该蛋白定位在细胞质与细胞核中, 属于细胞质与细胞核内的活性蛋白, 符合上述预测该蛋白质的具有亲水性特征, 正是参与甘蓝自交不亲和信号传导和生物抗逆反应所需要的。图9
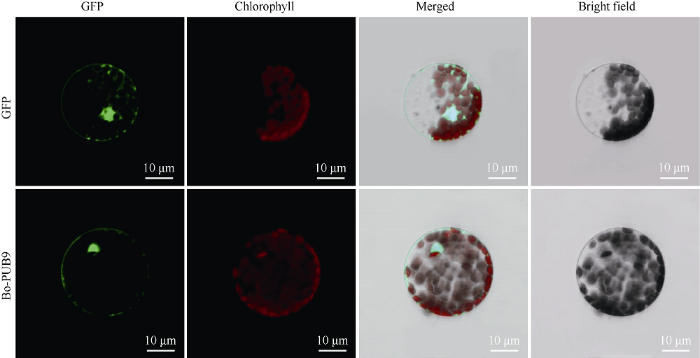 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9BoPUB9-GFP融合蛋白的亚细胞定位
标尺为10 μm。
Fig. 9Subcellular localization of BoPUB9-GFP fusion protein
Bar: 10 μm.
2.8 BoPUB9的原核表达
将pGEX-4T-1-BoPUB9重组菌株扩大培养, 并在16℃过夜诱导表达后收集菌体, 进行超声破碎, 破碎结束后离心收集上清液, 经海狸GST融合蛋白纯化磁珠纯化目的蛋白后, 用SDS-PAGE电泳检测空载与目的基因蛋白的表达如图10所示: 泳道1为诱导纯化的pGEX-4T-1空载; 泳道2为诱导纯化的pGEX-4T-1-BoPUB9; 泳道3为诱导未纯化的pGEX- 4T-1-BoPUB9; 泳道4为未诱导的pGEX-4T-1- BoPUB9。其中泳道1空载pGEX-4T-1表达蛋白大小约26 kD, 泳道2出现大小约77 kD左右的蛋白条带, BoPUB9蛋白预测约51 kD, 与空载融合后蛋白大小约77 kD, 蛋白相对分子质量与目的基因蛋白预测结果一致, 表明pGEX-4T-1-BoPUB9融合蛋白在原核细胞中成功诱导表达。图10
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图10BoPUB9 蛋白的原核表达
泳道1: 空载pGEX-4T-1; 泳道2: 诱导纯化的pGEX-4T- 1-BoPUB9; 泳道3: 诱导未纯化的的pGEX-4T-1-BoPUB9; 泳道4: 未诱导的pGEX-4T-1-BoPUB9。M: 蛋白分子量标准。
Fig. 10Prokaryotic expression profile of BoPUB9 protein
Lane 1: pGEX-4T-1; Lane 2: induced and purified pGEX-4T-1- BoPUB9; Lane 3: induced non-purified pGEX-4T-1-BoPUB9; Lane 4: non-induced pGEX-4T-1-BoPUB9. M: molecular weight standard of protein.
2.9 BoPUB9与BoSRK激酶域之间的相互作用
验证得到BoSRKj-pGBKT7重组质粒不存在自激活后(图11-a, b), 将阴性对照(pGADT7-T× pGBKT7-Lam)、阳性对照(pGADT7-T×pGBKT7-53)和重组载体组合(pGADT7-BoPUB9×pGBKT7- BoSRKj)利用PEG/LiAC法将共转化酵母感受态细胞, 涂布在DDO (SD/-Leu/-Trp/)平板上, 30℃倒置培养, 3 d后观察平板的生长情况。经PCR检测均能扩增出目的条带, 说明重组质粒同时转进酵母细胞, 根据图11-c~e初步推断BoPUB9在酵母水平上与BoSRKj之间可能存在相互作用。然后将单斑稀释后点涂于QDO (SD/-Leu/-Trp/-His/-Ade/)平板上, 倒置培养3~5 d, 发现BoPUB9×BoSRKj和阳性对照能在平板上能够生长, 并出现白色反应, 阴性对照无法正常生长(图11-f~h), 表明BoPUB9与BoSRKj之间存在相互作用。图11
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图11酵母融合株的相互作用检测
DDO/X: SD/-Leu/-Trp/X-α-Gal酵母二缺培养基; /DDO: SD/-Leu/-Trp酵母二缺培养基; QDO: SD/-Ade/-His/-Leu/-Trp酵母四缺培养基。
Fig. 11Protein-protein interactions
DDO/X: SD/-Leu/-Trp/X-α-Gal; DDO: SD/-Leu/-Trp; QDO: SD/-Ade/-His/-Leu/-Trp.
2.10 BoPUB9蛋白与BoSRKj相互作用的体外检测
根据His标签和Ni+鳌合, GST标签和GSH磁珠结合, GSH磁珠不能与His标签的特性, 通过SDS-PAGE 电泳得到结果如图12所示, 泳道1为BoPUB9-His, 泳道2为BoSRKj-GST, 泳道3为BoPUB9-His与GST 空载体共同孵育的结果, 泳道4为BoPUB9-His与BoSRKj-GST共同孵育的结果。与泳道1和泳道2纯化组相比较, 泳道3仅仅只有GST的表达产物条带, 而泳道4出现泳道1与泳道2的条带, 表明这2个蛋白能相互结合形成稳定的复合体与GSH磁珠一同被洗脱下来, 表明BoPUB9蛋白与BoSRKj蛋白发生相互作用, 被GSH磁珠结合一起沉淀下来。图12
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图12融合蛋白纯化与体外互作检测结果
泳道1: BoPUB9-His; 泳道2: BoSRKj-GST; 泳道3: BoPUB9-His×GST; 泳道4: BoPUB9-His×BoSRKj-GST。M: DNA分子量标准。
Fig. 12Detection of fusion protein purification and in vitro interaction
Lane 1: BoPUB9-His; Lane 2: BoSRKj-GST; Lane 3: BoPUB9- His×GST; Lane 4: BoPUB9-His×BoSRKj-GST. M: marker.
3 讨论
本研究基于自交不亲和甘蓝不同授粉处理后的柱头转录组数据, 成功筛选到1个在自花授粉后显著上调表达的基因BoPUB9, qRT-PCR结果表示, 该基因在自花授粉0~30 min内快速上调表达, 15 min后为异花授粉后表达量的20多倍, 在30 min后达到差异最大, 自花授粉后的表达量为异花授粉的40多倍, 该基因表达模式与转录组分析结果基本一致, 在表达量差异达到最大的时期正是SI反应的关键时期, 说明BoPUB9通过上调表达响应自交不亲和。甘蓝组织的表达量与GUS染色结果基本保持一致, 在该基因的启动子中存在与脱落酸、生长素、乙烯、高温胁迫响应及花粉特异性顺式作用, 乙烯可直接负调控白菜的自交不亲和, 花粉特异性顺式元件是在甘蓝花粉发育阶段至关重要的元件, 所以该基因可能通过植物激素调控或者影响花粉发育等防止参与自交不亲和[23]。E3泛素连接酶可以根据作用机制和特定域(例如RING、HECT、F-box和U-box)的存在进行分类, 而PUB蛋白属于含U-Box结构域的E3泛素连接酶, 在植物生命周期各个过程中都起到至关重要的作用[24,25]。在马铃薯中含U-Box结构的蛋白PHOR1在光和赤霉素(GA)的影响马铃薯块茎化[26], 在水稻中SPL11和拟南芥感官结合泛素连接酶1 (SAUL1)蛋白同属于PUB蛋白, 在植物的细胞死亡/衰老控制中具有调节功能。在水稻中SPL11通过其ARM重复序列与Rho-GAP SPIN6相互作用, 以介导细胞程序性死亡和植株先天免疫, 是水稻细胞死亡和防御的负调节剂, 但在拟南芥中最接近SPL11的同系物SAUL1却是水杨酸介导的与抑制早衰的关键新基因[27,28]。拟南芥在磷酸盐缺陷环境中, 通过上调PUB9蛋白以诱导产生更多的侧根适应环境胁迫。PUB除了受压力胁迫诱导外, 现在越来越多的研究揭示其与激酶域相结合, 导致自身磷酸化从而参与多种生理反应。鞭毛蛋白诱导拟南芥中FLS2受体复合物与2个E3泛素连接酶PUB12和PUB13结合, 当FLS2-PUB12/13结合后, BAK1磷酸化PUB12和PUB13, PUB12和PUB13泛素化FLS2并促进鞭毛蛋白诱导FLS2降解, 导致植株的免疫力下降[16]。在烟草中NtPUB4与CHRK1相互作用, 参与调节CHRK1介导的植物发育信号通路, 苜蓿PUB E3泛素连接酶在通过与激酶相互作用来控制植物与微生物的相互作用和植物自身发育[29,30]。在甘蓝中, E3泛素连接酶与激酶域相互作用最典型的例子就是ARC1参与的SI信号传导。除了参与自交不亲和, ARC1不仅靶向乙二醛酶GLO1, 该酶使糖酵解的副产物甲基乙二醛解毒, 还靶向磷脂酶D, 使其成为SI信号传导过程中被降解的底物[18,31]。Lian等[32]通过启动子融合GUS报告基因检测BoPUB7在不同组织器官中的表达模式发现BoPUB7是一个生殖发育相关基因, 能够与UBA2b相互作用, BoPUB7的表达会抑制花粉的萌发, 可能是通过与BoUBA2b的相互作用负调控花粉的萌发从而参与甘蓝SI。Shi等[33]发现BoPUB3 基因主要在甘蓝柱头中表达, 转录组数据和荧光定量 PCR 结果均显示随着自花授粉处理后表达量显著降低, 通过酵母双杂发现BoPUB3与SRK互作, 极可能参与甘蓝SI信号传导。Hu等[34]在通过基因组数据库及Tair基因组数据库共检索出103个BoPUB基因家族成员, 首次对甘蓝U-box基因家族中进行全面系统的生物信息学分析, 提出部分基因极有可能与SI反应相关。
本研究通过酵母双杂证明了BoPUB9与甘蓝自交不亲和信号传导过程中的关键因子SRK的激酶域相互作用, 可能直接通过SRK参与甘蓝SI信号传导途径。在拟南芥中AtPUB9可调节ABA相关的某些生理过程, 特别是种子发芽过程, 并且AtPUB9本身可能被ARK1受体激酶激活, 活性ARK1激酶结构域和ABA处理引起的AtPUB9定位至细胞膜[35]。PUB蛋白在激酶修饰后可能会引起亚细胞重新定位, 甘蓝型油菜SRK 910与ARC1在烟草BY-2细胞中的瞬时共表达导致ARC1从胞质和细胞核重新定位到内质网[36]。PUB蛋白的瞬时表达表明某些PUB基因的功能是与该蛋白定位于细胞内不同部位相关的, 此前大部分已被****鉴定的PUB蛋白是细胞膜上的固有蛋白, 这也与该蛋白参与膜上物质运输或者信号传递的功能相对应, 本研究表明BoPUB9蛋白定位于细胞质和细胞核, 与拟南芥中的核定位中不同, 但是与ARC1相同, 研究表明ARC1在SUMO前后分别定位于细胞质和细胞核, 所以BoPUB9蛋白很可能对甘蓝自交不亲和产生响应[37]。
BoPUB9基因在自花授粉0~30 min内快速上调表达, 在30 min后达到差异最大, 为异花授粉的40多倍, 此时正是甘蓝自交不亲和的关键时期[38], 表明该基因高度上调表达的原因是因为对于甘蓝SI信号的响应, 同时亚细胞定位也表明该蛋白可能具有核ARC1相似的功能, 酵母双杂交与poll-down实验更证明了该基因编码蛋白可直接和SRK激酶域相互作用, 可通过成为SRK下游底物参与甘蓝的自交不亲和。
4 结论
本研究通过挖掘转录组数据0~60 min自花与异花授粉的柱头转录组数据, 筛选到1个自花授粉后上调表达、异花授粉变化不明显的基因, 命名为BoPUB9, 该基因cDNA序列长度为1368 bp, gDNA序列全长1720 bp, 该基因编码蛋白具有1个臂重复结构域位于284~323位氨基酸残基, 具有1个U-box结构域位于79~142位氨基酸残基之间。BoPUB9基因在甘蓝不同组织中均有表达, 在萼片中的表达量最高, 花瓣、雄蕊、柱头中表达量次之, 在花粉、花蕾中表达量相对较低, 同GUS染色结果保持一致。BoPUB9基因在自花授粉0~30 min内相对表达量快速上调达到异花授粉的40多倍。BoPUB9蛋白在细胞核和细胞质中均有表达, BoPUB9蛋白大小约51 kD, 酵母双杂交和poll-down试验结果表明PUB9蛋白与SRK激酶域相互作用。根据以上结果推测BoPUB9可能是一种参与甘蓝自交不亲和反应过程的新基因。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
