 ,1,2, 冯文豪1, 张兴华1,2, 徐淑兔
,1,2, 冯文豪1, 张兴华1,2, 徐淑兔 ,1,2,*, 薛吉全
,1,2,*, 薛吉全 ,1,2,*
,1,2,*Dissecting the genetic architecture of maize kernel size based on genome-wide association study
QU Jian-Zhou ,1,2, FENG Wen-Hao1, ZHANG Xing-Hua1,2, XU Shu-Tu
,1,2, FENG Wen-Hao1, ZHANG Xing-Hua1,2, XU Shu-Tu ,1,2,*, XUE Ji-Quan
,1,2,*, XUE Ji-Quan ,1,2,*
,1,2,*通讯作者: *徐淑兔, E-mail:Shutuxu@nwafu.edu.cn;薛吉全, E-mail:xjq2934@163.com
收稿日期:2021-01-6接受日期:2021-04-26网络出版日期:2022-06-03
| 基金资助: |
Received:2021-01-6Accepted:2021-04-26Published online:2022-06-03
| Fund supported: |
作者简介 About authors
E-mail:qujz0220@163.com

摘要
玉米籽粒大小是产量重要构成因子之一, 也是受多基因调控的复杂数量性状, 挖掘玉米籽粒大小相关性状的关键调控基因, 将有助于提高玉米的产量。本研究以212份优良玉米自交系为材料, 于2018年和2019年分别对粒长、粒宽和粒厚进行测定, 并结合均匀分布于玉米基因组的73,006个单核苷酸多态性(single nucleotide polymorphism, SNP)标记进行全基因组关联分析。基于FarmCPU算法, 在玉米的10条染色体上检测到47个与籽粒大小相关性状关联的SNP。结合B73玉米自交系籽粒发育的动态时空转录数据, 在显著SNP标记的连锁不平衡区域内, 共检测到58个与籽粒大小相关的候选基因, 其编码的蛋白与多种蛋白存在互作关系, 参与并调控多个与籽粒发育密切相关的生物学过程。本研究为解析玉米籽粒发育的分子调控机制, 改良籽粒大小和提高作物产量提供了新的参考。
关键词:
Abstract
Kernel size related traits are one of the important compounds of yield, and they are also complex quantitative traits regulated by multiple genes. Mining the key regulatory genes of maize kernel size related traits will help to improve the yield. In this study, 212 excellent maize inbred lines were selected as materials. The kernel length, kernel width, and kernel thickness were measured in 2018 and 2019, respectively, and we performed genome-wide association study (GWAS) based on 73,006 single nucleotide polymorphic (SNP) markers uniformly distributed in maize genome. Based on the FarmCPU algorithm, 47 SNP markers associated with kernel size related traits were detected on 10 chromosomes in maize. Combined with the public dynamic spatio-temporal transcriptional data of kernel development of B73 maize inbred line, 58 candidate genes related to kernel size were detected in the linkage disequilibrium (LD) region marked by significant SNP. The proteins encoded by candidate genes interacted with multiple proteins and participated in and regulated many biological processes closely related to kernel development. These results provide a new reference for understanding the molecular regulation mechanism of maize kernel development, improving kernel size and increasing crop yield.
Keywords:
PDF (4960KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
渠建洲, 冯文豪, 张兴华, 徐淑兔, 薛吉全. 基于全基因组关联分析解析玉米籽粒大小的遗传结构. 作物学报, 2021, 48(2): 304-319 DOI:10.3724/SP.J.1006.2022.13002
QU Jian-Zhou, FENG Wen-Hao, ZHANG Xing-Hua, XU Shu-Tu, XUE Ji-Quan.
玉米(Zea mays L.)是世界上最重要的粮食作物之一, 也是世界上食品、饲料、工业产品和生物能源的重要的来源[1]。因此, 在全球耕地面积减少的情况下, 提高玉米产量是确保粮食安全的必要措施。籽粒大小, 包括粒长、粒宽和粒厚等性状, 是决定籽粒重量的关键因素, 也是最重要的产量构成因子之一[2,3]。这些构成因子均是复杂遗传性状, 具有较高的广义遗传力, 受多种生物学过程的协同调控[2,4]。因此, 阐明籽粒大小相关性状的遗传基础, 将有助于理解玉米籽粒发育的分子调控机制, 加速优化玉米增产的策略。
全基因组关联分析作为复杂数量性状遗传解析的有效工具, 为系统研究复杂性状的遗传结构提供了机会。玉米具有丰富的遗传变异和快速的连锁不平衡衰减, 是遗传学研究的重要模式作物[5]。目前, 以玉米为材料的全基因组关联分析已挖掘到了大量与农艺性状、产量、抗旱性、抗病性等复杂数量性状相关的候选基因[1]。同时, 许多研究通过全基因组关联分析和连锁分析鉴定到了多个与玉米籽粒大小相关的位点和候选基因[2,4,7-8]。例如, 前人利用11个RIL群体分别鉴定出与粒长、粒宽和粒厚相关的数量性状位点(QTL) 35、43和31个[2]; 也有研究通过连锁和关联分析相结合的方式鉴定了50个与籽粒大小相关的QTL和21个与籽粒大小相关的SNP[4]; 同时, 利用数量遗传学方法, 鉴定了137个与粒长相关的基因, 以及一个位于ECT2编码基因上的表达数量性状位点(eQTL)[8]。然而, 与水稻相比, 玉米对籽粒大小的研究仍相对较少, 到目前为止, 通过定位克隆鉴定了少数几个直接影响玉米籽粒大小的基因。如ZmCKX10, 是通过对粒长主效QTL (qKL1.07)的精细定位克隆鉴定的基因[9]。ZmGS3, 水稻GS3的同源基因, 与粒长显著相关, 并影响百粒重[10]; ZmGW2-CHR4和ZmGW2-CHR5是水稻GW2的同源基因, 均影响玉米籽粒的大小和重量, 但详尽的调控机制与功能仍不清楚[11]。因此, 当前仍亟须加强玉米籽粒大小的调控基因挖掘, 加深对玉米籽粒性状变异的分子调控机制的理解。
本研究以212份优良玉米自交系作为关联群体, 结合覆盖玉米全基因的73,006个SNP标记, 通过对玉米籽粒大小相关性状开展全基因组关联分析, 深入挖掘玉米种质资源中调控玉米籽粒大小的基因资源, 提高对玉米籽粒产量的遗传结构和分子机制的认识, 为克隆控制玉米籽粒大小的关键基因, 加速高产玉米新品种的选育提供宝贵的信息资源。
1 材料与方法
1.1 试验材料与田间设计
212份玉米自交系来源于西北农林科技大学玉米育种课题组构建的陕A群、陕B群以及国内外收集的部分自交系[12,13], 于2018年和2019年在西北农林科技大学官村玉米试验基地进行种植, 采用完全随机区组试验设计, 行长4.5 m, 行距0.6 m, 每个小区种植4行, 2次重复, 密度75,000株 hm-2, 田间管理条件与当地大田生产管理相同。1.2 性状测定与数据统计分析
所有材料均进行人工套袋自交授粉。收获后, 每个小区随机收取10个授粉良好的果穗, 并将10个果穗中部籽粒混合, 利用电子游标卡尺测定15个籽粒的长度(粒长)、宽度(粒宽)和厚度(粒厚), 并以测量值的平均值用于统计分析。其中, 2018年有表型的数据为196份玉米自交系, 2019年有表型的数据为163份玉米自交系, 2年相同的自交系为147份。利用R语言(1.3 基因型检测与质控
采用改良CTAB法[15]提取212份玉米自交系苗期叶片的基因组DNA, 利用NanoDrop 2000定量, 并用0.8%琼脂糖凝胶电泳检测DNA完整性。DNA质检合格后, 采用Affymetrix maize 6H90K芯片进行基因分型(北京康普森生物技术有限公司)。该芯片有效SNP标记为73,006个, 均匀覆盖玉米自交系B73全基因组。利用PLINK (v1.90b6.9)软件[16]对最小等位基因频率小于0.05和基因型缺失率大于0.2的SNP (single nucleotide polymorphism)标记进行过滤, 质控后保留的SNP标记用于后续全基因组关联分析。1.4 全基因组关联分析
利用Admixture (v1.3.0)进行群体遗传结构分析, GCTA软件[17]进行关联群体的主成分分析, 并通过PopLDdecay (v3.40)程序[18]计算关联群体的连锁不平衡平均衰减距离。同时, 利用GAPIT[19]工具计算关联群体的亲缘关系。由于FarmCPU[20]算法能够通过一个固定效应模型和一个随机效应模型的交替使用改善模型中的混杂问题, 显著提高统计效力和计算速度。因此, 本研究采用FarmCPU算法进行籽粒大小相关性状(包括所有年份和BLUP数据)与SNP标记的关联分析。以P值小于1/n (n为标记数)作为阈值, 筛选与籽粒大小性状显著关联的SNP标记[24]。1.5 候选基因的筛选
根据籽粒大小相关性状显著关联的SNP标记在玉米自交系B73参考基因组(ZmB73_RefGen_v3;1.6 候选基因的功能挖掘
基因表达数据来源于玉米自交系B73籽粒发育前期、中期和后期的21个时间点[21]。利用R语言中的Pheatmap包对候选基因进行表达水平聚类和可视化。利用STRING (v11.0,2 结果与分析
2.1 玉米籽粒大小性状的统计分析
不同年份籽粒大小相关性状的基本统计分析(表1)表明, 2018年关联群体的籽粒大小性状均值高于2019年, 并且关联群体中自交系的粒长(kernel length, KL)、粒宽(kernel width, KW)和粒厚(kernel thick, KT)在不同年份呈现广泛的连续变异, 变异系数为5%~10%。不同年份粒长、粒宽和粒厚的峰度和偏度的绝对值均小于1, 表明这些性状在不同年份均服从正态分布。粒长、粒宽和粒厚的广义遗传力分别为55.12%、80.76%和55.52% (表1)。不同年份关联群体粒长、粒宽和粒厚的Pearson相关分析表明, 粒厚和粒宽呈显著正相关, 粒长与粒宽和粒厚之间也存在显著的相关关系, 说明在玉米籽粒发育过程中, 粒长、粒宽和粒厚协调调控玉米籽粒大小。Table 1
表1
表1不同年份玉米籽粒大小相关性状的基本统计分析
Table 1
| 性状 Trait | 年份 Year | 平均值 Average (mm) | 标准差 SD | 变异范围 Range (mm) | 变异系数 CV | 峰度 Kurt. | 偏度 Skew. | 遗传力 H2 (%) | 相关系数 Correlation coefficient | ||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 粒长KL (mm) | 粒宽KW (mm) | 粒厚KT (mm) | |||||||||
| 粒长 KL (mm) | 2018 | 10.89 | 1.04 | 8.52-13.47 | 0.10 | -0.50 | 0.19 | 55.12 | 1.00 | 0.09 | -0.22** |
| 2019 | 8.96 | 0.73 | 6.39-11.87 | 0.10 | 0.77 | 0.04 | 1.00 | 0.31** | 0.04 | ||
| BLUP | 9.95 | 0.42 | 8.74-11.19 | 0.05 | 0.10 | 0.18 | 1.00 | 0.22** | 0.03 | ||
| 粒宽 KW (mm) | 2018 | 7.86 | 0.87 | 6.22-9.95 | 0.09 | -0.14 | 0.33 | 80.76 | 0.09 | 1.00 | 0.24** |
| 2019 | 7.45 | 0.66 | 6.13-9.32 | 0.09 | -0.13 | 0.35 | 0.31** | 1.00 | 0.43** | ||
| BLUP | 7.62 | 0.44 | 6.69-8.99 | 0.06 | -0.09 | 0.30 | 0.22** | 1.00 | 0.36** | ||
| 粒厚 KT (mm) | 2018 | 4.61 | 0.45 | 3.60-5.80 | 0.09 | -0.16 | 0.40 | 55.52 | -0.22** | 0.24** | 1.00 |
| 2019 | 4.59 | 0.47 | 3.41-5.80 | 0.10 | -0.34 | 0.37 | 0.04 | 0.43** | 1.00 | ||
| BLUP | 4.60 | 0.19 | 4.10-5.14 | 0.04 | -0.22 | 0.25 | 0.03 | 0.36** | 1.00 | ||
新窗口打开|下载CSV
2.2 玉米籽粒大小性状的全基因组关联分析
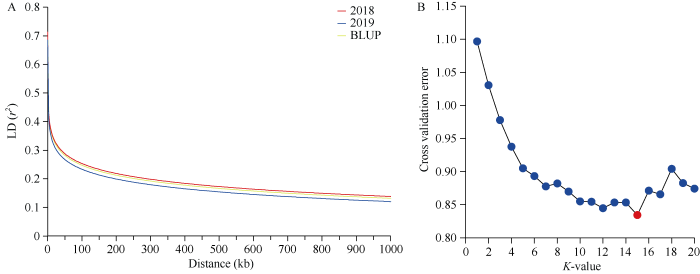
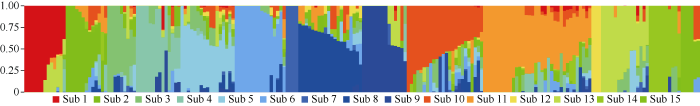
利用PLINK (v1.90b6.9)软件对212份玉米自交系的基因型进行质控, 并计算关联群体的连锁不平衡平均衰减距离(LD)。关联群体的LD分析表明, 随着物理距离的增加, r2值急剧下降, 当r2的截距设为0.2时, 不同年份关联群体的所有染色体的平均r2值约为200 kb (图1-A)。以群体的遗传距离矩阵为基础, 利用Admixture (v1.3.0)软件进行群体遗传结构分析, 可将212份玉米自交系材料分为15个亚群(图1-B和附图1)。利用PLINK (v1.90b6.9)软件, 分别对2018年196份玉米自交系、2019年163份玉米自交系和2年共同的147份玉米自交系的基因型进行质控, 分别获得高质量的SNP标记66,282、67,555和66,721个。通过GAPIT程序中FarmCPU模型, 对多样性玉米自交系连续2年的籽粒大小以及BLUP值进行了全基因组关联分析, 基于1/n (n为有效标记数)作为显著关联的SNP标记筛选阈值[24], 分别检测到与粒长、粒宽和粒厚显著关联的SNP标记14、15和18个(图2、表2和附图2-3)。其中, 与粒宽相关的Affx-291421141标记和与粒厚相关的Affx-291424033标记, 均在2018年和基于BLUP值的全基因组关联分析中被检测到(图2、表2和附图2-3)。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1群体连锁不平衡(LD)和群遗传结构分析
A: 横轴表示同一染色体上单核苷酸位点(SNP)之间的物理距离, 纵轴表示连锁不平衡参数r2值, 颜色条表示不同的关联群体。B: K = 1~20范围内的交叉验证误差值曲线图。
Fig. 1Population linkage disequilibrium (LD) and population genetic structure
The horizontal axis indicates the pairwise distance of SNPs within the same chromosome, and the vertical axis indicates the parameter r2 of LD. B: plot of the cross-validation error value in the range of K = 1 to 20.
图2
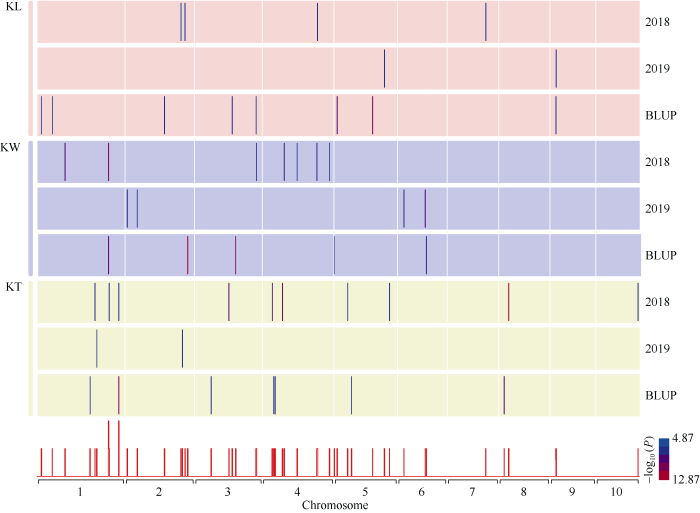 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2粒长、粒宽和粒厚显著相关的SNP分布统计
红色、蓝色和黄色背景分别代表粒长、粒宽和粒厚; 竖线表示籽粒大小性状关联到的位点在染色体上的位置; 比例尺表示关联位点的显著水平; 直方图的高度表示位点的频率。
Fig. 2Distribution of SNPs significantly correlated with kernel length (KL), kernel width (KW), and kernel thickness (KT)
Red, blue, and yellow backgrounds represent KL, KW, and KT, respectively. Vertical lines indicate the position on the chromosome of the loci associated with kernel size traits. The scale bar indicates the significance level of the association site; the height of the histogram indicates the frequency of the loci.
Table 2
表2
表2不同年份粒长、粒宽和粒厚显著相关的SNP
Table 2
| 标记名称 SNP ID | 年份 Year | 性状 Trait | 基因型 Genotype | 最小等位基因频率 MAF | 染色体 Chr. | 位置 Position | P值 P-value | 表型贡献率 R2 (%) |
|---|---|---|---|---|---|---|---|---|
| Affx-291392839 | 2018 | KL | C/T | 0.45 | 2 | 205,144,469 | 1.29E-05 | 0.36 |
| Affx-291410933 | 2018 | KL | A/C | 0.23 | 2 | 191,587,706 | 1.35E-05 | 4.52 |
| Affx-291385262 | 2018 | KL | C/T | 0.40 | 4 | 188,701,278 | 8.25E-06 | 6.96 |
| Affx-158858862 | 2018 | KL | A/G | 0.47 | 7 | 132,422,651 | 5.35E-07 | 20.90 |
| Affx-88989857 | 2019 | KL | C/A | 0.43 | 5 | 172,855,146 | 1.02E-05 | 11.64 |
| Affx-291391022 | 2019 | KL | T/C | 0.41 | 9 | 207,693,32 | 8.33E-06 | 14.53 |
| Affx-291390553 | BLUP | KL | C/T | 0.46 | 1 | 14,651,295 | 6.14E-06 | 1.24 |
| Affx-291430755 | BLUP | KL | T/G | 0.26 | 1 | 52,391,689 | 5.62E-07 | 13.31 |
| Affx-291442609 | BLUP | KL | G/A | 0.37 | 2 | 134,763,888 | 2.39E-06 | 8.31 |
| Affx-291378135 | BLUP | KL | C/T | 0.10 | 3 | 210,753,912 | 2.08E-07 | 5.97 |
| Affx-291412195 | BLUP | KL | C/T | 0.30 | 3 | 128,909,025 | 4.11E-07 | 16.92 |
| Affx-291417243 | BLUP | KL | G/T | 0.20 | 5 | 132,317,996 | 6.14E-10 | 13.73 |
| Affx-291423518 | BLUP | KL | A/G | 0.36 | 5 | 10,908,609 | 2.63E-09 | 22.90 |
| Affx-159157467 | BLUP | KL | G/A | 0.38 | 9 | 20,345,047 | 4.14E-06 | 12.08 |
| Affx-291421141 | 2018 | KW | T/C | 0.21 | 1 | 244,696,518 | 6.28E-11 | 18.69 |
| Affx-291429284 | 2018 | KW | C/A | 0.45 | 1 | 95,971,671 | 4.07E-08 | 18.38 |
| Affx-291434526 | 2018 | KW | C/T | 0.21 | 3 | 212,098,697 | 1.14E-05 | 0.62 |
| Affx-291383862 | 2018 | KW | T/C | 0.42 | 4 | 229,953,723 | 3.18E-06 | 14.42 |
| Affx-291396984 | 2018 | KW | T/C | 0.44 | 4 | 118,709,183 | 3.67E-06 | 2.38 |
| Affx-291430867 | 2018 | KW | G/A | 0.33 | 4 | 74,628,134 | 1.30E-05 | 11.78 |
| Affx-291433635 | 2018 | KW | G/A | 0.33 | 4 | 186,554,980 | 7.87E-06 | 19.65 |
| Affx-291390831 | 2019 | KW | A/G | 0.23 | 2 | 7,631,038 | 1.63E-07 | 6.09 |
| Affx-291416165 | 2019 | KW | G/A | 0.27 | 2 | 41,515,971 | 4.32E-07 | 1.50 |
| Affx-291388322 | 2019 | KW | A/G | 0.36 | 6 | 94,087,816 | 5.57E-08 | 5.08 |
| Affx-291396454 | 2019 | KW | G/A | 0.07 | 6 | 21,380,243 | 9.41E-07 | 15.80 |
| Affx-291421141 | BLUP | KW | T/C | 0.23 | 1 | 244,696,518 | 4.18E-09 | 18.51 |
| Affx-291391782 | BLUP | KW | G/A | 0.49 | 2 | 214,513,500 | 6.90E-12 | 18.84 |
| Affx-291386982 | BLUP | KW | G/A | 0.38 | 3 | 140,660,245 | 7.62E-10 | 4.90 |
| Affx-291443225 | BLUP | KW | C/T | 0.47 | 5 | 1,250,179 | 5.12E-07 | 1.73 |
| Affx-291416220 | BLUP | KW | G/A | 0.41 | 6 | 98,426,114 | 1.09E-05 | 1.67 |
| Affx-291424033 | 2018 | KT | G/A | 0.32 | 1 | 279,751,443 | 1.88E-06 | 7.04 |
| Affx-291437625 | 2018 | KT | C/T | 0.23 | 1 | 246,781,098 | 1.58E-06 | 8.62 |
| Affx-291442339 | 2018 | KT | T/G | 0.10 | 1 | 198,355,431 | 2.82E-06 | 13.41 |
| Affx-291423488 | 2018 | KT | G/A | 0.39 | 3 | 117,603,753 | 5.43E-08 | 14.05 |
| Affx-123595299 | 2018 | KT | T/C | 0.46 | 4 | 33,851,115 | 6.28E-08 | 3.06 |
| Affx-291407692 | 2018 | KT | C/T | 0.47 | 4 | 68,756,350 | 2.42E-10 | 7.53 |
| Affx-291421461 | 2018 | KT | T/G | 0.24 | 5 | 190,291,719 | 7.78E-07 | 12.80 |
| Affx-291440347 | 2018 | KT | T/C | 0.27 | 5 | 46,983,911 | 2.69E-07 | 3.51 |
| Affx-291431852 | 2018 | KT | C/T | 0.09 | 8 | 33,721,488 | 1.07E-12 | 11.05 |
| Affx-291393756 | 2018 | KT | T/C | 0.17 | 10 | 144,450,832 | 2.09E-06 | 4.11 |
| Affx-291392059 | 2019 | KT | A/T | 0.47 | 1 | 204,796,933 | 1.16E-05 | 19.87 |
| Affx-291380154 | 2019 | KT | T/C | 0.13 | 2 | 196,175,692 | 1.10E-05 | 19.15 |
| Affx-291409744 | BLUP | KT | A/G | 0.37 | 1 | 181,908,322 | 1.49E-06 | 3.75 |
| Affx-291424033 | BLUP | KT | G/A | 0.31 | 1 | 279,751,443 | 1.17E-09 | 10.20 |
| Affx-291384679 | BLUP | KT | C/A | 0.32 | 3 | 56,819,365 | 7.59E-06 | 8.77 |
| Affx-291424461 | BLUP | KT | T/C | 0.39 | 4 | 43,693,955 | 1.31E-06 | 1.38 |
| Affx-291442826 | BLUP | KT | G/A | 0.29 | 4 | 38,973,447 | 2.77E-06 | 15.91 |
| Affx-291427217 | BLUP | KT | T/C | 0.46 | 5 | 59,748,660 | 3.61E-06 | 6.66 |
| Affx-291432324 | BLUP | KT | A/G | 0.08 | 8 | 18,220,008 | 1.22E-07 | 14.41 |
新窗口打开|下载CSV
2.3 玉米籽粒大小性状的候选基因表达模式分析
以200 kb作为候选区间, 对粒长、粒宽和粒厚显著关联的SNP标记在基因组(B73参考基因组v3版本,Table 3
表3
表3玉米粒长、粒宽和粒厚的候选基因及功能注释
Table 3
| 标记名称 SNP ID | 基因 Gene | 性状 Trait | 染色体 Chr. | 位置 Position | 功能注释 Annotation | 模块 Module | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Affx-291430755 | GRMZM2G033570 | KL | 1 | 52,391,689 | ETHYLENE-INSENSITIVE3-like 1 protein | M1 | |||||||
| Affx-291410933 | GRMZM2G014313 | KL | 2 | 191,587,706 | Protein pob | M2 | |||||||
| Affx-291410933 | GRMZM2G007885 | KL | 2 | 191,587,706 | Myosin heavy chain-related protein | M3 | |||||||
| Affx-291410933 | GRMZM2G150319 | KL | 2 | 191,587,706 | O-fucosyltransferase family protein | M4 | |||||||
| Affx-291392839 | GRMZM5G821439 | KL | 2 | 205,144,469 | Chitin-inducible gibberellin-responsive protein 1 | M5 | |||||||
| Affx-291392839 | GRMZM2G046011 | KL | 2 | 205,144,469 | 60S ribosomal protein L38 | M6 | |||||||
| Affx-291392839 | GRMZM2G163233 | KL | 2 | 205,144,469 | Male sterile 32 | M7 | |||||||
| Affx-291412195 | GRMZM2G122443 | KL | 3 | 128,909,025 | OS-9-like protein | M8 | |||||||
| Affx-291412195 | GRMZM2G122481 | KL | 3 | 128,909,025 | Cytochrome c oxidase subunit 5C | M9 | |||||||
| Affx-291412195 | GRMZM2G138077 | KL | 3 | 128,909,025 | Uncharacterized protein | — | |||||||
| Affx-291378135 | AC230013.2_FG007 | KL | 3 | 210,753,912 | 60S ribosomal protein L18a | — | |||||||
| Affx-291378135 | GRMZM2G150631 | KL | 3 | 210,753,912 | Transducin/WD40 repeat-like superfamily protein | M10 | |||||||
| Affx-88989857 | GRMZM2G119517 | KL | 5 | 172,855,146 | ACT domain-containing protein ACR3 | M11 | |||||||
| Affx-291423518 | GRMZM2G145854 | KL | 5 | 10,908,609 | NADH-ubiquinone oxidoreductase subunit | M12 | |||||||
| Affx-291423518 | GRMZM2G569855 | KL | 5 | 10,908,609 | Threonine dehydratase 1 biosynthetic | M13 | |||||||
| Affx-291423518 | GRMZM2G109383 | KL | 5 | 10,908,609 | Phosphoglucomutase 2 | M14 | |||||||
| Affx-291391022 | GRMZM5G829544 | KL | 9 | 20,769,332 | Fatty acyl-ACP thioesterase 2 | M15 | |||||||
| Affx-291391022 | GRMZM2G147399 | KL | 9 | 20,769,332 | Early nodulin 93 | M16 | |||||||
| Affx-291391022 | GRMZM2G131421 | KL | 9 | 20,769,332 | Early nodulin 93 | M17 | |||||||
| Affx-291391022 | GRMZM2G404897 | KL | 9 | 20,769,332 | Zinc finger protein | M18 | |||||||
| Affx-159157467 | GRMZM2G173693 | KL | 9 | 20,345,047 | Pre-mRNA-splicing factor 38B-like | M19 | |||||||
| Affx-291416165 | GRMZM2G325131 | KW | 2 | 41,515,971 | Anthranilate synthase homolog 1 | M20 | |||||||
| Affx-291391782 | GRMZM2G049347 | KW | 2 | 214,513,500 | Synaptotagmin-5 | M21 | |||||||
| Affx-291434526 | GRMZM2G152686 | KW | 3 | 212,098,697 | Pyruvate kinase | M22 | |||||||
| Affx-291434526 | GRMZM2G070360 | KW | 3 | 212,098,697 | V-type proton ATPase subunit E-like | M23 | |||||||
| Affx-291430867 | GRMZM2G117755 | KW | 4 | 74,628,134 | Hypersensitive-induced response protein 2 | M24 | |||||||
| Affx-291383862 | GRMZM2G162992 | KW | 4 | 229,953,723 | KH domain-containing protein | M25 | |||||||
| Affx-291443225 | GRMZM2G034326 | KW | 5 | 1,250,179 | DNA-directed RNA polymerases II, IV, and V subunit 8B | M26 | |||||||
| Affx-291443225 | GRMZM2G069916 | KW | 5 | 1,250,179 | LUC7 related protein | M27 | |||||||
| Affx-291443225 | GRMZM2G138676 | KW | 5 | 1,250,179 | Shaggy-related protein kinase kappa | M28 | |||||||
| Affx-291396454 | GRMZM2G060856 | KW | 6 | 21,380,243 | Membrane protein | M29 | |||||||
| Affx-291442339 | GRMZM2G318475 | KT | 1 | 198,355,431 | Eukaryotic translation initiation factor 6 | M30 | |||||||
| Affx-291442339 | GRMZM2G017110 | KT | 1 | 198,355,431 | Glutamate decarboxylase | M31 | |||||||
| Affx-291437625 | GRMZM2G012119 | KT | 1 | 246,781,098 | Post-illumination chlorophyll fluorescence increase | M32 | |||||||
| Affx-291437625 | GRMZM2G013600 | KT | 1 | 246,781,098 | DNA-directed RNA polymerases I, II, and III polypeptide | M33 | |||||||
| Affx-291437625 | GRMZM2G146819 | KT | 1 | 246,781,098 | Uncharacterized protein | M34 | |||||||
| Affx-291437625 | GRMZM2G146818 | KT | 1 | 246,781,098 | 40S ribosomal protein | — | |||||||
| Affx-291437625 | GRMZM2G146589 | KT | 1 | 246,781,098 | Lysine-tRNA ligase | M35 | |||||||
| Affx-291437625 | GRMZM2G146486 | KT | 1 | 246,781,098 | P-loop containing nucleoside triphosphate hydrolase Superfamily protein | M36 | |||||||
| Affx-291437625 | GRMZM2G034417 | KT | 1 | 246,781,098 | Inositol-phosphate phosphatase | M37 | |||||||
| Affx-291424033 | GRMZM2G047456 | KT | 1 | 279,751,443 | Peroxidase 35 | M38 | |||||||
| Affx-291424033 | GRMZM2G307252 | KT | 1 | 279,751,443 | SecY protein transport family protein | M39 | |||||||
| Affx-291392059 | GRMZM2G357804 | KT | 1 | 204,796,933 | UDP-N-acetylglucosamine-peptide N-acetylglucosaminyltransferase SPINDLY | M40 | |||||||
| Affx-291392059 | GRMZM2G057608 | KT | 1 | 204,796,933 | 40S ribosomal protein S25-1 | M41 | |||||||
| Affx-291392059 | GRMZM2G167548 | KT | 1 | 204,796,933 | Multiple myeloma tumor-associated protein 2 | M42 | |||||||
| Affx-291392059 | GRMZM2G167836 | KT | 1 | 204,796,933 | Vesicle-associated membrane protein 727 | M43 | |||||||
| Affx-291380154 | GRMZM2G073942 | KT | 2 | 196,175,692 | Uncharacterized protein | — | |||||||
| Affx-291423488 | GRMZM2G138268 | KT | 3 | 117,603,753 | Auxin-responsive protein IAA 10 | M44 | |||||||
| Affx-123595299 | GRMZM2G139550 | KT | 4 | 33,851,115 | Aldolase superfamily protein | M45 | |||||||
| Affx-123595299 | GRMZM5G870572 | KT | 4 | 33,851,115 | BSD domain containing protein | M46 | |||||||
| Affx-291442826 | GRMZM2G064807 | KT | 4 | 38,973,447 | Pentatricopeptide repeat-containing protein | M47 | |||||||
| Affx-291442826 | GRMZM2G158153 | KT | 4 | 38,973,447 | KH domain-containing protein | M48 | |||||||
| Affx-291421461 | GRMZM2G004847 | KT | 5 | 190,291,719 | Pectin lyase-like superfamily protein | M49 | |||||||
| Affx-291427217 | GRMZM2G102447 | KT | 5 | 59,748,660 | WAS/WASL-interacting protein family member 1-like | M50 | |||||||
| Affx-291431852 | GRMZM2G180990 | KT | 8 | 33,721,488 | Ligatin | M51 | |||||||
| Affx-291432324 | GRMZM2G107696 | KT | 8 | 18,220,008 | Ubiquitin carboxyl-terminal hydrolase | M52 | |||||||
| Affx-291432324 | GRMZM2G107639 | KT | 8 | 18,220,008 | Transaminase | M53 | |||||||
| Affx-291393756 | GRMZM2G086030 | KT | 10 | 144,450,832 | Tetratricopeptide repeat (TPR)-like superfamily protein | M54 | |||||||
新窗口打开|下载CSV
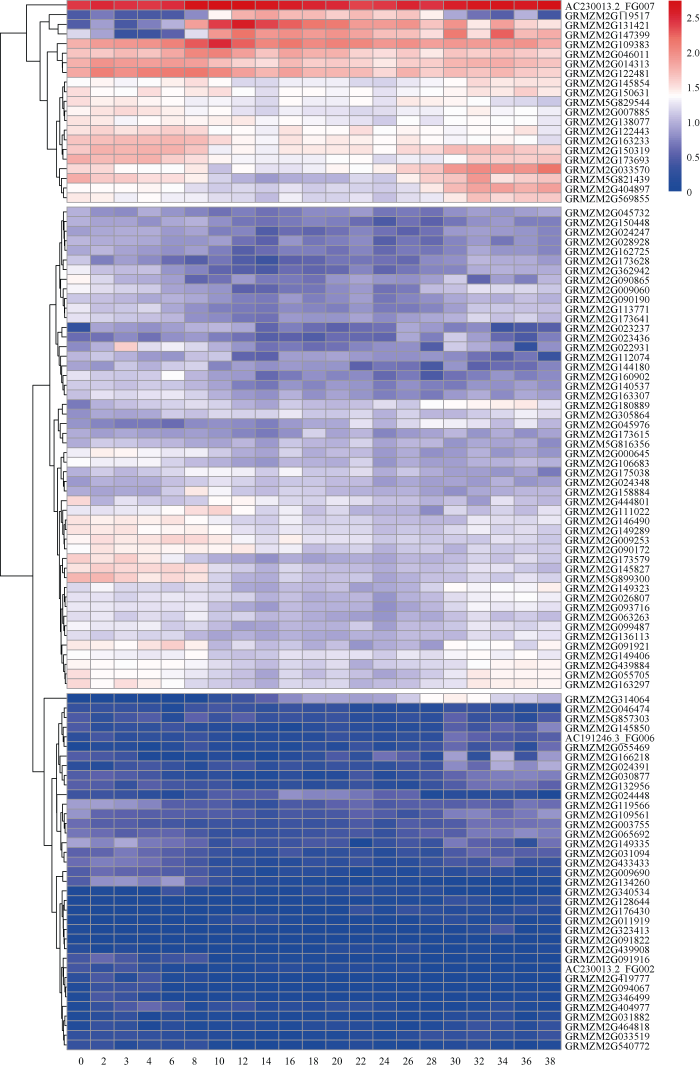
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3玉米粒长相关候选基因的动态表达模式
比例尺表示标准化的基因表达水平。
Fig. 3Dynamic expression patterns of candidate genes related to maize kernel length
The scale bar indicates normalized gene expression levels.
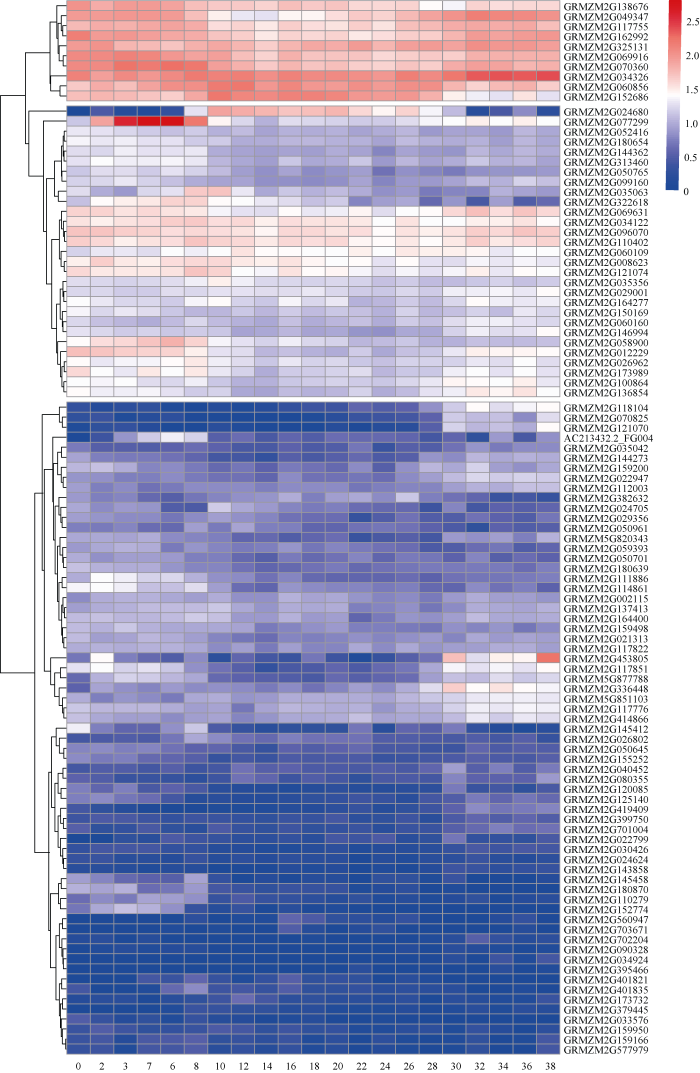
图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4玉米粒宽相关候选基因的动态表达模式
比例尺表示标准化的基因表达水平。
Fig. 4Dynamic expression patterns of candidate genes related to maize kernel width
The scale bar indicates normalized gene expression levels.
图5
 新窗口打开|下载原图ZIP|生成PPT
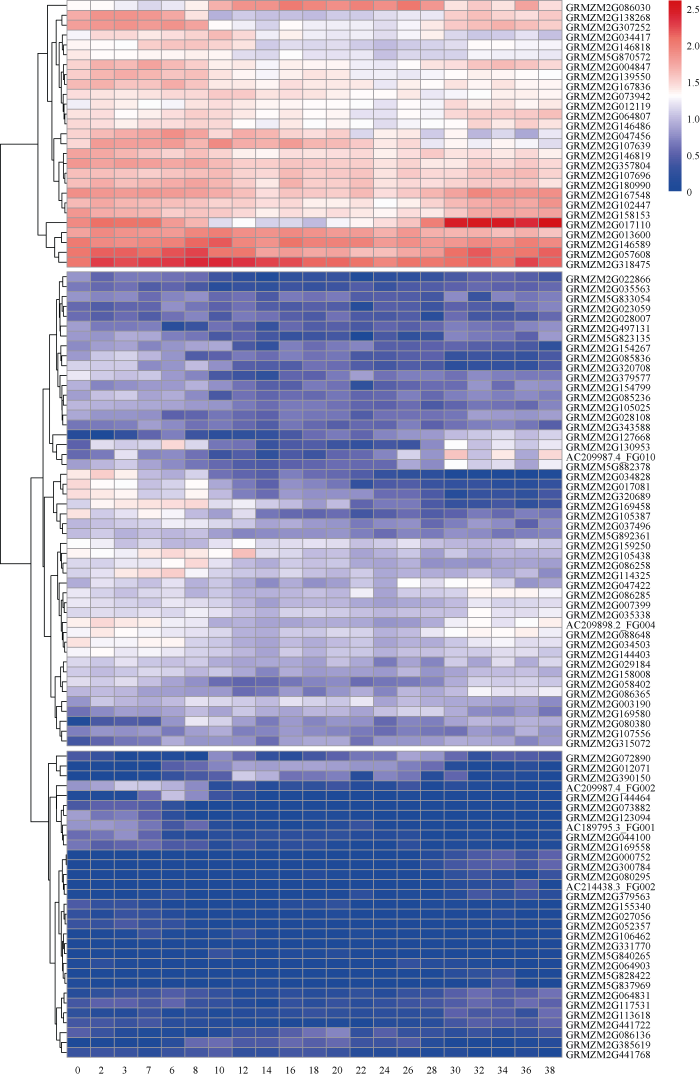
新窗口打开|下载原图ZIP|生成PPT图5玉米粒厚相关候选基因的动态表达模式
比例尺表示标准化的基因表达水平。
Fig. 5Dynamic expression patterns of candidate genes related to maize kernel thickness
The scale bar indicates normalized gene expression levels.
2.4 候选基因调控网络对玉米籽粒发育的响应分析
利用STRING (v11.0)数据库对58个与籽粒大小相关的候选基因的编码蛋白互作网络进行分析, 结果表明其编码蛋白参与多个生物学过程级联网络的调控(图6)。除候选基因GRMZM2G138077、AC 230013.2_ FG007、GRMZM2G146818和GRMZM2G073942外, 其他54个候选基因编码蛋白, 均与多种功能蛋白存在互作关系。互作蛋白的基因本体富集(GO)分析表明, 以候选基因编码蛋白为核心的互作蛋白, 可能通过参与生长速率的正向调控、细胞氨基酸生物合成、糖酵解过程、二糖代谢过程、组织形态发生、表皮发育等生物学过程调控玉米籽粒的发育(图7-A)。同时, 部分候选基因编码蛋白之间, 可能通过多个候选基因编码蛋白构成的互作网络调控玉米籽粒发育的多种代谢过程。例如, 以候选基因GRMZM2G046011 (M6)、GRMZM2G122443 (M8)、GRMZM2G117755 (M24)、GRMZM2G034326 (M26)、GRMZM2G060856 (M29)、GRMZM2G013600 (M33)、GRMZM2G307252 (M39)、GRMZM2G057608 (M41)、GRMZM2G064807 (M47)和GRMZM2G180990 (M51)编码蛋白为核心的蛋白互作网络主要参与了籽粒发育过程中转录延伸、翻译、蛋白质的靶向输送等生物学过程(图7-B); 以候选基因GRMZM2G119517 (M11)、GRMZM2G404897 (M18)、GRMZM2G017110 (M31)、GRMZM2G146486 (M36)、GRMZM2G004847 (M49)和GRMZM2G107639 (M53)编码蛋白为核心的蛋白互作网络, 在氧化还原过程、细胞氮化物分解过程、胺生物合成的过程等籽粒发育过程中发挥重要的作用(图7-B)。图6
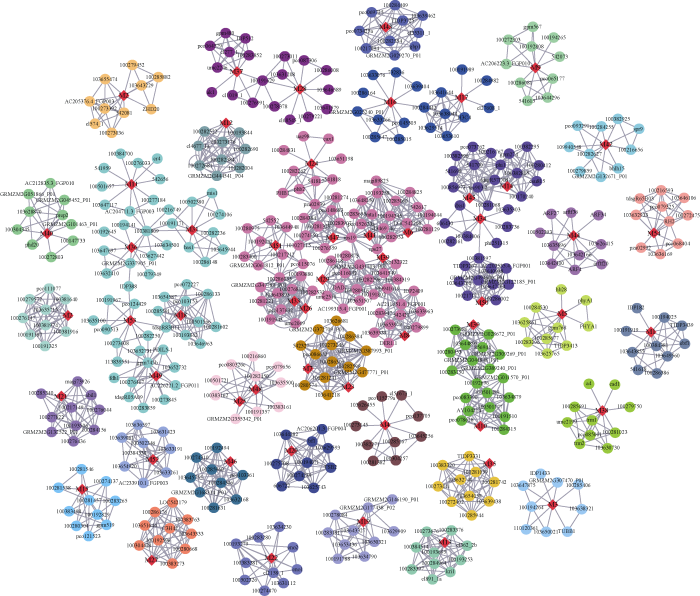 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图654个高表达的候选基因编码蛋白互作网络
节点表示蛋白, 连线表示蛋白之间的互作关系。红色表示候选基因, 其他颜色表示候选基因编码蛋白的互作网络。
Fig. 6Protein-protein interaction networks of 54 highly expressed candidate genes
The node indicates the proteins, and the line indicates the interaction between proteins. Red color indicates candidate gene encoded proteins, and the interactive proteins in different networks are distinguished by different colors.
图7
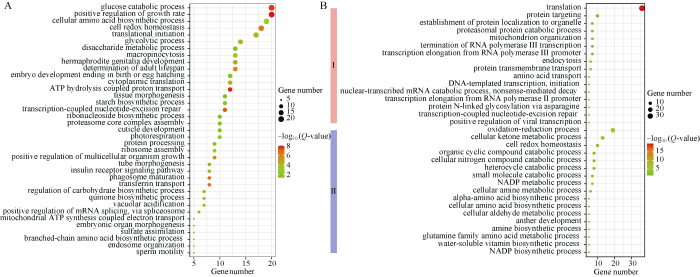 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7候选基因编码蛋白互作网络的功能富集分析
圆圈大小表示基因富集数目, 标准化的Q值表示富集的显著程度。A代表M6、M8、M24、M26、M29、M33、M39、M41、M47和M51中蛋白显著富集的生物学过程; B代表M11、M18、M31、M36、M49和M53中蛋白显著富集的生物学过程。
Fig. 7Functional enrichment analysis of protein-protein interaction networks for candidate genes
Circle size indicates the number of genes, the normalized Q-value indicates the significance of enrichment. The biological process of significant protein enrichment in M6, M8, M24, M26, M29, M33, M39, M41, M47, and M51 is in Fig. A; the biological process of significant protein enrichment in M11, M18, M31, M36, M49, and M53 is in Fig. B.
3 讨论
3.1 不同关联群体有利于籽粒大小相关性状关联位点的发掘
玉米籽粒产量是一个复杂性状的集合体, 其中籽粒大小是与产量最直接相关的性状之一。本研究通过对212份玉米自交系组成的关联群体的籽粒大小性状测定, 发现2018年关联群体籽粒大小性状的均值均高于2019年的均值, 并且籽粒大小性状之间的相关性上也存在差异, 但籽粒大小性状均具有较高的广义遗传力。由此可以推测2018年和2019年籽粒大小的性状差异和性状间的相关程度变化与关联群体的大小和环境因子有关, Liu等[4]的研究也进一步验证了这一推测。因此, 利用不同的群体在不同的环境开展籽粒大小性状的研究, 将有助于玉米籽粒大小性状的遗传构型解析。本研究采用212份玉米自交系组成的关联群体进行全基因组关联分析, 在2018年和2019年共检测到14个与粒长显著关联的SNP、15个与粒宽显著关联的SNP标记和18个与粒厚显著关联的SNP标记。其中, 部分SNP标记与先前通过多群体连锁定位或关联分析检测到的QTL相一致。例如, 1号染色体上定位的与玉米粒厚关联的标记Affx- 291424033位于Li等[2]利用11个RIL群体定位的区间npi282b-lim39内, 2号染色体上定位的与玉米粒宽关联的标记Affx-291391782位于Liu等[25]利用F2群体定位的区间bnlg1316-umc1464内; 与玉米粒厚关联的标记Affx-291407692和与玉米粒宽关联的标记Affx-291396984位于Li等[2]利用11个RIL群体定位的区间ntf2-umc1346内; 与玉米粒宽关联的标记Affx-291433635位于Liu等[25]利用F2群体定位的区间umc1667-umc2041内; 5号染色体上定位的与玉米粒长关联的标记Affx-291423518位于Raihan等[26]利用RIL群体定位的区间PZE-105017975-PZE- 105023645内; 与玉米粒厚关联的标记Affx- 291440347和Affx-291427217位于Li等[2]利用11个RIL群体定位的区间ras11E3-umc1355内, 其中Affx-291427217也位于Liu等[25]利用F2群体定位的区间umc1784-umc1747内; 9号染色体上定位的与玉米粒长关联的标记Affx-159157467和Affx-291391022位于Liu等[24]利用F2群体定位的区间umc1893- umc1634内, 这些结果进一步证实了本研究结果的可靠性。
此外, 本研究还检测到了37个与玉米籽粒大小相关的新位点, 这可能与玉米籽粒大小是由微效多基因控制有关。同时, 传统定位方法受遗传背景和分析方法的限制, 不能全面地定位这些微效基因, 而利用新的群体开展籽粒大小的全基因组关联分析, 可为解析玉米籽粒大小的遗传结构提供了新的参考。
3.2 多种代谢途径协同调控玉米籽粒大小
本研究根据与籽粒大小相关性状显著关联的SNP标记的物理位置及染色体上LD衰减距离, 从47个关联位点共检测到474个潜在的候选基因, 结合玉米自交系B73籽粒发育动态的转录组数据, 发掘到了58个与籽粒大小相关性状关联的候选基因, 包含5个转录因子和53个蛋白编码基因。GRMZM2G033570 (EIL1), 是一个与粒长关联的候选基因。该基因作为主要的调控因子, 在光形态建成[27]、根系发育[28]、生长素生物合成和转运[29]、细胞分裂素信号反应[30]和水杨酸合成[31]中发挥着重要的作用。GRMZM5G821439 (GRAS79)是一个与粒长关联的候选基因, 其家族基因编码蛋白在赤霉素信号转导[32]、光敏色素A信号转导[33]、腋生分生组织初始化[34]和芽分生组织维持[35]发挥着重要的作用。GRMZM2G163233 (bHLH66), 是一个与粒长关联的候选基因, 该基因在花药发育过程中起着重要的分裂和分化调节作用[36]。GRMZM2G138268 (IAA10)是一个与粒厚关联的候选基因, 通过作用于胚柄细胞介导胚根原的分化, 并阻止向胚发育[37]。GRMZM5G870572 (BSD), 是一个与粒厚关联的候选基因, 该基因在调控果实品质、营养生长和植物叶片的衰老方面发挥着重要的作用。因此, 我们推测这5个高表达的候选基因, 可能主要是通过调控植物内源激素以及细胞的分裂和分化进一步影响籽粒的大小。
此外, 其他53个高表达的蛋白编码基因, 也可能通过调控不同代谢途径影响籽粒的大小。例如, GRMZM2G150319编码一个岩藻糖基转移酶, 在调控细胞粘附、花粉管穿越雌蕊[38]和生物钟方面[39]发挥着重要的作用。GRMZM2G122443编码一个胞质内质网膜相关蛋白OS-9, 该蛋白通过降解错误折叠的受体激酶维持细胞蛋白质稳态[40]。GRMZM2G150631编码一个转导蛋白, 该蛋白含有一个由多个串联复制单元组成的WD基序, 并在信号转导、蛋白质运输、染色质修饰、细胞分裂和胞质分裂、细胞凋亡、细胞移动、花发育、分生组织发育等植物发育过程中发挥着重要的作用[41]。GRMZM2G109383编码一个葡萄糖磷酸变位酶, 催化葡萄糖6-磷酸(G6P)和葡萄糖1-磷酸的可逆转化反应, 影响配子体的发育、淀粉合成、植株的生长和种子的重量[42]。GRMZM5G829544编码一个脂肪酰基ACP硫酯酶, 影响脂肪酸的延伸循环, 释放游离脂肪酸, 并且诱导脂肪酸的降解[43]。GRMZM2G138676编码一个SHAGGY蛋白激酶, 参与油菜素内酯的信号转导[44], 并调控细胞的分裂和细胞周期[45]。GRMZM2G307252作为2年均检测到的候选基因, 其编码一个SecY蛋白, 该蛋白影响多种蛋白跨内质网膜转运, 并且影响拟南芥胚的正常发育[46]。在玉米中调控籽粒发育的详尽机制仍有待进一步研究。这些候选基因信息将为克隆玉米产量性状相关候选基因提供宝贵的信息资源。
4 结论
本研究对212个玉米自交系籽粒大小相关性状进行全基因组关联分析, 共检测到38个与玉米籽粒大小相关性状显著关联的SNP标记, 结合籽粒发育的动态转录组数据, 在显著SNP标记LD区域内发掘出58个与玉米籽粒大小相关的候选基因, 这些基因在调控细胞分裂和分化以及籽粒贮藏物质积累等多种生物学过程中发挥着重要作用。本研究为解析玉米籽粒大小的分子调控机制和分子标记辅助玉米籽粒性状的改良提供了新的参考。附图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT附图1群体遗传结构分析
基于k = 15的群体结构, 不同颜色片段的长度表示该个体基因组中某个亲本所占的比例。
Fig. S1Population genetic structure analysis
Population structure based on k = 15, the length of the different color segments represents the proportion of an ancestor in the individual genome.
附图2
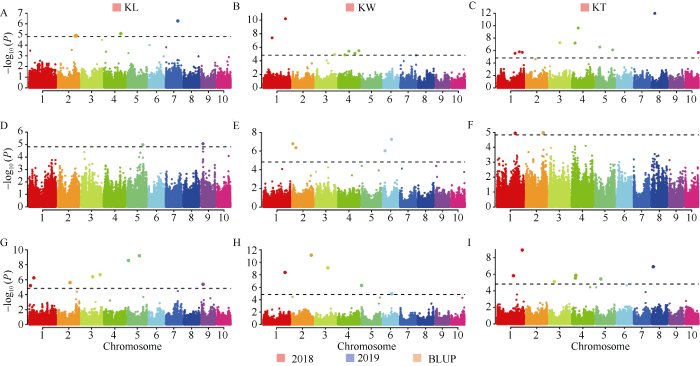 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT附图2全基因组关联分析曼哈顿图
A~C: 2018年的粒长、粒宽和粒厚; D~F: 2019年的粒长、粒宽和粒厚; G~I: 2年的粒长、粒宽和粒厚的BLUP值。黑色水平线代表全基因组关联分析的显著阈值。
Fig. S2Manhattan plot of genome-wide association study
A-C: KL, KW, and KT of 2018; D-F: KL, KW and KT of 2019; G-I: BLUP value of KL, KW, and KT for 2 years. Black horizontal line indicates the genome-wide significance threshold.
附图3
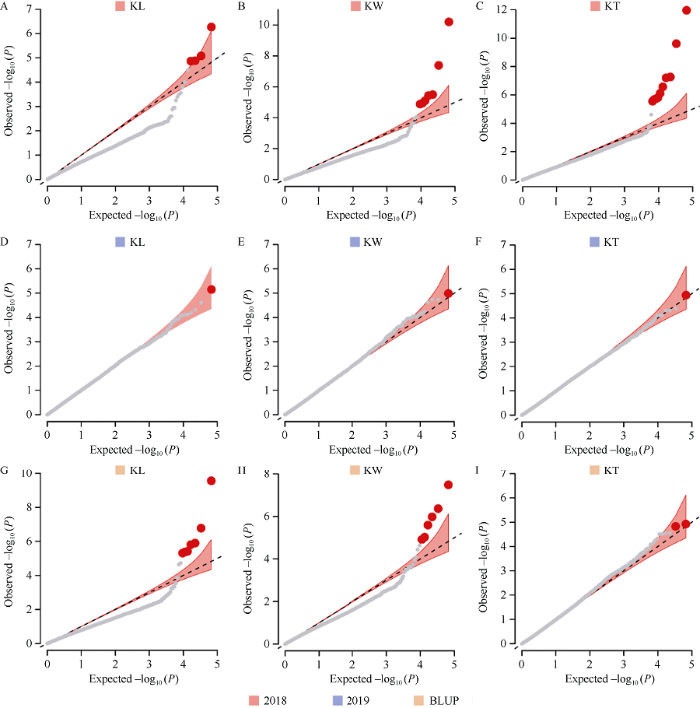 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT附图3全基因组关联分析QQ图
A~C: 2018年的粒长、粒宽和粒厚; D~F: 2019年的粒长、粒宽和粒厚; G~I: 2年的粒长、粒宽和粒厚的BLUP值。横轴表示标准化的期望P值, 纵轴表示标准化的观察到的P值。
Fig. S3Quantitle-quantitle plot of genome-wide association study
A-C: KL, KW, and KT of 2018; D-F: KL, KW and KT of 2019; G-I: BLUP value of KL, KW, and KT for 2 years. The horizontal axis shows normalized expected P-values, and the vertical axis shows normalized observed P-values.
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 7]
[本文引用: 1]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
