 ,1, 于美琴2, 张晓娇1, 王露露1, 张志勇1, 马新明1, 李会强2, 王小纯
,1, 于美琴2, 张晓娇1, 王露露1, 张志勇1, 马新明1, 李会强2, 王小纯 ,1,2,*
,1,2,*Alternative splicing analysis of wheat glutamine synthase genes
WEI Yi-Hao ,1, YU Mei-Qin2, ZHANG Xiao-Jiao1, WANG Lu-Lu1, ZHANG Zhi-Yong1, MA Xin-Ming1, LI Hui-Qing2, WANG Xiao-Chun
,1, YU Mei-Qin2, ZHANG Xiao-Jiao1, WANG Lu-Lu1, ZHANG Zhi-Yong1, MA Xin-Ming1, LI Hui-Qing2, WANG Xiao-Chun ,1,2,*
,1,2,*通讯作者: *王小纯, E-mail:xiaochun.w@163.com
收稿日期:2020-12-29接受日期:2021-04-14网络出版日期:2021-06-28
| 基金资助: |
Corresponding authors: *E-mail:xiaochun.w@163.com
Received:2020-12-29Accepted:2021-04-14Published online:2021-06-28
| Fund supported: |
作者简介 About authors
E-mail:yc_yihao@163.com

摘要
谷氨酰胺合成酶(glutamine synthetase, GS)是植物氮素同化的关键酶, 小麦(Triticum aestivum L.) GS由12个核基因编码, 即TaGS1;1-6A/6B/6D、TaGS1;2-4A/4B/4D、TaGS1;3-4A/4B/4D和TaGS2-2A/2B/2D。利用单分子测序技术获得了TaGS基因的全长转录本, 发现TaGS1;1-6A有1种可变剪接转录本、TaGS1;1-6B有2种可变剪接形式; 与正常转录本编码的TaGS相比, TaGS1;1-6A-1的Gln_synt_N结构域部分缺失, TaGS1;1-6B-3缺少Gln_synth_ gly_rich_site结构域; TaGS1;1-6B-4是新鉴定的转录本, 编码缺少Gln_synth_gly_rich_site和Gln_synth_cat_dom结构域的GS蛋白。进一步研究了氮肥对不同氮效率小麦品种可变剪接转录本表达的影响, 发现氮高效品种YM49叶片TaGS1;1-6A-1表达水平显著高于氮低效品种XN509, 且随着氮肥增加而升高; YM49根系TaGS1;1-6A-1表达随供氮量增加而升高, XN509却呈现相反趋势。TaGS1;1-6B-3在YM49和XN509中有相似的表达趋势, 高浓度NO3-促进TaGS1;1-6B-3在叶片中表达, 却抑制其在根中的表达; 高浓度NH4+抑制TaGS1;1-6B-3在叶片中表达, 但在根中表达影响较小。TaGS1;1-6B-4主要在低氮条件下表达, NH4+浓度不影响其在YM49根中的表达。了解TaGS基因可变剪接有助于阐明TaGS同工酶在小麦氮代谢中的功能。
关键词:
Abstract
As a key enzyme for nitrogen assimilation in plant, glutamine synthetase (TaGS) is encoded by 12 genes in common wheat (Triticum aestivum L.), namely TaGS1;1-6A/6B/6D, TaGS1;2-4A/4B/4D, TaGS1;3-4A/4B/4D, and TaGS2-2A/2B/2D. Transcripts of TaGS genes were obtained using the single molecular sequencing technology, and the results showed that TaGS1;1-6A had one alternative splicing (AS) version of transcripts and TaGS1;1-6B had two AS versions. Compared with the TaGS encoded by normal transcript, TaGS1;1-6A-1 lacked part of the Gln_synt_N domain, and TaGS1;1-6B-3 lacked the Gln_synth_gly_rich_ site domain; TaGS1;1-6B-4 was a new AS event, and encoded a truncated GS without Gln_synth_gly_rich_site and Gln_synth_ cat_dom domain. The relative expression level of TaGS alternative splicing transcripts of wheat cultivars with different nitrogen use efficiencies showed that the relative expression level of TaGS1;1-6A-1 in leaves of YM49 (N-efficient) was significantly higher than that of XN509 (N-inefficient), and increased with the increase of nitrogen supply. In roots, the relative expression of TaGS1;1-6A-1 increased with the increase of nitrogen supply in YM49, but decreased in XN509. TaGS1;1-6B-3 showed a similar expression trend in YM49 and XN509, and high concentration of NO3- promoted the expression of TaGS1;1-6B-3 in leaves while inhibited in roots. High concentration of NH4+ inhibited the expression of TaGS1;1-6B-3 in leaves, but had little effect in roots. TaGS1;1-6B-4 was mainly expressed under low nitrogen condition, and its expression in roots of YM49 was not affected by NH4+ supply. Understanding the AS transcript versions of TaGS genes is helpful to illustrate the functions of TaGS isozymes in nitrogen metabolism of wheat.
Keywords:
PDF (537KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
韦一昊, 于美琴, 张晓娇, 王露露, 张志勇, 马新明, 李会强, 王小纯. 小麦谷氨酰胺合成酶基因可变剪接分析. 作物学报, 2022, 48(1): 40-47 DOI:10.3724/SP.J.1006.2022.01103
WEI Yi-Hao, YU Mei-Qin, ZHANG Xiao-Jiao, WANG Lu-Lu, ZHANG Zhi-Yong, MA Xin-Ming, LI Hui-Qing, WANG Xiao-Chun.
谷氨酰胺合成酶(GS)是植物将NH4+同化为有机氮的关键酶, 催化ATP依赖的NH4+和谷氨酸(Glu)合成谷氨酰胺(Gln)[1]。依据亚细胞定位, 植物GS分为胞质型(GS1)和质体型(GS2)两类。GS1一般由3~5个核基因编码, 主要参与根系吸收NH4+同化以及叶片衰老过程中氮素再利用; GS2由1个核基因编码, 主要参与NO3-还原NH4+及光呼吸释放NH4+的同化[2]。小麦有TaGS1;1 (TaGS1)、TaGS1;2 (TaGSr)和TaGS1;3 (TaGSe)等3种胞质型GS, 1种质体型GS, 即TaGS2[3]。小麦是异源6倍体, 含A、B、D三套染色体, 每一种TaGS基因在A、B、D染色体上都有分布, 因此, TaGS同工酶由12个核基因编码, 数目远多于拟南芥、玉米、水稻等植物[4], TaGS基因可能具有更加复杂的表达调控模式。
可变剪接(alternative splicing, AS)是真核生物一种重要的mRNA转录后加工机制, 通过mRNA的可变剪接, 一个基因能产生多个转录本, 从而增加了蛋白质的多样性[5,6,7,8]。可变剪接也是植物基因表达调控的重要方式, 在生理代谢、信号转导以及生物和非生物胁迫响应中发挥作用[9,10,11,12]。可变剪接包括外显子跳跃、内含子保留、可变的5′端位点、可变的3′端位点、互斥外显子等5种主要方式, 其中内含子保留是植物最常见的剪接方式[13,14]。
随着第2代测序技术的快速推广, 许多植物基因组和转录组测序已经完成, 但2代测序技术无法提供足量的全长转录本, 一些重要的可变剪接信息可能丢失[15]。基于单分子测序的第3代测序技术可以获得更长乃至全长转录本序列, 为基因的可变剪接研究提供了技术支撑[16]。不同染色体编码的同一类TaGS基因, 如TaGS1;1-6A/6B/6D, 其启动子序列不尽相同, 表明它们具有不同的表达特性和功能[17]。全面了解小麦12个TaGS基因的转录概况, 是研究其功能的前提。为此, 本研究利用单分子测序技术对小麦转录组进行了序列测定, 通过分析TaGS基因全长转录本, 不仅了解了小麦12个TaGS基因的转录概况, 还发现了其可变剪接形式, 并发现氮处理影响可变剪接在不同氮效率小麦中的表达, 为进一步探究TaGS同工酶在小麦氮代谢中的功能奠定了基础。
1 材料与方法
1.1 试验材料
试验所用材料为氮高效品种豫麦49-198 (YM49)及氮低效品种西农509 (XN509)。用75% (v/v)乙醇将大小均匀一致的小麦种子浸泡1 min, 用蒸馏水清洗3次, 在湿润滤纸上萌发。待根长达到1 cm后, 将植株悬浮到含0.5 L Hoagland营养液杯子中, 每个杯子种10株小麦, 每3 d更换一次营养液。Hoagland营养液以NH4+或NO3-为唯一氮源, 氮浓度分别设置为0、0.2、2、5、10、20 mmol L-1。在光周期16 h/8 h、光照强度300 μmol photons m-2 s-1、温度(22 ± 2)℃、湿度50%~70%的光照培养箱中将小麦培养至三叶一心后, 分别取其根系和叶片, 液氮速冻后于-80℃保存。大田试验在河南农业大学许昌实验农场进行, 上季作物为玉米。基础肥力为有机质1.21%, 全氮1.5 mg g-1, 全磷0.7 mg g-1, 全钾1.5 mg g-1, 有效磷6.4 mg kg-1, 速效钾201 mg kg-1。每公顷施P2O5 120 kg、K2O 120 kg, 作基肥一次施入; 每公顷施纯氮225 kg, 60%作基肥, 40%在拔节期结合浇水施入。豫麦49-198于10月16日播种, 其他管理同大田。在开花期、花后8、16和24 d分别取旗叶、穗下节、穗轴和籽粒, 清洗剪碎后于液氮中速冻, -80℃保存。
1.2 转录组测序
以YM49为材料进行单分子全长转录组测序分析。取-80℃保存的不同处理不同生育时期不同组织样品各3份, 送百迈客生物科技公司, 分别提取各样品RNA, 等质量混合后测序。采用新一代基于纳米孔的单分子实时电信号测序技术Oxford Nanopore Technologies (ONT)平台测序, 即第3代测序技术。1.3 TaGS基因的染色体定位
利用TBtools软件, 从中国春小麦基因组数据库(IWGSC RefSeq v2.0)中获得TaGS基因的染色体位置信息, 并对其进行可视化。1.4 TaGS基因转录本的提取与分析
从第3代转录组测序结果中筛选注释为“glutamine synthetase”的转录本, 对所有转录本进行序列比对, 确定“CACTACAAGGC”为其共有序列。以“CACTACAAGGC”序列为筛选条件, 从clean data文件中提取出TaGS基因全长转录本序列。Table 1
表1
表1不同氮处理营养液组成
Table 1
| 氮含量 Nitrogen content | KH2PO4 | MgSO4 | KCl | CaCl2 | Ca(NO3-)2 | NH4Cl |
|---|---|---|---|---|---|---|
| N0 | 0.2 | 1 | 1.5 | 2.5 | 0 | 0 |
| 0.2 mmol L-1 NO3- | 0.2 | 1 | 1.5 | 2.4 | 0.1 | 0 |
| 2 mmol L-1 NO3- | 0.2 | 1 | 1.5 | 1.5 | 1 | 0 |
| 5 mmol L-1 NO3- | 0.2 | 1 | 1.5 | 0 | 2.5 | 0 |
| 10 mmol L-1 NO3- | 0.2 | 1 | 1.5 | 0 | 5 | 0 |
| 20 mmol L-1 NO3- | 0.2 | 1 | 1.5 | 0 | 10 | 0 |
| 0.2 mmol L-1 NH4+ | 0.2 | 1 | 1.5 | 2.5 | 0 | 0.2 |
| 2 mmol L-1 NH4+ | 0.2 | 1 | 1.5 | 2.5 | 0 | 2 |
| 5 mmol L-1 NH4+ | 0.2 | 1 | 1.5 | 2.5 | 0 | 5 |
| 10 mmol L-1 NH4+ | 0.2 | 1 | 1.5 | 2.5 | 0 | 10 |
| 20 mmol L-1 NH4+ | 0.2 | 1 | 1.5 | 2.5 | 0 | 20 |
新窗口打开|下载CSV
1.5 基因可变剪接分析
从IWGSC RefSeq v2.0中提取TaGS的基因序列, 结合全长转录本序列, 分析转录本的基因结构。1.6 蛋白结构域分析
运用Interpro (1.7 RNA提取与逆转录
称取小麦样品约0.1 g, 加液氮研磨, 用TRIzol Reagent (Vazyme)提取总RNA, 用HiScript II Q RT SuperMix for qPCR (+gDNA wiper) (Vazyme)对所提取的总RNA进行逆转录。实验按说明书进行操作。1.8 TaGS1;1-6A-1和TaGS1;1-6B-4克隆鉴定
利用Phanta Max Super-Fidelity DNA Polymerase (Vazyme)试剂盒, 以10 mmol L-1 NH4+处理的YM49根的cDNA为模板扩增TaGS1;1-6A-1, 以无氮培养的YM49根的cDNA为模板扩增TaGS1;1-6B-4。PCR程序为95℃预变性2 min, 95℃变性15 s, 退火 15 s (退火温度见表2), 72℃延伸(延伸时间按照1 kb min-1), 循环35次, 72℃延伸5 min。Table 2
表2
表2RT-PCR引物
Table 2
| 转录本名称 Transcript name | 引物名称 Primer name | 引物序列 Primer sequence (5′-3′) | 退火温度 Annealing temperature (℃) | PCR产物长度 PCR fragment (bp) |
|---|---|---|---|---|
| TaGS1;1-6A-1 | TaGS1;1-6A-1-F | GGCCAATACCCATATTTTCACTGTG | 60 | 1199 |
| TaGS1;1-6A-1-R | CAGTGGCCACCCACCAAATC | |||
| TaGS1;1-6B-3 | TaGS1;1-6B-3-F | GCCGGAGTTGTCATCGTGG | 65 | 460 |
| TaGS1;1-6B-3-R | CAGACACCCTGATGACGACG | |||
| TaGS1;1-6B-4 | TaGS1;1-6B-4-F | CCTCAGTCAGCCGGCCAT | 59 | 708 |
| TaGS1;1-6B-4-R | GATGATTACACGACGAGAAGGTAGC | |||
| TaTEF1 | TaTEF1-F | GGTGACAACATGATTGAGAGGTCC | 60 | 678 |
| TaTEF1-R | AACAGCCACAGTTTGCCTCATG |
新窗口打开|下载CSV
利用Mini BEST DNA Fragment Purification KitVer.4.0 (TaKaRa)纯化PCR产物, 将纯化的DNA片段与ZTOPO-Blunt/TA载体连接, 然后热激转化DH5α感受态细胞。利用含100 μg mL-1氨苄青的LB平板筛选阳性克隆。挑取单克隆摇菌, 提取质粒电泳检测, 进一步测序鉴定(华大基因公司)。
1.9 TaGS1;1基因可变剪接转录本的表达量测定
据转录本TaGS1;1-6A-1、TaGS1;1-6B-3、TaGS1;1-6B-4及TaTEF1 (Translation Elongation Factor 1)序列设计并合成引物(表2, 生工生物工程(上海)股份有限公司)。以cDNA为模板进行PCR扩增: 95℃预变性2 min, 95℃变性30 s, 退火20 s (退火温度见表2), 72℃延伸(延伸时间按照1 kb min-1), 循环25次, 72℃延伸5 min。取5 μL电泳检测。2 结果与分析
2.1 转录组测序结果
转录组测序clean data为26.76 GB, 其中全长序列24,481,537个。对全长序列进行polish获得一致性转录本序列, 将一致性转录本序列与小麦参考基因组进行比对去冗, 最终获得转录本序列94,074个。转录本与小麦参考基因组已知注释进行比较, 发现新基因位点12,827个, 新转录本69,191个。对新发现的转录本进行序列结构分析, 预测出36,227个完整的ORF, 含3259个转录因子和1847个lncRNA, 并完成了58,237个新转录本的功能注释。2.2 TaGS基因在染色体上的分布
从IWGSC RefSeq v2.0中筛选出12个TaGS基因, 包括9个TaGS1基因TaGS1;1-6A/6B/6D、TaGS1;2-4A/4B/4D、TaGS1;3-4A/4B/4D和3个TaGS2基因TaGS2-2A/2B/2D。TaGS1;1基因分别定位于6A、6B和6D染色体的长臂; TaGS1;2基因分别位于4A染色体的短臂和4B、4D染色体的长臂; TaGS1;3基因分别定位于4A染色体的长臂和4B、4D染色体的短臂; TaGS2基因分别位于2A、2B和2D染色体的长臂上(图1)。图1
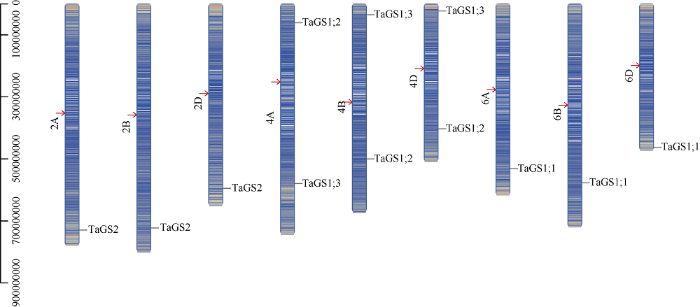 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1TaGS基因在染色体上的位置分布
箭头所示位置是染色体着丝粒。
Fig. 1Location of TaGS genes on chromosome in wheat
The position shown by the arrows are the chromosomal centromere.
2.3 TaGS基因转录本差异分析
为了分析小麦TaGS基因转录概况和可变剪接种类, 从转录组clean data中提取TaGS基因全长转录本序列2299条, 其中TaGS1;1转录本940条、TaGS1;2转录本884条、TaGS1;3转录本41条、TaGS2转录本434条。TaGS1;1、TaGS1;2、TaGS1;3和TaGS2基因中表达量最高的转录本分别为TraesCS6B02G327500.1、TraesCS4D02G240700.1、TraesCS4D02G047400.1和TraesCS2B02G528300.2。与IWSGC RefSeq v2.0中的TaGS基因转录本序列进行比对, 没有检测到定位于6B及6D染色体的TaGS1;1转录本TraesCS6B02G327500.2和TraesCS6D02G383600LC.2及定位于2A和2B染色体的TaGS2转录本TraesCS2A02G500400.2和TraesCS2B02G528300.1 (表3)。Table 3
表3
表3TaGS转录本在IWGSC RefSeq v2.0和第3代测序中的差异
Table 3
| TaGS同工酶 TaGS isoforms | 基因编号 Gene ID | IWGSC RefSeq2.0 | 第3代测序分析 Third-generation sequencing analysis | |||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 转录本编号 Transcript ID | 蛋白质 Protein (aa) | 分子量 MW (kD) | 转录本编号 Transcript ID | 蛋白质 Protein (aa) | 分子量 MW (kD) | CPM* | ||||
| TaGS1;1 | TraesCS6A02G298100 | TraesCS6A02G298100.1 | 349 | 38.4 | TraesCS6A02G298100.1 | 359 | 39.7 | 6.4 | ||
| TraesCS6A02G298100.2 | 356 | 39.2 | TraesCS6A02G298100.2 | 356 | 39.2 | 103.5 | ||||
| TraesCS6B02G327500 | TraesCS6B02G327500.1 | 356 | 39.2 | TraesCS6B02G327500.1 | 356 | 39.2 | 154.9 | |||
| TraesCS6B02G327500.2 | 325 | 35.8 | 0 | |||||||
| TraesCS6B02G327500.3 | 322 | 35.6 | TraesCS6B02G327500.3 | 322 | 35.6 | 7.6 | ||||
| ONT.45646.3 | 223 | 24.5 | 3.8 | |||||||
| TraesCS6D02G383600LC | TraesCS6D02G383600LC.1 | 356 | 39.2 | TraesCS6D02G383600LC.1 | 356 | 39.2 | 16 | |||
| TraesCS6D02G383600LC.2 | 325 | 35.8 | 0 | |||||||
| TaGS1;2 | TraesCS4A02G063800 | TraesCS4A02G063800.1 | 354 | 38.7 | TraesCS4A02G063800.1 | 354 | 38.7 | 114.6 | ||
| TraesCS4B02G240900 | TraesCS4B02G240900.1 | 354 | 38.7 | TraesCS4B02G240900.1 | 354 | 38.7 | 134.2 | |||
| TraesCS4D02G240700 | TraesCS4D02G240700.1 | 354 | 38.7 | TraesCS4D02G240700.1 | 354 | 38.7 | 170 | |||
| TaGS1;3 | TraesCS4A02G266900 | TraesCS4A02G266900.1 | 362 | 39.6 | TraesCS4A02G266900.1 | 362 | 39.6 | 8.8 | ||
| TraesCS4B02G047400 | TraesCS4B02G047400.1 | 362 | 39.5 | TraesCS4B02G047400.1 | 362 | 39.5 | 3.3 | |||
| TraesCS4D02G047400 | TraesCS4D02G047400.1 | 362 | 39.5 | TraesCS4D02G047400.1 | 362 | 39.5 | 13 | |||
| TaGS2 | TraesCS2A02G500400 | TraesCS2A02G500400.1 | 427 | 46.7 | TraesCS2A02G500400.1 | 427 | 46.7 | 9.7 | ||
| TraesCS2A02G500400.2 | 423 | 46.09 | 0 | |||||||
| TraesCS2B02G528300 | TraesCS2B02G528300.1 | 423 | 46.08 | 0 | ||||||
| TraesCS2B02G528300.2 | 369 | 40.63 | TraesCS2B02G528300.2 | 427 | 46.7 | 53 | ||||
| TraesCS2D02G500600 | TraesCS2D02G500600.1 | 427 | 46.7 | TraesCS2D02G500600.1 | 427 | 46.7 | 27.3 | |||
新窗口打开|下载CSV
依据第3代测序获得的TaGS全长转录本序列, 对IWGSC RefSeq v2.0公布的相应基因转录本进行了完善, 更新序列已提交到GenBank (MW419935- MW419949)。补充了转录本TraesCS4A02G063800.1和TraesCS2A02G500400.1的5′UTR序列, 将转录本TraesCS6A02G298100.1的5′端向上游延长了80个核苷酸, 与之对应蛋白质在N端多出9个氨基酸残基, 所有转录本3′末端更新到以polyA结束。首次鉴定到TaGS1;1-6B的一个新转录本ONT.45646.3, 并将其命名为TraesCS6B02G327500.4 (TaGS1;1-6B-4), 其基因结构如图2所示, 与转录本TraesCS6B02G327500.1 (TaGS1;1-6B-1)相比, 新转录本缺失E9和E10外显子, E1和E11外显子部分缺失, 是一个新的可变剪接形式。
图2
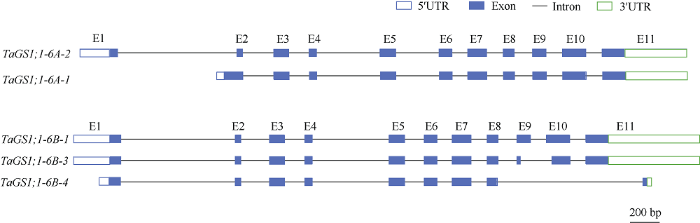 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2TaGS可变剪接的基因结构比较
Fig. 2Comparison of gene structure of TaGS alternative splicing
为了验证3代测序所更新的TaGS1;1-6A-1和新发现的可变剪接形式TaGS1;1-6B-4, 利用其特异引物, 分别以10 mmol L-1 NH4+及无氮处理的YM49根cDNA为模板扩增到了长度约为1199 bp和708 bp的片段(图3), 克隆鉴定发现与3代转录组测序结果一致。
图3
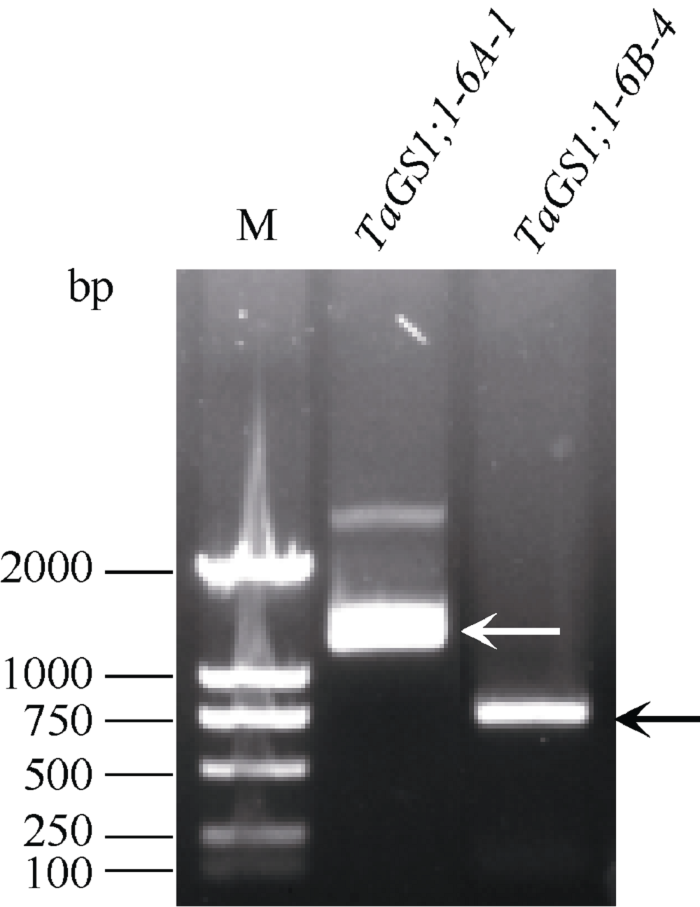 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3转录本TaGS1;1-6A-1和TaGS1;1-6B-4 PCR扩增
M: DNA分子量标记。箭头所指为目的条带。
Fig. 3PCR amplification of TaGS1;1-6A-1, and TaGS1;1-6B-4 transcripts
M: marker. The arrow points to the target segment.
2.4 TaGS转录本对应的蛋白质结构域分析
TaGS含Gln_synt_N、Gln_synt_b-grasp、Gln_synth_N_conserv_site、Gln_synth/guanido_kin_cat_dom、Gln_synth_cat_dom和Gln_synth_gly_rich_site等6个结构域。Gln_synt_N是GS的N-端结构域, 包含催化Glu与NH4+连接的Gln_synt_b-grasp结构域和N-端保守结构域Gln_synth_N_conserv_site。Gln_synth/guanido_kin_cat_dom和Gln_synth_cat_dom是C-端的催化结构域,有重叠区域。Gln_synth_gly_rich_site是C-端富含甘氨酸区域,与ATP结合有关。TaGS1;1-6A-1包含所有的6种蛋白结构域;TaGS1;1-6B-3缺少Gln_synth_gly_rich_site结构域,可能影响其ATP结合效率;TaGS1;1-6B-4缺少Gln_synth_gly_rich_site和Gln_synth_cat_dom结构域, C-端催化结构域缺失。2.5 氮处理对不同氮效小麦TaGS1;1可变剪接转录本表达的影响
为了解TaGS1;1可变剪接本在氮同化中的作用, 研究了不同氮处理对氮高效品种YM49和氮低效品种XN509的TaGS1;1-6A-1、TaGS1;1-6B-3和TaGS1;1-6B-4表达的影响(图5)。发现随着氮浓度(NO3-或NH4+)升高, TaGS1;1-6A-1在YM49叶片中表达不断升高, 而在根中表达逐渐降低; TaGS1;1-6A-1主要在XN509根中表达, 且表达量随着氮浓度升高而增加。YM49的TaGS1;1-6B-3表达量比XN509高, TaGS1;1-6B-3在YM49和XN509中表达趋势一致: 在叶片中表达被高浓度NO3-诱导, 却被高浓度NH4+所抑制; 在根中表达随着NO3-浓度的升高而降低, 但NH4+浓度影响甚微。TaGS1;1-6B-4在3个转录本中表达水平最低, 且主要在低氮条件下表达, NH4+浓度不影响其在YM49根中的表达。图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5不同氮处理下TaGS1;1可变剪接转录本的表达
Fig. 5Expression of TaGS1;1 alternative splicing transcripts under different nitrogen conditions
3 讨论
第2代测序技术读长短, 序列拼接时导致一些转录本信息缺失。利用第3代全长转录组测序技术, 可以获取转录本的完整信息, 对于研究基因可变剪接有明显的优势。本研究利用单分子全长转录组测序技术对TaGS基因的可变剪接进行了分析, 发现了3个可变剪接转录本, TaGS1;1-6A-1、TaGS1;1- 6B-3和TaGS1;1-6B-4。其中TaGS1;1-6B-4是新发现的TaGS1;1-6B可变剪接形式。小麦作是异源六倍体, 基因组复杂庞大。基因表达受时序和环境条件调控, 因实验组织种类、发育阶段及处理方式限制, 不能排除TaGS基因存在其他可变剪接形式。植物基因可变剪接受生物或非生物胁迫影响。GS是植物无机氮同化的关键酶, 其表达首先受组织器官及外源氮素供应的调控。与TaGS1;1-6A-2相比, TaGS1;1-6A-1的E1外显子缺失和I1内含子部分保留, 导致TaGS1;1结构域Gln_synt_N部分缺失, 表达量约是TaGS1;1-6A-2的5% (图2、图4和表3)。高浓度的NO3-和NH4+抑制叶片中TaGS1;1表达[18], 却诱导TaGS1;1-6A-1表达, 且氮高效小麦YM49叶片TaGS1;1-6A-1表达量更高(图5), 推测TaGS1;1- 6A-1在高氮条件下发挥氮同化功能, 进而提高氮素利用效率。与TaGS1;1-6B-1相比, TaGS1;1-6B-3的E9外显子部分缺失, 导致TaGS1;1缺失与ATP结合结构域Gln_synth_gly_rich_site (图2和图4), 推测其可能有特殊的催化特性。TaGS1;1-6B-4的3′端大量缺失, 其编码蛋白仅包含Gln_synt_N及Gln_synt_ b-grasp结构域(图2和图4)。GS的催化活性中心非常保守, 一般由一个亚基N-端Gln_synt_b-grasp结构域与另一个相邻亚基C-端催化结构域(Gln_synth_ cat_dom)组成[19,20,21]。小麦胞质型GS同工酶是12聚体[22], TaGS1;1-6B-4编码蛋白C-端大量缺失, 可能会影响酶活性中心形成和调节GS同工酶全酶组装。外源氮素调控TaGS基因3个可变剪接转录本表达, 表明TaGS存在更为复杂的转录水平调控。进一步研究TaGS可变剪接转录本在小麦不同发育时期不同组织器官的表达动态, 将有助于深入了解TaGS的功能。
图4
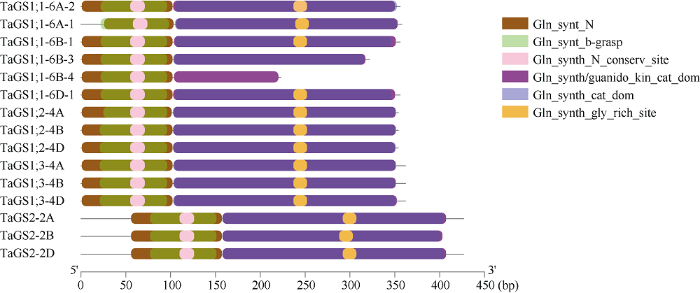 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4TaGS转录本对应的蛋白质结构域
Fig. 4Functional domain of proteins corresponding to transcripts of TaGS
4 结论
TaGS由12个核基因编码, 单分子测序技术显示其有15个转录本, 存在3种可变剪接形式。TaGS1;1-6B-4是新鉴定转录本, 编码缺少Gln_synth_gly_rich_site和Gln_synth_cat_dom结构域的GS蛋白, 预测分子量24.5 kD。不同氮处理不同氮效小麦品种的不同可变剪接转录本表达特点不同, 推测TaGS基因可变剪接在小麦氮同化过程中发挥不同的功能。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
