 ,1,2,3,**, 尚丽娜
,1,2,3,**, 尚丽娜 ,1,2,3,**, 金中辉1,2,3, 丁艺1, 李燕1,3, 王季春1,2,3, 胡柏耿4, Vadim Khassanov5, 吕典秋
,1,2,3,**, 金中辉1,2,3, 丁艺1, 李燕1,3, 王季春1,2,3, 胡柏耿4, Vadim Khassanov5, 吕典秋 ,1,2,3,*
,1,2,3,*Genome-wide identification and characterization of PIF genes and their response to high temperature stress in potato
JIAN Hong-Ju ,1,2,3,**, SHANG Li-Na
,1,2,3,**, SHANG Li-Na ,1,2,3,**, JIN Zhong-Hui1,2,3, DING Yi1, LI Yan1,3, WANG Ji-Chun1,2,3, HU Bai-Geng4, Vadim Khassanov5, LYU Dian-Qiu
,1,2,3,**, JIN Zhong-Hui1,2,3, DING Yi1, LI Yan1,3, WANG Ji-Chun1,2,3, HU Bai-Geng4, Vadim Khassanov5, LYU Dian-Qiu ,1,2,3,*
,1,2,3,*通讯作者: *吕典秋, E-mail:smallpotatoes@126.com
第一联系人:
收稿日期:2020-12-29接受日期:2021-04-14网络出版日期:2021-05-20
| 基金资助: |
Corresponding authors: *smallpotatoes@126.com
First author contact:
Received:2020-12-29Accepted:2021-04-14Published online:2021-05-20
| Fund supported: |
作者简介 About authors
荐红举,E-mail:hjjian518@swu.edu.cn
尚丽娜,E-mail:cxldshanglina@163.com

摘要
植物光敏色素作用因子(phytochrome interacting factors, PIFs)属于碱性-螺旋-环-螺旋(basic helix-loop-helix, bHLH)转录因子家族, 通过将光和温度等外部环境信号与植物体内源信号途径相整合, 进而形成复杂的信号转导网络来精密调控植物的生长发育进程。目前, 关于马铃薯PIF家族基因的研究较少, 鉴定和分析StPIF家族成员有助于进一步提高马铃薯的产量和品质。本研究运用生物信息学方法, 以拟南芥PIF家族成员蛋白序列作为源序列, 通过在马铃薯基因组数据库中进行BlastP分析鉴定出7个StPIFs家族成员, 并对其进行系统进化、染色体分布、复制事件、蛋白理化性质、基因结构、Motif预测、启动子顺式作用元件、基因表达模式以及对高温胁迫的响应分析。结果显示, StPIF家族所有成员均含有Motif 1 (bHLH结构域)、Motif 2 (APB结构域)基序; 在StPIF基因的启动子区域预测到多个参与光响应、激素、干旱、低温、昼夜节律以及防御和应激反应调控元件; 基因表达模式和现蕾期高温胁迫响应分析表明, 家族成员具有明显的组织表达特异性, 基因存在功能分化, 且大部分StPIF成员对生物胁迫和高温等非生物胁迫具有明显响应。以上研究结果极大丰富了我们对StPIF家族的认识, 为进一步探究StPIF基因在马铃薯生长期应对生物胁迫以及在结薯期应对高温等非生物胁迫中发挥的功能奠定了理论基础。
关键词:
Abstract
Phytochrome interacting factors (PIFs) belong to the basic helix-loop-helix (bHLH) transcription factor family, which integrates external environmental signals such as light and temperature with plant endogenous signal pathways, and then form a complex signal transduction network to precisely regulate the growth and development of plants. Up to now, there are few studies of PIF family genes in potato. Identification and analysis of StPIF family members will help to further improve the yield and quality of potatoes. In this study, BlastP analysis was performed in the potato genome database using Arabidopsis PIFs family member protein sequences as source sequences and seven StPIFs family members were identified. Systemic evolution, chromosome distribution, replication events, protein physicochemical properties, gene structure, motif prediction, promoter cis-acting elements, gene expression pattern, and the response to high temperature stress were also conducted. These results showed that all members of the StPIFs gene family contain Motif 1 (bHLH domain) and Motif 2 (APB domain); multiple cis-regulatory elements involved in light response, hormones, drought, low temperature, and circadian rhythm as well as defense and stress response regulatory elements were predicted in the promoter regions of the StPIF genes. Results of gene expression patterns and high temperature stress response at budding stage revealed that StPIFs family members had obvious tissue expression specificity, indicating their functional differentiation, and most StPIFs members had obvious responses to biotic stress and abiotic stresses including high temperature. In conclusion, these foundlings greatly enriched the understanding of the members of the StPIF family, and laid a theoretical foundation for further exploring the functions of StPIFs genes in responses to biotic stress and abiotic stress during potato growth period including high temperature at tuber stage.
Keywords:
PDF (6218KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
荐红举, 尚丽娜, 金中辉, 丁艺, 李燕, 王季春, 胡柏耿, Vadim Khassanov, 吕典秋. 马铃薯PIF家族成员鉴定及其对高温胁迫的响应分析. 作物学报, 2022, 48(1): 86-98 DOI:10.3724/SP.J.1006.2022.04285
JIAN Hong-Ju, SHANG Li-Na, JIN Zhong-Hui, DING Yi, LI Yan, WANG Ji-Chun, HU Bai-Geng, Vadim Khassanov, LYU Dian-Qiu.
植物光敏色素作用因子(plant phytochrome interacting factors, PIF)是连接植物体内外信号的桥梁, 通过将植物体外部光、温度、胁迫等环境信号与激素、生物钟等内部信号整合, 在调控植物种子萌发[1]、开花[2]、庇荫反应[3]、抗逆[4,5,6,7]等途径发挥重要功能。PIFs家族成员有保守的N端APB结构域(active phytochrome B-binding Motif)和C端bHLH (basic helix-loop-helix)结构域, 部分成员N端还含有1个APA结构域(active phytochrome A-binding Motif)[8]。PIF家族成员通过APB或APA结构域分别与光敏色素B (phytochrome B, phyB)或光敏色素A (phytochrome A, phyA)相结合来调控植物形态建成[9]。
在水稻中, 过表达玉米ZmPIF3基因能提高转基因水稻对干旱和盐的抵抗能力, 进一步研究发现, ZmPIF3在胁迫条件下通过促进气孔的闭合来减少水分的蒸发, 从而提高转基因植物的抗逆性[4,6]。与ZmPIF3功能相似, ZmPIF1也能通过调控气孔的关闭来提高转基因水稻的抗旱能力[7]。AtPIF3能够直接结合在CBF (CREPEAT BINDING FACTOR)基因的启动子区, 抑制CBF及其下游低温响应基因表达, 负调控植株的抗冻性[5]。拟南芥突变体pif4在高温条件下出现晚开花的表型, 进一步研究发现, 温度升高促进PIF4的表达, PIF4通过结合开花相关的正调控因子FT (FLOWERING LOCUS T)基因的启动子, 调节FT基因的表达促进开花[2]。PIF4在响应高温环境时, 能够激活生长素生物合成基因YUC8、TAA1, 促进生长素应答基因IAA19、IAA29的表达, 调节植物下胚轴和叶柄伸长, 从而使植物提高对热环境的抵抗能力[10,11]。高温还能够诱导PIF4在气孔前体细胞中积累, 并与SPEECHLESS (SPCH)基因启动子结合抑制该基因的表达, 从而限制高温下气孔的产生, 提高植物的耐热性[12]。在棉花中, GhPIF4参与调控高温下的花药败育[13]。油菜中, BnaPIF4和BnaBZR1基因通过相互拮抗作用来调控甘蓝型油菜弱光光效[14]。拟南芥PIF5能够与2个编码DELLA蛋白的基因RGA1和GAI的启动子结合并激活其表达, 进而抑制黑暗中种子萌发[1]。AtPIF7在昼夜节律的作用下负调控DREB1的表达[15]。然而, 在马铃薯中, 仅有报道StPIF3参与光周期调控马铃薯块茎形成[16], 并对StPIF4基因进行克隆和初步分析[17], 关于马铃薯中PIFs基因家族成员鉴定的研究, 以及其他成员是否能够整合外部环境信号来调控马铃薯植株的生长发育以及块茎形成仍未可知。
本研究运用生物信息学方法, 以拟南芥PIF家族成员蛋白序列作为源序列, 在马铃薯基因组数据库中进行BlastP分析, 鉴定出7个StPIF家族成员, 并对其进行系统进化、染色体分布、复制事件、蛋白理化性质、基因结构、Motif预测、启动子顺式作用元件、基因表达模式以及高温胁迫后不同组织部位的响应分析, 以期为进一步探究StPIF基因在马铃薯生长期应对生物胁迫以及在结薯期应对高温等非生物胁迫中发挥的功能奠定理论基础。
1 材料与方法
1.1 试验材料
选用马铃薯‘中薯20号’在光照培养箱中进行盆栽试验(塑料盆直径24 cm), 培养箱温度22℃/18℃ (白天/黑夜), 光周期16 h/8 h, 光照强度270 μmol m-2 s-1, 相对湿度60%±5%, 将生长至现蕾期的马铃薯植株分别置于35℃/30℃和22℃/18℃进行不同温度处理(其他条件保持一致), 并对不同组织部位进行取材, 液氮速冻后保存于-80℃冰箱, 备用。1.2 马铃薯PIF家族成员的鉴定
从拟南芥数据库TAIR (1.3 多序列比对
为明确PIF基因家族成员的蛋白保守结构域, 利用软件MEGA7.0和Jalview对拟南芥、马铃薯和番茄的PIFs蛋白序列进行多序列比对并展示。1.4 马铃薯PIF家族成员的系统进化分析
利用MEGA7.0软件采用邻接法(Neighbor- Joining, NJ)对鉴定出的7条马铃薯PIF蛋白序列和通过ClustalW (v1.81)比对得到的7条拟南芥PIF蛋白序列、8条番茄PIF蛋白序列、7条水稻PIF蛋白序列以及6条玉米PIF蛋白序列构建系统发育进化树。设置参数为: Passion model、partial deletion以及1000 bootstrap replications。1.5 马铃薯PIF家族成员的基因结构、保守基序分析
从马铃薯数据库PGSC (1.6 马铃薯PIFs基因家族共线性分析
从马铃薯数据库PGSC (1.7 启动子顺式作用元件分析
通过TBtools提取StPIF基因起始密码子上游2000 bp的序列在PlantCARE网站(1.8 不同胁迫下的表达模式分析
从马铃薯数据库PGSC (1.9 高温胁迫后不同组织部位RNA的提取与转录水平分析
分别提取现蕾期马铃薯的根、茎、叶、匍匐茎全长、匍匐茎尖、小薯等不同组织部位RNA, 总RNA提取纯化试剂盒购自生工生物工程(上海)股份有限公司, 反转录试剂盒Hifair II 1st Strand cDNA Synthesis SuperMix for qPCR (gDNA digester plus)购自重庆沐生生物科技有限公司。在CFX Connect Real-time PCR仪(成都百乐科技有限公司)上进行qRT-PCR, 程序如下: 95℃ 30 s; 95℃ 5 s, 60℃ 30 s, 39个循环; 95℃ 5 s; 60℃ 1 min; 熔解曲线60~95℃, 增量0.5℃。以elongation factor 1-α (ef1α)基因为内参基因, 按2-ΔΔCt方法分析基因的相对表达量, 每个样品重复3次。由生工生物工程(上海)股份有限公司合成qRT-PCR相关引物, 序列如表1。Table 1
表1
表1本研究所用引物
Table 1
| 引物 Primer | 正向序列 Forward sequence (5'-3') | 反向序列 Reverse sequence (5'-3') |
|---|---|---|
| ef1α StPIF1a StPIF1b StPIF3 StPIF4 StPIF8 | ATTGGAAACGGATATGCTCCA GTTTCACACGTACCCGATAATG GTACTTCGACTGTAGAGATCCG GAGTTTAGGTATGGGTATGGGG TGAACTTCCTGTTCCACATCAA AATCAGTGTGTACCGAGTTG | TCCTTACCTGAACGCCTGTCA GAAATGTCACATGTAGTCGTCG TTTGAATCCACAATCGTCGATG TACAGCTATTCCTGGATAAGCG TTCATTAGGATCATAGCCTGGC CTAACCCATGCATTGCTAAC |
新窗口打开|下载CSV
2 结果与分析
2.1 马铃薯PIF家族成员的鉴定与分析
本研究以确定的拟南芥PIF蛋白序列作为源序列, 通过BlastP分析, 获得7个马铃薯PIF基因家族成员(表2)。基于马铃薯PIF蛋白序列与拟南芥、番茄、水稻和玉米的PIF蛋白序列的聚类分析, 可将马铃薯PIF基因分别命名为StPIF1a、StPIF1b、StPIF3、StPIF4、StPIF7a、StPIF7b、StPIF8。对马铃薯PIFs家族成员的序列长度、分子量、等电点以及在染色体上的位置等基本特性的分析表明, 7个StPIFs成员共分布在5条染色体上。其中, StPIF3和StPIF8共同分布在染色体ch01上, StPIF7a在染色体ch03上, StPIF1b和StPIF7b同在染色体ch06上, StPIF4在染色体ch07上, StPIF1a在染色体ch09上。其分子量在46.81~75.83 kD之间, 氨基酸大小在416~708 aa之间, 等电点介于5.13~8.71。每条StPIF蛋白序列上都含有bHLH保守结构域。Table 2
表2
表2马铃薯PIFs家族成员的序列特性
Table 2
| 基因 Gene | 基因编号 Gene ID | 氨基酸数目 Number of amino acids (aa) | 分子量Molecular weight (kD) | 等电点Isoelectric point (pI) | 位置 Location | 功能域 Functional domains (Start-End, bp) |
|---|---|---|---|---|---|---|
| StPIF1a | PGSC0003DMG400018950 | 578 | 62.94 | 6.42 | ch09:45450532-45460120 | 343-406/bHLH |
| StPIF1b | PGSC0003DMG400014705 | 515 | 56.41 | 5.13 | ch06:2038519-2047215 | 309-372/bHLH |
| StPIF3 | PGSC0003DMG400018280 | 708 | 75.83 | 7.26 | ch01:79007967-79013234 | 450-512/bHLH |
| StPIF4 | PGSC0003DMG401015926 | 517 | 57.44 | 7.09 | ch07:42074703-42078252 | 331-394/bHLH |
| StPIF7a | PGSC0003DMG400024554 | 437 | 48.17 | 6.79 | ch03:55395567-55398080 | 237-298/bHLH |
| StPIF7b | PGSC0003DMG400033087 | 416 | 46.81 | 8.71 | ch06:51466651-51468759 | 195-252/bHLH |
| StPIF8 | PGSC0003DMG400025976 | 464 | 50.75 | 7.62 | ch01:70077080-70081427 | 281-344/bHLH |
新窗口打开|下载CSV
2.2 拟南芥、马铃薯、番茄PIF蛋白序列比对
对拟南芥、马铃薯和番茄PIF蛋白序列进行比对(图1)发现, 所有马铃薯PIF成员均含有保守的APB结构域和bHLH结构域, PIF1和PIF3成员还同时含有APA结构域。结构域中的保守区段存在个别氨基酸的差异。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1拟南芥、马铃薯和番茄PIF氨基酸多序列比对
A: APB结构域; B: APA结构域; C: bHLH结构域。
Fig. 1Alignment of PIFs amino acid sequences from Arabidopsis thaliana, Solanum tubersum, and Solanum lycopersicum
A: active phytochrome B-binding (APB) domain; B: active phytochrome A-binding (APA) domain; C: basic helix-loop-helix (bHLH) DNA-binding domain.
2.3 马铃薯PIFs家族成员的系统进化分析
对马铃薯StPIF蛋白序列与拟南芥、番茄、水稻和玉米中的PIF蛋白序列进行系统进化分析有助于预测其在马铃薯中发挥的功能。分析结果表明, PIF家族成员大致可以分为5个亚家族(图2)。第I亚家族中包括拟南芥、马铃薯和番茄的PIF7和PIF8成员; 第II亚家族中只有拟南芥的PIF6成员; 第III亚家族包括拟南芥、马铃薯、番茄和玉米的PIF3成员, 以及OsPIL16、OsPIL15、ZmPIF1和ZmPIF2; 第IV亚家族中包括拟南芥、马铃薯和番茄的PIF1成员; 第V亚家族不仅包括马铃薯和番茄的PIF4, 拟南芥的PIF4、PIF5成员, 还包括OsPIL11、OsPIL12、OsPIL13、OsPIL14以及ZmPIF4、ZmPIF5、ZmPIF6。其中第I和第IV亚家族类是双子叶特有的, 第III和第V亚家族类是单、双子叶共有的。PIF4和PIF5在拟南芥和玉米中聚在一起, 在马铃薯和番茄中只发现了PIF4成员。马铃薯PIF家族成员与拟南芥和番茄的同源性明显大于单子叶植物水稻和玉米。图2
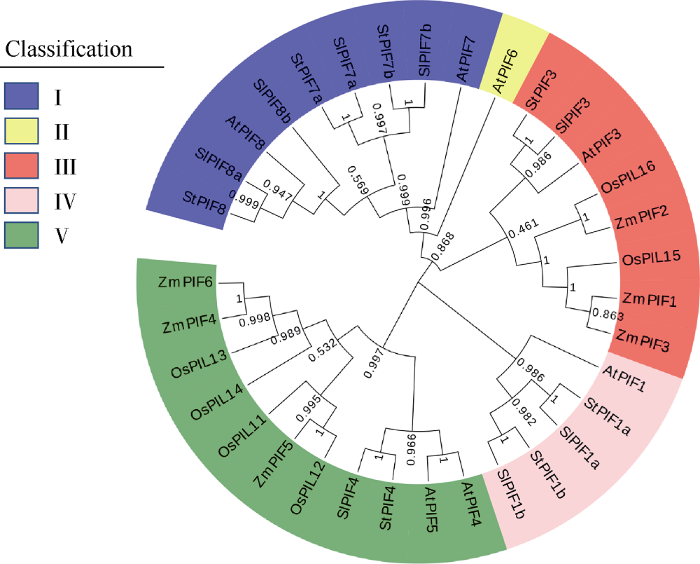 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2PIF基因家族系统发育进化树
At: 拟南芥; St: 马铃薯; Sl: 番茄; Os: 水稻; Zm: 玉米。
Fig. 2Phylogenic tree of PIFs gene family
At: Arabidopsis thaliana; St: Solanum tubersum; Sl: Solanum lycopersicum; Os: Oryza sativa; Zm: Zea mays.
2.4 马铃薯PIFs家族成员的基因结构、保守基序及结构域分析
编码区的差异, 特别是可以改变基因功能的差异, 可能是由基因结构上氨基酸的变化引起的[19]。对马铃薯StPIF家族成员基因结构分析发现(图3), 7个StPIF基因均含有5'UTR和3'UTR, 除StPIF7a、StPIF7b、StPIF8含有6个外显子外, 其余4个基因均含有7个外显子。结合进化分析发现, 在同一分支的StPIF基因结构相似, 且外显子区段的大小和分布基本相近。图3
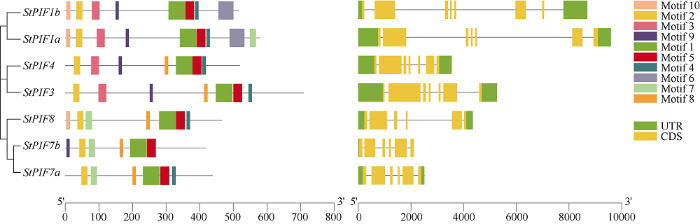 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3马铃薯PIF基因结构和保守基序
Motif: 保守基序; UTR: 非翻译区; CDS: 编码区序列。
Fig. 3Structure and conserved motif of PIFs gene in potato
Motif: conservative base sequence; UTR: untranslated region; CDS: coding region sequence.
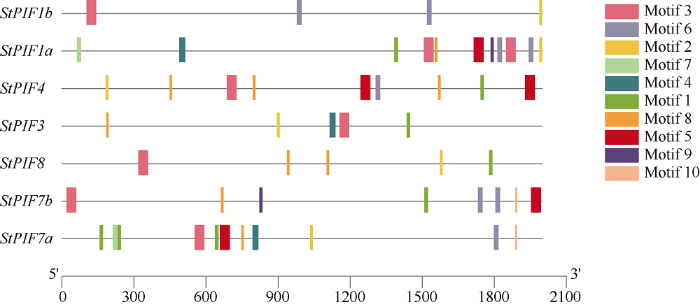
利用MEME在线网站对马铃薯PIF蛋白序列进行保守基序分析(图3)发现, Motif 1、Motif 2、Motif 4和Motif 5在所有StPIF蛋白中均存在, 其中Motif 1对应bHLH结构域, Motif 2对应APB结构域(图4), 说明马铃薯PIF基因家族的基序具有高度保守性。除此之外, 7个成员中, 有4个StPIF序列含有Motif 3, 2个StPIF序列含有Motif 6, 4个StPIF序列含有Motif 7, 5个StPIF序列含有Motif 8, 5个StPIF序列含有Motif 9, 3个StPIF序列含有Motif 10。
图4
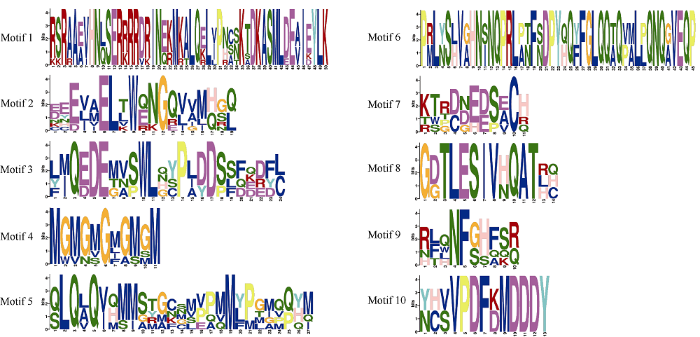 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4马铃薯PIFs家族成员保守基序
Fig. 4Conserved motifs of PIFs family members in potato
2.5 马铃薯PIFs家族成员共线性分析
基因复制在生物进化过程中起着关键的作用, 为研究马铃薯PIF基因间可能的关系和潜在的基因重复, 运用TBtools鉴定的马铃薯PIF家族成员中只有StPIF7a和StPIF7b存在部分重复(图5)。图5
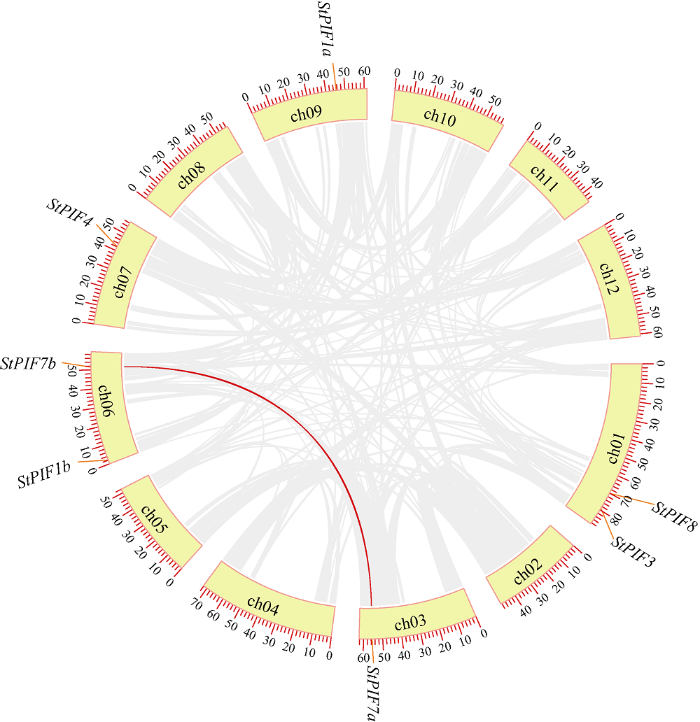 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5马铃薯PIF基因家族共线性分析
Fig. 5Collinearity analysis of the PIFs gene family in potato
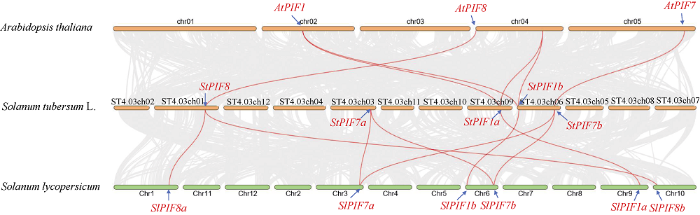
为研究马铃薯PIF基因的进化起源和过程, 利用TBtools对马铃薯、拟南芥和番茄的PIF基因进行共线性分析并绘图(图6)。分析结果表明, 4个配对的AtPIF-StPIF基因(AtPIF1-StPIF1b、AtPIF1- StPIF1a、AtPIF7-StPIF7b、AtPIF8-StPIF8)位于拟南芥和马铃薯基因组之间具有共线性的基因组区域。8个配对的StPIF-SlPIF基因(StPIF1a-SlPIF1a、StPIF1b-SlPIF1b、StPIF7a-SlPIF7a、StPIF7b- SlPIF7a、StPIF7a-SlPIF7b、StPIF7b-SlPIF7b、StPIF8- SlPIF8a、StPIF8-SlPIF8b)位于马铃薯和番茄基因组之间具有共线性的基因组区域。
图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6马铃薯、拟南芥和番茄PIF基因的共线性分析
Fig. 6Collinearity analysis of PIFs genes among Solanum tubersum, Arabidopsis thaliana, and Solanum lycopersicum
2.6 启动子顺式作用元件分析
为研究StPIF家族成员潜在的调控机制, 进一步对起始密码子上游2000 bp的序列进行顺式作用元件分析。结果表明, 马铃薯StPIF家族基因启动子区域除了有TATA-box和CAAT-box等一些基本的保守区外, 还有很多光响应元件以及与胁迫、激素和植物生长发育相关等共137个(16种)重要顺式作用元件(图7)。分别为分生组织表达元件(3个CAT-box)、昼夜节律调控元件(4个circadian)、防御和胁迫响应元件(1个TC-rich repeats)、低温应答元件(3个LTR)、受伤应答元件(7个WUN-Motif)、缺氧诱导响应元件(6个ARE)、干旱应答元件(21个MYC、15个MYB和6个MBS)以及生长素应答元件(1个TGA-element和3个AuxRR-core)、脱落酸应答元件(6个ABRE)、乙烯应答元件(16个ERE)、茉莉酸甲酯应答元件(11个CGTCA-Motif、11个TGACG-Motif和11个as-1)、赤霉素应答元件(5个P-box、1个TATC-box和1个GARE-Motif)和水杨酸应答元件(5个TCA-element)。图7
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7马铃薯StPIF基因启动子中与胁迫反应和植物发育相关的顺式作用元件
Fig. 7Cis-elements in the promoter of StPIFs genes in responses to various stresses and plant development in potato
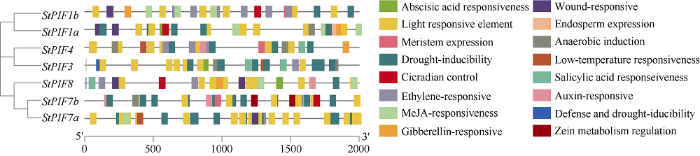
利用MEME在线网站对马铃薯PIFs家族成员启动子序列进行保守基序分析(图8和图9)表明, StPIF1a启动子序列含有Motif 1~9; StPIF1b启动子序列含有Motif 2、Motif 3、Motif 6; StPIF3启动子序列含有Motif 1~3、Motif 4、Motif 8; StPIF4启动子序列含有Motif 1~3、Motif 5、Motif 6、Motif 8, StPIF7a启动子序列含有Motif 1~8、Motif 10, StPIF7b启动子序列含有Motif 1、Motif 3、Motif 5~6, Motif 8~10, StPIF8启动子序列含有Motif 1~3和Motif 8。
图8
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8马铃薯PIF启动子序列保守基序
Fig. 8Conserved motif of PIFs promoter sequence in potato
图9
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9马铃薯PIF家族成员启动子保守基序
Fig. 9Conserved motifs of promoters of PIFs family in potato
利用PlantPAN对马铃薯PIF家族成员启动子序列可能结合的转录因子进行分析(图10)表明, StPIF家族成员启动子顺式作用元件能结合包含bZIP、NAC、NF-YB等31种类型的转录因子。其中StPIF1a的启动子区含有可以被MYB、G2-like、WRKY、SBP等14种转录因子结合的顺式作用元件; StPIF1b的启动子区含有可以被ARID、AP2/ERF、TCP等14种转录因子结合的顺式作用元件; StPIF3 的启动子区含有可以被SBP、NF-Y、bHLH等14种转录因子结合的顺式作用元件; StPIF4的启动子区含有可以被MYB、bZIP等13种转录因子结合的顺式作用元件; StPIF7a的启动子区含有可以被HSF、WRKY等11种转录因子结合的顺式作用元件; StPIF7b的启动子区含有可以被EIN3、WRKY等16种转录因子结合的顺式作用元件; StPIF8的启动子区含有可以被MADS box、WRKY、NF-Y等14种转录因子结合的顺式作用元件。
图10
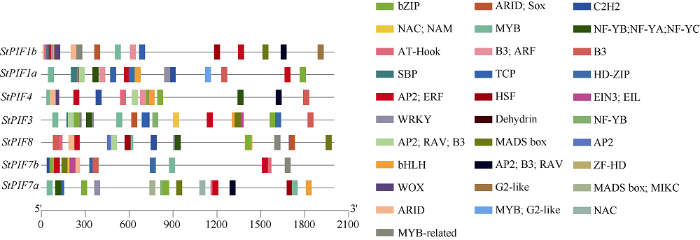 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图10马铃薯PIF启动子序列结合转录因子
Fig. 10PIFs promoter sequence binding transcription factor in potato
2.7 不同胁迫下的表达模式分析
基于从马铃薯PGSC数据库中下载的转录组数据, 对StPIFs家族成员在6种环境胁迫(高温、盐渍、干旱、晚疫病以及诱导因子苯并噻二唑和β-氨基丁酸)和4种外源激素(脱落酸、生长素、赤霉素和细胞分裂素)处理后进行表达模式分析(图11)。结果显示, 在环境胁迫中, StPIF1a本体表达水平较高, 对高温、盐渍和干旱响应不明显; StPIF1b和StPIF3对高温有明显响应, 对盐渍和干旱响应不明显; StPIF4对高温有强烈响应, 对盐渍和干旱有明显响应; StPIF7a和StPIF7b对高温、盐渍和干旱均有明显响应, 但本体表达量较低, 几乎不表达(不进行后续分析); StPIF8对干旱有明显响应, 但对高温和盐渍响应不明显。对马铃薯晚疫病的响应过程中, StPIF4响应最强烈, StPIF1a、StPIF1b、StPIF3和 StPIF8均有不同程度响应; 对苯并噻二唑和β-氨基丁酸的响应过程中, StPIF3响应最强烈, StPIF1a、StPIF1b、StPIF4和StPIF8均有不同程度响应。外源施加激素的响应分析表明, 该家族成员对激素均有不同程度响应, 其中StPIF1a、StPIF1b和StPIF4 对细胞分裂素有明显响应, StPIF3对细胞分裂素和脱落酸响应强烈, StPIF8对脱落酸和赤霉素有明显响应。图11
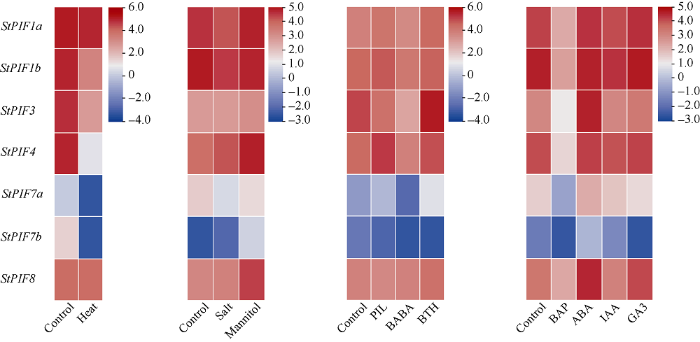 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图11马铃薯PIF基因对环境胁迫的响应
Fig. 11Heat map of PIFs genes in response to environmental stress in potato
2.8 高温胁迫下不同组织部位的转录水平分析
温度是影响马铃薯后期结薯的较为关键的不可控因素之一。为分析StPIF家族成员在马铃薯后期应对非生物胁迫中的作用, 结合家族基因在转录组数据中不同胁迫下的表达模式, 进一步对StPIF1a、StPIF1b、StPIF3、StPIF4和StPIF8进行35℃高温胁迫下不同组织部位的转录水平分析(图12), 其中StPIF7a、StPIF7b在不同组织部位表达量均较低, 数据未显示; 现蕾期植株在22℃下生长10 d后, 部分匍匐茎尖膨大成小薯, 而在35℃高温处理10 d后, 匍匐茎尖未见膨大。StPIF整个家族成员在马铃薯的根中几乎不表达, StPIF1a、StPIF1b、StPIF3、StPIF4和StPIF8在叶片和茎中均高表达, 且StPIF1a、StPIF1b、StPIF3和StPIF4在匍匐茎中均有表达。经35℃高温处理10 d后, StPIF1a、StPIF3、StPIF4和StPIF8表达量均明显下调, 无论叶片、茎还是匍匐茎中, StPIF4、StPIF8表达下调均较为明显; 然而, StPIF1b在叶片等地上部位及匍匐茎中表达均发生上调。图12
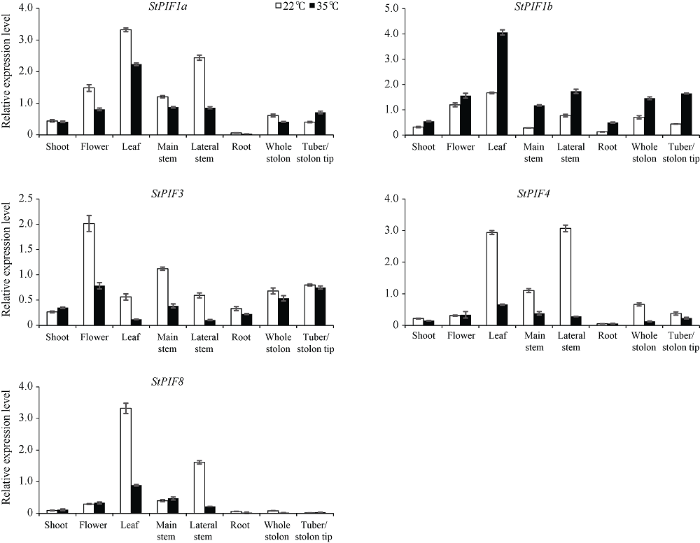 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图12高温胁迫下StPIF家族基因在不同组织部位的表达分析
Fig. 12Relative expression level of StPIFs genes in different tissues under high temperature stress
3 讨论
PIF家族成员在植物的生长发育中发挥着重要的作用, 不同物种中PIF家族成员的数量也不相同。单子叶植物水稻[21]和玉米[22]中均为6个, 葡萄[25]、拟南芥[20]、番茄[23]、杨树[24]中分别有4、7、8、10个, 然而, 对于马铃薯PIF家族成员功能知之甚少。本研究运用生物信息学方法, 对马铃薯PIF蛋白序列与拟南芥、番茄、水稻和玉米中的PIF蛋白序列比对, 在基因组数据库中共鉴定出7个StPIF成员, 归为5个亚家族, 分别将其命名为StPIF1a、StPIF1b、StPIF3、StPIF4、StPIF7a、StPIF7b、StPIF8。基因结构与保守结构域的差异是遗传变异的基础, 对StPIF家族成员基因结构与进化分析表明, 同一分支上StPIF基因结构相似, 外显子区段大小和分布基本相近; 蛋白序列保守基序分析, StPIF成员均含有bHLH和APB结构域, 部分成员含有APA结构域。表明该家族成员在进化过程和功能上具有高度保守性。
启动子顺式作用元件分析有助于预测基因潜在的功能, 转录因子通过与下游靶基因启动子中的顺式作用元件结合, 进而对靶基因起到转录调控作用[26]。研究表明, 油菜BnaPIF4和BnaBZR1基因通过相互拮抗作用来调控甘蓝型油菜的弱光光效[14]。过表达ZmPIF3能提高转基因水稻对干旱和盐的抵抗能力[4,6]; AtPIF3通过抑制CBF及其下游低温响应基因表达, 负调控植株的抗冻性[5]; AtPIF7在昼夜节律的作用下负调控DREB1的表达[15]; 拟南芥PIF5能够与RGA1和GAI的启动子结合并激活其表达, 进而抑制黑暗中种子萌发[1]。启动子顺式作用元件分析表明, StPIF家族7个成员中均含有多个光响应和干旱应答元件, StPIF4和StPIF8中含有低温应答元件, StPIF1a、StPIF1b、StPIF7b、StPIF8含有时钟响应元件, StPIF3中含有防御和胁迫应答元件, StPIF1a、StPIF1b、StPIF4、StPIF8中含有赤霉素应答元件, StPIF1b、StPIF3、StPIF4、StPIF7a、StPIF8中含有多个乙烯应答元件, StPIF4、StPIF7b、StPIF8含有生长素响应元件。StPIF不同家族成员中含有多个胁迫响应元件, 表明该家族成员可能对调控植物的生长发育发挥着重要作用。对启动子区域可能结合的转录因子进行预测分析发现, StPIF家族成员启动子区域含有多个bZIP、NAC、WARKY、TCP等转录因子结合位点, 已知在胁迫条件下, bZIP转录因子可以与ABA诱导基因启动子区域的ABRE结合来调节下游靶基因的表达[27]; NAC转录因子直接或间接参与干旱、高盐、冷害应答基因的表达, 在植物抗干旱、高盐等非生物逆境胁迫中起重要作用[28]; WRKY转录因子通过调控植物细胞壁的合成、开花时间、种子储藏、物质代谢、种子萌发、植物衰老等过程参与植物生长发育的各个阶段[29]; TCP家族转录因子不仅影响细胞增殖的定位模式, 控制叶形、茎分枝等形态特征, 还能够参与激素的合成和昼夜节律调控[30]。因此, 推测StPIF家族可能参与植物对外界环境的响应, 与启动子区域顺式作用元件预测相一致。
PGSC数据库表达数据显示, StPIF家族成员对温度、干旱等非生物胁迫以及晚疫病等生物胁迫有着明显的响应, 其中, StPIF1b、StPIF3、StPIF4、StPIF7a、StPIF7b在高温处理24 h后明显下调。马铃薯植株对高温较为敏感[31], 若在生长前期遭遇高温天气会推迟块茎的发生, 缩短膨大时间, 进而降低净同化率, 导致产量明显下降[32]。据预测, 由于气候变化带来的生物和非生物胁迫, 到2055年马铃薯产量将大幅度下降[33], 预计2040—2069年世界马铃薯产量将下降18%~32% [34]。因此, 研究高温对马铃薯产量及品质的影响具有重要意义。结合组织表达分析表明, StPIF家族基因在马铃薯的根中几乎不表达, StPIF1a、StPIF1b、StPIF3、StPIF4和StPIF8在叶片和茎中高表达, 但不同家族成员之间存在明显的表达差异(StPIF7a、StPIF7b在不同组织部位表达量均较低, 数据未显示)。对现蕾期的马铃薯植株35℃高温处理10 d后, StPIF1a、StPIF3、StPIF4和StPIF8表达量在叶片、茎及匍匐茎中均明显下调, 然而, StPIF1b在叶片等地上部位及匍匐茎中表达明显上调。在StPIF家族启动子元件中未预测到与高温响应相关元件, 但是存在与胁迫相关转录因子的结合位点。因此, 在StPIF家族启动子区域是否存在新的高温响应元件, StPIF家族是否通过其他转录因子对高温的应答间接响应温度的变化, 以及不同成员之间对高温条件下结薯调控机制是否一致, 值得我们进一步深入探究。
4 结论
本研究鉴定出7个马铃薯PIF家族成员, 分属于5个亚家族, 所有StPIF成员均含有保守的APB结构域和bHLH结构域, 部分成员同时含有APA结构域。基因表达分析表明StPIF家族成员具有明显的组织特异性, 35℃高温处理后StPIF成员有不同的应答模式。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 3]
[本文引用: 2]
[本文引用: 1]
[本文引用: 3]
[本文引用: 3]
[本文引用: 3]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
