 ,, 贾思思, 王加峰, 黄翠红, 王慧, 陈志强, 郭涛
,, 贾思思, 王加峰, 黄翠红, 王慧, 陈志强, 郭涛 ,*华南农业大学/国家植物航天育种工程技术研究中心, 广东广州 510642
,*华南农业大学/国家植物航天育种工程技术研究中心, 广东广州 510642Construction and identification of haploid induction gene OsMATL mutants in rice
WEN Qin ,, JIA Si-Si, WANG Jia-Feng, HUANG Cui-Hong, WANG Hui, CHEN Zhi-Qiang, GUO Tao
,, JIA Si-Si, WANG Jia-Feng, HUANG Cui-Hong, WANG Hui, CHEN Zhi-Qiang, GUO Tao ,*National Engineering Research Center of Plant Space Breeding, South China Agricultural University, Guangzhou 510642, Guangdong, China
,*National Engineering Research Center of Plant Space Breeding, South China Agricultural University, Guangzhou 510642, Guangdong, China通讯作者:
收稿日期:2020-06-29接受日期:2020-11-13网络出版日期:2021-05-12
| 基金资助: |
Received:2020-06-29Accepted:2020-11-13Online:2021-05-12
| Fund supported: |
作者简介 About authors
E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (2474KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
文钦, 贾思思, 王加峰, 黄翠红, 王慧, 陈志强, 郭涛. 水稻单倍体诱导基因OsMATL突变体的创制与分析[J]. 作物学报, 2021, 47(5): 827-836. doi:10.3724/SP.J.1006.2021.02044
WEN Qin, JIA Si-Si, WANG Jia-Feng, HUANG Cui-Hong, WANG Hui, CHEN Zhi-Qiang, GUO Tao.
水稻(Oryza sativa L.)是我国重要的粮食作物之一, 水稻的产量和品质直接影响着我国粮食安全与人民生活品质[1]。水稻品种的高效选育是提高水稻产量、品质和抗性的关键环节。传统育种中需要6~8个世代连续自交和不断选择才能获得相对稳定的材料, 周期较长, 难以满足水稻育种需求。双单倍体育种(double haploid, DH技术)只需2个世代就能获得纯系, 大大缩短了育种进程, 是纯系选育的一次技术革命[2]。单倍体的产生是双单倍体育种的第一步, 自然发生单倍体的频率很低, 仅为0.1%, 无法满足育种的大量需求[3]。在水稻中, 利用花药进行离体培养, 诱导花药产生愈伤组织再分化成单倍体植株, 是产生水稻单倍体的主要方法。但是, 水稻的花药培养具有严格的基因型依赖且操作复杂, 如籼稻的花药培养能力显著弱于粳稻[4]。因此, 水稻单倍体的产生一直是制约着水稻双单倍体育种的瓶颈。
近年来, 在玉米中以生物诱导为基础的DH技术已被国内外种业公司及育种单位广泛的应用, 成为了与分子标记辅助育种和转基因技术并称的现代玉米三大育种技术[5,6]。生物诱导产生孤雌生殖单倍体是利用单倍体诱导系作父本, 与目标选系基础材料进行杂交, 在杂交当代的籽粒上就能获得一定比例的母本血缘单倍体。利用诱导系产生单倍体具有不受基因型依赖、诱导过程简单、成本低廉等优势。在玉米中, 诱导单倍体产生的性状受主效QTL控制, 其中qhir1能够解释66%遗传变异[5]。2017年美国、中国和法国科学家几乎同时克隆qhir1位点的花粉特异性磷脂酶A基因MTL/ZmPLA1/NLD (GRMZM2G471240)[7,8,9]。在玉米单倍体诱导系中, 该基因在第4外显子1572~1573之间有个4 bp (CGAG)插入, 造成20个氨基酸移码突变, 导致翻译提前终止, 产生单倍体诱导功能。
MTL在作物中高度保守, 利用基因编辑技术已成功在玉米[7-8,10]、水稻[11,12]和小麦[13,14]中实现以诱导基因MTL为基础的单倍体诱导。先正达公司[11]对籼稻IR58025B中MTL同源基因OsMATL (Os03g0393900)第1外显子和第4外显子进行编辑, 突变体可诱导产生2%~6%的单倍体。王春等[12]对籼粳杂交稻春优84中OsMATL第4外显子进行编辑后单倍体诱导率为4.44%。玉米中MTL基因不同位点突变后单倍体诱导率不同, 水稻中OsMATL基因不同位点的突变是否会影响单倍体诱导率还有待探索。此外, 水稻材料的不同遗传背景对单倍体诱导率的影响也未知, 因此获得一个高诱导率的水稻单倍体诱导系对水稻双单倍体育种有着重要的意义。本研究以粳稻日本晴和籼稻华航48为研究材料, 利用CRISPR/Cas9技术对OsMATL基因启动子区和编码区的多个靶点进行编辑, 获得了籼粳背景下不同类型的OsMATL突变体, 进一步分析了不同位点的突变效应, 为深入研究水稻孤雌生殖单倍体诱导的机制提供了基础材料。
1 材料与方法
1.1 材料
研究材料为粳稻日本晴和籼稻华航48, 日本晴是典型的粳型纯系品种, 华航48是国家植物航天育种工程技术研究中心选育的综合性状优良的籼型纯系品种。试验材料水培种植于人工气候室(温度28℃, 光照10 h, 黑暗14 h), 所有材料常规管理。1.2 OsMATL生物信息学分析
利用Ensembl数据库(1.3 靶点及引物设计
利用CRISPR-GE网站(图1
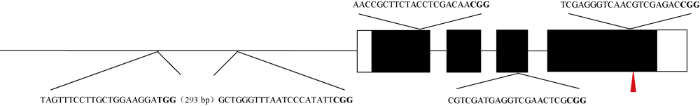 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1OsMATL基因靶点序列及位置
黑色加粗的字母为PAM序列; 红色三角形指向玉米MTL天然突变位点。
Fig. 1Sequence and positions of targets in OsMATL
The black bold letters are the protospacer adjacent motif (PAM) sequences; the red triangle points to the mutation site in the native MTL allele in maize.
Table 1
表1
表1本研究所用引物
Table 1
| 引物名称 Primer name | 引物序列Primer sequence (5'-3') |
|---|---|
| OsMATL-P1-F | GCATCCATGTGTTCCTGATCC |
| OsMATL-P1-R | GGTCCCATTAAGTTTGGCGG |
| OsMATL-P2-F | GGGAGAGTTGGGAACGTTGA |
| OsMATL-P2-R | TGTCGAGGTAGAAGCGGTTG |
| Recom-F | GTGCAGATGATCCGT GGCAACAAAGCACCAGTGGT |
| Recom-R | CTATTTCTAGCTCTAAAAC AAAAAAAAAAGCACCGACTCGGTG |
| OsMATL-Cas9-1-F | taGGTCTCCCTACCTCGACAAgttttagagctagaa |
| OsMATL-Cas9-1-R | cgGGTCTCAGTAGAAGCGGTTtgcaccagccgggaa |
| OsMATL-Cas9-3-F | taGGTCTCCAGGTCGAACTCGgttttagagctagaa |
| OsMATL-Cas9-3-R | cgGGTCTCAACCTCATCGACGtgcaccagccgggaa |
| OsMATL-Cas9-4-F | taGGTCTCCCAACGTCGAGACgttttagagctagaa |
| OsMATL-Cas9-4-R | cgGGTCTCAGTTGACCCTCGAtgcaccagccgggaa |
| OsMATL-Cas9-P1-F | taGGTCTCCTTGCTGGAAGGAgttttagagctagaa |
| OsMATL-Cas9-P1-R | cgGGTCTCAGCAAGGAAACTAtgcaccagccgggaa |
| OsMATL-Cas9-P2-F | taGGTCTCCTAATCCCATATTgttttagagctagaa |
| OsMATL-Cas9-P2-R | cgGGTCTCAATTAAACCCAGCtgcaccagccgggaa |
| Cas9-J-F | AAGCATTTCGTAGTGGGCCA |
| Cas9-J-R | TTTGGTGGTCGCCGTTAGGA |
| OsMATL-1-F | GCATCAGGGGCCTCATCC |
| OsMATL-1-R | GCCCAGCATCTTCCTGATCT |
| OsMATL-3-F | GCGTTGATTCCATCCGCATT |
| OsMATL-3-R | GCTCCTCCTTGTCCTTCACC |
| OsMATL-4-F | GCGCCCATCATCGACATCTT |
| OsMATL-4-R | TGCGTGTACAGTTACTAACGCT |
| OsMATL-P-F | GATCGATCGCCCATAAGCCA |
| OsMATL-P-R | TGACCAACTTGGCGTCATGA |
| q-ACTIN-F | GAATGCTAAGCCAAGAGGAG |
| q-ACTIN-R | AATCACAAGTGAGAACCACAG |
| q-OsMATL-F | CCGAGGTACAACGGCAAGTA |
| q-OsMATL-R | AGAAGATGGTTGGCTGGAGC |
新窗口打开|下载CSV
1.4 CRISPR/Cas9载体构建
参照谢卡斌等[19]利用TPG系统构建CRISPR/Cas9载体的方法。试验步骤如下: (1)初级片段获取。以pGTR质粒为模板, PCR扩增出含有靶点和PTG载体的片段; (2) PTG片段组装。利用Golden Gate克隆方式将初级片段连接起来; (3) pRGEB32载体酶切。利用Bsa I限制性内切酶酶切pRGEB32载体, 获得与PTG相同的黏性末端; (4)重组载体构建。将PTG片段插入到酶切后pRGEB32载体中Bas I克隆位点; (5)重组载体转化。用热激法将重组的载体转入大肠杆菌感受态细胞DH5α中; (6)阳性克隆检测。挑取单个菌落接种培养, 通过测序验证载体正确性。CRISPR/Cas9载体构建如图2所示。图2
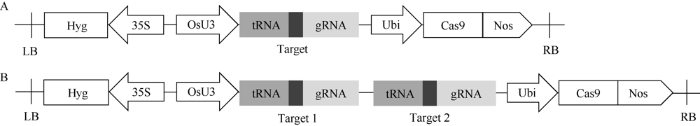 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2CRISPR/Cas9载体构建示意图
A: 单靶点载体; B: 双靶点载体。
Fig. 2Schematic diagram of the CRISPR/Cas9 vector construction
A: single-target vector; B: dual-target vector.
1.5 农杆菌介导遗传转化
参照Hiei等[20]建立的水稻高效农杆菌介导的遗传转化法, 将构建好的CRISPR/Cas9载体通过电击转化农杆菌EHA105中, 3个编码区的单靶点载体分别转化日本晴和华航48成熟胚诱导的愈伤组织中, 1个启动子区的双靶点载体转化到华航48成熟胚诱导的愈伤组织中, 通过潮霉素抗性筛选T0代组培苗。1.6 靶点检测
提取转基因水稻基因组DNA, 在靶点上下游200 bp处设计引物, 对包含靶点目的片段进行扩增和测序, 将测序的结果放入在线解码工具DSDecode (1.7 OsMATL基因表达量分析
收集OsMATL启动子区突变体和野生型散粉前新鲜的花药, 于-80℃保存。采用TRIzol法提取成熟花粉RNA, 参照EvoM-MLV反转录试剂盒II说明书反转录合成cDNA。以cDNA为模板, actin为内参, 参照AceQ qPCR SYBR Green Master Mix试剂说明书, 在荧光定量PCR仪(ABI StepOnePlus)上进行qRT-PCR分析, 采用2-ΔΔCt法计算基因的相对表达量。1.8 突变体结实情况调查
调查OsMATL编码区纯合突变体和OsMATL启动子区大片段缺失突变体主穗上结实情况, 分为实粒(籽粒饱满透明)、败育(只剩薄薄种皮或籽粒皱缩)以及空粒(颖壳内无籽粒), 并统计结实率和败育率。利用SPSS进行单因素方差分析及Duncan’s法多重比较, 差异显著性水平为P≤0.01。2 结果与分析
2.1 OsMATL生物信息学分析
对玉米MTL基因与水稻、高粱、小麦、大麦、小米和拟南芥中的MTL同源基因进行氨基酸序列多重比对(图3)。结果显示, MTL在谷类作物中的同源性较高, 达到70%以上, 玉米中MTL基因与水稻中同源基因OsMATL氨基酸序列相似性为79%, 粳稻和籼稻中OsMATL基因氨基酸序列相似性高达99%。保守的氨基酸序列暗示MTL在谷类作物中具有相似的功能。水稻中OsMATL基因位于3号染色体上, 含有4个外显子, 编码432个氨基酸。该基因编码花粉特异性磷脂酶, 有1个类磷脂酶A2结构域、1个酰基转移酶/酰基水解酶/溶血磷脂酶结构域、1个聚乳酸结构域、3个Motif (GXGXXG、GXSXG和DGA/G), 在C430处有S-棕榈酰化或S-法尼酰化位点, 具有脂类分解代谢和植物防御的功能。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3谷物和拟南芥中MTL同源基因氨基酸序列多重比对
红色方框处为玉米MTL天然突变位点。
Fig. 3Multiple alignment of MTL homologous genes amino sequences in cereals and A. thaliana
The red box highlights the mutation site in the native MTL allele of maize.
OsMATL启动子与其他真核启动子相似, 包含了典型的基础转录调控的顺式作用元件(图4), 有47个TATA-box和36个CAAT-box, 其中TATA-box主要集中在0至-1000 bp处, CAAT-box分散分布在启动子区。还有4种逆境响应元件, 分别为厌氧诱导的ARE元件、GC-motif元件、低温胁迫相关的LTR元件和应激胁迫相关的TC-rich repeats元件, 这些元件可能参与了植物防御。5种(8个)光响应元件, 如Box 4、G-box、MRE、TCT-motif和TCCC-motif。此外还有4种(6个)激素响应元件、1种(2个)生理调控相关元件、1种(2个)蛋白结合位点相关元件和若干功能未知的顺式作用元件。
图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4OsMATL启动子序列及主要顺式作用元件
蓝色字母为TATA-box; 灰色阴影为CAAT-box; 其余元件均用下画线标出, 元件名字在序列下方。
Fig. 4Promoter sequence and major cis-acting elements of OsMATL
The blue letters are TATA-box; the gray shadow shows CAAT-box; other elements are underlined whose name remarks under the sequence.
2.2 CRISPR/Cas9靶点分析
为了获得不同位点的OsMATL突变体, 在OsMATL基因编码区设计3个单靶点(图1), 第1外显子上的靶点距陈绍江团队报道的CRISPR/Cas9靶点下游11 bp, 较为彻底的破坏OsMATL的功能; 第3外显子上的靶点在DGA/G motif处; 第4外显子上的靶点距玉米天然突变位点上游8 bp处, 与先正达公司和王克剑团队在第4外显子上的靶点接近, 此靶点能够减少非预期的影响, 获得具有单倍体诱导功能的突变体。在启动子区域-1200 bp到-1500 bp设计双靶点敲除多个顺式作用元件, 2个靶点之间距离293 bp (图1), 预期获得OsMATL基因表达量具有差异的等位突变。2.3 OsMATL突变位点分析
所有转化均成功获得T0代转基因苗, 对T0代转基因苗进行测序, 筛选OsMATL编码区纯合突变的植株和OsMATL启动子区大片段缺失的植株。OsMATL编码区上一共获得了56种纯合突变体(表2), 纯合突变率为19%。在第1外显子的靶点位置, 日本晴中获得4种纯合突变, 分别为单碱基A和T的插入、8个碱基的缺失、3个碱基的替换和10个碱基的缺失; 华航48中只获得了单碱基A插入的纯合突变。这些突变类型均导致了移码突变, 其中10个碱基的缺失使得翻译在第112个氨基酸处提前终止, 截短了303个氨基酸。对比蛋白质预测的结构(图5), 发现第1外显子上突变体的三级结构与野生型的完全不同, 蛋白结构更简单。Table 2
表2
表2T0代编码区纯合突变及启动子区大片段缺失突变类型
Table 2
| 植株编号 Number of plants | 品种 Variety | 序列 Sequence (5′-3′) | 突变类型 Mutation type |
|---|---|---|---|
| Exon 1 | WT | AACCGCTTCTACCTCGACAACGGCCCACTCATC | |
| 1-1-1 | Nipponbare | AACCGCTTCTACCTCGAACAACGGCCCACTCATC | +1 bp |
| 1-1-2 | Nipponbare | AACCGCTTCTACCTCGATCAACGGCCCACTCATC | +1 bp |
| 1-1-3 | Nipponbare | AACCGCTTCTACCTCGAATTCG --------------------------- ATC | -8 bp and substitute 3 bp |
| 1-1-4 | Nipponbare | AACCGCTT --------------------------------- AACGGCCCACTCATC | -10 bp |
| 1-2-1 | Huahang 48 | AACCGCTTCTACCTCGATCAACGGCCCACTCATC | +1 bp |
| Exon 3 | WT | CGTCGATGAGGTCGAACTCGCGGACCTTGCCGG | |
| 3-1-1 | Nipponbare | CGTCGATGAGGTCGAACTTCGCGGACCTTGCCGG | +1 bp |
| 3-1-2 | Nipponbare | CGTCGATGAGGTCGA-CTCGCGGACCTTGCCGG | -1 bp |
| 3-1-3 | Nipponbare | CGTCGATGAGGTCGAA-TCGCGGACCTTGCCGG | -1 bp |
| 3-1-4 | Nipponbare | CGTCGATGAGGTCGA ------ TCGCGGACCTTGCCGG | -2 bp |
| 3-1-5 | Nipponbare | CGTCGATGAGGTCG -------------------- CGGACCTTGCCGG | -6 bp |
| 3-1-6 | Nipponbare | CGTCGATGAGG ------------------------------ CGGACCTTGCCGG | -9 bp |
| 3-2-1 | Huahang 48 | CGTCGATGAGGTCGAAACTCGCGGACCTTGCCGG | +1 bp |
| 3-2-2 | Huahang 48 | CGTCGATGAGGTCGAA-TCGCGGACCTTGCCGG | -1 bp |
| 3-2-3 | Huahang 48 | CGTCGATGAGGTCGA ----- TCGCGGACCTTGCCGG | -2 bp |
| 3-2-4 | Huahang 48 | CGTCGATGAGGTC ------------ TCGCGGACCTTGCCGG | -4 bp |
| 3-2-5 | Huahang 48 | CGTCGATGAGGT --------------- TCGCGGACCTTGCCGG | -5 bp |
| Exon 4 | WT | TCGAGGGTCAACGTCGAGACCGGCAGGTACGTC | |
| 4-1-1 | Nipponbare | TCGAGGGTCAACGTCGAAGACCGGCAGGTACGTC | +1 bp |
| 4-1-2 | Nipponbare | TCGAGGGTCAACGTCGATGACCGGCAGGTACGTC | +1 bp |
| 4-1-3 | Nipponbare | TCGAGGGTCAACGTC-AGACCGGCAGGTACGTC | -1 bp |
| 4-1-4 | Nipponbare | TCGAGGGTCAACGTC ------- GACCGGCAGGTACGTC | -2 bp |
| 4-2-1 | Huahang 48 | TCGAGGGTCAACGTCGAAGACCGGCAGGTACGTC | +1 bp |
| 4-2-2 | Huahang 48 | TCGAGGGTCAACGTCGATGACCGGCAGGTACGTC | +1 bp |
| 4-2-3 | Huahang 48 | TCGAGGGTCAACGT ------- AGACCGGCAGGTACGTC | -2 bp |
| 4-2-4 | Huahang 48 | TCGAGGGTCAACGTC ------ GAACCGGCAGGTACGTC | -2 bp |
| 4-2-5 | Huahang 48 | TCGAGGGT --------------------------------------------------------------------- ACGTC | -20 bp |
| Promoter | WT | CCATCCTTCCAGCAAGGAAACTA(293 bp)CCGAATATGGGATTAAAC CCAGC | |
| P-1 | Huahang 48 | CCATCCT ------------------------------------- (-292 bp) -------------- ATGGGATTAAACCCAGC | -292 bp |
| P-2 | Huahang 48 | CCATCC ---------------------------------------- (-293 bp) ------------- ATGGGATTAAACCCAGC | -293 bp |
| P-3 | Huahang 48 | CCA -------------------------------------------------- (-304 bp) --------------------------------------- AAACCCAGC | -304 bp |
| P-4 | Huahang 48 | TTAGTTA -------------------------------------- (-306 bp) ------------------- GGGATTAAACCCAGC | -306 bp |
| P-5 | Huahang 48 | TTAGT --------------------------------------------- (-315 bp) ------------------------------------------ AACCCAGC | -315 bp |
| P-6 | Huahang 48 | TTAGT --------------------------------------------- (-363 bp) -------------------- AGGAGTATATAATTA | -363 bp |
| P-7 | Huahang 48 | CCAT ------------------------------------------------ (-722 bp) --------------------------------- TAGTTACGGAG | -722 bp |
新窗口打开|下载CSV
图5
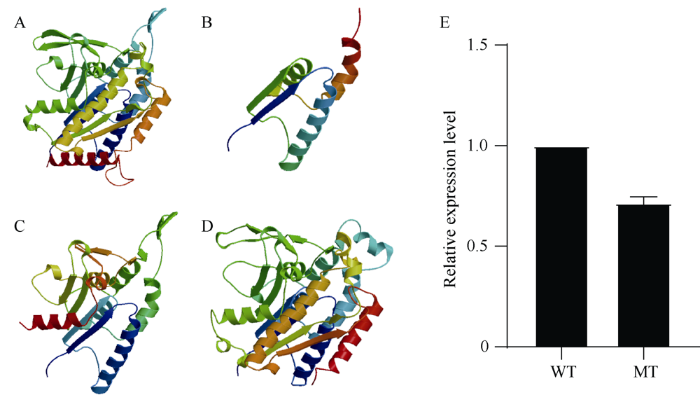 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5野生型和突变体OsMATL基因蛋白三级结构预测及相对表达量
A: 野生型OsMATL基因蛋白三级结构; B: 第1外显子上突变体OsMATL基因蛋白三级结构; C: 第3外显子上突变体OsMATL基因蛋白三级结构; D: 第4外显子上突变体OsMATL基因蛋白三级结构; E: 启动子区293 bp缺失突变体OsMATL基因相对表达量。WT为野生型, MT为突变体。
Fig. 5Tertiary structure prediction and relative expression levels of OsMATL gene in wild type and mutant
A: tertiary structure prediction of OsMATL gene in wild type; B: tertiary structure prediction of OsMATL gene in the first exon mutant; C: tertiary structure prediction of OsMATL gene in the third exon mutant; D: tertiary structure prediction of OsMATL gene in the fourth exon mutant; E: relative expression levels of OsMATL gene in promoter region 293 bp deletion mutant. WT is wild type and MT is mutant.
在第3外显子靶点位置, 获得了单碱基A和T的插入、单碱基和多个碱基缺失的纯合突变。在日本晴中获得6种类型的纯合突变, 其中6个碱基和9个碱基的缺失导致该位点编码氨基酸的缺失, 而其他突变类型均引起移码突变, 使得DGA/G motif丧失, 蛋白翻译出现不同程度的提前终止。对比蛋白质预测的结构(图5), 第3外显子上突变体的蛋白结构较野生型简单, 但比第1外显子上突变体的蛋白结构复杂。
在第4外显子靶点位置, 日本晴和华航48中均获得了单碱基A和T的插入以及2个碱基缺失的纯合突变。此外, 在日本晴中获得了单个碱基缺失的纯合突变, 华航48中获得了20个碱基缺失的纯合突变。这些突变类型均导致了移码突变, 其中单个碱基的缺失导致翻译在第378个氨基酸处提前终止, 其他的移码突变均未导致翻译提前终止。第4外显子上的突变只有少量C端氨基酸改变, 蛋白三级结构与野生型相似(图5), 突变后的蛋白结构细微的差异可能导致催化位点的丧失, 使得蛋白失去了原有的功能。
启动子区获得了7种含有大片段缺失的突变体(表2), 占33.33%。主要为300 bp左右的大片段缺失, 敲除了多个顺式作用元件, 分别为3个TATA-box、2个CAAT-box、4个光响应元件和1个激素响应元件。 此外, 还有1个缺失722 bp的突变体, 删除了多个TATA-box和多种顺式作用元件。进一步对启动子区大片段缺失突变体OsMATL基因进行实时荧光定量PCR分析(图5-E), 结果显示启动子区域293 bp的缺失显著降低了OsMATL基因表达量, 该种类型的突变可能影响OsMATL基因的功能。
2.4 OsMATL突变体结实情况分析
对OsMATL突变体结实情况进行调查(图6和图7), 日本晴和华航48中OsMATL编码区突变体结实率分别为2.87%~6.92%和12.46%~15.26%, 均极显著低于野生型, OsMATL编码区突变后对籼稻结实的影响比粳稻小, 暗示籼稻更能忍OsMATL基因的功能缺失。日本晴中OsMATL编码区突变体败育率极显著增加, 高达51%以上; 华航48中OsMATL编码区突变体败育率为11%~18%, 其中第4外显子上突变体败育率极显著高于野生型。在日本晴和华航48编码区突变体的后代中均发现不同程度的败育籽粒, 大部分只剩薄薄的种皮内部无内含物, 小部分胚乳出现不同程度的皱缩(图7-C, D)。华航48启动子区域大片段的缺失对植株的结实几乎没有影响, 暗示OsMATL基因表达量的改变与编码氨基酸的改变调控双受精的机制可能不同。图6
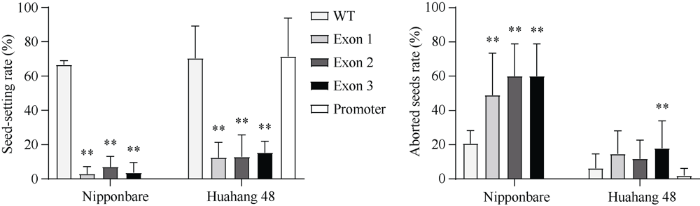 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6野生型和OsMATL突变体的结实率和败育率
**表示OsMATL突变体与野生型之间存在显著差异(P < 0.01)。
Fig. 6Seed setting rate and abortion seed rate of wild type and OsMATL mutant
** indicates significant differences at P < 0.01 between OsMATL mutant and wild type.
图7
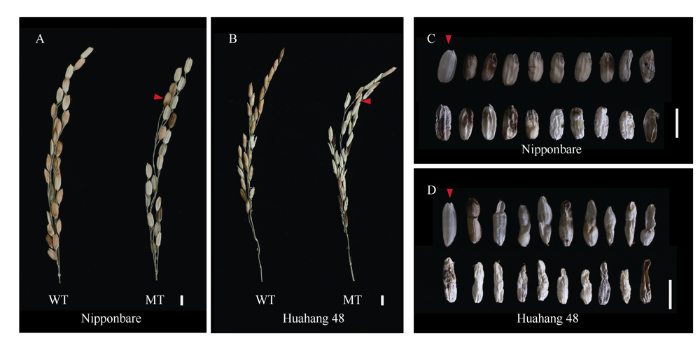 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7结实表型及不同类型败育籽粒表型
A, B: 结实表型; C, D: 败育表型; 红色三角形指向为实粒。标尺为0.5 cm。
Fig. 7Seed setting phenotype and different types of abortion seed phenotypes
A, B: the phenotype of seed setting; C, D: the phenotype of abortion seed; the red triangles point to solid grains. Bar = 0.5 cm.
3 讨论
2017年, 控制玉米单倍体诱导的关键基因MTL几乎同时被3个国家的科学家独立克隆, 单倍体诱导系该基因第4外显子处发生了4 bp的插入, 引起氨基酸的移码突变, 导致MTL蛋白被截短[7,8,9]。全长的MTL蛋白被定位在精细胞膜上, 而突变后截短的MTL蛋白在精细胞膜上并未观察到信号, MTL基因可能参与信号转导或配子融合的过程, 突变后MTL蛋白在精细胞膜上的缺失可能是单倍体诱导的原因[22]。利用基因编辑技术对玉米中的MTL基因进行突变, 证明了编码磷脂酶的MTL基因功能减弱或者丧失能够诱导单倍体的产生。Kelliher等[7]利用TALEN技术对玉米NP2222在自然突变位点附件进行编辑, 使得部分肽段缺失, 单倍体诱导率为6.7%。刘晨旭等[8,23]对ZmPLA1第1外显子进行编辑, 获得单碱基A插入、单碱基缺失和11个碱基缺失的等位突变, 造成较为彻底的基因突变, 单倍体诱导率为2%左右与Stock 6诱导率基本一致, 胚乳败育籽粒频率也显著升高, 证明了MTL基因突变会导致玉米胚乳败育。随后董乐等[10]利用CRISPR/Cas9技术对玉米自交系ZC01的MTL基因天然突变位点上游5 bp处进行靶向突变, 获得了一系列的MTL突变体, 以杂交种ZY7、ZY8和单99为受体测定单倍体诱导率, 验证了所获得的突变体具有较高的单倍体诱导能力, 平均单倍体诱导率为7.32%, 此研究结果与Kelliher等的结果相近, 推测MTL基因突变位点和材料可能会影响单倍体诱导率。MTL在作物中功能保守, 为以单倍体诱导基因为基础的单倍体诱导系统在其他作物中的建立提供了途径。先正达公司率先在水稻中实现单倍体诱导, 通过氨基酸同源序列比对在水稻中找到了MTL同源基因OsMATL, 对籼稻自交系IR58025B中OsMATL基因的第1外显子和第4外显子上天然突变位点下游7 bp处进行编辑, 突变体的结实率为20%, 单倍体诱导率2%~6%左右, 且单倍体诱导率受母本遗传背景的影响, 与玉米MTL突变后结果相似, 证明了MTL在作物中功能保守[10,11]。王春等[12]对籼粳杂交稻春优84中OsMATL基因第4外显子上天然突变位点上游5 bp处进行编辑, 对单碱基C插入纯合突变体以及单碱基C插入和单碱基缺失双等位突变体进行分析, 突变体的结实率为11.5%, 单倍体诱导率为4.4%。刘晨旭等[13]将单倍体诱导系统扩展到六倍体小麦中, 利用CRISPR/Cas9技术对小麦CB037中TaPLA第1外显子进行编辑, 成功获得了小麦A、D基因组磷脂酶基因TaPLA的突变体, 该突变体结实率为30%~60%, 可诱导产生约5%~15%的单倍体, 但同时在自交和杂交过程中还会产生大量的败育籽粒和一定频率的非整倍体籽粒。刘会云等[14]利用优化的SpCas9系统对小麦Fielder和宁春4号的TaMTL基因设计双靶点进行高效的编辑, 获得了小麦A、B和D基因组3个TaMTL基因的突变体, T0代突变体结实率明显降低, 三突结实率仅为19.8%~22.4%, T1代突变体单倍体诱导率可达到18.9%, 并在后代中发现了胚缺失、胚和胚乳双缺失的籽粒表型。利用CRISPR/Cas9技术对作物中单倍体诱导基因进行编辑可快速获得单倍体诱导系, 至此单倍体诱导系统在三大粮食作物上均得以验证。玉米、水稻和小麦中诱导基因突变后单倍体诱导率并不同, 单倍体诱导率可能与突变位点和遗传背景有关, 在不同突变位点和遗传背景的材料中可能存在一些单倍体诱导的修饰位点。本研究对粳稻日本晴和籼稻华航48两个遗传背景不同的材料中OsMATL第1外显子、第3外显子、第4外显子及启动子进行不同位置的编辑, 获得了不同位点和不同遗传背景的OsMATL突变体。本研究中OsMATL编码区的移码突变, 使得氨基酸在不同位置开始发生改变以及蛋白翻译提前终止, 导致蛋白结构发生不同程度的改变, 其中第1外显子上突变后蛋白结构与刘晨旭[23]对玉米MTL基因第1外显子突变后蛋白结构变化相似, 突变后蛋白结构更简单。蛋白结构不同程度的改变可能会影响OsMATL蛋白与细胞膜的结合能力, 从而诱导产生不同频率的单倍体, 为在水稻中获得高频单倍体诱导系奠定了基础。
单倍体诱导系在诱导单倍体的过程中会引起结实率的降低, 同时, 会产生较高频率的败育籽粒。本研究获得的OsMATL编码区纯合突变体的结实率均显著极降低, 首次对粳稻中OsMATL基因进行突变, 发现粳稻OsMATL编码区突变后对结实的影响比籼稻大, 粳稻对OsMATL基因缺失的耐受性较差。先正达公司[11]和王春等[12]对水稻中OsMATL基因进行突变后结实率为10%~20%, 并可诱导产生2%~6%的单倍体。本研究中籼稻突变体的结实率与前人研究结果相似, 可推测本研究中籼稻突变体的单倍体诱导率约为5%。本研究在水稻OsMATL突变体后代中发现了不同程度的败育籽粒, 前人对玉米[7,8,9]和小麦[13,14]中MTL基因进行编辑后均伴随着败育籽粒的产生, 败育与单倍体的形成密切相关[24], 说明MTL基因突变后在不同的作物中均发生了异常双受精。OsMATL编码区突变体结实率的降低和败育籽粒的产生, 暗示本研究在籼稻和粳稻上获得的OsMATL编码区突变体均可能具有单倍体诱导的功能。OsMATL启动子区域大片段的缺失对植株结实并没有显著影响, 启动子大片段缺失的纯合突变是否会造成结实率的降低需要进一步的研究。本研究筛选到不同突变位点和不同遗传背景的OsMATL突变体, 为水稻单倍体诱导系创制提供了基础, 也为进一步研究OsMATL基因的功能提供了材料。
4 结论
以粳稻日本晴和籼稻华航48为研究材料, 本研究利用CRISPR/Cas9基因编辑技术获得OsMATL移码突变和启动子大片段缺失的突变体, OsMATL编码区突变极显著降低了结实率, 但突变效应受到不同位点及遗传背景的影响, 籼稻对于突变的忍受程度高于粳稻。此外, OsMATL启动子区域大片段的缺失对植株的结实几乎没有影响, 暗示OsMATL基因表达量的改变与编码氨基酸的改变调控双受精的机制可能不同。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 5]
[本文引用: 5]
[本文引用: 3]
[本文引用: 3]
[本文引用: 4]
[本文引用: 4]
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
