 ,1,2,**, 毕真真
,1,2,**, 毕真真 ,1,2,**, 孙超1,2, 秦天元1,2, 梁文君1,2, 王一好1,2, 许德蓉1,2, 刘玉汇1,2, 张俊莲1,2, 白江平
,1,2,**, 孙超1,2, 秦天元1,2, 梁文君1,2, 王一好1,2, 许德蓉1,2, 刘玉汇1,2, 张俊莲1,2, 白江平 ,1,2,*
,1,2,*Key genes mining of DNA methylation involved in regulating drought stress response in potato
LI Peng-Cheng ,1,2,**, BI Zhen-Zhen
,1,2,**, BI Zhen-Zhen ,1,2,**, SUN Chao1,2, QIN Tian-Yuan1,2, LIANG Wen-Jun1,2, WANG Yi-Hao1,2, XU De-Rong1,2, LIU Yu-Hui1,2, ZHANG Jun-Lian1,2, BAI Jiang-Ping
,1,2,**, SUN Chao1,2, QIN Tian-Yuan1,2, LIANG Wen-Jun1,2, WANG Yi-Hao1,2, XU De-Rong1,2, LIU Yu-Hui1,2, ZHANG Jun-Lian1,2, BAI Jiang-Ping ,1,2,*
,1,2,*通讯作者:
收稿日期:2020-07-11接受日期:2020-10-14网络出版日期:2021-04-12
| 基金资助: |
First author contact:
Received:2020-07-11Accepted:2020-10-14Online:2021-04-12
| Fund supported: |
作者简介 About authors
李鹏程, E-mail:
毕真真, E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (3516KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李鹏程, 毕真真, 孙超, 秦天元, 梁文君, 王一好, 许德蓉, 刘玉汇, 张俊莲, 白江平. DNA甲基化参与调控马铃薯响应干旱胁迫的关键基因挖掘[J]. 作物学报, 2021, 47(4): 599-612. doi:10.3724/SP.J.1006.2021.04152
LI Peng-Cheng, BI Zhen-Zhen, SUN Chao, QIN Tian-Yuan, LIANG Wen-Jun, WANG Yi-Hao, XU De-Rong, LIU Yu-Hui, ZHANG Jun-Lian, BAI Jiang-Ping.
干旱胁迫通常发生在土壤水分不足以维持植物正常的生命活动且蒸发量大的区域[1]。水分缺乏是限制植物生长发育的关键因素, 影响植物的伸长和膨大生长[2,3]以及土壤养分供应的不平衡等[4]。马铃薯作为四大主粮作物之一, 也是水分敏感作物。中国的马铃薯主要种植在西北干旱和半干旱地区, 因此干旱严重影响了马铃薯的植株生长、块茎大小和商品属性[5]。
近年来, 除了阐明响应干旱胁迫的信号转导机制外, 越来越多的研究表明, 表观遗传调控在植物干旱胁迫响应中具有重要的作用[6,7]。DNA甲基化是发现最早、研究最深入的表观遗传修饰中的一种。DNA甲基化的修饰往往与基因的转录相关, 即DNA甲基化程度越高, 基因的转录水平越低[8,9]。干旱胁迫会引起植物体内DNA甲基化水平的改变, 激活胁迫相关基因的表达、转座子的活性以及调控启动子区域的开关, 以增强植物适应及抵御胁迫的能力[10]。Fei等[11]研究表明, 2个不同耐旱性的小麦品种中的胞质甘油醛-3-磷酸脱氢酶(GAPC)[12]启动子中不同类型和水平的DNA甲基化动态变化与干旱胁迫反应中基因表达之间存在显著关系。Liang等[13]研究表明, 转录因子可能通过DNA甲基化调控从而在毛果杨干旱胁迫反应中发挥重要作用。对耐旱(Bachar)和干旱敏感(F177)蚕豆基因型的全基因组DNA甲基化多态性进行分析, 确定了6个与干旱胁迫相关的差异甲基化区域, 且发现干旱胁迫下Bachar的抗旱相关基因的表达水平高于F177。这表明全基因组DNA甲基化变化可能是蚕豆对响应干旱胁迫的重要调控机制[14]。本试验前期通过5-azadC处理不同干旱敏感的马铃薯品种发现, 干旱胁迫下DNA去甲基化参与调控马铃薯试管苗表型性状及生理生化的响应[15]。而有关DNA甲基化如何影响干旱胁迫下马铃薯相关基因的变化尚不清楚。
通过转录组测序比较不同物种中不同基因型的转录表达是探索非生物胁迫下抗性基因、阐明代谢途径以及解析响应胁迫的耐受性机制的最合适技术之一[16,17]。因此, 本研究通过比较转录组学分析以探讨耐旱型品种青薯9号和干旱敏感型品种大西洋在干旱胁迫和DNA甲基化抑制剂处理下相关基因的表达特性, 以期获得受DNA甲基化调控响应干旱胁迫的相关基因, 从而为更好地阐明DNA甲基化参与调控马铃薯响应干旱胁迫的分子机制, 扩展对马铃薯耐旱机制的认识, 为马铃薯抗旱分子育种的新方法提供一定的理论依据。
1 材料与方法
1.1 试验材料及处理方法
马铃薯耐旱品种为青薯9号(Qingshu 9), 干旱敏感品种为大西洋(Atlantic), 均由甘肃省作物遗传改良与种质创新重点实验室提供。使用纸桥法将2个品种的马铃薯组培苗茎段接种到MS液体培养基(不含琼脂), 每个茎段带有1个腋芽, 生长环境条件为温度(22±2)℃, 光强25.0~37.5 μmol m-2 s-1, 光照16 h d-1。培养24 d后, 用200 mmol L-1甘露醇和60 μmol L-1 DNA甲基化抑制剂(5-azadC)处理马铃薯试管苗, 分别取处理2、6、12和24 h以及未进行处理(对照, 0 h)试管苗的茎叶, 迅速使用液氮冷冻, 存储在-80℃超低温冰箱备用。每个处理3个生物学重复。5-azadC (5-aza-2’-deoxycytidine)购于Sigma公司。1.2 茎叶总RNA提取、文库构建及转录组测序
使用植物总RNA提取TRIzol试剂(Invitgen, 美国)分别提取54个样品?2个品种×[对照+(2种处理×4个时间点)]×3次生物学重复}的总RNA, 用Turbo DNase I (Ambion, 美国)处理30 min后使用RNeasy Plant Mini Kit (QIAGEN, 德国)进行纯化; 然后使用TruSeq RNA sample Prep V2 kit (Illumina, 美国)构建转录组测序cDNA文库, 并利用Agilent 2200 TapeStation (Agilent, 美国)系统检测cDNA文库质量; 最后使用Illumina HiSeqX10测序仪上进行双端测序。1.3 转录组数据分析
首先使用FASTQC对下机数据进行质控并计算Q20、Q30和GC含量。然后从ENSEMBL植物数据库中下载了马铃薯参考基因组序列(SolTub 3.0)和注释文件(gtf), 利用HISAT2将测序reads比对到马铃薯参考基因组中, 获得sam文件; 利用Samtools 工具将sam文件转换为bam文件并排序, 使用Cufflinks对排序后的bam文件进行转录本的组装和定量; 使用HTseq-count计算各样本转录本的counts数, 利用Python脚本计算参考基因组各基因的长度, 使用R包Dseq2计算各基因的TPM (Transcripts Per Million, 每百万条reads的转录本)和样本间的差异表达分析。将校正后的P<0.01和变化倍数大于2定义为差异表达基因(DEG), 通过比较青薯9号和大西洋在同一处理同一时间点的表达水平来确定差异表达基因。采用在线工具g:Profiler[18] (1.4 启动子区甲基化CpG岛预测和顺式作用元件分析
先使用perl脚本批量提取所分析基因上游1500 bp启动子序列, 然后利用在线软件MethPrimer[19] (1.5 实时荧光定量PCR验证
随机选择22个差异基因, 使用Primer Express 3.0.1设计引物(表1), 进行实时荧光定量PCR分析。采用cDNA合成试剂盒9 (TaKaRa, 日本)将各样品3次生物学重复的RNA反转录为cDNA, 以Actin为内参, 利用QuantStudio5实时荧光定量PCR系统 (ABI, 美国)进行荧光定量检测。每个样品3次生物学重复, 按2-ΔΔCT相对定量法计算相对表达量。Table 1
表1
表1实时荧光定量PCR所用的引物
Table 1
| 基因 Gene | 正向引物 Forward primer (5'-3') | 反向引物 Reverse primer (5'-3') |
|---|---|---|
| Actin | AGGAGCATCCTGTCCTCCTAA | CACCATCACCAGAGTCCAACA |
| PGSC0003DMG400001333 | CTCAATTGCACCTACTAAGCAC | GTGATATTCTTGCATGCAGAGG |
| PGSC0003DMG400005108 | GTTGTGTTCTTGGTTTTAGGCT | GTGGCATATTCATGGGCATAAG |
| PGSC0003DMG400005112 | TCGAATTCTTGTCAACCTGGTA | CCTGGTGGATCATAATTGCAAG |
| PGSC0003DMG400005116 | CACAATGAAGCTCGTAGACAAG | CACATACTCTACCTGCTTGACA |
| PGSC0003DMG400017231 | CACGAATTCCAAGAAAAGCAGT | CCCCAAATCTTCAAACACAACT |
| PGSC0003DMG400023921 | CAACAAATGTGCTAGTGGGAAA | TGTTTCCTGAAGGAGCATAGTT |
| PGSC0003DMG400002176 | GCAGTCAAGTCAAGAAATATCTACC | TGAACACGTACAAGTAAGTGGA |
| PGSC0003DMG400004977 | GATTGATGATGTGAAGAGTGCC | TGTCATAATGAAGGCCTTGAGT |
| PGSC0003DMG400011012 | AATTTTTGTGTGTGGAGGGATG | TATTCATTTGAGAGTAGCCGCA |
| PGSC0003DMG400016722 | AACTAACAGGAACCACACTG | GTTGTGTCCCATTTTAGGGA |
| PGSC0003DMG400020253 | GCTGGAAGGTTCATGAAAGATG | GGAATGGATATTCTCAATGGCG |
| PGSC0003DMG400024232 | GAGCAGGATTTAGCCGTTTTAG | AGGTGCAATTACTCCGTAGAAA |
| PGSC0003DMG400006270 | ATGCAATTGCTGGTGCTTATAG | AGCCTGATAGCAAGTAACAGTT |
| PGSC0003DMG400030038 | ATGAAGTTTAGGGGTGACTCAG | TACACAAACTTCCCGTATGTGA |
| PGSC0003DMG400030624 | TGCTGAAAGTTCATATCCTGGT | GAAGTGTCTTGATGCATAGCAG |
| PGSC0003DMG403001316 | GATGCTGTGAATCACCCAAATT | GATGGTGAACGTATACAACACG |
| PGSC0003DMG401000287 | CTTCATTTGACTGGCCTCATTC | TCATCAAAAGCACAACCAAGAG |
| PGSC0003DMG401023603 | TTAACTGATGAACCGACTTGGA | CAATTGTGAGACCAATCGAGAC |
| PGSC0003DMG400013765 | TAAGTGCCAGAAGATTGGTCAT | TACATCTCTTGCTCCTCAATCG |
| PGSC0003DMG400019274 | CTGGGCTTTATAGGTATCGTGT | ATATGAAGTTGAATTGAGGCGC |
| PGSC0003DMG400020139 | AGCAACAAAAGACCTTCATCAC | CATGCTCACTTTAACGTAGCTC |
| PGSC0003DMG400026261 | GGATTTGAATTCGAGACGTCTG | TCGTTGTAGAAATCATCGCAAC |
新窗口打开|下载CSV
2 结果与分析
2.1 不同马铃薯品种干旱和DNA甲基化处理下差异表达基因的鉴定与分析
在数据过滤和质量评估后, 54个样品产生了大约2547百万的reads数, 平均每个样本约产生7.1 G碱基, GC含量均在44.17%~46.83%范围之间。应用RNA-seq分析比对软件HISAT2将各样本所有的reads与马铃薯参考基因组序列进行比对, 比对率为75.56%~94.64% (表2)。Table 2
表2
表2测序reads数比对到参考基因组的基本统计
Table 2
| 样品ID Sample ID | 总reads数 Total reads | 总碱基数 Total bases (G) | GC含量 GC content (%) | Q20 (%) | Q30 (%) | 比对率 Mapped reads (%) |
|---|---|---|---|---|---|---|
| AtC_0h | 132,318,050 | 19.85 | 44.33 | 99.93 | 96.45 | 82.13 |
| AtA_2h | 149,098,932 | 22.36 | 44.33 | 99.95 | 96.58 | 89.28 |
| AtA_6h | 157,099,124 | 23.56 | 44.50 | 99.94 | 96.40 | 84.40 |
| AtA_12h | 137,418,600 | 20.61 | 44.67 | 99.94 | 96.42 | 91.26 |
| AtA_24h | 146,797,368 | 22.02 | 45.67 | 99.95 | 96.59 | 91.93 |
| AtM_2h | 137,686,150 | 20.65 | 46.83 | 99.95 | 96.76 | 94.64 |
| AtM_6h | 147,166,226 | 22.07 | 45.67 | 99.91 | 96.45 | 79.18 |
| AtM_12h | 132,018,290 | 19.80 | 44.17 | 99.91 | 96.20 | 78.02 |
| AtM_24h | 143,830,794 | 21.57 | 44.83 | 99.94 | 96.43 | 87.10 |
| QsC_0h | 137,904,066 | 20.69 | 44.67 | 99.90 | 96.41 | 82.46 |
| QsA_2h | 133,480,780 | 20.02 | 45.67 | 99.93 | 96.68 | 92.56 |
| QsA_6h | 143,833,564 | 21.58 | 45.00 | 99.94 | 96.47 | 92.32 |
| QsA_12h | 150,781,586 | 22.62 | 44.67 | 99.93 | 96.41 | 84.35 |
| QsA_24h | 134,038,164 | 20.11 | 44.17 | 99.92 | 96.42 | 75.56 |
| QsM_2h | 145,819,266 | 21.87 | 44.83 | 99.92 | 96.43 | 81.50 |
| QsM_6h | 136,496,172 | 20.47 | 44.33 | 99.92 | 96.30 | 89.37 |
| QsM_12h | 130,599,502 | 19.59 | 44.50 | 99.93 | 96.40 | 85.07 |
| QsM_24h | 150,207,156 | 22.53 | 44.33 | 99.94 | 96.42 | 90.72 |
新窗口打开|下载CSV
为了分析不同耐旱型马铃薯品种在甘露醇和5-Azadc处理期间转录水平的差异, 使用R包Dseq2对大西洋和青薯9号在同一时间样本的TPM值计算了所有差异基因的表达。以Flod-change>2和校正后P<0.01作为差异表达基因 (DEG)的筛选标准。结果表明, 在对照 (0 h)下, A vs. Q中筛选到1030个DEG, 其中有515个上调和515个下调表达基因。干旱处理下, 在A vs. Q 2 h中筛选到568个DEG, 其中有384个上调表达基因, 184个下调表达基因, 在A vs. Q 6 h中筛选到994个DEG, 其中有560个上调表达基因, 434个下调表达基因, 在A vs. Q 12 h中筛选到519个DEG, 其中有321个上调表达基因, 198个下调表达基因, 在A vs. Q 24 h中筛选到1251个DEG, 其中有471个上调表达基因, 780个下调表达基因。DNA去甲基化处理下, 在A vs. Q 2 h中筛选到1603个DEG, 其中有1033个上调表达基因, 570个下调表达基因。在A vs. Q 6 h中筛选到501个DEG, 其中有641个上调表达基因, 434个下调表达基因。在A vs. Q 12 h中筛选到1153个DEG, 其中有576个上调表达基因, 577个下调表达基因。在A vs. Q 24 h中筛选到981个DEG 其中有465个上调表达基因, 516个下调表达基因(表3)。
Table 3
表3
表3干旱、DNA去甲基化处理下不同品系间差异表达基因数量
Table 3
| 处理 Treatment | 比较组合 Comparison | 差异基因数 Number of DEGs | 上调差异基因数 Up-regulated DEGs | 下调差异基因数 Down-regulated DEGs |
|---|---|---|---|---|
| 对照Control | A vs. Q_0h | 1030 | 515 | 515 |
| 干旱Drought | A vs. Q_2h | 568 | 384 | 184 |
| A vs. Q_6h | 994 | 560 | 434 | |
| A vs. Q_12h | 519 | 321 | 198 | |
| A vs. Q_24h | 1251 | 471 | 780 | |
| DNA去甲基化 Demethylation | A vs. Q_2h | 1603 | 1033 | 570 |
| A vs. Q_6h | 1142 | 501 | 641 | |
| A vs. Q_12h | 1153 | 576 | 577 | |
| A vs. Q_24h | 981 | 465 | 516 |
新窗口打开|下载CSV
为了进一步分析马铃薯不同品种在不同处理下特定和共同差异表达基因变化。利用R语言分别绘制了干旱和DNA去甲基化处理下的Upset图(图1-A, B)。与对照相比, 干旱处理下, A vs. Q在2、6、12和24 h时差异表达基因总数为1790, 在2、6、12和24 h中特异表达的差异基因为170、432、175和507个, 在4个时间点共同表达的基因有17个; DNA去甲基化处理下, A vs. Q在2、6、12和24 h中差异表达基因总数为2617, 在2、6、12和24 h中特异表达的差异基因为890、435、345和291个, 在这4个时间点共同表达的基因有29个。同时绘制了对照、干旱和DNA甲基化抑制剂处理下A vs. Q差异表达基因的韦恩图(图1-C), 干旱和DNA甲基化抑制剂处理下共同表达的基因有1354个, 表明这1354个基因可能受DNA甲基化调控参与马铃薯干旱响应, 可作为后续基因挖掘的重点部分。
图1
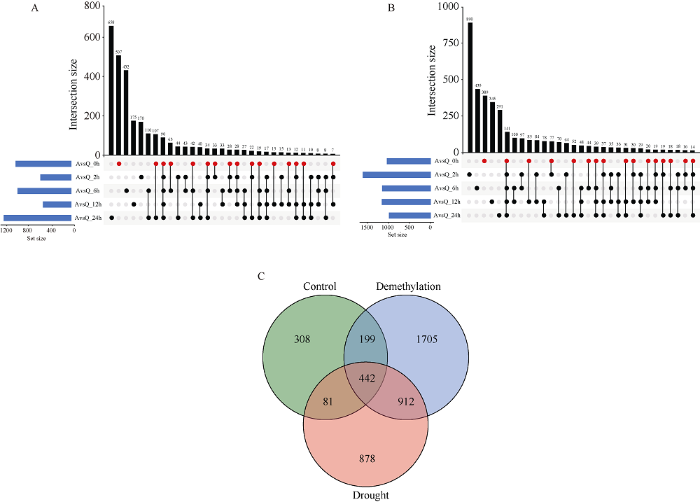 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1马铃薯干旱胁迫和DNA去甲基化处理下差异基因表达谱
A和B是UpSet图, 分别表示干旱和DNA去甲基化处理下AvsQ各时间点比较得到的集合的重叠和DEG的数量。C是韦恩图。
Fig. 1Expression profile of regulated genes under drought stress and DNA demethylation in potato
A and B are UpSet diagrams, showing the overlap of sets and the number of DEG obtained by comparison of AvsQ at each time point under drought and DNA demethylation, respectively. C is the venn diagram.
2.2 不同马铃薯品种干旱和DNA甲基化处理下差异表达基因GO分析
甘露醇处理下, 对A vs. Q在2、6、12和24 h中共1790个差异表达基因进行GO功能富集分析发现, 这些差异基因主要富集包括生物过程、分子功能、细胞组分三大分类下的42个GO term (图2-A)。生物学过程共富集到15个GO term, 主要是苯丙烷类分解代谢过程(GO:0046274和GO:0009698)、木质素代谢过程(GO:0009808)、氧化还原过程(GO:0055114)、蛋白质磷酸化(GO:0006468)、植物型细胞壁组织或生物发生(GO:0071669)和碳水化合物代谢过程(GO:0005975)。分子功能共富集到22个GO term, 与催化活性(GO:0003824)、氧化还原酶活性(GO:0016491和GO:0016682)、碳水化合物结合(GO:0030246)、水解O-糖基化合物的水解酶活性(GO:0004553和GO:0016798)、天冬氨酸肽酶活性(GO:0070001和GO:0004190)、蛋白丝氨酸/苏氨酸激酶活性(GO:0004674)、木糖基转移酶活性(GO:0016762)、蛋白激酶活性(GO:0004672)、内肽酶抑制剂活性(GO:0004866)、钙调素结合(GO: 0005516)和磷酸转移酶活性(GO:0016773)相关。细胞组分共富集到5个GO term, 主要是质外体(GO:0005576)、细胞膜(GO:0005576和GO:0030312)和细胞壁(GO:0005618)。同样地, 对DNA甲基化抑制剂处理下, A vs. Q在2、6、12和24 h共2617个差异表达基因进行GO功能富集分析(图2-B)发现, 这些差异基因也显著富集到生物过程、分子功能、细胞组分下的39个GO term。其中富集到生物学过程的共16个, 主要包括氧化还原过程(GO: 0055114)、碳水化合物代谢过程(GO:0005975)、糖代谢过程(GO:0010411、GO:0006073、GO:0044042、GO: 0010410、GO:0044264、GO:0010383和GO:0005976)和蛋白水解(GO:0045861)过程。分子功能共富集到18个GO term, 与催化活性(GO:0003824)、加氧酶活性(GO:0004497)、氧化还原酶活性(GO:0016491)、血红素结合(GO:0020037)、木糖基转移酶活性(GO: 0016762)、辅酶因子结合(GO:0048037)、水解O-糖基化合物的水解酶活性(GO:0004553)和内肽酶抑制剂活性(GO:0004866)相关。细胞组分共富集到5个GO term, 主要是是质外体(GO:0005576)、细胞壁(GO: 0005618)和细胞膜(GO:0005576和GO:0030312)。图2
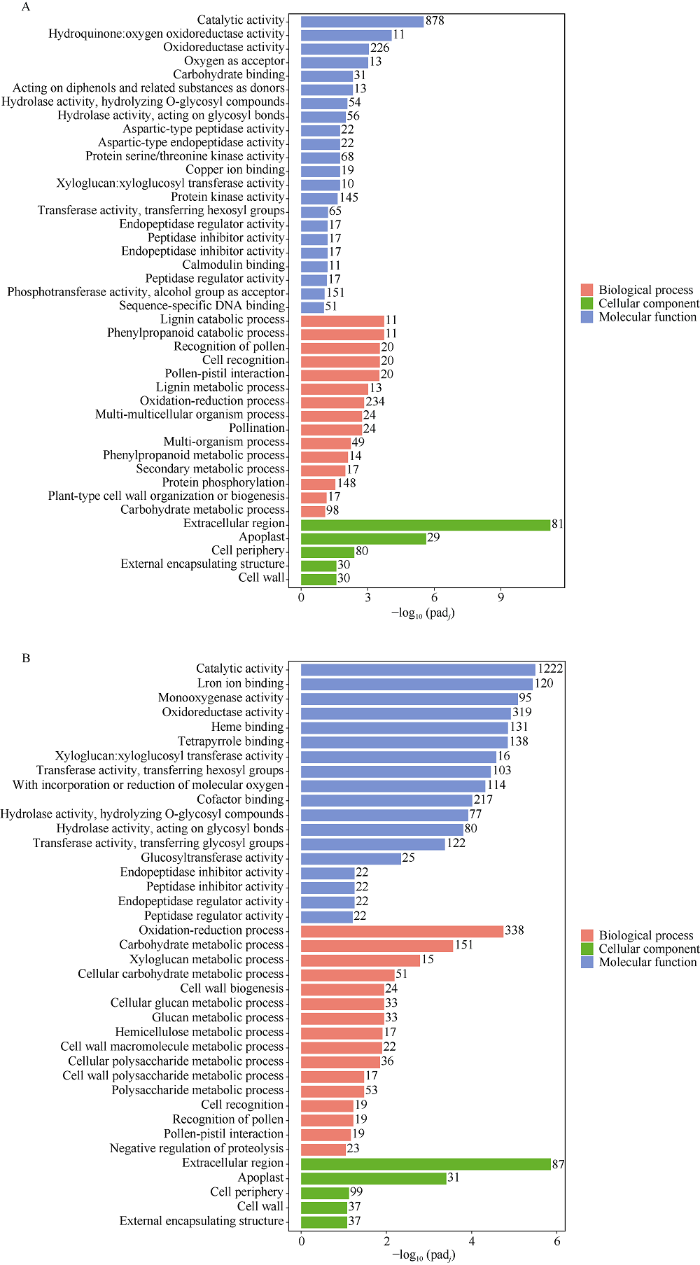 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2差异基因的GO功能富集分析
Fig. 2GO functional enrichment analysis of differential expressed genes
同时比较干旱处理和去甲基化处理下所富集的GO term发现, 2种处理都共同显著富集到催化活性(GO:0003824)、氧化还原酶活性(GO:0016491和GO:0016682)、木糖基转移酶活性(GO:0016762)、内肽酶抑制剂活性(GO:0004866)、碳水化合物代谢过程、质外体(GO:0005576)、细胞壁(GO:0005618)和细胞膜(GO:0005576和GO:0030312)。
2.3 两种处理下共同差异表达基因表达聚类分析
聚类分析主要是把表达模式一致的差异表达基因归为一类。以干旱和DNA甲基化抑制剂处理下A vs. Q共同差异表达的1354个基因的表达量为目标, 对各样本进行表达层次聚类分析。由图3可知, A vs. Q在干旱和DNA甲基化抑制剂处理下的表达模式基本一致。表达趋势主要分为: 2种处理下持续下调表达基因, 说明与马铃薯响应干旱胁迫相关的负向调节基因受到DNA去甲基化的负调控; 以及2种处理下持续上调表达基因, 说明与马铃薯响应干旱胁迫相关的正向调节基因受到DNA去甲基化的正调控。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3两种处理下共同差异基因层次聚类分析
Fig. 3Hierarchical cluster analysis of common differential genes under two treatments
2.4 两种处理下共同差异表达基因KEGG功能富集分析
为了进一步分析DNA甲基化参与调控马铃薯干旱胁迫响应的相关基因的生物学功能, 利用KEGG数据库对2个马铃薯品种在甘露醇和DNA甲基化抑制剂处理下共同差异表达的基因进行通路分析发现, 1345个差异表达基因显著富集到12个KEGG pathways (图4-A), 其中与植物抗旱相关的通路有5个, 分别是植物MAPK信号通路(KO04016, 富集到DEG有11个)、谷胱甘肽代谢(KO00480, 富集到DEG有8个)、糖酵解/糖异生(KO00010, 富集到DEG有8个)、磷酸肌醇代谢(KO00562, 富集到DEG有5个)、植物激素信号转导通路(KO04075, 富集到DEG有14个)。通过pathview绘制富集通路的基因在干旱和去甲基化处理下的表达模式和调控网络发现, 大西洋和青薯9号在响应干旱处理和DNA去甲基化处理下, 谷胱甘肽代谢(图4-B)和磷酸肌醇代谢(图4-C)通路中差异基因多为上调表达基因, 且2种处理下各时间点表达趋势类似。而MAPK信号(图4-D)、糖酵解/糖异生代谢(图4-E)和植物激素信号转导(图4-F)通路中差异基因多为下调表达基因。这些基因的功能注释信息如表4所示。图4
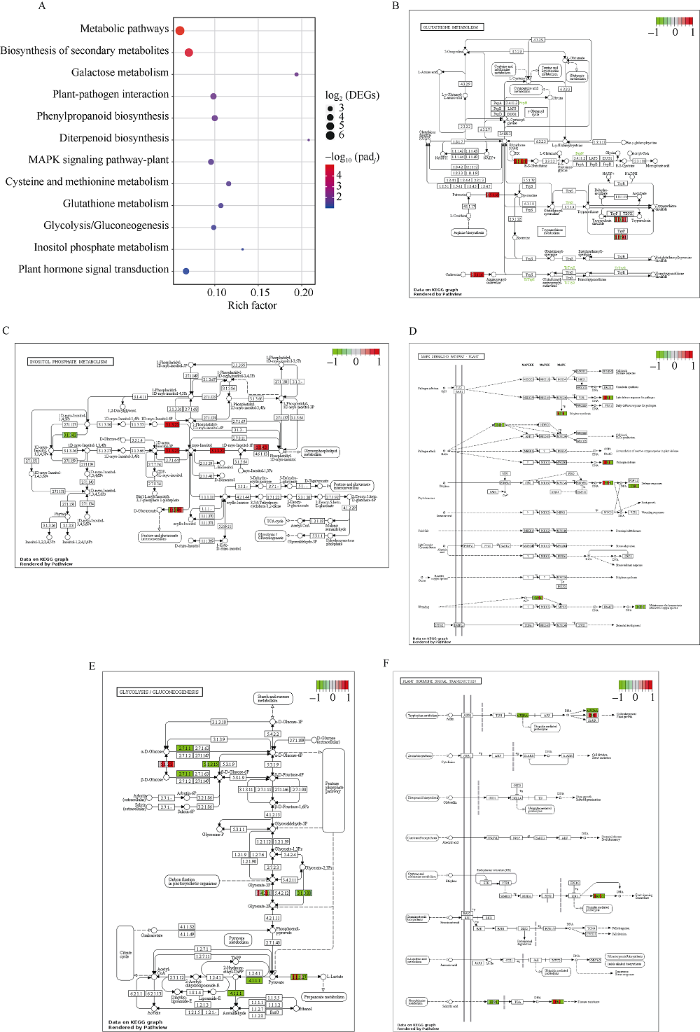 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4两种处理下共同差异表达基因KEGG功能富集分析
Fig. 4KEGG functional enrichment analysis of common differential genes under two treatments
Table 4
表4
表4共同富集KEGG与干旱相关通路基因功能注释
Table 4
| KEGG通路ID KEGG ID | 基因ID Gene ID | 对应拟南芥基因ID Gene in A. thaliana | 基因功能 Gene function |
|---|---|---|---|
| KO04016 | PGSC0003DMG400000193 | AT3G61510 | 1-氨基环丙烷-1-羧酸合酶2 1-aminocyclopropane-1-carboxylate synthase 2 |
| KO04016 | PGSC0003DMG400006639 | AT3G25250 | 类丝氨酸/苏氨酸蛋白激酶 OXI1 serine/threonine-protein kinase OXI1-like |
| KO04016 | PGSC0003DMG400001333 | AT5G39670 | Ca2+结合蛋白 Ca2+ binding protein |
| KO04016 | PGSC0003DMG400001529 | AT3G12500 | 酸性27-kD内切酶 Acidic 27-kD endochitinase |
| KO04016 | PGSC0003DMG400005108 | AT3G19690 | 致病相关蛋白1 pathogenesis-related protein 1-like |
| KO04016 | PGSC0003DMG400005112 | AT4G33720 | 碱性PR-1蛋白 Basic PR-1 protein |
| KO04016 | PGSC0003DMG400005116 | AT2G14580 | PR1蛋白 PR1 protein |
| KO04016 | PGSC0003DMG400017231 | AT3G23240 | 转录因子TSRF1 Transcription factor TSRF1 |
| KO04016 | PGSC0003DMG400001204 | AT5G49480 | Ca2+结合蛋白1 Ca2+-binding protein 1 |
| KO04016 | PGSC0003DMG400005110 | AT3G19690 | PR1蛋白 PR1 protein |
| KO04016 | PGSC0003DMG400023921 | AT2G14610 | 细胞质小热休克蛋白I类 Cytoplasmic small heat shock protein class I |
| KO00480 | PGSC0003DMG400031091 | AT1G17180 | 谷胱甘肽S转移酶 Glutathione S-transferase |
| KO00480 | PGSC0003DMG400024232 | AT1G23820 | 亚精胺合成酶1 Spermidine synthase 1 |
| KO00480 | PGSC0003DMG400011012 | AT2G29490 | 谷胱甘肽S转移酶 Glutathione S-transferase |
| KO00480 | PGSC0003DMG400016722 | AT3G03190 | 谷胱甘肽S转移酶 Glutathione S-transferase |
| KO00480 | PGSC0003DMG400004977 | AT5G41210 | 谷胱甘肽S转移酶 Glutathione S-transferase |
| KO00480 | PGSC0003DMG400002169 | AT3G09270 | 谷胱甘肽S转移酶 Glutathione S-transferase |
| KO00480 | PGSC0003DMG400002176 | AT3G09270 | 谷胱甘肽S转移酶 Glutathione S-transferase |
| KO00480 | PGSC0003DMG400020253 | AT3G27060 | 核糖核苷二磷酸还原酶小链 Ribonucleoside-diphosphate reductase small chain |
| KO00010 | PGSC0003DMG400009472 | AT1G22170 | 磷酸甘油酸变位酶 Phosphoglycerate mutase |
| KO00010 | PGSC0003DMG400006270 | AT4G33070 | 丙酮酸脱羧酶 Pyruvate decarboxylase |
| KO00010 | PGSC0003DMG400030038 | AT1G09870 | 多肌醇多磷酸磷酸酶1 Multiple inositol polyphosphate phosphatase 1 |
| KO00010 | PGSC0003DMG400030624 | AT4G37840 | 己糖激酶 Hexokinase |
| KO00010 | PGSC0003DMG400018084 | AT3G47800 | 醛糖-1-表异构酶 Aldose-1-epimerase |
| KO00010 | PGSC0003DMG403001316 | AT5G15140 | 非细胞自主蛋白途径2 Non-cell-autonomous protein pathway 2 |
| KO00010 | PGSC0003DMG400012308 | AT5G57330 | 醛糖1-表异构酶 Aldose 1-epimerase |
| KEGG通路ID KEGG ID | 基因ID Gene ID | 对应拟南芥基因ID Gene in A. thaliana | 基因功能 Gene function |
| KO00010 | PGSC0003DMG400004800 | AT4G17260 | L-乳酸脱氢酶B L-lactate dehydrogenase B |
| KO00562 | PGSC0003DMG400022715 | AT2G26870 | 磷酸酯酶家族蛋白 Phosphoesterase family protein |
| KO00562 | PGSC0003DMG401000287 | AT4G26260 | 肌醇加氧酶 Myo-inositol oxygenase |
| KO00562 | PGSC0003DMG400004872 | AT1G14520 | 肌醇加氧酶 Myo-inositol oxygenase |
| KO00562 | PGSC0003DMG400030038 | AT1G09870 | 多肌醇多磷酸磷酸酶1 Multiple inositol polyphosphate phosphatase 1 |
| KO00562 | PGSC0003DMG401023603 | AT3G02870 | 肌醇单磷酸酶1 Inositol monophosphatase 1 |
| KO04075 | PGSC0003DMG400019274 | AT5G54510 | 吲哚-3-乙酸酰胺合成酶GH3.6 Indole-3-acetic acid-amido synthetase GH3.6 |
| KO04075 | PGSC0003DMG400005108 | AT3G19690 | 致病相关蛋白1 pathogenesis-related protein 1-like |
| KO04075 | PGSC0003DMG400005112 | AT4G33720 | 碱性PR-1蛋白 Basic PR-1 protein |
| KO04075 | PGSC0003DMG400021210 | AT5G45110 | NIM1 1 |
| KO04075 | PGSC0003DMG400020139 | AT5G43700 | 生长素诱导蛋白22B Auxin-induced protein 22B |
| KO04075 | PGSC0003DMG400005116 | AT2G14580 | PR1蛋白 PR1 protein |
| KO04075 | PGSC0003DMG400017231 | AT3G23240 | 转录因子TSRF1 Transcription factor TSRF1 |
| KO04075 | PGSC0003DMG400005110 | AT3G19690 | PR1蛋白 PR1 protein |
| KO04075 | PGSC0003DMG400021676 | AT4G03400 | 吲哚-3-乙酸酰胺合成酶GH3.5 Indole-3-acetic acid-amido synthetase GH3.5 |
| KO04075 | PGSC0003DMG400025856 | AT2G14960 | 生长素和乙烯响应性GH3 Auxin and ethylene responsive GH3 |
| KO04075 | PGSC0003DMG400023921 | AT2G14610 | 细胞质小热休克蛋白I类 Cytoplasmic small heat shock protein class I |
| KO04075 | PGSC0003DMG400026261 | AT5G47220 | ATERF-2/ATERF2/ERF2 |
| KO04075 | PGSC0003DMG400024978 | AT2G14960 | 吲哚-3-乙酸酰胺合成酶GH3.3 Indole-3-acetic acid-amido synthetase GH3.3 |
| KO04075 | PGSC0003DMG400013765 | AT4G32280 | ATP结合蛋白 ATP binding protein |
新窗口打开|下载CSV
2.5 两种处理下共同差异表达基因启动子分析
对KEGG富集到与植物抗旱相关的5个通路中的39个基因, 进行启动子区CpG岛预测和顺式作用元件分析。首先, 从马铃薯基因组序列中提取39个基因1500 bp的启动子序列。使用在线软件MethPrimer预测这些基因的启动子区域的CpG岛发现, 只有1个与植物谷胱甘肽代谢通路相关的基因 (PGSC0003DMG400004977)含有CpG岛, 位于该基因启动子序列的-736~ -866之间, 大小为131 bp (图5)。同时, 使用在线数据库Plant Care对该基因启动子序列进行顺式作用元件分析发现, 在PGSC 0003DMG400004977基因的CpG岛区域有ABRE、CAAT-box、A-box元件, 其中ABRE和CAAT-box与植物响应干旱胁迫相关。表明, 马铃薯在响应干旱胁迫时, 谷胱甘肽转移酶基因启动子区的ABRE和CAAT-box作用元件通过DNA去甲基化激活该基因的表达以应对逆境胁迫。图5
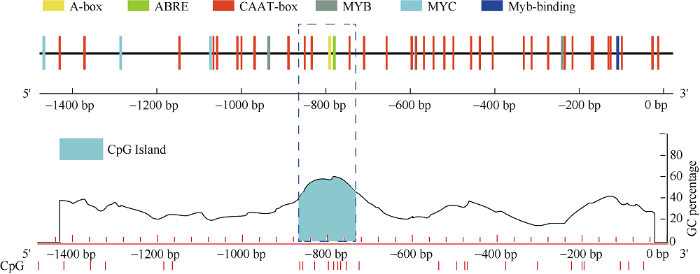 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5谷胱甘肽S转移酶基因启动子分析
Fig. 5Analysis of glutathione S-transferase gene promoter
2.6 qRT-PCR验证
从KEGG显著富集到的5条干旱相关通路的基因中随机挑选22个基因进行实时荧光定量PCR验证。利用反转录cDNA试剂盒将转录组测序时保存的RNA样品进行反转录, 使用qRT-PCR检测这些基因在不同处理下在大西洋和青薯9号中的相对表达量。结果表明, qRT-PCR结果和转录组测序结果变化趋势基本一致(图6)。说明转录组测序数据的可靠性, 并进一步验证了这些基因对干旱和DNA甲基化抑制剂的响应。图6
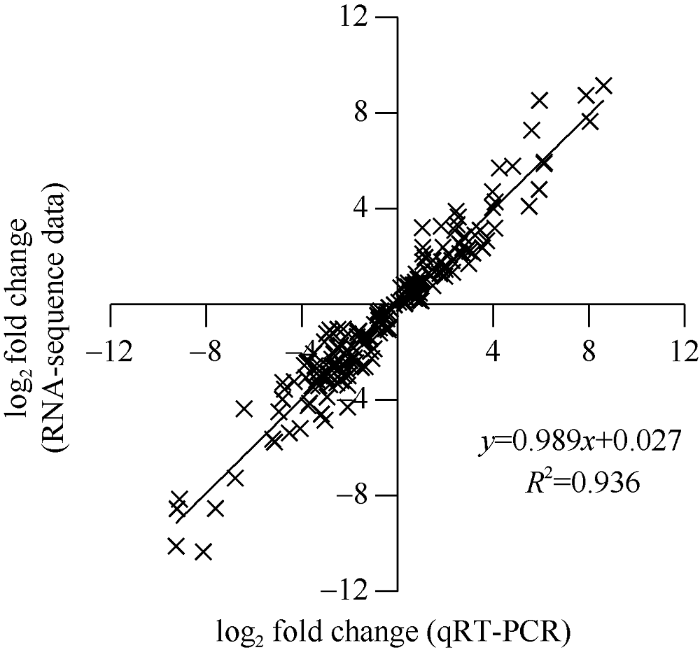 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6干旱和5-azadC处理下差异表达基因的qRT-PCR验证
Fig. 6qRT-PCR validation of differentially expressed genes under drought and 5-azadC treatment
3 讨论
植物对干旱胁迫反应的分子机制包括转录前表观遗传调控和转录后激素、转录因子、胁迫反应基因等信号分子的参与。研究发现, 植物通过基因组DNA甲基化和去甲基化的动态变化来迅速适应外部干旱环境, 表明DNA甲基化调控在植物响应干旱胁迫中起重要作用[21]。本研究利用甘露醇和DNA甲基化抑制剂分别处理2种耐旱性不同的马铃薯试管苗, 通过比较RNA-seq分析2个品种转录本在相同处理和不同处理下5个时间点的基因表达变化, 在分子水平探究DNA甲基化如何参与调控马铃薯对干旱胁迫的响应。植物通过升高或降低DNA在特定位点发生特定类型的甲基化水平, 来打开或关闭一系列与逆境响应相关的基因, 进而启动胁迫应答通路, 提高响应逆境相关酶活性, 从而防御逆境胁迫对植物功能基因表达造成的影响, 保护植物自身的生长发育[15]。植物对干旱胁迫的耐受性是由复杂的多组分信号通路触发, 以恢复细胞动态平衡并促进植物生存[22]。信号转导的一般途径包括由应激特异性受体(如G蛋白偶联受体、蛋白激酶受体或磷酸肌醇)检测和接受信号, 然后由第二信使扩增信号, 导致参与调控响应干旱胁迫功能基因的激活或表达调节蛋白活性的激活与抑制[23]。Molina等[24]对鹰嘴豆根部在干旱处理下的转录组测序结果发现, 蛋白激酶基因和Ca2+应答基因在感知水分胁迫信号中发挥重要作用。本研究也发现, 马铃薯在干旱胁迫下, 蛋白激酶 (PGSC0003DMG400006639)、Ca2+结合蛋白(PGSC 0003DMG400001333和PGSC0003DMG400001204)、肌醇加氧酶 (PGSC0003DMG401000287和PGSC 0003DMG400004872)相关基因响应了干旱胁迫, 同时这些基因也响应了DNA去甲基化处理, 且2种处理下的表达模式基本一致, 说明马铃薯在响应干旱胁迫时蛋白激酶、Ca2+结合蛋白、肌醇加氧酶相关基因的表达受到DNA甲基化的调控。
植物激素是一类小的内源性信号分子, 如生长素(IAA)、赤霉素(GA)、细胞分裂素(CK)、脱落酸(ABA)、乙烯(ET)、茉莉酸(JA)和水杨酸(SA)。植物激素是一种介质, 不仅作用于内源性发育过程, 而且还向多种激素反应途径提供环境刺激, 以适应不利条件[25,26]。Jain和Khurana [27]发现, 水稻生长素反应相关基因在生殖发育和非生物胁迫过程中受到不同程度的调控。本研究也得到了类似的结果, 在“植物激素信号转导”中富集到14个基因, 其中5个是生长素信号转导基因, 编码“生长素响应蛋白”和“GH3生长素响应基因”。因此, 我们推测生长素可能在马铃薯响应干旱胁迫中起重要作用。本研究还鉴定到与ABA、ET和SA调控相关的基因。共有9个差异表达基因, 分别编码ERF2转录因子(PGSC0003 DMG400026261)、PR1蛋白(PGSC0003DMG40000 5108、PGSC0003DMG400005112、PGSC0003DMG 400005116和PGSC0003DMG400005110)和热休克蛋白(PGSC0003DMG400023921); 同时PR1蛋白和热休克蛋白也被富集到植物MAPK信号转导通路。这些基因在2种处理下的表达模式基本一致, 这是因为在马铃薯响应干旱胁迫时, DNA甲基化参与了这些基因的表达调控。此外, 本研究还发现了参与植物糖酵解和糖异生途径的相关基因, 如磷酸甘油酸变位酶(PGSC0003DMG400009472)、丙酮酸脱羧酶(PGSC0003DMG400006270)、己糖激酶(PGSC0003 DMG400030624), 在马铃薯响应干旱胁迫时受DNA甲基化的调控。
植物谷胱甘肽代谢中有3种重要的酶, 分别是谷胱甘肽还原酶(GR)、谷胱甘肽过氧化物酶(GP)和谷胱甘肽S转移酶(GST)。这3种酶活性的变化与植物应对逆境胁迫密切相关[28]。其中GST是在动植物中编码的多功能蛋白的一个基因家族。同时GST也在参与干旱、盐、重金属等氧化应激的反应中发挥重要功能。过量的ROS诱导GST水平升高, 使得GST对外源物质的解毒和应激代谢中发挥关键作用[29]。Xu等[30]获得了高表达番茄谷胱甘肽S-转移酶的转基因拟南芥, 命名为LeGSTU2, 该转基因拟南芥具有较强的耐盐性和抗旱性, 同时也发现转基因植株中脯氨酸、丙二醛含量以及抗氧化酶活性的变化与植物抗逆性相关。与野生型相比, 干旱处理过的拟南芥, AtGSTF14启动子区重复序列的DNA甲基化水平显著降低, 同时AtGSTF14基因表达水平显著升高[31]。本研究也得到类似结果, 对抗旱性不同的马铃薯品种共同响应干旱和DNA去甲基化处理下的差异表达基因进行功能富集, 结果显示, 有8个DEG显著富集到植物谷胱甘肽代谢通路, 其中有6个属于谷胱甘肽S转移酶基因。同时差异基因启动子区分析表明, 在谷胱甘肽S转移酶基因(PGSC0003DMG400004977)的CpG岛区域有与植物响应干旱胁迫相关的ABRE和CAAT-box作用元件。推测马铃薯在响应干旱胁迫时, 可能通过DNA去甲基化来降低该基因启动子区的ABRE和CAAT-box作用元件的甲基化水平, 进而激活该基因的表达以应对干旱胁迫(图7)。
图7
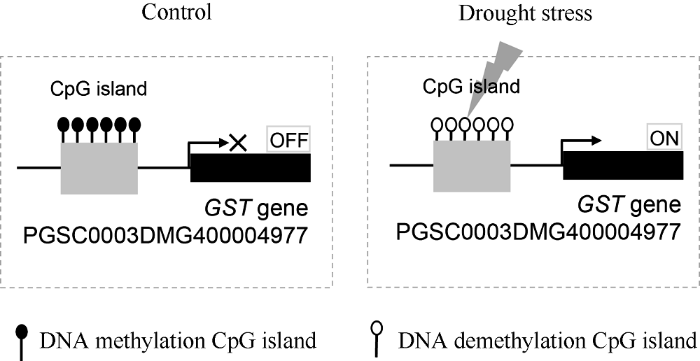 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7干旱胁迫下马铃薯GST (PGSC0003DMG400004977)基因DNA去甲基化调控模式
Fig. 7Regulatory pattern of DNA demethylation of potato GST (PGSC0003DMG400004977) gene under drought stress
以上结果表明, 利用比较转录学分析干旱和DNA去甲基化处理下差异表达基因, 可挖掘到通过DNA甲基化参与调控马铃薯响应干旱胁迫的相关基因, 为研究马铃薯干旱胁迫响应的表观遗传学机理提供新的研究思路。同时, 本研究中有关DNA甲基化具体调控马铃薯响应干旱胁迫的机制尚不清楚, 后续研究可通过检测差异表达基因启动子区DNA甲基化水平来进一步解析其蕴含的生物学意义。
4 结论
本研究对甘露醇和5-azadC处理下抗旱性不同的2个马铃薯品种进行比较转录组分析发现, 1345个DEG既响应干旱又响应DNA去甲基化。这些基因功能富集结果显示, 与植物抗旱相关的通路有植物MAPK信号通路、谷胱甘肽代谢、糖酵解/糖异生、磷酸肌醇代谢和植物激素信号转导通路。差异基因启动子分析表明, 干旱胁迫下GST基因通过DNA去甲基化来降低启动子区ABRE和CAAT-box作用元件的甲基化水平, 进而激活该基因的表达以应对干旱胁迫。本研究结果将为进一步解析马铃薯响应干旱胁迫的表观遗传学调控机理提供理论依据。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/j.bbagrm.2011.04.006URLPMID:21549230 [本文引用: 1]

A relatively high degree of nuclear DNA (nDNA) methylation is a specific feature of plant genomes. Targets for cytosine DNA methylation in plant genomes are CG, CHG and CHH (H is A, T, C) sequences. More than 30% total m(5)C in plant DNA is located in non-CG sites. DNA methylation in plants is species-, tissue-, organelle- and age-specific; it is involved in the control of all genetic functions including transcription, replication, DNA repair, gene transposition and cell differentiation. DNA methylation is engaged in gene silencing and parental imprinting, it controls expression of transgenes and foreign DNA in cell. Plants have much more complicated and sophisticated system of the multicomponent genome methylations compared to animals; DNA methylation in plant mitochondria is performed in other fashion as compared to that in nuclei. The nDNA methylation is carried out by cytosine DNA methyltransferases of, at least, three families. In contrast to animals the plants with the major maintenance methyltransferase MET1 (similar to animal Dnmt1) inactivated do survive. One and the same plant gene may be methylated at both adenine and cytosine residues; specific plant adenine DNA methyltransferase was described. Thus, two different systems of the genome modification based on methylation of cytosines and adenines seem to coexist in higher plants. This article is part of a Special Issue entitled: Epigenetic control of cellular and developmental processes in plants.
URLPMID:27379113 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
URLPMID:26759178 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1186/s12870-016-0771-yURL [本文引用: 1]
DOI:10.1111/j.1742-4658.2009.07033.xURLPMID:19490115 [本文引用: 1]

Auxin influences growth and development in plants by altering gene expression. Many auxin-responsive genes have been characterized in Arabidopsis in detail, but not in crop plants. Earlier, we reported the identification and characterization of the members of the GH3, Aux/IAA and SAUR gene families in rice. In this study, whole genome microarray analysis of auxin-responsive genes in rice was performed, with the aim of gaining some insight into the mechanism of auxin action. A comparison of expression profiles of untreated and auxin-treated rice seedlings identified 315 probe sets representing 298 (225 upregulated and 73 downregulated) unique genes as auxin-responsive. Functional categorization revealed that genes involved in various biological processes, including metabolism, transcription, signal transduction, and transport, are regulated by auxin. The expression profiles of auxin-responsive genes identified in this study and those of the members of the GH3, Aux/IAA, SAUR and ARF gene families were analyzed during various stages of vegetative and reproductive (panicle and seed) development by employing microarray analysis. Many of these genes are, indeed, expressed in a tissue-specific or developmental stage-specific manner, and the expression profiles of some of the representative genes were confirmed by real-time PCR. The differential expression of auxin-responsive genes during various stages of panicle and seed development implies their involvement in diverse developmental processes. Moreover, several auxin-responsive genes were differentially expressed under various abiotic stress conditions, indicating crosstalk between auxin and abiotic stress signaling.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1371/journal.pone.0136960URLPMID:26327625 [本文引用: 1]

Although glutathione S-transferases (GST, EC 2.5.1.18) are involved in response to abiotic stress, limited information is available regarding gene function in tomato. In this study, a GST gene from tomato, designated LeGSTU2, was cloned and functionally characterized. Expression profile analysis results showed that it was expressed in roots and flowers, and the transcription was induced by salt, osmotic, and heat stress. The gene was then introduced to Arabidopsis by Agrobacterium tumefaciens-mediated transformation. Transgenic Arabidopsis plants were normal in terms of growth and maturity compared with wild-type plants. Transgenic plants also showed an enhanced resistance to salt and osmotic stress induced by NaCl and mannitol. The increased tolerance of transgenic plants was correlated with the changes in proline, malondialdehyde and antioxidative emzymes activities. Our results indicated that the gene from tomato plays a positive role in improving tolerance to salinity and drought stresses in Arabidopsis.
[本文引用: 1]
[本文引用: 1]
