 ,1,*, 刘晓东
,1,*, 刘晓东 ,1,*
,1,*Enhancing CRISPR/Cas9 genomic editing efficiency based on optimization of sgRNA of Gossypium barbadense L.
LI Ji-Yang1,2,**, HU Yan1,**, YAO Rui2, DAI Pei-Hong ,1,*, LIU Xiao-Dong
,1,*, LIU Xiao-Dong ,1,*
,1,*通讯作者:
第一联系人:
收稿日期:2018-10-16接受日期:2019-05-12网络出版日期:2019-05-31
| 基金资助: |
Received:2018-10-16Accepted:2019-05-12Online:2019-05-31
| Fund supported: |

摘要
关键词:
Abstract
Keywords:
PDF (8059KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李继洋, 胡燕, 姚瑞, 代培红, 刘晓东. 基于优化sgRNA系统提高海岛棉CRISPR/Cas9基因组编辑功效的研究[J]. 作物学报, 2019, 45(10): 1522-1534. doi:10.3724/SP.J.1006.2019.84130
LI Ji-Yang, HU Yan, YAO Rui, DAI Pei-Hong, LIU Xiao-Dong.
目前, CRISPR/Cas9基因组编辑体系已经在多种作物中被建立[1], 然而对转基因后代靶位点突变情况检测发现, 部分物种存在低编辑效率、同义突变等现象[2], 尤其在拟南芥等植物中, 转基因后代还往往伴随高频脱靶效应[2,3]。影响CRISPR/Cas9介导的基因组编辑效率的因素主要源于组成其系统的2个重要元件(Cas9和sgRNA)。前人研究发现, 密码子不同优化程度的Cas9核酸酶对编辑效率和脱靶效应都具有一定的影响[4,5]。负责招募Cas9的sgRNA包含一段约20 nt的guide RNA, 是引导Cas9靶向切割基因组的重要组成部分, guide RNA的特异性也决定了其脱靶效率[6,7]。
脱靶效应是指由于引导核酸酶的向导DNA (gDNA)或RNA (gRNA)在对基因组靶标序列识别的过程中出现了差错, 使得核酸酶在与guide RNA高度同源位点处对基因组双链进行切割, 从而造成非目标位点突变。事实上, 这一现象并不仅存在于CRISPR/Cas9系统介导的基因组编辑, 锌指核酸酶(zinc-finger nucleases, ZFNs)和类转录激活效应物核酸酶(transcription activator-like effector nucleases, TALENs)等基因组编辑技术或NgAgo介导的基因沉默技术同样存在脱靶效应[8,9,10,11], 除此之外, 脱靶效应也影响基因组编辑效率。因此, 靶向编辑效率和脱靶效应是CRISPR/Cas9基因组编辑过程中1对相互对立的结果[12]。
CRISPR/Cas9技术已经在突变体创制过程中表现出绝对优势, 但由于广泛存在的脱靶效应, 使其在农业育种研究工作中留下潜在隐患, 对与此相关的农业分子育种工作造成负面影响。为了高效安全地使用这一技术, 就必须采取有效手段, 在保证其靶向编辑效率的同时尽可能降低脱靶效率, 为选育用于种质创新的低脱靶效应的靶向突变体提供技术保障[13,14]。到目前为止已有多个实验室研究出提高基因组编辑效率和增加基因靶向特异性的方法。Cong等[15]构建了Cas9核酸酶的一种变体Cas9 (D10A), 该变体Cas9只能切割单链DNA, 如果要产生双链断裂, 引发基因突变, 就必须在基因靶位点区结合2条sgRNA。实验证明, 上述方法能有效降低转基因T1代的脱靶效应, 或采用基因组双向编辑技术在目的基因靶位点被编辑的同时使Cas9也被编辑[16,17], 通过减少后代Cas9表达累积量降低脱靶效应。
通过对驱动Cas9蛋白表达的启动子进行改造及优化, 能显著提高编辑效率, 降低脱靶效应。例如, 选择卵细胞特异性表达的启动子EC1.2p或EC1.1p替代常规的CAMV35S或UBQ类启动子, 可以在T1代就能获得3个基因同时突变的纯合体植株, 突变率高达8.3%, 进一步用融合了这2个启动子关键元件的新启动子去驱动Cas9蛋白的表达, 可以获得比单个启动子更高的突变效率, 并且这些突变体中均未检测到脱靶效应。DMC1在玉米愈伤组织中高表达, 应用DMC1启动子驱动Cas9, 也能显著提高基因组编辑的效率, 并且获得的突变体中也均未检测到脱靶效应[18]。除此之外, 增加PAM位点的长度也是一种减少脱靶效应的策略。目前已经从不同的细菌物种中分离得到不同的Cas9蛋白, 除SpCas9[19]识别NGG类的PAM位点外, 还有NmeCas9识别NNNNGATT[20]; SaCas9识别NNGRRT[21]; St1Cas9和St2Cas9分别识别NNAGAAW和NGGNG[22]。与SpCas9蛋白相比, 这些不同来源的Cas9均可以增加基因靶向的特异性, 减少脱靶效应。CRISPR-Cpf1是一种V型核酸内切酶, 与Cas9蛋白相比, CRISPR-Cpf1系统在动植物应用中表现出更低的或未发现脱靶效应[23]。除了上述不同来源的Cas9可以增加基因靶向的特异性, 减少脱靶效应外, 在野生型SpCas9蛋白的基础上, 筛选高特异性Cas9蛋白变异体的研究也取得了良好的结果, 如eSpCas9[5]、Cas9- HF[3]、evoCas9[24]、HypaCas9[25]和Sniper-Cas9[26]。其中evoCas9、HypaCas9和Sniper-Cas9不仅显著增加了基因靶向的特异性, 同时也提高了基因的编辑效率。上述提高基因组编辑效率和增加基因靶向特异性的方法, 多集中在Cas9蛋白方面, 而从sgRNA入手的研究还鲜见报道。2015年在线虫中研究发现与单个PAM位点相比, 连续2个PAM位点可以显著提高基因的编辑效率, 但脱靶率是否也同步提高并不清楚[27]。
在植物中, CRISPR/Cas9系统在拟南芥、水稻应用较为广泛, 但在棉花[28,29]中CRISPR/Cas9系统的报道还较少见, 其中在海岛棉中还未见有报道能高效表达的CRISPR/Cas9系统。海岛棉的组织再生存在体细胞胚胎发育困难、转化期长、效率低等一些问题, 并且有研究发现陆地棉经Cas9编辑后, 组织再生的过程中有大量的体细胞变异会影响编辑效率[28], 这对初步构建海岛棉CRISPR/Cas9基因组编辑增进了困难, 因此, 使用瞬时转化方法快速验证构建的CRISPR/Cas9基因组编辑载体的有效性是最佳的方法。本研究在基于前期海岛棉原生质体中建立的CRISPR/Cas9基因组编辑体系[30], 针对Cas9表达量和sgRNA的优化设计, 对比转化不同sgRNA个数和不同类型sgRNA对基因组编辑效率和脱靶效应的影响进行了初步分析和试验来验证上述假设。以期为今后利用CRISPR/Cas9基因组编辑技术进行精确的分子设计育种奠定一定的理论依据。
1 材料与方法
1.1 供试材料
新海16胚性愈伤组织由新疆农业大学农业生物技术重点实验室提供, 用于制备海岛棉原生质体, 载体GbU6-4P::GUS-sgRNA、GbU6-5P::GUS-sgRNA、pBlueScript SK(+)(简称SK载体)、pUC19均由本实验室保存, 2种不同密码子优化的Cas9基因(Cas9-I和Cas9-II)分别由中国科学院上海生命科学研究院和中国农业科学院生物技术研究所惠赠, 限制性内切酶和DNA聚合酶均为NEB和Thermo公司产品, 纤维素酶和离析酶由IUZMI株式会社提供, 引物合成及测序由武汉昆泰瑞有限公司(新疆分公司)完成。1.2 海岛棉编辑载体构建
1.2.1 基础载体构建 以新海16基因组DNA为模板克隆获得GbGGB和GbERA1基因部分序列用于靶序列设计, 靶序列的设计除满足其基本要求外[29], 选择在GbGGB和GbERA1基因组CDS区域不超过20 bp长度任意片段的3°末端含有连续2个5°NGG3° (N代表任意碱基)位点的区域, 分别设计包含2个NGG位点(No shift sgRNA)和包含1个NGG (Shift sgRNA)位点的sgRNA序列, 引物序列如表1, 靶序列设计模式如图1。Table 1
表1
表1本研究使用的引物信息
Table 1
| 序列名称 Sequence name | 序列 Sequence (5'-3') | PAM位点序列 PAM site sequence |
|---|---|---|
| Shift GbU6-4PERA1-sg2F | GATTGTCTTTCGCAGAATGCATGA | CGG |
| Shift GbU6-4PERA1-sg2R | AAACTCATGCATTCTGCGAAAGAC | |
| GbU6-4PERA1-sg2F | GATTGTTCGCAGAATGCATGACGG | TGG |
| GbU6-4PERA1-sg2R | AAACCCGTCATGCATTCTGCGAAC | |
| GbU6-5PGGB-sg1F | AAGTGTGTCGAAGTACTGAAGCGG | CGG |
| GbU6-5PGGB-sg1R | AAACCCGCTTCAGTACTTCGACAC | |
| GbU6-4PGGB-sg2F | GATTGTCTTTCAGTCTTATGATGG | TGG |
| GbU6-4PGGB-sg2R | AAACCCATCATAAGACTGAAAGAC | |
| Shift GbU6-5PGGB-sg1F | AAGTGCTCTGTCGAAGTACTGAAG | CGG |
| Shift GbU6-5PGGB-sg1R | AAACCTTCAGTACTTCGACAGAGC | |
| Shift GbU6-4PGGB-sg2F | GATTGTGTTCTTTCAGTCTTATGA | TGG |
| Shift GbU6-4PGGB-sg2R | AAACTCATAAGACTGAAAGAACAC | |
| Test GGB-sg1F | AAGTGGAAAGAGAATGGCGAC | |
| Test GGB-sg1R | AGCTAATTCGCTATTGCAATCAATC | |
| Test GGB-sgR | ACCATGTGATTCTAAACCAGG | |
| Test ERA1-sg1F | TAATAGGTGGTCAGGTTATGC | |
| Test ERA1-sg1R | AGGAGACTTGCAACCTGATC | |
| FJ-1F | GTAAAACGACGGCCAG | |
| FJ-1R | CAGGAAACAGCTATGAC | |
| FJ-2F | TAAACTGAAGGCGGGAAACG | |
| FJ-2R | CGGTTCTGTCAGTTCCAAACG |
新窗口打开|下载CSV
图1
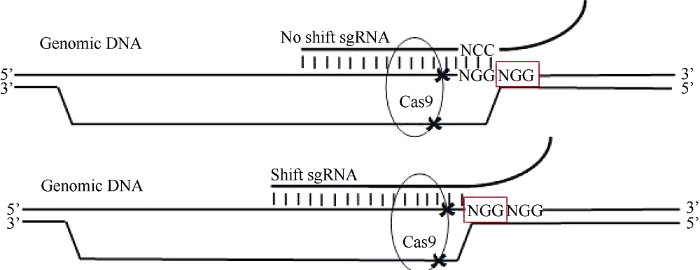 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1No shift sgRNA和Shift sgRNA靶序列特点及其作用模式
红框标识为CRISPR/Cas9作用的PAM位点。
Fig. 1No shift sgRNA and Shift sgRNA target sequence characteristics and mode of action
The red box identifies the PAM site for the action of CRISPR/Cas9.
将经过DNA复性反应后的靶序列分别连接于以Bbs I酶切获得的GbU6-4P::GUS-sgRNA和GbU6-5P::GUS-sgRNA线性化载体。退火反应体系为20 μL, 包括10 μmol L-1的上下游靶序列各10 μL。复性程序为95℃至20℃, 每5 s降低0.5℃。经测序验证上述(shift)GbU6-4P(5P)::GGB-sgRNA和(shift)GbU6-4P::ERA1-sgRNA2质粒构建正确后备用。1.2.2 编辑载体组装 以Kpn I、Xba I酶切GbU6-5P::GGB-sgRNA1载体, 同时以Xba I、Hind III 酶切GbU6-4P::GGB-sgRNA2和GbU6-4P::ERA1- sgRNA2载体, 以Kpn I、Hind III酶切pUC19载体, 三片段连接构建GbU6-5P::GGB-sgRNA1-GbU6- 4P::GGB-sgRNA2-pUC19-GbU6-5P::GGB-sgRNA1-GbU6-4P:ERA1sgRNA2-pUC19过渡载体, 以Kpn I、Hind III酶切上述过渡载体和Cas9-I编辑载体, 构建GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2- Cas9-I和GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1- sgRNA2-Cas9-I编辑载体(图2-A)。以Kpn I、Hind III酶切GbU6-5P::GGB-sgRNA1载体, 同时以Xba I、Hind III酶切GbU6-4P::GGB-sgRNA2和GbU6-4P::ERA1-sgRNA2载体, 以Kpn I、Xba I酶切SK载体, 3个片段连接构建GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2- SK和GbU6-5P::GGB-SgRNA1-GbU6-4P::ERA1-sgRNA2- SK过渡载体, 以Kpn I、Xba I酶切上述过渡载体和Cas9II编辑载体, 构建GbU6-5P::GGB-sgRNA1-GbU6-4P:: GGB-sgRNA2-Cas9II和GbU6-5P::GGB-sgRNA1-GbU6- 4P::ERA1-sgRNA2-Cas9II编辑载体(图2-B), 以GbU6- 5P::GGB-sgRNA1-Cas9-I和GbU6-5P::GGB-sgRNA1- Cas9II及其连接2种Cas9基因不含靶序列GbU6- 5P(4P):: sgRNA为对照。构建Shift sgRNA类型编辑载体方法同上。
图2
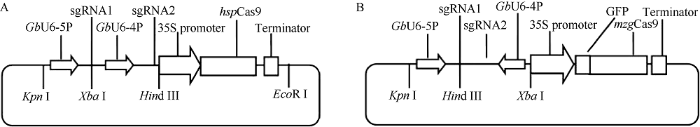 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2海岛棉基因组编辑载体结构示意图
A: 含Cas9I编辑载体; B: 含Cas9II编辑载体。
Fig. 2Sketch map of genome editing structure of cotton (Gossypium barbadense L.)
A: editing vector containing Cas9I; B: editing vector containing Cas9II.
1.3 海岛棉原生质体制备及转化
1.3.1 富集编辑载体核心片段 经酶切检测正确的编辑载体利用富集引物(Cas9I编辑载体采用FJ-1引物, Cas9II编辑载体采用FJ-2引物), 采用fast pfu fly超保真DNA聚合酶(北京全式金)扩增编辑载体的核心区域, 扩增体系参照说明书推荐反应体系, PCR产物纯化参照本实验室已有成熟体系[29], 纯化后的PCR产物用双蒸水溶解并调整浓度至0.5~1.0 μg μL-1, -20℃保存备用。1.3.2 原生质体的制备与转化 取新海16扩繁培养约15 d的胚性愈伤组织, 利用本实验室建立的改良CPW双酶法制备原生质体[31]。取10 μg富集的PCR产物直接转化原生质体, 方法参照拟南芥原生质体转化方法[32], 过程略有改进(悬浮原生质体不使用MMG溶液), 为获得较高转化率, 本研究在原有成熟体系基础上优化了海岛棉胚性愈伤组织与酶解液最适反应比例(每毫升酶解液加入约0.45 g海岛棉胚性愈伤组织), 转化后的原生质体于28℃, 50 r min-1避光孵育48 h。1.4 内源靶基因编辑效应检测
将孵育48 h的原生质体悬浮液以1000 × g离心1 min, 去尽上清, 用于提取原生质体沉淀的基因组DNA, 方法参照Trans Easypure plant Genomic DNA Kit (北京全式金)说明书要求。除转化GbU6- 5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I和GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2- Cas9II载体提取的基因组DNA直接用相应的Test引物(Test GGB-sg1F和Test GGB-sgR)进行PCR扩增外, 转化其余编辑载体提取的基因组DNA采用先酶切后PCR方式检测, PCR体系共20 μL, 包含ddH2O 11.2 μL、5×Phusion buffer 4 μL、2.5 mmol L-1 dNTP 1.6 μL、上下游引物各1 μL、DNA模板1 μL、Phusion超保真DNA 聚合酶0.2 μL。扩增程序为94℃ 2 min; 98℃ 30 s, 60℃ 30 s, 72℃ 4 min 30 s, 35个循环; 72℃ 7 min。扩增得到的PCR产物经EasyPure Quick Gel Extraction Kit (北京全式金)回收后, 连接pEASY-Blunt Zero Cloning (北京全式金)载体, 转化大肠杆菌Trans T1, 经菌落PCR和酶切鉴定正确后, 将鉴定正确的单克隆菌液进行序列测定, 测序序列采用DNAstar和DNAMAN软件进行比对分析, 统计基因组靶序列20 bp区域碱基突变个数并计算相应区域碱基突变频率, 通过计算得到编辑效率, 利用SPSS 19.0统计软件进行差异显著性分析, 对比每种Cas9类型编辑载体转化Shift sgRNA和NO shift sgRNA靶序列类型对应的单靶序(GbU6-5P::GGB- sgRNA1)以及双靶序(GbU6-5P::GGB-sgRNA1- GbU6-4P::GGB-sgRNA2-GbU6-5P::GGB-sgRNA1- GbU6-4P::ERA1-sgRNA2)编辑效率差异的显著性。1.5 脱靶效率分析
根据设计的Guide RNA序列, 浏览华中农业大学CRISPR sgRNA设计网站(http://crispr.hzau.edu. cn/cgi-bin/CRISPR2/CRISPR)[6], 寻找本研究设计使用的GGB-sgRNA1和Shift GGB-sgRNA1靶序列, 并分别预测得到脱靶率较高的2种靶序列(表2), 利用该序列, 登录中国农业科学院棉花基因组数据库(https:// cgp.genomics.org.cn/), 通过BLAST获得每条预测的脱靶序列对应的同源序列, 再通过Mapping View将上述核苷酸序列定位到具体的染色体进而获得涵盖该脱靶序列的基因组序列, 设计用于检测脱靶率的引物(表3)。Table 2
表2
表2脱靶序列预测信息
Table 2
| 脱靶序列名称 Name for sequence of off target | 序列 Sequence (5°-3°) | 预测脱靶率 Predict the off target rate |
|---|---|---|
| OT-GGB-sgRNA1-1 | GGTCCAAGGACTGAAGCTGTGG | 0.311 |
| OT-GGB-sgRNA1-2 | GGTTGAAGTAGTAAAGCGGAGG | 0.177 |
| OT-Shift GGB-sgRNA1-1 | TCCAGTCGAAATACTGAAGAGG | 0.408 |
| OT-Shift GGB-sgRNA1-2 | CTCTGTTGATGAACTGATGGGG | 0.287 |
新窗口打开|下载CSV
Table 3
表3
表3脱靶序列检测引物信息
Table 3
| 序列名称 Sequence name | 序列 Sequence (5°-3°) |
|---|---|
| OTtest-G1-1F | CCTTGAAAGTTGCTTCCGAC |
| OTtest-G1-1R | GAAACTGCCTTTTTTGACACC |
| OTtest-G1-2F | CGAAAAATGGTTAGTGGTGAC |
| OTtest-G1-2R | GTGGATGTTACTGTAGCAATG |
| OTtest-SG1-1F | AGAGGACCAAACACATTAACC |
| OTtest-SG1-1R | TTTGGGGGAGAGCACTTTTG |
| OTtest-SG1-2F | ATCACCTTGCTACTCTTTCTG |
| OTtest-SG1-2R | GTTTAGTTGACCAAGCATCTC |
新窗口打开|下载CSV
以上述转化不同编辑载体核心片段原生质体提取的基因组DNA为模板, 扩增获得涵盖脱靶序列的片段, 克隆该序列, 方法参照编辑效应分析方法, 将菌落PCR鉴定正确的单克隆菌液进行序列测定, 测序序列采用DNAstar软件进行比对分析, 统计基因组涵盖脱靶序列20 bp区域中碱基突变个数并计算相应区域碱基突变频率, 利用SPSS 19.0统计软件进行单因素方差分析, 对比分别转化Shift sgRNA和No shift sgRNA靶序列类型对应的单靶序列和双靶序列目标片段脱靶效率的差异是否具有显著性。
1.6 sgRNA及其Cas9表达分析
收集转化后孵育48 h的原生质体, 参照Biospin Plant Total RNA Extration Kit (DNA-free)试剂盒(Bioer Technology Co, Ltd.)说明书立即提取总RNA, 将质检合格用于合成cDNA第1条链的总RNA质量调整至1 μg逆转录为cDNA, 方法参照TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix (北京全式金)试剂盒说明书, 将合成的cDNA用于Cas9、GGB-sgRNA1、GGB-sgRNA2、ERA1-sgRNA2半定量RT-PCR检测分析。2 结果与分析
2.1 编辑载体的构建
采用双酶切方式(Kpn I、Hind III)检测上述构建的编辑载体, 酶切结果符合预期设计, 证明分别连接不同Cas9的GbU6-5P-GGB-sgRNA1、GbU6- 5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2和GbU6- 5P::GGB-sgRNA1-GbU6-4P::ERA1-sgRNA2的编辑载体均构建成功(图3)。连接Shift相关靶序列的上述编辑载体鉴定结果与预期相符。图3
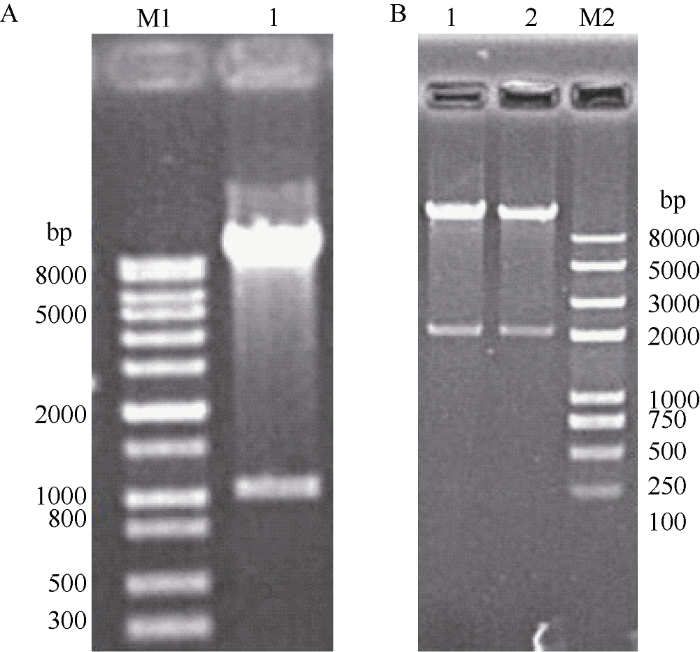 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3海岛棉编辑载体酶切检测结果
A: GbU6-5P-GGB-sgRNA1编辑载体酶切检测结果; M1为1 kb plus marker。B: 1和2分别为GbU6-5P::GGB-sgRNA1-GbU6- 4P::GGB-sgRNA2和GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1- sgRNA2编辑载体酶切检测结果; M2为2 kb plus II marker。
Fig. 3Result of gene editing vector restriction enzyme digestion of Gossypium barbadense L.
A: GbU6-5P-GGB-sgRNA 1 editing vector digestion test results; M1 is 1 kb plus marker. B: GbU6-5P::GGB-sgRNA1-GbU6- 4P::GGB-sgRNA2 and GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1- sgRNA2 edit vector restriction test results; M2 is 2 kb plus II marker.
2.2 原生质体制备
基于前期本实验室建立的较成熟的原生质体制备体系, 转化前后原生质体形态如图4, 经计算每毫升转化前原生质体数量达到约4×106个左右, 镜检视野内, 原生质体未出现明显畸形或破壁现象, 表明制备的原生质体能够用于编辑效率、脱靶效率以及半达量RT-PCR的检测分析。图4
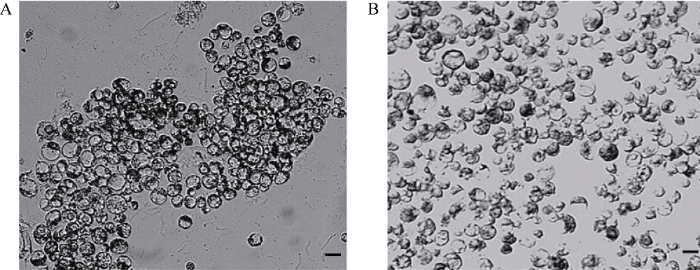 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4海岛棉原生质体制备及其转化结果
A: 海岛棉转化前原生质体镜检结果; B: 海岛棉转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I载体后原生质体镜检结果。标尺为40 μm。
Fig. 4Protoplast preparation and transformation results of Gossypium barbadense L.
A: microscopic examination of Gossypium barbadense L. protoplast before transformation; B: after protoplast microscopy results transformation of G. barbadense GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I vector. Bar = 40 μm.
2.3 编辑效率结果分析
2.3.1 海岛棉基因组编辑检测分析 将转化GbU6-5P-GGB-sgRNA1和转化GbU6-5P::GGB- sgRNA1-GbU6-4P::ERA1-sgRNA2提取的基因组DNA采用酶切(Kpn I、Sac I)后PCR方式检测, 转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2采用直接PCR方式检测, 结果如图5, 转化Cas9 I型靶序列编辑载体的原生质体DNA酶切后PCR检测存在扩增条带, 对照则未出现明显条带, 初步证明, 转化GbU6-5P-GGB-sgRNA1和转化GbU6-5P::GGB- sgRNA1-GbU6-4P::ERA1-sgRNA2编辑载体造成海岛棉基因组相应的靶位点突变, 将上述PCR产物克隆后测序结果(图6和图7)分析发现, 所有转化靶序列的原生质体均检测到碱基突变现象, 转化与之对应空载体的原生质体未检测到碱基突变现象, 对比转化不同载体的原生质体, 虽然靶序列作用位点相同, 但引发碱基突变的个数和位置均不同。分析后认为, 可能是转化不同编辑载体引起不同程度编辑效应造成的结果。针对转化Shift相关靶序列的基因组编辑载体, 也获得与上述相同的实验结果, 相比同一位点2种(No shift sgRNA和shift sgRNA)不同靶序列类型, 转化No Shift靶序列的原生质体其编辑效率高于转化Shift靶序列, 增幅为20.0%~49.6% (表4)。转化Cas9II型编辑载体结果与上述类似。图5
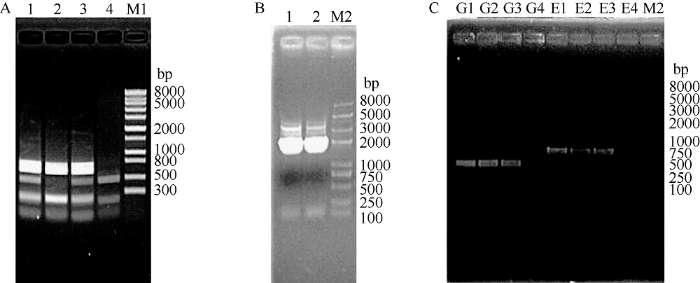 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5海岛棉转化不同靶序列编辑效应检测结果
A: 1为转化GbU6-5P-GGB-sgRNA1-Cas9I载体酶切前直接PCR扩增结果, 2为转化GbU6-5P-GGB-sgRNA1-Cas9I载体酶切后PCR扩增结果, 3为转化GbU6-5P-sgRNA1-Cas9I载体酶切前直接PCR扩增结果, 4为转化GbU6-5P-sgRNA1-Cas9I载体酶切后PCR扩增结果。B: 1为转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I直接PCR扩增结果, 2为转化GbU6-5P-sgRNA1-GbU6-4P-sgRNA2- Cas9I直接PCR扩增结果。C: G1、E1为转化GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1-sgRNA2-Cas9I酶切前直接PCR扩增结果, G2、E2为转化GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1-sgRNA2-Cas9I酶切后PCR扩增结果, G3、E3为转化GbU6-5P-sgRNA1-GbU6-4P- sgRNA2-Cas9I酶切前直接PCR扩增结果, G4、E4为转化GbU6-5P-sgRNA1-GbU6-4P-sgRNA2-Cas9I酶切后PCR扩增结果。M1为1 kb plus marker, M2为2 kb plus II marker。
Fig. 5Transformation of different target sequence editing effect test results of Gossypium barbadense L.
A: 1 is the result of direct PCR before transformation of GbU6-5P-GGB-sgRNA1-Cas9I vector, 2 is PCR amplification after transformation of GbU6-5P-GGB-sgRNA1-Cas9I vector As a result, 3 was obtained by direct PCR amplification before transformation of GbU6-5P-sgRNA1- Cas9I vector and 4 was PCR amplification after transformation with GbU6-5P-sgRNA1-Cas9I vector. B: 1 was transformed with GbU6- 5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I direct PCR amplification results, 2 for the transformation of GbU6-5P-sgRNA1-GbU6- 4P-sgRNA2-Cas9I direct PCR amplification results. C: G1 and E1 are the results of direct PCR before transformation of GbU6-5P:: GGB-sgRNA1-GbU6-4P::ERA1-sgRNA2-Cas9I, G2 and E2 are the results of transformation of GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1- sgRNA2-Cas9 digestion after PCR amplification results, G3 and E3 to transform GbU6-5P-sgRNA1-GbU6-4P-sgRNA2-Cas9I digestion before direct PCR amplification results, G4 and E4 to transform GbU6-5P-sgRNA1-PCR amplification results after GbU6-4P-sgRNA2-Cas9 digestion. M1 is 1 kb plus marker, M2 is 2 kb plus II marker.
图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6海岛棉转化不同类型靶序列碱基突变测序结果
A: 转化GbU6-5P-GGB-sgRNA1-Cas9I载体靶位点编辑效应测序检测结果; B: 转化GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1- sgRNA2-Cas9I载体GGB靶位点编辑效应测序检测结果; C: 转化GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1-sgRNA2-Cas9I载体ERA1靶位点编辑效应测序检测结果; D: 转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I载体GGB-sgRNA2靶位点编辑效应测序检测结果; E: 转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I载体GGB-sgRNA1靶位点编辑效应测序检测结果。红色框为PAM位点的位置。
Fig. 6Sequencing results of base mutation of different types of target sequences of Gossypium barbadense L.
A: Transformation of GbU6-5P-GGB-sgRNA 1-Cas9I vector target site editing effect sequencing detection results; B: Transformation GbU6- 5P::GGB-sgRNA1-GbU6-4P::ERA1-sgRNA2-Cas9I vector GGB target site editing effect sequencing detection results; C: GbU6-5P::GGB- sgRNA1-GbU6-4P::ERA1-sgRNA2-Cas9I vector ERA1 target site editing sequencing results; D: transformation GbU6-5P::GGB-sgRNA1-GbU6- 4P::GGB-sgRNA2-Cas9I vector GGB-sgRNA2 target site editing effect sequencing test results; E: transformation GbU6-5P::GGB-sgRNA1-GbU6- 4P::GGB-sgRNA2-Cas9I vector GGB-sgRNA1 target site editing effect sequencing test results. The red box is the location of the PAM site.
图7
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7海岛棉转化不同类型靶序列碱基突变部分测序峰图
A: 转化GbU6-5P-GGB-sgRNA1-Cas9I靶序列基因组碱基突变测序峰图; B: 转化GbU6-5P::GGB-sgRNA1-GbU6-4P::ERA1- sgRNA2-Cas9I靶序列基因组碱基突变测序峰图; C: 转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I靶序列基因组碱基突变测序峰图。
Fig. 7Sequencing peak map of base mutation of different types of target sequences of Gossypium barbadense L.
A: baseline mutation map of genomes transformed with GbU6-5P-GGB-sgRNA1-Cas9I target sequence; B: transformation of GbU6- 5P::GGB-sgRNA1-GbU6-4P::ERA1-sgRNA2-Cas9I target sequence; C: baseline mutation sequencing of the genome of transformed GbU6- 5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I target sequence.
Table 4
表4
表4转化不同靶序列编辑效率统计结果
Table 4
| 转化载体名称 Conversion carrier name | 一个位点碱基突变个数 A base muta- tion number | 2个位点同时突变个数 Number of mutations for two sites at the same time | 突变率 Mutation rate | ||
|---|---|---|---|---|---|
| sgRNA1位点 sgRNA1 site | sgRNA2位点 sgRNA2 site | sgRNA1位点sgRNA1 site | sgRNA2位点sgRNA2 site | ||
| GGB-sgRNA1 | 12/95 | — | — | 0.126 | — |
| Shift GGB-sgRNA1 | 10/95 | — | — | 0.105 | — |
| GGB-sgRNA1-ERA1-sgRNA2 | — | 15/70 | 15/70 | 0.214 | 0.214 |
| Shift GGB-sgRNA1-ERA1-sgRNA2 | — | 11/70 | 10/70 | 0.157 | 0.143 |
| GGB-sgRNA1-GGB-sgRNA2 | — | 9/57 | 7/57 | 0.157 | 0.123 |
| Shift GGB-sgRNA1-GGB-sgRNA2 | — | 7/60 | 5/60 | 0.117 | 0.083 |
新窗口打开|下载CSV
2.3.2 Cas9及其相关sgRNA表达量检测 半定量RT-PCR检测转化靶序列和对照编辑载体原生质体中Cas9及其对应sgRNA表达量, 结果如图8, 在转化包括对照编辑载体的原生质体中, Cas9和相应sgRNA表达量均无明显差异, 表明不同载体转化的原生质体中Cas9和相应sgRNA表达量均无明显差异。
图8
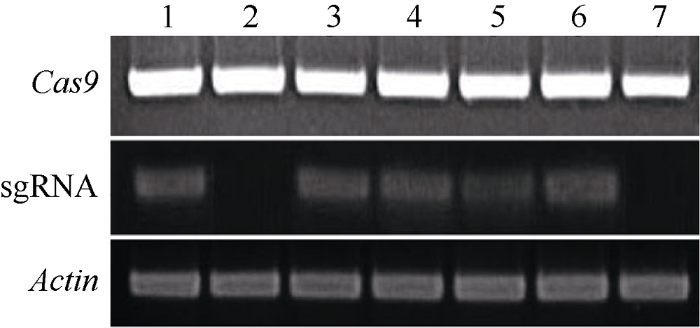 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8转化海岛棉不同类型靶序列的原生质体Cas9及对应sgRNA半定量检测结果
1、2分别为转化GbU6-5P-GGB-sgRNA1-Cas9I和对照编辑载体检测GGB-sgRNA1, 3、4为GbU6-5P-GGB-sgRNA1-GbU6-4P- ERA1-sgRNA2-Cas9I编辑载体分别检测GGB-sgRNA1、ERA1- sgRNA2, 5、6为转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB- sgRNA2-Cas9I分别检测GGB-sgRNA1、GGB-sgRNA2, 7为转化与GbU6-5P-sgRNA1-4P-sgRNA1Cas9I对照编辑载体检测GGB- sgRNA1。
Fig. 8Semi-quantitative detection of protoplast Cas9 and corresponding sgRNA in different types of target sequences transformed in Gossypium barbadense L.
1 and 2 is transformed GbU6-5P-GGB-sgRNA1-Cas9I edit vector and Control edit vector were detected GGB-sgRNA1, 3 and 4 is transformed GbU6-5P-GGB-sgRNA1-GbU6-4P::ERA1-sgRNA1- Cas9I edit vector were detected GGB-sgRNA1, ERA1-sgRNA2, 5 and 6 is transformed GbU6-5P-GGB-sgRNA1-GbU6-4P::GGB- sgRNA2-Cas9I edit vector were detected GGB-sgRNA1, GGB- sgRNA2, 7 is transformed GbU6-5P-sgRNA1-4P-sgRNA1-Cas9I control edit vector were detected GGB-sgRNA1.
2.4 脱靶效应检测结果
对转化不同类型靶序列编辑载体的海岛棉基因组进行脱靶率鉴定后发现, 在海岛棉原生质体瞬时表达的基因组编辑过程中也存在一定概率的脱靶现象(图9、图10和图11), 转化不同类型靶序列的编辑载体, 脱靶效应存在差异, 这说明不同类型靶序列对海岛棉基因组编辑效应影响不同, 对比编辑效率较高的靶序列类型发现其脱靶效率并未相应升高, 同时对比转化单靶序列和双靶序列类型的编辑载体, 其脱靶效率同样存在差异且转化单靶序列脱靶效率高于双靶序列(表5), 同时, 转化No shift sgRNA靶序列脱靶率显著低于Shift sgRNA (表5)。图9
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9海岛棉脱靶目标序列扩增检测
转化No shift sgRNA类型靶序列: 1、2分别为转化GbU6-5P-GGB-sgRNA1-Cas9I和对照编辑载体, 3、4为GbU6-5P- GGB-GbU6-4P-sgRNA1- ERA1-sgRNA2-Cas9I和对照编辑载体, 5、6为转化GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2 -Cas9I和对照编辑载体。转化Shift sgRNA类型靶序列: 1、2分别为转化Shift GbU6-5P-GGB-sgRNA1-Cas9I和对照编辑载体, 3、4为Shift GbU6-5P-GGB-GbU6-4P-sgRNA1-ERA1-sgRNA2- Cas9I和对照编辑载体, 5、6为转化Shift GbU6-5P::GGB- sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I和对照编辑载体。
Fig. 9Off target sequence amplification test of Gossypium barbadense L.
Transform No shift sgRNA type target sequence: 1 and 2 is transformed GbU6-5P-GGB-sgRNA1-Cas9I edit vector and Control edit vector, 3 and 4 is transformed GbU6-5P-GGB-sgRNA1-GbU6- 4P::ERA1-sgRNA1-Cas9I edit vector and Control edit vector, 5 and 6 is transformed GbU6-5P-GGB-sgRNA1-GbU6-4P::GGB-sgRNA2- Cas9I edit vector and Control edit vector. Transform Shift sgRNA type target sequence: 1 and 2 is transformed Shift GbU6-5P- GGB-sgRNA1-Cas9I edit vector and Control edit vector, 3 and 4 is transformed Shift GbU6-5P-GGB-sgRNA1-GbU6-4P::ERA1-sgRNA1- Cas9I edit vector and Control edit vector, 5 and 6 is transformed Shift GbU6-5P-GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I edit vector and Control edit vector.
图10
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图10海岛棉脱靶目标序列突变检测
A: OTtest-SG1-2引物检测; 1为转化Shift GbU6-5P-GGB- sgRNA1-Cas9I编辑载体, 2为Shift GbU6-5P-GGB-GbU6-4P- sgRNA1-ERA1-sgRNA2-Cas9I编辑载体, 3为转化Shift GbU6- 5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I编辑载体。 B: OTtest-G1-2引物检测; 1为转化GbU6-5P-GGB-sgRNA1-Cas9I编辑载体, 2为GbU6-5P-GGB-GbU6-4P-sgRNA1-ERA1-sgRNA2- Cas9I编辑载体, 3为转化 GbU6-5P::GGB-sgRNA1-GbU6-4P:: GGB-sgRNA2-Cas9I编辑载体。C: OTtest-SG1-1引物检测; 1为转化Shift GbU6-5P-GGB-sgRNA1-Cas9I编辑载体, 2为Shift GbU6-5P-GGB-GbU6-4P-sgRNA1-ERA1-sgRNA2-Cas9I编辑载体, 3为转化Shift GbU6-5P::GGB-sgRNA1-GbU6-4P::GGB-sgRNA2- Cas9I编辑载体。
Fig. 10Off target sequence mutation detection of Gossypium barbadense L.
A: OTtest-SG1-2 primer detection; 1 is transformed Shift GbU6- 5P-GGB-sgRNA1-Cas9I edit vector, 2 is transformed Shift GbU6- 5P-GGB-sgRNA1-GbU6-4P::ERA1-sgRNA1-Cas9I edit vector, 3 is transformed Shift GbU6-5P-GGB-sgRNA1-GbU6-4P::GGB-sgRNA2- Cas9I edit vector. B:OTtest-G1-2 primer detection; 1 is transformed GbU6-5P-GGB-sgRNA1-Cas9I edit vector, 2 is transformed GbU6- 5P-GGB-sgRNA1-GbU6-4P::ERA1-sgRNA1-Cas9I edit vector, 3 is transformed GbU6-5P-GGB-sgRNA1-GbU6-4P::GGB-sgRNA2-Cas9I edit vector. C: OTtest-SG1-2 primer detection; 1 is transformed Shift GbU6-5P-GGB-sgRNA1-Cas9I edit vector, 2 is transformed Shift GbU6-5P-GGB-sgRNA1-GbU6-4P::ERA1-sgRNA1-Cas9I edit vector, 3 is transformed Shift GbU6-5P-GGB-sgRNA1-GbU6-4P::GGB-sgRNA2- Cas9I ditvector.
图11
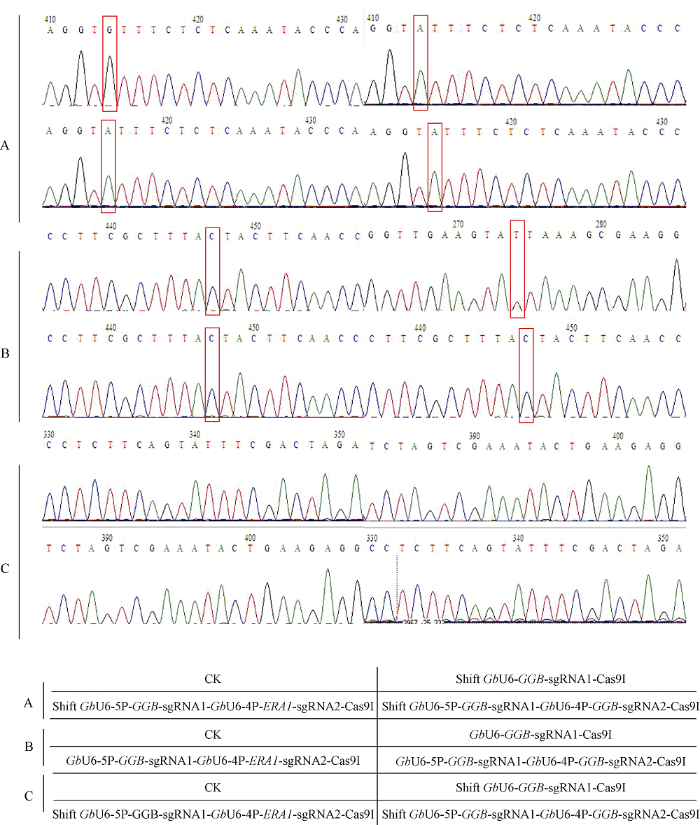 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图11海岛棉脱靶目标序列测序峰图
A、B、C分别为OTtest-SG1-2、OTtest-G1-2、OTtest-SG1-1引物扩增转化下图对应载体得到的目标片段测序结果。红框标识为碱基突变位点。
Fig. 11Off target sequence sequencing peak map of Gossypium barbadense L.
A, B, and C are OTtest-SG1-2, OTtest-G1-2, and OTtest-SG1-1 primers to transform the sequencing result of the target fragment amplified by the corresponding vector in the figure below, respectively. The red box identifies the base mutation site.
Table 5
表5
表5转化不同靶序列脱靶效率统计结果
Table 5
| 转化载体名称 Conversion carrier name | 脱靶序列检测Detection of off-target sequences | ||
|---|---|---|---|
| OTtest-G1-1 | OTtest-SG1-1 | OTtest-SG1-2 | |
| GGB-sgRNA1 | 6/50 | — | — |
| Shift GGB-sgRNA1 | — | 0 | 18/50 |
| GGB-sgRNA1-ERA1-sgRNA2 | 0 | — | — |
| Shift GGB-sgRNA1-ERA1-sgRNA2 | — | 0 | 6/50 |
| GGB-sgRNA1-GGB-sgRNA2 | 0 | — | — |
| Shift GGB-sgRNA1-GGB-sgRNA2 | — | 0 | 6/50 |
新窗口打开|下载CSV
3 讨论
3.1 靶序列类型对编辑效率影响
CRISPR/Cas9基因组编辑技术已在各种动物、植物、微生物和人中得到了广泛的应用。但基因编辑的脱靶效应和编辑效率低, 甚至常常不编辑的现象也引起了科学家们的关注。一系列提高基因编辑效率和增加基因靶向特异性的方法陆续研发出来, 这些方法多集中在Cas9蛋白方面[19,20,21,22,23,24,25,26], 而从sgRNA入手的研究还鲜见报道。在线虫中研究发现, 与单个NGG的PAM位点(Shift)相比, 连续2个NGG位点(No shift)可以显著提高基因的编辑效率。那么在植物体内No shift是否也能显著提高基因的编辑效率?为此本文开展了相关的研究工作。根据在3个基因靶位点、5组独立的转化编辑实验数据结果显示, 在海岛棉原生质体内优化后, No shift编辑效率较对应的Shift的编辑效率有所提高, 提高幅度为20.0%~49.6%。这与在秀丽隐杆线虫中报道的结果相似[33], 只是提高的幅度低于线虫。连续2个NGG位点(No shift)可以显著提高基因的编辑效率, 推测No shift sgRNA类型靶序列引导Cas9发挥基因组编辑时, Cas9在确定最适作用位点时需要在基因组的作用区域发生滑动, 该过程类似于RNA聚合酶识别启动子的过程[34,35], 连续的NGG碱基组成相当于双PAM结构, 可能增加了Cas9识别切割位点的效率, 从而使得Cas9-sgRNA-DNA编辑复合体更稳定, 编辑效率也因此更高。另外在对编辑效率统计时还发现, 转化不同类型双靶序列编辑效率均高于单靶序列, 原因可能是更多的DNA双链断裂激活了细胞体内的错误倾向修复的SOS应急反应, 使细胞有了更高的突变率[36]。但该结论还需要进一步扩大实验规模来验证。Cas9和相应sgRNA的表达量在不同载体转化原生质体细胞内没有明显差异, 表明出现的编辑效率的差异不是因为基因表达差异引起的, 而是因为sgRNA的选择不同。3.2 靶序列类型对脱靶效率影响
对转化不同类型靶序列后脱靶效应测序统计发现, 转化双靶序列脱靶效率显著低于单靶序列, 推测上述原因, 一方面, 由转化不同数量靶序列结果分析, 转化双靶序列进行基因组编辑时, 由于Cas9在细胞内的表达量基本相似(从Cas9半定量RT-PCR结果可以得出), Cas9核酸酶在同一细胞内需同时编辑2个基因组位点, 相比单一位点基因组编辑(转化一个靶序列类型), Cas9作用更加分散, 冗余性更低, 在发挥基因组编辑效应时, 双靶序列对Cas9蛋白的竞争作用强于单靶序列, 作用于脱靶靶序列的Cas9蛋白效应相比单靶序列更弱, 因此造成的脱靶效率相应较低。另一方面, No shift sgRNA类型靶序列在设计过程中选择3°末端带有连续“NGG”结构作为候选靶序列, 使得No shift sgRNA靶序列与基因组匹配特异性较Shift sgRNA靶序列类型特异性提高约16倍(基因组具有连续5°-NGG-3°结构的位点数目少于单一NGG结构), 同时由No shift sgRNA靶序列产生的脱靶序列与基因组匹配度特异性降低了约256倍, No shift sgRNA靶序列与基因组匹配度提高有利于增强sgRNA与基因组结合稳定性, 同时细胞内相应sgRNA表达量仍处于同一水平(从sgRNA半定量RT-PCR结果可以得出), 因此编辑效率也相应提高, 从降低脱靶效率角度, No shift sgRNA靶序列对避免脱靶的贡献率更加显著。长期以来, 棉花功能基因组学研究由于缺乏适宜创制突变体的方法, 使得对棉花功能基因挖掘及其分子育种工作都造成严重影响。棉花遗传转化由于操作困难, 受基因型影响限制了棉花功能基因组学研究工作。CRISPR/Cas9基因组编辑技术的出现为棉花功能基因组学研究拓宽了思路, 有利于针对性地创制棉花突变体。随着该技术在近几年发展逐渐趋于成熟, CRISPR/Cas9技术也正如当初人类基因组计划, 可以畅想, CRISPR/Cas9技术也必将呈现出一个以高效、精准、高通量基因编辑为代表的后基因组编辑时代。然而目前, 棉花基因组编辑研究面临着与拟南芥等物种相同的现象, 如何在棉花中实现精准高效的基因组编辑, 需要对CRISPR/Cas9基因组编辑系统进一步优化, 提高编辑效率, 降低脱靶效率, 本研究为利用CRISPR/Cas9技术对棉花功能基因组学研究奠定了一定的基础。
4 结论
转化不同类型双靶序列编辑效率大部分显著高于单靶序列(转化GGB-sgRNA1-GGB-sgRNA2和Shift GGB-sgRNA1-GGB-sgRNA2差异不显著), 其对应的脱靶效率较单靶序列均有所降低, 转化No shift sgRNA类型靶序列编辑效率显著高于转化Shift sgRNA。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
