 ,2,*, 张洁夫
,2,*, 张洁夫 ,1,*, 傅廷栋2
,1,*, 傅廷栋2Genome-wide association study of silique length in rapeseed (Brassica napus L.)
SUN Cheng-Ming1,2, CHEN Song1, PENG Qi1, ZHANG Wei1, YI Bin ,2,*, ZHANG Jie-Fu
,2,*, ZHANG Jie-Fu ,1,*, FU Ting-Dong2
,1,*, FU Ting-Dong2通讯作者:
收稿日期:2019-02-12接受日期:2019-05-12网络出版日期:2019-05-17
| 基金资助: |
Received:2019-02-12Accepted:2019-05-12Online:2019-05-17
| Fund supported: |
作者简介 About authors
E-mail:suncm8331537@gmail.com。

摘要
关键词:
Abstract
Keywords:
PDF (1365KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
孙程明, 陈松, 彭琦, 张维, 易斌, 张洁夫, 傅廷栋. 甘蓝型油菜角果长度性状的全基因组关联分析[J]. 作物学报, 2019, 45(9): 1303-1310. doi:10.3724/SP.J.1006.2019.94021
SUN Cheng-Ming, CHEN Song, PENG Qi, ZHANG Wei, YI Bin, ZHANG Jie-Fu, FU Ting-Dong.
油菜是我国重要的油料作物, 种植面积约690万公顷, 产油量占国产植物油总量的55%以上, 是我国最大的食用植物油来源[1]。作为食用油消费大国, 我国食用油自给率仅35%左右, 因此, 提高产量成为油菜育种最迫切的目标。角果长度是油菜重要的产量相关性状, 解析其遗传基础对提高油菜产量具有重要意义。
角果是油菜重要的光合产物贮存器官, 适度增加角果长度, 可增加角果库容量, 有利于灌浆物质向籽粒中运输, 每角粒数和籽粒大小也随之提高[2]。此外, 角果还是油菜成熟后期重要的光合器官, 在角果完全伸展后, 角果皮面积占全株光合面积的60.6%[3]; 对角果遮光处理导致油菜的千粒重降低54.7%、籽粒产量降低62.1%、含油量降低44.2%[3]。冷锁虎等[2]研究表明, 角果长度每增加1 cm, 群体中角果皮面积占总光合面积的比例增加2.64%。因此适度增加角果长度有利于扩大角果库容量, 增加全株光合面积, 从而提高油菜的籽粒产量。
油菜角果长度是受多基因调控的数量性状, 众多研究者采用遗传差异较大的双亲构建连锁群体, 进行角果长度的QTL定位。Udall等[4]定位的角果长度QTL分布在A09、C02和C08连锁群, 其中A09连锁群的QTL ln9.5贡献率为25.3%[4]; Yang等[5]定位到的QTL分布在A01、A02、A06、A07、A09、C02、C03和C04连锁群, 其中A09连锁群的QTL qSLA9.3能解释65.6%的表型变异; 漆丽萍[6]在A01、A07、A09和A10连锁群检测到11个QTL, 其中贡献率最大的2个QTL均位于A09, 在3个环境中分别解释38.3%~60.0%和11.2%~16.5%的表型变异; Wang等[7]在A05、A06、C01、C05和C06检测到11个QTL, 解释3.4%~14.36%的表型变异; Fu等[8]在A01、A05、A09和C08连锁群上检测到20个角果长度QTL, 效应最大的2个QTL均位于A09; Yang等[9]在A05、A09、C02、C08和C09检测到7个角果长度QTL, 位于C09的主效位点解释26.51%的表型变异。
近年来, 随着高密度SNP基因分型芯片和全基因组测序等技术的发展, 全基因组关联分析(genome-wide association study, GWAS)已广泛应用于玉米[10,11]、水稻[12,13]、油菜[14,15]等作物复杂性状的遗传结构解析。本研究利用60K SNP芯片对群体进行基因型分析, 并对角果长度进行全基因组关联分析, 旨在挖掘显著的SNP位点, 分析其候选基因, 为油菜角果长度的遗传改良奠定基础。
1 材料与方法
1.1 试验材料
用于关联分析的496份甘蓝型油菜包括地方品种、育成品种及高世代育种材料。其中国内资源444份, 主要来自湖北、重庆、江苏、湖南、四川、陕西等油菜主产省市; 国外资源52份, 主要来自德国、瑞典、朝鲜、加拿大等国家(附表1)。所有材料均由华中农业大学国家油菜工程技术研究中心提供。Table 1
表1
表1关联群体角果长度性状的统计分析
Table 1
| 环境 Environment | 最小值 Min. | 最大值 Max. | 平均值±标准差 Mean ± SD | 变异系数 CV |
|---|---|---|---|---|
| 2013/2014 Nanjing | 3.42 | 11.37 | 5.40±1.01 | 0.19 |
| 2013/2014 Nanjing | 3.28 | 15.80 | 6.39±1.25 | 0.20 |
| 2014/2015 Taizhou | 3.31 | 10.18 | 5.79±0.88 | 0.15 |
| 2015/2016 Taizhou | 4.01 | 9.94 | 5.90±0.77 | 0.13 |
新窗口打开|下载CSV
1.2 田间试验与表型调查
2013年、2014年于江苏南京(13NJ和14NJ), 2015年、2016年于江苏泰州(15TZ和16TZ), 采用完全随机区组设计种植自然群体, 在南京设置3次重复, 在泰州设置2个重复。单个材料每重复种2行, 每行15株, 行宽1.5 m, 行距40 cm, 行长1.5 m。每年10月上旬大田直播, 田间管理按当地常规方式进行。在油菜成熟期, 取每小区8株长势一致的植株, 选主花序中部10个角果测量角果长度。利用R软件对各环境的角果长度表型进行统计分析和相关性分析, 并计算广义遗传力和最佳线性无偏预测值(best linear unbiased prediction, BLUP)[16,17]。1.3 基因型检测与分析
利用Illumina 60K SNP芯片分析基因型。在Genome Studio软件中, 剔除Call Freq < 0.75, Minor Freq < 0.05, AA, BB frequencies < 0.03及GenTrain Scores < 0.50的SNP, 基因型杂合的SNP按缺失值处理。将过滤后的33,218个SNP序列比对至油菜Darmor-bzh基因组, E-value阈值设为10-12, 保留19,167个单拷贝、位置清楚的SNP标记用于后续分析。1.4 全基因组关联分析
用Structure 2.3.3计算群体结构Q矩阵 [18], 用SPAGeDi计算材料间的亲缘关系K矩阵[19]。将基因型、表型、Q矩阵和K矩阵导入Tassel 4.0后, 以Q矩阵和Q+K矩阵作协变量, 分别进行基于一般线性模型(General Linear Model, GLM)和混合线性模型(Mixed Linear Model, MLM)的全基因组关联分析[20]。关联分析的显著性阈值 (Bonferroni threshold) 定为1/总标记数, 即-lg (P) = 4.28。当1 Mb区间内存在多个显著SNP时, 如果两两间的r2 ≥ 0.1, 则将这些SNP归为一个关联位点, 以最小P值的SNP作为代表。用R软件包qqman绘制曼哈顿图和QQ (Quantile-Quantile)图[21]。用R软件中逐步回归分析的lm功能分析关联位点解释的总表型变异。首先, 挑选出关联区间的lead SNP, 将其中一种等位基因型标记成1, 另外一种标记成-1, 杂合和缺失位点标记成0; 利用“stepAIC”函数, 通过选择最小的AIC (Akaike information criterion)信息统计量, 逐步增加或剔除lead SNP, 最终确定最适宜的模型, 模型的R2即为总表型变异。1.5 已报道QTL的信息整理
目前有2篇角果长度的关联分析研究[22,23], 2个群体分别包含300个和157个株系, 整理其显著位点的信息并与本研究的结果比较。角果长度的连锁分析报道很多, 但多数研究的标记为SSR和AFLP等传统标记, 数目少且无法准确确定其物理位置。漆丽萍[6]和Yang等[9]分别用SNP芯片对DH群体进行基因型分析, 本研究将其角果长度QTL两侧的SNP标记比对至油菜Darmor-bzh参考基因组以获得QTL的物理区间。此外, Fu等[8]利用混池测序将一个角果长度主效QTL定位至油菜A09染色体27.21~ 28.26 Mb区段。1.6 候选基因挖掘
以r2 = 0.1作为衰减阈值, 用Tassel计算显著关联位点的LD 衰减距离作为其置信区间。提取所有显著位点置信区间内的基因CDS序列, 与拟南芥的基因CDS进行BLAST比对, E-value阈值设为10-10, 以相似度最高的拟南芥基因信息来注释油菜基因。2 结果与分析
2.1 表型统计分析
496份油菜资源的角果长度在4个环境中具有广泛的变异, 单个环境的角果长度极差范围是5.93~ 12.52 cm。单个环境表型平均值范围是(5.40±1.01) ~ (6.39±1.25) cm, 变异系数范围是0.13~ 0.20 (表1和图1)。由表2可知, 4个环境的角果长度存在极显著相关(P ≤ 0.001), 表明本研究考察的表型数据具有较高的可靠性和重复性。根据R软件包lem4的计算, 角果长度在4个环境的广义遗传力为86.0%。图1
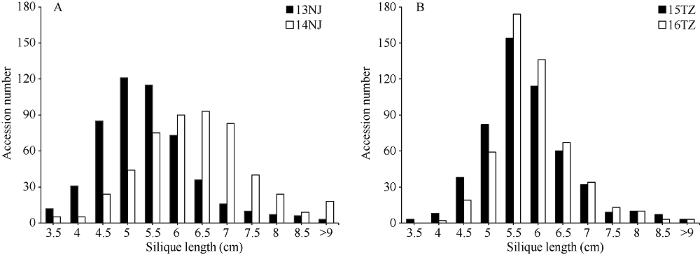 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1关联群体在4个环境的角果长度分布
A和B分别指群体在南京和泰州2年的角果长度频数分布图。
Fig. 1Distribution of silique length of the association panel in four environments
A and B, histogram of silique length in Nanjing and Taizhou in two years, respectively.
Table 2
表2
表24个环境的角果长度相关系数
Table 2
| 2012/2013 Nanjing | 2013/2014 Nanjing | 2014/2015 Taizhou | |
|---|---|---|---|
| 2013/2014 Nanjing | 0.57*** | ||
| 2014/2015 Taizhou | 0.64*** | 0.70*** | |
| 2015/2016 Taizhou | 0.56*** | 0.57*** | 0.75*** |
新窗口打开|下载CSV
2.2 角果长度全基因组关联分析
利用MLM模型, 当阈值为-lg (P) = 4.28时, 检测到15个显著SNP。合并存在连锁不平衡的SNP (r2 ≥ 0.1), 得到7个显著位点, 分布在A07、A09、C02、C04和C09染色体(表3和图2)。效应最大的位点Bn-A09-p29991443位于A09染色体[-lg (P) = 13.59], 对表型变异的贡献率为13.89%; 携带其等位基因型A的材料(48份)角果长度均值为6.62 cm, 比携带基因型T的材料(389份)角果长度均值增加0.89 cm。商品种如华油6号、华油12号、中双5号、中双7号、中双11号、宁油6号、湘油13号等, 地方品种包括南川长角、湖北白花油菜等均携带该位点的优异等位基因型(附表2)。其余位点的-lg (P)值范围是4.29~ 5.80, 可解释3.21%~5.10%的表型变异。利用一般线性模型计算, 7个位点联合解释25.01%的表型变异。与单环境的关联结果比较, 5个BLUP位点在单环境中被重复检测到, 3个位点在2个环境中得到验证, Bn-A09-p29991443位点在3个环境中被重复检测到。Table 3
表3
表3MLM角果长度显著关联位点(BLUP)
Table 3
| 标记 Marker | 染色体 Chr. | 位置 Position | -lg (P) | 表型变异 R2 (%) | 环境 Environment | 已报道QTL Reported QTL |
|---|---|---|---|---|---|---|
| Bn-A07-p8572785 | A07 | 10,018,929 | 5.80 | 5.10 | 13NJ/15TZ | |
| Bn-A09-p28925363 | A09 | 26,858,796 | 5.15 | 4.92 | 13NJ/15TZ | [8, 22] |
| Bn-A09-p29991443 | A09 | 27,815,620 | 13.59 | 13.89 | 13NJ/14NJ/15TZ | [6, 8, 22, 23] |
| Bn-scaff_15712_2-p492440 | C02 | 38,697,584 | 4.67 | 3.54 | ||
| Bn-scaff_17869_1-p813291 | C04 | 9,884,407 | 5.41 | 4.73 | 14NJ /15TZ | |
| Bn-scaff_20817_1-p60579 | C04 | 48,635,772 | 4.56 | 4.27 | 15TZ | |
| Bn-scaff_15576_1-p68473 | C09 | 41,121,951 | 4.29 | 3.21 | 13NJ/15TZ |
新窗口打开|下载CSV
图2
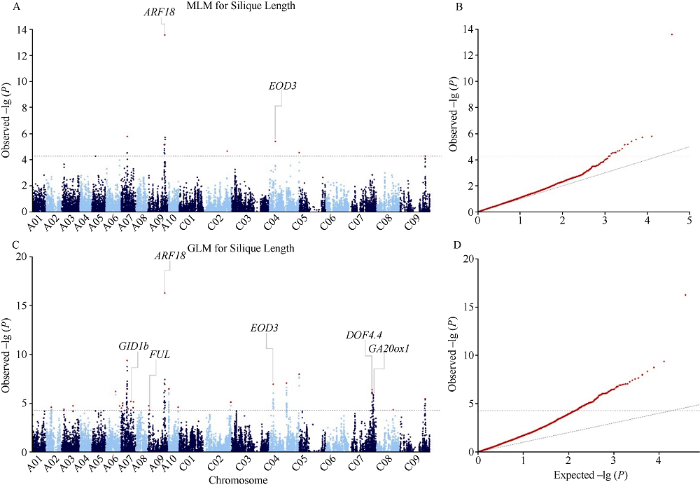 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2油菜角果长度全基因组关联分析(BLUP)
A: 角果长度MLM曼哈顿图; B: 角果长度MLM QQ图; C: 角果长度GLM曼哈顿图; D: 角果长度GLM QQ图。水平线代表Bonferroni阈值。
Fig. 2Genome-wide association study of rapeseed silique length (BLUP)
A: Manhattan plot of MLM for silique length; B: Quantile-quantile plot of MLM for silique length; C: Manhattan plot of GLM for silique length; D: Quantile-quantile plot of GLM for silique length. The dashed horizontal line depicts the Bonferroni significance threshold.
通过GLM模型共检测到136个显著SNP, 合并存在连锁不平衡的SNP后得到25个显著位点, 分布在A02、A03、A06、A07、A09、A10、C02、C04、C07、C08和C09染色体(表4和图2)。效应最大的位点和MLM相同, 在GLM模型中Bn-A09- p29991443的-lg (P) = 16.27, 对表型变异的贡献率为12.86%。其余的位点-lg (P)值范围是4.31~9.38, 可解释2.91%~7.76%的表型变异。利用一般线性模型计算, 25个位点联合解释41.77%的表型变异。与单环境的关联结果比较, 23个BLUP位点在单环境中被重复检测到, 其中8、6和5个位点分别在2、3和4个环境中得到验证。比较2个关联模型的结果, 5个MLM位点与GLM重叠, 合并重叠的位点共得到27个位点。利用一般线性模型计算, 27个位点联合解释44.04%的表型变异。
Table 4
表4
表4GLM角果长度显著关联位点(BLUP)
Table 4
| 标记 Marker | 染色体 Chr. | 位置 Position | -lg (P) | 表型变异 R2 (%) | 环境 Environment | 已报道QTL Reported QTL |
|---|---|---|---|---|---|---|
| Bn-A02-p11454573 | A02 | 8,166,563 | 4.61 | 3.72 | 13NJ/14NJ/15TZ | |
| Bn-A03-p4408895 | A03 | 3,938,915 | 4.39 | 2.91 | ||
| Bn-scaff_16092_1-p866917 | A03 | 18,418,252 | 4.74 | 4.66 | 13NJ/15TZ | |
| Bn-scaff_27914_1-p34836 | A06 | 15,544,511 | 6.21 | 5.30 | 15TZ/16TZ | |
| Bn-A06-p23042849 | A06 | 22,092,944 | 4.72 | 3.85 | 15TZ/16TZ | |
| Bn-A07-p1228602 | A07 | 860,252 | 4.51 | 3.76 | 16TZ | |
| Bn-A10-p12099059 | A07 | 2,762,935 | 5.02 | 3.38 | 15TZ/16TZ | |
| Bn-A07-p7606228 | A07 | 9,162,030 | 6.03 | 4.90 | 13NJ/15TZ/16TZ | [23] |
| Bn-A07-p8572785 | A07 | 10,018,929 | 9.38 | 7.76 | 13NJ/15TZ/16TZ | |
| Bn-A07-p10340211 | A07 | 11,509,208 | 4.45 | 3.70 | 15TZ/16TZ | |
| Bn-A07-p13957160 | A07 | 15,884,413 | 5.18 | 4.20 | 15TZ/16TZ | [23] |
| Bn-A07-p18319586 | A07 | 20,221,220 | 5.14 | 4.33 | 14NJ/15TZ | [6, 23] |
| Bn-A09-p3051349 | A09 | 2,971,335 | 4.31 | 3.32 | 15TZ | |
| Bn-A09-p28925363 | A09 | 26,858,796 | 6.08 | 4.89 | 13NJ/15TZ/16TZ | [8, 22] |
| Bn-A09-p29991443 | A09 | 27,815,620 | 16.27 | 12.86 | 13NJ/14NJ/15TZ/16TZ | [6, 8, 22, 23] |
| Bn-A10-p3966740 | A10 | 885,133 | 6.47 | 4.46 | 13NJ/14NJ/15TZ/16TZ | [6] |
| Bn-A10-p15793623 | A10 | 15,756,213 | 4.60 | 3.65 | ||
| Bn-scaff_17177_1-p381225 | C02 | 44,843,894 | 5.12 | 4.13 | 15TZ | |
| Bn-A05-p4304172 | C04 | 6,449,454 | 6.96 | 5.75 | 14NJ/15TZ/16TZ | |
| Bn-scaff_27914_1-p9919 | C04 | 28,255,420 | 7.06 | 6.09 | 13NJ/14NJ/15TZ/16TZ | |
| Bn-scaff_20817_1-p60579 | C04 | 48,635,772 | 7.98 | 6.89 | 13NJ/14NJ/15TZ/16TZ | |
| Bn-scaff_16069_1-p1668600 | C07 | 38,086,007 | 6.38 | 5.29 | 15TZ/16TZ | |
| Bn-scaff_16069_1-p3731985 | C07 | 40,142,298 | 5.92 | 4.63 | 13NJ/14NJ/15TZ/16TZ | |
| Bn-scaff_16361_1-p241830 | C08 | 27,753,144 | 4.34 | 3.48 | 13NJ | [9] |
| Bn-scaff_15576_1-p68473 | C09 | 41,121,951 | 5.47 | 3.72 | 14NJ/15TZ/16TZ |
新窗口打开|下载CSV
2.3 与前人报道QTL的比较
综合MLM和GLM的结果, 与已报道的角果长度QTL结果比较, 本研究发现的7个位点与前人研究结果一致(表3和表4)。周庆红等[22]、漆丽萍[6]、Fu等[8]、Dong等[23]通过不同的定位策略检测到效应最大的位点均落在A09染色体27.26~28.61 Mb区段。本研究中效应最大的位点Bn-A09-p29991443也同样落在该区间。位于A07的位点Bn-A07- p18319586物理位置是20.22 Mb, 与Dong等[23]检测到的角果长度显著位点(范围为19.76~20.57 Mb)重叠, 紧邻漆丽萍[6]检测到的QTL qSL.A7.2 (范围为21.04~22.52 Mb)。位于C08的位点Bn-scaff_16361_ 1-p241830物理位置是27.75 Mb, 与Yang等[9]检测到的QTL cqSL.C08a (范围为25.92~27.79 Mb)重叠。此外, 还有4个位点与前人检测的QTL重叠或紧邻。这些结果证实了本研究结果的可靠性。此外, 综合MLM和GLM的结果, 本研究共检测到20个新位点。多个位点效应较高, 如位于A07的Bn-A07-p8572785表型贡献率为9.38%, 位于C04的Bn-scaff_20817_1-p60579表型贡献率为7.98%。多个位点在多环境中被检测到, 可信度高, 例如位于C04染色体的位点Bn-scaff_27914_1-p9919和Bn- scaff_20817_1-p60579及位于C07染色体的位点Bn- scaff_16069_1-p3731985, 这3个位点在4个环境中均被检测到。这些位点值得进一步验证和研究。2.4 候选基因挖掘
基于中双11号基因组注释信息, 在Bn-A09- p29991443位点下游153 kb和184 kb处分别找到油菜角果长度已知基因ARF18和BnaA9.CYP78A9 (表5)。ARF18编码一个生长素响应因子, 通过生长素信号途径调节角果皮的细胞生长[24]。BnaA9.CYP78A9编码一个细胞色素P450单加氧酶, 通过调节细胞的伸长影响角果长度[25]。基于Darmor-bzh基因组注释信息, 在Bn-A09-p3051349位点上游252 kb处找到一个候选基因BnaA09g05500, 该基因的拟南芥同源基因FUL编码一个MADS-box转录因子, 该基因突变导致植株角果无法伸长, 果实不能正常开裂[26]。在Bn-scaff_ 20817_1-p60579位点上游293 kb处找到一个候选基因BnaC04g50960, 该基因的拟南芥同源基因EOD3编码一个细胞色素P450单加氧酶, 其功能缺失突变体角果长度缩短、种子变小[27]。在Bn-scaff_16069_1-p1668600位点下游490 kb处找到一个候选基因BnaC07g36530, 该基因在拟南芥中的同源基因DOF4.4属于Dof转录因子家族, RNAi株系角果增长、产量增多[28]。此外, 本研究还找到别的候选基因, 如拟南芥已知基因GID1b和ga20ox1在油菜中的同源拷贝[29,30]。Table 5
表5
表5角果长度关联位点候选基因信息
Table 5
| 标记 Marker | 油菜基因 Rapeseed gene | 染色体 Chr. | 位置 Position | 拟南芥同源基因 Ar. homolog | 参考基因组 Ref. genome |
|---|---|---|---|---|---|
| Bn-A07-p13957160 | BnaA07g19530 | A07 | 15,590,253 | GID1b | Darmor-bzh |
| Bn-A09-p3051349 | BnaA09g05500 | A09 | 2,718,832 | FUL | Darmor-bzh |
| Bn-scaff_20817_1-p60579 | BnaC04g50960 | C04 | 48,343,005 | EOD3 | Darmor-bzh |
| Bn-scaff_16069_1-p1668600 | BnaC07g36530 | C07 | 38,575,790 | DOF4.4 | Darmor-bzh |
| Bn-scaff_16069_1-p3731985 | BnaC07g39650 | C07 | 40,392,394 | GA20ox1 | Darmor-bzh |
| Bn-A09-p29991443 | BnA09g0377700 | A09 | 36,712,502 | ARF18 | ZS11 |
| Bn-A09-p29991443 | BnA09g0377760 | A09 | 36,740,578 | CYP78A9 | ZS11 |
新窗口打开|下载CSV
3 讨论
角果长度是油菜重要的农艺性状, 适度增加角果长度有利于扩大角果库容量, 增加植株光合面积, 从而提高油菜的籽粒产量。因此, 众多研究者围绕角果长度开展了一系列的定位工作, 在油菜19条染色体上均检测到QTL, 对表型变异的贡献率为3.4%~65.5%[4-9,22,23]。本研究对496份油菜资源的角果长度进行4个环境的考察, 通过全基因组关联分析挖掘到27个显著位点, 分布在A02、A03、A06、A07、A09、A10、C02、C04、C07、C08和C09共11条染色体, 对表型变异的贡献率为2.91%~ 13.89%。多个研究者在A09染色体27.26~28.61 Mb区段检测到主效QTL, 最高可解释超过60%的表型变异[5,6,8,22-23]。本研究中效应最大的位点Bn-A09- p29991443同样落在该区间。此外, 本研究还检测到另外6个与前人结果共定位的位点及20个新位点, 所有位点联合解释44.04%的表型变异。可将携带不同有利等位基因的材料杂交, 利用与上述QTL紧密连锁的分子标记进行辅助选择, 聚合更多有利等位基因, 培育角果长度理想的油菜新品种。目前油菜中已克隆的角果长度基因较少, Liu等[24]和Shi等[25]分别报道在A09染色体克隆了角果长度基因ARF18和BnaA9.CYP78A9。2个基因均落在本研究最显著的位点Bn-A09-p29991443所在区段, 相距28.1 kb, 处于紧密连锁状态。可能由于2个基因效应叠加, 致使该区间的显著性很高。此外, 在对应的C08同源区段检测到微弱的关联信号(GLM, -lg (P) = 3.72), 表明ARF18和BnaA9. CYP78A9位于C08的同源拷贝, 对角果长度的表型贡献很小, 与Liu等[24]和Shi等[25]的研究结果一致。Bn-A07-p8572785是用MLM和GLM检测到显著性较高的位点, 但在附近未找到角果长度已知基因。分析其附近基因的注释信息, 找到一个候选基因BnaA07g10150, 该基因位于Bn-A07-p8572785上游339 kb处, 其拟南芥同源基因PIN7编码一个生长素运输基因。生长素调节细胞的伸长生长, ARF18通过生长素途径调节角果长度, 因此BnaA07g10150的功能值得进一步研究。Bn-scaff_20817_1-p60579是GLM模型中显著性较高的位点, 根据基因注释信息在该位点附近发现重要候选基因BnaC04g50960, 其拟南芥同源基因EOD3同时调控角果长度和籽粒大小[27], 后续研究可以通过转基因或CRISPR敲除验证该基因的功能。
4 结论
用MLM检测到7个位点, 联合解释25.01%的表型变异; 用GLM检测到25个位点, 联合解释41.77%的表型变异。合并共同的位点后得到27个位点, 其中7个位点与前人QTL共定位, 其余20个是新位点。效应最大的位点Bn-A09-p29991443位于A09染色体, 在其附近检测到油菜已克隆的角果长度基因ARF18和BnaA9.CYP78A9。此外, 在其他多个位点附近发现拟南芥已知角果长度基因GID1b、FUL、EOD3、DOF4.4和GA20ox1的同源拷贝。附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical-zuowxb. aspx。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
.
[本文引用: 2]
[本文引用: 3]
[本文引用: 2]
[本文引用: 5]
[本文引用: 5]
[本文引用: 1]
[本文引用: 4]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
.
[本文引用: 4]
[本文引用: 5]
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
