 ,*河北师范大学生命科学学院, 河北石家庄 050024
,*河北师范大学生命科学学院, 河北石家庄 050024Expression of OsUBA and its function in promoting seed germination and flowering
ZHANG Shuang-Shuang**, WANG Li-Wei**, YAO Nan, GUO Guang-Yan, XIA Yu-Feng, BI Cai-Li ,*Life Science College, Hebei Normal University, Shijiazhuang 050024, Hebei, China
,*Life Science College, Hebei Normal University, Shijiazhuang 050024, Hebei, China通讯作者:
第一联系人:
收稿日期:2018-12-25接受日期:2019-05-12网络出版日期:2019-05-17
| 基金资助: |
Received:2018-12-25Accepted:2019-05-12Online:2019-05-17
| Fund supported: |
作者简介 About authors
E-mail:zhangshuang1597@126.com。

摘要
关键词:
Abstract
Keywords:
PDF (1763KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
张双双, 王立伟, 姚楠, 郭光艳, 夏玉凤, 秘彩莉. 水稻OsUBA基因的表达及其在促进种子萌发和开花中的功能[J]. 作物学报, 2019, 45(9): 1327-1337. doi:10.3724/SP.J.1006.2019.82069
ZHANG Shuang-Shuang, WANG Li-Wei, YAO Nan, GUO Guang-Yan, XIA Yu-Feng, BI Cai-Li.
在真核生物中, 自噬是在液泡(植物和酵母)或溶酶体(动物)中将胞内组分降解的细胞学过程。参与自噬途径的基因称为自噬相关基因(autophagy-associated genes, ATG)[1]。在营养缺乏时, 生物通过非选择性自噬途径将胞内组分(包括蛋白质和细胞器)大规模降解, 以实现营养物质的回收和再利用[2]。此外, 通过自噬途径还可以将受损的细胞器和有毒的大分子选择性地清除, 即选择性自噬。选择性自噬由自噬受体介导, 自噬受体不仅能识别自噬底物, 还能与自噬体的标记蛋白ATG8 (在哺乳动物中也称LC3, microtubule-associated protein 1 light chain 3)相互作用, 从而将自噬底物运送至自噬体进行降解[3]。
p62 (也称Sequestosome-1, SQSTM1)和NBR1 (Next to BRCA1 gene 1, NBR1)是哺乳动物中最主要的选择性自噬受体[4,5], 两者的结构域组成和排列顺序相似——N端的PB1 (Phox and Bem1p, PB1)结构域, 中间的锌指结构域ZZ, C端的UBA (Ubiquitin associated, UBA)结构域。UBA结构域介导了p62和NBR1与单泛素和/或多聚泛素链间的结合, PB1结构域则介导其自身的多聚化及与其他有PB1结构域的蛋白间的相互作用[5,6,7]。
虽然p62在人类中的研究已经非常广泛而深入, 但其在植物中同源蛋白的研究却屈指可数, 迄今只在拟南芥、烟草和马铃薯中有过相关报道。与p62相比, 拟南芥中的NBR1无论在结构域组成还是氨基酸序列上都与哺乳动物中的NBR1更相似。AtNBR1有2个UBA 结构域, 但只有C末端的UBA结构域能够与泛素结合[8]。AtNBR1在热、氧化、盐和干旱等非生物胁迫反应中发挥着重要作用, 而且这些生物学功能依赖于NBR1与ATG8的相互作用[9]。Ntjoka2是烟草中的NBR1蛋白。Ntjoka2能够与ATG8f 结合; 过量表达Ntjoka2的烟草植株对营养缺乏的反应减弱[10]。Ntjoka2可以以单体或寡聚体的形式存在; Ntjoka2通过PB1-PB1间的相互作用参与自噬体的形成[11]。此外, Joka2类蛋白(即NBR1蛋白)也参与植物的防御反应。PexRD54是来源于爱尔兰马铃薯致病疫霉的效应蛋白, PexRD54与寄主ATG8CL间的结合刺激了自噬体的形成。PexRD54可以将Joka2从ATG8CL复合体中解离出来从而干扰Joka2在植物病原防御反应中的积极作用[12]。综上所述, 已报道的植物中的NBR1类蛋白参与了生物/非生物胁迫反应以及对营养缺乏的响应, 但尚未有此类蛋白参与植物发育过程的相关报道。
本实验室前期从小麦中克隆了一个含有UBA结构域的基因, 将其命名为TaUBA。分析表明, TaUBA的结构域组成和排列方式与人类的p62及NBR1类蛋白非常相似。研究表明, 过量表达TaUBA的拟南芥株系耐盐性下降; 去掉UBA结构域或ZZ结构域的点突变可解除TaUBA对盐胁迫反应的抑制效应[13]。由于小麦的转化效率较低, 在小麦中研究基因的功能比较困难。为了深入研究NBR1类蛋白在植物中的功能, 本实验室通过Blast找到了TaUBA在水稻中的同源蛋白并将其命名为OsUBA (XP_015625351)。结构域分析表明, OsUBA与TaUBA、AtNBR1以及Ntjoka2的结构域组成一致, 是水稻中的NBR1类蛋白, 即水稻中的自噬受体。
本研究拟对水稻中的NBR1类蛋白OsUBA进行生物信息学分析, 通过对OsUBA p:Gus水稻株系的Gus染色研究OsUBA的表达特性, 通过在水稻中过量表达OsUBA研究OsUBA的功能。
1 材料与方法
1.1 试验材料
1.1.1 植物材料及处理 水稻(Oryza sativa L.)日本晴种子由本院朱正歌教授惠赠。水稻日本晴和各转基因水稻株系均种植于河北师范大学(河北省石家庄市)院内的水稻池中。用于开花期统计的OsUBA过表达水稻株系及野生型分别于2017年6月3日种植、11月10日收获; 2018年5月17日种植、11月8日收获。在种子萌发率统计试验中用到的水稻材料种植于2017年5月18日、收获于11月10日。1.1.2 主要试剂和菌株 RNA 提取试剂RNAiso Plus、cDNA第1链合成试剂盒Reverse Transcriptase M-MLV (RNase H-)、限制性核酸内切酶和T4 DNA连接酶均购自Takara (大连)公司; pGEM-T载体购自Promega生物技术有限公司, TransTaq HiFi DNA Polymerase购自北京全式金生物技术有限公司; 大肠杆菌感受态细胞 DH5α感和农杆菌感受态细胞EHA105为实验室自行制备, SanPrep柱式DNA胶回收试剂盒、普通Taq酶及其他生化试剂购自生工生物工程(上海)股份有限公司。
1.2 试验方法
1.2.1 水稻OsUBA基因的克隆与序列分析 基于 OsUBA的mRNA序列(XM_015769865), 应用Primer 5设计引物OsUBA-F1 (5′-ATGTCCGGCCGG AGCTCG-3′)和OsUBA-R1 (5′-TCACTGGTCTTTCT TCTCCCT-3′)用于OsUBA的扩增。提取正常培养5 d的水稻日本晴根的总 RNA, 然后将其反转录为cDNA第1链作为模板扩增OsUBA的开放阅读框ORF。PCR 扩增程序为94℃预变性5 min; 94℃变性30 s, 58℃退火30 s, 72℃延伸2 min, 30个循环; 72℃再延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测及胶回收纯化后, 与pGEM-T载体16℃连接过夜(约10 h), 转化大肠杆菌DH5α杆感受态细胞, 将PCR鉴定呈阳性的克隆送生工生物工程(上海)股份有限公司测序, 测序正确的阳性克隆命名为T-OsUBA。利用在线分析软件SignalP 4.1 Server (http:// www.cbs.dtu.dk/services/SignalP/)、TMHMM Server v. 2.0 (http://www.cbs.dtu.dk/services/TMHMM/)和NetPhos 2.0 Server (http://www.cbs.dtu.dk/services/NetPhos/)分别分析OsUBA基因编码蛋白的信号肽序列、跨膜结构域以及磷酸化位点数目; 利用Psort (http://www. psort.org/) 软件预测OsUBA蛋白的亚细胞定位情况。
1.2.2 水稻OsUBA基因启动子区域的克隆与序列分析 根据水稻参考基因组(Oryza sativa japonica Group cultivar Nipponbare chromosome 2, IRGSP- 1.0) Os02g0593700上游的基因组序列信息 (22,981,857~22,989,595)设计引物OsUBAp-F1 (5'-AA GCTTCACAAAGTACAATGGGAACAAA-3', Hind III)和OsUBAp-R1 (5'-GTCGACCGCGGGGGTGCGG GC-3', Sal I), 以正常培养2周的日本晴幼苗叶片的 DNA 为模板进行PCR扩增, PCR程序为: 94℃预变性5 min; 94℃变性30 s, 60℃退火30 s, 72℃延伸4 min, 35个循环; 72℃再延伸10 min。PCR产物经电泳、胶回收、连接pGEM-T载体和转化大肠杆菌DH5α后测序验证, 将验证正确的克隆命名为T-OsUBAp1, 并利用PlantCARE[14]在线分析软件分析OsUBA启动子的序列特征。
1.2.3 OsUBA p:Gus载体和OsUBA基因过量表达载体的构建 以含T-OsUBAp1的菌液为模板, 以OsUBAp-F2 (5'-AAGCTTCACAAAGTACAATGGGA ACAAA-3', Hind III)和OsUBAp-R2 (5'-GTCGACCG CGGGGGTGCGGGC-3', Sal I)扩增带有Hind III/Sal I酶切位点的OsUBA启动子片段, 将PCR产物胶回收后连接pGEM-T载体, 转化E. coli DH5α感受态细胞, 挑取阳性克隆测序验证, 验证正确的克隆命名为T-OsUBAp2。提取pCAMBIA1391Z和T-OsUBAp2的质粒, 分别用Hind III/Sal I进行双酶切, 电泳回收目的条带, 连接、转化大肠杆菌DH5α, 阳性克隆经菌液PCR和Hind III/Sal I双酶切进行鉴定, 鉴定正确的克隆即为构建成功的OsUBA p:Gus载体。
以含T-OsUBA的菌液为模板, 以OsUBA-F2 (5'-TCTAGAATGTCCGGCCGGAGCT-3', Xba I)和OsUBA-R2 (5'-GGTACCTCACTGGTCTTTCTTCTC- 3', Kpn I)扩增带有Xba I和Kpn I酶切位点的OsUBA基因的 CDS片段, 将PCR产物胶回收后连接pGEM-T载体, 转化E. coli DH5α, 阳性克隆经测序验证后命名为T-OsUBA-OX。提取pCAMBIA1300和T-OsUBA-OX的质粒, Xba I/Kpn I双酶切(37℃, 3 h)后电泳并回收目的条带, 16℃连接过夜(约10 h), 将连接产物转化大肠杆菌DH5α, 阳性克隆经菌液PCR和Xba I/Kpn I双酶切鉴定, 鉴定正确的克隆命名为35S: OsUBA, 即构建成功的OsUBA过表达载体。
1.2.4 OsUBA p:Gus和35S:OsUBA表达载体转化水稻 提取OsUBA p:Gus和35S:OsUBA的质粒, 分别转化根癌农杆菌菌株EHA105感受态细胞, 然后将转化产物涂布于含有卡那霉素(50 μg mL-1)和利福平(50 μg mL-1 )的YEP固体培养基上, 经28℃培养72 h, 挑取单克隆分别进行PCR检测及质粒回转大肠杆菌后的双酶切鉴定。鉴定正确的农杆菌克隆可用于转化水稻。
利用农杆菌介导法[15]将含有OsUBA p:Gus和35S:OsUBA质粒的EHA105重组菌株分别转化水稻日本晴, 转化得到T0代转基因水稻株系。将收获的T0转基因水稻种子经过连续2代潮霉素(50 μg mL-1)筛选直至获得T2代转基因水稻纯合株系。
1.2.5 OsUBA p:Gus和35S:OsUBA水稻株系RNA水平的鉴定 对按上述方法筛选到的T2代转基因水稻纯合株系进行RNA水平检测。
采用TRIzol法提取正常培养10 d的T2代OsUBA p:Gus水稻株系根的总RNA并反转录成cDNA, 利用RT-PCR检测Gus (所用引物为Gus F: 5'-GGGCAGGCCAGCGTATCG-3'和Gus R: 5'-CGGC GAAATTCCATACCTG-3')基因的表达水平, 以水稻Actin 基因(GenBank登录号为AK100267.1)为内对照(引物序列为Actin F: 5'-CTTCAACACCCCTGCT ATG-3'和Actin R: 5'-TCCATCAGGAAGCTCGTAG-3')。
采用TRIzol法提取正常培养10 d的T2代35S:OsUBA水稻株系叶片的总RNA, 按照PrimeScript RT reagent Kit with gDNA Eraser (Takara, 中国大连)说明书合成cDNA第1链, 稀释10倍后作为qRT-PCR的模板。在 Primer Premier 5.0软件中设计检测OsUBA基因的qRT-PCR引物(引物序列为OsUBA qF: 5'-CGTCGTCCTCAAGGTGAAGTA-3'和OsUBA qR: 5'-CATCCAGCATGACAATATCCC-3'), 以水稻Actin 基因(GenBank登录号为AK100267.1)为内参(引物序列为: Actin-qF: 5'-CGGGAAATTGT GAGGGACAT-3'和Actin-qR: 5'-AGGAAGGCTGGA AGAGGACC-3')。采用 ABI 7300 Real-time PCR System (美国)进行qRT-PCR检测, 并按照TB Green Premix Ex Taq (Tli RNaseH Plus) (Takara, 中国大连)说明书配置反应体系。qRT-PCR 扩增程序为95℃ 30 s; 95℃ 5 s, 60℃ 34 s, 45个循环。每个样品设3个技术重复。利用2-ΔΔCt[16]计算OsUBA基因的相对表达量, 利用Graphpad Prism7软件绘制柱状图。
1.2.6 利用OsUBA p:Gus转基因水稻株系研究OsUBA的表达特性 将OsUBA p:Gus水稻植株的花、开花期主茎的茎节和第2节间的切片、萌发1 d及3 d的种子、生长7 d的幼苗分别移入加有1 mL Gus染液(Gus染色液配方: 100 mmol L-1磷酸缓冲液pH 7.0, 0.5% Triton X-100, 20%甲醇和0.5 mg mL-1 X-Gluc)的离心管中, 37℃染色, 待染出理想效果后, 吸去反应液, 用无水乙醇脱色1~6 h, 吸去乙醇, 体式显微镜下拍照记录。
用200 μmol L-1 ABA和100 μmol L-1 GA分别处理萌发2 d的种子根24 h, 然后Gus染色25 min, 通过对比Gus染色情况研究ABA和GA处理对OsUBA表达的影响。
1.2.7 OsUBA过表达水稻株系开花时间和种子萌发率的统计 本实验室于2017年和2018年连续2个年度分别对野生型水稻日本晴及3个过表达OsUBA水稻株系(L10、L14和L15)的开花时间进行了田间调查。每个植株的第1个穗子开花即记录为开花植株; 以所有株系中最早开花的植株出现的那一天定为第1天, 以后每天统计一次各株系中开花的株数, 直至所有植株开花结束为止。2个年度统计的各水稻株系及野生型均为30株。
为了研究ABA及GA处理对OsUBA过表达水稻株系(L10、L14和L15)及野生型种子萌发的影响, 将水稻日本晴及3个OsUBA过表达水稻株系的种子(各100粒)经消毒后分别点播于1/2 MS培养基、含3 μmol L-1 ABA或100 μmol L-1 GA的1/2 MS培养基上, 在光照培养箱(28℃, 16 h光照/8 h黑暗)中萌发, 每12 h统计一次萌发情况, 直至所有种子萌发为止。绘制各株系的种子萌发曲线图, 独立实验重复3次。
2 结果与分析
2.1 OsUBA基因的克隆与序列分析
水稻OsUBA基因的CDS全长为2538 bp, 编码845个氨基酸残基(图1)。其编码蛋白质的相对分子质量为93.754 kD, 理论等电点为5.31, 其中脯氨酸和亮氨酸含量最高, 分别为10.18%和8.64%; 此外, 有 107个带负电荷的氨基酸残基(Asp和Glu)和79个带正电荷的氨基酸残基(Arg和Lys), 总体带负电荷, 属亲水蛋白。生物信息学分析表明, OsUBA没有信号肽序列及跨膜结构, 预测有3个磷酸化位点(丝氨酸、酪氨酸、苏氨酸各一个)。亚细胞定位预测显示, OsUBA蛋白位于细胞核中的概率最大, 为64.3%。图1
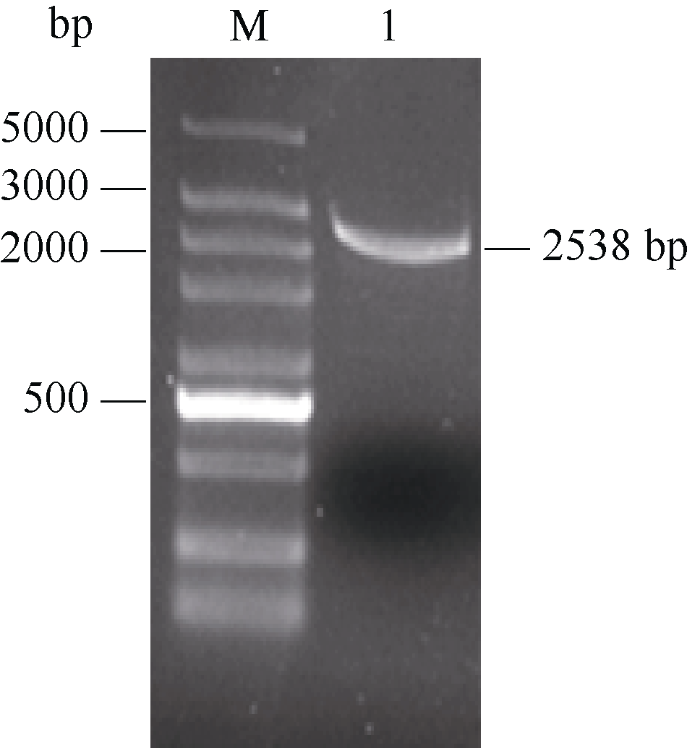 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1水稻OsUBA基因的PCR扩增
M: 5 kb DNA marker; 1: PCR 产物。
Fig. 1PCR amplification of OsUBA gene in rice
M: 5 kb DNA marker; 1: PCR product.
在NCBI网站进行的保守结构域预测显示, 该蛋白具有多个介导蛋白相互作用的结构域: 位于氨基端的PB1结构域(21~107 aa)、中间的ZZ型锌指结构域(430~461 aa)和NBR1结构域(500~613 aa)以及羧基端的2个UBA结构域(769~796 aa和800~838 aa)(图2), 预示其可与多种蛋白质相互作用, 生物学功能复杂。
图2
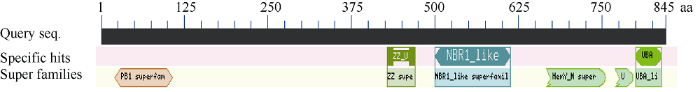 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2水稻OsUBA蛋白的保守结构域预测
Fig. 2Conserved domain prediction of OsUBA protein
将OsUBA的氨基酸序列提交至NCBI数据库, 利用Blast搜索其在水稻(Oryza sativa L.)、小麦(Triticum aestivum L.)、玉米(Zea mays L.)、谷子(Setaria italica L.)、短柄草(Brachypodium distachyon L.)、高粱(Sorghum bicolor L.)、拟南芥(Arabidopsis thaliana L.)及烟草(Nicotiana tabacum L.)等植物中的同源蛋白, 并利用MEGA6.0软件构建系统进化树(图3)。系统分析表明, 所有蛋白聚为两个大的分支, 其中, 玉米、高粱、谷子、水稻、小麦、短柄草等单子叶植物的NBR1类蛋白聚为一个分支; 拟南芥和烟草等双子叶植物的NBR1蛋白聚成另一分支。OsUBA即为水稻中的NBR1 X1亚型, 与水稻中的NBR1 X2亚型相似性最高, 氨基酸水平的一致性高达96.37%。上述结果说明, OsUBA编码水稻中的NBR1类蛋白。
图3
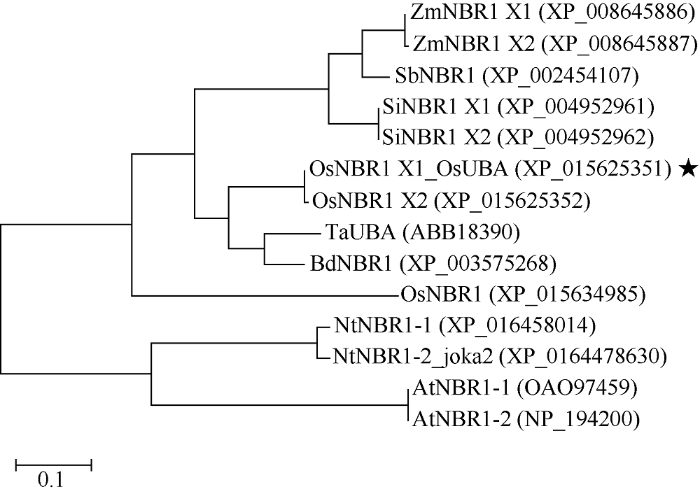 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3OsUBA蛋白及其他植物中的同源蛋白的系统进化分析
星形所示为OsUBA。
Fig. 3Phylogenetic tree of OsUBA and its homologues in plants
The star indicates OsUBA.
2.2 OsUBA基因启动子区域的克隆与分析
以水稻日本晴基因组DNA为模板扩增OsUBA启动子区域, 扩增片段长1810 bp (图4), 将该PCR产物克隆、测序, 并与数据库中的序列比对, 确认该序列为OsUBA的启动子序列。图4
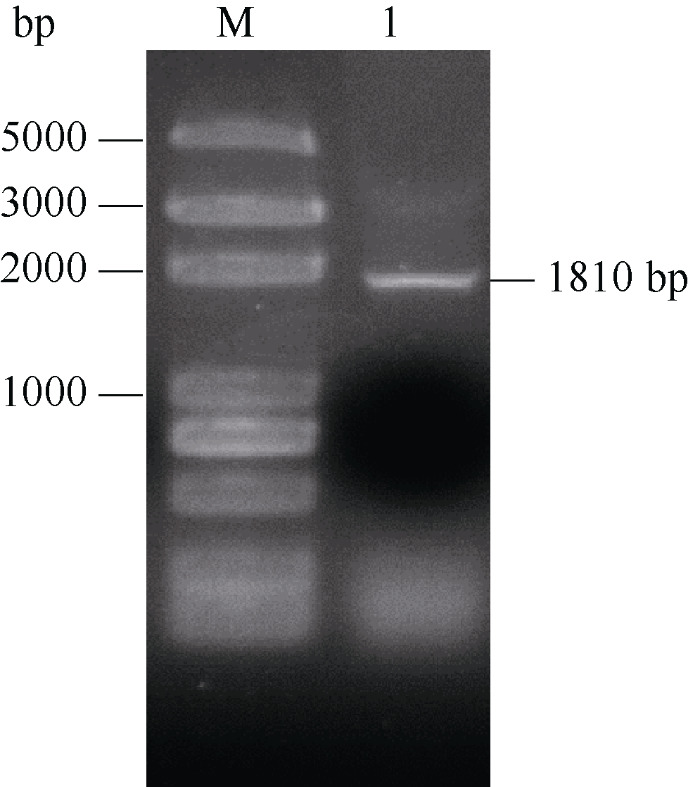 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4OsUBA基因启动子的PCR扩增
M: 5 kb DNA marker; 1: PCR产物。
Fig. 4PCR amplification of OsUBA promoter
M: 5 kb DNA marker; 1: PCR product.
在PlantCARE网站的分析显示, 在该启动子区域除含有启动子基本元件TATA-box和CAAT-box外, 还有光响应元件G-box和Sp1, 与激素相关的顺式作用元件有ABRE、CGTCA-motif、TATC-box、P- box、GARE-motif和TCA-element, 与逆境响应相关的GC-motif和LTR, 及其他组织特异性启动子元件等(表1)。
Table 1
表1
表1OsUBA基因启动子调控元件预测
Table 1
| 功能 Function | 元件名称(核心序列) Element (core sequence) |
|---|---|
| ABA响应元件 cis-acting element involved in the abscisic acid responsiveness | ABRE (ACGTG) |
| 分生组织表达调控元件 cis-acting regulatory element related to meristem expression | CAT-box (GCCACT) |
| 茉莉酸甲酯响应元件 cis-acting regulatory element involved in the MeJA-responsiveness | CGTCA-motif (CGTCA) |
| 赤霉素响应元件 cis-acting element involved in gibberellin-responsiveness | TATC-box (TATCCCA), P-box (CCTTTTG), GARE-motif (TCTGTTG) |
| 水杨酸响应元件 cis-acting element involved in salicylic acid responsiveness | TCA-element (CCATCTTTTT) |
| 缺氧特异诱导增强子样元件 Enhancer-like element involved in anoxic specific inducibility | GC-motif (CCCCCG) |
| 功能 Function | 元件名称(核心序列) Element (core sequence) |
| 低温响应元件 cis-acting element involved in low-temperature responsiveness | LTR (CCGAAA) |
| 光响应相关元件 cis-acting regulatory element involved in light responsiveness | G-box (CACGTC), Sp1 (CC(G/A)CCC) |
| 部分光反应元件 Part of a light responsive element | Box 4 (ATTAAT), GATA-motif (GATAGGG), I-box (GATAAGGTG), LAMP-element (CCTTATCCA), chs-CMA1a (TTACTTAA), chs-CMA2c (ATATACGTGAAGG) |
新窗口打开|下载CSV
2.3 OsUBA p:Gus载体的构建及转基因水稻的鉴定
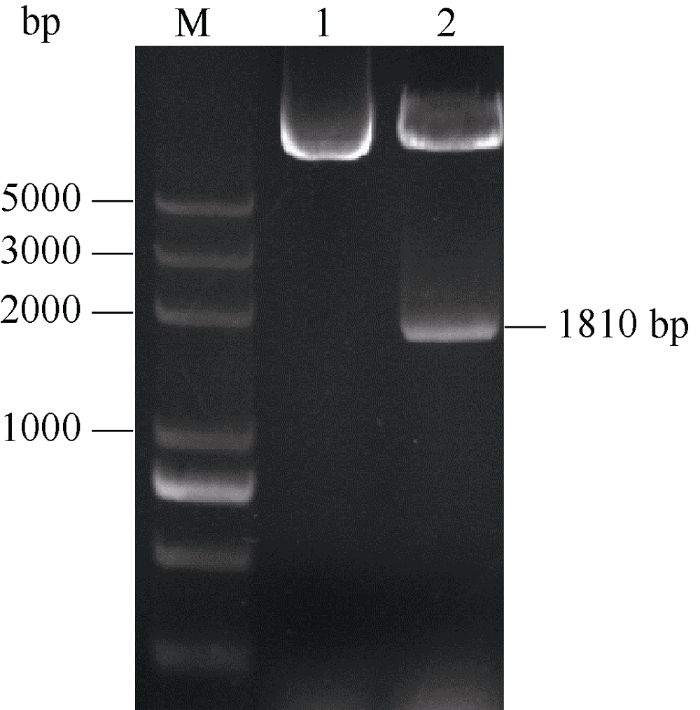
为了系统研究OsUBA基因的表达特性, 构建了OsUBA p:Gus载体。按前述方法将OsUBA启动子连接至pCAMBIA1391载体的Hind III和Sal I酶切位点之间, 阳性克隆经Hind III/Sal I酶切电泳后出现一条1810 bp的条带(图5), 说明OsUBA p:Gus载体构建成功。图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5OsUBA p:Gus载体的酶切鉴定
M: 5 kb DNA marker; 1: pCAMBIA1391-OsUBAp质粒(未酶切); 2: pCAMBIA1391-OsUBAp质粒的Hind III/Sal I双酶切鉴定。
Fig. 5Identification of OsUBA p:Gus vector by double digestion
M: 5 kb DNA marker; 1: plasmid of pCAMBIA1391-OsUBAp (undigested); 2: identification of pCAMBIA1391-OsUBAp plasmid by Hind III/Sal I digestion.
将OsUBA p:Gus载体转化水稻日本晴, 通过连续的潮霉素筛选获得2个转基因纯合株系L6和L12。提取正常培养10 d的OsUBA p:Gus水稻株系的根的总RNA, 检测转基因株系中Gus的表达。检测表明这2个株系中Gus均能正常表达(图6), 可用于后期对OsUBA表达的分析。
图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6OsUBA p:Gus水稻株系中Gus的表达分析
WT: 野生型水稻日本晴; L6和L12: 2个OsUBA p:Gus水稻纯合株系。
Fig. 6Analysis of Gus expression in OsUBA p:Gus transgenic rice plants
WT: rice cv. Nipponbare; L6 and L2: two homozygous rice lines harboring OsUBA p:Gus.
2.4 OsUBA基因的表达特性分析
按前述方法对OsUBA p:Gus水稻株系L6的各器官及正在萌发的种子进行Gus染色表明, OsUBA在水稻的根(包括种子根、冠根和侧根)和幼叶(图7-A)、茎(包括茎节和茎的节间, 图7-B, C)、花(主要是花药, 图7-D)中的表达均较强; 在萌发1 d种子的胚(图7-E)以及萌发3 d种子的胚芽鞘和胚根中的表达量也很高, 暗示其是一个遍在表达的基因, 有可能参与植物生长发育的多个生物学过程。图7
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7OsUBA在不同器官和组织中的表达特性
A: 正常生长7 d水稻的根和叶; B: 开花期主茎的茎节; C: 开花期主茎的第2节间的横切面; D: 花; E: 萌发1 d的种子; F: 萌发3 d的种子。A图中的标尺代
Fig. 7Expression of OsUBA in different organs and tissues
A: roots and leaf from rice seedlings grown in normal conditions for 7 d; B: the main stem node during flowering; C: the 2nd internode of the main stem during flowering; D: flower; E: seed germinated for 1 d; F: seed germinated for 3 d. The scale in A represents 1 cm, and the scale in B and C equals to 2 mm, the scale in D, E, and F means 1 mm.
利用200 μmol L-1 ABA和100 μmol L-1 GA分别处理萌发2 d的OsUBA p:Gus水稻株系24 h, 利用Gus染色研究了ABA和GA对OsUBA表达的影响。与处理前相比(图8-A), ABA处理显著抑制了OsUBA的表达(图8-B); GA处理后, OsUBA的表达略有升高, 但不显著(图8-C)。
图8
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8ABA和GA处理对OsUBA表达的影响
A: 未处理对照, 正常生长24 h; B: 200 μmol L-1 ABA处理24 h; C: 100 μmoL L-1 GA处理24 h; Bar = 1 mm。
Fig. 8Expression of OsUBA under ABA and GA treatments
A: untreated control, normally grown for 24 h; B: 200 μmol L-1 ABA treatment for 24 h; C: 100 μmoL L-1 GA treatment for 24 h; Bar = 1 mm.
2.5 OsUBA基因过量表达载体的构建及转基因水稻表达水平鉴定
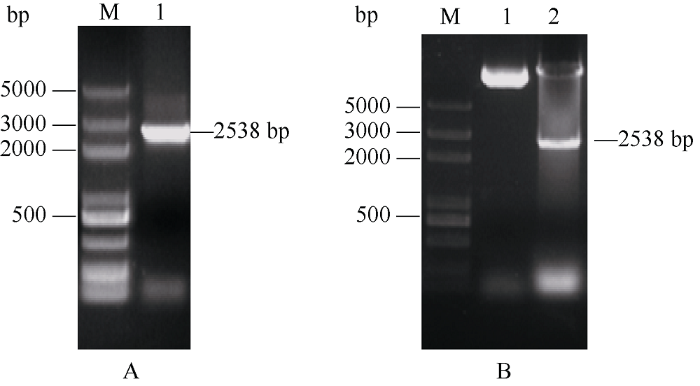
按前述方法将OsUBA基因的CDS连接至pCAMBIA1300 载体, 所得阳性克隆经菌液PCR (图9-A)和Xba I/Kpn I双酶切(图9-B)鉴定, 电泳检测结果均显示存在一条约2500 bp的目的基因条带, 表明35S:OsUBA载体构建成功。图9
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图935S:OsUBA载体的PCR及双酶切鉴定
A: pCAMBIA1300-OsUBA的菌液PCR; M: 5 kb marker; 1: 阳性克隆; B: pCAMBIA1300-OsUBA质粒的Xba I/Kpn I双酶切鉴定; M: 5 kb marker; 1: 阳性克隆质粒(未酶切); 2: 阳性克隆质粒的Xba I/Kpn I双酶切。
Fig. 9Identification of 35S:OsUBA vector by PCR and double digestion
A: amplification of positive clone harboring pCAMBIA1300- OsUBA; M: 5 kb marker; 1: positive clone; B: identification of pCAMBIA1300-OsUBA positive clone by Xba I/Kpn I digestion; M: 5 kb marker; 1: plasmid of pCAMBIA1300-OsUBA (undigested); 2: Xba I/Kpn I digestion of pCAMBIA1300-OsUBA plasmid.
将35S:OsUBA载体转化水稻日本晴, 通过连续2代的潮霉素筛选,获得3个带潮霉素抗性的T2代纯合水稻株系(L10、L14和L15)。提取这3个株系和日本晴水稻正常生长10 d的叶片的总RNA并反转录为 cDNA, 利用qRT-PCR分析OsUBA在各株系中的表达水平。结果表明, OsUBA在3个过量表达OsUBA的水稻株系中表达量都很高, 分别是野生型的31.4 (L10)、32.4 (L14)和38.4 (L15)倍(图10), 这些株系可用于后续对OsUBA功能的研究。
图10
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图10过量表达OsUBA的水稻株系中OsUBA的表达水平分析
Fig. 10Relative expression analysis of OsUBA in OsUBA- overexpressing rice plants
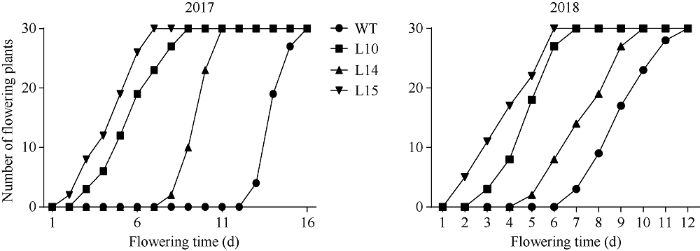
2.6 过量表达OsUBA水稻株系的开花时间统计
在2016年, 本实验室发现OsUBA过表达株系L15的开花时间明显早于野生型, 同时, 另2个株系L10和L14的开花时间虽晚于L15, 但仍比野生型早。当时发现这个现象时L15株系大部分已经开花, 因此未能在2016年完整记录这些过表达株系的开花时间的数据。本研究在随后的2017和2018连续2年继续观察、统计OsUBA过表达株系及野生型的开花时间, 获得了完整的统计数据。整体来看, 各株系在这2个年度的开花时间数据比较一致: OsUBA过表达株系中, L15开花最早, 其次是L10, 然后是L14; 2个年度的统计结果均表明这3个OsUBA过表达水稻株系的开花时间均明显早于野生型对照(图11)。图11
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图11OsUBA过表达水稻株系和野生型的开花时间比较
Fig. 11Comparison of flowering time between OsUBA-overexpressing rice plants and the wild type control
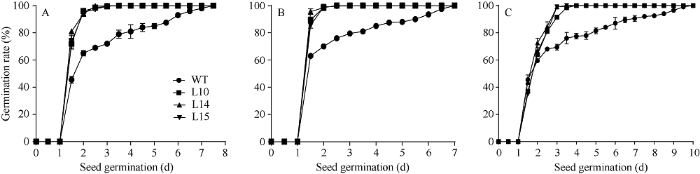
2.7 ABA和GA处理对过量表达OsUBA水稻株系种子萌发过程的影响
按前述方法研究了在正常生长条件下以及ABA (3 μmol L-1)和GA (100 μmol L-1)处理对OsUBA过表达水稻株系及野生型萌发的影响。结果表明, OsUBA过表达水稻株系的种子比野生型萌发更快, 这种差异在萌发的第1.5天、2.0天和2.5天均非常显著, 此后差距逐渐缩小。3个OsUBA过表达株系中, L10萌发最快, L15次之, L14最慢, 但3个过表达株系的萌发始终比野生型快, 到7.5 d时所有种子全部萌发(图12-A); ABA处理均抑制了OsUBA过表达水稻株系及野生型种子的萌发, 但对OsUBA过表达水稻株系的影响更大。在萌发的第1.5天影响最显著, 此时,野生型的萌发率与对照基本相同, 但3个OsUBA过表达株系仅有对照条件下的51.4% (L10)、74.9% (L14)和53.5% (L15), 可以看出, ABA对L10种子萌发的抑制效应最显著, 对L14的影响最小(图12-C); GA处理均促进了OsUBA过表达水稻株系及野生型种子的萌发。在萌发的1.5 d, GA对野生型的促进效应比L10和L15稍大一些, 但对L14的促进效应稍高于野生型, 但差异都微乎其微(图12-B)。以上结果说明, 过表达OsUBA促进了种子的萌发, OsUBA过表达株系对ABA处理更敏感, GA处理对OsUBA过表达株系种子的萌发稍有促进, 但与野生型相比差异不大。3个OsUBA过表达株系中, L14的表型最弱。图12
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图12ABA和GA处理对OsUBA过表达水稻株系和野生型种子萌发的影响
A: 1/2 MS培养基上Osuba过表达的水稻株系种子萌发过程和野生型对照; B: 1/2 MS培养基添加100 μmol L-1 GA; C: 1/2 MS培养基添加3 μmol L-1 ABA。
Fig. 12ABA and GA treatment affects seed germination of OsUBA-overexpressing rice plants and the wild type control
A: The seed germination course of the OsUBA-overexpressing rice plants and the wild type control on 1/2 MS medium; B: 1/2 MS medium supplemented with 100 μmol L-1 GA; C: 1/2 MS medium supplemented with 3 μmol L-1 ABA.
3 讨论
NBR1类自噬受体通过与ATG8蛋白间的相互作用参与自噬体膜的形成和延伸[5,7], 在自噬过程中扮演重要角色。迄今为止, 植物中有关NBR1类自噬受体的研究相对较少。已有研究表明, 拟南芥、烟草等植物中的NBR1蛋白参与了植物的非生物胁迫反应以及对营养缺乏的响应[9,10]。本实验室前期有关小麦中NBR1蛋白TaUBA的研究表明, TaUBA在植物的盐和干旱胁迫反应中起负调控作用[13]。但水稻中此类蛋白的研究还未见报道。本研究表明, OsUBA基因在水稻花药中的表达较高(图7), 过量表达OsUBA的水稻株系开花时间明显提前(图11)。这些结果说明, OsUBA可能参与对水稻开花时间的调控。开花是植物由营养生长转向生殖生长的重要转折点。在许多植物物种中, 开花时间受到光(光周期)和温度的精确调控[17,18,19]。在OsUBA基因启动子区发现的G-box和Sp1元件是常见的光反应元件[20]; 此外, 该启动子区还有多个部分光反应元件, 如Box 4、GATA-motif、I-box、LAMP- element、chs-CMA1a、chs-CMA2c等, 这些元件常见于光调控基因的启动子区, 对光调控下基因的转录至关重要[21], 推测光照影响OsUBA的表达, 进而影响转基因水稻的开花时间。此外, 有研究表明, 自噬途径对花药发育中的代谢调控和营养供应非常重要。在拟南芥花粉萌发过程中, 自噬相关基因ATG6是必需的[22]; 在水稻自噬突变体Osatg7-1中, 花药发育受损[23]; OsATG7在花药中表达较高; OsATG7介导的自噬依赖的脂质代谢在花药发育中是必需的[24]。本研究中的OsUBA基因编码选择性自噬受体, 推测OsUBA也可能通过自噬途径介导的能量代谢影响开花时间。
种子萌发是一个复杂的细胞学过程, 脂质、碳水化合物和蛋白质等的代谢为种子萌发过程提供能量[25,26]; 此外, 各类激素在种子萌发中也发挥重要作用[27,28], 其中, ABA是种子萌发的负调节剂[29,30], GA能促进种子萌发和拮抗ABA的抑制作用[31]。本研究发现, OsUBA在萌发种子的胚中表达量很高(图7); 在OsUBA基因启动子区发现了ABA反应元件ABRE以及GA反应元件TATC-box、P-box和GARE-motif等; 进一步的实验也表明, ABA处理显著抑制OsUBA的表达, GA 处理后OsUBA的表达略有升高(图8); 在正常条件下, 3个过表达OsUBA水稻株系种子萌发明显比野生型快(图12-A), ABA处理显著抑制OsUBA过表达水稻株系的萌发(图12-C), GA处理对OsUBA过表达水稻株系的萌发稍有促进(图12-B), 这些结果与ABA和GA对OsUBA表达的影响是一致的, 即过量表达OsUBA促进种子的萌发; ABA处理抑制OsUBA的表达, 进而使OsUBA过表达水稻株系的种子萌发速率也显著下降; GA处理对OsUBA的表达略有增强, 相应地, GA处理后OsUBA过表达水稻株系的种子萌发略快, 但与野生型间的差异不显著。此外, 也有研究表明自噬途径参与了种子的萌发过程。AT1和AT2是拟南芥中两个与ATG8f相互作用的蛋白, AT1和AT2在种子发育的晚期表达升高; 过表达AT1促进了拟南芥种子的萌发; 而抑制AT1的表达会延迟种子萌发过程[32]; 在小麦种子萌发过程中, 胚和胚乳中代谢产物的变化差异巨大, 在胚乳中, 贮藏淀粉及小蛋白类代谢物呈现上升的趋势; 而在胚中, 蛋白质和脂质代谢产物较多, 推测胚中的能量代谢对种子的萌发更重要[33]。据文献报道, Ntjoka2的作者曾拿到拟南芥AtNBR1突变体的种子, 但他们无法获得nbr1的纯合株系, 推测AtNBR1的突变影响了种子发育[10]; 但令人不解的是, 在有关AtNBR1的研究中, 作者得到了nbr1突变体[9]。综上所述, 过量表达OsUBA促进种子萌发, 这种影响可能是OsUBA通过对ABA和GA的合成/反应实现的; 本研究中的OsUBA编码自噬受体, 因此, 也有可能是OsUBA通过自噬介导的能量代谢影响了种子的萌发, 抑或是两者兼而有之。
本研究在OsUBA基因的启动子区发现了许多与逆境胁迫有关的元件, 如与缺氧有关的GC- motif、与低温胁迫有关的LTR元件以及与ABA反应相关的ABRE元件; 此外, OsUBA基因的启动子区还有与MeJA和SA有关的反应元件。MeJA广泛参与了各种植物逆境胁迫反应及与其他激素的相互作用[34]; SA在植物的抗病反应中发挥重要作用[35], ABA与植物非生物胁迫的关系密切[36], 推测OsUBA可能也参与植物生物/非生物胁迫反应。此前文献中已有此类蛋白参与植物逆境胁迫反应的相关报道, 如拟南芥中AtNBR1的表达受热胁迫诱导, nbr1对热胁迫的敏感性增强, 盐和干旱胁迫抗性降低[9]; 小麦中的NBR1类蛋白TaUBA也参与了盐和干旱胁迫反应[13]。
在对OsUBA及其同源蛋白进行进化分析时, 发现在水稻中除了OsUBA外还有与之同源的蛋白(图3)。我们在前期研究中曾根据OsUBA的不同区域设计/构建了3个RNAi载体, 并分别得到了3个载体的纯合株系, 但遗憾的是各株系的表型与野生型相比差异均不显著(未发表数据), 估计是因为有同源蛋白的存在, 推测文中提及的水稻中的3个NBR1蛋白可能功能冗余; 因OsUBA与OsNBR1 X2序列相似性更高, 两者的功能可能也更相似。综上所述, 本文中的OsUBA除了参与对花期和种子萌发的调控外还可能参与植物的非生物/生物胁迫反应, 具有广泛的生物学功能, 这需要后期进一步的功能验证。
4 结论
本研究克隆了水稻NBR1类自噬受体蛋白基因OsUBA, 该基因的启动子区含有与各种激素和光反应等有关的顺式作用元件。OsUBA基因在各器官中均有表达, 但在花药、根以及萌发中的种子中表达量较高, ABA抑制OsUBA的表达; OsUBA基因在水稻中的过量表达使种子萌发加快、开花时间提前, ABA处理显著抑制了过表达OsUBA水稻株系种子的萌发, 推测OsUBA基因的表达和功能可能与花的发育、种子萌发以及生物/非生物胁迫反应有关。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 4]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
