 ,2,*, 李志新
,2,*, 李志新 ,1,*
,1,*Potassium uptake and genome-wide association analysis of rice under different nitrogen levels
ZOU Wei-Wei1, LU Xue-Li2, WANG Li1, XUE Da-Wei3, ZENG Da-Li ,2,*, LI Zhi-Xin
,2,*, LI Zhi-Xin ,1,*
,1,*通讯作者:
收稿日期:2018-11-22接受日期:2019-01-19网络出版日期:2019-03-11
| 基金资助: |
Received:2018-11-22Accepted:2019-01-19Online:2019-03-11
| Fund supported: |
作者简介 About authors
E-mail:1058510926@qq.com。

摘要
关键词:
Abstract
Keywords:
PDF (2296KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
邹伟伟, 路雪丽, 王丽, 薛大伟, 曾大力, 李志新. 不同氮水平下水稻钾吸收及全基因组关联分析[J]. 作物学报, 2019, 45(8): 1189-1199. doi:10.3724/SP.J.1006.2019.82058
ZOU Wei-Wei, LU Xue-Li, WANG Li, XUE Da-Wei, ZENG Da-Li, LI Zhi-Xin.
水稻的正常生长离不开氮、钾等营养元素, 氮、钾的养分平衡及交互作用对水稻的生长发育和产量形成起着重要的调控效应[1]。虽然目前关于施肥对水稻产量及肥料利用率影响的研究很多, 但是大部分以研究单一肥料的效应为主[2,3]。有研究表明, 增施钾肥可以提高大麦的氮肥农学利用率和生理利用率, 氮、钾肥平衡施用能显著提高谷子的产量和肥效, 但却并未对肥料间的交互作用进行详细分析[4]。我国约有三分之一的可耕作土壤中钾含量不足[5]。土壤中钾肥的缺乏使得作物产量和品质受到严重的影响[7]。因而了解氮钾之间的协同吸收, 选育出钾高效吸收和利用的水稻品种, 对提高水稻品质减少钾肥施用量具有重要意义。
研究显示, 氮主要从3个方面影响钾吸收及利用。首先, 在田间土壤条件下, NH+通过土壤对NH+、K+之间吸附固定存在的竞争来影响水稻对钾的吸收; 其次, NH4+通过对水稻根系钾高亲和转运系统和低亲和转运系统的影响来抑制水稻根系对钾离子的吸收; 第三, 由于NO3-与K+具有相反的电荷, NO3-通过离子补偿来促进钾离子的吸收; 另外植物体内的钾与许多营养元素存在互作关系, 钾通过影响作物的氮代谢和碳代谢, 来加快植物体内氮化合物向蛋白质合成场所运输, 以及氨基酸合成蛋白质和稳定蛋白质的结构, 有利于植物体内有机质的转化及累积, 提高氮和磷养分的利用率[8,9,10,11,12,13,14]。
目前认为植物体内存在两种不同类型的钾吸收系统, 即高亲和与低亲和两种机制。在低钾浓度小于0.2 mmol L-1条件下, 由钾载体蛋白(carrier protein)组成的高亲和转运系统起作用; 在高浓度(1~10 mmol L-1)条件下由通道蛋白(channel protein)组成的低亲和吸收系统(1ow-affinity K channe1)起作用[15,16,17]。许多研究发现, 不同基因型水稻之间的钾吸收利用效率显著不同, 说明植物钾营养性状是受遗传控制的。为获取水稻的全部HAK (High Affinity K transporter, 高亲和钾离子转运蛋白)基因, Amrutha等[18]以拟南芥HAK转运蛋白作为检索序列, 利用Blastp搜索TIGR水稻基因组注释数据库, 水稻基因组计划(Rice Genome Project, RGP)数据库和NCBI数据库, 在水稻基因组中鉴定出27个HAK基因。Li等[19]发现OsCIPK23的RNAi转基因株系表现出钾离子缺失性状, 与osakt1突变体在低钾离子浓度环境下表型相同。Yu等[20]通过T-DNA插入WOX11基因的第2外显子得到突变体wox11; Chen等[21]和Zhao等[22]对WOX11基因研究发现, WOX11基因是不定根生长发育关键调节因子, 增强植株根部生长和发育, 增加低钾土壤条件下植株的钾吸收效率。另外研究者利用各种群体定位了许多与钾素相关QTL, 这些QTL在水稻的12条染色体上都有分布[23,24,25,26]。如Wu等[23]用籼稻IR64与粳稻Azucena杂交的水稻HD群体(共123个株系)为试验材料, 共检测到20个与低钾胁迫相关性状的QTL, 主要位于第3、第5、第8染色体; Shimizu等[25]共检测到20个与低钾胁迫相关性状的QTL, 主要位于第1、第2、第4、第6、第8、第9、第10、第12染色体。近年来, 全基因组关联分析(genome-wide association study, GWAS)成功地应用在一些模式生物研究和农作物的育种当中[27,28,29,30]。
在水稻中, 利用GWAS分析了包括产量、品质、抽穗期、株型等多种重要的农艺性状, 同时也对病虫害抗性等生物胁迫和盐碱耐受性等非生物胁迫性状进行了全基因组关联分析, 并鉴定了许多重要的SNP位点[28,31-33]。但是, 与钾吸收及氮响应相关的GWAS研究尚未见报道。本研究选取134份来自不同地区的水稻品种作为关联分析的群体, 利用在不同氮肥条件下钾吸收的变化, 从氮、钾吸收互作角度来解释水稻的钾吸收及品种间钾利用差异; 通过全基因组关联分析筛选钾吸收和利用的基因, 为进一步的功能研究和育种应用奠定基础。
1 材料与方法
1.1 供试材料
134个地方品种来自中国水稻研究所(CNRRI)、国际水稻研究所(IRRI)种质资源库及多个国家和地区。包括各省农业科学院的地方品种41份, 其他国家36份, 国内大面积推广种植品种57份。其中籼稻71份, 即宝大粒、七桂B、窄叶青8号、潮安柑园、七语冷禾、浙733、澄海白壳矮、齐眉1号、浙场3号、川大粒、青种、浙辐、大粒红、秋布了、制4、二九青、仁化九工几、中白花、赣早籼37、三朝齐、中156、广陆矮4号、胜利籼、舟903、桂朝2号、蜀恢527、湖南早、苏御糯、花占、泰国香稻、黄黏5号、天丰、火烧禾、万利籼、加拿大1号、湘矮早9号、嘉育948、湘早籼、粳籼89、湘早籼3号、垦稻8号、湘早籼7号、况丝佳帝、小白芒、柳条子、小毛香、陆财号、扬辐籼5号、罗大穗、扬尖糯、罗长大粒、一红嘴、密阳46、英德大叶拍、埝口黄、原丰早、488、Albolia-P、Arborio、C8CU80-S、C91 C-S、CEYSVONI-S、DR32、ECIA 66、Ichsannde、IFON、IR36、MEDIO 18、PANAMA1537、RD25、SELECCION; 粳稻63份, 即阿尔巴尼亚、农虎6号、矮子黄壳糯、秋光、奥羽326、热研、北陆129、软乌稻、边别、三粒寸、楚粳26、沈长粳、粗粳23、台中65、大粒瓜糯、太湖青、稻花香2号、特大粘、东农416、晚百歌、鄂晚5号、武育粳3号、鄂宜105、武运粳7号、蜂子糯、祥湖84、凤尾6号、新长粳、高粱稻、新竹4号、哥罗27、秀水8号、合江23、秀水9号、黑猪尿、秀水63、黄丝糯2号、徐稻3号、空育131、粤T2567、兰稻、早丰9号、老龙须、早熟刚糯2号、木邦谷、长白9号、南洋占、中242、Acc.14725、Baldo、Chabli-c-G、Barah、Gamilunan、IRAT104、Taihuqing IRAT243、Tedazhan ITA233、Hrborio-c、Kasinkan、Kenteng、Kutube-n、Labelle、Radi-P和SLG-1 [34]。1.2 试验设计
试验于2015年在中国水稻研究所实验基地(浙江富阳)完成, 试验田土壤为水稻土(土壤含有机质36.9 g kg-1、全氮2.73 g kg-1、速效钾104 mg kg-1、速效磷52 mg kg-1、碱解氮156 mg kg-1, pH 6.24)。设不施氮(N0), 施96 kg hm-2纯氮(N1)和施192 kg hm-2纯氮(N2) 3种氮水平; 以施氮量为主区, 品种为裂区, 3次重复。每个品种种植4行, 每行种6株, 株行距为20 cm × 20 cm。采用尼龙塑料薄膜覆盖田埂, 防止漏水漏肥, 保证单独排灌。以尿素为氮肥, 按基肥∶分蘖肥 = 5∶3分2次施入, 其中播种前1 d施基肥、播种后25 d施分蘖肥。分蘖肥施用期间(6月中旬至7月中旬)保持畦面湿润并无水层, 且仅保证沟中有水以防止水分串流导致的串肥。磷、钾施用量为P2O5 75 kg hm-2、K2O 150 kg hm-2, 其中磷肥为底肥一次施入, 钙、镁、钾肥按基、蘖肥各50%分2次施入。其他栽培管理措施同一般大田。于水稻移栽后32 d (分蘖盛期)地上部钾含量、干重和钾积累调查。1.3 植株样品采集及分析
分蘖盛期取每品种中间的6株无边际效应植株的地上部位, 装于纸袋中105℃下杀青30 min, 80℃烘干至恒重后称重, 即得出地上部位干重量, 由M (g plant-1)表示。将地上部位经磨碎过0.5 mm筛后, 采用H2SO4-H2O2消煮, 火焰光度计法测定植物全钾含量, 由KC (mg g-1)表示。地上部钾素积累由KA (mg plant-1)表示, 即干重M与钾含量KC的乘积。钾含量在N0处理下的相对变化用KCN1-N0表示,即N1与N0水平下的钾含量差值, 钾含量在N2处理下的相对变化用KCN2-N1表示, 即N2与N1水平下的钾含量差值; 类似地, 钾积累在N0和N2处理下的相对变化则分别用KAN1-N0和KAN2-N1表示。1.4 数据分析
1.4.1 性状相关性分析 使用Microsoft Excel 2010整理和分析数据, 并计算其平均值和标准差; 使用DPS9.50进行相关性分析和显著水平检验。1.4.2 基因型测定 采用CTAB法提取134份品种的DNA[35], 样品检测合格后高通量测序。每品种测序的reads数据量保证在5 Gb以上。之前的研究中, 已利用BWA和GATK软件对SNP提取, 使用最小等位基因频率大于0.03 (MAF>0.03)过滤SNP, 并使用ANNOVAR 软件注释SNP数据[34]。
1.4.3 群体结构与亲缘关系分析 之前的研究中, 已利用提取到的3,356,591个SNP位点开展亲缘关系和系统进化分析[34]。
1.4.4 全基因组关联分析 采用混合线性模型(mixed linear model, MLM)的EMMAX (Efficient Mixed-Model Association eXpedited)软件包对SNP标记与目标性状进行关联分析, 将EMMAX软件计算的亲缘关系K矩阵及基于EIGENSOFT软件计算的群体结构Q值纳入分析模型中, 设定关联分析参数为emmax -v, -t, -k, -p, -d 10, -c, -o。关于关联分析的显著性, 采取P<10-6作为宽松阈值, 进一步的严格阈值采取Bonferroni校验阈值[34,35,36]。
1.5 RNA的提取和RT-PCR
在3个氮水平下, 分别取分蘖盛期TN1 (台中本地1号)中间的6株无边际效应植株的全展叶。用Total RNA Kit (AXYGEN)试剂盒提取植株总RNA, 并且反转录成cDNA。采用SYBR Green PCR Master Mix (Thermo Fisher Scientific)试剂盒配成每孔10 μL体系, 在ABI 7900进行RT-PCR, 水稻泛素5基因作为内参。引物序列见表1。Table 1
表1
表1RT-PCR引物
Table 1
| 候选基因 Candidate gene | 上游引物序列 Forward primer sequences (5°-3°) | 下游引物序列 Reverse primer sequences (5'-3') |
|---|---|---|
| LOC_Os10g05730 | GGCATCAAAACAGGCTCACA | GGCCAACTTTACAGGTACACA |
| LOC_Os10g05600 | GCTGTTCTTGGTCCACGC | GGCCTTACTGAGTCTCTCCC |
| LOC_Os10g05540 | CTCGGCTTCTTCCACAACTC | CCTGGATCTGGCTGGAGAC |
| LOC_Og05620 | CACGAGCACCCACAATGTAG | AACGGTCACCCCTTCATCTT |
新窗口打开|下载CSV
2 结果与分析
2.1 3种氮水平下钾含量、干重和钾积累的分布
钾含量在3个氮水平上的变幅分别为22.2~35.1 mg g-1、20.0~37.0 mg g-1和19.2~38.9 mg g-1(表2), 表明该群体的钾含量、干重和钾积累变异非常丰富。Table 2
表2
表23个氮水平下钾含量、干重和钾积累变化
Table 2
| 性状 Trait | 参数 Parameter | 氮水平 N level | ||
|---|---|---|---|---|
| N0 | N1 | N2 | ||
| K含量K content (mg g-1) | 均值Mean | 28.87±2.85 | 28.05±3.35 | 28.05±3.20 |
| 变幅Range | 22.21-35.12 | 20.02-37.03 | 19.23-38.92 | |
| 干重Dry weight (g plant-1) | 均值Mean | 4.68±1.29 | 6.30±1.69 | 7.70±2.02 |
| 变幅Range | 1.94-8.12 | 2.66-10.30 | 3.68-14.40 | |
| 钾积累K accumulation (mg plant-1) | 均值Mean | 26.47±7.07 | 34.91±8.76 | 42.51±10.89 |
| 变幅Range | 11.41-48.26 | 15.58-53.72 | 21.71-72.93 | |
新窗口打开|下载CSV
在不同氮水平下, 钾含量、植株干重和钾积累在134份品种组成的自然群体中呈正态分布。其中, 钾含量在N0水平下主要集中在27~33 mg g-1, 而N1、N2水平下主要集中在25~31 mg g-1 (图1-a); 钾积累在N0、N1、N2水平上分别集中分布在16.5~ 33.0、27.5~44.0和33.0~49.5 mg plant-1 (图1-e)。在反应氮响应变化中, 只在N0水平下对钾含量的影响比较大, 表现为钾含量增加(图1-b); 并且从图1-b、d和f可以看出, 钾含量整体上受氮水平的影响不明显, 而干重和钾积累明显随着氮水平的增加而增加, 说明高氮条件下引起的生物量增加是导致钾积累增加的关键。
图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图13 种氮水平下水稻钾含量、干重和钾积累的分布
a、c和e分别为N0、N1和N2条件下的钾含量、干重和钾积累分布; b、d、f 为N0、N1和N2条件下钾含量、干重和钾积累分布的气泡图, 气泡宽度代表株系数, 红色虚线为3 种氮条件下各性状的平均值。
Fig. 1Distribution of K content, dry weight, and K accumulation under three nitrogen levels
a, c, and e are the distribution of K content, dry weight, and K accumulation under three nitrogen levels, respectively; b, d, and f are the distribution of K content, dry weight, and K accumulation in bubble chart under three nitrogen levels, respectively; the width of bubble represents the number of lines, and the red dotted line is the average value of every character.
2.2 钾含量和钾积累在不同氮处理下的相对变化
在不同的氮处理下, 将钾含量和钾积累的相对变化分为4种响应模式。在钾含量相对变化中, 14.1%品种表现为在N0条件下钾含量相对降低和在N2条件下相对增加, 35.4%品种表现为在N0和N2条件下钾含量都相对增加, 27.4%品种表现为在N0条件下钾含量相对增加而在N2条件下相对降低, 23%品种表现为在N0和N2条件下钾含量都相对降低(图2-a); 在钾积累相对变化中, 78.8%品种表现为在N0条件下钾积累相对降低和在N2条件下相对增加, 11.5%品种表现为在N0和N2条件下钾积累都相对增加, 0.9%品种表现为在N0条件下钾积累相对增加而在N2条件下相对降低, 8.9%品种表现为在N0和N2条件下钾积累都相对降低(图 2-b)。由此可知,该关联群体在不同氮处理下钾含量和钾积累相对变化均呈现丰富的变异, 预示着钾响应氮变化的相关基因位点在不同品种中存在丰富的变异。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2不同氮处理下钾含量和钾积累相对变化的分布
a和b是分别相对N0N1-N0和N2N2-N1钾含量和钾积累相对变化分布。KC: 钾含量; KA: 钾积累。
Fig. 2Distributions of relative changes of K content and K accumulation under different nitrogen levels
a and b are the distributions of relative changes of K content and K accumulation under N0N1-N0 and N2N2-N1; KC: K content; KA: K accumulation.
2.3 钾含量、植株干重和钾积累的籼粳差异比较
由图3所示, 在不同氮水平下, 参试材料的钾含量、干重和钾积累的变异范围和平均值在籼粳亚种间存在较大差异。3个氮水平中籼稻的钾含量均极显著低于粳稻, 例如在N0水平下籼和粳稻中钾含量分别为27.82 mg g-1和30.26 mg g-1, 表明不同的种质资源对钾的吸收能力存在差异; 在3个氮水平中, 籼稻的干重显著高于粳稻的干重, 籼稻的钾积累也显著高于粳稻, 说明在3个氮水平下, 籼稻的生物量增加是导致钾积累增加的主要原因。其中籼粳的干重和钾积累随着施氮用量的增加, 其指标也随之增加, 但是籼粳的钾含量并没有随着施氮水平的变化而出现明显的变化。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3钾含量、干重和钾积累在3种氮水平处理下籼粳亚种间的比较
a、b、c是钾含量、干重和钾积累在3种氮水平处理下籼粳亚种间的比较。C0i、C1i、C2i分别表示籼稻在N0、N1、N2下的钾含量, C0j、C1j、C2j分别表示梗稻在N0、N1、N2下的钾含量; W0i、W1i、W2i分别表示籼稻在N0、N1、N2下的植株干重, W0j、W1j、W2j分别表示粳稻在N0、N1、N2下的植株干重; A0i、A1i、A2i分别表示籼稻在N0、N1、N2下的钾积累, A0j、A1j、A2j分别表示粳稻在N0、N1、N2下的钾积累。
Fig. 3Comparisons of K content, dry weight and K accumulation between indica and japonica under three nitrogen levels
a, b, and c are comparisons of K content, dry weight and K accumulation between indica and japonica under three nitrogen levels. C0i, C1i, and C2i are the K content of indica under N0, N1, and N2 nitrogen levels respectively; C0j, C1j, and C2j are the K content of japonica under N0, N1, and N2 nitrogen levels respectively; W0i, W1i, and W2i are the dry weight of indica under N0, N1, and N2 nitrogen levels respectively; W0j, W1j, and W2j are the dry weight of japonica under N0, N1, and N2 nitrogen levels respectively; A0i, A1i, and A2i are the K accumulation of indica under N0, N1, and N2 nitrogen levels respectively; A0j, A1j, and A2j are the K accumulation of japonica under N0, N1, and N2 nitrogen levels, respectively.
2.4 钾含量、植株干重和钾积累的相关性分析
钾含量随着施氮量的增加变化不明显, 植株干重和钾积累都随着施氮量的增加而增加(表3)。钾含量与干重在3个氮水平上均呈现出极显著的负相关, 相关系数分别为-0.33、-0.38和-0.30, 说明水稻植株干重越大, 钾含量就越低; 钾含量与钾积累在3个氮水平上均无显著相关性, 而干重与钾积累在3个氮水平中均呈极显著正相关, 相关系数分别达0.92、0.88和0.89, 表明水稻钾积累主要受水稻干重影响, 即水稻干重越大, 水稻钾积累水平就越高(表4)。这说明施用高氮引起的生物量增加是导致钾积累增加的关键。Table 3
表3
表3参试水稻材料钾含量、干重和钾积累与氮水平的相关系数
Table 3
| 氮水平 N level | 钾含量 K content | 干重 Dry weight | 钾积累 K accumulation | |||
|---|---|---|---|---|---|---|
| N0 | N1 | N0 | N1 | N0 | N1 | |
| N1 | -0.11 | 0.48** | 0.46** | |||
| N2 | -0.14 | -0.02 | 0.67** | 0.35** | 0.66** | 0.37** |
新窗口打开|下载CSV
Table 4
表4
表43个氮水平下参试水稻材料3个目标性状间的相关系数
Table 4
| 性状 Trait | N0 | N1 | N2 | |||||
|---|---|---|---|---|---|---|---|---|
| K含量 K content | 干重 Dry weight | K含量 K content | 干重 Dry weight | K含量 K content | 干重 Dry weight | |||
| 干重Dry weight | -0.33** | -0.38** | -0.30** | |||||
| K积累 K accumulation | 0.06 | 0.92** | 0.09 | 0.88** | 0.16 | 0.89** | ||
新窗口打开|下载CSV
2.5 不同氮水平钾含量、植株干重和钾积累的全基因组关联分析
对134份水稻钾含量、钾积累和干重, 在3个氮处理条件下, 以-lg P大于6为阈值, 共检测到12个与目标性状显著相关的SNP位点, 包括钾含量和干重的5个显著位点, 钾积累的显著位点2个; 在N0和N2水平, 共检测到3个与目标性状相关的SNP位点, 但在N1水平下, 检测到的与目标性状相关的显著位点最多, 为9个; 在2个氮水平上均检测到的与钾积累、钾含量和干重显著关联的位点一共有3个, 说明此类与目标相关的3个位点受施氮量影响较小(表5和图4-a~c)。Table 5
表5
表53种氮处理下钾含量、干重和钾积累的显著性关联位点
Table 5
| 性状 Trait | 氮水平 Nitrogen level | 染色体 Chr. | 物理距离 Position (bp) | 主要等位基因 Major allele | 次要等位基因 Minor allele | 最小等位基因频率 Minor allele frequency | P-value (-lg P) |
|---|---|---|---|---|---|---|---|
| 钾积累 | N0 | 6 | 21,362,789 | G | A | 0.087 | 6.803 |
| K accumulation | N1 | 6 | 21,336,732 | A | G | 0.387 | 6.130 |
| 钾含量 | N1 | 1 | 2,908,421 | A | G | 0.302 | 6.313 |
| K content | N1 | 2 | 7,907,735 | A | C | 0.477 | 6.900 |
| N1 | 3 | 5,568,543 | A | G | 0.317 | 6.791 | |
| N1 | 6 | 1,524,776 | G | A | 0.228 | 7.643 | |
| N1 | 6 | 26,011,593 | C | T | 0.077 | 6.400 | |
| 植株干重 | N0 | 6 | 21,337,192 | T | C | 0.380 | 6.265 |
| Plant weight | N1 | 6 | 21,337,192 | T | C | 0.378 | 7.303 |
| N1 | 6 | 23,166,060 | C | T | 0.049 | 6.267 | |
| N1 | 10 | 19,108,030 | C | A | 0.105 | 6.248 | |
| N2 | 10 | 19,108,030 | C | A | 0.105 | 6.282 |
新窗口打开|下载CSV
图4
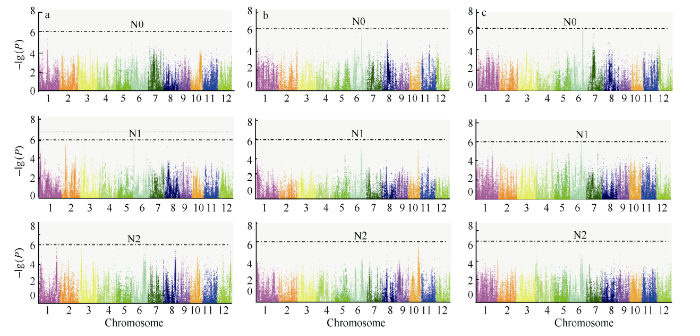 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图43个氮水平下水稻钾含量、干重和钾积累的全基因组关联分析
a、b和c分别是3个氮水平下水稻钾含量、干重和钾积累的全基因组关联分析。
Fig. 4Genome-wide association studies of K content, dry weight, and K accumulation under three nitrogen levels
a, b, and c are genome-wide association studies of K content, dry weight, and K accumulation under three nitrogen levels, respectively.
基因的多效性是生物学上的普遍现象。本研究发现第6染色体上的Chr6_21,337,192位点在干重和钾积累两个性状中均被检测到, 表明该位点既控制干重也影响钾积累, 从遗传上也进一步说明了钾积累与干重紧密相联。此外, 第6染色体上检测到的显著位点最多, 一共有7个, 均位于21.3~26.0 Mb区间(表5)。
另外检测到在N1水平上干重的1个显著关联位点MAF (Minor Allele Frequency)为0.049, 小于0.05, 表明此位点为1个稀有的突变位点; 而其他的显著位点MAF均大于0.05, 表明此类SNP在关联群体中分布较为广泛(表5)。
进一步对不同的氮处理下钾含量和钾积累相对变化进行全基因组关联分析, 共检测到了4个钾含量相对变化的显著关联位点, 而没有检测到钾积累的相对变化; 其中有3个位点处于第10染色体上, 而且在相对N0和N2处理下均检测到位于第10染色体同一位点Chr10_2,822,026, 表明此位点相对于N0和N2都表现出了响应, 是后续进行候选基因分析的重要依据(表6和图5-a, b)。
Table 6
表6
表6钾含量在N0和N2处理下的相对变化的显著关联位点
Table 6
| 性状 Trait | 染色体 Chr. | 物理距离 Position (bp) | 主要等位基因 Major allele | 次要等位基因 Minor allele | 最小等位基因频率 Minor allele frequency | P-value (-lg P) |
|---|---|---|---|---|---|---|
| KCN1-N0 | 9 | 6,934,774 | T | A | 0.149 | 6.672 |
| KCN1-N0 | 10 | 2,822,026 | C | T | 0.066 | 6.296 |
| KCN1-N0 | 10 | 4,669,497 | G | A | 0.030 | 6.201 |
| KCN2-N1 | 10 | 2,822,026 | C | T | 0.066 | 6.938 |
新窗口打开|下载CSV
图5
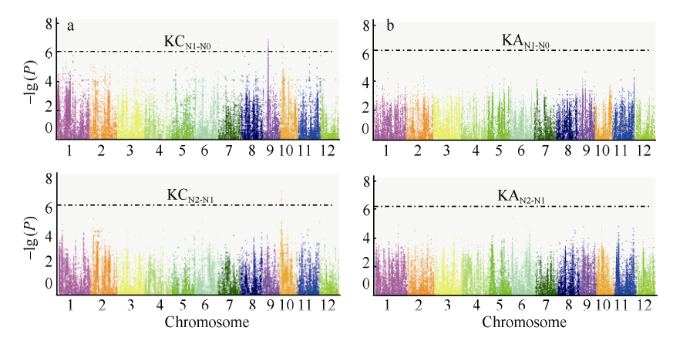 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5钾含量和钾积累在N0和N2相对变化的全基因组关联分析
a和b分别为钾含量和钾积累在N0和N2相对变化的全基因组关联分析。KC: 钾含量; KA: 钾积累。
Fig. 5Genome-wide association analysis of K content and K accumulation relative changes under N0 and N2 levels
a and b are Genome-wide association analysis of K content and K accumulation relative changes under N0 and N2 levels. KC: K content; KA: K accumulation.
2.6 候选基因分析
在N1水平下, 第6染色体上显著性SNP (chr6_ 1,524,776)位点的P值(-lg P)为7.64, 属于最高P值; 对此位点进行候选基因分析, 在该位点±200 kb的候选区间内, 找到LOC_Os06g03610, 即为RUPO基因, RUPO与OsHAK1/19/20 3个钾离子转运蛋白互作, 与OsHAK1互作最强, 调控K+稳态[37]。另外, 在N0和N2处理下钾含量的相对变化, 均检测到了位点Chr10_2,822,026 (表6); 对该位点上下游200 kb区间在水稻基因组注释计划数据库进行候选基因搜索, 发现共有62个候选基因。通过生物信息学分析, 确定18个可能的候选基因。通过对该位点不同品种的钾含量分析, 选取1个在N0、N1、N2钾含量差异较明显的TN1品种, 进行分蘖期的定量表达分析, 对候选基因进行验证。与N1相比, 大部分候选基因在N0和N2下的表达量显著降低(图6)。其中LOC_Os10g05730在N0的表达量是N1的0.413倍, 而N2是N1的0.0626倍; LOC_Os10g05540在N0和N2的表达量分别是N1的0.209倍和0.214倍; LOC_Os10g05620在N0的表达量是N1的0.154倍, 在N2下降为0.00451倍; LOC_Os10g05600在N0和N2的表达量分别是N1的0.146倍和0.112倍(图5); 通过对候选基因在N0和N2相对N1的表达分析, 进一步缩小钾含量响应基因的范围, 为后续的基因克隆及功能研究奠定了基础。图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6在3个氮水平下4个候选基因相对表达水平
在3个氮水平中, 雷达图以N1水平基因的表达量为1。
Fig. 6Four candidate genes relative expression levels under three nitrogen levels
Radar chart is based on expression level of 1 under N1 level among three nitrogen levels.
3 讨论
氮和钾都是水稻生长和发育的大量必须营养元素, 两者之间存在着一定的互作效应。但目前大部分以研究单一肥料的效应为主, 有关氮肥对钾吸收的影响鲜有报道[1]。本研究对134份水稻品种在不同氮水平下的钾含量、植株干重和钾积累分析表明, 水稻植株钾积累随着施氮水平的提高和植株生物量的增加而增加, 而钾含量则没有明显的变化。梁健等[7]的研究也发现随着氮肥群体生产力等级的提高, 钾总积累量显著增多; 在N1水平下, 位于第6染色体上与钾积累显著相关的Chr6_21,336,732位点和植株干重显著相关的Chr6_21,337,192位点相邻, 而在此位点附近并未检测到与钾含量显著相关的SNP位点, 这也解释了钾积累与植株干重之间存在相关的内在原因。刘国栋等[38]用水培方法对86种不同基因型籼稻筛选比较发现, 不同基因型间吸钾效率差异十分显著; 李华等[39]对在不同氮源和钾水平下杂交稻及其父母本和常规稻的钾吸收和积累的研究认为不同类型水稻对钾素的吸收可能存在差异。本研究发现在不同的氮水平下粳稻品种的钾含量均普遍高于籼稻品种; 另外, 在籼型和粳型品种中均存在钾积累和钾吸收显著差异的种质资源, 这种不同水稻基因型在吸钾能力和钾素利用效率上的差异将为培育钾高效利用率水稻提供有益的种质资源。不同的研究者采用不同的群体在不同的环境下定位了一些与水稻钾吸收性状相关的QTL, 目前在水稻12条染色体上均检测到了与钾素吸收相关的QTL[23,24,25,26]; Shimizu等[25]通过低钾胁迫共检测到分布于8条染色体上20个与钾吸收性状相关的QTL; Pandit等[26]利用重组自交系在第1、第8和第12染色体都检测到与钾吸收相关的QTL。本研究在第1、第2、第3、第6、第9和第10染色体均找到了与钾吸收相关的显著位点, 并且这些位点在第6染色体上分布最多; 其中一些已克隆的与钾素相关基因位于本研究关联到的显著性位点区间内, 如在N1下, RUPO基因与OsHAK1/19/20 3个钾离子转运蛋白互作调控K+稳态, 位于显著位点Chr6_1,524,776区间内[37]。同时, 钾素营养与水稻的根系生长也具有密切的关系, 显著位点Chr2_7,907,735附近的OsRR1基因参与水稻不定根的发育[40]; 在N0处理下钾含量相对变化中, 在Chr9_6,934,774位点附近检测到1个与最大根长度相关的QTL[41]; 该结果进一步验证了全基因组关联分析对遗传变异检测的有效性和可靠性。
本研究在2个氮水平下都检测到的显著性位点有3个, 并且钾含量在N0和N2处理下的相对变化中均检测到了同一位点Chr10_2,822,026, 表明这些位点附近的候选基因能在不同氮水平下稳定表达, 附近可能存在效应较大的候选基因, 利用这些稳定表达的位点进行聚合育种, 提高群体内优势等位基因频率, 可能取得较好的育种效果。另外本文找到了4个在不同氮水平下具有较大表达量差异的候选基因, 对后期候选基因鉴定、分析、克隆、表达水平变化、基因敲除及功能验证十分重要, 将有助于揭示水稻钾素吸收的遗传机制。
4 结论
本研究群体在3种氮水平下表型丰富。施氮量对钾含量影响不明显, 而施氮量的增加引起植株干重的增加导致了钾积累的增加; 另外, 在相同氮水平下籼稻的钾含量显著低于粳稻, 而籼稻的干重和钾积累极显著高于粳稻。GWAS分析检测到16个显著关联位点, 其中2、5和5个分别与钾积累、钾含量和植株干重显著关联, 另外4个是钾含量相对变化的关联位点; 并且在两种氮响应下均检测到的同一显著关联位点附近找到了4个表达量差异较大的候选基因。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.17221/PSEURL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/j.jplph.2008.12.009URL [本文引用: 1]
[本文引用: 1]
DOI:10.1146/annurev.arplant.54.031902.134831URL [本文引用: 1]
DOI:10.1104/pp.122.3.823URL [本文引用: 1]
DOI:10.2134/agronj1969.00021962006100010009xURL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.2135/cropsci1998.0011183X003800060009xURL [本文引用: 3]
[本文引用: 2]
[本文引用: 4]
[本文引用: 3]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 4]
DOI:10.1093/nar/8.19.4321URL [本文引用: 2]
DOI:10.1002/(ISSN)1098-2272URL [本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
