 ,1,*, 刘风珍
,1,*, 刘风珍 ,2,*
,2,*Transcriptome analysis of the peanut transgenic offspring with depressing AhPEPC1 gene
PAN Li-Juan1, CHEN Na1, CHEN Ming-Na1, WANG Tong1, WANG Mian1, CHEN Jing1, YANG Zhen1, WAN Yong-Shan2, YU Shan-Lin1, CHI Xiao-Yuan ,1,*, LIU Feng-Zhen
,1,*, LIU Feng-Zhen ,2,*
,2,*通讯作者:
收稿日期:2018-09-20接受日期:2019-01-19网络出版日期:2019-03-22
| 基金资助: |
Received:2018-09-20Accepted:2019-01-19Online:2019-03-22
| Fund supported: |
作者简介 About authors
E-mail:panlijuan_2008@aliyun.com。

摘要
关键词:
Abstract
Keywords:
PDF (1938KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
潘丽娟, 陈娜, 陈明娜, 王通, 王冕, 陈静, 杨珍, 万勇善, 禹山林, 迟晓元, 刘风珍. 花生AhPEPC1基因抑制表达的转基因后代转录组分析[J]. 作物学报, 2019, 45(7): 993-1001. doi:10.3724/SP.J.1006.2019.84122
PAN Li-Juan, CHEN Na, CHEN Ming-Na, WANG Tong, WANG Mian, CHEN Jing, YANG Zhen, WAN Yong-Shan, YU Shan-Lin, CHI Xiao-Yuan, LIU Feng-Zhen.
目前我国植物油供需矛盾突出, 自给率不足35%。提高油料作物的含油量是增加油脂产量的有效途径。花生是我国最重要的油料作物之一, 与大豆、油菜等主要油料作物相比, 花生单位面积产油量最高[1]。花生种子含油量为50%左右[2], 具有进一步提高的潜力。
近年来, 随着花生基因组测序工作的开展、花生转基因技术的逐步完善, 通过转基因、分子育种手段培育高油花生品种成为可能[2,3]。油脂合成和蛋白质合成存在一定的竞争性, 2个代谢过程均需要利用磷酸烯醇式丙酮酸(phosphoenolpyruvate, PEP)做底物。乙酰辅酶A羧化酶(ACCase)催化丙酮酸合成乙酰-CoA, 进入脂肪酸合成途径, 而磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase, PEPC)可催化丙酮酸合成草酰乙酸, 进入蛋白质合成途径[4,5]。研究者发现, 油菜、大豆籽仁油脂和蛋白质含量与磷酸烯醇式丙酮酸羧化酶(PEPC)活性密切相关[6,7]。本实验室前期构建了AhPEPC1基因片段反义表达载体, 通过农杆菌介导法转入花生, 经遗传转化、温室种植、分子水平检测筛选等, 获得3个稳定遗传的转基因株系[8]。本研究在此基础上, 分析了转基因花生含油量、脂肪酸组分和蛋白质含量的变化。
转录组测序技术不仅可以用于已知参考基因组的物种测序, 也可用于无参考基因组的物种测序, 进行基因表达水平、新转录本预测、可变剪切以及SNP和InDel等诸多方面的分析, 具有结果准确性高、敏感度强、重复性好等优点。转录组测序可以快速掌握目标植物性状中起重要作用的基因, 促进育种或者相关农业应用研究的进程[9,10]。本研究进一步利用转录组测序(RNA-Seq)技术, 通过转录组分析来研究转基因花生中基因表达的变化, 在一定程度上解析了AhPEPC1基因调控花生种子含油量的分子机制。
1 材料与方法
1.1 供试材料
花生(Arachis hypogaea L.)品种花育26号由山东省花生研究所提供。本试验分析的转基因株系编号为L6、L11、L21, 非转基因对照编号为WT。1.2 稳定遗传转基因株系的获得
实验室前期以花育26号花生种子中胚轴作为外植体, 采用农杆菌介导法将AhPEPC1基因片段反义转入受体花生。转基因T0代植株经3代温室种植, 田间取样PCR检测、DNA序列测定以及转录水平分析等筛选过程, 最终获得稳定遗传的转基因株系3个, 用于后续试验。1.3 转基因花生含油量、脂肪酸组分和蛋白质含量的测定
1.3.1 花生种子含油量测定 采用索氏抽提法测定花生种子含油量, 将预先烘好的滤纸包称重(M1), 种子在70℃烘烤后磨样, 准确称量0.5~1.0 g转入滤纸包中, 于105℃烘烤3 h, 称重(M2), 然后置石油醚中浸泡过夜, 于索氏抽提器中50℃回流8 h, 待油脂完全浸出后, 干燥残渣, 105℃烘烤3 h并称重(M3)。种子含油量 = (M2-M3)/(M2-M1) × 100%。每个样品测定3次, 求其平均值。1.3.2 脂肪酸组分测量 用Agilent 7890A型气相色谱仪进行相对含量测定, FID检测器, FFAP弹性石英毛细管柱(50 m × 0.2 mm × 0.33 μm), 恒压模式, 载气为高纯N2, 流速1 mL min-1; H2流速10 mL min-1, 空气流速350 mL min-1。进样量1 μL, 分流比10:1, 进样口温度250℃, 检测器温度250℃。程序升温自210℃, 9 min; 以9℃ min-1升至230℃, 保持8 min。
1.3.3 花生种子蛋白含量测定 取0.5 g左右的花生种子磨碎样品, 采用凯氏定氮仪(Kjeltec TM2300, FOSS, Danmark)测定蛋白含量。
1.4 转录组测序
取T3代转基因株系L21与对照花生发育中期的种子(荚果形成40 d), 各3个生物学重复(测序代号分别为TL1、TL2、TL3、WT1、WT2、WT3), 委托北京诺禾致源生物信息科技有限公司利用Illumina HiSeq 4000测序。高通量测序得到的原始图像数据文件经CASAVA碱基识别(Base Calling)分析转化为原始测序序列(Raw Data或Raw Reads)。1.5 转录组数据分析
将测序过滤后得到的 clean data 数据与花生(A. duranensis)参考基因组序列(Gene Ontology (简称 GO,
KEGG (Kyoto Encyclopedia of Genes and Genomes)是系统分析基因功能、基因组信息数据库, 它有助于研究者把基因及表达信息作为一个整体网络进行研究。通过Pathway显著性富集能确定差异表达基因参与的最主要生化代谢途径和信号转导途径。
1.6 实时荧光定量PCR分析
选择15个差异表达基因, 用Primer Premier 5.0软件设计引物(表1), 以Actin为内参基因, 进行实时荧光定量PCR分析。每个样品进行3次生物学重复, 用2-ΔΔCT 法对样本基因进行表达差异相对定量分析。Table 1
表1
表1荧光定量PCR的引物序列
Table 1
| 引物名称 Primer name | 引物序列 Primer sequence (5°-3°) | 引物名称 Primer name | 引物序列 Primer sequence (5°-3°) |
|---|---|---|---|
| Aradu.2N2A3-F | TTCATTCATCCCCCTTTTCAG | Aradu.CZ64T-F | GGAGTAAACGACCCGATAAGC |
| Aradu.2N2A3-R | GGCTGGTTTTGGTTGCTCC | Aradu.CZ64T-R | GTCCTACGATGCCCGAGAACG |
| Aradu.32P5Q-F | TTGTTTTTGGGTCGGTTGA | Aradu.FE0Z7-F | TTTTCTCCGCACTCACCTAA |
| Aradu.32P5Q-R | TTAGGTGTTGTGTAGGGTG | Aradu.FE0Z7-R | TGCCGACTCTTCTTCACCTT |
| Aradu.38MP3-F | TGATTGGTGTGGTGTGTTGC | Aradu.G395P-F | CGTTCTTGGTGTCCCTGTG |
| Aradu.38MP3-R | CTCCTTATGTGTTCGTGTTTTG | Aradu.G395P-R | TTCCTTGTGCTTGAGCCTT |
| Aradu.3UA04-F | AGTGAAAATAAGGGAAAGAC | Aradu.JE7IW-F | GCAATTTGTCCTGATGACCCTA |
| Aradu.3UA04-R | CAACTGAAACTAAACGGGAT | Aradu.JE7IW-R | CCTCCACTGTGCTCCTCCCT |
| Aradu.8XK1K-F | CTCAAGCGTTCTGCATTGCC | Aradu.M0JX8-F | CCATTCATGTCTTCGTCGT |
| Aradu.8XK1K-R | CCCTGAAGAGTTTTCCCCCA | Aradu.M0JX8-R | CTCTTCTTCACCTTCTGCG |
| Aradu.91JGK-F | ATTCAACGAGTTATCCACGG | Aradu.PU7RJ-F | AGAGGAAGGGTTATGCATT |
| Aradu.91JGK-R | GGATTCCACAACATCAGCAG | Aradu.PU7RJ-R | AACTTTCGATTTGGGGCTG |
| Aradu.94LL9-F | ATTGAAAGTGGTGGCGAAGT | Aradu.Y0LYZ-F | GGTGGTGTCACTGCTGGTA |
| Aradu.94LL9-R | TTGACGGAATGGGTGAGG | Aradu.Y0LYZ-R | GAACTCGTGGCATTGTCC |
| Aradu.AAS86-F | TGGGCTTCCAGGAGTTACAGG | β-actin-F | TTGGAAT GGGTCAGAAGGATGC |
| Aradu.AAS86-R | GTTCTTCCAGTGTCCACAGTGT | β-actin-R | AGTGGTGCCTCAGTAAGAAGC |
新窗口打开|下载CSV
1.7 数据统计分析
用Microsoft Excel对试验结果相关数据进行统计分析。2 结果与分析
2.1 AhPEPC1基因的抑制表达对花生种子含油量、脂肪酸含量和蛋白含量的影响
非转基因花生(对照)种子含油量为50.5%, 转基因系L6、L11和L21种子的含油量分别为53.4%、54.2%、55.7%。与对照相比, 转基因花生的含油量提高了5.7%~10.3% (图1-A)。棕榈酸、硬脂酸、油酸、亚油酸、花生酸、花生四烯酸等脂肪酸成分相对含量在转基因株系和对照中没有明显差异(图1-C)。说明转基因只提高了花生种子含油量, 没有改变脂肪酸成分组成。图1
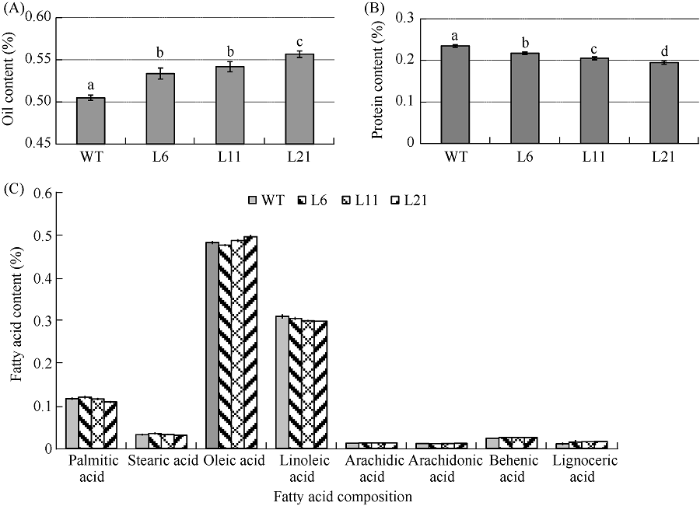 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1转基因株系和对照花生种子含油量(A)、蛋白含量(B)和脂肪酸含量(C)
WT: 对照; L6、L11、L21: 转基因株系。a、b、c、d的柱值在处理间差异显著(P = 0.05)。
Fig. 1Contents of lipid (A), protein (B), and fatty acid (C) in transgenic and control plants
WT: control plants; L6, L11, L21: transgenic plants. Bars superscripted by different letters are significant different at P = 0.05.
对照种子蛋白含量为23.5%, 转基因系L6、L11和L21种子的蛋白含量分别为21.7%、20.5%和19.5%。与对照相比, 转基因花生的蛋白含量降低了7.5%~17.0% (图1-B)。上述结果表明, 转基因花生的成熟种子随着油脂含量的增加, 蛋白含量有所下降。可推测AhPEPC1基因在花生中抑制表达, 减少了碳源向蛋白合成方向的分配, 从而降低了种子蛋白含量, 同时在一定程度上增加了合成油脂的碳流分配量, 提高了种子含油量。
2.2 转基因花生的RNA-Seq测序及分析
利用转录组测序技术, 分析转基因花生中AhPEPC1基因的抑制表达是否影响其他基因的功能。分析结果显示, 对照花生样品WT中共有17,928个基因表达, 转基因花生样品TL中共有17,839个基因表达, 其中有17,418个基因是WT和TL共同表达的, 有510个基因是WT特有表达, 有421个基因是TL特有表达(图2)。在转基因和对照花生样品中, 筛选到110个基因差异表达, 其中25个基因表达上调, 85个基因表达下调(图3)。图2
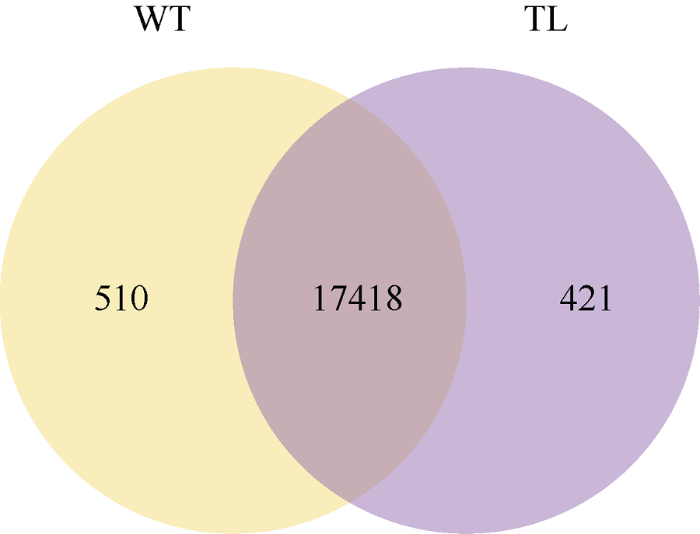 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2基因表达维恩图
WT: 对照; TL: 转基因株系。每个大圆圈中的数字之和代表该group表达了的基因总个数, 圆圈交叠的部分表示group之间共有的表达基因。
Fig. 2Venn diagram of the gene expression
WT: control plants; TL: transgenic plants. The sum of the numbers in each large circle represents the total number of genes expressed in the group, the overlapping circles represent common genes between groups.
图3
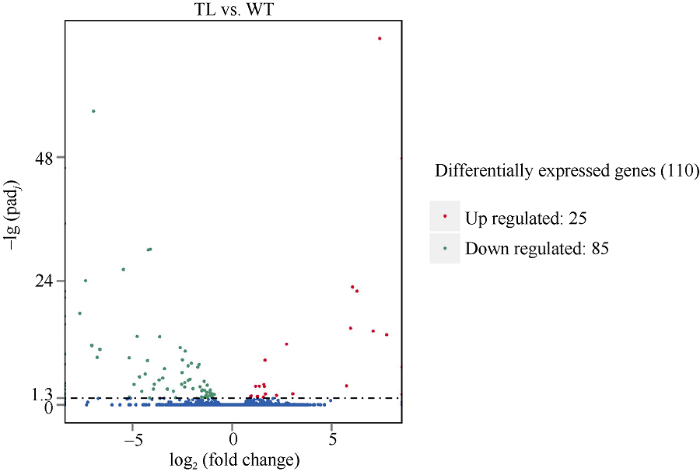 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3差异基因火山图
Padj < 0.05为差异基因筛选标准。有显著性差异表达的基因用红色点(上调)和绿色点(下调)表示, 无显著性差异表达的基因用蓝色点表示。
Fig. 3Volcano plot of differentially expressed gene distribution
The screening standard of differential genes is Padj < 0.05. Red dots indicate the up-regulated genes with significant differences, green dots indicate the down regulated genes with significant differences, no significant difference in the expression of genes with blue dots.
2.3 差异基因GO富集分析
对110个差异表达基因进行GO功能显著性富集分析, 差异基因GO富集柱状图, 直观地反映出在生物过程、细胞组分和分子功能富集的GO term上差异基因的个数分布情况。本研究挑选了富集最显著的30个GO term (图4)。在生物过程部分中, 差异表达基因几乎平均分布在23个GO term中, 其中己糖代谢过程(hexose metabolic process)功能组中含有2个基因, 磷脂酰肌醇(-3)激酶信号(phosphatidylinositol 3-kinase signaling)、磷脂酰肌醇(-3)激酶信号的调控(regulation of phosphatidylinositol 3-kinase signaling)等22个功能组各含有1个基因。在细胞组成部分, 差异表达基因分布在4个GO term中,细胞质(cytosol)及胞质部分(cytosolic part)功能组各含有2个基因。在分子功能部分, 差异表达基因分布在3个GO term中,其中核苷磷酸酶活性(nucleotide phosphatase activity)功能组含有3个基因, 磷脂酰肌醇(-3)激酶结合(phosphatidylinositol 3-kinase binding)和磷酸甘油酸变位酶活性(phosphoglycerate mutase activity)功能组各含有1个基因。图4
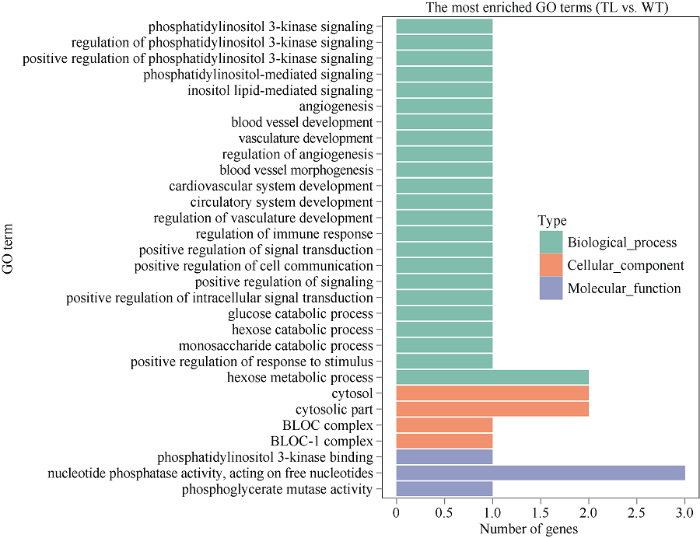 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4GO富集分类图
纵坐标为富集的GO term, 横坐标为该term中差异基因个数。绿色表示生物过程, 橙色表示细胞组分, 紫色表示分子功能。
Fig. 4GO annotation of differentially expressed genes
The ordinate is the enriched GO term, the abscissa is the number of differential genes in term. The green represents the biological process, the orange represents the cell component, and the purple represents the molecular function.
2.4 差异基因KEGG富集分析
对110个差异表达基因进行了KEGG功能注释和pathway显著性富集分析, 其中有34个基因成功获得了 KEGG注释(表2)。Pathway显著性富集分析结果显示, 差异基因多定位在光合作用、嘌呤代谢、半胱氨酸和蛋氨酸代谢、氨基酸的生物合成、次生代谢产物生物合成、植物激素信号转导等23个途径。其中氨基酸的生物合成中有2个基因(Aradu.M0JX8, Aradu. FE0Z7)下调表达(图5和图6)。说明转基因花生中AhPEPC1基因的抑制表达, 可能引起碳源向蛋白合成方向分配的减少, 从而使氨基酸生物合成途径中有些基因的表达下降。这与转基因花生中蛋白含量降低的结果吻合。这个结果需要后续功能试验进一步验证。Table 2
表 2
表 2差异基因KEGG富集情况
Table 2
| 条目 Term | 数据库 Database | 编号 ID | 差异基因个数 Sample number | 调控模式Regulation | 注释基因个数 Background number | 显著水平 P-value | 矫正显著水平 Corrected P-value |
|---|---|---|---|---|---|---|---|
| Photosynthesis - antenna proteins | KEGG PATHWAY | adu00196 | 2 | Up | 18 | 0.000093 | 0.000560 |
| Purine metabolism | KEGG PATHWAY | adu00230 | 2 | Up | 194 | 0.008769 | 0.026306 |
| DNA replication | KEGG PATHWAY | adu03030 | 1 | Up | 90 | 0.065891 | 0.116645 |
| Peroxisome | KEGG PATHWAY | adu04146 | 1 | Up | 107 | 0.077763 | 0.116645 |
| Pyrimidine metabolism | KEGG PATHWAY | adu00240 | 1 | Up | 140 | 0.100427 | 0.120512 |
| Metabolic pathways | KEGG PATHWAY | adu01100 | 2 | Up | 2197 | 0.508576 | 0.508576 |
| Selenocompound metabolism | KEGG PATHWAY | adu00450 | 2 | Down | 20 | 0.000926 | 0.015741 |
| Cysteine and methionine metabolism | KEGG PATHWAY | adu00270 | 2 | Down | 108 | 0.021504 | 0.182780 |
| Stilbenoid, diarylheptanoid and gingerol biosynthesis | KEGG PATHWAY | adu00945 | 1 | Down | 18 | 0.038622 | 0.218860 |
| Base excision repair | KEGG PATHWAY | adu03410 | 1 | Down | 46 | 0.092940 | 0.284623 |
| Biosynthesis of amino acids | KEGG PATHWAY | adu01230 | 2 | Down | 250 | 0.095036 | 0.284623 |
| Phenylalanine metabolism | KEGG PATHWAY | adu00360 | 1 | Down | 50 | 0.100455 | 0.284623 |
| Flavonoid biosynthesis | KEGG PATHWAY | adu00941 | 1 | Down | 62 | 0.122643 | 0.297848 |
| RNA degradation | KEGG PATHWAY | adu03018 | 1 | Down | 111 | 0.207912 | 0.441813 |
| Pyrimidine metabolism | KEGG PATHWAY | adu00240 | 1 | Down | 140 | 0.254567 | 0.471897 |
| RNA transport | KEGG PATHWAY | adu03013 | 1 | Down | 166 | 0.294144 | 0.471897 |
| Plant-pathogen interaction | KEGG PATHWAY | adu04626 | 1 | Down | 182 | 0.317487 | 0.471897 |
| Phenylpropanoid biosynthesis | KEGG PATHWAY | adu00940 | 1 | Down | 193 | 0.333104 | 0.471897 |
| Endocytosis | KEGG PATHWAY | adu04144 | 1 | Down | 217 | 0.365995 | 0.478609 |
| Plant hormone signal transduction | KEGG PATHWAY | adu04075 | 1 | Down | 314 | 0.483750 | 0.543526 |
| Biosynthesis of secondary metabolites | KEGG PATHWAY | adu01110 | 3 | Down | 1282 | 0.505639 | 0.543526 |
| Ribosome | KEGG PATHWAY | adu03010 | 1 | Down | 340 | 0.511554 | 0.543526 |
| Metabolic pathways | KEGG PATHWAY | adu01100 | 4 | Down | 2197 | 0.693074 | 0.693074 |
新窗口打开|下载CSV
图5
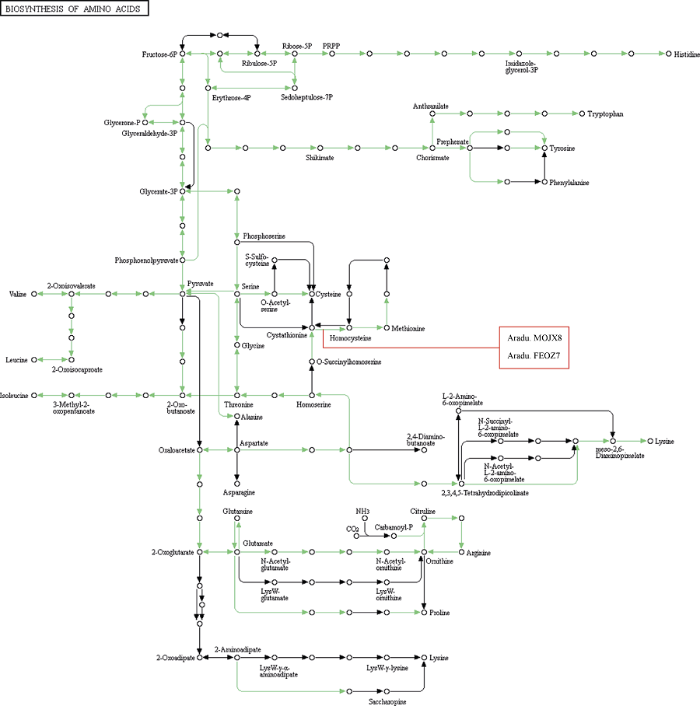 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5氨基酸生物合成途径的转录变化
Fig. 5Transcriptional changes in biosynthesis of amino acids pathways
图6
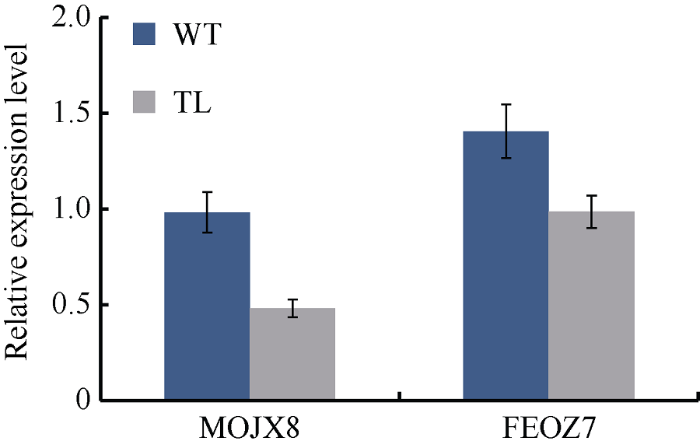 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6氨基酸合成途径中基因相对表达量
Fig. 6Relative expression of genes (MOJX8, FEO27) in the biosynthesis of amino acids
2.5 转录组数据验证
为了验证RNA-seq所测结果的准确性, 利用荧光定量PCR分析了15个DEG (differential expressed gene)在非转基因对照和转基因花生荚果形成中期的种子中的表达情况。结果表明, 15个DEG在RNA- Seq和 RT-PCR中的表达模式基本是一致的, 但基因表达水平的变化幅度有一定差异(表3), 说明转录组测序分析结果是可靠的。Table 3
表3
表3RNA-seq与RT-PCR验证倍数差别
Table 3
| 基因编号 Gene ID | 基因描述 Gene description | RNA-seq变化倍数 RNA-seq fold change | 调控模式 Regulation | RT-PCR变化倍数 RT-PCR fold change |
|---|---|---|---|---|
| Aradu.2N2A3 | Protein IQ-DOMAIN 14 | 53.7 | Up | 2.2 |
| Aradu.32P5Q | Transketolase, chloroplastic | 164.3 | Down | 2.1 |
| Aradu.38MP3 | — | 61.8 | Up | 1.7 |
| Aradu.3UA04 | Pollen-specific protein SF21 | 11.2 | Down | 16.8 |
| Aradu.8XK1K | DNA polymerase alpha catalytic subunit | 2.3 | Up | 1.1 |
| Aradu.91JGK | Probable leucine-rich repeat receptor-like serine/threonine-protein kinase | 10.5 | Down | 1.6 |
| Aradu.94LL9 | F-box/FBD/LRR-repeat protein | 4.7 | Up | 1.9 |
| Aradu.AAS86 | ABC transporter G family member 36 | 2.4 | Up | 3.0 |
| Aradu.CZ64T | Protein FAR1-RELATED SEQUENCE 12 | 5.2 | Down | 2.4 |
| Aradu.FE0Z7 | Cystathionine beta-lyase, chloroplastic | 137.4 | Down | 1.4 |
| Aradu.G395P | Copper-transporting ATPase PAA1, chloroplastic | 30.4 | Down | 1.5 |
| Aradu.JE7IW | Pollen-specific protein SF21 | 25.1 | Down | 2.9 |
| Aradu.M0JX8 | Cystathionine beta-lyase, chloroplastic | 201.1 | Down | 2.0 |
| Aradu.PU7RJ | MADS-box transcription factor 6 | 2.9 | Down | 3.4 |
| Aradu.Y0LYZ | Myb-like protein | 2.0 | Up | 2.4 |
新窗口打开|下载CSV
3 讨论
花生籽仁中贮藏的物质(包括油脂、蛋白质、糖类等)来源于光合碳同化的原初产物, 花生油脂代谢与其他能量物质的代谢是普遍联系和交织的, 抑制这些代谢竞争途径可以增加参与油脂合成的碳流, 提高含油量[12,13]。油脂合成与蛋白质合成2个代谢过程均需要利用糖酵解产物磷酸烯醇式丙酮酸(phosphoenolpyruvate, PEP)作底物。研究者发现, 大豆籽仁油脂和蛋白质含量与磷酸烯醇式丙酮酸羧化酶(PEPC)活性密切相关[14,15,16]。也有研究指出, 油菜种子中PEPC活性与种子的TAG和脂肪酸含量之间有一定的关系[17,18]。Chen等[6]提出“底物竞争”假说, 认为油脂和蛋白质均来自葡萄糖糖酵解产物丙酮酸, PEPC酶催化丙酮酸合成草酰乙酸, 进入蛋白质合成途径; 而ACCase酶可催化丙酮酸合成乙酰-CoA,进入脂肪酸合成途径。一般认为PEPC酶和ACCase酶的相对活性决定了二者竞争的平衡点。在油菜中通过反义抑制PEPC基因的表达, 获得了含油量提高6.4%~18.0%的转基因油菜, 转化植株油脂含量与蛋白质含量呈显著负相关。本研究将反义AhPEPC1基因转化花生, 获得了含油量提高5.7%~10.3%的转基因花生。转基因花生的脂肪酸成分相对含量未发生改变, 但蛋白含量降低了7.5%~17.0%。研究结果也说明了花生种子中油脂含量与蛋白含量是相互制约的。AhPEPC1基因的抑制表达能明显提高花生含油量, 转基因植株又能正常生长发育, 表明利用该基因通过转基因途径提高油料作物种子含油量具有良好的科研价值和应用前景。
RNA-Seq测序技术可以在植物转录组水平上对基因的表达情况进行准确、全面的研究, 特别是对转基因植株或目标性状突变体植株的转录组分析, 可以揭示单个目标基因的变化对植物体性状带来的各种影响[19]。本研究对转基因株系和对照花生发育中期的种子进行了RNA-Seq测序分析, 筛选到110个基因差异表达, 其中25个基因上调表达, 85个基因表达下调, 但是其中有28.2%的基因在花生基因组数据库中没有找到注释信息, 说明存在着大量未知基因信息。差异基因的KEGG富集分析结果显示, 氨基酸的生物合成途径中有2个基因(Aradu.M0JX8, Aradu.FE0Z7)下调表达, 说明转基因花生中AhPEPC1基因的抑制表达, 可能引起碳源向蛋白合成方向分配的减少, 使氨基酸生物合成途径中有些基因的表达下降, 从而使转基因花生蛋白质含量降低, 这些分析结果还需开展后续试验进一步验证。
4 结论
花生AhPEPC1基因的抑制表达可影响转基因后代种子含油量和蛋白含量。与对照相比, 转基因花生含油量提高了5.7%~10.3%, 蛋白质含量降低了7.5%~17.0%。转录组测序数据初步分析了AhPEPC1基因抑制表达对相关基因表达的影响, 其中转基因花生氨基酸的生物合成途径中有2个基因(Aradu. M0JX8, Aradu.FE0Z7)下调表达, 与转基因花生蛋白含量降低的结果吻合。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/S1872-2075(08)60016-3URL [本文引用: 1]
[本文引用: 2]
[本文引用: 2]
DOI:10.1007/BF00046459URL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
DOI:10.1038/nbt.1621 [本文引用: 1]
[本文引用: 1]
DOI:10.1104/pp.107.110361URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1046/j.1365-2486.1999.00270.xURL [本文引用: 1]
DOI:10.1016/j.envexpbot.2005.06.021URL [本文引用: 1]
[本文引用: 1]
