 ,2,*
,2,*Identification of rice stress-related gene co-expression modules by WGCNA
LI Xu-Kai1, LI Ren-Jian2, ZHANG Bao-Jun ,2,*
,2,*通讯作者:
收稿日期:2018-12-10接受日期:2019-04-15网络出版日期:2019-04-18
| 基金资助: |
Received:2018-12-10Accepted:2019-04-15Online:2019-04-18
| Fund supported: |
作者简介 About authors
E-mail:xukai_li@sxau.edu.cn。

摘要
关键词:
Abstract
Keywords:
PDF (2211KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李旭凯, 李任建, 张宝俊. 利用WGCNA鉴定非生物胁迫相关基因共表达网络[J]. 作物学报, 2019, 45(9): 1349-1364. doi:10.3724/SP.J.1006.2019.82061
LI Xu-Kai, LI Ren-Jian, ZHANG Bao-Jun.
干旱、冷害和高盐等非生物逆境会对植物的生长和发育产生不利影响, 造成植物生长发育缓慢, 严重时可导致植物死亡[1]。干旱、冷害和高盐导致的植物应激可破坏其细胞结构和影响关键的生理功能[1], 使植物细胞产生渗透胁迫, 水分外流, 蛋白失活或变性, 产生大量的活性氧(reactive oxygen species, ROS)进而损伤细胞, 最终导致植物光合作用被抑制, 代谢功能紊乱[2]。在农业方面, 这些逆境反应造成了作物产量和品质的降低。植物的耐受性会随着环境的改变而改变[3], 同时受许多基因控制[4], 基因工程为发展多耐性作物提供了方法, 可将多个基因组装导入作物, 实现作物更高的耐受性[5]。高通量测序技术的飞速发展, 也为基因的挖掘提供了快捷的方法。研究人员利用转录组学和代谢组学分析了烟草对低温胁迫的反应[6]。王涛研究团队通过建立黄花苜蓿非生物胁迫转录组数据库, 揭示了植物膜锚定转录因子在干旱胁迫下重定位进入细胞核调控机体氧化还原平衡的分子机制[7]。
加权共表达网络分析(Weighted Gene Co-expression Network Analysis, WGCNA)利用植物体内各个生命活动互相关联的特点, 通过高通量测序技术获得基因的表达情况, 对基因间的连接系统进行幂次处理, 使网络内部的基因符合无尺度网络拓扑结构, 即网络中存在的少数基因与大多数基因存在联系的特点[8], 该中心基因即为起关键调控的基因(hub基因)。该方法将具有相似表达模式的基因聚类到同一个模块, 多被用于研究共表达网络与植物性状之间的生物学关系, 挖掘与性状之间高度关联的关键基因。本研究利用水稻的根、叶、花蕊、种子等材料, 以及在干旱、冷、盐胁迫下的不同处理时间共47份转录组数据, 构建了共表达网络, 得到了15个模块。从国家水稻数据中心(http://www.ricedata.cn/)获取已克隆的干旱、冷、盐相关的406个基因(附表1), 通过查看已知基因所在的模块, 并通过对冷、干旱、盐胁迫处理的不同时间段的转录组数据的差异表达分析, 根据已报道相关胁迫基因在模块中的数量和各胁迫不同处理中差异表达基因存在的模块数量, 确定并对数目较多的模块进行生物学功能分析。通过对干旱、冷、盐胁迫共同存在的模块进行了功能分析, 预测水稻耐受性关联的关键基因, 为提高水稻耐受性方面的研究提供了有效参考, 打开了该研究方向的新思路。
1 材料与方法
1.1 水稻转录组数据的获取与分析
水稻干旱胁迫、冷胁迫、盐胁迫处理的100个转录组数据来源于NCBI的SRA (Sequence Read Archive)数据库(附表2)。首先利用SRAtoolkit[9]软件的fast- dump将SRA格式文件转换为序列文件fastq, 利用FastQC[10]软件对原始测序数据进行质量评估, 紧接着用Trimmonatic[11]软件去除接头、过滤低质量的reads, 得到clean data。利用Hisat2[12]软件将clean data比对到水稻基因组。水稻基因组序列和注释信息下载自Rice Genome Annotation Project (http://rice.plantbiology. msu.edu)数据库。用featureCounts[13]获得数据的reads计数, 之后根据基因表达的TPM值(transcripts per million)公式 $\text{TP}{{\text{M}}_{i}}=\frac{{{X}_{1}}}{{{L}_{1}}}\times \frac{1}{\sum{\frac{{{X}_{j}}}{{{L}_{j}}}}}\times {{10}^{6}}$, 编写计算TPM值的R语言代码, 构建基因表达矩阵, R语言代码如下:df<-read.delim("counts.txt",sep="\t",header=T,
skip=1, row.names=1)
len<-as.vector(df[,5]);
df<-df[,6:ncol(df)];
TPM <- function(counts,len) {
x <- counts/len
return(t(t(x)*1e6/colSums(x)))
} res<-TPM(df,len);
write.table(res,file="TPM.txt",sep="\t",quote=F)。
1.2 共表达网络构建和已知基因模块的选取
基因的表达量矩阵来自不同胁迫处理下的基因表达量, 带重复的样本进行均值预处理。使用R软件(R version 3.4.4)和WGCNA (R version 1.6.6)包, 构建加权基因共表达网络与划分相关模块[14]。使用WGCNA包中的pickSoftThreashold计算权重值, 本实验选取power值为12, 使用blockwiseModules构建无尺度网络, 参数按照默认设置[14]。利用edgeR[15]分别对数据库中下载到的冷胁迫处理3 h、6 h、24 h的水稻幼苗和干旱胁迫处理1 h、6 h、24 h的水稻幼苗以及盐胁迫处理1 h、5 h、24 h的水稻幼苗进行差异表达分析, 分析差异表达所在的模块, 并结合水稻已克隆报道的胁迫基因, 确定已报道基因所在模块及数量, 确定各胁迫下的特异性模块。1.3 模块的GO富集分析
首先提取模块的中基因, 利用TBtools (A Toolkit for Biologists integrating various biological data handling tools with a user-friendly interface)进行GO富集分析[16]。使用水稻基因组作为参考数据库, P值小于0.005。利用Cytoscape (version 3.6.1)对模块中的网络进行可视化[17]。1.4 转录因子注释分析
通过PlantTFDB数据库(http://planttfdb.cbi.pku. edu.cn/index.php?sp=Osj), 获得水稻中的转录因子共2409个, 分属于52个家族[18]。与模块中预测的基因进行比较, 得到共表达模块中预测的转录因子信息(附表3)。2 结果与分析
2.1 水稻加权基因共表达网络构建
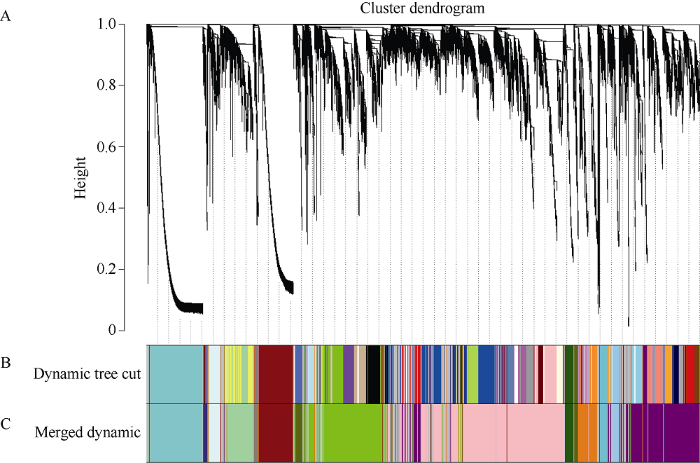
对水稻表达矩阵中表达量较低的基因进行过滤, 获得30,339个高表达基因。选择权重值β=12来构建无尺度网络, 之后按照混合动态剪切的标准划分模块, 随之依次计算每个模块的特征向量值(eigengenes), 对距离较近的模块进行合并, 最终获得15个共表达模块(图1), 每个模块用不同的颜色来表示聚类到一起的基因。其中pink模块聚类到的基因数目最多, 为7386个, bisque4模块包含的基因最少, 有286个。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1基因聚类树和样品分割
A: 样品的层次聚类树; B: dynamic tree cut表示根据各个基因的表达量计算划分的模块; C: merged dynamic是根据dynamic tree cut合并相似模块的结果。
Fig. 1Gene cluster dendrograms and module detecting
A: a hierarchical clustering tree of samples; B: the dynamic tree cut represents the module divided according to the expression of each gene; C: the merged dynamic is the result of merging similar modules according to the dynamic tree cut.
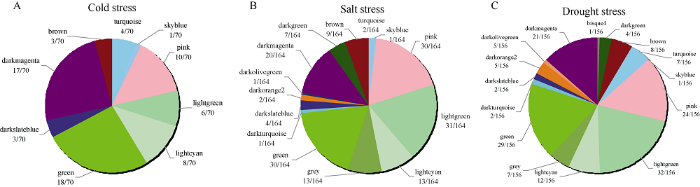
将水稻数据库中已报道与干旱、冷、盐胁迫相关的基因与模块中的基因比较。已报道的干旱基因在共表达网络中有156个, 其中lightgreen模块和green模块存在较多, 分别为32个和29个; 已报道的冷胁迫相关基因共有164个, 其中lightgreen和green模块存在基因数目最多, 分别为31个和32个; 盐胁迫的已报道基因在共表达模块中共有70个, 在green模块和darkmagenta模块中分别存在18个和17个(图2)。干旱、冷、盐胁迫相关基因被聚在各个模块中, 其中在green模块中大量的聚集。
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2报道基因分布情况
图中英文表示模块的颜色, 数字表示已报道相关基因所属模块的数目及总数目。A: 已报道冷胁迫相关基因在各个模块中所占的比例; B: 已报道干旱胁迫相关基因在各个模块中所占的比例; C: 已报道盐胁迫相关基因在各个模块中所占的比例。
Fig. 2Distribution of opented genes
The English words in the figure indicate the color of the module, and the numbers mean the relevant genes number and total number of modules. A: the proportion of cold stress related genes in each module reported; B: the proportion of drought stress related genes in each module reported; C: the proportion of salt stress related genes in each module reported.
为了验证和确定模块选取的准确性, 同时对数据中冷胁迫处理3 h、6 h、12 h处理的水稻幼苗, 干旱处理1 h、6 h、24 h的水稻幼苗和盐胁迫处理1 h、5 h、24 h的水稻幼苗3份转录组数据进行了差异表达基因分析。在对冷胁迫的差异表达分析中, 模块darkmagenta中不同处理下的差异基因数目占所有基因总数的17%左右, green模块中占18%左右。在干旱胁迫差异表达分析中, 模块green中的差异基因占总差异基因数目的13%左右, lightgreen模块差异基因约占总数目的16%左右。在对盐胁迫的差异表达分析中发现, green模块中的差异基因约占总差异基因数目的13%左右, lightgreen模块中5 h处理下的差异基因数目约占40%左右(附表4)。
2.2 模块的GO富集分析
选取已报道基因所在最多的模块, 结合不同胁迫下差异表达基因在模块中的分布结果(附表4), 干旱选取lightgreen模块和green模块, 冷胁迫选取green模块和darkmagenta模块, 盐胁迫选取lightgreen模块和green模块。对这些已报道基因有关联的基因进行GO功能富集分析, 富集结果涉及到了生物学过程(biological process, BP)、分子功能(molecular function, MF)和细胞组分(cellular component, CC)。与干旱相关的lightgreen和green模块都富集到了与干旱相关的调控通路, 主要是与脂肪酸分解过程(GO:0009062)、活性氧的响应(GO:0000302)、脱落酸响应(GO:0000302)、渗透胁迫响应(GO:00009737)等相关。与冷相关的green模块和darkmagenta模块富集到了与钙依赖性蛋白激酶活性(GO:0010857)、细胞酰胺代谢过程(GO:0044106)等, 以及茉莉酸介导的信号转导通路的调控(GO:2000022)。与盐胁迫相关的green模块和lightgreen模块也富集到了盐胁迫响应(GO:0009651)、内源性激素的响应(GO: 0009719)和激素应答(GO:0009725)的一些响应胁迫相关的功能(表1和附表5)。
Table 1
表1
表1模块GO富集情况(部分)
Table 1
| 胁迫 Stress | 模块 Module | 基因数目 Number of genes | GO | 显著富集的项目 Significantly enriched term | P值 P-value | |
|---|---|---|---|---|---|---|
| 干旱 Drought | Lightgreen | 33 | GO:0009062 | 脂肪酸分解过程 Fatty acid catabolic process | 0 | |
| 330 | GO:1901700 | 对含氧化合物的响应 Response to oxygen-containing compound | 0 | |||
| 65 | GO:0000302 | 活性氧的响应 Response to reactive oxygen species | 1.11E-16 | |||
| 132 | GO:0009737 | 脱落酸响应 Response to abscisic acid | 1.38E-10 | |||
| 160 | GO:0006970 | 渗透胁迫响应 Response to osmotic stress | 6.47E-13 | |||
| 103 | GO:0009415 | 对水的响应 Response to water | 5.72E-09 | |||
| Green | 23 | GO:0043069 | 程序性细胞死亡的负调控 Negative regulation of programmed cell death | 8.84E-09 | ||
| 203 | GO:0006979 | 氧化应激反应 Response to oxidative stress | 4.41E-12 | |||
| 胁迫 Stress | 模块 Module | 基因数目 Number of genes | GO | 显著富集的项目 Significantly enriched term | P值 P-value | |
| 冷 Cold | Green | 25 | GO:0010857 | 钙依赖性蛋白激酶活性 Calcium-dependent protein kinase activity | 1.12E-08 | |
| 31 | GO:0044106 | 细胞酰胺代谢过程 Cellular amine metabolic process | 2.16E-07 | |||
| 20 | GO:0060548 | 细胞死亡负调控 Negative regulation of cell death | 2.21E-07 | |||
| 14 | GO:0003714 | 转录辅阻遏活性 Transcription corepressor activity | 2.64E-07 | |||
| 16 | GO:2000022 | 茉莉酸介导的信号转导通路的调控 Regulation of jasmonic acid mediated signaling pathway | 3.58E-06 | |||
| Darkmagenta | 70 | GO:0008299 | 异戊二烯生物合成过程 Isoprenoid biosynthetic process | 0 | ||
| 167 | GO:0009266 | 温度刺激响应 Response to temperature stimulus | 0 | |||
| 167 | GO:0006091 | 前体代谢物和能量的产生 Generation of precursor metabolites and energy | 0 | |||
| 32 | GO:0016116 | 类胡萝卜素代谢过程 Carotenoid metabolic process | 2.22E-16 | |||
| 盐 Salt | Green | 261 | GO:0009651 | 盐胁迫响应 Response to salt stress | 3.81E-11 | |
| 586 | GO:0009719 | 内源性刺激响应 Response to endogenous stimulus | 1.94E-08 | |||
| 547 | GO:0009725 | 激素应答 Response to hormone | 2.68E-07 | |||
| Lightgreen | 244 | GO:1901575 | 有机物分解代谢过程 Organic substance catabolic process | 0 | ||
| 10 | GO:0046487 | 乙醛酸代谢过程 Glyoxylate metabolic process | 1.04E-08 | |||
新窗口打开|下载CSV
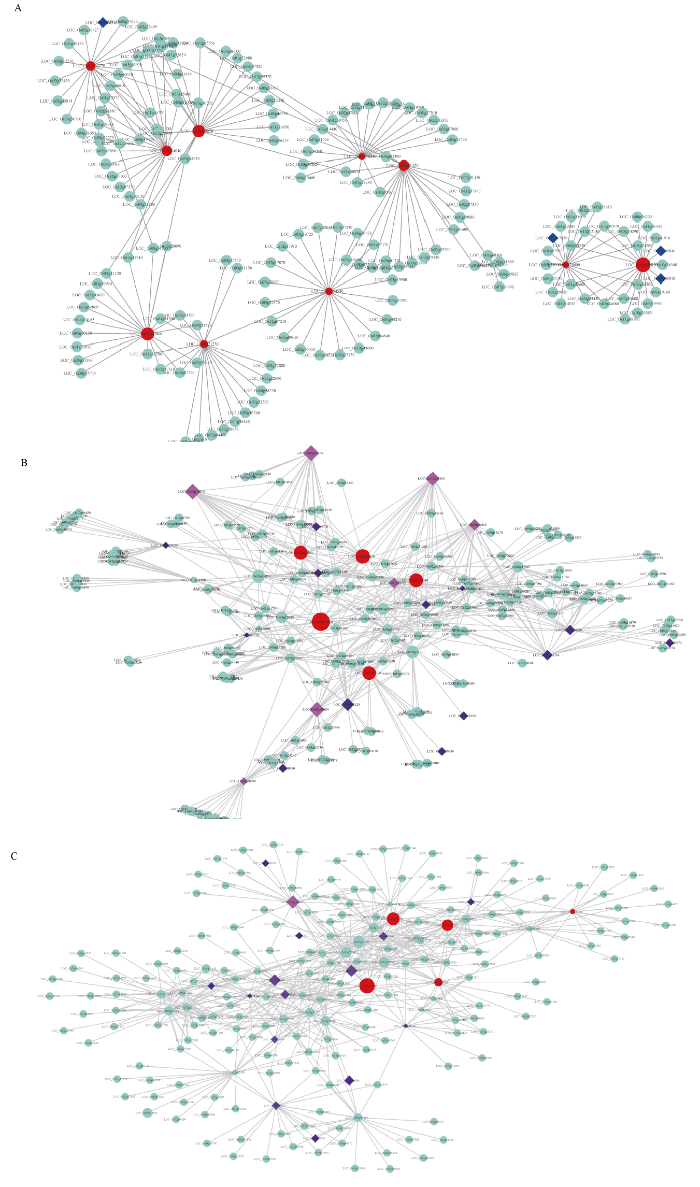
植物对逆境的响应是多个基因调控的, 在目前已报道与逆境胁迫相关的基因中, 仍有部分潜在的基因其在逆境胁迫中发挥的具体作用机制尚不明确。本研究对干旱胁迫、冷胁迫、盐胁迫中已报道逆境相关基因存在较多的6个模块, 通过权重共表达网络分析, 并使用Cytoscape软件对网络进行可视化(图3)。其中, 在darkmagenta中的WLP1[19] (LOC_ Os01g54540)、OsPDS[20] (LOC_Os03g08570)、OsCYL4[21] (LOC_Os09g02270)、hbd2[22] (LOC_Os02g40860); lightgreen模块中的OsDREB1G[23] (LOC_Os08g 43210)、OCPI2[24] (LOC_Os01g42860)、OsMSR15[25] (LOC_Os03g41390)、SRWD1[26] (LOC_ Os08g38880)、OsNHX1[27] (LOC_Os07g47100)都处于网络的核心位置(图3)。对处于网络中心权重值较高的基因在水稻中进行了注释和在拟南芥中进行了同源基因的注释(表2)。
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3冷、干旱、盐胁迫相关的共表达网络
图中点的大小表示输入基因在网络中的权重值大小, 红色表示属于该共表达网络的核心基因, 紫色菱形表示注释到的转录因子, 粉色菱形表示既注释到转录因子又属于网络中的核心基因。A: 模块darkmagenta中与冷胁迫相关的基因共表达网络; B: 模块lightgreen中与干旱胁迫相关的基因共表达网络; C: 模块lightgreen中与盐胁迫相关的基因共表达网络。
Fig. 3Coexpression network related to cold, drought, and salt stress
The size of dots in the graph indicates the weight of the input gene in the network. Red represents the core gene belonging to the co-expression network. The purple diamond indicates the annotated transcription factor, and the pink diamond indicates the core gene that is both annotated to the transcription factor and belongs to the network. A: Module darkmagenta coexpression network of genes related to cold stress; B: Module lightgreen is a co-expression network of genes related to drought stress; C: Gene co-expression network related to salt stress in module lightgreen.
Table 2
表2
表2模块核心基因功能注释
Table 2
| 模块分类 Module name | 基因名称 Gene name | 水稻中基因注释 Annotation in rice | 拟南芥同源基因注释 Homologous gene in A. thaliana | 功能注释 Gene function | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Darkmagnenta | LOC_Os03g08570 | 胺氧化酶 Amine oxidase | AT4G14210 | 一种在类胡萝卜素生物合成过程中催化八氢番茄红素致zeta-胡萝卜素去饱和的酶 An enzyme that catalyzes the desaturation of phytoene to zeta-carotene during carotenoid biosynthesis | ||||||||||||||||||
| LOC_Os01g01420 | 表达蛋白 Expressed protein | AT3G54360 | 编码一个过氧化氢酶活性相关的过氧化氢酶伴侣, 可响应多重胁迫 Encodes a catalase chaperon that is essential for catalase activity. Required for multiple stress responses. | |||||||||||||||||||
| LOC_Os04g39970 | 含有结构域的液泡分选蛋白9 Vacuolar sorting protein 9 domain-containing protein | |||||||||||||||||||||
| 模块分类 Module name | 基因名称 Gene name | 水稻中基因注释 Annotation in rice | 拟南芥同源基因注释 Homologous gene in A. thaliana | 功能注释 Gene function | ||||||||||||||||||
| LOC_Os05g34040 | 单加氧酶 Monooxygenase | AT4G30720 | 编编码在叶绿体基质中检测到的推定的氧化还原酶/电子载体 Encodes a putative oxidoreductase/electron carrier detected in the chloroplast stroma | |||||||||||||||||||
| LOC_Os02g40860 | 酪蛋白激酶1 CK1_CaseinKinase_1 | |||||||||||||||||||||
| LOC_Os09g21250 | 表达蛋白 Expressed protein | AT5G23040 | 编码一种蛋白质, 使原卟啉抗体能够与pPORA的转运序列结合, 调节pPORA向质体基质的易位, 并阻止易位多肽链运回细胞质 Encodes a protein that enables protochlorophillide's binding to pPORA’s transit sequence, regulating Ppora’s translocation into the plastid stroma, and blocking movement of the translocating polypeptide chain back into the cytosol. | |||||||||||||||||||
| LOC_Os09g02270 | 环化酶家族蛋白 Cyclase family protein | AT4G35220 | 环化酶家族蛋白 Cyclase family protein | |||||||||||||||||||
| LOC_Os01g54540 | 核糖体蛋白L13 Ribosomal protein L13 | AT1G78630 | 核糖体蛋白L13家族蛋白 Ribosomal protein L13 family protein | |||||||||||||||||||
| Lightgreen | LOC_Os10g28600 | PPR重复结构域含有蛋白质 PPR repeat domain containing protein | AT4G31850 | 编码含有27个五肽-重复序列(PPR)基序的蛋白质。 Encodes a protein containing 27 pentatrico-peptide repeat (PPR) motifs. | ||||||||||||||||||
| LOC_Os01g07120 | 含有AP2结构域的蛋白质 AP2 domain containing protein | AT2G40340 | 编码ERF/AP2转录因子家族的DREB亚家族A-2的成员。DREB2A和DREB2B参与对干旱的反应。 Encodes a member of the DREB subfamily A-2 of ERF/AP2 transcription factor family. DREB2A and DREB2B that are involved in response to drought. | |||||||||||||||||||
| LOC_Os08g43210 | 含有AP2结构域的蛋白质 AP2 domain containing protein | |||||||||||||||||||||
| LOC_Os08g43334 | 含有HSF型DNA结合域的蛋白质 HSF-type DNA-binding domain containing protein | AT4G11660 | 热应激转录因子(HSF)家族成员 Member of Heat Stress Transcription Factor (Hsf) family | |||||||||||||||||||
| LOC_Os03g41390 | ZOS3-15-C2H2锌指蛋白 ZOS3-15-C2H2 zinc finger protein | AT1G27730 | 与高等植物中的Cys2/HIS2型锌指蛋白有关。结果表明, ZAT10产生的耐盐性部分依赖于EnA1/PMR2。 Related to Cys2/His2-type zinc-finger proteins found in higher plants. responses. Salt tolerance produced by ZAT10 appeared to be partially dependent on ENA1/PMR2 | |||||||||||||||||||
| LOC_Os12g39400 | ZOS2-02-C2H2锌指蛋白 ZOS12-09-C2H2 zinc finger protein | |||||||||||||||||||||
| LOC_Os05g34830 | 无顶端分生组织蛋白 No apical meristem protein | |||||||||||||||||||||
| 模块分类 Module name | 基因名称 Gene name | 水稻中基因注释 Annotation in rice | 拟南芥同源基因注释 Homologous gene in A. thaliana | 功能注释 Gene function | ||||||||||||||||||
| LOC_Os01g42860 | 抑制剂I家族蛋白 Inhibitor I family protein | |||||||||||||||||||||
| LOC_Os02g09830 | BZIP转录因子结构域蛋白 bZIP transcription factor domain containing protein | |||||||||||||||||||||
| LOC_Os08g38880 | WD40重复家族蛋白 WD-40 repeat family protein | |||||||||||||||||||||
| LOC_Os07g47100 | 质子逆向转运蛋白-2家族 Proton antiporter-2 family | AT3G05030 | 编码液泡K+/H+交换器, 该交换器对液泡膜上活跃的K+摄取必不可少, 并参与调节气孔关闭。 Encodes a vacuolar K+/H+ exchanger essential for active K+ uptake at the tonoplast and involved in regulating stomatal closure. | |||||||||||||||||||
新窗口打开|下载CSV
2.3 干旱、冷、盐胁迫相关基因功能富集分析
对不同胁迫在共表达网络分析中得到的基因进行韦恩图分析, 发现干旱、冷、盐胁迫的相关报道基因大量被聚类到green模块(图3)。冷胁迫共有3006个基因被富集到了green模块, 其中与干旱共有的基因数目为2680个, 与冷胁迫相关的基因数目为2716个, 特有的基因有209个; 干旱胁迫在绿色模块富集到的基因数目多达4226个, 与盐胁迫的共有基因比与冷胁迫共有的基因数目要多, 有3663个, 特有的基因数目为462个; 盐胁迫总共有4057个基因被富集到绿色模块; 三者共有的基因数目为2599个(图4)。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4绿色模块中与3种胁迫相关的共表达基因
绿色模块中冷、干旱、盐胁迫下, 预测的与已报道基因共表达基因的韦恩图。A: 黄色表示与已报道冷胁迫基因共表达的基因, 蓝色表示与已报道干旱胁迫基因共表达的基因, 绿色表示与已报道盐胁迫基因共表达的基因; B: 柱状图中分别表示已报道的相关基因的总数目; C: 分别表示3种已报道胁迫基因的交集数目、存在两两交集的数目的总个数、各自特有未被取到交集的基因数目总和。
Fig. 4Modular network gene in co-expression network
In the green module of Wean map the predicted co-expressed gene with the reported genes under cold, drought and salt stresses. A: yellow indicates a gene co-expressed with a cold stress gene, blue indicates a gene co-expressed with a drought stress gene, and green indicates a gene co-expressed with a salt stress gene. B: the total number of reported genes. C: the total number of related genes reported which shows the sum of the three kinds of reported stress genes, the total number of pairs of intersections, and the sum of genes that are not uniquely intersected.
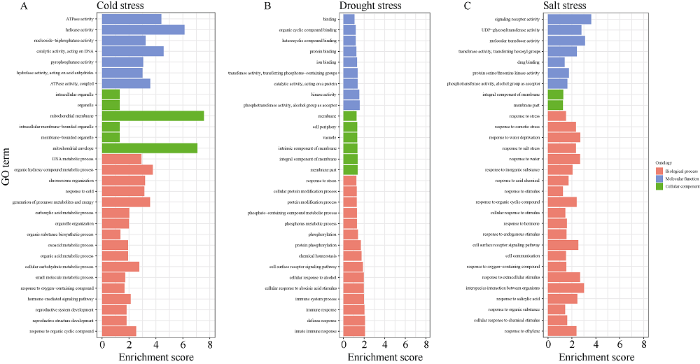
对冷胁迫富集到了焦磷酸酶活性(GO: 0016462)、核苷三磷酸酶活性(nucleoside-triphosphatase activity)等分子功能(molecular function, MF), 与样本具有较高生物学关系的响应冷胁迫(GO: 0009409)等生物学过程(biological process, BP), 以及有机生物合成过程(GO:1901576)、细胞糖类代谢过程(GO:0044262)等生物学过程(图5)。
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5绿色模块中3种胁迫特有共表达基因的GO功能富集
不同胁迫下特有的共表达基因GO富集结果。横轴表示富集分数, 纵轴表示富集到的GO通路。A: 冷胁迫下特有的共表达基因GO功能富集结果; B: 干旱胁迫下特有的共表达基因GO功能富集结果; C: 盐胁迫下特有的共表达基因GO富集结果。
Fig. 5GO functional enrichment of three kinds of stress-specific co-expressed genes in the green module
The results of GO enrichment of unique co-expressed genes under different stresses. The horizontal axis represents the enrichment score and the vertical axis represents the enriched GO pathway. A: the results of GO functional enrichment of the unique co-expressed genes under cold stress; B: the GO functional enrichment result of the unique co-expressed genes under drought stress; C: the GO enrichment result of the unique co-expressed genes under salt stress.
对干旱胁迫特有的基因, 富集到一些与蛋白质相关的功能, 包括蛋白质结合(GO:0005515)、作用于蛋白质的催化活性(GO:0140096)等; 在细胞组分中, 显著富集到4个与细胞膜相关的功能; 在生物学过程中, 富集到蛋白质修饰(GO:0006464)、蛋白质磷酸化(GO:0006468)等功能, 也富集到与信号转导相关的细胞表明受体信号通路(GO:0007166), 脱落酸刺激的细胞反应(GO:0071215), 和一些与免疫相关的功能, 包括免疫系统过程(GO:0002376)、免疫应答(GO:0006955)、防御反应(GO:0006952)、固有免疫反应(GO:0045087)等(图5)。
对盐胁迫特有的基因, 最显著富集到的是信号受体活性(GO:0038023)、分子传感性活性(GO: 0060089); 细胞组分中富集到一些与膜组分相关的功能, 包括膜的整体组份(GO:0016021)、膜部分(GO:0044425); 生物学功能富集到较多与盐胁迫相关的功能, 包括渗透胁迫反应(GO:0006970)、盐胁迫反应(GO:0009651)、对水的反应(GO:0009415)、对水杨酸的反应(GO:0009751)、对乙烯的反应(GO: 0009723)(图5)。
在3种胁迫共有的基因中有20个注释到了已报道与冷胁迫相关的基因, 35个注释到了已报道与干旱胁迫相关的基因, 35个注释到了已报道与盐胁迫相关的基因, 其中已报道的同时参与3种胁迫的基因有3个, 分别是编码AT-hook蛋白的OsAHL1[28]、编码丝裂原活化蛋白激酶的OsMAPK5[29]和NAC转录因子OsNAC6[30]; 同时参与调控干旱和盐胁迫的基因有17个, 同时参与调控干旱和冷胁迫的基因有6个, 同时参与调控冷和盐胁迫的基因有2个(表3)。
Table 3
表3
表3多种胁迫调控基因功能注释
Table 3
| 候选基因 Candidate gene | 转录因子 Transcription factor | 冷胁迫 Cold | 干旱 Drought | 盐 Salt | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| LOC_Os11g05160 | AT-hook蛋白 AT-hook content nuclear localized protein | AT-hook蛋白 AT-hook content nuclear localized protein | AT-hook蛋白 AT-hook content nuclear localized protein | |||||||||||||
| LOC_Os03g17700 | 丝裂原活化蛋白激酶 Mitogen-activated protein kinase | 丝裂原活化蛋白激酶 Mitogen-activated protein kinase | 丝裂原活化蛋白激酶 Mitogen-activated protein kinase | |||||||||||||
| LOC_Os01g66120 | NAC | NAC转录因子 NAC domain transcription factor | NAC转录因子 NAC domain transcription factor | NAC转录因子 NAC domain transcription factor | ||||||||||||
| LOC_Os03g12820 | SRO蛋白基因 Similar to RCD1 gene | SRO蛋白基因 Similar to RCD1 gene | ||||||||||||||
| 候选基因 Candidate gene | 转录因子 Transcription factor | 冷胁迫 Cold | 干旱 Drought | 盐 Salt | ||||||||||||
| LOC_Os03g60560 | C2H2 | TFIIIA型锌指蛋白基因 TFIIIA-type zinc finger protein gene | TFIIIA型锌指蛋白基因 TFIIIA-type zinc finger protein gene | |||||||||||||
| LOC_Os02g33820 | ABA-胁迫-成熟诱导基因 ABA, stress, ripening gene | ABA-胁迫-成熟诱导基因 ABA, stress, ripening gene | ||||||||||||||
| LOC_Os07g39870 | TFIIIA型锌指蛋白基因 TFIIIA-type zinc finger protein gene | TFIIIA型锌指蛋白基因 TFIIIA-type zinc finger protein gene | ||||||||||||||
| LOC_Os06g36670 | ABA受体 Pyrabactin resistance-like abscisic acid receptor | ABA受体 Pyrabactin resistance-like abscisic acid receptor | ||||||||||||||
| LOC_Os06g43660 | 液泡膜质子转运无机焦磷酸酶 H+-pyrophosphatase in tonoplasts | 液泡膜质子转运无机焦磷酸酶 H+-pyrophosphatase in tonoplasts | ||||||||||||||
| LOC_Os07g03710 | 病程相关蛋白 Pathogenesis-related protein 1a | 病程相关蛋白 Pathogenesis-related protein 1a | ||||||||||||||
| LOC_Os02g03410 | 钙依赖性蛋白激酶 Calcium-dependent protein Kinase | 钙依赖性蛋白激酶 Calcium-dependent protein kinase | ||||||||||||||
| LOC_Os03g53200 | 钙调素 Calmodulin | 钙调素 Calmodulin | ||||||||||||||
| LOC_Os08g39450 | A20/AN1型锌指蛋白 A20/AN1 zinc-finger protein | A20/AN1型锌指蛋白 A20/AN1 zinc-finger protein | ||||||||||||||
| LOC_Os01g74410 | MYB | MYB转录因子 MYB family transcription factor | MYB转录因子 MYB family transcription factor | |||||||||||||
| LOC_Os02g52780 | bZIP | bZIP转录因子 bZIP transcription factor | bZIP转录因子 bZIP transcription factor | |||||||||||||
| LOC_Os03g60080 | NAC | 胁迫响应的NAC转录因子 基因 Stress-responsive NAC 1 | 胁迫响应的NAC转录因子 基因 Stress-responsive NAC 1 | |||||||||||||
| LOC_Os02g52780 | bZIP | bZIP转录因子 bZIP transcription factor | bZIP转录因子 bZIP transcription factor | |||||||||||||
| LOC_Os11g08210 | NAC | NAC转录因子 NAC domain transcription factor | NAC转录因子 NAC domain transcription factor | |||||||||||||
| LOC_Os04g50060 | GRAS | GRAS转录因子 GRAS transcription factor gene | GRAS转录因子 GRAS transcription factor gene | |||||||||||||
| LOC_Os01g74410 | MYB | MYB转录因子 MYB family transcription factor | MYB转录因子 MYB family transcription factor | |||||||||||||
| LOC_Os03g53200 | 钙调蛋白 Calmodulin | 钙调蛋白 Calmodulin | ||||||||||||||
| LOC_Os02g50970 | 丝裂原活化蛋白激酶 Mitogen-activated protein kinase | 丝裂原活化蛋白激酶Mitogen-activated protein kinase | ||||||||||||||
| 候选基因 Candidate gene | 转录因子 Transcription factor | 冷胁迫 Cold | 干旱 Drought | 盐 Salt | ||||||||||||
| LOC_Os07g03710 | 病程相关蛋白 Pathogenesis-related protein 1a | 病程相关蛋白 Pathogenesis-related protein 1a | ||||||||||||||
| LOC_Os01g32660 | 丝裂原活化蛋白激酶 Mitogen activated protein kinase | 丝裂原活化蛋白激酶 Mitogen activated protein kinase | ||||||||||||||
| LOC_Os01g73770 | ERF | AP2/EREBP转录因子基因 AP2/EREBP transcription factor gene | AP2/EREBP转录因子基因 AP2/EREBP transcription factor gene | |||||||||||||
新窗口打开|下载CSV
通过GO功能富集分析, 干旱、冷、盐胁迫在分子功能中富集到了与氧化反应相关的氧化还原酶活性(GO:0016491)、过氧化物酶活性(GO:0004601)、氧化还原酶活性对过氧化物作为受体的作用(GO:0016684)、抗氧化活性(GO:0016209)等。在受到干旱、冷、盐胁迫后, 酰胺的生物合成过程(GO:0043604)、细胞酰胺代谢(GO:0043603), 过氧化氢分解代谢过程(GO:0042744)、氧化还原过程(GO:0055114)、过氧化氢代谢过程(GO:0072593)、毒素代谢过程(GO:0009404); 苯丙烷代谢过程(GO:0009698)和木质素代谢过程(GO:00009808)也得到了富集, 也与一些细胞程序性死亡负调节(GO:0043069)和细胞分解代谢过程(GO:0044248)显著富集(表4和附表6)。
Table 4
表4
表4模块GO富集情况(部分)
Table 4
| 分类 Class | GO分类 GO term | 显著富集的term Significantly enriched term | P值 P-value |
|---|---|---|---|
| 分子功能 Molecular function | GO:0005198 | 结构分子活性 Structural molecule activity | 0 |
| GO:0003735 | 核糖体的结构组成 Structural constituent of ribosome | 0 | |
| GO:0016491 | 氧化还原酶活性 Oxidoreductase activity | 5.69E-13 | |
| GO:0004601 | 过氧化物酶活性 Peroxidase activity | 1.07E-11 | |
| GO:0016684 | 氧化还原酶活性对过氧化物作为受体的作用 Oxidoreductase activity, acting on peroxide as acceptor | 3.13E-11 | |
| GO:0016209 | 抗氧化活性 Antioxidant activity | 4.28E-11 | |
| GO:0020037 | 血红素结合 Heme binding | 4.68E-09 | |
| 细胞组分 Cellular component | GO:0022625 | 胞浆大核糖体亚基 Cytosolic large ribosomal subunit | 0 |
| GO:0071944 | 细胞外周 Cell periphery | 0 | |
| GO:0044391 | 核糖体亚基 Ribosomal subunit | 0 | |
| GO:0044445 | 胞质部分 Cytosolic part | 0 | |
| GO:0015934 | 大核糖体亚基 Large ribosomal subunit | 0 | |
| GO:0005840 | 核糖体 Ribosome | 0 | |
| 生物学过程 Biological process | GO:0043604 | 酰胺生物合成过程 Amide biosynthetic process | 0 |
| GO:0043603 | 细胞酰胺代谢过程 Cellular amide metabolic process | 2.22E-16 | |
| GO:0042744 | 过氧化氢分解代谢过程 Hydrogen peroxide catabolic process | 2.34E-10 | |
| GO:0055114 | 氧化还原过程 Oxidation-reduction process | 3.03E-09 | |
| GO:0072593 | 过氧化氢代谢过程 Reactive oxygen species metabolic process | 1.05E-06 | |
| GO:0009698 | 苯丙烷代谢过程 Phenylpropanoid metabolic process | 4.01E-05 | |
| GO:0009808 | 木质素代谢过程 Lignin metabolic process | 6.62E-05 | |
| GO:0006970 | 对渗透压的反应 Response to osmotic stress | 4.15E-04 | |
| GO:0009404 | 毒素代谢过程 Toxin metabolic process | 4.23E-04 | |
| GO:0043069 | 细胞程序性死亡负调节Negative regulation of programmed cell death | 5.87E-04 | |
| GO:0044248 | 细胞分解代谢过程 Cellular catabolic process | 0.00136054 |
新窗口打开|下载CSV
以已报道的对3种胁迫都具有调控作用的OsAHL1、DSM1和ONAC095为核心基因, 构建共表达网络, 利用Cytoscape对网络进行可视化。其中LOC_Os02g30900编码的蛋白激酶结构域含蛋白质是NAC转录因子(LOC_Os01g66120)和丝裂原活化蛋白激酶(LOC_Os03g17700)的枢纽基因(图6)。对这些预测到的枢纽基因在玉米数据库中进行检索, 得到同源基因, 并进行了功能注释(表5)。
图6
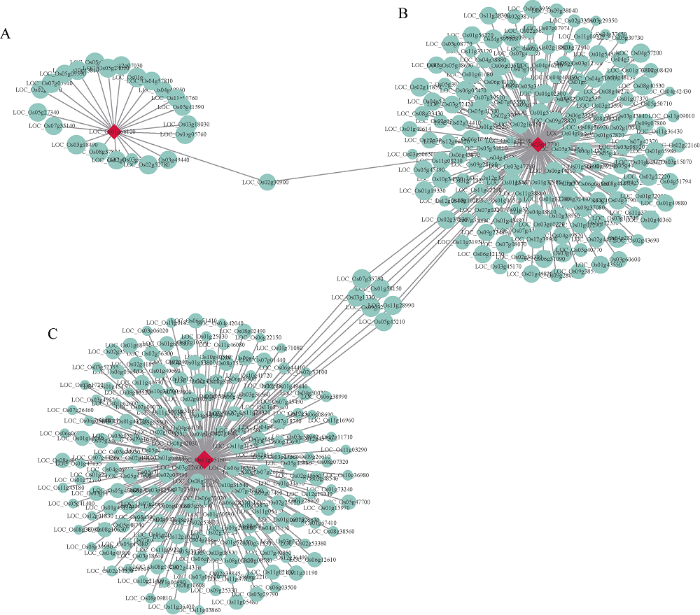 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6Green模块中3种胁迫下共有基因的共表达网络
图中红色表示同时参与3种胁迫响应的基因, 蓝色表示共表达基因, 点的大小表示该基因的权重值。A: 冷胁迫下的共表达网络; B: 干旱胁迫下的共表达网络; C: 盐胁迫下的共表达网络。
Fig. 6Co-expression network of shared genes under three stresses in green module
The red color in the figure indicates the genes involved in the three stress responses simultaneously, the blue color indicates the co-expressed gene, and the size of the dot indicates the weight value of the gene. A: the co-expression network under cold stress; B: the co-expression network under drought stress; C: the co-expression network under salt stress.
Table 5
表5
表5候选基因功能注释
Table 5
| 候选基因 Candidate gene | 候选基因在水稻中注释 Annotation of candidate genes in rice | 候选基因在玉米同源基因 Homology gene in maize | 基因功能 Gene function |
|---|---|---|---|
| LOC_Os02g30900 | 激酶结构域蛋白 Protein kinase domain containing protein | GRMZM2G137468 | 蛋白激酶APK1A Protein kinase APK1A |
| LOC_Os11g28990 | TBC结构域蛋白质 TBC domain containing protein | GRMZM2G122935 | 含有RabGAP/TBC结构域的蛋白质 RabGAP/TBC domain-containing protein |
| LOC_Os09g32040 | 无顶端分生组织蛋白 No apical meristem protein | GRMZM2G004531 | N-乙酰-L-蛋氨酸/N-乙酰-L-甲硫氨酸 Nam 7 |
| LOC_Os07g35750 | DUF26激酶 DUF26 kinases | GRMZM2G075438 | 受体激酶RK20-1 Receptor kinase RK20-1 |
| LOC_Os05g45210 | 呼吸爆发氧化酶 Respiratory burst oxidase | GRMZM2G043435 | 呼吸爆发氧化酶C Respiratory burst oxidase C |
| 候选基因 Candidate gene | 候选基因在水稻中注释 Annotation of candidate genes in rice | 候选基因在玉米同源基因 Homology gene in maize | 基因功能 Gene function |
| LOC_Os01g58150 | 表达蛋白质 Expressed protein | GRMZM2G139460 | 功能未知的保守基因 Conserved gene of unknown function |
| LOC_Os03g13300 | 谷氨酸脱羧酶 Glutamate decarboxylase | GRMZM2G098875 | 谷氨酸脱羧酶 Glutamate decarboxylase |
新窗口打开|下载CSV
3 讨论
本研究通过对干旱、冷、盐胁迫不同处理下47个不同材料建立共表达网络, 根据聚类情况划分为15个模块, 对已克隆报道的水稻数据库中与干旱、冷、盐胁迫相关基因进行统计, 并与划分的模块进行比较, 发现各个模块都能得到与各胁迫相关的基因。在对每个胁迫下存在的基因数目最多的2个模块进行了GO功能富集分析, 各个模块都得到了具有相关生物学意义的功能富集结果, 并且发现不同的胁迫下寄主的响应也有所不同。如与干旱胁迫相关的lightgreen模块中的渗透胁迫响应(GO: 0009737)、对水的响应(GO:0006970), 与冷胁迫相关的茉莉酸介导的信号转导通路的调控(GO:2000022), darkmagenta模块中的对温度刺激响应(GO:0009266)通路等; 盐胁迫相关的green模块中的盐胁迫响应(GO:0009651), 这些富集到的功能与样品的处理方式生物功能基本吻合, 证明了WGCNA方法在对大数据分析时具有较高的可靠性。在对模块green中3种胁迫下各自特有的基因进行富集也表现出功能上的明显不同, 其中对冷胁迫的响应较敏感。对共表达网络中的核心基因分析发现, 在darkmagenta中, OsPDS (LOC_Os03g08570)编码的八氢番茄红素脱氢酶, 在水稻中报道为对干旱敏感性增加, 而耐冷性和氧化胁迫性增强[31]。OsCOIN (LOC_ Os01g01420)编码的锌指蛋白, 过量表达可增强水稻对冷、盐和干旱的抗性, 对冷胁迫较为敏感[32]。在lightgreen中, 处于表达网络中心的OsMSR15 (LOC_ Os03g41390)是一类具有转录激活活性锌指蛋白, 在冷、干旱、盐胁迫中均能强烈响应, 是植物响应干旱的重要调节因子[33]。SRWD1 (LOC_Os08g 38880)编码盐应答WD40蛋白, 受盐胁迫调节, 具有多种调节模式[34]。对预测的连通性较高的基因进行注释发现, LOC_Os01g01420在水稻中功能尚不明确, 拟南芥中注释为编码过氧化氢酶, 参与多重胁迫反应[35]。对green模块分析和共有的基因富集分析中发现, 酰胺生物合成、细胞酰胺代谢被显著富集, 而在拟南芥中, IAA酰胺合成酶GH3-6负调控干旱和盐胁迫的反应[36]。苯丙烷代谢中的木质素生物合成在非生物胁迫下被诱导, 通过积累保护寄主细胞[37]。在构建的共表达网络中, 处于枢纽位置的基因被注释到了DUF26激酶, 该激酶在拟南芥中被报道参与防御和程序化细胞的调节[38]。本研究对干旱、冷、盐这3种胁迫下共同存在的调控机制进行了分析, 但鉴定到的结果都是直接或间接地参与胁迫调控, 需进一步挖掘和验证这些基因的具体生物功能。总之, 利用WGCNA共表达网络对干旱、冷、盐胁迫相关功能基因进行挖掘, 对水稻抗逆的研究具有重要的意义。
4 结论
通过对冷、干旱、盐胁迫数据的共表达网络分析, 得到了15个模块。对这3种胁迫下各自的生物学意义进行了描述, 对参与2种或3种胁迫的基因进行了鉴定, 构建了具有生物学意义的共表达网络。本研究结果可为后续的水稻多抗逆性和综合抗逆性相关的调控机制研究提供重要的参考依据。附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical-zuowxb. aspx。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
