 ,1,2,*
,1,2,*Identification and function analysis of AVP1, VHA-a2, and VHA-a3 genes in Brassica napus L.
YAO Jun-Yue1,2, HUA Ying-Peng1,2, ZHOU Ting1,2, WANG Tao1,2, SONG Hai-Xing1,2, GUAN Chun-Yun3, ZHANG Zhen-Hua ,1,2,*
,1,2,*通讯作者:
收稿日期:2018-09-20接受日期:2019-04-15网络出版日期:2019-04-30
| 基金资助: |
Received:2018-09-20Accepted:2019-04-15Online:2019-04-30
| Fund supported: |
作者简介 About authors
E-mail:yjy950606@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (3126KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
姚珺玥, 华营鹏, 周婷, 王涛, 宋海星, 官春云, 张振华. 甘蓝型油菜AVP1、VHA-a2和VHA-a3基因的鉴定及功能性研究[J]. 作物学报, 2019, 45(8): 1146-1157. doi:10.3724/SP.J.1006.2019.84124
YAO Jun-Yue, HUA Ying-Peng, ZHOU Ting, WANG Tao, SONG Hai-Xing, GUAN Chun-Yun, ZHANG Zhen-Hua.
植物的生长和发育离不开对必需营养元素的吸收及代谢产物的转运, 植物通过调控转运蛋白的活性来感知并应对不断变化的营养水平和环境压力[1]。这一基本调节过程需要大量的能量来维持平衡, 而ATP水解所形成的能量在这一过程中起着重要的作用, 如果没有主要的质子动力来激活耦合的载体或离子通道, 所有的运输和代谢活动会受到抑制甚至停止。
在植物体中存在着3种不同的质子泵, 即定位在细胞质膜上的P型质子泵(H+-ATPase)、液泡膜上利用焦磷酸水解释放能量驱动的H+-PPase (V-H+-PPase)和广泛存在于内膜系统的液泡型质子泵(V-H+-ATPase), 3种类型的质子泵协同调控着细胞内各区室间pH的动态平衡, 对细胞内的物质代谢及维持其他转运蛋白的正常运转等具有重要的作用[1]。H+-PPase和H+-ATPase是作用于细胞液泡膜上进行H+代谢的主要作用酶, 两者都是将细胞质内的H+转运到液泡内, 酸化液泡内的许多离子(如Ca2+等), 为代谢物的生成提供能量, 由此产生的离子梯度形成渗透势差, 影响多种细胞功能[2]。液泡H+-ATP酶(V-H+-ATPase)和液泡H+-PPase (V-H+-PPase)都是高度丰富的液泡蛋白, 所有液泡功能都需要穿过液泡膜, 处理代谢过程中产生的大量质子, 这一过程被认为是由这2个质子泵组合活动形成的质子梯度和膜电位效应调控[3]。1992年, 在拟南芥中克隆出完整的H+-PPase的cDNA序列, 命名为AVP1, 目前已经确定了3个基因编码H+-PPase的同源物(AVP1~AVP3)[4]。2002年, 在拟南芥基因组中, 鉴定出至少26个编码液泡型H+-ATPase亚单位的基因, 并命名这些基因为VHA-X, X代表每个亚单位的字母代码, 定位于膜上的为小写字母。参与植物主要活性运输的基因被命名为VHA-a, 其中VHA-a2和VHA-a3定位于液泡膜上[1]。到目前为止, 在很多植物中已经克隆出完整的H+-PPase cDNA序列[5], 但是在甘蓝型油菜中却少见报道。V-ATPase定位于分泌系统的液泡膜、内质网(ER)膜、高尔基体膜、小囊泡以及质膜, 且V-ATPase复合物在所有真核生物中都是保守的[3]。3种质子泵中, 液泡型H+-ATP酶功能和结构最为复杂。
甘蓝型油菜(Brassica napus L.)是由白菜(B. rapa)和甘蓝(B. oleracea)远缘杂交形成的异源四倍体作物, 全基因组的编码基因数量庞大, 其进化、遗传的复杂性在一定程度上影响了各种功能基因在甘蓝型油菜生长和发育中的作用[6]。本研究利用拟南芥AVP1、VHA-a2和VHA-a3基因序列, 在甘蓝型油菜全基因组数据中鉴定其基因序列, 通过生物信息学方法研究甘蓝型油菜AVP1、VHA-a2和VHA-a3基因的分子特征、序列信息、保守基序特征、染色体定位、系统进化关系、蛋白质的结构, 旨在系统研究甘蓝型油菜AVP1、VHA-a2和VHA-a3基因的遗传信息、编码的蛋白结构, 为进一步研究该家族基因的生物学功能奠定理论基础。
1 材料与方法
1.1 试验材料
拟南芥AVP1、VHA-a2和VHA-a3蛋白氨基酸序列下载自美国国立生物信息中心(NCBI)数据库, 以AtAVP1、AtVHA-a2和AtVHA-a3为比对序列, 对NCBI收录的甘蓝型油菜基因组数据库进行BLASTN[7]搜索, 下载筛选到BnaAVP1、BnaVHA-a2和BnaVHA-a3基因编码的氨基酸序列。水培实验所用的甘蓝型油菜品种为湘油15。
1.2 试验方法
1.2.1 甘蓝型油菜AVP1、VHA-a2和VHA-a3基因的鉴定 首先以AtAVP1、AtVHA-a2和AtVHA-a3为原始序列, 使用在线工具Pfam[8] (http://pfam.xfam. org/)和SMART [9] (http://smart.em-bl-heidelberg.de/)分析AtAVP1、AtVHA-a2和AtVHA-a3序列, 分析结果显示, AtAVP1、AtVHA-a2和AtVHA-a3具有的跨膜结构域; 其次, 下载甘蓝型油菜、白菜、甘蓝全基因组数据(http://brassicadb.org/brad/), 并利用Bio Edit软件构建本地数据库, 以AtAVP1、AtVHA-a2和AtVHA-a3的Mem-trans为检索序列, 设定检索参数为E值<e-6, 在构建的本地数据库中进行BLASTN检索, 手动删除<50个氨基酸和氨基酸同源性<35%的序列, 将所有鉴定的甘蓝型油菜、白菜、甘蓝AVP1、VHA-a2和VHA-a3基因的编码氨基酸序列置于Pfam和SMART工具中, 检验候选序列是否具有跨膜结构域; 最后, 运用NCBI数据库(http://www. ncbi.nlm.nih.gov/)中BLASTN工具命名符合上述条件的基因序列。1.2.2 甘蓝型油菜AVP1、VHA-a2和VHA-a3蛋白的分子特性分析 采用ExPASy ProtoParam (https://web.expasy.org/protparam/)[10]在线软件预测甘蓝型油菜AVP1、VHA-a2和VHA-a3蛋白的氨基酸数目和组成、相对分子量(MW)、理论等电点(pI)、蛋白质的亲水性(GRAVY)及稳定性等理化性质。
1.2.3 甘蓝型油菜AVP1、VHA-a2和VHA-a3蛋白的进化选择压力分析 为了研究AVP1、VHA-a2和VHA-a3蛋白在物种进化过程中是否涉及达尔文的选择, 对甘蓝型油菜中AVP1、VHA-a2和VHA-a3的CDS序列和蛋白序列进行比对, 利用在线软件PAL2NAL (http://www.bork.embl.de/pal2nal/)计算核苷酸的同义突变频率(synonymous, Ks)和非同义突变频率(non-synonymous, Ka), 并计算非同义突变频率与同义突变频率的比值即Ka/Ks。
1.2.4 保守结构域和保守残基的鉴定 使用SMART (http://smart.emblheidelberg.de/)[9]鉴定保守结构域, 并使用Pfam (http://pfam.xfam.org/)注释[8]。使用Vector NTI对保守残基进行多重比对分析。
1.2.5 甘蓝型油菜AVP1、VHA-a2和VHA-a3蛋白保守基序分布信息获取 运用MEME Version 4.9.1 (http://meme.nbcr.net/meme/cgi-bin/meme.cgi)[11]在线工具分析甘蓝型油菜AVP1、VHA-a2和VHA-a3蛋白的保守基序信息, 设置最大基序检索值为10。
1.2.6 AVP1、VHA-a2和VHA-a3的系统进化树构建 运用Clustal X version 2.1[12]软件对甘蓝型油菜、拟南芥、甘蓝、白菜以及部分其他物种的AVP1、VHA-a2和VHA-a3蛋白的氨基酸序列进行多重比对, 使用 MEGA6.0[13]软件分析比对结果并采用邻接法(Neighbor-Joining, NJ)构建系统进化树, 设定泊松校正法计算进化距离和进行重复1000次的自展法(bootstrap)检验。
1.2.7 甘蓝型油菜AVP1、VHA-a2和VHA-a3基因染色体定位分析 选取甘蓝型油菜全基因组数据库(http://brassicadb.org/brad/)用于甘蓝型油菜AVP1、VHA-a2和VHA-a3基因的筛选。利用Pfam[8] (http://pfam.xfam.org/)对筛选到的AVP1、VHA-a2和VHA-a3基因进行结构域判定, 去除冗余基因, 确定位置信息, 利用Circos 0.6[14]构建AVP1、VHA-a2和VHA-a3基因染色体分布图谱。
1.2.8 甘蓝型油菜AVP1、VHA-a2和VHA-a3蛋白的二级结构分析及三维结构预测 使用GOR4 (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)[15]和PSIPRED (http://bioinf.cs. ucl.ac.uk/web_servers/)[16-17] 2种不同的工具分析二级结构。使用Phyre2 (http://www.sbg.bio.ic.ac.uk/~ phyre2/html/)[18]预测BnaAVP1、BnaVHA-a2和BnaVHA-a3蛋白的三维结构。
1.2.9 营养液培养实验 油菜苗期营养液培养试验在湖南农业大学植物营养与养分高效利用课题组光照培养室中进行。光照培养室的温度设置为22oC, 光照周期为14 h (光照)/10 h (黑暗), 光照强度为300~320 μmol m-2 s-1, 湿度为60%~75%。选取大小一致的油菜种子, 使用1% NaClO灭菌处理10 min, 将种子表面冲洗干净后, 于4oC下用灭菌超纯水(>18.25 MΩ)浸泡24 h。将浸泡过后的种子均匀播种到塑料育苗盘表面固定的纱布上, 育苗盘中加适量超纯水。育苗1周后, 将长势一致的幼苗移栽至盛有10 L营养液的黑色塑料盆中, 营养液为1/4浓度采用Hoagland营养液[19]。
1.2.10 低氮处理高通量转录组测序试验 油菜幼苗先在正常氮条件下培养10 d, 然后转至0.3 mmol L-1 NO3-培养, 0、3和72 h后用离心管取样, 样品先装入液氮罐中速冻, 后移入-80oC冰箱保存待测, 每个处理3个独立的生物学重复。测序平台为 Illumina Hiseq 2000[20], 其产生6.0 Gb测序数据, 每个样品具有150 bp配对末端(PE)读数, 转录组丰度(FPKM值)由RNA-seq数据确定[21]。使用(Mev, http://www.tm4.org/mev.html)[22]绘制基因表达谱的热图, 使用错误发现率(FDR)≤0.05和log2(倍数变化)≥1作为鉴定差异表达基因的阈值。
1.2.11 低磷处理高通量转录组测序试验 油菜幼苗先在250 μmol L-1 Pi (KH2PO4)营养液中培养10 d, 再将油菜样品分成2组, 一组继续在正常营养液中培养, 另一组转移至无磷的营养液中培养5 d, 分别对2组样品的地上部(S)和根(R)取样, 将样品先装入液氮罐中速冻, 后移入-80oC冰箱保存待测, 每个处理3个独立的生物学重复。测序平台为Illumina Hiseq 2000[20], 其产生6.0 Gb测序数据, 每个样品具有150 bp配对末端(PE)读数, 转录组丰度(FPKM值)由RNA-seq数据确定[21]。使用(Mev, http://www.tm4. org/mev.html)[22]绘制基因表达谱的热图, 使用错误发现率(FDR)≤0.05和log2(倍数变化)≥1作为鉴定差异表达基因的阈值。
2 结果与分析
2.1 十字花科作物AVP1、VHA-a2和VHA-a3基因的拷贝数变异
基因的拷贝数与基因组大小密切相关, 为了比较十字花科作物AVP1、VHA-a2和VHA-a3基因的进化多样性, 以AtAVP1、AtVHA-a2和AtVHA-a3的氨基酸序列为查询对象, 对NCBI数据库进行BLASTN搜索, 在异源四倍体甘蓝型油菜中鉴定了9个BnaAVP1、3个BnaVHA-a2和4个BnaVHA-a3, 且基因数量明显多于甘蓝和白菜(图1)。可能是由于甘蓝型油菜是由白菜和甘蓝远缘杂交形成的异源四倍体作物, 其进化和遗传远比白菜和甘蓝复杂[6]。图1
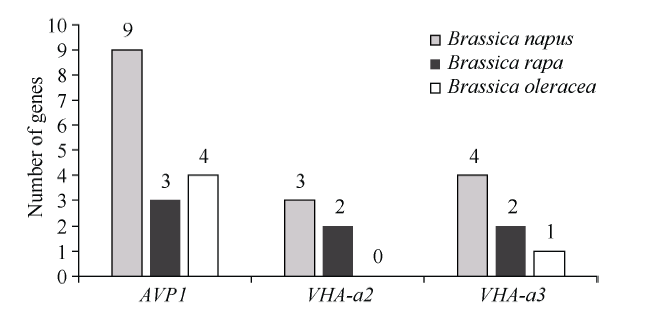 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1甘蓝型油菜、甘蓝、白菜AVP1、VHA-a2和VHA-a3基因的拷贝数变异
柱状图顶部数值为该物种拷贝的基因数目。
Fig. 1Copy number variation of AVP1, VHA-a2, and VHA-a3 genes in Brassica napus, B. rapa, and B. olerace
The number at the top of the histogram is the number of genes copied for that species.
2.2 甘蓝型油菜AVP1、VHA-a2和VHA-a3基因分子特征
由表1可知, BnaAVP1基因编码区长度在1821 bp (BnaAVP1-2)~3525 bp (BnaAVP1-3)之间, 编码606~1174个氨基酸, 预测蛋白质的理论分子量为64.61~126.46 kD; 理论等电点(pI)为5.01~6.29, 为酸性蛋白; BnaVHA-a2基因编码区长度平均为821, 编码823个氨基酸, 预测其分子量是92.96 kDa, 理论等电点(pI)为5.61左右, 为酸性蛋白; BnaVHA-a3基因编码区长度在2454 bp (BnaVHA-a3-4)~2463 bp (BnaVHA-a3-1)之间, 编码817~820个氨基酸, 预测蛋白质的理论分子量为92.32~92.85 kD; 理论等电点(pI)为5.66~5.86, 为酸性蛋白。根据亲水性指数介于-0.5~0.5为两性蛋白(GRAVY为负值表示亲水性, 正值表示疏水性)的原则[23], 对于BnaAVP1成员, 仅有BnaAVP1-2和BnaAVP1-3是两性蛋白外, 其余均为疏水性蛋白, 而BnaVHA-a2和BnaVHA-a3蛋白均为两性蛋白。Table 1
表1
表1甘蓝型油菜AVP1、VHA-a2和VHA-a3基因成员信息
Table 1
| 基因名称 Gene name | 基因库编号 Database No. | 基因编号 Gene ID | 基因长度 CDS (bp) | 氨基酸长度 Amino acids (aa) | 分子量 MW (kD) | 理论等电点 pI | 亲水性指数 GRAVY |
|---|---|---|---|---|---|---|---|
| BnaAVP1-1 | GSBRNA2T00010424001 | BnaA09g56670D | 2409 | 802 | 83.97 | 5.06 | 0.620 |
| BnaAVP1-2 | GSBRNA2T00033628001 | BnaA06g38620D | 1821 | 606 | 64.61 | 5.00 | 0.281 |
| BnaAVP1-3 | GSBRNA2T00136659001 | BnaC08g38730D | 3525 | 1174 | 126.46 | 6.29 | 0.269 |
| BnaAVP1-4 | GSBRNA2T00139755001 | BnaC08g16730D | 2310 | 769 | 80.59 | 5.03 | 0.611 |
| BnaAVP1-5 | GSBRNA2T00068861001 | BnaC06g40530D | 2310 | 769 | 80.61 | 5.00 | 0.574 |
| BnaAVP1-6 | GSBRNA2T00054245001 | BnaC05g11930D | 2316 | 771 | 80.83 | 5.08 | 0.618 |
| BnaAVP1-7 | GSBRNA2T00103071001 | BnaA08g23780D | 2310 | 769 | 80.52 | 5.03 | 0.616 |
| BnaAVP1-8 | GSBRNA2T00146278001 | BnaA07g35600D | 2310 | 769 | 80.70 | 5.01 | 0.572 |
| BnaAVP1-9 | GSBRNA2T00062272001 | BnaA06g10340D | 2316 | 771 | 80.83 | 5.08 | 0.621 |
| BnaVHA-a2-1 | GSBRNA2T00137010001 | BnaC08g35720D | 2466 | 821 | 92.97 | 5.61 | 0.060 |
| BnaVHA-a2-2 | GSBRNA2T00108093001 | BnaC04g33510D | 2472 | 823 | 92.96 | 5.61 | 0.056 |
| BnaVHA-a2-3 | GSBRNA2T00090255001 | BnaA04g12160D | 2460 | 819 | 92.80 | 5.56 | 0.059 |
| BnaVHA-a3-1 | GSBRNA2T00061106001 | BnaC03g60340D | 2463 | 820 | 92.85 | 5.86 | 0.041 |
| BnaVHA-a3-2 | GSBRNA2T00075109001 | BnaC01g00150D | 2457 | 818 | 92.50 | 5.80 | 0.080 |
| BnaVHA-a3-3 | GSBRNA2T00150888001 | BnaA08g16770D | 2460 | 819 | 92.79 | 5.75 | 0.043 |
| BnaVHA-a3-4 | GSBRNA2T00073949001 | BnaA01g05300D | 2454 | 817 | 92.32 | 5.66 | 0.071 |
新窗口打开|下载CSV
2.3 甘蓝型油菜AVP1、VHA-a2和VHA-a3蛋白的进化选择压力分析
在遗传学中认为, Ka/Ks的比例是判断这个基因在进化中是否受到选择压力的一个指标。当Ka/Ks>1.0时, 认为有正选择效应; 当Ka/Ks=1.0时, 认为该蛋白存在中性选择; 当Ka/Ks<1.0时, 则认为存在纯化选择作用[24]。由图2可知, AVP1、VHA-a2和VHA-a3在甘蓝型油菜中的Ka/Ks值均小于1.0, 表明这3种蛋白在物种进化过程中为保留其重要功能而存在纯化选择作用。图2
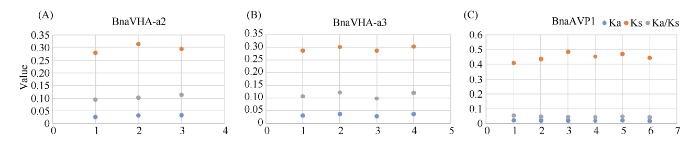 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2甘蓝型油菜中BnaAVP1、BnaVHA-a2和BnaVHA-a3蛋白的同义突变频率和非同义突变频率
Fig. 2Synonymous nucleotide substitution rates (Ks) and non-synonymous nucleotide substitution rates (Ka) of the BnaAVP1, BnaVHA-a2, and BnaVHA-a3 proteins in Brassica napus L.
2.4 甘蓝型油菜AVP1、VHA-a2和VHA-a3基因的染色体定位
甘蓝型油菜AVP1、VHA-a2和VHA-a3基因染色体定位分析发现, 3个基因的成员分布于甘蓝型油菜基因组A/C亚基因组上, 但不是均匀分布。AVP1的9个成员中, BnaAVP1-2和BnaAVP1-9位于A6染色体上, BnaAVP1-8位于A7染色体上, BnaAVP1-7位于A8染色体上, BnaAVP1-1位于A9染色体上, BnaAVP1-6位于C5染色体上, BnaAVP1-5位于C6染色体上, BnaAVP1-4和BnaAVP1-3位于C8染色体上。VHA-a2的3个成员中, BnaVHA-a2-3位于A4染色体上, BnaVHA-a2-2位于C4染色体上, BnaVHA- a2-1位于C8染色体上。VHA-a2的4个成员中, BnaVHA-a3-4位于A1染色体上, BnaVHA-a3-3位于A8染色体上, BnaVHA-a3-2位于C1染色体上, BnaVHA-a3-1位于C3染色体上(图3)。图3
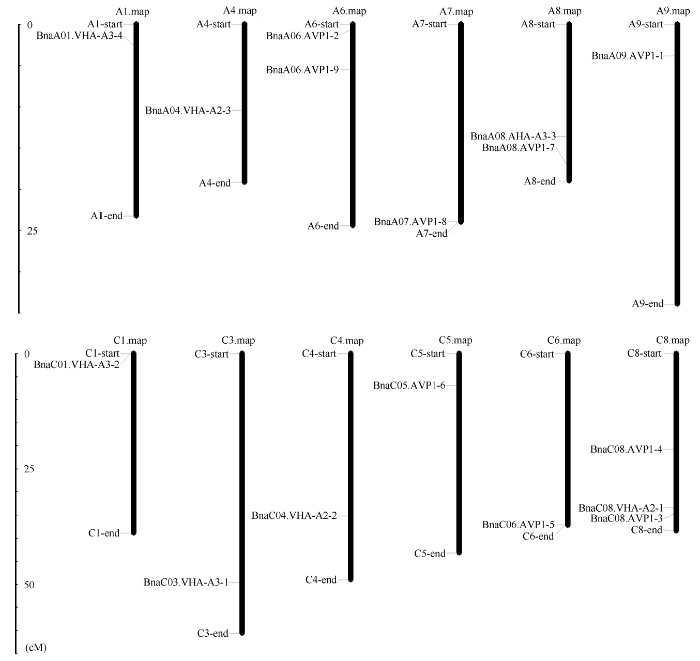 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3甘蓝型油菜AVP1、VHA-a2和VHA-a3基因染色体定位
Fig. 3Chromosome localization of the BnaAVP1s, BnaVHA-a2s, and BnaVHA-a3s of Brassica napus
2.5 甘蓝型油菜、拟南芥、白菜以及甘蓝的AVP1、VHA-a2和VHA-a3蛋白的保守基序分布
保守基序分析发现甘蓝型油菜AVP1蛋白的N末端具有保守基序数1 (Motif 1), 而VHA-a2和VHA-a3则具有相同的N末端保守蛋白和保守基序数。对于甘蓝型油菜AVP1蛋白, 其保守基序数量变化范围为1~10, 具有一定规律: 除了BnaAVP1-2外, 其他系列都含有10个基序, 此外BnaAVP1-3的Motif8所在保守序列的位置不同于其他蛋白。此外, 研究还发现BraAVP1-3的保守基序数8缺失。对于甘蓝型油菜蛋白VHA-a2和VHA-a3, 均有相同的N末端保守蛋白和保守基序数(图4)。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4甘蓝型油菜、拟南芥、白菜以及甘蓝的AVP1、VHA-a2和VHA-a3蛋白家族成员的保守基序分布
Fig. 4Distribution of conserved motifs in BnaAVP1s, BnaVHA-a2s, and BnaVHA-a3s of Brassica napus
2.6 甘蓝型油菜、拟南芥、白菜以及甘蓝的AVP1、VHA-a2和VHA-a3基因外显子-内含子结构的比对分析
通过将编码序列与芸薹属作物中相应基因组序列的比对来确定AVP1、VHA-a2和VHA-a3基因的外显子-内含子结构, 由图5可知, 芸薹属作物中AVP1基因的内含子-外显子结构与拟南芥中的同源物相似, 大多数AVP1基因被6~10个内含子间隔; VHA-a2和VHA-a3基因的外显子-内含子结构与拟南芥中的同源物也相似, 均含有17个内含子。图5
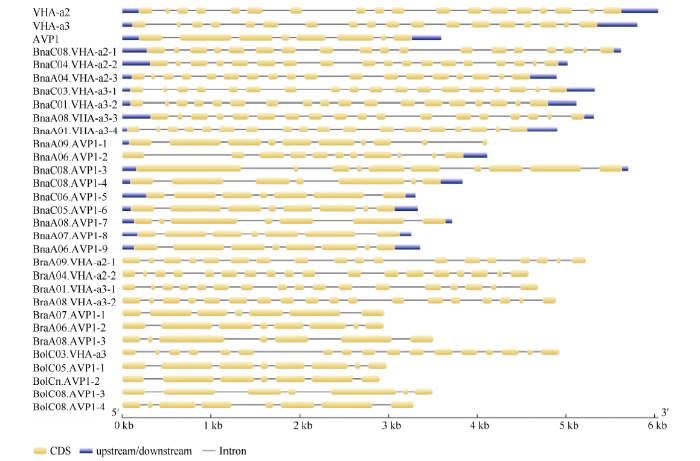 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5甘蓝型油菜、拟南芥、白菜以及甘蓝的AVP1、VHA-a2和VHA-a3基因外显子-内含子结构
Fig. 5Exon-intron structure of BnaAVP1, BnaVHA-a2, and BnaVHA-a3 of Brassica napus
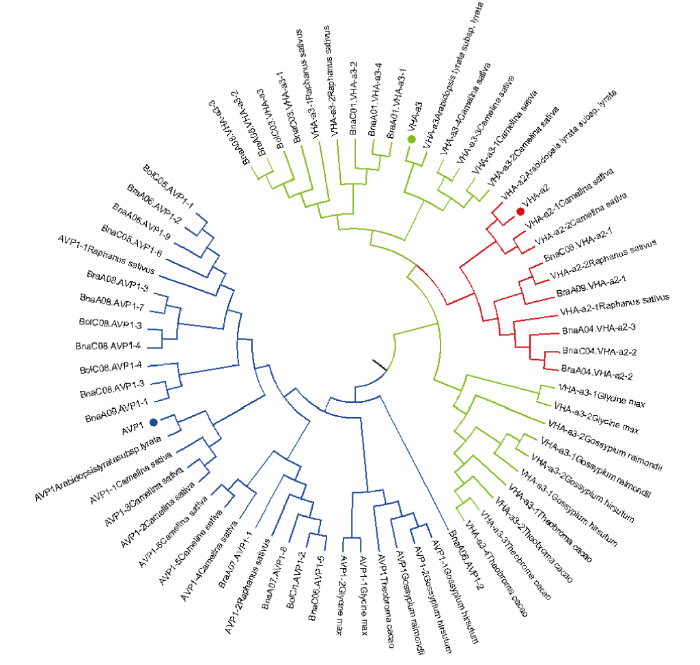
2.7 AVP1、VHA-a2和VHA-a3蛋白的系统进化分析
为确定不同物种中AVP1、VHA-a2和VHA-a3蛋白质之间的分子进化和系统发育关系, 基于来自14种植物的蛋白质序列构建了无根系统发育树(图6)。结果表明, 对于BnaAVP1的成员, BnaAVP1-1和BnaAVP1-3、BnaAVP1-4、BnaAVP1-5分别与BolAVP1-4、BolAVP1-3、BolAVP1-2关系紧密, BnaAVP1-6和BnaAVP1-9与BolAVP1-1和BraAVP1-2关系紧密, BnaAVP1-7与BraAVP1-3关系紧密, BnaAVP1-8与BraAVP1-1关系紧密, 而BnaAVP1-2则与其他成员关系不紧密。对于BnaVHA-a2成员, BnaVHA-a2-1、BnaVHA-a2-2和BnaVHA-a2-3与BraVHA-a2-1、BraVHA-a2-2关系紧密。BnaVHA-a3成员中, BnaVHA-a3-1和BolVHA-a3关系紧密, BnaVHA-a3-3与BraVHA-a3-2, 而BnaVHA-a3-2、BnaVHA-a3-4与BraVHA-a3-1关系紧密, 说明甘蓝型油菜VHA-a2和VHA-a3蛋白更多地遗传于白菜。图 6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图 6甘蓝型油菜BnaAVP1、BnaVHA-a2和BnaVHA-a3蛋白的进化分析
蓝色代表BnaAVP1家族, 绿色代表BnaVHA-a3家族, 红色代表BnaVHA-a2家族。
Fig. 6Evolutionary relationships of BnaAVP1, BnaVHA-a2, and BnaVHA-a3 proteins of Brassica napus
Blue represents the BnaAVP1 family, green represents the BnaVHA-a3 family, and red represents the BnaVHA-a2 family.
2.8 甘蓝型油菜AVP1、VHA-a2和VHA-a3 蛋白二级结构分析和三级结构预测
使用GOR4和SPIPRED进行BnaVHA-a2蛋白的二级结构分析, GOR4和SPIPRED预测结构类似(图7)。运用Phyre2数据库、分析的保守氨基酸残基和AVP1、VHA-a2和VHA-a3的蛋白二级结构对BnaAVP1、BnaVHA-a2和BnaVHA-a3蛋白进行三级结构预测(图8)。由BnaVHA-a2-1蛋白二级结构和BnaVHA-a2-1、BnaVHA-a3-1、BnaAVP1-1蛋白三级预测结构可知, α螺旋是BnaAVP1s、BnaVHA-a2s和BnaVHA-a3s 蛋白的主要结构, BnaVHA-a2s和BnaVHA-a3s蛋白三级结构相似, 与BnaAVP1s结构有较大差异, 这可能导致了其功能的差异性。图7
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7甘蓝型油菜BnaVHA-a2-1蛋白二级结构分析
GOR4 (A)和PSIPRED (B)分别是进行二级结构分析的2种不同的工具。
Fig. 7Secondary structure analysis of BnaVHA-a2-1 protein
GOR4 (A) and PSIPRED (B) are two tools for the secondary structure analysis.
图8
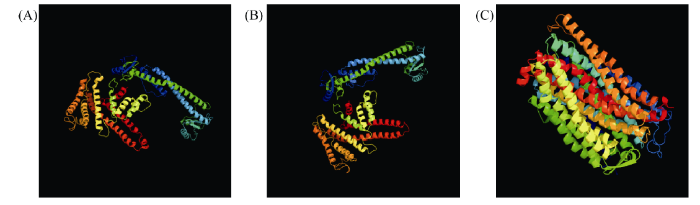 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8甘蓝型油菜BnaAVP1、BnaVHA-a2和BnaVHA-a3蛋白三级结构预测
Fig. 8Prediction of three-dimensional structure of BnaAVP1, BnaVHA-a2, and BnaVHA-a3 protein
A: BnaVHA-a2-1; B: BnaVHA-a3-1; C: BnaAVP1-1.
2.9 甘蓝型油菜AVP1、VHA-a2和VHA-a3基因在低氮条件下的表达分析
氮元素是蛋白合成的基本元素, 也是植物生长发育所必需的营养元素。为了确定AVP1、VHA-a2和VHA-a3对低氮是否存在响应, 本研究对短期(3 h)和长期(72 h)低氮培养的甘蓝型油菜地上部和根进行了高通量转录组测序。结果表明, BnaAVP1s基因主要在地上部表达, 其中BnaC05g11930D和BnaA06g10340D在根部和地上部均有表达, BnaAVP1s在低氮处理3 h后地上部表达下调, 在低氮处理72 h后根中表达上调, 部分有显著差异(图9-C)。BnaVHA-a2s和BnaVHA-a3s基因在地上部和根中均有表达, BnaVHA-a2s在低氮处理72 h后表达量基本呈上调趋势, BnaC04g33510D达到显著差异(图9-A), BnaVHA-a3s在低氮3 h后基本呈下调趋势, 少数达到显著差异(图9-B)。图9
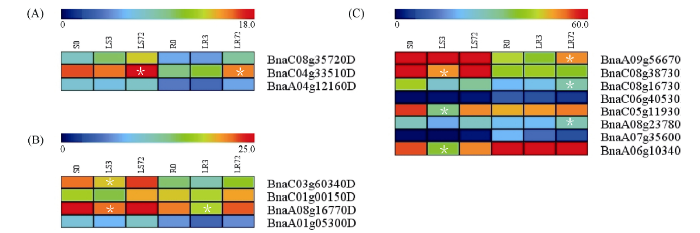 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9在低氮条件下甘蓝型油菜BnaAVP1、BnaVHA-a2和BnaVHA-a3家族的转录组分析。
S: 地上部; R: 根; 0、3、72 (h)代表缺氮胁迫的时间。图中结果表示3次生物学重复的平均值。*代表有显著差异。
Fig. 9Transcriptome analysis of BnaAVP1, BnaVHA-a2, and BnaVHA-a3 family genes in Brassica napus
S: shoot; R: root; 0, 3, and 72 (h) represent the time course of nitrogen deficiency. The results represent the average of three biological replicates. * represent significant differences. (A): BnaVHA-a2s; (B): BnaVHA-a3s; (C): BnaAVP1s.
2.10 甘蓝型油菜AVP1、VHA-a2和VHA-a3基因在低磷条件下的表达分析
磷元素是植物生长发育所必需的营养元素。为了确定AVP1、VHA-a2和VHA-a3对低磷是否存在响应, 本研究对低磷培养的甘蓝型油菜地上部和根进行了高通量转录组测序。结果表明, 低磷处理后, BnaAVP1s基因在根中大部分表达上调, 而在地上部基因表达基本无差异(图10-C)。BnaVHA-a2s在低磷处理后基本无差异(图10-A)。BnaVHA-a3s基因在地上部和根中均有表达, 在低磷处理后表达上调较多, 且部分达到显著差异(图10-B)。图10
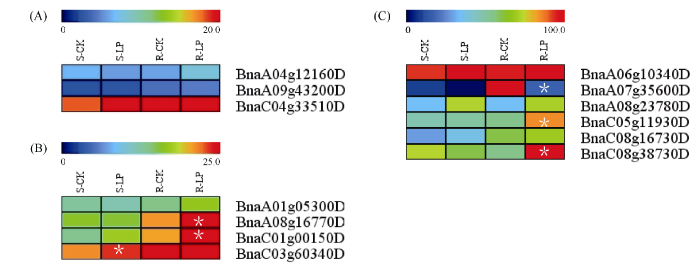 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图10在低磷条件下甘蓝型油菜BnaAVP1、BnaVHA-a2和BnaVHA-a3家族的转录组分析
S: 地上部; R: 根。CK代表正常条件, LP代表低磷胁迫。图中结果表示3次生物学重复的平均值。*代表有显著差异。
Fig. 10Transcriptome analysis of BnaAVP1, BnaVHA-a2, and BnaVHA-a3 family genes in Brassica napus
S: shoot; R: root; CK represent normal condition and LP represent low phosphorus stress. The results represent the average of three biological replicates. * represent significant differences. (A): BnaVHA-a2s; (B): BnaVHA-a3s; (C): BnaAVP1s.
3 讨论
细胞的伸长和分裂是植物生长发育的必然过程, 在这一过程中, 液泡是调控细胞内外压力平衡以及物质代谢的主要器官。植物体内3种质子泵中VHA (V-H+-ATPase)的功能和结构最为复杂; 对于AVP即H+-PPase (V-H+-PPase), 目前认为有3种编码H+-PPase的基因, 其中1种基因编码I型酶, 叫做AVP1, 定位于液泡, 另2种编码II型酶, 叫做AVP2-1和AVP2-2, 定位于高尔基体膜[25]。AVP1蛋白水解产生能量并使液泡酸化, 有研究表明, 拟南芥avp1-1突变体中根、芽、花的生长受到严重影响并导致败育, 主要是因为拟南芥AVP1除了酸化液泡外, 还控制植物生长素的运输[26,27]。甘蓝型油菜作为旱地作物, 主要以硝态氮为氮素营养, 并累积在液泡中, 这种氮素利用特性导致根际土壤pH升高, 而液泡型质子泵的水解会减轻盐分过高造成的生理干旱[28], AVP1基因的表达, 也调控着甘蓝型油菜的氮代谢循环。本文以基因功能已知的拟南芥基因为参考序列, 筛选出9条BnaAVP1基因, 发现甘蓝型油菜BnaAVP1在白菜和甘蓝远缘杂交后遗传完好, 且数量明显多于甘蓝和白菜; 进一步的信息和序列分析发现, 其编码的氨基酸均为酸性氨基酸, 且大部分为疏水性蛋白; 染色体定位表明, BnaAVP1基因分布于甘蓝型油菜基因组A/C亚基因组上, 且甘蓝型油菜的BnaAVP1基因与甘蓝AVP1基因关系紧密。近年来, 已在植物基因功能的发现和挖掘方面做了大量的研究工作, 从转录合成到对植物的生理调控都有了一定的了解, 甜菜中VHA基因的A亚基的启动子在NaCl存在下活性增高, 转录量增加[29]; 盐胁迫下, 拟南芥和竹子A亚基转录水平增加2~4倍, 而番茄中A亚基转录水平瞬时增加, 且在持续处理3 d后恢复到对照水平[30]。本文筛选出的3条BnaVHA-a2和4条BnaVHA-a3甘蓝型油菜基因, 在白菜和甘蓝远缘杂交后遗传完好, 且数量多于甘蓝和白菜, BnaVHA-a2和BnaVHA-a3均为两性蛋白, 且甘蓝型油菜BnaVHA-a2和BnaVHA-a3基因也与甘蓝关系紧密。
旱地作物主要吸收硝态氮来合成体内有机代谢物, 而质子泵在这一过程中起着重要的调控作用, 质子泵的活性在铵态氮营养下明显高于在硝态氮下, 这与铵和硝态氮的吸收方式有关[31], 同时也与这2种氮素形态在细胞内的同化过程有关。在铵态氮营养下根系质子泵提高是一种必然的现象, 主要是因为铵态氮的吸收促进了质子泵的活性, 进而分泌质子来平衡细胞内外电荷和离子平衡[32]。硝酸盐从细胞质进入液泡主要靠液泡膜上的H+/NO3-通道运输来完成, 而液泡膜上硝酸盐与其他阴离子构成的通道则有利于液泡中硝酸盐向细胞质的转移, 细胞质中高浓度的NO3--N对液泡膜H+-ATPase有抑制作用, 随着细胞质内NO3–趋于耗尽, 抑制作用被解除, H+-ATPase活性恢复, 建立起新的质子梯度, 从而推动NO3-/H+共运转, 使液泡内的NO3-外排至细胞质[33,34]。这一观点与本文的研究结果一致, 本研究中, 随着低氮处理的时间增加, 3种质子泵的表达量均表现为先下降后上升的趋势。基于目前的研究, 不同质子泵的不同家族成员在甘蓝型油菜体内的表达部位不同, 我们将进一步探究甘蓝型油菜生长过程中质子泵家族基因之间的关系及其在氮素吸收利用过程中的调控机制, 以期从质子调控角度为甘蓝型油菜氮素养分利用过程提供理论指导。
4 结论
十字花科作物的AVP1、VHA-a2和VHA-a3拷贝数存在着差异, 异源四倍体作物甘蓝型油菜的BnaAVP1和BnaVHA-a3同源基因数量明显多于系统进化关系相近的甘蓝和白菜, BnaAVP1、BnaVHA-a2和BnaVHA-a3基因分别位于A/C亚基因组的不同染色体上, 均有N末端保守结构, 所翻译蛋白多为两性蛋白; 另一方面, 甘蓝型油菜的BnaAVP1基因多与甘蓝关系紧密, 而BnaVHA-a2和BnaVHA-a3基因与白菜关系更紧密。在低氮和低磷条件下, 3个基因的表达也存在着差异, BnaAVP1s基因在低氮条件下地上部表达下调, 而根中BnaAVP1s基因在2个处理下表达均上调; BnaVHA-a2s在低氮条件下基本呈上调趋势, 但在低磷处理后却无明显差异; BnaVHA- a3s在低氮条件下下调, 而低磷处理条件下表达上调较多。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 3]
[本文引用: 1]
DOI:10.1016/j.pbi.2014.09.005URL [本文引用: 2]
DOI:10.1016/S1360-1385(01)01923-9URL [本文引用: 1]
DOI:10.1146/annurev.arplant.52.1.469URL [本文引用: 1]
DOI:10.1111/pbi.2017.15.issue-12URL [本文引用: 2]
DOI:10.1016/S0022-2836(05)80360-2URL [本文引用: 1]
[本文引用: 3]
[本文引用: 2]
DOI:10.1093/nar/gkg563URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1093/bib/bbn017URL [本文引用: 1]
DOI:10.1101/gr.092759.109URL [本文引用: 1]
DOI:10.1016/S0968-0004(99)01540-6URL [本文引用: 1]
DOI:10.1006/jmbi.1999.3091URL [本文引用: 1]
DOI:10.1093/nar/gkt381URL [本文引用: 1]
DOI:10.1016/j.jmb.2015.10.017URL [本文引用: 1]
[本文引用: 1]
DOI:10.2144/000112900URL [本文引用: 2]
[本文引用: 2]
DOI:10.1073/pnas.95.25.14863URL [本文引用: 2]
DOI:10.1007/978-1-60761-987-1URL [本文引用: 1]
[本文引用: 1]
DOI:10.1093/pcp/pcq096URL [本文引用: 1]
DOI:10.1126/science.1115711URL [本文引用: 1]
DOI:10.1016/j.febslet.2007.03.050URL [本文引用: 1]
DOI:10.1242/jeb.02014URL [本文引用: 1]
DOI:10.1104/pp.97.2.562URL [本文引用: 1]
[本文引用: 1]
DOI:10.1080/00380768.1995.10419566URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
