张少飞1,3, 张园1,2, 申传璞3

 , 陈奇2,4
, 陈奇2,4

1. 淮北师范大学生命科学学院, 安徽 淮北 235000;
2. 安徽医科大学生命科学学院, 安徽 合肥 230032;
3. 安徽医科大学药学院, 安徽 合肥 230032;
4. 中国科学院南海海洋研究所, 中国科学院热带海洋生物资源与生态重点实验室, 广东省海洋药物重点实验室, 中国科学院海洋微生物研究中心, 广东 广州 510301
收稿日期:2020-01-13;修回日期:2020-03-19;网络出版日期:2020-06-15
基金项目:国家自然科学基金(41806158);广东省海洋药物重点实验室开放课题基金及中国科学院热带海洋生物资源与生态重点实验室开放课题基金(2018011007)
*通信作者:陈奇, E-mail:chenqi@ahmu.edu.cn;
申传璞, Tel/Fax:+86-551-65172130;E-mail:scp1001@126.com.
摘要:[目的] 分离Streptomyces sp.NO1W98中的杀黑星菌素并鉴定其生物合成基因簇。[方法] 利用有机溶剂萃取法对Streptomyces sp.NO1W98放大规模发酵产物进行提取;以正向、反向色谱柱层析进行化合物的分离纯化;借助波谱学手段进行单体化合物的结构鉴定;采用Illumina Hiseq技术进行基因组序列测定,对得到的序列进行生物信息学分析、注释并定位杀黑星菌素的生物合成基因簇vtd,利用基于PCR-targeting的遗传操作系统构建vtd内相关基因的阻断突变株,同时利用pSET152AKE进行基因回补,并分析与野生菌株的发酵产物差异。[结果] 从NO1W98发酵产物提取物中初步分离鉴定了2个大环内酯类化合物杀黑星菌素A(1)和B(2);NO1W98的基因组大小约为11.6 Mb,蕴涵49个次级代谢产物生物合成基因簇,其中scaffold 3上的Region 3.3可能负责杀黑星菌素的生物合成;基因阻断和回补实验初步鉴定了杀黑星菌素的生物合成基因簇,包含6个骨架基因、5个转运基因、2个调控基因以及9个后修饰基因。[结论] 杀黑星菌素的分离、结构鉴定和基因簇的鉴定以及生物合成途径的推导为其遗传改造和工程菌株的构建奠定了分子基础。
关键词:Streptomyces sp.NO1W98杀黑星菌素基因簇PCR-targeting生物合成途径
Isolation and identification of venturicidins biosynthetic gene cluster from Streptomyces sp. NO1W98
Shaofei Zhang1,3, Yuan Zhang1,2, Chuanpu Shen3

 , Qi Chen2,4
, Qi Chen2,4

1. School of Life Sciences, Huaibei Normal University, Huaibei 235000, Anhui Province, China;
2. School of Life Sciences, Anhui Medical University, Hefei 230032, Anhui Province, China;
3. School of Pharmacy, Anhui Medical University, Hefei 230032, Anhui Province, China;
4. CAS Key Laboratory of Tropical Marine Bio-resources and Ecology, Guangdong Key Laboratory of Marine Materia Medica, RNAM Center of Marine Microbiology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, Guangdong Province, China
Received: 13 January 2020; Revised: 19 March 2020; Published online: 15 June 2020
*Corresponding author: Qi Chen, E-mail:chenqi@ahmu.edu.cn;
Chuanpu Shen, Tel/Fax:+86-551-65172130;E-mail:scp1001@126.com.
Foundation item: Supported by the National Natural Science Foundation of China (41806158) and by the Open Project Program of Guangdong Key Laboratory of Marine Materia Medica & Key Laboratory of Tropical Marine Bio-resources and Ecology, SCSIO, CAS (2018011007)
Abstract: [Objective] The aim of this study was to isolate venturicidins from Streptomyces sp. NO1W98 and identify their biosynthetic gene cluster. [Methods] The secondary metabolites were extracted from enlarged-scale fermentation broth of Streptomyces sp. NO1W98 with organic solvent, purified with normal and reverse phase silica gel chromatography and structure elucidated with spectroscopic approaches. The genomic DNA of Streptomyces sp. NO1W98 was extracted and sequenced by Illumina Hiseq technology. Gene cluster of venturicidins (vtd) was preliminarily located by bioinformatic analysis and verified by gene disruption and trans complementation. Different in fermentation extracts between Streptomyces sp. NO1W98 wild type and related mutants were analyzed with HPLC. [Results] Two macrolides, venturicidins A and B were isolated and their structures were identified from Streptomyces sp. NO1W98. Draft genome of Streptomyces sp. NO1W98 harbors 49 proposed secondary metabolite biosynthetic gene clusters, including the vtd located in Region 3.3 of scaffold 3 that is responsible for the biosynthesis of venturicidins. The vtd was preliminarily characterized by gene disruption of vtdA1, orf(-1), orf(+1) and then verified by gene complementation of vtdA1 to ΔvtdA1. The vtd was found to include 6 PKS skeleton genes, 5 transporter genes, 2 regulator genes and 9 post-PKS tailoring genes. [Conclusion] The isolation and identification of venturicidins and their biosynthetic gene cluster from Streptomyces sp. NO1W98 provide the basis for future genetic engineering and strain improvement.
Keywords: Streptomyces sp. NO1W98venturicidinsgene clusterPCR-targetingbiosynthetic pathway
锥虫病是一种因寄生于人和动物血液或组织中的锥虫引起的感染性疾病,其中引起关注最多的是一种易被忽视的热带寄生虫病——非洲锥虫病(Human African Trypanosomiasis,HAT),一直以来严重威胁着撒哈拉以南非洲30多个国家近6000万人民的健康和生命[1-3]。HAT也称睡眠病,是被布氏锥虫罗德西亚种(Trypanosoma brucei rhodesiense)或布氏锥虫冈比亚种感染的舌蝇(俗称采采蝇)叮咬传播引起的一种寄生虫病。针对HAT的治疗,目前注册在用的药物有4种,分别为苏拉明(suramin)、戊烷脒(pentamidine)、美拉胂醇(melarsoprol)和依氟鸟氨酸(eflornithine),各自适用的病症和作用机制不尽相同[1-4]。然而,由于这些药物开发的年代较为久远,出现的问题也越来越显著,如用药困难、副作用大、产生耐药性等等,在治疗的过程中给病人带来很多痛苦[2-4]。此外,目前虽然还有一些进入临床实验阶段的候选药物和一些作用靶点不明确的活性化合物,但它们仍未成药,远不能满足人们对HAT防治的需求[1, 3-4]。
杀黑星菌素(venturicidin,VTD)也叫阿博霉素,属于20-元大环内酯类抗生素(图 1),最初分离自3株不同的链霉菌菌株Streptomyces sp. AA32、AA117、AA590,对一系列植物病原菌具有显著的抑制活性[5-6]。由于杀黑星菌素分子量相对较大,在植物体表一般具有较强的渗透性,但无内吸性,因此,发病后用药量大且防治效果不好,被开发成为农用抗生素的前景并不广阔[7]。随着研究的深入,研究者们发现杀黑星菌素在体外能显著抑制T. b. brucei GUTat3.1亚株和T. b. rhodesiense STIB900亚株(IC50达到0.12-0.54 μg/mL),同时不具细胞毒活性[3, 8]。杀黑星菌素还能够通过抑制ATP驱动的质子转运和ATP的水解抑制E. coli H+-ATP酶[9-10],在一些真菌和细菌中,能够抑制ATP的合成[11-12]。另据研究表明,杀黑星菌素通过抑制大鼠肝脏线粒体的磷酰基转移反应发挥作用,抑制梨形四膜虫(Tetrahymena pyriformis ST)的线粒体ATP酶,具有抗疟活性[13-15]。因此,杀黑星菌素抗锥虫活性的作用机制与线粒体ATP合成酶系统有着密切联系,有望被开发成为具有新作用位点的抗锥虫药物。此外,Shaaban等还从Streptomyces sp. TS-2-2中分离得到了该家族的一个新成员杀黑星菌素C (图 1)[8]。
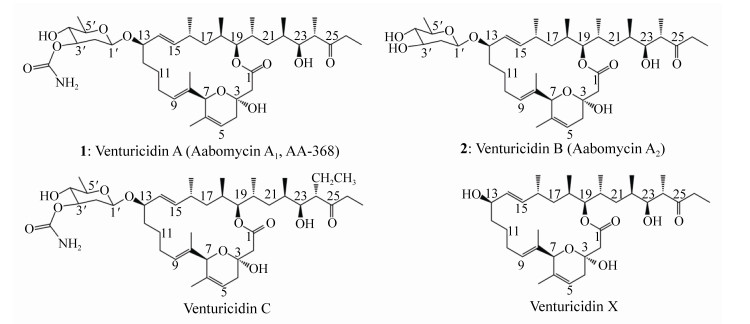 |
| 图 1 大环内酯类化合物杀黑星菌素A、B、C和X的化学结构 Figure 1 Chemical structures of venturicidins A (1), B (2), C and X. |
| 图选项 |
然而,到目前为止,杀黑星菌素的生物基因簇并未被鉴定,限制了人们从分子遗传水平上操纵相应产生菌的代谢途径,构建适于发酵的基因工程菌株,也限制了利用组合生物合成技术对其生物合成途径进行改造,获得相应的结构类似物用于抗锥虫活性的筛选。鉴于此,本文采用放线菌大规模发酵培养和产物的提取分离、基因组序列测定、文库构建和基于PCR-targeting的遗传操作等方法,结合相关生物信息学分析,从一株放线菌Streptomyces sp. NO1W98中分离得到了杀黑星菌素并鉴定了其生物合成基因簇,为后续的遗传改造和工程菌株的构建奠定了基础。
1 材料和方法 1.1 材料
1.1.1 质粒、菌株: Streptomyces sp. NO1W98 (NO1W98)由华北制药集团有限责任公司分离、鉴定和馈赠,菌株保存和传代使用固体ISP2培养基,于28 ℃下培养;生产代谢产物使用液体A-培养基。包含有目的基因的cosmid 18H11[阻断突变orf(–1)和vtdA1]、11H10 [阻断突变orf(+1)]为本研究中构建,研究中使用的其他菌株和质粒参见表 1。
表 1. 文中所用的菌株和质粒 Table 1. Strains and plasmids used in this study
| Strains/Plasmids | Description | Reference/Source |
| Strains | ||
| ??E. coli LE392 | Host strain of cosmid vector SuperCos 1 | Stratagene |
| ??E. coli DH5α | Host strain for general clone | Stratagene |
| ??E. coli ET12567/pUZ8002 | Host strain for conjugation | [16] |
| ??E. coli BW25113/pIJ790 | Host strain for PCR-targeting | [16] |
| ??Streptomyces sp. NO1W98 | VTDs biosynthesis stain Streptomyces sp. NO1W98 | Laboratory stock |
| ??ΔvtdA1 | The vtdA1gene disrupted mutant of Streptomyces sp. NO1W98 | This study |
| ??Δorf(–1) | The orf(–1) gene disrupted mutant of Streptomyces sp. NO1W98 | This study |
| ??Δorf(+1) | The orf(+1) gene disrupted mutant of Streptomyces sp. NO1W98 | This study |
| ??ΔvtdA1:: vtdA1 | ΔvtdA1 complemented with vtdA1 | This study |
| Plasmids | ||
| ??SuperCos1 | Ampr, Kanr, cosmid vector | Stratagene |
| ??pIJ773 | Apmr, source of acc(3)IV and oriT fragment | [16] |
| ??pIJ790 | Cmlr, including λ-RED (gam, bet, exo) for PCR-targeting | [16] |
| ??pUZ8002 | Kanr, including tra for conjugation | [16] |
| ??pSET152AKE | Apmr, Kanr, int C31, ermE* | [17] |
| ??pCJ 2001 | Cosmid18H11derivative in which vtdA1 was disrupted by aac(3)IV | This study |
| ??pCJ 2002 | Cosmid 18H11 derivative in which orf(–1) was disrupted by aac(3)IV | This study |
| ??pCJ 2003 | Cosmid 11H10 derivative in which orf(+1) was disrupted by aac(3)IV | This study |
表选项
1.1.2 培养基和培养条件: (1) LB培养基:蛋白胨10 g,酵母提取物5 g,NaCl 10 g,加ddH2O至1 L,pH 7.0,固体添加1.5%–2.0%琼脂粉,121 ℃灭菌30 min备用;(2) M-ISP4培养基:可溶性淀粉10 g,K2HPO4 1 g,MgSO4·7H2O 1 g,NaCl 1 g,蛋白胨1 g,酵母粉0.5 g,(NH4)2SO4 2 g,CaCO3 2 g,自来水1 L,pH 7.2–7.4,固体添加1.5%–2%琼脂粉,121 ℃灭菌30 min备用;(3) ISP2培养基:葡萄糖4 g,酵母粉4 g;麦芽提取粉10 g,自来水1 L,pH 7.2–7.4,固体添加1.5%–2.0%琼脂粉,115 ℃灭菌30 min备用;(4) A-培养液[8]:葡萄糖10 g,酵母粉5 g,可溶性淀粉20 g,蛋白胨5 g,NaCl 4 g,K2HPO4 0.5 g,MgSO4·7H2O 0.5 g,CaCO3 2 g,自来水1 L,pH 7.0,121 ℃灭菌30 min备用。
大肠杆菌菌株DH5α和ET12567/pUZ8002用LB于37 ℃下培养,而BW25113/pIJ790相关菌株使用30 ℃或37 ℃培养。
1.1.3 试剂和主要仪器: 限制性核酸内切酶和DNA Marker等购自上海生工生物工程有限公司;高保真DNA聚合酶购自TransGen公司;长片段DNA的扩增使用宝生物公司的PrimeSTAR? GXL DNA Polymerase;DNA回收试剂盒和质粒提取试剂盒购自上海生工生物工程有限公司。其他常规试剂均为国产分析纯级产品,各种抗生素均购自国内的试剂公司,使用浓度为氨苄青霉素(Ampicillin,Amp) 100 μg/mL,卡那霉素(Kanamycin,Kan) 50 μg/mL,安普霉素(Apramycin,Apm) 50 μg/mL,氯霉素(Chloramphenicol,Cml) 25 μg/mL,甲氧苄氨嘧啶(Trimethoprim,Tmp) 50 μg/mL。PCR引物由上海生工生物工程有限公司合成,DNA测序由上海生工生物工程有限公司完成。Eppendorf 5331梯度PCR仪,Eppendolf 5810R台式大容量多功能冷冻离心机,Eppendolf 5418小型台式高速离心机,BIO-RAD Molecular Image Gel DocTM XR+ Imaging System凝胶成像系统,SONICS超声波破碎仪VCX-130,Eppendorf Concentrator plus旋转浓缩仪,Heidolf Laborota4000 eco/WB/G3旋转蒸发仪,Agilent 1260 infinity高效液相色谱仪(High Performance Liquid Chromatography,HPLC),分析型色谱柱为Agilent Zorbax SB-C18 (150 mm×4.6 mm,5 μm),Bruker maXis超高分辨液质联用仪,Bruker AVANCE DRX 500 M核磁共振波谱仪(500/125 MHz,TMS为内标)。
表 2. 本研究中使用的引物 Table 2. Primer pairs used in this study
| Primers | Sequence (5′→3′) | Purpose |
| orf(–1)-ts-F | GACCATCGCGTACATCAACG | Δorf(–1) confirmation and genomic library screening, 1497 bp |
| orf(–1)-ts-R | GCGAGCGACACATAGAGCAT | |
| vtdA1-ts-F | TGAGGAGTCGAGGAGTTCGG | ΔvtdA1 confirmation and genomic library screening, 1675 bp |
| vtdA1-ts-R | CGCAGTAGTCGGGATATCCG | |
| orf(+1)-ts-F | GATCAAGGTGGGGATCACC | Δorf(+1) confirmation and genomic library screening, 644 bp |
| orf(+1)-ts-R | CAACTCGTCGTTCTCGCCTA | |
| orf(–1)-del-F | CGGTTCTTCTACGCCACCGTCATGCTGCCGCTCGAGCACattccggggatccgtcgacc | orf(–1) disruption |
| orf(–1)-del-R | CTTCGACGTGGCCAGGCCCTTGATGACGTAGCGGATGTAtgtaggctggagctgcttc | |
| vtdA1-del-F | GACCCCCAGCAGCGACTCGCCCTCGAACTCGGCTGGGAGattccggggatccgtcgacc | vtdA1 disruption |
| vtdA1-del-R | TGCGTCGGGCCGCGGCCACGGGGTCAGCCCGGTCTGCACtgtaggctggagctgcttc | |
| orf(+1)-del-F | ACCGAGCGCGAGGGCGCGCGGCTGCTCGAACAGGCCGCGattccggggatccgtcgacc | orf(+1) disruption |
| orf(+1)-del-R | GGCCACCTCCGCCGTGGTGGCGGCGTCCGCGGGGGCCGCtgtaggctggagctgcttc |
表选项
1.2 Streptomyces sp. NO1W98野生型菌株、基因阻断突变株的培养和发酵 Streptomyces sp. NO1W98野生型和相关的基因阻断突变株培养时使用ISP2固体培养基,根据需要在28 ℃下培养3–7 d,与相应的E. coli ET12567/pUZ8002系列菌株进行接合转移实验时使用M-ISP4固体培养基。NO1W98野生型和相应突变株发酵生产相应代谢产物时使用A-培养液[8],小瓶发酵时采用一步培养法,在250 mL锥形瓶中装入50 mL A-培养液(无菌),接入适当NO1W98或突变株的菌体,28 ℃下200 r/min振荡培养约1周;放大规模发酵时采用两步培养法,以小瓶振荡培养36–48 h的产物作为种子,转入到含200 mL A-培养液的1 L锥形瓶中(无菌),28 ℃下200 r/min继续振荡培养约1周,以实际检测结果为准。
1.3 Streptomyces sp. NO1W98发酵产物的提取与分离 对Streptomyces sp. NO1W98的发酵产物(约8 L)进行离心得到菌液和菌体两部分,分别用等体积的乙酸乙酯和丙酮萃取3次,得到各自对应的粗提物。HPLC检测后合并两份粗提物,得到总浸膏约6 g。将合并的粗提物用适量氯仿-甲醇混合溶剂和硅胶进行拌样,挥干后装柱进行柱层析分离。采用氯仿-甲醇系统(A柱)进行梯度洗脱,二者比例依次为100/0、98/2、96/4、94/6、92/8、90/10和80/20,分别得到Fr.1、Fr.2、Fr.3、Fr.4、Fr.5、Fr.6和Fr.7共计7个组分。含有目标物的组分再用石油醚-乙酸乙酯系统进行梯度洗脱(B柱),二者比例依次为90/10、80/20、70/30、60/40、50/50、40/60、30/70、20/80、10/90和0/100,共得到10个组分(B1–B10);将含有目标物的组分再用乙腈-水系统进行MPLC (C柱),每10 min收集1瓶,TLC和HPLC检测后旋干,含目标物的组分用甲醇溶解并用孔径为0.45 μm的滤膜过滤除去不溶物,高速离心后进行半制备,半制备的条件为:Hitachi半制备HPLC,装配有Hitachi泵、二极管阵列检测器和D2000模块精英数据工作站,采用YMC-Pack ODS的半制备柱(YMC,250 mm×10 mm,5 μm)。流动相A相为15%乙腈,流动相B相为85%乙腈,均含0.1%冰乙酸(glacial acetic acid,GAA);流速为2.5 mL/min,检测波长为210 nm和254 nm。HPLC走样程序:HPLC走样程序:0–20 min,30%–90% B相;20.1–21.0 min,90%–100% B相;21.1–26.0 min,100% B相;26.1–30.0 min,0% B相。
1.4 Streptomyces sp. NO1W98基因组DNA的提取、测序及蕴含基因簇的生物信息学分析 Streptomyces sp. NO1W98基因组DNA的提取参考链霉菌实验手册[18],并略有改动;得到的基因组DNA分别进行全基因组扫描、序列拼接和基因功能注释,由上海凌恩生物科技有限公司采用HiSeq 4000测序技术完成。基因组序列使用antiSMASH软件(https://antismash.secondarymetabolites.org/)进行蕴藏的次级代谢产物的预测,各目标基因簇利用2ndFind在线软件(https://biosyn.nih.go.jp/2ndfind/)分析内部涵盖的次级代谢产物生物合成基因。读码框的分析采用FramePlot 4.0 beta软件(https://nocardia.nih.go.jp/fp4/),对应的基因功能注释采用BLAST (https://blast.ncbi.nlm.nih.gov/);PKS (polyketide synthase,聚酮合酶)内部的结构域预测采用PKS/NRPS Analysis软件(https://nrps.igs.umaryland.edu/)。
1.5 Streptomyces sp. NO1W98基因组文库的构建和筛选 参考SuperCos1 Cosmid Vector Kit和Gigapack Ⅲ XL Packaging Extracts操作手册,构建Streptomyces sp. NO1W98的基因组文库,挑取2400个克隆子于96孔板中并保存到超低温冰箱中,对应制成混合模板500个。设计3对引物筛选orf(–1)-ts-F/R、vtdA1-ts-F/R和orf(+1)-ts-F/R分别筛选含有orf(–1)、vtdA1和orf(+1)的cosmid。最后对得到的cosmid进行末端测序,以确定各自的末端序列,选择合适的cosmid进行后续的基因阻断实验。
1.6 Streptomyces sp. NO1W98的抗生素敏感性实验及基因阻断突变株的构建 测试Streptomyces sp. NO1W98对安普霉素(Apm)、氨苄西林(Amp)、卡那霉素(Kan)、氯霉素(Cml)和三甲氧苄胺嘧啶(Tmp)等抗生素的敏感性,作为后续基因阻断时的抗性筛选标记。
基因阻断突变株的构建流程参考文献[19],以vtdA1的阻断实验为例。首先用vtdA1-ts-F/R这对引物进行筛选,得到目标大小为1675 bp的阳性克隆子分别对应划线至Kan+Amp的固体LB平板上,置于37 ℃培养箱中过夜培养。分别从固体平板上挑取单克隆摇菌,菌液PCR进一步验证。提取cosmid,用BamH I及EcoR I/Bgl II进行酶切分析,选择cosmid 18H11转化至E. coli BW25113/pIJ790得到E. coli BW25113/pIJ790/ 18H11,成功转入相应cosmid的菌株分别制备感受态细胞备用。接下来的步骤参照基于λ-RED介导的PCR-Targeting技术[16],vtdA1被Apm抗性片段置换掉的重组cosmid分别标记为pCJ 2001。将这些重组的cosmid分别转化E. coli ET12567/pUZ8002感受态细胞,与Streptomyces sp. NO1W98进行接合转移并挑选双交换突变株。利用微波法[20]对疑似双交换突变株进行快速抽提基因组DNA以及PCR验证:以vtdA1-ts-F/R作为其阻断突变前后的验证引物,扩增产物野生型大小为1675 bp,相应突变型大小为2168 bp。
1.7 vtdA1基因阻断突变株(ΔvtdA1)的回补 vtdA1基因阻断突变株(ΔvtdA1)的回补参考文献[17],简要流程为:以Streptomyces sp. NO1W98的基因组DNA为模板,用引物vtdA1-c-F (CGCCATATGGTGACCCCGGTGCCGCCCGG)和vtdA1-c-R (TGCTCTAGATCAGGAGATCCCGAA TTCCT)进行PCR扩增,经Nde I和Xba I双酶切后,连接至同样酶切处理的载体pSET152AKE,得到pSET152AKE-vtdA1,转化E. coli ET12567/ pUZ8002感受态细胞,与ΔvtdA1进行接合转移并挑选双交换突变株,得到对应的回补突变株ΔvtdA1::vtdA1。
1.8 Streptomyces sp. NO1W98野生型及其突变株的HPLC检测 采用1.2部分进行Streptomyces sp. NO1W98野生菌株及其相应的基因阻断和回补突变株的小瓶发酵,用100 mL乙酸乙酯萃取发酵产物,上清于旋转蒸发仪上蒸干,得到的提取物以1 mL甲醇作为溶剂制成相应的粗提物,离心后取20 μL进行HPLC检测。条件为:流动相A相为15%乙腈,流动相B相为85%乙腈,均含0.1%冰乙酸(glacial acetic acid,GAA);流速为1 mL/min,检测波长为210 nm和254 nm。HPLC走样程序:0–20 min,0%–70% B相;20–21 min,70%–100% B相;21–26 min,100% B相;26.0–26.1 min,100%–0%;26.1–30.0 min,0% B相。
2 结果和分析 2.1 化合物1和2的分离和结构鉴定 经过正向柱层析,HPLC制备分离得到化合物1约20.5 mg,化合物2约5.6 mg。结构解析如下。
化合物1白色粉末,(+)ESI-MS给出的准分子离子峰m/z 772 [M+Na]+,提示其分子量为749,提示化合物中可能还有一个氮原子。根据氢谱和碳谱的信息可以推测其分子式为C41H67NO11,不饱和度为9。在化合物1的1H NMR中可以发现5个双峰甲基质子信号,4个单峰甲基信号,2个孤立的烯质子信号(δH 5.49, br m; δH 5.44, dd, J= 10.5, 5.5 Hz)以及一组反式烯质子信号(δH 5.39, dd, J=15.0, 7.5 Hz; δH 5.34, dd, J=15.0, 8.5 Hz)。在化合物1的13C NMR谱中可以观察到41个碳信号,其中有6个烯碳信号,1个酰胺羰基信号以及1个酮羰基碳信号。综合以上信息,通过文献查阅[8]确定化合物1为venturicidins A。
化合物2白色粉末,(+)ESI-MS给出的准分子离子峰m/z 729 [M+Na]+,提示其分子量为706。根据氢谱和碳谱的信息可以推测其分子式为C40H66O10,不饱和度为8。在化合物2的1H NMR中可以发现5个双峰甲基质子信号,4个单峰甲基信号,2个孤立的烯质子信号(δH 5.48, m; δH 5.44, m)以及一组反式烯质子信号(δH 5.38, dd, J= 15.0, 8.5 Hz; δH 5.30, dd, J=15.0, 8.5 Hz)。在化合物2的13C NMR谱中可以观察到41个碳信号,其中有6个烯碳信号以及1个酮羰基碳信号。综合以上信息,可以推导出该化合物依然为venturicidin型的化合物,通过数据的比对发现该化合物与化合物1之间结构上的区别是在C-3′位置上的酰胺基团。通过文献查阅[8]确定化合物2为venturicidins B。
2.2 Streptomyces sp. NO1W98菌株的生物信息学分析和杀黑星菌素生物合成基因簇的初步定位 根据上海凌恩生物科技有限公司对NO1W98进行基因组测序和拼接的结果,我们得到107个scaffold文件,约11.6 Mb个碱基序列。利用antiSMASH软件对基因组序列进行分析,结果表明NO1W98中共有49个cluster (基因簇),其中包括18个Ⅰ型PKS及相关杂合形式。Ⅰ型PKS中与杀黑星菌素的生物合成可能相关的为位于Scaffold3上面的Region 3.3, 该段序列与已鉴定的氯丝菌素(chlorothricin)有着25%的基因相似性[21],与伴刀球霉素A (concanamycin A)有着35%的基因相似性[22]。我们进一步利用2ndFind软件进行分析,结果表明位于scaffold3上从179998 bp到295375 bp的DNA序列与杀黑星菌素的生物合成可能相关,但在该段序列内部含有多个gap。鉴于此,我们对gap进行填补,最终拼接得到约为90.5 kb的DNA序列,命名为vtd。接下来,利用FramePlot 4.0 beta和Blast软件对该段基因进行功能注释,同时结合PKS/NRPS Analysis软件对相应PKS和模块的结构域组成进行分析,表明该段基因含有6个PKS编码基因,对应含有13个模块,与杀黑星菌素的骨架结构匹配。另外,杀黑星菌素和氯丝菌素在结构上除了共有大环内酯骨架外,还共有D-olivose结构单元,对应在该段序列中我们也得到了相应的糖基修饰基因。最后,该段序列中还含有1个糖基转移酶和1个氨甲酰转移酶编码基因,可以跟杀黑星菌素的结构中的糖基的装载以及氨甲酰化修饰对应起来。综上所述,我们推测vtd与杀黑星菌素的生物合成密切相关。杀黑星菌素疑似生物合成基因簇(vtd)的组织排布和基因功能注释结果见图 2和表 3。
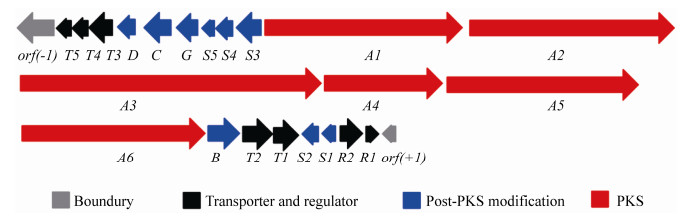 |
| 图 2 Streptomyces sp. NO1W98中vtd的基因组织排布图(相应基因的预测功能见表 3) Figure 2 Genetic organization of the vtd cluster. Proposed functions of individual orfs are summarized in Table 3. |
| 图选项 |
表 3. Streptomyces sp. NO1W98菌株杀黑星菌素生物合成基因簇内各基因的功能注释 Table 3. Gene annotation of venturicidins' biosynthetic gene cluster from Streptomycessp. NO1W98
| Protein | Size/aa | Proposed function |
| Orf(-1) | 819 | Arginine-tRNA ligase |
| Vtd T5 | 305 | ABC transporter permease |
| Vtd T4 | 323 | Transporter |
| Vtd T3 | 445 | Transporter |
| Vtd D | 352 | 2-hydroxyacid dehydrogenase |
| Vtd C | 514 | (2Fe-2S)-binding protein |
| Vtd G | 417 | Glycosyltransferase |
| Vtd S5 | 245 | 4-ketoreductase |
| Vtd S4 | 341 | 3-ketoreductase |
| Vtd S3 | 477 | NDP-hexose 2, 3-dehydratase |
| Vtd A1 | 3590 | Type Ⅰ PKS: KS-AT-PP-KS-AT-PP-KS-AT-KR-PP |
| Vtd A2 | 3723 | Type Ⅰ PKS: KS-AT-DH-ER-KR-PP-KS-AT-KR-PP |
| Vtd A3 | 5476 | Type Ⅰ PKS: KS-AT-DH-ER-KR-PP-KS-AT-DH-KR-PP-KS-AT-KR-PP |
| Vtd A4 | 2171 | Type Ⅰ PKS: KS-AT-DH-ER-KR-PP |
| Vtd A5 | 3588 | Type Ⅰ PKS: KS-AT-DH-KR-PP-KS-AT-DH-KR-PP |
| Vtd A6 | 3336 | Type Ⅰ PKS: KS-AT-DH-KR-PP-KS-AT-DH-PP-Te |
| Vtd B | 597 | Carbamoyltransferase |
| Vtd T2 | 570 | Transporter |
| Vtd T1 | 487 | Transporter |
| Vtd S2 | 325 | dNDP-glucose 4, 6-dehydratase |
| Vtd S1 | 289 | NDP-glucose synthase |
| Vtd R2 | 433 | Two component system sensor kinase |
| Vtd R1 | 230 | LuxR family transcriptional regulator |
| Orf(+1) | 257 | Disabled homolog 2-interacting protein |
表选项
2.3 Streptomyces sp. NO1W98中杀黑星菌素基因簇和边界基因的初步鉴定 为证实vtd确实负责杀黑星菌素的生物合成,我们对其内部的结构基因vtdA1进行了阻断突变。为此,首先探究了NO1W98对常用抗生素的敏感性,发现安普霉素(50 μg/mL)、氯霉素(25 μg/mL)、氨苄青霉素(100 μg/mL)和卡那霉素(50 μg/mL)对NO1W98具有较好的杀灭作用;三甲氧苄胺嘧啶(50 μg/mL)对NO1W98的生长无影响,但对E. coli具有杀灭作用。接下来,参考SuperCos1 Cosmid Vector Kit和Gigapack Ⅲ XL Packaging Extracts操作手册,成功构建了NO1W98的基因组文库,共得到约2400个克隆,制成混合模板500个。针对该段序列设计了相应的文库筛选引物,得到了相应的cosmids。
进一步利用PCR-targeting技术,构建得到了vtdA1被阻断的突变cosmid,将其转化E. coli ET12567/pUZ8002并与Streptomyces sp. NO1W98进行接合转移,通过抗性筛选和PCR验证得到了vtdA1的基因阻断突变株ΔvtdA1 (图 3)。对得到的突变株ΔvtdA1进行摇瓶发酵、萃取和HPLC分析,与经同样条件进行发酵和分析的Streptomyces sp. NO1W98野生型进行对比,发现突变株中完全不产生任何杀黑星菌素(图 4)。最后,对ΔvtdA1进行基因回补验证,发现回补菌株ΔvtdA1::vtdA1恢复了杀黑星菌素的产生(图 4)。由此,我们证实该段DNA序列vtd确实负责杀黑星菌素的生物合成。
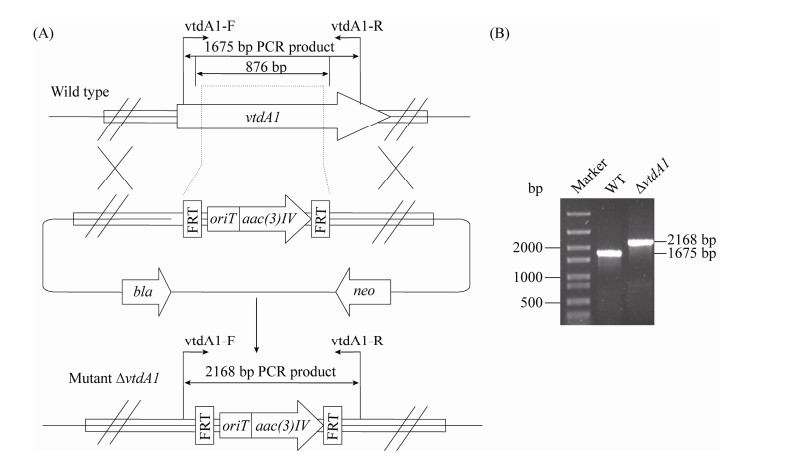 |
| 图 3 vtdA1基因阻断突变株的构建 Figure 3 Construction of vtdA1 gene disruption mutant. A: Schematic diagram. B: Gel electrophoresis analyses of PCR products. Marker: DNA molecular ladder; WT: Streptomyces sp. NO1W98, 1675 bp; ΔvtdA1: mutant strain, 2168 bp. |
| 图选项 |
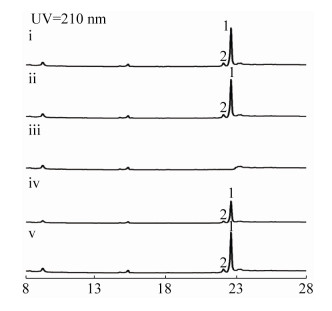 |
| 图 4 不同菌株提取物的HPLC检测 Figure 4 HPLC analysis of the fermentation extracts of Streptomyces sp. NO1W98 wild-type and its mutant strains. ⅰ: Streptomyces sp. NO1W98; ⅱ: Δorf(–1); ⅲ: ΔvtdA1; ⅳ: ΔvtdA1::vtdA1; ⅴ: Δorf(+1). |
| 图选项 |
为初步确定vtd基因簇的边界,我们又对其内部的orf(–1)和orf(+1)进行了阻断突变,分别得到对应的基因阻断突变株Δorf(–1)和Δorf(+1),并与Streptomyces sp. NO1W98同时进行摇瓶发酵、萃取和HPLC分析,结果发现这些突变株均能正常产生杀黑星菌素,与野生型没有任何区别(图 4),这说明orf(–1)和orf(+1)不是杀黑星菌素合成的必需基因,可能是其边界基因(图 2,表 3;相应vtd基因簇的GenBank登录号为MN914689)。为最终确定其边界基因和最小生物合成基因簇,还需对vtdT5、vtdT4、vtdT3、vtdD、vtdR1、vtdR2和vtdS1逐个进行阻断突变。
2.4 杀黑星菌素生物合成途径的推导 根据Ⅰ型PKS的装配原理和vtd内部基因功能注释的结果,结合Ⅰ型PKS的装配机制[23],我们推测杀黑星菌素的生物合成途径包括骨架的形成和后修饰途径两大部分(图 5),骨架的形成涉及vtdA1、vtdA2、vtdA3、vtdA4、vtdA5、vtdA6共计6个基因。接着通过结构域组成及功能分析,发现该组装线涵盖13个模块(module)。合成途径起始于1个丙酰-CoA的装载,酰基转移酶(acyltransferase,AT)特异性识别丙酰-CoA,将其装载于loading module的ACP上作为一个三碳单元的起始点进行后续的装配步骤。在随后的骨架分子延伸释放过程中,可以大致分为6种类型:(1)模块1起始于三碳单元,由甲基丙二酰-CoA在酮基合成酶(keto-synthase,KS)催化下脱羧形成;(2)模块2、4和7的结构域组成相同,比模块1多了一个酮基还原酶(keto reductase,KR)结构域,可将对应的酮基还原为羟基,其中模块4的羟基作为后续成环的位点;(3)模块3、5和8的结构域组成相同,比模块1多了一个脱水酶结构域(dehydratase,DH)、一个烯醇还原酶结构域(enoyl reductase,ER)和一个KR,可将对应的酮基形成饱和烃结构单元;(4)模块6和9的结构域组成相同,比模块1多了一个DH和一个KR,可将对应的酮基形成烯烃结构单元;(5)模块10和11的结构域组成相同,比模块1多了一个DH和一个KR,同时依据化合物的分子结构推测其中的DH功能失活,未形成对应的烯键而保留了β-羟基;(6)模块12比模块1多了一个DH,但功能失活,可加载上最后一个二碳单元。最后,装配链末端的硫酯酶结构域(thioesterase,TE)催化碳链上模块4的羟基亲核攻击模块12中ACP上的硫酯键,最终环化解离形成大环内酯骨架结构。再经后修饰形成骨架内的吡喃环结构得到VTD-X。
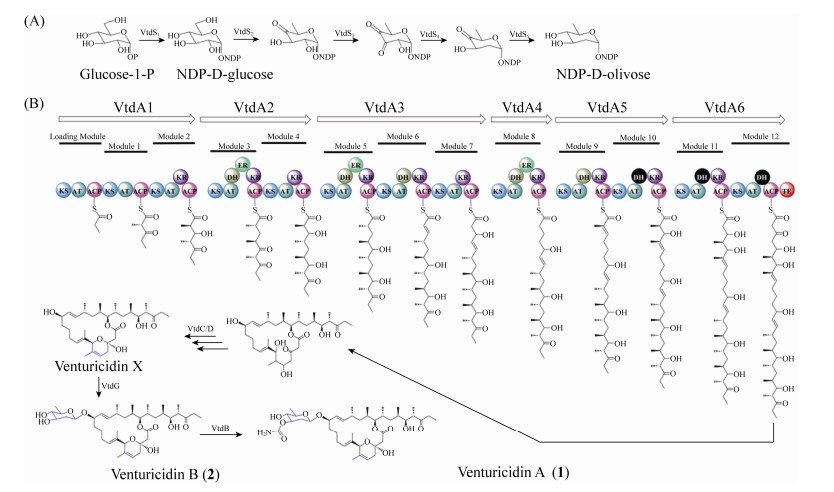 |
| 图 5 杀黑星菌素可能的生物合成途径 Figure 5 Proposed biosynthetic pathway of venturicidins. A: olivose sugar moiety; B: PKS assembly and biosynthetic route to venturicidin A (1). |
| 图选项 |
VTD结构中的后修饰途径为:葡萄糖-1-磷酸在NDP-葡萄糖合成酶VtdS1的作用下活化成为NDP-D-葡萄糖,接着依次在dNDP-葡萄糖4, 6-脱水酶VtdS2、NDP-己糖2, 3-脱水酶VtdS3、3-酮基还原酶VtdS4和4-酮基还原酶VtdS5的作用下形成NDP-D-olivose;VTD-X被糖基转移酶VtdG催化NDP-D-olivose连接上糖基形成VTD-B,最后氨甲酰转移酶VtdB对VTD-B进行氨甲酰化修饰形成终产物VTD-A (图 5)。
3 讨论 本研究首先从常规发酵分离着手得到化合物实体,经由基因组测序,结合相关生物信息学分析,初步定位了杀黑星菌素的生物合成基因簇,最后经体内基因阻断和遗传互补实验确证,从链霉菌NO1W98中分离鉴定了杀黑星菌素,并鉴定了其生物合成基因簇。
Streptomyces sp. NO1W98中VTD的分离鉴定提供了1株VTD的产生菌,可以用于后续基因工程菌株的构建。已有报道证实VTD类化合物具有显著的抗真菌和锥虫抑制活性[3, 5-6, 8],后续可以利用已得到的化合物实体拓展VTD的活性筛选范围,以获得更为广泛的活性。另外,相应的化合物实体可以作为构效关系研究的基础,为相关的化学合成研究提供帮助,从而为活性筛选提供更多的化合物实体。
鉴定生物合成基因簇最为常用的两种方法是基因阻断(敲除)和异源表达。本文使用的是前者,鉴于该基因簇比较大,而基于SuperCos1的cosmid文库容量通常只有20–40 kb,单一cosmid难以涵盖vtd的所有生物合成基因。因此,我们同步构建了容量更大的PAC文库,后续可广泛应用于该菌株的基因组挖掘。NO1W98相应遗传操作体系的建立,可为后续基因簇内其他基因的阻断突变提供实例,也为相应菌株的基因组挖掘打下基础。
杀黑星菌素生物合成基因簇的鉴定,确定了其产生的分子基础,可为后续产量优化和结构改造奠定基础。簇内涵盖的调控和转运基因可以借助相应的技术进行产量优化。另外,VTD蕴含的Ⅰ型PKS的机制可以借助代谢工程优化前体的供给,从而提高产量,构建高产基因工程菌株,有利于后续大规模生产和研究开发。相应后修饰基因的确定,可以依赖于组合生物合成技术得到不同修饰程度的VTD衍生物,为相关的活性筛选提供化合物实体,应用于农业和医药卫生领域。而在初步鉴定的杀黑星菌素生物合成基因簇内,除了负责核心骨架形成的vtdA1–vtdA6,负责后修饰的vtdS1–vtdS5、vtdB、vtdG,以及调控转运相关的vtdR1、vtdR2、vtdT1–T5外,余下的(2Fe-2S)结合蛋白编码基因vtdC和2-羟基酸脱氢酶编码基因vtdD的具体功能无法预测,推测其最有可能负责杀黑星菌素大环骨架初步装配之后的修饰(图 5),与吡喃环的形成密切相关,具体机制有待于进一步研究。与调控转运相关的vtdR1、vtdR2和vtdT1–T5后续需要逐个进行阻断突变,从而确定其具体功能。
总之,本研究通过“经典”的生物合成研究思路,从一株链霉菌NO1W98中分离得到了杀黑星菌素,并鉴定了其生物合成基因簇。研究结果扩充了Ⅰ型PKS途径的家族成员,为杀黑星菌素基因簇内其他基因的功能研究奠定了基础。后续可通过分子遗传学手段对杀黑星菌素进行结构修饰、改造以及产量优化,以扩充该家族化合物成员,为系统的活性筛选和构效关系研究提供支撑。
References
| [1] | Li GQ, He XZ. Progress in the immune mechanism of trypanosomiasis. Chinese Journal of Veterinary Medicine, 1996, 22(7): 44-46. (in Chinese) 李国清, 何晓竹. 锥虫病免疫机理的研究进展. 中国兽医杂志, 1996, 22(7): 44-46. |
| [2] | Du J, Zhu YM. Progress in the treatment of Human African Trypanosomiasis. Progress in Modern Biomedicine, 2015, 15(6): 1154-1159. (in Chinese) 杜金, 朱明彦. 人类非洲锥虫病的治疗研究进展. 现代生物医学进展, 2015, 15(6): 1154-1159. |
| [3] | Otoguro K, Ishiyama A, Namatame M, Nishihara A, Furusawa T, Masuma R, Shiomi K, Takahashi Y, Yamada H, ōmura S. Selective and potent in vitro antitrypanosomal activities of ten microbial metabolites. The Journal of Antibiotics, 2008, 61(6): 372-378. |
| [4] | Patrick DA, Gillespie JR, McQueen J, Hulverson MA, Ranade RM, Creason SA, Herbst ZM, Gelb MH, Buckner FS, Tidwell RR. Urea derivatives of 2-aryl-benzothiazol-5-amines:a new class of potential drugs for Human African Trypanosomiasis. Journal of Medicinal Chemistry, 2017, 60(3): 957-971. |
| [5] | Rhodes A, Fantes KH, Boothroyd B, McGonagle MP, Crosse R. Venturicidin:a new antifungal antibiotic of potential use in agriculture. Nature, 1961, 192(4806): 952-954. DOI:10.1038/192952a0 |
| [6] | Aizawa S, Nakamura Y, Shirato S, Taguchi R, Yamaguchi I. Aabomycin A, a new antibiotic. Ⅰ. Production, isolation and properties of aabomycin A. The Journal of Antibiotics, 1969, 22(10): 457-462. DOI:10.7164/antibiotics.22.457 |
| [7] | Ma CZ, Wei CM, Qian ZG. Activity of agricultural antifungal agent "Nongkang 653" against some phytopathogenic fungi in laboratory assay and control effect in field test. Acta Agriculture Shanghai, 2000, 16(S1): 54-56. (in Chinese) 马承铸, 魏春妹, 钱振官. 农抗653对几种真菌的抗菌活性和田间防效. 上海农业学报, 2000, 16(S1): 54-56. |
| [8] | Shaaban KA, Singh S, Elshahawi SI, Wang XC, Ponomareva LV, Sunkara M, Copley GC, Hower JC, Morris AJ, Kharel MK, Thorson JS. Venturicidin C, a new 20-membered macrolide produced by Streptomyces sp. TS-2-2. The Journal of Antibiotics, 2014, 67(3): 223-230. |
| [9] | Nesci S, Ventrella V, Trombetti F, Pirini M, Pagliarani A. Thiol oxidation is crucial in the desensitization of the mitochondrial F1FO-ATPase to oligomycin and other macrolide antibiotics. Biochimica et Biophysica Acta (BBA)-General Subjects, 2014, 1840(6): 1882-1891. DOI:10.1016/j.bbagen.2014.01.008 |
| [10] | Perlin DS, Latchney LR, Senior AE. Inhibition of Escherichia coli H+-ATPase by venturicidin, oligomycin and ossamycin. Biochimica et Biophysica Acta (BBA)-Bioenergetics, 1985, 807(3): 238-244. DOI:10.1016/0005-2728(85)90254-3 |
| [11] | Morales-Ríos E, de La Rosa-Morales F, Mendoza-Hernández G, Rodríguez-Zavala JS, Celis H, Zarco-Zavala M, García-Trejo JJ. A novel 11-kDa inhibitory subunit in the F1FO ATP synthase of Paracoccus denitrificans and related α-proteobacteria. The FASEB Journal, 2010, 24(2): 599-608. |
| [12] | Griffiths DE, Houghton RL. Studies on energy-linked reactions:modified mitochondrial ATPase of oligomycin-resistant mutants of Saccharomyces cerevisiae. European Journal of Biochemistry, 1974, 46(1): 157-167. |
| [13] | Walter P, Lardy HA, Johnson D. Antibiotics as tools for metabolic studies. Ⅹ. Inhibition of phosphoryl transfer reactions in mitochondria by peliomycin, ossamycin, and venturicidin. The Journal of Biological Chemistry, 1967, 242(21): 5014-5018. |
| [14] | Unitt MD, Lloyd D. Effects of inhibitors on mitochondrial adenosine triphosphatase of Tetrahymena pyriformis ST. Journal of General Microbiology, 1981, 126(2): 261-266. |
| [15] | Warhurst DC, Thomas SC. The chemotherapy of rodent malaria, XXXI. The effect of some metabolic inhibitors upon chloroquine-induced pigment clumping (CIPC) in Plasmodium berghei. Annals of Tropical Medicine & Parasitology, 1978, 72(3): 203-211. |
| [16] | Gust B, Kieser T, Chater KF. REDIRECT technology:PCR-targeting system in Streptomyces coelicolor. Norwich: John Innes Center, 2002. |
| [17] | Ma JY, Wang ZW, Huang HB, Luo MH, Zuo DG, Wang B, Sun AJ, Cheng YQ, Zhang CS, Ju JH. Biosynthesis of himastatin:assembly line and characterization of three cytochrome P450 enzymes involved in the post-tailoring oxidative steps. Angewandte Chemie International Edition, 2011, 50(34): 7797-7802. DOI:10.1002/anie.201102305 |
| [18] | Kieser T, Bibb MJ, Buttner MJ, Chater KF, Hopwood DA. Practical streptomyces genetics. Norwich: The John Innes Foundation, 2000. |
| [19] | Chen Q, Ji CT, Song YX, Huang HB, Ma JY, Tian XP, Ju JH. Discovery of McbB, an enzyme catalyzing the β-carboline skeleton construction in the marinacarboline biosynthetic pathway. Angewandte Chemie International Edition, 2013, 52(38): 9980-9984. DOI:10.1002/anie.201303449 |
| [20] | Xu P, Li WJ, Xu LH, Jiang CL. A microwave-based method for genomic DNA extraction from actinomycetes. Microbiology China, 2003, 30(4): 82-84. (in Chinese) 徐平, 李文均, 徐丽华, 姜成林. 微波法快速提取放线菌基因组DNA. 微生物学通报, 2003, 30(4): 82-84. |
| [21] | Jia XY, Tian ZH, Shao L, Qu XD, Zhao QF, Tang J, Tang GL, Liu W. Genetic characterization of the chlorothricin gene cluster as a model for spirotetronate antibiotic biosynthesis. Chemistry & Biology, 2006, 13(6): 575-585. |
| [22] | Haydock SF, Appleyard AN, Mironenko T, Lester J, Scott N, Leadlay PF. Organization of the biosynthetic gene cluster for the macrolide concanamycin A in Streptomyces neyagawaensis ATCC 27449. Microbiology, 2005, 151(10): 3161-3169. DOI:10.1099/mic.0.28194-0 |
| [23] | 王以光, 王勇. 抗生素生物技术. 第2版. 北京: 化学工业出版社, 2019. |
