 ,1,2,3
,1,2,3GSA: Genome Sequence Archive
Sisi Zhang1,2, Tingting Chen1,2, Junwei Zhu1,2, Qing Zhou1,3, Xu Chen1,2, Yanqing Wang1,2, Wenming Zhao ,1,2,3
,1,2,3通讯作者:
编委: 张勇
收稿日期:2018-06-29修回日期:2018-09-6网络出版日期:2018-11-20
| 基金资助: |
Editorial board:
Received:2018-06-29Revised:2018-09-6Online:2018-11-20
| Fund supported: |
作者简介 About authors
张思思,博士,工程师,研究方向:生物信息学E-mail:zhangss@big.ac.cn。
陈婷婷,硕士,工程师,研究方向:生物信息学E-mail:chentt@big.ac.cn,张思思和陈婷婷并列第一作者。

摘要
关键词:
Abstract
Keywords:
PDF (318KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
张思思, 陈婷婷, 朱军伟, 周晴, 陈旭, 王彦青, 赵文明. GSA:组学原始数据归档库[J]. 遗传, 2018, 40(11): 1044-1047 doi:10.16288/j.yczz.18-178
Sisi Zhang, Tingting Chen, Junwei Zhu, Qing Zhou, Xu Chen, Yanqing Wang, Wenming Zhao.
基因组测序技术的迅速发展,使生命科学研究产生的组学数据呈现爆炸性增长的态势,生命科学亦进入大数据时代。“数据堪比石油”,是国家重要战略资源,生物数据尤为如此。未来,生物数据的存储、管理、共享和开发利用将在一定程度上反应国家科技发展实力和水平。
国际上,美国、欧洲和日本于20世纪80年代相继建立核酸序列数据库[1,2,3],并于2002年成立了国际核酸序列共享联盟(International Nucleotide Sequence Database Collaboration, INSDC)[4],制定了生命科学研究领域数据管理和共享标准,收集并存储来自全世界科学家提交的组学数据,提供共享服务。随着数据量的持续增加和学术论文发表的数据共享要求,大量组学数据通过国际互联网递交到INSDC变得十分困难。我国国际网络出口带宽的瓶颈问题,更使得数据传输效率低下。以中国科学院北京基因组研究所150 Mbs出口带宽为例,向美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)数据库递交1 TB的数据需要花费两周甚至更长的时间。数据下载亦是如此,国内科研人员饱受从国际数据库下载数据效率低下的困扰。这种低下的数据传输效率以及对国际数据库使用的不便,使得我国生物学家在一定程度上失去了生命科学研究领域的竞争力。
随着国家一系列重大生命科学研究计划的部署和实施,预期我国每年将产生超过100 PB的组学数据。为解决这些海量数据存储、管理、共享与发布,中国科学院北京基因组研究所生命与健康大数据中心(BIG Data Center, BIGD)[5,6],研发并构建了组学原始数据归档系统(Genome Sequence Archive, GSA)[7],
专注于生命组学原始数据收集与管理,并提供免费的数据存储、共享与访问服务。GSA遵循国际INSDC的数据标准及数据库建设标准,可收集来自不同测序平台产出的数据,并存储序列数据及其对应的元数据信息,确保数据的完整性。本文主要从数据类别与使用、运行与效果等方面,全面解析GSA数据库在数据汇交、存储及共享各项功能,以便更高效为用户提供数据服务。
1 GSA的数据类别与使用
1.1 数据库建设
GSA系统建设采用数据分类存储、集中管理的指导思想及高内聚、低耦合的程序设计原则,既确保各类数据的独立性及关联性,又保证整体系统的完整性和有效性,避免信息孤岛的存在,可实现任一数据的向上回溯及向下追踪。所谓数据分类是根据表现形式将数据分为元数据信息和测序数据文件,集中管理则指通过内在的关联信息,对数据进行关联显示、调用与管理。GSA数据类别主要包括项目信息(BioProject)、样本信息(BioSample)、实验信息(Experiment)、测序反应(Run)信息以及测序数据文件。项目信息是用来描述所开展研究的目的、涉及物种、数据类型、研究思路等信息;样本信息是指本研究涉及的生物样本描述,如样本类型、样本属性等;实验信息包括实验目的、文库构建方式、测序类型等信息;测序反应信息包括测序文件和对应的校验信息。各类数据之间采用线性、一对多的模式进行关联,从而形成“金字塔”式的信息组织与管理模式。
为确保与国际同类数据库系统的兼容性,GSA遵循INSDC联盟的数据标准和命名规范。如BioProject序列号(accession number)以PRJC开头,前3位字母PRJ为Project的缩写,第4位字母C代表中国(China),第5位是英文字母A~Z,其余为6位自然数,例如PRJCA000001;BioSample序列号以SAMC开头,前3位字母SAM为Sample的缩写,第4位字母C代表中国(China),第5位是英文字母A~Z,其余为6位自然数,例如SAMCA000001。其他数据类型的编码遵循相同标准,既确保数据编码的全局性与唯一性,又便于后续数据使用者的信息检索与访问。
1.2 数据汇交与审核
2017年6月,数据递交系统(BIG Sub)正式上线,作为生命与健康大数据中心数据统一汇交入口,BIG Sub承载GSA系统的数据汇交功能,并为用户提供一站式数据递交服务。在元数据信息汇交方面,BIG Sub提供两种数据递交方式:在线递交和离线递交。在线递交即通过WEB页面实现信息输入,GSA系统提供可视化及向导化的操作模式,最大限度地规范信息录入并实现各类数据的质量控制;离线递交即采用离线模板的形式,由科研人员事先整理文件,然后通过GSA系统或GSA审编人员进行数据批量导入。在线递交较适合小量样本的数据递交(如样本数小于10个),而离线递交适合大量样本的数据递交,这两种互为补充的提交方式为科研人员提供简单、便利、高效的数据递交服务。在测序序列文件汇交方面,提供在线FTP上传服务和邮寄硬盘两种服务模式,如超过500 GB的序列文件,数据递交者可以选择采用邮递硬盘的模式,由GSA系统审编人员协助上传数据。针对FTP数据上传服务,BIG Sub为每位数据递交者提供独立的数据存储空间,以防止不同递交者之间的数据干扰及信息泄露,充分确保数据的隐私性和安全性。GSA系统具有元数据审核和序列数据质量控制功能。针对元数据信息,采用自动校验和人工校验相结合的模式进行审核,以保证信息的有效性。而对于测序序列数据,GSA内置数据质量审核的标准化流程,以防止序列文件在处理、传输、压缩、拷贝等过程中出现损坏。审核内容包括:(1) 文件压缩的正确性;(2) 文件格式的正确性;(3) 序列的测序质量。针对某一数据递交,只有当元数据和序列数据均审核通过,GSA系统方可为该数据分配正式的访问序列号(accession number)。
1.3 数据发布与管理
GSA系统提供两种数据状态控制权限:公开访问(public)或受控访问(confidential),公开即意味着数据可被任何人访问或下载使用,受控即在数据公开发布前,他人的访问将被限制,且无法通过系统检索获取相关的信息,更无法下载相关数据文件。同时,GSA系统提供个性化的数据状态及权限管理方案,即由数据递交者自行设置数据受控保护期限,最大限度的满足论文发表前的数据保密需求,亦可方便论文审稿人对数据在线访问与审核(peer reviewer link),还可快速实现文章发表后的数据发布与共享。2 GSA的运行效果
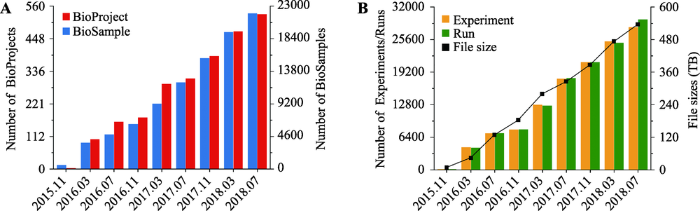
GSA系统自2015年上线运行以来,获得包括Cell、Nature、PNAS、AJHG、GPB、Cell Research等在内的30余个国内外期刊的认可,支持40余篇科研论文的数据归档与发布任务。截止2018年7月,GSA接收的数据来自国内外93个机构的300余名科研用户,累计递交项目信息达535个,涵盖的生物物种数量超过178个,涉及的生物学样本21 843个,生物学实验28 050个,测序反应29 624个,测序序列数据总量超过 TB,且各类数据呈现显著增长的趋势(图1)。同时,GSA系统收录的数据受到国内外科研人员的广泛关注,经统计发现,GSA系统访问用户来自于70余个国家/地区,累计访问量超过13 305人次。数据下载用户来自39个国家/地区,日平均上传下载量达到1 TB。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1GSA数据量统计图
A: GSA数据库中项目和样本数量统计图;B: GSA数据库中实验、测序反应和文件数量统计图。数据统计截至2018年7月。
Fig. 1Statistics of data submissions of GSA
GSA系统不仅为国内科研人员提供了良好的数据存储和共享服务,同时也为国家的重大科研计划提供了良好的数据管理和支撑平台。如为国家科技部重点研发计划“精准医学”专项提供数据集中存储和管理保障,为中国科学院战略先导专项“分子模块设计育种创新体系”和“动物复杂性状的进化解析与调控”等项目提供数据集中存储、统一管理和共享服务,并取得了较好的应用效果。
3 结语与展望
GSA是一个公共的、免费的组学原始数据存储库,在建设标准上遵循国际INSDC联盟的数据标准和数据库结构标准,在内容上收集生命科学研究中产生的组学测序数据及其元数据信息,并且接受来自全世界科研人员的数据递交与获取请求。在组学大数据时代,GSA不仅作为当前INSDC联盟体系的补充以缓解组学大数据远距离传输与储存的压力,而且承担推动国际组学大数据共享的责任。GSA致力于我国组学数据汇交、管理、共享与应用体系的建设,促进我国在生命组学大数据领域的发展,力争我国在国际组学数据共享领域的地位,为国内外生命科学研究与产业创新应用提供服务。2017年,中国科学院北京基因组研究所发起“中国基因组数据共享倡议”(http://bigd.big.ac.cn/gdsd),呼吁中国产出的组学数据递交GSA进行统一存储、管理与共享。截止2018年6月,得到国内570多个机构1400余人支持。
立足现在、着眼未来,GSA将不断完善系统的功能,更加重视数据资源使用者的需求,开发类似fastq-dump (如SRA toolkit)的辅助数据下载,实现数据便捷共享。此外,还将开发基于数据分析的云计算平台,提供免费数据在线分析服务,届时,用户可以不用下载数据便可利用云计算资源进行数据分析。顺应国家大数据发展战略及科技创新和产业发展需求,存好、管好国家生物数据资源,推动“国家生物信息中心”的建立。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
URL [本文引用: 1]
URLPMID:5753375 [本文引用: 1]

Abstract For 35 years the European Nucleotide Archive (ENA; https://www.ebi.ac.uk/ena) has been responsible for making the world's public sequencing data available to the scientific community. Advances in sequencing technology have driven exponential growth in the volume of data to be processed and stored and a substantial broadening of the user community. Here, we outline ENA services and content in 2017 and provide insight into a selection of current key areas of development in ENA driven by challenges arising from the above growth.
URLPMID:4702806 [本文引用: 1]

The DNA Data Bank of Japan Center (DDBJ Center;http://www.ddbj.nig.ac.jp) maintains and provides public archival, retrieval and analytical services for biological information. The contents of the DDBJ databases are shared with the US National Center for Biotechnology Information (NCBI) and the European Bioinformatics Institute (EBI) within the framework of the International Nucleotide Sequence Database Collaboration (INSDC). Since 2013, the DDBJ Center has been operating the Japanese Genotype-phenotype Archive (JGA) in collaboration with the National Bioscience Database Center (NBDC) in Japan. In addition, the DDBJ Center develops semantic web technologies for data integration and sharing in collaboration with the Database Center for Life Science (DBCLS) in Japan. This paper briefly reports on the activities of the DDBJ Center over the past year including submissions to databases and improvements in our services for data retrieval, analysis, and integration.
URLPMID:29420816 [本文引用: 1]

Abstract Splicing is an essential and highly regulated process in mammalian cells. We developed a synthetic riboswitch that efficiently controls alternative splicing of a cassette exon in response to the small molecule ligand tetracycline. The riboswitch was designed to control the accessibility of the 3' splice site by placing the latter inside the closing stem of a conformationally controlled tetracycline aptamer. In the presence of tetracycline, the cassette exon is skipped, whereas it is included in the ligand's absence. The design allows for an easy, context-independent integration of the regulatory device into any gene of interest. Portability of the device was shown through its functionality in four different systems: a synthetic minigene, a reporter gene and two endogenous genes. Furthermore, riboswitch functionality to control cellular signaling cascades was demonstrated by using it to specifically induce cell death through the conditionally controlled expression of CD20, which is a target in cancer therapy.
URLPMID:27899658 [本文引用: 1]

Abstract Biological data are generated at unprecedentedly exponential rates, posing considerable challenges in big data deposition, integration and translation. The BIG Data Center, established at Beijing Institute of Genomics (BIG), Chinese Academy of Sciences, provides a suite of database resources, including (i) Genome Sequence Archive, a data repository specialized for archiving raw sequence reads, (ii) Gene Expression Nebulas, a data portal of gene expression profiles based entirely on RNA-Seq data, (iii) Genome Variation Map, a comprehensive collection of genome variations for featured species, (iv) Genome Warehouse, a centralized resource housing genome-scale data with particular focus on economically important animals and plants, (v) Methylation Bank, an integrated database of whole-genome single-base resolution methylomes and (vi) Science Wikis, a central access point for biological wikis developed for community annotations. The BIG Data Center is dedicated to constructing and maintaining biological databases through big data integration and value-added curation, conducting basic research to translate big data into big knowledge and providing freely open access to a variety of data resources in support of worldwide research activities in both academia and industry. All of these resources are publicly available and can be found at http://bigd.big.ac.cn. The Author(s) 2016. Published by Oxford University Press on behalf of Nucleic Acids Research.
URLPMID:29140455 [本文引用: 1]

Abstract Cell types in cell populations change as the condition changes: some cell types die out, new cell types may emerge and surviving cell types evolve to adapt to the new condition. Using single-cell RNA-sequencing data that measure the gene expression of cells before and after the condition change, we propose an algorithm, SparseDC, which identifies cell types, traces their changes across conditions and identifies genes which are marker genes for these changes. By solving a unified optimization problem, SparseDC completes all three tasks simultaneously. SparseDC is highly computationally efficient and demonstrates its accuracy on both simulated and real data. The Author(s) 2017. Published by Oxford University Press on behalf of Nucleic Acids Research.
URLPMID:5339404 [本文引用: 1]

With the rapid development of sequencing technologies towards higher throughput and lower cost, sequence data are generated at an unprecedentedly explosive rate. To provide an efficient and easy-to-use platform for managing huge sequence data, here we presentGenome Sequence Archive(GSA;http://bigd.big.ac.cn/gsaorhttp://gsa.big.ac.cn), a data repository for archivingraw sequence data. In compliance with data standards and structures of the International Nucleotide Sequence Database Collaboration (INSDC), GSA adopts four data objects (BioProject, BioSample, Experiment, and Run) for data organization, accepts raw sequence reads produced by a variety of sequencing platforms, stores both sequence reads and metadata submitted from all over the world, and makes all these data publicly available to worldwide scientific communities. In the era ofbig data, GSA is not only an important complement to existing INSDC members by alleviating the increasing burdens of handling sequence data deluge, but also takes the significant responsibility for global big data archive and provides free unrestricted access to all publicly available data in support of research activities throughout the world.
