 ,*华南农业大学生命科学学院, 亚热带农业生物资源保护与利用国家重点实验室, 广州 510642
,*华南农业大学生命科学学院, 亚热带农业生物资源保护与利用国家重点实验室, 广州 510642CRISPR-based DNA Fragment Deletion in Plants
Xianrong Xie, Dongchang Zeng, Jiantao Tan, Qinlong Zhu, Yaoguang Liu ,*State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresources, College of Life Sciences, South China Agricultural University, Guangzhou 510642, China
,*State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresources, College of Life Sciences, South China Agricultural University, Guangzhou 510642, China通讯作者: E-mail:ygliu@scau.edu.cn
责任编辑: 朱亚娜
收稿日期:2020-12-11接受日期:2021-02-25网络出版日期:2021-01-01
| 基金资助: |
Corresponding authors: *E-mail:ygliu@scau.edu.cn
Received:2020-12-11Accepted:2021-02-25Online:2021-01-01

摘要
基于CRISPR/Cas9系统的基因组编辑技术已成为基因功能研究和遗传修饰的重要工具。在引导RNA的引导下, Cas9蛋白对基因组靶位点进行精准切割产生DNA双链断裂(DSB), 借助细胞内的DSB修复机制, 可实现基因组靶位点碱基的缺失、插入或者替换, 甚至发生片段删除。该文介绍了基于CRISPR/Cas9基因组编辑系统的DSB微同源末端连接修复方式(MMEJ)提高基因组DNA片段删除效率的方法, 主要从靶点设计和突变检测方面展开详细描述。
关键词:
Abstract
CRISPR/Cas9-based genome editing technology has been an important tool to study the gene function and genomic modification. Directed by a guide RNA, Cas9 protein can cleavage the genomic DNA at the target site, and produce mutations, including deletion, insertion, substitution and fragment deletion, by DNA double strand break (DSB) repair mechanism. In this protocol, we introduce the method to use CRISPR/Cas9 system to increase the efficiency of genomic DNA fragment deletion with microhomology-mediated end joining, especially the details in target design and detection of mutant plants.
Keywords:
PDF (794KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
谢先荣, 曾栋昌, 谭健韬, 祝钦泷, 刘耀光. 基于CRISPR编辑系统的DNA片段删除技术. 植物学报, 2021, 56(1): 44-49 doi:10.11983/CBB20203
Xie Xianrong, Zeng Dongchang, Tan Jiantao, Zhu Qinlong, Liu Yaoguang.

基于成簇的规律间隔的短回文重复序列(clu- stered regularly interspaced short palindromic repeats, CRISPR)系统的基因组编辑技术因其简便、高效的优势, 已成为生物学功能研究和遗传改良的一项革命性技术(Ma et al., 2016; 苏钺凯等, 2019)。CRISPR相关蛋白(CRISPR associated, Cas)在单链引导RNA (single guide RNA, sgRNA)的引导下, 在靶点处切割产生DNA双链断裂(double strand break, DSB)。在植物细胞中, DSB主要经由细胞内的非同源末端连接(non-homologous end-joining, NHEJ)进行修复(Manova and Gruszka, 2015)。借助NHEJ介导的修复方式对DSB末端进行微小处理后重新连接, 这种易错的修复方式在修复后往往会导致1个或几个核苷酸的缺失或插入, 从而达到敲除基因功能的目的。然而, 由于这种方式产生的碱基插入和删除数目较小, 易使基因产生不正常的转录本或异常蛋白, 影响科研人员对基因功能的判断(Tuladhar et al., 2019)。
植物基因组中存在多种多样的调控元件或非编码DNA, 包括启动子的顺式元件、miRNA序列或其结合位点以及其它具有调控作用的基因间区, 这些元件在基因转录或翻译水平行使重要功能(Burgess et al., 2015)。利用CRISPR/Cas9技术对这些调控序列进行编辑, 也是目前用于功能基因研究和相关数量性状遗传改良的热点(Zhang et al., 2018)。然而, 借助NHEJ介导的单靶点编辑, 仅产生少数碱基的插入/缺失突变, 往往很难有效破坏这些非编码DNA序列的功能。因此, 可以在目标DNA序列的两侧设计2个靶点, 以达到将整个目标序列删除的目的。然而, 借助NHEJ介导的修复机制, 实现片段删除的效率较低(Canver et al., 2014)。
近期, Owens等(2019)发现在细胞和小鼠(Mus musculus)胚胎中, CRISPR/Cas9介导的大片段缺失连接点处出现高频率的微同源序列(microhomolo- gous sequence, MHS), 暗示微同源介导的末端连接(microhomology-mediated end joining, MMEJ)修复方式提高了大片段DNA缺失的发生率。本课题组前期研究发现, 如果在水稻(Oryza sativa)基因组靠近2个MHS位点内侧位置设计2个CRISPR/Cas9靶点, 基于MMEJ的DSB修复系统能够比NHEJ更加高效地删除DNA片段(Tan et al., 2020); 进一步利用MMEJ删除DNA片段的原理, 高效地将一个转基因水稻T-DNA内的潮霉素筛选标记基因(HPT)删除(Tan et al., 2020)。为了便于研究人员设计基于MMEJ修复方式进行片段删除的靶点, 我们进一步开发了一个在线工具MMEJ-KO (
1 实验材料
本课题组开发的整套适用于单、双子叶植物的CRISPR/Cas9基因组编辑载体系统(Ma et al., 2015) (可以向本课题组索取, 也可通过Addgene (2 试剂
试剂包括BsaI限制性内切酶、Phanta Max Super-Fidelity DNA Polymerase或KOD-FX等高保真酶以及其它分子生物学常用试剂。3 仪器与软件
仪器包括台式离心机、超净工作台、电激转化仪和移液器等。软件包括靶点设计软件MMEJ-KO (
4 实现DNA片段删除的靶点设计思路
利用MMEJ实现基因组片段的高效删除, 在设计编辑切割靶点时需要在目标删除片段两侧寻找1对MHSs, 然后设计1对位于MHSs内侧且尽量靠近MHSs的靶点。利用CRISPR/Cas9或CRISPR/Cpf1介导的编辑造成2个MHSs内侧附近的DSBs, 有助于断裂点两侧的MHSs产生配对, 提高细胞内MMEJ修复效率, 高效删除2个MHSs之间的片段(图1)。图1
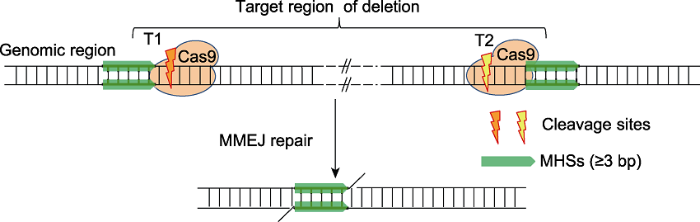 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1利用微同源介导的末端连接(MMEJ)修复进行DNA片段删除的原理(改自Xie et al., 2020)
MHS: 微同源序列
Figure 1The principle for microhomology-mediated end joining (MMEJ)-based DNA fragment deletion(modified from Xie et al., 2020)
MHS: Microhomologous sequence
5 实验流程
具体操作流程见图2。图2
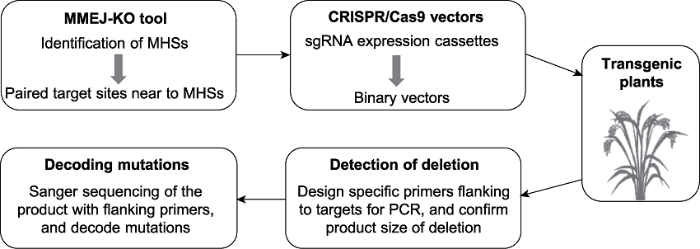 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2基于微同源介导的末端连接(MMEJ)介导DNA片段删除的实验流程
Figure 2The experimental workflow for microhomology-mediated end joining (MMEJ)-mediated DNA fragment deletion
5.1 靶点对的设计
5.1.1 MMEJ-KO在线设计工具基于上述设计原则, 如果通过人工查找MHSs (BLASTN等比对工具不支持对MHSs的查找), 并在MHSs两内侧寻找合适靶点, 将费时费力。因此, 本团队开发了一款在线工具MMEJ-KO (
该工具目前支持41个植物基因组、5个非植物基因组和少量线粒体、叶绿体基因组的靶点设计。此外, 软件也支持对于尚未加入参考基因组的序列进行靶点设计, 用户只需在靶向的参考基因组处选择“None”即可, 但输出的结果将不包含对靶点的脱靶位点评估。
5.1.2 靶点对的选择
MMEJ-KO的结果页面是一个可交互的图表, 页面中直观显示所输入的序列或选定基因序列中存在的MHS组。如果指定了希望删除的区间, 则只会显示位于删除区间两侧的MHSs对。序列下方的表则显示具体的MHS组和对应的靶点对信息; 如果将鼠标置于图中的某一组MHS上, 则表中只会显示该MHS组附近可用的靶点。点击某组MHS下面的“show”, 即显示目标序列上MHS和靶点PAM的位置。
为了方便选择靶点对, MMEJ-KO对GC含量高于80%或低于30%、靶序列与sgRNA序列有8个以上配对碱基(容易产生阻碍转录的RNA茎-环结构)、连续4个或以上T碱基(可能被认作转录终止信号), 或脱靶值高于0.7的候选靶点判定为编辑效率可能较差, 并给出特别的警示(!、!!或!!!)。用户在选择靶点对时, 应尽量避免选择使用这类有警示标识的靶点。在MMEJ-KO结果页面中, 勾选用于载体构建的靶点对 (2个靶点均要勾选)后, 点击primerDesign可跳转至sgRNA表达盒引物构建工具primerDesign-V, 进行引物序列设计。
5.2 编辑载体的构建与转化
由于基于MMEJ实现片段删除, 需要2个靶点共同参与, 因此需要构建一个双靶点的编辑载体并用于遗传转化。实验过程与普通的植物基因组编辑载体构建相同, 基本步骤包括sgRNA表达盒的构建、将表达盒克隆到CRISPR/Cas9双元载体以及遗传转化获得转基因阳性植株(图2)。具体可参考曾栋昌等(2018)的操作方法。5.3 转化植株的突变类型检测
在获得阳性的T0转化植株后, 需要进一步确认植株是否发生目标基因组片段删除或靶位点序列是否存在突变。根据实验目的和目标删除片段的大小, 设计相应的扩增引物和测序引物进行检测。根据目标删除片段的大小(本方法以800 bp为界限, 可适当调整), 提供以下2种扩增方法作为参考(图3A-C)。图3
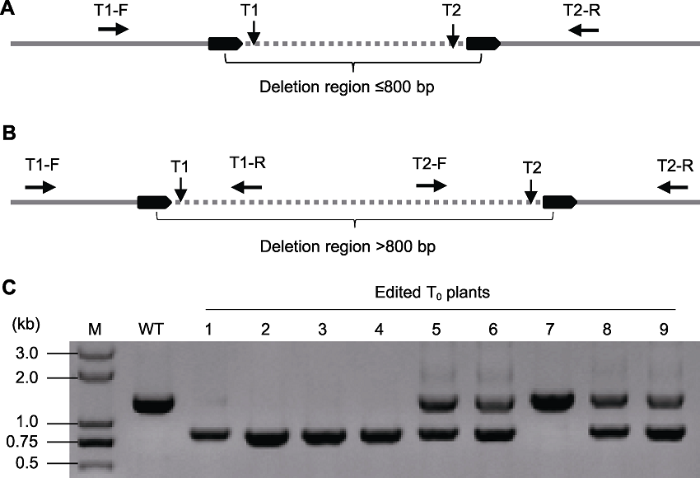 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3再生植株突变体突变类型的鉴定策略
(A) 如果删除片段小于或约等于800 bp, 在靶点1 (T1)的上游和靶点2 (T2)的下游设计正向引物(T1-F)和反向引物(T2-R)进行PCR扩增; (B) 如果删除片段大于800 bp, 合成4条引物(使以下各扩增产物长度相互有约70-100 bp的差别), 设3组PCR反应(T1-F/T1-R、T2-F/T2-R和T1-F/T2-R), 也可以在同一个PCR反应使用4条引物(如果只扩增出T1-F/T1-R片段和T2-F/T2-R片段而扩增不出T1-F/T2-R小片段, 表明没有发生片段删除; 如果只扩增出T1-F/T2-R小片段而没有扩增出T1-F/T1-R片段和T2-F/T2-R片段, 表明2个等位序列都产生了片段删除; 如果3个片段都被扩增出, 表明一个等位序列产生了片段删除而另一等位序列没有片段删除); (C) 按策略(A)的引物设计的PCR扩增电泳检测图。WT: 未编辑的野生型; 1-9为T0编辑植株, 其中植株1-4的2个等位序列都发生了片段删除; 植株5、6、8、9有1个等位序列发生了片段删除, 植株7未发生片段删除。
Figure 3The strategy for identification of the mutant types in transgenic plants
(A) Amplification of the target region using the forward primer (T1-F) upstream the T1 and reverse primer (T2-R) downstream T2 if the length of desirable deleted fragment is less than 800 bp; (B) Four primers (with the length of the following amplification products is about 70-100 bp different from each other) with three combinations (T1-F/T1-R, T2-F/T2-R, and T1-F/T2-R) or pooled in an amplification are suggested if the length of desirable deleted fragment is more than 800 bp (there is no deletion of the target region if the resulting bands are produced with primer groups (T1-F/T1-R and T2-F/T2-R) but not T1-F/T2-R; both alleles had fragment deletion if the resulting bands are produced with primer group T1-F/T2-R but not T1-F/T1-R and T2-F/T2-R; one allele had fragment deletion and the other allele had no fragment deletion if all three fragments were produced; (C) The example of gel electrophoresis according to the detection method of (A). WT: Wild type; 1-9 indicate edited T0 plants in which 1-4 with biallelic fragment deletion, 5, 6, 8 and 9 with heterozygous deletion and 7 with no deletion.
5.3.1 目标删除片段在800 bp以内的检测方法
由于目标删除片段较小, 可以设计1对包含2个靶点的检测引物直接进行PCR扩增, 根据凝胶电泳后条带大小判断片段是否发生删除(对于删除片段小于50 bp的难以通过琼脂糖凝胶电泳区分, 需进一步测序或借助其它检测方法判别)。对于二倍体生物, 如果只有其中1条等位染色体发生了片段删除, 则电泳后会出现一大一小2条条带(较大的为未发生删除的条带, 较小的为发生删除的条带)。如果2条染色体均发生了删除(删除片段的长度和连接点可能并不完全相同), 则电泳的条带长度均小于野生型。
5.3.2 目标删除片段在800 bp以上的检测方法
对于删除片段较大的序列, 可以先设计1对包含整个目标删除片段在内(在2个靶点序列的外侧)的引物直接PCR扩增后进行电泳, 以初步判断2个靶点间是否存在片段删除。然而, 由于扩增片段较大, 未发生删除的等位染色体可能会因扩增困难导致不能扩增出对应大小的条带或因浓度太低不易观察, 从而对突变类型产生错误判断。因此, 可以进一步在每个靶点的两侧分别设计检测引物进行扩增(扩增片段大小在500 bp-1 kb之间); 也可以将4条引物在同一个PCR反应中进行扩增。如果可以扩增出预期条带, 说明其中至少有1条等位染色体未发生删除, 但靶位点序列是否发生突变及突变类型等具体情况, 需要进一步测序分析。
通过PCR扩增可以初步判断目标片段是否发生删除, 但要想获得具体的突变后序列或删除后连接点序列则需要通过进一步的测序分析进行确认。对于二倍体物种的编辑, 对扩增产物直接进行普通Sanger测序, 如果是杂合或双等位突变会产生重叠的波峰图, 而无法直接判别突变类型和序列。本课题组前期开发了一套用于突变序列解码的在线分析工具DSDecode (Liu et al., 2015)及其升级版DSDecodeM(
6 注意事项
借助MMEJ修复介导的片段敲除在一定程度上可以提高目标片段的删除效率, 但删除效果会受编辑的靶物种、遗传转化方法及靶点编辑效率等因素的影响。对于一段目标删除序列, 在序列两侧设计1对靶点, 使其各自的切点尽可能靠近MHSs, 可以提高DSB后MHSs对相互配对的几率, 从而在一定程度上提高删除的效率。而MHSs的长度在4 bp或以上(最短为3 bp)就可以介导MMEJ的修复, 其长度可能并不是影响删除效率的关键因素。此外, 如果期望获得更多不同位置或范围的删除事件, 可以采用多组靶点的形式。例如, 在1组或多组MHSs内侧设计2个以上的靶点, 可能会提高基因组片段删除的效率。(责任编辑: 朱亚娜)
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/j.pbi.2015.07.001URL [本文引用: 1]
DOI:10.1074/jbc.M114.564625URLPMID:24907273 [本文引用: 1]

The clustered regularly interspaced short [corrected] palindromic repeats (CRISPR)/CRISPR-associated (Cas) 9 nuclease system has provided a powerful tool for genome engineering. Double strand breaks may trigger nonhomologous end joining repair, leading to frameshift mutations, or homology-directed repair using an extrachromosomal template. Alternatively, genomic deletions may be produced by a pair of double strand breaks. The efficiency of CRISPR/Cas9-mediated genomic deletions has not been systematically explored. Here, we present a methodology for the production of deletions in mammalian cells, ranging from 1.3 kb to greater than 1 Mb. We observed a high frequency of intended genomic deletions. Nondeleted alleles are nonetheless often edited with inversions or small insertion/deletions produced at CRISPR recognition sites. Deleted alleles also typically include small insertion/deletions at predicted deletion junctions. We retrieved cells with biallelic deletion at a frequency exceeding that of probabilistic expectation. We demonstrate an inverse relationship between deletion frequency and deletion size. This work suggests that CRISPR/Cas9 is a robust system to produce a spectrum of genomic deletions to allow investigation of genes and genetic elements.
[本文引用: 1]
URLPMID:25917172 [本文引用: 1]
URLPMID:27108381 [本文引用: 1]
URLPMID:26557130 [本文引用: 1]
[本文引用: 1]
DOI:10.1111/pbi.13390URLPMID:32336015 [本文引用: 2]
URLPMID:31492834 [本文引用: 1]
URLPMID:33569675 [本文引用: 5]
DOI:10.1016/j.molp.2017.06.004URLPMID:28624544 [本文引用: 1]
[本文引用: 1]
CRISPR/ Cas9系统在植物基因组编辑中技术改进与创新的研究进展
1
2019
... 基于成簇的规律间隔的短回文重复序列(clu- stered regularly interspaced short palindromic repeats, CRISPR)系统的基因组编辑技术因其简便、高效的优势, 已成为生物学功能研究和遗传改良的一项革命性技术(
植物CRISPR/Cas9多基因编辑载体构建和突变分析的操作方法
1
2018
... 由于基于MMEJ实现片段删除, 需要2个靶点共同参与, 因此需要构建一个双靶点的编辑载体并用于遗传转化.实验过程与普通的植物基因组编辑载体构建相同, 基本步骤包括sgRNA表达盒的构建、将表达盒克隆到CRISPR/Cas9双元载体以及遗传转化获得转基因阳性植株(
Advances in understanding cis regulation of the plant gene with an emphasis on comparative genomics
1
2015
... 植物基因组中存在多种多样的调控元件或非编码DNA, 包括启动子的顺式元件、miRNA序列或其结合位点以及其它具有调控作用的基因间区, 这些元件在基因转录或翻译水平行使重要功能(
Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRIS- PR)/Cas9 nuclease system in mammalian cells
1
2014
... 植物基因组中存在多种多样的调控元件或非编码DNA, 包括启动子的顺式元件、miRNA序列或其结合位点以及其它具有调控作用的基因间区, 这些元件在基因转录或翻译水平行使重要功能(
DSDecode: a web-based tool for decoding of sequencing chromatograms for genotyping of targeted mutations
1
2015
... 通过PCR扩增可以初步判断目标片段是否发生删除, 但要想获得具体的突变后序列或删除后连接点序列则需要通过进一步的测序分析进行确认.对于二倍体物种的编辑, 对扩增产物直接进行普通Sanger测序, 如果是杂合或双等位突变会产生重叠的波峰图, 而无法直接判别突变类型和序列.本课题组前期开发了一套用于突变序列解码的在线分析工具DSDecode (
A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants
1
2015
... 本课题组开发的整套适用于单、双子叶植物的CRISPR/Cas9基因组编辑载体系统(
CRISPR/Cas9 platforms for genome editing in plants: developments and applications
1
2016
... 基于成簇的规律间隔的短回文重复序列(clu- stered regularly interspaced short palindromic repeats, CRISPR)系统的基因组编辑技术因其简便、高效的优势, 已成为生物学功能研究和遗传改良的一项革命性技术(
DNA damage and repair in plants-from models to crops
1
2015
... 基于成簇的规律间隔的短回文重复序列(clu- stered regularly interspaced short palindromic repeats, CRISPR)系统的基因组编辑技术因其简便、高效的优势, 已成为生物学功能研究和遗传改良的一项革命性技术(
Microhomologies are prevalent at Cas9-induced larger deletions
1
2019
... 近期,
Efficient CRISPR/Cas9-based plant genomic fragment deletions by microhomology- mediated end joining
2
2020
... 近期,
... )删除(
CRISPR-Cas9-based mu- tagenesis frequently provokes on-target mRNA misregulation
1
2019
... 基于成簇的规律间隔的短回文重复序列(clu- stered regularly interspaced short palindromic repeats, CRISPR)系统的基因组编辑技术因其简便、高效的优势, 已成为生物学功能研究和遗传改良的一项革命性技术(
MMEJ-KO: a web tool for designing paired CRISPR guide RNAs for microhomology-mediated end joining fragment deletion
5
2020
... 近期,
... 软件包括靶点设计软件MMEJ-KO (
... 利用MMEJ实现基因组片段的高效删除, 在设计编辑切割靶点时需要在目标删除片段两侧寻找1对MHSs, 然后设计1对位于MHSs内侧且尽量靠近MHSs的靶点.利用CRISPR/Cas9或CRISPR/Cpf1介导的编辑造成2个MHSs内侧附近的DSBs, 有助于断裂点两侧的MHSs产生配对, 提高细胞内MMEJ修复效率, 高效删除2个MHSs之间的片段(
... MHS: 微同源序列
... 基于上述设计原则, 如果通过人工查找MHSs (BLASTN等比对工具不支持对MHSs的查找), 并在MHSs两内侧寻找合适靶点, 将费时费力.因此, 本团队开发了一款在线工具MMEJ-KO (
CRISPR-GE: a convenient software toolkit for CRISPR- based genome editing
1
2017
... 软件包括靶点设计软件MMEJ-KO (
Applications and potential of genome editing in crop improvement
1
2018
... 植物基因组中存在多种多样的调控元件或非编码DNA, 包括启动子的顺式元件、miRNA序列或其结合位点以及其它具有调控作用的基因间区, 这些元件在基因转录或翻译水平行使重要功能(
