 ,*中国科学院种子创新研究院, 遗传与发育生物学研究所, 植物细胞与染色体工程国家重点实验室, 基因组编辑中心, 北京 100101
,*中国科学院种子创新研究院, 遗传与发育生物学研究所, 植物细胞与染色体工程国家重点实验室, 基因组编辑中心, 北京 100101Saturation Mutagenesis Using Dual Cytosine and Adenine Base Editors
Rui Zhang, Caixia Gao ,*Center for Genome Editing, State Key Laboratory of Plant Cell and Chromosome Engineering, Institute of Genetics and Developmental Biology, Innovation Academy for Seed Design, Chinese Academy of Sciences, Beijing 100101, China
,*Center for Genome Editing, State Key Laboratory of Plant Cell and Chromosome Engineering, Institute of Genetics and Developmental Biology, Innovation Academy for Seed Design, Chinese Academy of Sciences, Beijing 100101, China通讯作者: E-mail:cxgao@genetics.ac.cn
责任编辑: 孙冬花
收稿日期:2021-01-11接受日期:2021-02-25网络出版日期:2021-01-01
| 基金资助: |
Corresponding authors: *E-mail:cxgao@genetics.ac.cn
Received:2021-01-11Accepted:2021-02-25Online:2021-01-01

摘要
遗传性变异是表型多样性的基础, 靶向饱和突变作物基因可以促进产生具有优异农艺性状的突变体。相较于传统诱变育种和异源物种中的定向进化方法, 基于双碱基编辑系统的植物基因靶向随机突变技术可对植物内源基因产生高效突变, 从而实现原位定向进化, 加快植物育种及功能基因研究进程。该文介绍了使用饱和靶向内源基因突变编辑器(STEME)对植物功能基因进行靶向随机突变的具体操作方法, 从靶点设计和突变体检测方面进行了详细描述。
关键词:
Abstract
Because the genome of an organism determines its primary phenotype, evolutionary principles suggest that genetic variations enhance phenotypic diversity towards increased fitness. Targeted saturation mutagenesis of crop genes could be used to screen for genetic variants with improved agronomic traits. Compared to traditional mutational breeding or directed evolution in heterologous organisms, targeted mutagenesis via dual cytosine and adenine base editors effectively generates endogenous mutagenesis and facilitates in vivo directed evolution of plant genes. In this protocol, we detail the process towards using saturated targeted endogenous mutagenesis editors (STEMEs) to generate targeted, random mutagenesis of plant genes. In particular, we focus on the process of designing targets, screening and genotyping the resulting evolved variants.
Keywords:
PDF (703KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
张瑞, 高彩霞. 基于双碱基编辑系统的植物基因靶向随机突变技术. 植物学报, 2021, 56(1): 50-55 doi:10.11983/CBB21009
Zhang Rui, Gao Caixia.
遗传变异是自然界物种形成和进化的基础, 也是物种性状多样性产生的原因。目前, 在作物中通过突变筛选优异性状的方法主要是诱变育种, 即通过物理及化学方法(如辐射和EMS诱变)产生全基因组的随机突变, 再从突变体中选取表现优异的个体进行新品种的培育(Henikoff et al., 2004; Slade et al., 2005; Pacher and Puchta, 2017)。由于诱变产生频率较低, 且变异方向和性质难以控制, 因此传统的诱变育种方法通常费时费力。定向进化则通过创制目标基因的突变文库, 在施加一定选择压力下能够快速获得目的突变体(Packer and Liu, 2015)。植物基因的定向进化通常先通过易错PCR、DNA合成或DNA重组等方法在体外产生目标基因的突变文库, 之后转化到大肠杆菌(Escherichia coli)或者酵母(Saccharomyces cerevisiae)中进行功能筛选(Engqvist and Rabe, 2019)。然而, 由于离开原始的基因组和细胞环境, 筛选出来的基因突变可能不能完全反映出其在植物中的真实功能。更重要的是, 大多数重要农艺性状无法在大肠杆菌或酵母中进行筛选。基于双碱基编辑系统的植物基因靶向随机突变技术, 可在植物中直接对特定内源基因进行饱和突变, 从而定向筛选具有所需优异农艺性状的突变体, 该方法有助于加快植物育种及功能基因研究进程(Li et al., 2020)。
基于双碱基编辑系统的植物基因靶向随机突变技术所使用的基因编辑工具称为“饱和靶向内源基因突变编辑器(saturated targeted endogenous mutagenesis editor, STEME)”, 主要包括STEME-1和STEME-NG两种(Li et al., 2020)。STEME-1编辑器中实现双碱基编辑功能的元件主要包括胞嘧啶脱氨酶APOBEC3A (Gehrke et al., 2018; Zong et al., 2018)、腺嘌呤脱氨酶ecTadA-ecTadA7.10 (Gaudelli et al., 2017; Li et al., 2018)和Cas9蛋白的切口酶形式nCas9 (D10A) 3个功能蛋白。它们通过48或32个氨基酸的连接序列从N端到C端依次连接为一个大的融合蛋白。在融合蛋白的C端带有抑制体内尿嘧啶糖基化酶活性的UGI蛋白和核定位信号。融合蛋白在单链引导RNA (single guide RNA, sgRNA)的作用下, 可以实现靶向区域一定范围内的C:G>T:A和A:T>G:C突变。STEME-NG是将STEME-1中的nCas9 (D10A)替换Cas9-NG变体(VRVRFRR)的切口酶形式nCas9-NG (D10A) (Nishimasu et al., 2018)。水稻(Oryza sativa)中更偏好在PAM (pro-tospacer adjacent motif)序列为NGD (NGA、NGT和NGG)的靶序列中产生碱基编辑, 而在PAM为NGC的靶序列中编辑效率很低。相较于STEME-NG, STEME-1可在PAM为NGG的靶序列上产生更高的碱基编辑效率。
本文以水稻为例, 介绍靶向随机突变水稻基因的具体操作流程, 并从操作过程中靶点设计和突变体检测两方面进行详细阐述。
1 实验材料
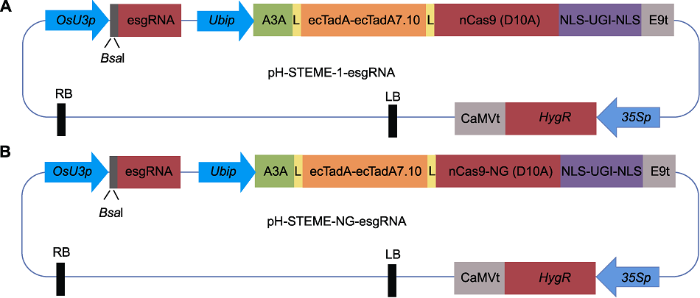
水稻(Oryza sativa L.)愈伤组织及本课题组开发的用于水稻靶向随机突变的双元载体pH-STEME-1-esg RNA (图1A)和pH-STEME-NG-esgRNA (图1B) (Li et al., 2020) (可以向本课题组索取, 也可通过Addgene (图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1靶向随机突变的双元载体图
(A) pH-STEME-1-esgRNA; (B) pH-STEME-NG-esgRNA。OsU3p: 水稻OsU3启动子; Ubip: 玉米Ubi-1启动子; A3A: 胞嘧啶脱氨酶APOBEC3A; ecTadA-ecTadA7.10: 腺嘌呤脱氨酶ecTadA-ecTadA7.10; NLS: 核定位信号; UGI: 尿嘧啶DNA糖基化酶抑制剂; E9t: 豌豆rbcS-E9终止子; 35Sp: 花椰菜花叶病毒35S启动子; HygR: 潮霉素抗性基因; CaMVt: 花椰菜花叶病毒终止子信号; LB: T-DNA左臂重复序列; RB: T-DNA右臂重复序列; L: 连接序列
Figure 1Architectures of the binary vectors
(A) pH-STEME-1-esgRNA; (B) pH-STEME-NG-esgRNA. OsU3p: Rice OsU3 promoter; Ubip: Maize Ubi-1 promoter; A3A: Cytidine deaminase APOBEC3A; ecTadA-ecTadA7.10: Adenosine deaminase ecTadA-ecTadA7.10; NLS: Nuclear localization signal; UGI: Uracil DNA glycosylase inhibitor; E9t: Pea rbcS-E9 terminator; 35Sp: Cauliflower mosaic virus 35S promoter; HygR: Hygromycin resistance gene; CaMVt: Cauliflower mosaic virus terminator; LB: T-DNA left border repeat; RB: T-DNA right border repeat; L: Linker sequence
2 试剂
试剂包括BsaI限制性内切酶、大肠杆菌感受态细胞(XL10)、根癌农杆菌(Agrobacterium tumefaciens)感受态细胞(AGL1)、水稻愈伤组织转化相关试剂及其它生物学常用试剂。3 仪器设备
仪器设备包括台式离心机、超净工作台、电击转化仪和移液器等。4 实现植物基因靶向随机突变的设计思路
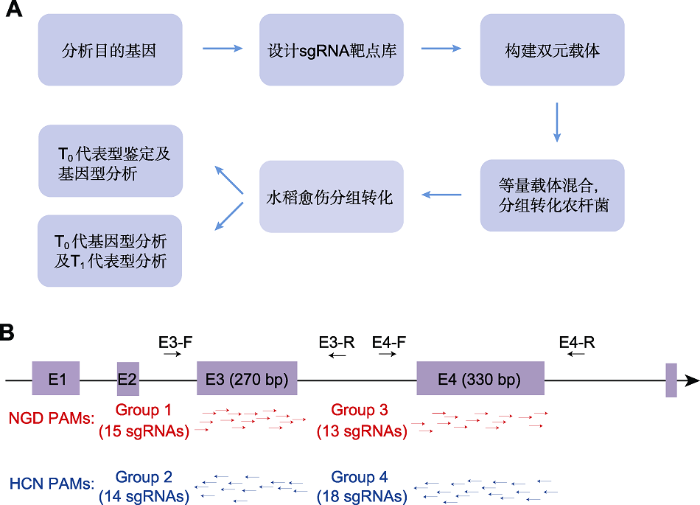
利用STEME-1和STEME-NG两个编辑载体对目的基因进行靶向随机突变, 需要先对目的基因序列进行分析, 并综合STEME-1和STEME-NG所识别PAM序列及碱基编辑窗口范围特性, 设计覆盖整个目的基因的靶点sgRNA, 构建水稻农杆菌转化载体; 根据sgRNA的方向和位置, 将载体分组并转化农杆菌感受态; 农杆菌转化水稻愈伤组织后, 对再生植物进行基因型或表型鉴定(图2A)。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2靶向随机突变的实验流程及sgRNA靶点库设计示例
(A) 靶向随机突变水稻基因的实验流程; (B) sgRNA靶点库设计示例。确定目标基因靶向随机突变区域为第3、第4外显子E3和E4 (270和330 bp), 设计60个sgRNA靶点并分为4组。其中组1、组2靶点PAM序列分别为NGD和HCN, 可由引物E3-F/E3-R扩增获得长度约700 bp的片段, 以便后续使用T7E1酶切法检测突变体植株; 组3、组4靶点PAM序列分别为NGD和HCN, 可由引物E4-F/E4-R扩增获得长度约700 bp的片段, 以便后续使用T7E1酶切法检测突变体植株。
Figure 2Schematic of the procedure for targeted random mutagenesis and illustration of design for sgRNA pool
(A) Schematic of the procedure for targeted random mutagenesis; (B) Illustration of design for sgRNA pool. Chose the target region of 3rd and 4th exons (E3: 270 bp and E4: 330 bp), then design 60 sgRNAs divided into 4 groups. sgRNAs in group 1 and 2 target NGD and HCN PAM sequences and the target region (700 bp) can be amplified using primers E3-F/E3-R. sgRNAs in group 3 and 4 target NGD and HCN PAM sequences and the target region (700 bp) can be amplified using primers E4-F/E4-R. Amplicons are about 700 bp and suitable for T7E1 assay.
5 实验流程
5.1 靶向随机突变靶点的设计
5.1.1 对目的基因进行分析对目的基因进行靶向随机突变, 首先要对目的基因的结构功能进行分析, 根据现有基因的研究进展, 确定靶向随机突变目的基因的最小区域。
5.1.2 靶点的设计
STEME产生的突变主要为碱基替换, 部分靶点会产生低频的碱基插入或缺失。由于碱基插入或缺失发生的频率较低, 因此设计sgRNA时只需要考虑碱基替换。根据氨基酸变化结果碱基突变分为3种: 错义突变、沉默突变和无义突变。错义突变是指DNA的突变引起mRNA中密码子改变, 编码另一种氨基酸; 沉默突变也称同义突变, 是指DNA的突变虽引起mRNA中密码子改变为另一种密码, 但由于密码子的简并性, 并未使编码的氨基酸改变; 无义突变指DNA的突变引起mRNA中的密码子改变为终止密码子。
如果设定sgRNA离PAM最远端的碱基位置为+1, 则STEME碱基编辑窗口为C1-C17和A4-A8。此外, STEME-1和STEME-NG所识别的PAM序列分别为NGG和NGD。基于STEME的编辑特性, 在靶基因区域设计多个sgRNA, 以每个sgRNA均可产生不同类型的错义突变为宜。
下面以STEME-1为例, 详述具体的设计步骤:
(1) 在目的基因选定区域查找所有满足PAM序列为NGG且在1-17位包含胞嘧啶C或4-8位包含腺嘌呤A的可能sgRNA靶点序列;
(2) 根据C>T/G/A和A>G的突变原则, 分析可能的sgRNA靶点产生的氨基酸突变类型, 剔除产生重复氨基酸突变、沉默突变和无义突变的sgRNA靶点;
(3) 在靶基因选定区域查找所有满足PAM序列为CCN且在1-17位包含鸟嘌呤G或4-8位包含胸腺嘧啶T的可能sgRNA靶点序列;
(4) 根据G>A/C/T和T>C的突变原则, 分析可能的sgRNA靶点产生的氨基酸突变类型, 剔除产生重复氨基酸突变、沉默突变和无义突变的sgRNA靶点。
(2)和(4)步分析所得的sgRNA总和即构成有效的sgRNA靶点库。
此外, STEME编辑工具的窗口虽为C1-C17和A4-A8, 但在编辑窗口外沿位置的编辑效率较低。例如, C1和C17位的编辑效率要低于处于编辑窗口中心位置的C5-C13的效率。因此, 在设计靶点时, 可设定更小的碱基编辑窗口范围参数(如C5-C13和A5-A7), 以提高整体靶向随机突变的效率; 针对邻近的sgRNA产生相同氨基酸突变的情况, 选择可突变碱基处于编辑窗口中心位置的sgRNA, 以提高整体靶向随机突变的效率。STEME编辑工具虽然可产生C>G/A或G>C/T的突变, 但相较于C:G>T:A突变的概率低。因此, 在剔除产生重复氨基酸突变、沉默突变和无义突变的sgRNA靶点时, 可仅考虑C:G>T:A和A:T>G:C两种碱基突变类型, 以提高整体靶向随机突变的效率。针对可能同时产生错义突变和无义突变的sgRNA靶点, 可根据具体实验需求确定sgRNA的选择。如期望获得更多的氨基酸突变类型, 可选择保留该sgRNA。STEME-NG在PAM序列为NGC的靶序列产生碱基编辑效率很低。因此, 设计sgRNA时, 可只考虑STEME-NG所偏好的NGD PAM序列。
5.2 载体的构建与转化
(1) 载体的构建STEME编辑器包括STEME-1和STEME-NG两种, 所识别PAM序列分别为NGG和NGD。STEME- NG识别的PAM序列范围包含STEME-1所识别的NGG PAM, 因此可只使用STEME-NG进行sgRNA的构建。由于相较于STEME-NG, STEME-1可以在PAM为NGG的靶序列上产生更高的碱基编辑效率, 故在载体构建时也可以选择同时使用STEME-1和STEME-NG, 即在PAM为NGA和NGT的靶序列中使用STEME-NG, 在PAM为NGG的靶序列中使用STEME-1。
载体具体构建方法是使用BsaI限制性内切酶将双元载体pH-STEME-1-esgRNA或pH-STEME-NG- esgRNA酶切后, 分别与退火后的sgRNA靶点序列进行连接, 随后将连接产物转化大肠杆菌感受态(大肠杆菌感受态菌株推荐使用XL10, 确保外源DNA质粒的稳定性), 经PCR和测序验证正确后, 逐一提取载体质粒以备农杆菌转化。
(2) 载体转化农杆菌感受态细胞
如果靶向目的基因所设计的sgRNA数量较少, 可将质粒载体逐一转化农杆菌感受态后, 分别进行水稻愈伤组织转化。
如果靶向目的基因所设计的sgRNA数量较多, 可将质粒载体逐一转化农杆菌感受态后, 分别进行水稻愈伤组织转化; 也可将sgRNA靶点库划分为若干小组, 每组包含多个质粒载体, 将组内多个质粒载体等比例混合后电击转化农杆菌感受态, 再分组进行水稻愈伤组织转化。
分组转化可根据所设计的sgRNA数量、sgRNA靶点的PAM序列方向和后续检测方案等实际情况进行具体划分; 遵循的原则是在保证较高转化效率和获得足量突变体植株的前提下, 最大限度地简化植物转化、突变体检测和目的突变体的获得过程。如后续使用T7E1检测法对突变体进行检测, 可将长度约400 bp的目标区域内的sgRNA划分为一组; 如拟使用Hi-TOM (Liu et al., 2019)等高通量测序方法对突变体进行检测, 可将长度约200 bp的目标区域内的sgRNA划分为一组。
以使用T7E1检测法为例, 假设以某一基因的2个外显子序列为目标突变区域, 共设计60个sgRNA靶点, 则可将sgRNA靶点简单划分为4个小组(图2B)。其中, 组1和组2的sgRNA在一个PCR扩增子内, PAM序列分别为NGD和HCN; 组3和组4的sgRNA在另一个PCR扩增子内, PAM序列分别为NGD和HCN。每组内的sgRNA均可通过一个PCR扩增子(约700 bp)进行后续T7E1酶切法检测突变体植株; 每组内sgRNA的PAM序列方向相同, 避免多个质粒进入单个农杆菌细胞后不同PAM方向的sgRNA在nCas9 (D10A)作用下发生靶序列DNA双链断裂。
(3) 农杆菌转化植物
水稻农杆菌转化可参考Shan等(2013)和Meng 等(2017)的操作方法。针对包含多个质粒载体的分组转化方法, 等比例增加起始转化水稻愈伤组织的数目, 可确保再生植株群体中包含所有转化质粒。
5.3 植物靶向随机突变的检测及表型鉴定
在获得阳性T0转化植株后, 可根据靶基因的特性和实验目的对突变体进行鉴定及表型分型。具体可分为两种情况, 一种为T0代可直接进行表型鉴定, 另一种是先在T0代转化植株中检测出突变体, 之后经种植保留突变体, 再对其后代进行表型鉴定。(1) T0代进行表型鉴定及目的突变体基因型分析
如果目的基因突变体可在T0代进行表型鉴定, 可根据表型鉴定结果直接从阳性转化植株中确定所需突变体。根据突变体的靶点序列或所在的转化分组信息进行PCR扩增, 经桑格测序即可确定目的突变体的基因型。与此同时, 可挑取几十个单个sgRNA转化植株或1-2个分组转化植株, 进行T7E1酶切或使用Hi-Tom等高通量测序方法进行检测, 以评估本次靶向随机突变实验产生突变体的效率。
(2) T1代进行表型鉴定及目的突变体基因型分析
如果目标基因突变体的表型鉴定不能在T0代进行, 则需要先从阳性的T0转化植株中检测出突变体, 然后经种植产生后代再进行表型鉴定。T0转化植株中突变体的检测包括混池检测和单株检测两步: 第1步, 将T0植株样品以3-6株混合取样, 使用T7E1酶切法或Hi-tom等高通量测序方法检测每个混样中是否存在突变体, 剔除无突变体的混样编号; 第2步, 选取存在突变体的混样编号, 再次使用T7E1酶切法或Hi-tom检测法进行单株鉴定, 可根据实验需求, 决定是否对T7E1酶切法阳性单株突变体进行基因型鉴定。将所有T0突变体通过种植保留下来, 并使用后代进行表型鉴定。表型鉴定确定所需目的突变体后, 可使用桑格测序等方法再次确认具体的基因型。
6 注意事项
基于双碱基编辑系统STEME的植物基因靶向随机突变技术, 可在植物原位进行基因突变及功能筛选的定向进化, 可应用于植物快速育种及重要功能基因研究, 该技术也有望应用于不同细胞系、酵母或动物中的非编码区顺式作用元件的调控、动物致病SNV的修正和抗药位点的筛选等。本文所介绍的STEME植物转化载体pH-STEME-1-esgRNA和pH-STEME-NG- esgRNA适用于水稻转化。如使用STEME系统对其它植物基因进行靶向随机突变, 需对STEME载体启动子和遗传转化标记基因等元件进行改造, 以适配目标突变植物。STEME系统在使用过程中, 可根据具体实验需求对sgRNA库的设计参数进行调整, 如设计sgRNA时设定的PAM序列、编辑窗口范围和碱基编辑类型。如果期望获得更多的氨基酸突变类型, 可适度放宽sgRNA设计条件, 增加植物转化数量以获得低频突变类型。
现有的STEME系统是基于胞嘧啶脱氨酶APOBEC3A、腺嘌呤脱氨酶ecTadA-ecTadA7.10、nCas9或nCas9-NG。如果期望获得更高的编辑效率和更广的编辑范围, 可使用更高效的脱氨酶和PAM识别序列更广的nCas9变体进行优化。
(责任编辑: 孙冬花)
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
DOI:10.1038/nbt.4199URLPMID:30059493 [本文引用: 1]

Base editor technology, which uses CRISPR-Cas9 to direct cytidine deaminase enzymatic activity to specific genomic loci, enables the highly efficient introduction of precise cytidine-to-thymidine DNA alterations. However, existing base editors create unwanted C-to-T alterations when more than one C is present in the enzyme's five-base-pair editing window. Here we describe a strategy for reducing bystander mutations using an engineered human APOBEC3A (eA3A) domain, which preferentially deaminates cytidines in specific motifs according to a TCR>TCY>VCN hierarchy. In direct comparisons with the widely used base editor 3 (BE3) fusion in human cells, our eA3A-BE3 fusion exhibits similar activities on cytidines in TC motifs but greatly reduced editing on cytidines in other sequence contexts. eA3A-BE3 corrects a human beta-thalassemia promoter mutation with much higher (>40-fold) precision than BE3. We also demonstrate that eA3A-BE3 shows reduced mutation frequencies on known off-target sites of BE3, even when targeting promiscuous homopolymeric sites.
[本文引用: 1]
URLPMID:31932727 [本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1111/tpj.13469URLPMID:28027431 [本文引用: 1]

Production of mutants of crop plants by the use of chemical or physical genotoxins has a long tradition. These factors induce the natural DNA repair machinery to repair damage in an error-prone way. In the case of radiation, multiple double-strand breaks (DSBs) are induced randomly in the genome, leading in very rare cases to a desirable phenotype. In recent years the use of synthetic, site-directed nucleases (SDNs) - also referred to as sequence-specific nucleases - like the CRISPR/Cas system has enabled scientists to use exactly the same naturally occurring DNA repair mechanisms for the controlled induction of genomic changes at pre-defined sites in plant genomes. As these changes are not necessarily associated with the permanent integration of foreign DNA, the obtained organisms per se cannot be regarded as genetically modified as there is no way to distinguish them from natural variants. This applies to changes induced by DSBs as well as single-strand breaks, and involves repair by non-homologous end-joining and homologous recombination. The recent development of SDN-based 'DNA-free' approaches makes mutagenesis strategies in classical breeding indistinguishable from SDN-derived targeted genome modifications, even in regard to current regulatory rules. With the advent of new SDN technologies, much faster and more precise genome editing becomes available at reasonable cost, and potentially without requiring time-consuming deregulation of newly created phenotypes. This review will focus on classical mutagenesis breeding and the application of newly developed SDNs in order to emphasize similarities in the context of the regulatory situation for genetically modified crop plants.
[本文引用: 1]
[本文引用: 1]
URLPMID:15580263 [本文引用: 1]
[本文引用: 1]
Applications of protein engineering and directed evolution in plant research
1
2019
... 遗传变异是自然界物种形成和进化的基础, 也是物种性状多样性产生的原因.目前, 在作物中通过突变筛选优异性状的方法主要是诱变育种, 即通过物理及化学方法(如辐射和EMS诱变)产生全基因组的随机突变, 再从突变体中选取表现优异的个体进行新品种的培育(
Programmable base editing of A?T to G?C in genomic DNA without DNA cleavage
1
2017
... 基于双碱基编辑系统的植物基因靶向随机突变技术所使用的基因编辑工具称为“饱和靶向内源基因突变编辑器(saturated targeted endogenous mutagenesis editor, STEME)”, 主要包括STEME-1和STEME-NG两种(
An APOBEC3A-Cas9 base editor with minimized bystander and off-target activities
1
2018
... 基于双碱基编辑系统的植物基因靶向随机突变技术所使用的基因编辑工具称为“饱和靶向内源基因突变编辑器(saturated targeted endogenous mutagenesis editor, STEME)”, 主要包括STEME-1和STEME-NG两种(
TILLING. Traditional mutagenesis meets functional genomics
1
2004
... 遗传变异是自然界物种形成和进化的基础, 也是物种性状多样性产生的原因.目前, 在作物中通过突变筛选优异性状的方法主要是诱变育种, 即通过物理及化学方法(如辐射和EMS诱变)产生全基因组的随机突变, 再从突变体中选取表现优异的个体进行新品种的培育(
Targeted, random mutagenesis of plant genes with dual cytosine and adenine base editors
3
2020
... 遗传变异是自然界物种形成和进化的基础, 也是物种性状多样性产生的原因.目前, 在作物中通过突变筛选优异性状的方法主要是诱变育种, 即通过物理及化学方法(如辐射和EMS诱变)产生全基因组的随机突变, 再从突变体中选取表现优异的个体进行新品种的培育(
... 基于双碱基编辑系统的植物基因靶向随机突变技术所使用的基因编辑工具称为“饱和靶向内源基因突变编辑器(saturated targeted endogenous mutagenesis editor, STEME)”, 主要包括STEME-1和STEME-NG两种(
... 水稻(Oryza sativa L.)愈伤组织及本课题组开发的用于水稻靶向随机突变的双元载体pH-STEME-1-esg RNA (
Expanded base editing in rice and wheat using a Cas9-adenosine deaminase fusion
1
2018
... 基于双碱基编辑系统的植物基因靶向随机突变技术所使用的基因编辑工具称为“饱和靶向内源基因突变编辑器(saturated targeted endogenous mutagenesis editor, STEME)”, 主要包括STEME-1和STEME-NG两种(
Hi-TOM: a platform for high-throughput tracking of mutations induced by CRISPR/Cassystems
1
2019
... 分组转化可根据所设计的sgRNA数量、sgRNA靶点的PAM序列方向和后续检测方案等实际情况进行具体划分; 遵循的原则是在保证较高转化效率和获得足量突变体植株的前提下, 最大限度地简化植物转化、突变体检测和目的突变体的获得过程.如后续使用T7E1检测法对突变体进行检测, 可将长度约400 bp的目标区域内的sgRNA划分为一组; 如拟使用Hi-TOM (
Construction of a genome-wide mutant library in rice using CRISPR/Cas9
1
2017
... 水稻农杆菌转化可参考
Engineered CRISPR-Cas9 nuclease with expanded targeting space
1
2018
... 基于双碱基编辑系统的植物基因靶向随机突变技术所使用的基因编辑工具称为“饱和靶向内源基因突变编辑器(saturated targeted endogenous mutagenesis editor, STEME)”, 主要包括STEME-1和STEME-NG两种(
From classical mutagenesis to nuclease-based breeding-directing natural DNA repair for a natural end-product
1
2017
... 遗传变异是自然界物种形成和进化的基础, 也是物种性状多样性产生的原因.目前, 在作物中通过突变筛选优异性状的方法主要是诱变育种, 即通过物理及化学方法(如辐射和EMS诱变)产生全基因组的随机突变, 再从突变体中选取表现优异的个体进行新品种的培育(
Methods for the directed evolution of proteins
1
2015
... 遗传变异是自然界物种形成和进化的基础, 也是物种性状多样性产生的原因.目前, 在作物中通过突变筛选优异性状的方法主要是诱变育种, 即通过物理及化学方法(如辐射和EMS诱变)产生全基因组的随机突变, 再从突变体中选取表现优异的个体进行新品种的培育(
Rapid and efficient gene modification in rice and Brachypodium using TALENs
1
2013
... 水稻农杆菌转化可参考
A reverse genetic, nontransgenic approach to wheat crop improvement by TILLING
1
2005
... 遗传变异是自然界物种形成和进化的基础, 也是物种性状多样性产生的原因.目前, 在作物中通过突变筛选优异性状的方法主要是诱变育种, 即通过物理及化学方法(如辐射和EMS诱变)产生全基因组的随机突变, 再从突变体中选取表现优异的个体进行新品种的培育(
Efficient C-to-T base editing in plants using a fusion of nCas9 and human APOBEC3A
1
2018
... 基于双碱基编辑系统的植物基因靶向随机突变技术所使用的基因编辑工具称为“饱和靶向内源基因突变编辑器(saturated targeted endogenous mutagenesis editor, STEME)”, 主要包括STEME-1和STEME-NG两种(
