 ,*浙江师范大学, 化学与生命科学学院, 金华 321004
,*浙江师范大学, 化学与生命科学学院, 金华 321004Design and Practice of a New Teaching Project of the Map-based Cloning Experiment in Genetics
Xifeng Chen, Yaping Liu, Bojun Ma ,*College of Chemistry and Life Science, Zhejiang Normal University, Jinhua 321004, China
,*College of Chemistry and Life Science, Zhejiang Normal University, Jinhua 321004, China通讯作者:
责任编辑: 孙冬花
收稿日期:2019-06-11接受日期:2019-09-17网络出版日期:2019-11-01
| 基金资助: |
Corresponding authors:
Received:2019-06-11Accepted:2019-09-17Online:2019-11-01

摘要
关键词:
Abstract
Keywords:
PDF (1141KB)摘要页面多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
陈析丰, 刘亚萍, 马伯军. 图位克隆技术新型遗传学实验教学项目的设计与实践. 植物学报, 2019, 54(6): 797-803 doi:10.11983/CBB19107
Chen Xifeng, Liu Yaping, Ma Bojun.
遗传学是当今生命科学领域一门重要的基础且前沿学科, 是生物学专业综合性较强的核心课程, 也是高等院校本科学习整个理论体系中的后续课程。遗传学具有很强的理论性和实践性, 内容跨度大且抽象、复杂, 学生不易理解, 只有使用多元化教学手段, 才能提高教学效率(杜少陵等, 2010)。教学与科研相结合是教学多元化的一种重要途径。将科研的最新进展和先进技术有机地融入教学中, 帮助学生掌握教学内容的同时, 又能让学生了解科学前沿的最新动态, 从而为其今后考研深造打下良好的基础。
图位克隆(map-based cloning)又称定位克隆(positional cloning), 由剑桥大学的Coulson等(1986)首先提出。该技术是利用遗传图谱上已知的染色体位置标记, 分析其与某一质量性状基因的连锁关系, 对该未知DNA序列的目的基因进行精细定位; 之后利用物理图谱通过染色体步移(chromosome walking)逐渐接近目的基因, 最终确定目的基因并通过遗传转化实验验证目的基因的功能(景润春等, 2000)。前期, 图位克隆主要依靠形态学标记、细胞学标记和生化标记, 但这些标记数目偏少且不稳定。随着RFLP (restriction fragment length polymorphism)、AFLP (amplified fragment length polymorphism)、RAPD (random amplified polymorphic DNA)、SSR (simple sequence repeats)、STS (sequence-tagged site)和SNP (single nucleotide polymorphism)等分子标记的不断更新及基因组测序技术的迅速发展, 图位克隆已成为当今植物重要性状调控基因分离的主要方法之一, 同时也是直接解析遗传重组与性状表型之间关系的重要技术, 在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、小麦(Triticum aestivum)、番茄(Solanum lycopersicum)和玉米(Zea mays)等植物中被广泛应用(景润春等, 2000; 何俊平等, 2010)。此外, 图位克隆技术也应用于数量性状位点(quantitative trait loci, QTL)基因的克隆, 通过回交构建各种定位群体, 将多个QTL分解为单个孟德尔因子, 再利用图位克隆技术进行分离和克隆(姜树坤等, 2008)。
鉴于图位克隆涉及遗传学中基因重组、连锁互换和遗传作图等学生较难理解的相关知识, 且涉及多项遗传学和分子生物学实验技术, 故掌握了图位克隆的技术原理可大大提高学生对遗传学重点知识的理解。我们前期利用图位克隆技术在水稻7号染色体上精细定位并克隆了1个调控类病变(lesion mimic)表型的基因SPL5 (Chen et al., 2009, 2012)。基于该项科研成果, 根据图位克隆的技术原理, 我们设计了一个新的综合型遗传学实验, 即利用DNA分子标记对基因进行定位, 旨在进一步加强遗传学课程教学, 切实提高大学生的自主学习、实践和科研能力, 积极推进遗传学教学的改革与创新。
1 教学实验设计
1.1 实验目的
(1) 从DNA分子水平理解染色体的分离与自由组合、连锁与互换规律;(2) 了解DNA分子标记及其多态性分析方法;
(3) 掌握基于图位克隆技术的基因连锁分析与初步定位。
1.2 实验原理
图位克隆技术是基于孟德尔遗传学定律, 通过寻找与目标基因紧密连锁的分子标记, 对调控某一表型的单个基因进行精确定位。根据孟德尔定律, 如果2个非等位基因不连锁(即位于不同染色体上), 在减数分裂过程中这2个非等位基因会发生自由组合, 产生重组基因型, 且重组率接近50%; 如2个非等位基因连锁(即位于同一染色体上), 在减数分裂过程中这2个非等位基因不能自由组合, 但可以通过染色体交换产生重组基因型, 而重组率远小于50%, 且2个基因距离越近, 重组率越低, 紧密连锁的2个基因几乎不发生交换, 重组率接近0%。本质上, 基因和分子标记都是染色体上的一段DNA序列, 两者在减数分裂过程中都有可能通过自由组合或连锁互换发生重组。因此, 利用染色体上已知位置的分子标记, 在减数分裂后分析不同位点的分子标记与目标基因的重组率, 就可对目标基因进行染色体连锁分析及精细定位。如图1所示, A与a为1对等位基因, 即未知序列的目标基因, A对于a为显性, AA、Aa为显性表型, aa为隐性表型。M与m为染色体上已知位置的分子标记, M与m位于同一位点, 但两者DNA序列存在碱基替换或片段长度差异, 可通过分子生物学手段鉴定M与m。假设基因型AAMM和aamm作为两亲本(P)进行杂交, 获得杂交后代F1的基因型为AaMm; F1减数分裂过程中染色体发生重组, 自交产生的F2群体中AA、Aa基因型都为显性表型, 无法区分, 而aa基因型为隐性表型, 可直接判断。因此, 取F2群体中的隐性个体(aa)用于基因定位分析: 如果A/a与M/m在不同染色体上(不连锁), 则减数分裂过程中A/a与M/m发生自由组合, F2群体aa基因型个体中MM、Mm与mm的比例接近1:2:1; 如果A/a与M/m在同一染色体上(连锁), 则减数分裂过程中基因A/a与M/m不能自由组合, A与M一起分离, a与m一起分离, 除非基因与标记之间发生染色体交换, 而发生交换的重组率远小于自由组合的重组率。因此, F2群体aa基因型个体中mm很多, Mm很少, MM更少, 而且Mm+MM的出现概率反映了分子标记与目标基因的重组率, 发生概率越低, 分子标记与目标基因的重组率越低, 分子标记就越接近目标基因。可见, 通过鉴定F2群体的隐性(aa)个体中分子标记MM、Mm与mm的比例, 即可对目的基因A/a进行染色体连锁分析与定位。
图1
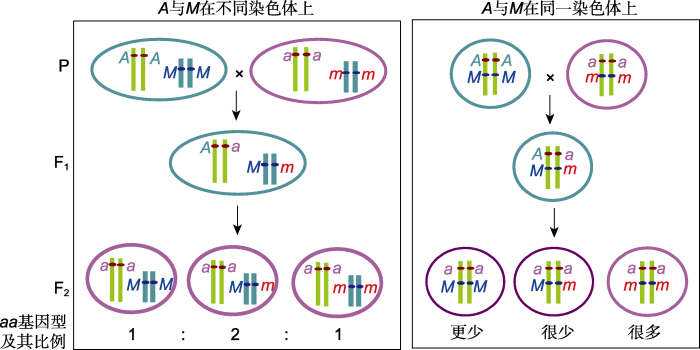 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1减数分裂过程中目标基因与分子标记的重组方式
A与a代
Figure 1Recombination patterns between the target gene and the molecular markers during meiosis
A and a represent a pair of dominant and recessive alleles, of which AA and Aa show dominant phenotypes and aa shows a recessive phenotype; M and m represent a given molecular marker at the same locus derived from the dominant parent (AA) and the recessive parent (aa), respectively.
1.3 实验材料
类病变指一类在没有逆境或病原物侵害时就能自发形成类似病变坏死斑的植物突变体。spl5 (spotted leaf 5)是典型的水稻类病变突变体, 其叶片上有明显的红褐色斑点, 甚至布满整个叶片(图2A)。本实验以粳稻(O. sativa L. ssp. janponica)品种日本晴背景的spl5突变体(aa基因型) (图2A, B)和籼稻(O. sativa L. ssp. indica)野生型品种9311 (AA基因型)为材料(图2C)。将spl5与9311进行杂交, 获得F1代(Aa基因型)并进一步自交, 得到F2代群体。由于基因的连锁分析涉及水稻所有染色体, 分子标记的PCR扩增工作量较大, 故一般会先选择10-20个F2代隐性单株进行基因的初步定位。从F2代群体中选取20个spl5突变型单株(aa基因型)及其亲本9311与spl5突变体作为实验材料, 用于后续分析(图2C)。图2
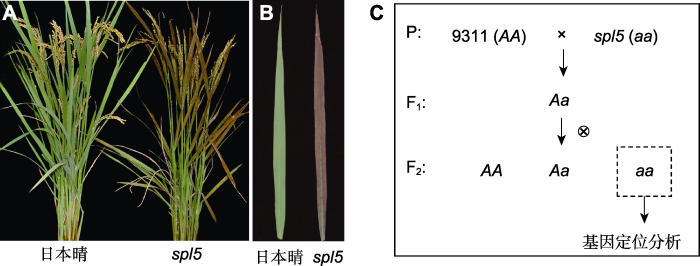 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2水稻类病变spl5突变体的表型与基因定位群体的构建
(A) 对照粳稻日本晴及spl5突变体植株表型; (B) 日本晴及spl5突变体叶片表型; (C) 野生型籼稻品种9311与粳稻日本晴背景的spl5突变体杂交构建F2代定位群体流程。
Figure 2The phenotype of the rice spl5 lesion mimic mutant and a schematic workflow of generation of the genetic mapping population
(A) The plant phenotype of wild-type control Nipponbare, a Japonica rice cultivar, and the spl5 mutant; (B) The leaf phenotype of wild-type control and the spl5 mutant; (C) A workflow showing the generation of an F2 mapping population by crossing an Indica cultivar 9311 with the spl5 mutant.
1.4 实验方法
1.4.1 目标基因的遗传分析根据2个亲本的表型特征, 对F1代植株和F2群体植株的表型进行鉴定, 区分野生型与突变型, 用卡方检验(公式(1))分析F2群体中野生型与突变型的分离比例是否符合3:1, 确定目标基因与表型的显、隐性关系及是否为单基因调控。
1.4.2 目标基因定位群体的PCR分析
取亲本9311、spl5突变体及其杂交F2代20个突变型单株的新鲜叶片, 分别提取其基因组DNA, 参考赵国珍等(2012)的方法。选取6个不同的SSR分子标记(其PCR引物序列见表1)分别对提取的DNA进行PCR扩增, 可采用PCR试剂盒(如2×Taq PCR预混试剂II, 天根生物), PCR体系根据产品使用说明配制。扩增程序为: 94°C预变性3分钟; 94°C变性30秒, 55°C退火30秒, 72°C延伸30秒, 35个循环; 72°C延伸5分钟。采用3%琼脂糖凝胶电泳30-60分钟, 电压100 v。结束后, 将凝胶放入Gel-red荧光染料溶液中染色5分钟, 在凝胶成像仪中拍照。
Table 1
表1
表1本研究SSR标记的PCR引物
Table 1
| SSR标记 | 正向引物 (5°-3°) | 反向引物 (5°-3°) |
|---|---|---|
| RM21103 | ACAAGTCCACAAGGACCACAACC | TGCTCCACCCAAAGATACAGAGC |
| RM21309 | GAGTGGATTTACTGACAAGCTCTTCC | GATTCTCTTGGAGCCCATACTCC |
| RM21470 | TCTTGCCATCACATAGCAACAGG | ACTCGGTGAGCATCCAATGTCC |
| RM22426 | ATTTCGTCGTCGTGTCTGTCC | TTAAAGCAGCCTGATCGAAAGC |
| RM22648 | AGCAGCTCCGATTGTGTTAGCC | AATGGCGTCTGTGGTACTTTGC |
| RM22925 | CGATTACCAGGACAACAGATCACG | GTGGCTCTTGAGTGTACGTGTGC |
新窗口打开|下载CSV
1.4.3 目标基因的连锁分析与初步定位
(1) 根据水稻基因组的SSR标记信息(International Rice Genome Sequencing Project, 2005), 搜索本实验SSR标记所在的染色体及其位置。
(2) 比较20个F2代突变型单株与2个亲本的电泳条带, 确定每个SSR标记与目标基因间是否发生染色体重组及其基因型: 仅含有spl5突变体带型的个体为非重组型, 无配子发生重组, 基因型为mm; 同时含有2个亲本带型的为单染色体重组型, 其中1个配子发生过重组, 基因型为Mm; 仅含有9311野生型带型的个体为双染色体重组型, 2个配子均发生过重组, 基因型为MM。
(3) 用卡方检验(公式(1))验证每个SSR标记的MM、Mm和mm基因型是否符合1:2:1比例; 同时, 根据遗传重组的基本公式(公式(2)), 计算每个SSR标记与目标基因的重组率。
(4) 根据每个SSR标记与目标基因的重组率和卡方检验结果, 确定与目标基因连锁的SSR标记, 将目的基因定位在水稻某个染色体上的2个标记之间, 并绘制染色体连锁图。
式中, O为观察值, E为预期值。
1.5 实验安排与材料准备
本实验分3次课, 共9课时。由于是综合设计性实验, 工作量相对较大, 建议4-6个学生组成1个小组, 共同完成实验。不同小组使用不同的F2代突变型单株。DNA提取、PCR及电泳等经典实验技术, 在《分子生物学实验》课上都已学过, 因此由学生自己设计。第1次课(3课时), 提取水稻叶片DNA, 包括2个亲本和20个F2代突变型单株, DNA样品于-20°C保存备用; 第2次课(3课时), 用表1所示的6个SSR标记分别对22个DNA样品进行PCR扩增, PCR产物4°C保存备用; 第3次课(3课时), 对132 (6×22)个PCR样品进行电泳检测, 通过电泳条带的统计分析, 计算每个SSR标记与目标基因的重组率, 对目标基因进行连锁分析和初步定位, 然后由各小组汇报实验结果, 教师进行点评, 并针对学生在实验中遇到的问题展开讨论, 提高学生解决问题的能力。由于前期的材料准备需要进行水稻杂交和繁种, 相对比较复杂。如果按照每次实验课平均5个小组计算, 则需要100个F2代的突变型单株。1株水稻F1代植株上可收获几百到上千粒种子(即F2代群体), 发育成植株后, 每株的叶子足够提取上百次DNA。因此, 只需获得几株F1代, 就可收获2 000多粒种子(F2代)。将种子播种在苗床上, 在苗期就可根据叶片的类病变表型将F2代突变型单株筛选出来; 按照10 cm×10 cm进行密植插秧, 只需面积为1 m×5 m的大田, 就能够获得可同时供5次平行实验课使用的500个F2代的突变型单株。可见, 虽然前期材料准备周期长, 水稻杂交、自交至少需要两季, 但是工作量并不多, 如果有-80°C低温储存条件, 1次准备的材料可供很多年使用, 效率极高。
科研工作中, 基因初步定位一般采用覆盖所有染色体的分子标记进行分析, 因此涉及的标记较多, PCR扩增和电泳的工作量较大。但对于本科实验教学来说, 每个实验的课时非常有限, 如何合理安排很关键。目前, 已公布的水稻SSR标记有上万个, 完全覆盖了水稻的12条染色体。由于SPL5基因在水稻7号染色体上, 因此本实验在挑选SSR标记时, 选择了与目标基因连锁的7号染色体上的3个SSR标记(分布于目标基因的两侧, 且与目标基因的连锁程度不同)。此外, 考虑到学生实验课的工作量不能太大, 本实验只随机挑选了1条与目标基因不连锁的染色体(8号); 且选择了8号染色体上的3个SSR标记(其相互间距离不宜太近, 避免紧密连锁)。以上所选的SSR标记必须在亲本9311和spl5突变体间具有较好的多态性, 可用琼脂糖凝胶电泳分离鉴定。
1.6 实验结果与讨论
1.6.1 实验结果(1) 学生根据2个水稻亲本植株的表型区分野生型与spl5突变型, 并对其杂交F1代与F2代群体植株的表型进行鉴定和数量统计。结果为F1代植株表现野生型, F2代群体中既有野生型也有spl5突变型, 且野生型与突变型的比例接近3:1, 经卡方检验分析其表型分离比符合单基因控制的遗传规律, 表明spl5突变表型受单个隐性基因的控制, 可以采用图位克隆技术对目的基因进行定位。
(2) 通过查阅文献(International Rice Genome Sequencing Project, 2005), 学生可确定本实验SSR标记所在的染色体及其物理位置(图3A)。用选取的6个SSR标记对20个F2代突变型单株及其亲本进行PCR扩增和电泳分析(图3B)。针对每一SSR标记, 确定每个F2代突变型单株在该SSR标记位点的基因型: mm (非重组)、Mm (单染色体重组)和MM (双染色体重组), 并将重组率的基本公式(2)转换成公式(3), 之后将该SSR标记的各基因型个体数代入公式(3), 计算出该SSR标记与目的基因的重组率。
图3
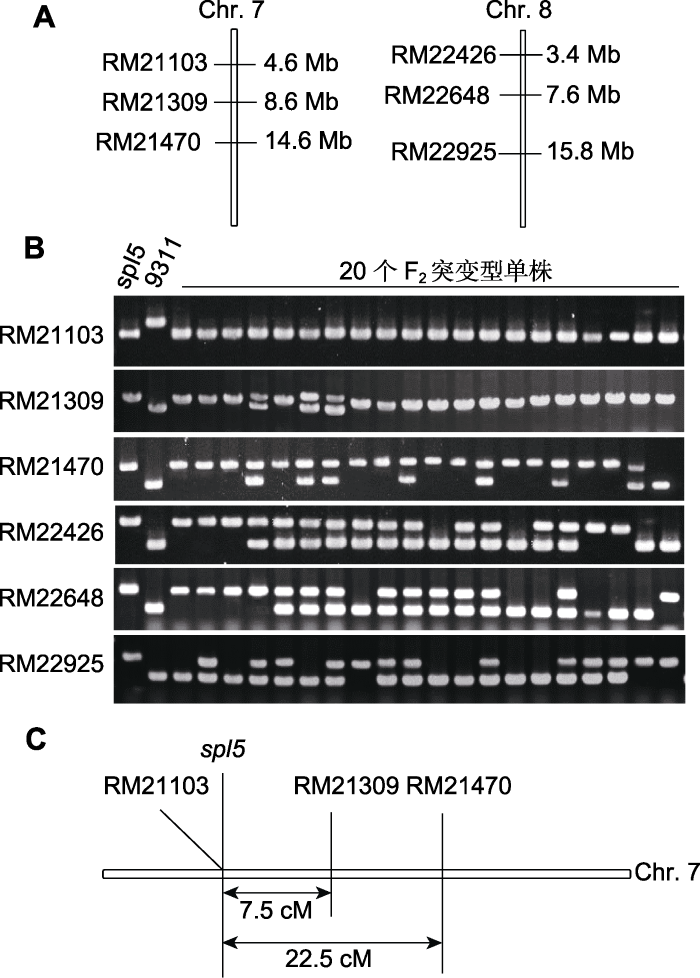 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3SPL5基因的连锁分析与遗传定位
(A) 本实验SSR标记所在的染色体及其物理距离; (B) 本实验SSR标记PCR扩增条带的电泳图; (C) SPL5基因的染色体遗传连锁图。
Figure 3Linkage analysis and genetic mapping of the SPL5 gene
(A) A physical map of the SSR markers on chromosomes used in this study; (B) Analysis of the PCR products of the SSR markers by electrophoresis; (C) A genetic linkage map of the SPL5 gene.
计算结果显示, RM22426、RM22648和RM22925与目标基因的重组率分别为47.5%、52.5%和60%, 接近50%, 且卡方检验其基因型分离比也符合1:2:1的遗传规律(P>0.05) (表2), 表明这3个SSR标记与目标基因不连锁, 发生了自由组合; 而RM21103、RM21309和RM21470与目标基因的重组率分别为0%、7.5%和22.5%, 远小于50%, 且卡方检验其基因型分离比也不符合1:2:1的遗传规律(P<0.01) (表2), 表明这3个SSR标记与目标基因连锁, 不能发生自由组合。将目标基因与连锁SSR标记的重组率转换成遗传距离后, 发现RM21103与目标基因的遗传距离为0 cM, 表明该SSR标记与目标基因非常近, 为紧密连锁或共分离标记。而RM21309和RM21470的重组率分别为7.5和22.5 cM, 与RM21103相比, 这两个标记与目标基因距离较远, RM21470距离目标基因最远。基于上述分析可绘制出目标基因的染色体遗传连锁图(图3C)。
Table 2
表2
表2本研究SSR标记基因型分离比的卡方(χ2)检验
Table 2
| 分子标记 | 20个F2突变型单株中各基因型的数量 | χ2值 | P值 | ||
|---|---|---|---|---|---|
| MM | Mm | mm | |||
| RM21103 | 0 | 0 | 20 | 60 | <0.01 |
| RM21309 | 0 | 3 | 17 | 38.7 | <0.01 |
| RM21470 | 1 | 7 | 12 | 13.9 | <0.01 |
| RM22426 | 4 | 11 | 5 | 0.3 | >0.05 |
| RM22648 | 5 | 9 | 6 | 0.3 | >0.05 |
| RM22925 | 7 | 10 | 3 | 1.6 | >0.05 |
新窗口打开|下载CSV
1.6.2 实验讨论
组织学生汇报实验结果, 并进行总结, 讨论对遗传学三大定律的认识, 同时反馈实验中遇到的困难和问题, 引导学生自己提出解决方法。例如, (1) 某些SSR标记PCR产物电泳时出现与亲本条带迁移率不同的其它条带, 该如何处理? 这主要是由PCR非特异性扩增引起, 在不影响目的条带观察的情况下, 可直接对目的条带进行分析; 若影响目的条带的观察, 可通过增加退火温度提高PCR的特异性。(2) 不同小组虽然采用相同的SSR标记, 但计算出的与目标基因的重组率为什么会有不同? 原因是分子标记与目标基因的重组是随机的, 各小组间使用的F2代突变型单株不同, 所以发生重组的事件也不同, 计算出的重组率也会有一定差异。(3) 个别SSR标记计算出的重组率可能大于50%, 如何解释? 这主要是由于F2代群体的样本数太少, 使得抽样误差大大增加, 从而导致计算的误差增大。将各组的数据合并再计算, 抽样误差将会减小。
2 教学实践效果分析
2.1 锻炼学生的科研实践能力
传统的遗传学实验主要是减数分裂的染色体行为和染色体失活的观察及利用果蝇(Drosophilia melanogaster)分析单基因和多基因的遗传规律等。在遗传学教材中也有相应的实例, 对学生来说属于验证性实验, 缺乏思考和自主设计等过程。本实验要求学生运用所学的遗传学理论知识和实验技术, 解决实际的科研问题, 从实验设计(包括植物DNA提取、PCR及电泳实验方法)到实验操作(包括移液器、离心机、PCR仪、电泳及凝胶成像设备等的使用), 再到结果分析(包括遗传学的计算与作图等), 整个实验课的规划都可由学生主导完成。因此, 提高了学生的自主学习和解决问题的能力, 拓宽了学生的知识面, 锻炼了学生的科研实践能力。2.2 加深学生对遗传三大定律的理解
遗传学是生物学本科生的一门专业必修课, 涉及的原理比较深。其中, 孟德尔的分离与自由组合、摩尔根的连锁互换是遗传学的三大定律, 贯穿整个遗传学的知识体系。虽然, 通过果蝇单因子、双因子和三点测交等传统遗传学实验能帮助学生加深认识, 但学生只能通过对杂交后代的表型观察来推测基因的重组方式和类型, 不仅比较抽象, 而且无法区别显性纯合与显性杂合的基因型, 也无法验证基因型分离的孟德尔比例(1:2:1)。此外, 连锁互换的理论较为抽象, 学生对利用分子标记构建的遗传连锁图谱难以理解。本实验借助图位克隆的技术原理, 让学生利用分子标记对F2代群体的基因型进行分析, 验证基因型1:2:1的孟德尔比例, 并通过电泳观察减数分裂后染色体的DNA序列发生分离与自由组合、连锁与互换, 非常直观且易于理解, 从而大大提高了学生对遗传学理论的认识。2.3 提高学生的团队合作能力
本实验工作量大, 且实验周期相对较长, 涉及DNA提取、PCR扩增、制胶及电泳等多种实验技术和生物统计学计算, 是一个综合性大型实验, 每个步骤都非常关键, 稍有差错就无法得到理想的结果。例如, DNA提取的时候个别样品没有成功、部分PCR扩增没有产物或出现非特异扩增、制胶不当导致电泳条带不清楚等, 都有可能影响重组率的计算。因此, 学生需要具备比较全面的实验技术, 熟练掌握相关操作, 才能独立完成这项实验。对部分学生而言有一定难度, 如果成功率偏低就会影响教学效果。因此, 由4-6个学生组成一个团队, 根据每个人的知识特点和技术优势, 进行分工合作, 共同完成此实验, 成功率会更高, 还可以培养学生协作互助的精神, 提高团队合作能力。3 结语
开设这门综合性遗传学实验不仅可在一定程度上提升遗传学教学的质量, 弥补传统实验教学的不足, 而且有利于开阔学生的视野、提高学生设计实验、运用理论知识分析和解决实际问题的能力, 也有利于培养学生的协作精神。与传统模式不同, 本实验着重培养学生的动手能力和创新意识。教师可从多个方面启发和引导学生, 即查询资料、选择材料和仪器、制定实验方案、实验操作和最后的结果分析, 整个实验过程可由学生自行完成。不仅符合循序渐进、因材施教和发展性教学原则, 还能充分调动学生的积极性和创造性, 使学生有独立思考、自主学习的空间和时间, 从而大大提高学生的实验技能和综合应用知识能力, 同时也可有效提高教师实验教学的效果。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
医学遗传学多元式教学的实践与探讨
1
2010
... 遗传学是当今生命科学领域一门重要的基础且前沿学科, 是生物学专业综合性较强的核心课程, 也是高等院校本科学习整个理论体系中的后续课程.遗传学具有很强的理论性和实践性, 内容跨度大且抽象、复杂, 学生不易理解, 只有使用多元化教学手段, 才能提高教学效率(
图位克隆技术在农作物基因分离中的应用与评价
1
2010
... 图位克隆(map-based cloning)又称定位克隆(positional cloning), 由剑桥大学的
水稻QTL图位克隆的特征分析
1
2008
... 图位克隆(map-based cloning)又称定位克隆(positional cloning), 由剑桥大学的
图位克隆技术在分离植物基因中的应用
2
2000
... 图位克隆(map-based cloning)又称定位克隆(positional cloning), 由剑桥大学的
... )等植物中被广泛应用(
一种高效便捷的水稻DNA提取法及其应用
1
2012
... 取亲本9311、spl5突变体及其杂交F2代20个突变型单株的新鲜叶片, 分别提取其基因组DNA, 参考
SPL5, a cell death and defense-related gene, encodes a putative splicing factor 3b subunit 3 (SF3b3) in rice
1
2012
... 鉴于图位克隆涉及遗传学中基因重组、连锁互换和遗传作图等学生较难理解的相关知识, 且涉及多项遗传学和分子生物学实验技术, 故掌握了图位克隆的技术原理可大大提高学生对遗传学重点知识的理解.我们前期利用图位克隆技术在水稻7号染色体上精细定位并克隆了1个调控类病变(lesion mimic)表型的基因SPL5 (
Fine genetic mapping and physical delimitation of the lesion mimic gene spotted leaf 5 (spl5) in rice(Oryza sativa L.)
1
2009
... 鉴于图位克隆涉及遗传学中基因重组、连锁互换和遗传作图等学生较难理解的相关知识, 且涉及多项遗传学和分子生物学实验技术, 故掌握了图位克隆的技术原理可大大提高学生对遗传学重点知识的理解.我们前期利用图位克隆技术在水稻7号染色体上精细定位并克隆了1个调控类病变(lesion mimic)表型的基因SPL5 (
Toward a physical map of the genome of the nematode Caenorhabditis elegans
1
1986
... 图位克隆(map-based cloning)又称定位克隆(positional cloning), 由剑桥大学的
The map-based sequence of the rice genome
2
2005
... (1) 根据水稻基因组的SSR标记信息(
... (2) 通过查阅文献(
备案号: 京ICP备16067583号-21
版权所有 © 2021 《植物学报》编辑部
地址:北京香山南辛村20号 邮编:100093
电话:010-62836135 010-62836131 E-mail:cbb@ibcas.ac.cn
本系统由北京玛格泰克科技发展有限公司设计开发
