0 引言
【研究意义】天然反义转录本(natural antisense transcripts,NATs)是一类内源性转录的可以与其他RNA转录本互补配对的RNA[1],它既可以是编码蛋白的RNA,也可以是非编码的RNA。NATs根据其转录来源可以分为顺式NATs(cis-NATs)和反式NATs(trans-NATs)[2]。cis-NATs转录自基因的互补链而trans-NATs从基因组其他位置转录。研究表明NATs在人、鼠、猪、酵母和拟南芥等真核生物组织中均广泛存在[3,4]。由此可见cis-NATs是基因表达调控的重要因素,本研究的意义在于通过对不同品种猪背最长肌组织中cis-NATs的研究进一步解析猪骨骼肌生长发育的分子机理。【前人研究进展】骨骼肌占哺乳动物总体重的50%左右[5]。研究发现骨骼肌发育过程中的多个关键基因都存在cis-NATs。BRAUNSCHWEIG等研究指出IGF2与其cis-NATs在猪的肌肉组织中协同表达[6]。SUN等在猪的肌肉和肝脏等多个组织中均检测到了FGF2的天然反义转录基因NUDT6,在肌纤维的生长发育过程中两者呈相反表达[7]。PANDORF等发现大鼠MHC基因簇的cis-NATs分别与Myh1和Myh2协同表达和相反表达[8],从而影响骨骼肌的肌纤维类型。CHEN等在全基因组水平上对猪的cis-NATs进行了研究,该研究表明猪骨骼肌中约30%的转录本具有cis-NATs[4]。NATs主要参与其正义基因转录水平和转录后水平的调控,目前已报道的调控机制主要包括:转录干扰[9]、RNA封闭[10]、RNA编辑[11]、RNA干扰[12]、染色质重塑[13,14]和兼并异染色质机制[15]。 【本研究切入点】猪骨骼肌的生长发育与其生长速度和瘦肉率息息相关,直接关系着生猪养殖的生产效益。中外猪种在生长速度和瘦肉率性状上具有明显的遗传差异,长白猪作为高度选育的瘦肉型外国猪种,具有生长快,瘦肉率高等特点,而中国地方猪种蓝塘猪的胴体瘦肉率相对较低,生长速度也相对较慢[16]。因此,以蓝塘猪和长白猪作为试验对象来探索cis-NATs与不同猪品种骨骼肌发育差异的关系,有助于进一步了解瘦肉型猪和肥胖型猪之间骨骼肌生长发育遗传差异的分子机制。虽然多项研究成果表明cis-NATs广泛存在于猪的骨骼肌中,但是cis-NATs在中外猪种骨骼肌生长发育过程中是否存在差异以及其所参与的生物学过程和代谢通路仍有待于研究。【拟解决的关键问题】运用生物信息学分析手段,系统比较多个时间点蓝塘猪和长白猪骨骼肌组织中差异表达cis-NATs,并以此为基础利用cis- NATs与相关表达的正义基因进行功能聚类分析,探索cis-NATs参与蓝塘猪和长白猪生长发育过程中品种间骨骼肌发育差异的分子机理。1 材料与方法
试验于2017年5—8月在华中农业大学农业动物遗传育种与繁殖教育部重点实验室、生猪健康养殖协同创新中心进行。1.1 试验材料
本试验中表达谱及cis-NATs数据[17,18]来自于笔者已发表的长白猪和蓝塘猪的背最长肌组织基因表达谱及cis-NATs表达谱数据。该套数据共有10个发育时期,分别为胚胎期35 d、胚胎期49 d、胚胎期63 d、胚胎期77 d、胚胎期91 d、生后期2 d、生后期28 d、生后期90 d、生后期120 d及生后期180 d。本研究基于本实验室前期已发表的已注释基因表达、cis-NATs表达及相关数据,进一步探索了品种间基因表达及cis-NATs差异及相关功能。1.2 试验方法
1.2.1 差异表达分析 本研究使用edger R语言包[19]鉴定蓝塘猪和长白猪两品种间差异表达的cis-NATs及基因,差异表达的阈值为|log2FC|≥1且FDR<0.01。1.2.2 功能注释分析 为了解析两品种间差异表达cis-NATs的生物学功能,我们首先运用DAVID [20] 对两品种的差异表达基因及差异表达cis-NATs对应的正义基因进行了GO分析和KEGG分析。筛选GO生物学过程和KEGG通路的阈值分别为P<0.01和P<0.05。然后,利用cis-NATs与其对应的正义基因表达相关(|R|>0.70且FDR<0.05)进一步筛选cis-NATs显著富集(cis-NATs个数大于10个)的GO生物学过程。同时,运用Cytoscape[21] 对显著富集的KEGG通路(P<0.05)进行整合分析,根据每个通路所包含的有相关表达的cis-NATs的基因数目填充节点颜色,以通路间共有的基因数目填充线的宽度,最后结合节点的颜色和线的宽度将代谢通路进行归类。
1.2.3 通路可视化分析 根据 KEGG通路整合分析对联系最紧密的模块进行了通路可视化分析,并用Cystoscape软件进行了绘图。首先,过滤掉通路中没有表达量的基因节点;然后根据各通路间的连接节点合并通路;进一步根据表达相关添加cis-NATs节点;用表达差异倍数填充节点颜色。
2 结果
2.1 猪骨骼肌生长发育过程中共有5 350个基因和738个cis-NATs在蓝塘猪与长白猪两品种间差异表达
根据10个时间点猪骨骼肌组织基因和cis-NATs表达谱,在蓝塘猪与长白猪两品种间进行差异表达分析,共鉴定出5 350个差异表达基因和738个差异表达cis-NATs(| log2FC |≥1,FDR<0.01)。各个时间点差异表达基因及cis-NATs数目如表1所示;出生后早期2—90 d,蓝塘猪和长白猪之间的差异表达基因及cis-NATs数目相对出生前明显下降(表1);MAplot显示品种间cis-NATs差异表达倍数分布类似于正义基因的差异表达倍数分布(图1)。各时间点差异倍数前十(5个蓝塘猪上调表达和5个蓝塘猪下调表达)的差异表达基因及cis-NATs如表2所示。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1蓝塘猪和长白猪差异表达基因及差异表达cis-NATs MAplot图
-->Fig. 1MAplot of DEGs and differential expression cis-NATs between LT and LR
-->
Table 1
表1
表1蓝塘猪和长白猪差异表达基因及差异表达cis-NATs数目统计
Table 1Statistics of DEGs and differential expression cis-NATs between LT and LR
| 时间点 Time point | 差异表达基因 DEGs | 差异表达cis-NATs Differential Expression cis-NATs | ||||
|---|---|---|---|---|---|---|
| 上调 Up | 下调 Down | 合计 Total | 上调 Up | 下调 Down | 合计 Total | |
| 胚胎期35d Fetal 35 day | 519 | 406 | 925 | 64 | 44 | 108 |
| 胚胎期49d Fetal 49 day | 1004 | 950 | 1954 | 102 | 148 | 250 |
| 胚胎期63d Fetal 63 day | 921 | 802 | 1723 | 101 | 106 | 207 |
| 胚胎期77d Fetal 77 day | 818 | 643 | 1461 | 104 | 91 | 195 |
| 胚胎期91d Fetal 91 day | 776 | 627 | 1403 | 79 | 130 | 209 |
| 生后期2d Postnatal 2 day | 417 | 476 | 893 | 47 | 99 | 146 |
| 生后期28d Postnatal 28 day | 373 | 400 | 773 | 43 | 57 | 100 |
| 生后期90d Postnatal 90 day | 386 | 374 | 760 | 76 | 33 | 109 |
| 生后期120d Postnatal 120 day | 903 | 636 | 1539 | 71 | 154 | 225 |
| 生后期180d Postnatal 180 day | 1166 | 829 | 1995 | 81 | 222 | 303 |
新窗口打开
Table 2
表2
表2骨骼肌发育过程中蓝塘猪与长白猪排在前10的差异表达基因及cis-NATs
Table 2Top10 of DEGs and differential expression cis-NATs between LT and LR during the skeletal muscle development
| 时间点 Time point | 差异倍数排在前十的差异表达基因 Top10 of DEGs | 差异倍数排在前十的差异表达cis-NATs对应的正义基因 Sense Genes for Top10 of Differential Expression cis-NATs |
|---|---|---|
| 胚胎期35d Fetal 35 day | CYP17A1,NCL,SLC4A11,CYP11A1,CYP21A2,MRPL22FETUB,SVEP1,HBE1,GBP7 | TAF10,DIRAS3,PTPRK,MAP2,BCLAF1,MICU2,PDGFRA,DNM3OS,PIK3R4,RICTOR |
| 胚胎期49d Fetal 49 day | GAS2,ISLR,MYO10,MAN1A1,LDHB,EIF2S3,DDX3X,RPS13,VEGFB,NOL8 | HMGB1,AP1G1,TNMD,ZNF277,PTPRK,SKIV2L,RPS19,FKBP1A,WDR34,LENG9 |
| 胚胎期63d Fetal 63 day | TFRC,GBE1,RIOK3,NCL,ZNF277,RPS13,TMSB15A,DOK4,PURA,TNFRSF12A | ZFR,EML4,RPS24,GAPVD1,MLLT10,SKIV2L,MICU2,RBM24,BLCAP,EIF2B1 |
| 胚胎期77d Fetal 77 day | COL1A1,ASPN,COL3A1,FN1,MYL2,KIF2C,USP22,PRMT9,MYCN,TMEM199 | SPARC,CENPP,SPARC,FHL1,C2CD3,ZNF142,REEP5,TRAPPC13,PITHD1,ZFR |
| 胚胎期91d Fetal 91 day | STK31,HLA-A,ARID1A, TMEFF2, FAM13C,RPS13,RPL23A,NOL8,VEGFB,ENY2 | TNPO1,STK31,IVNS1ABP,C2CD3,SF3B1,SKIV2L,FKBP1A,FAM49B,TM2D1,NOP56 |
| 生后期2d Postnatal 2 day | SYN1,MLH1,SPIDR,THBS1,AMPD3,SKIV2L,RNF103,SLC4A7,PSAT1,CSNK1G3 | SLC4A11,RPL23A,MT1A,MT1E,NCL,RPS13,MRPL22,EPC1,EXTL1,HAND2-AS1 |
| 生后期28d Postnatal 28 day | NCL,KCNAB1,MYH6,POLR2KMETTL21EP,PPP1R1A,RPS13,PFKFB1,ENO1,ATF3 | ANXA1,RP11-451G4.2, GFM1,BOLA3,MACF1, KCNMA1,OTUD1,ATF4,TMOD4,RBM24 |
| 生后期90d Postnatal 90 day | TMED10,MGEA5,UBR3,MARCKS,COPG1,FHL1,FAM104A,SLC2A1,RYR1,CENPP | RPLP0,RPSA,PGM1,ACTA1, SUCLA2,FARSA,EXT2,RRAGD,SMTN,KBTBD4 |
| 生后期120d Postnatal 120 day | PHKA1,CMYA5,ART3,NDUFB5,ACTC1,RPS13,RP11-96C21.2, LINC00493,CXCL2,BTG2 | GOLGB1,FKBP2,MTMR9LP,CALM1,DHX32,TM2D1,ANKRD1,DDIT4,CUL2,WDR34 |
| 生后期180d Postnatal 180 day | ACTC1,C7orf73,AMPD3,CLTC,CCT2,RPL15,STX10,RP11-96C21.2,ENY2,MMP7 | GOLGB1,CCNI,FKBP2,MTMR9LP,AMPD3,DDIT4,FKBP1A,ATPIF1,TM2D1,HACD1 |
新窗口打开
2.2 品种间骨骼肌组织差异表达cis-NATs主要富集在骨骼肌发育及能量代谢的GO生物学过程中
利用DAVID功能注释平台对差异表达cis-NATs及相关正义基因进行功能注释分析,结合表达相关性分析结果发现包含10个以上差异表达cis-NATs的正义基因的GO生物学过程有两类,第一类是肌肉发育,包含肌肉收缩(muscle contraction)、肌肉器官发育(muscle organ development)、骨骼肌细胞分化(skeletal muscle cell differentiation)和横纹肌收缩(striated muscle contraction)等11个GO生物学过程;第二类是能量代谢,主要包括糖酵解过程(glycolytic process)、糖异生(gluconeogenesis)、氢离子跨膜运输(hydrogen ion transmembrane transport)和ATP生物合成过程(ATP biosynthetic process)等6个GO生物学过程(表3)。Table 3
表3
表3包含10个以上差异表达cis-NATs的GO生物学过程
Table 3GO terms of muscle development and energy metabolism with up 10 cis-NATS
| 条目 Term | F35 | F49 | F63 | F77 | F91 | P2 | P28 | P90 | P120 | P180 | 合计 Total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 肌肉发育相关条目Terms associated with muscle developmental | |||||||||||
| GO:0006936~肌肉收缩 Muscle contraction | — | 10 | 8 | 8 | 6 | 8 | 9 | 6 | 16 | 17 | 88 |
| GO:0030049~肌纤维滑动 Muscle filament sliding | — | 7 | 2 | 3 | 5 | 8 | 4 | 6 | 9 | 11 | 55 |
| GO:0045214~肌节组织 Sarcomere organization | — | 5 | 4 | 4 | 5 | 4 | 5 | 4 | 8 | 9 | 48 |
| GO:0060048~心肌收缩 Cardiac muscle contraction | 1 | 3 | 1 | 2 | 3 | 4 | 1 | 3 | 4 | 6 | 28 |
| GO:0007517~肌肉器官发育 Muscle organ development | — | 6 | 4 | 5 | 2 | 3 | — | — | 4 | 2 | 26 |
| GO:0007519~骨骼肌发育 Skeletal muscle tissue development | 2 | 3 | 4 | 3 | 2 | 3 | 2 | 2 | 4 | — | 25 |
| GO:0007507~心脏发育 Heart development | 1 | 6 | 4 | 5 | 4 | 4 | — | — | — | — | 24 |
| GO:0007015~肌动蛋白纤维组织 Actin filament organization | — | 4 | 1 | 3 | 3 | — | 1 | — | 4 | 6 | 22 |
| GO:0035914~骨骼肌细胞分化 Skeletal muscle cell differentiation | — | — | 5 | 2 | — | 3 | 3 | 2 | 6 | — | 21 |
| GO:0006941~横纹肌收缩 Striated muscle contraction | — | 2 | 2 | 2 | 2 | 2 | 3 | 1 | 3 | 3 | 20 |
| GO:0030239~肌原纤维组装 Myofibril assembly | — | — | 3 | — | — | — | 4 | 1 | 2 | 4 | 14 |
| 能量代谢相关条目Terms associated with energy metabolism | |||||||||||
| GO:0006096~糖酵解过程 Glycolytic process | — | — | — | — | — | 5 | 3 | 3 | 7 | 9 | 27 |
| GO:0006094~糖异生 Gluconeogenesis | — | — | — | — | — | 4 | 5 | 3 | 7 | 8 | 27 |
| GO:0061621~经典糖酵解 Canonical glycolysis | — | — | — | — | — | 3 | 2 | 3 | 5 | 7 | 20 |
| GO:0005980~糖原分解代谢过程 Glycogen catabolic process | — | — | — | — | — | 3 | 3 | 3 | 5 | 5 | 19 |
| GO:0006754~ATP 生物合成过程 Biosynthetic process ATP | — | — | — | — | — | — | 2 | 4 | 3 | 4 | 13 |
| GO:1902600~氢离子跨膜运输 Hydrogen ion transmembrane transport | — | 2 | — | 1 | — | — | 2 | — | 2 | 3 | 10 |
新窗口打开
2.3 KEGG通路整合分析发现能量代谢通路之间关联性最强
利用Cytoscape软件根据KEGG通路间相同基因的数目进行归类,最终将通路分为三大类:能量代谢相关通路、肌肉生长发育相关通路及其他。其中能量代谢相关通路包括糖酵解/糖异生、三羧酸循环及氧化磷酸化等,肌肉生长发育相关的通路则有心肌收缩、肥厚性心肌病及扩张型心肌病等(图2)。进一步分析显示能量代谢相关的通路之间相同数目基因较多,关系更紧密。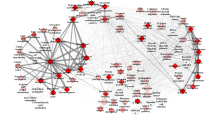 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2KEGG通路关联图
节点颜色的深浅代表通路中包含有相关表达的cis-NATs的基因数目;线的宽度代表节点间所含相同基因的数目;有相关表达的cis-NATs的基因数目小于3的通路用“”表示;其余则用“”表示
-->Fig. 2Association network of KEGG pathways
The color depth of the node reflects the number of gene that including expressed correlatively cis-NATs in the pathway. The width of the line indicates the overlap number of gene between nodes. “” indicates the pathway that contain less than 3 genes related to cis-NATs, and the rest of pathways were present with “”
-->
2.4 线粒体中三羧酸循环通路的cis-NATs及基因在仔猪出生后早期表达量更高
根据通路整合结果筛选出线粒体中三羧酸循环代谢通路的差异表达基因及其相关表达的cis-NATs,利用perl语言提取这些基因在长白猪和蓝塘猪10个时间点的表达量,使用R语言pheatmap包绘制热图(图3)。在胚胎期,蓝塘猪的线粒体中三羧酸循环通路的基因及cis-NATs表达量整体稍高于长白猪。仔猪出生后,基因及cis-NATs的表达量显著上升。在出生后2 d,长白猪的表达量普遍高于蓝塘猪。到第28天时大部分基因及cis-NATs表达量出现反转,蓝塘猪的表达量更高。在第90天时,蓝塘猪和长白猪的表达量都有所下降。第120天和180天两个时间点的差别不大,整体上都是蓝塘猪稍高于长白猪。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3三羧酸循环通路中基因及cis-NATs差异表达热图
LD代表长白猪;LT代表蓝塘猪
-->Fig. 3of genes and cis-NATs in citrate cycle pathway
LD means Landrace and LT means Lantang
-->
2.5 通路可视化分析发现cis-NATs在品种间的差异表达具有时效性
根据不同KEGG通路之间的节点基因合并能量代谢相关通路,结合通路中的基因和cis-NATs的表达量以及两者在中外猪种间的差异表达倍数,绘制出生后不同时期的能量代谢通路。在胚胎期第49天和胚胎期第77天蓝塘猪的基因及cis-NATs的表达量显著高于长白猪,其中,胚胎期第49天蓝塘猪和长白猪能量代谢通路中相关基因及其cis-NATs的差异表达情况如图4-A所示。仔猪出生后第2天,蓝塘猪与能量代谢相关的通路中基因及cis-NATs的表达量显著低于长白猪(图4-B)。生后期28 d,蓝塘猪的表达量明显上升,大部分基因及cis-NATs的表达量高于长白猪(图5-A),而生后期90 d,蓝塘猪糖酵解通路及氧化磷酸化通路中基因及cis-NATs的表达量相对于长白猪显著下调(图5-B)。图4和图5说明cis-NATs在品种间的差异表达具有时效性,不同时间点上蓝塘猪和长白猪的差异表达情况有所不同。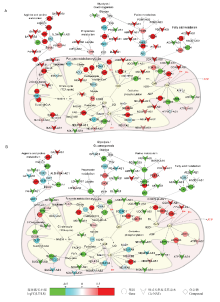 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4蓝塘猪和长白猪在胚胎期49 d(A)和生后期2 d(B)背最长肌组织能量代谢相关通路中基因及其相关表达cis-NATs的表达模式
A. 胚胎期49 d时蓝塘猪长白猪的对比 Fetal 49 day LT vs. LD;B. 生后期2 d时蓝塘猪与长白猪的对比 Postnatal 2 day LT vs. LD
-->Fig. 4The expression patterns of genes involved in energy metabolic pathways and their correlated cis-NATs between LT and LR longissimus dorsi on 49dpc (A) and 2dpn (B)
-->
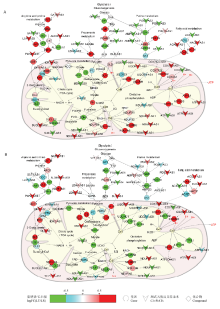 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5蓝塘猪和长白猪在生后期28 d(A)和90 d(B)背最长肌组织能量代谢相关通路中基因及其相关表达cis-NATs的表达模式
A.生后期28 d时蓝塘猪与长白猪的对比 Postnatal 28 day LT vs. LD;B.生后期90 d时蓝塘猪与长白猪的对比 Postnatal 90 day LT vs. LD
-->Fig. 5The expression patterns of genes involved in energy metabolic pathways and their correlated cis-NATs between LT and LR longissimus dorsi on 28 dpn (A) and 90 dpn (B)
-->
3 讨论
3.1 cis-NATs差异表达分析
目前关于骨骼肌中cis-NATs的研究主要集中于鉴定、发育过程中表达模式及与其正义基因间表达关系等方面,不同猪种cis-NATs表达模式的比较却鲜有研究。因此品种间cis-NATs的差异表达模式是否影响猪骨骼肌生长发育过程仍有待于研究。长白猪和蓝塘猪在肌肉生长发育方面存在较大差异,前人研究发现两品种间肌纤维数目及横截面积都存在差异[17, 22],中外猪种肌纤维类型也有较大差异[23]。本研究通过对胚胎期至成年共10个时间点的长白猪和蓝塘猪骨骼肌组织中差异表达的cis-NATs进行分析,总共鉴定出738个差异表达cis-NATs。为了研究这些cis-NATs的生物学功能,本研究首先对差异表达的基因及cis-NATs对应的基因进行了功能聚类分析,并在此基础上筛选出包含有显著相关表达的cis-NATs的正义基因显著富集的GO生物学过程和KEGG通路,以此来推断猪骨骼肌生长发育过程中蓝塘猪和长白猪间差异表达cis-NATs参与的主要通路,解析cis-NATs调控品种间骨骼肌发育差异的分子机理。3.2 功能注释及通路可视化
GO分析结果显示,蓝塘猪和长白猪的差异表达基因及其相关表达的cis-NATs富集在与肌肉生长发育及能量代谢相关的生物学过程中。由于能量代谢相关KEGG通路之间的关联性更为紧密(图2),因此本研究对蓝塘猪和长白猪之间能量代谢相关通路进行了进一步的比较及可视化分析。能量代谢通路可视化分析结果显示:胚胎期49 d和77 d时蓝塘猪的cis-NATs的表达量显著高于长白猪(图4-A)。已有研究显示猪在出生前肌纤维数量已经基本确定[24],胚胎期第49和77天分别处于猪初级肌管和次级肌管增殖的高峰期内,初级肌管和次级肌管分别发育成为初级肌纤维和次级肌纤维,研究表明长白猪的次级肌纤维数目多于蓝塘猪,长白猪在胚胎期生肌速度更快[17],这暗示cis-NATs可能参与猪胚胎期肌纤维的生长调控。此外,本研究统计结果显示:与能量代谢相关的生物学过程中差异表达的cis-NATs主要富集在生后期而胚胎期较少(表3),且三羧酸循环通路中的基因及cis-NATs在蓝塘猪和长白猪的生后期表达量也都显著高于胚胎期(图3),这可能与仔猪在出生后能量代谢较为旺盛有关。猪在出生后骨骼肌发育主要是肌纤维类型的转变,肌纤维根据收缩功能和所含酶系及活性可以划分为四种类型:慢收缩氧化型(I型)、快收缩氧化型(IIa型)、中间型(IIx型)和快收缩酵解型(IIb型)[25]。I型肌纤维富含线粒体,有氧代谢的酶系活性很高,主要通过有氧代谢供能,纤维细小、收缩慢而持久;IIb型肌纤维含少量线粒体,糖酵解酶系和ATP酶活性较高,主要进行厌氧代谢,纤维粗大、收缩快但不持久;IIa型和IIx型肌纤维的特征介于I 型和IIb型之间[26]。刚出生的仔猪肌纤维类型是氧化型,有氧代谢占主导[27] 。猪骨骼肌肌纤维类型的分化主要发生在出生后早期,从出生开始部分氧化型(I型)肌纤维逐渐转化成酵解型(II型)肌纤维,即I型肌纤维数目开始下降,II型肌纤维开始出现并逐渐增加[28]。在出生后早期这一转化就会完成,且肌纤维类型比例基本确定[29,30,31]。本研究发现在出生后早期,蓝塘猪和长白猪的差异表达模式在28 d和90 d发生了两次转变:在生后期2 d,蓝塘猪与能量代谢相关的通路中基因及其相关表达的cis-NATs的表达量显著低于长白猪(图4-B),到生后期28 d发生转变(图5-A)。而生后期90 d时再次发生反转,与生后期2 d类似,蓝塘猪的能量代谢相关通路中基因及其相关表达的cis-NATs相对于长白猪显著下调(图5-B)。研究表明长白猪较中国地方猪种拥有更多的酵解型肌纤维[23]。与本研究相对应的发育时间点的研究表明,长白猪能量代谢相关通路中的基因及其相关表达的cis-NATs在生后期2 d到28 d间发生剧烈下调并一直持续到90 d,而蓝塘猪类似的下调则晚于长白猪[18]。这表明长白猪的肌纤维类型转化可能早于蓝塘猪,并且这种差异与品种间差异表达的cis-NATs相关。因此推测蓝塘猪和长白猪出生后cis-NATs的差异表达与两品种猪肌纤维类型的转变调控相关。
4 结论
在蓝塘猪和长白猪两品种背最长肌中总共筛选了5 350个差异表达基因和738个差异表达cis-NATs。功能分析结果表明品种间差异表达基因及cis-NATs主要富集到与肌肉生长发育和能量代谢相关的通路中。通过可视化分析结果显示cis-NATs参与猪骨骼肌组织中能量代谢相关基因的表达调控,这可能是导致品种间骨骼肌纤维数目及类型差异的原因之一。The authors have declared that no competing interests exist.
