0 引言
【研究意义】猪肉是人类摄取蛋白质的第一来源,尤其对于中国而言,更是在国民经济中有着至关重要的地位。但是中国对家猪育种改良起步比较晚,在此方面比较落后。巴马小型猪是地方选育出来的小型猪品系,其心脏等各脏器结构与人极为相似,因此作为重要的大动物模型被应用于生命科学研究,并且得到了较好的开发利用[1]。ZBED6是新近发现的一个能够调控IGF2表达和肌肉生长的转录因子,其调节肌肉生长发育等功能引起了广泛关注[2,3]。ZBED6已经被证明是包括人类在内的胎盘类哺乳动物的进化物,并且与发育类疾病相关[3,4]。本试验以ZBED6基因敲除巴马小型猪为试验载体,从体内水平研究转录因子ZBED6在心肌中的功能,进一步解析该转录因子调控肌肉生长发育等重要生物学功能的分子机理,从而为研究相关疾病提供理论基础,同时为瘦肉型家猪的育种改良做出贡献。【前人研究进展】在家猪中,IGF2基因的内含子3检测到一个突变位点G→A,该突变导致骨骼肌中IGF2的表达量上调3倍,IGF2的上调对肌肉生长、心脏大小以及脂肪沉淀有重要的影响[3, 5]。ZBED6在不同物种的细胞和成体水平都有一定的研究,细胞水平研究显示,ZBED6能抑制小鼠和牛肌原细胞中IGF2的表达水平,抑制肌管的形成,最终导致肌肉细胞的生长发育受阻[2, 6-11];成体水平上的研究显示,ZBED6基因的遗传变异与绵羊的眼肌面积、尾脂重、背部肌肉厚、背最长肌重等主要胴体性状显著相关[12]。【本研究切入点】目前ZBED6基因在家猪方面的研究比较缺乏,ZBED6基因影响家猪心肌发育的分子机制,目前尚未有相关报道。【拟解决的关键问题】本试验以ZBED6基因敲除巴马小型猪为载体,对ZBED6-KO猪和ZBED6-WT猪的心脏组织进行表型特征鉴定、组织学分析以及转录组水平分析,以期揭示ZBED6在心脏发育中的调控机制,为进一步揭示ZBED6的分子调控机制提供更多依据。1 材料与方法
试验于2016—2017 年在中国农业科学院北京畜牧兽医研究所畜禽遗传资源创新实验室完成。1.1 实验动物
本试验中的ZBED6基因敲除(ZBED6-KO)巴马小型猪和同日龄正常巴马小型猪(ZBED6-WT)来自于中国农业科学院北京畜牧兽医研究所大兴猪场实验基地,ZBED6基因的敲除由中国农业科学院北京畜牧兽医研究所潘登科课题组利用CRISPR/Cas9技术完成。1.2 样品采集及表型特征鉴定
对3头ZBED6-KO猪(x1、x3、x6)和3头ZBED6-WT猪(x2、x4、x5)进行屠宰,称取心脏重量,同时采集心脏组织样品,一份置于RNA-later中,用于RNA的提取;一份于4%多聚甲醛中固定,用于组织切片的制作。1.3 q-PCR分析IGF2在心脏组织中的相对表达量
1.3.1 心脏组织总RNA提取与反转录 使用Qiagen公司的组织RNA提取试剂盒进行心脏组织RNA的提取,操作步骤参照该试剂盒使用说明书。将每个样品的1 μg RNA采用TaKaRa公司的RT-PCR kit试剂盒(TaKaRa,大连,中国)进行反转得到cDNA。1.3.2 定量引物的设计及合成 下载NCBI网站上IGF2基因mRNA序列,同时选用GAPDH作为内参基因,利用软件primer 5.0设计引物(表1)。引物由上海生工生物工程股份有限公司合成。
Table 1
表1
表1本研究采用的 qRT-PCR 鉴定引物
Table 1qRT-PCR primers used in this study
| 基因名称 Gene name | 引物序列 Sequence of primer (5′-3′) | 扩增长度 Amplification length (bp) |
|---|---|---|
| GAPDH-F | GGGCATGAACCATGAGAAGT | 187 |
| GAPDH-R | GTCTTCTGGGTGGCAGTGAT | |
| IGF2-F | ACACCCTCCAGTTTGTCTGC | 154 |
| IGF2-R | AAGCAGCACTCTTCCACGAT | |
| IFIT3-F | ACTGCAGCCCAACAGTCTTT | 104 |
| IFIT3-R | TTGTGGAAGCTGATTTGCAG | |
| MX1-F | CACAGAACTGCCAAGTCCAA | 82 |
| MX1-R | GCAGTACACGATCTGCTCCA | |
| ISG12(A)-F | GCCTGGCTTCCAAGATACTG | 145 |
| ISG12(A)-R | CTGAAGGAAGTCCTGCCAAG | |
| P2RY1-F | TTCCTGACTTGCATCAGTGC | 139 |
| P2RY1-R | CAGTGCCCGAGTAGAAGAGG | |
| TNNT1-F | GGTCAAGGCAGAACAGAAGC | 162 |
| TNNT1-R | ATCCAATCCGACAGTTCCTG | |
| MYL7-F | CCCATCAACTTCACCGTCTT | 132 |
| MYL7-R | GGAGAAGCTGCTTGAACTGG | |
| XAF1-F | GGAGATCCATGAGCACCACT | 164 |
| XAF1-R | TTGCTTTCAGGAGCTGGAAT | |
| CEL-F | CATGGACGGTCACTTGTTTG | 139 |
| CEL-R | CACGTCAAAGGTGATGTTGG | |
| ATP1A2-F | CTGTCACGGTGTGTTTGACC | 162 |
| ATP1A2-R | AACAGATGGTGGAGGTGGAG |
新窗口打开
1.3.3 qRT-PCR 用SYBR Green Master Mix(TaKaRa,大连,中国)试剂,cDNA作为模板,利用ABI7500 实时荧光定量仪(Applied Biosystems, Forest City, CA, SA)对cDNA进行基因表达分析,每个样品设置三个生物学重复,每个基因(包括内参)的3个Ct值的平均值作为该基因的平均Ct值;差异表达基因的qRT-PCR表达量计算采用2-ΔΔct方法。
1.4 石蜡组织切片的制备
将心肌组织切割成大小约1 cm3小组织块,置于4%多聚甲醛溶液中固定24 h,然后进行修块、脱水、石蜡包埋,将石蜡包埋组织作4 μm厚的连续切片,H.E染色,中性树胶封片,切片镜检拍照[13]。1.5 RNA-seq
1.5.1 总RNA提取 RNA提取方法同1.3.1,通过Agilent 2100 bioanalyzer检测RNA的浓度和RIN值,浓度>100 ng·μL-1,总量≥2.0 μg,且RIN值>8.0,则可用于后续转录组分析。1.5.2 RNA-seq建库及测序 根据Illumina TruSeq RNA样品制备试剂盒操作说明书[14]进行RNA-seq测序所需的cDNA文库的构建。
严格按照TruSeq PE Cluster Kit v3-cBot-HS试剂盒说明,使用cBot Cluster Generation系统对含有索引接头的样品进行聚类。形成cluster之后在Illumina Hiseq 2500测序平台分别对上述样品进行读长为125bp的双末端测序。
1.5.3 转录组测序原始数据的质控 原始数据质控:使用NGSQC Toolkit v2.3.3软件对原始reads进行过滤,去除接头序列,reads末端的低质量碱基(Phred质量值<20),以及过滤后长度小于25 bp的reads。
1.5.4 测序reads的比对与拼接 reads比对与拼接:从Ensemble数据库下载猪参考基因组以及基因组注释文件。用TopHat v2.0.11软件[15]将质控后的clean data比对到参考基因组上。然后用Cufflink软件[16]进行转录本的组装。用Cuffmerge将所有样本的转录本及参考基因组组装成新的注释文件,用于后续分析。
1.5.5 差异表达基因的筛选及层次聚类分析 用Cufflink软件的Cuffdiff包对ZBED6-KO猪和对ZBED6-WT猪心脏组织进行差异基因分析。用FDR法进行多重校正,筛选同时满足FDR<0.05和Fold change>2的基因作为显著表达基因。
利用R包中的聚类分析程序对ZBED6-KO猪和ZBED6-WT猪心脏组织差异表达的基因进行层次聚类分析[17]。
1.5.6 差异表达基因的GO和IPA富集分析 采用g:Profiler在线分析软件进行差异表达基因GO富集分析[18],按照富集倍数(Fold-enrichment)从小到大的原则,使用P<0.05作为显著富集基因的阈值;将差异表达基因导入IPA软件,通过“Core Analysis”和“Enrichment Analysis”获得pathway通路,根据-ln (P-value)值的大小进行排序,使用-ln(P-value)>1.30(P<0.05)作为显著富集基因的阈值。
1.6 差异表达基因qRT-PCR验证
随机选取9个差异表达基因采用qRT-PCR方法,检验RNA-seq结果的准确性。方法同1.3。2 结果
2.1 表型特征的鉴定结果
统计分析结果如图1所示,ZBED6-KO猪的心脏重高于ZBED6-WT猪,且差异显著(P<0.05),由此可知ZBED6基因的敲除对家猪心脏的生长发育有一定的促进作用。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1心脏组织表型特征鉴定结果
-->Fig. 1Results of the identification of cardiac tissue phenotype
*P<0.05
-->
2.2 IGF2在心脏组织中的相对表达量
IGF2在ZBED6-KO猪和ZBED6-WT猪心脏组织中的相对表达量结果如图2所示,ZBED6-KO组IGF2的表达量高于ZBED6-WT组,且差异显著(P<0.05)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2IGF2的相对表达量
-->Fig. 2The relative expression of IGF2
*P<0.05
-->
2.3 组织学分析结果
心脏组织的H.E染色(图3)结果表明,与ZBED6- WT猪相比,ZBED6-KO猪的肌纤维较宽,结缔组织较少,说明ZBED6的敲除在一定程度上促进家猪心肌的发育。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3心肌切片(H.E染色 100×)
-->Fig. 3Myocardial paraffin section
-->
2.4 RNA-seq 原始测序数据质量控制
各样本的测序所得原始数据组成如表2所示。每个样本测序获得至少10 G的数据量,各样本clean ratio和Q30 data都在90%以上。测序数据符合后续生物信息分析要求。Table 2
表2
表2转录组测序数据质量检测分析
Table 2The quality analysis of sequencing data of transcription group
| 样品 Sample | 原始序列 Raw reads | 质控后序列 Clean reads | 质控率 Clean ratio | Q30数据 Q30 data |
|---|---|---|---|---|
| x1 | 40241698 | 38971687 | 96.84 | 95.76;88.49 |
| x2 | 56977284 | 54157864 | 95.05 | 95.94;89.49 |
| x3 | 44460101 | 42764905 | 96.19 | 96.21;91.73 |
| x4 | 45403659 | 43618327 | 96.07 | 96.20;91.71 |
| x5 | 49894338 | 45301686 | 90.8 | 95.89;88.46 |
| x6 | 54203615 | 51972678 | 95.88 | 96.19;90.55 |
新窗口打开
2.5 参考基因组比对
通过Tophat比对,mapping结果如表3,62.8%— 80.1%的reads能比对到猪的基因组上,可以进行下一步分析。Table 3
表3
表3转录组测序基因组比对分析
Table 3The genome mapping analysis of transcriptome sequencing
| 样品 Sample | 质控后序列 clean reads | 比对序列(左端) mapped reads(left) | 比对率 Ratio | 比对序列(右端) mapped reads(right) | 比对率 Ratio |
|---|---|---|---|---|---|
| x1 | 38971687 | 31235281 | 80.10% | 28229426 | 72.40% |
| x2 | 54157864 | 37851248 | 69.90% | 33992351 | 62.80% |
| x3 | 42764905 | 33394956 | 78.10% | 30626858 | 71.60% |
| x4 | 43618327 | 31898918 | 73.10% | 29036097 | 66.60% |
| x5 | 45301686 | 32559473 | 71.90% | 28791916 | 63.60% |
| x6 | 51972678 | 40927677 | 78.70% | 37010351 | 71.20% |
新窗口打开
2.6 差异表达基因的筛选
本研究共鉴定到25 213个基因,以FDR<0.05,Fold Change>2的标准进行筛选,得到184个差异基因,筛选结果如图4所示,其中上调的基因114个,下调的基因70个,注释的(已知)141个,未注释的(未知)43个。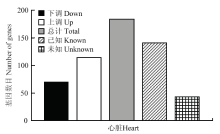 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4差异表达基因数统计
-->Fig. 4The number statistics of differentially expressed genes
-->
2.7 差异表达基因的聚类分析
利用ZBED6-KO猪和ZBED6-WT猪心脏组织之间的差异基因进行聚类分析。结果如图5所示:x4和x5首先聚在一起,然后和x2再聚在一起,x1和x3首先聚在一起,然后和x6再聚在一起。这表明ZBED6-KO组(x1、x3、x6)的3个个体表达模式更相似,ZBED6-WT组(x2、x4、x5)的3个个体表达模式更相似,证明实验样品和RNA-seq数据都是可靠的。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5184个差异表达基因的聚类结果
-->Fig. 5The clustering results of 184 differentially expressed genes
-->
2.8 差异表达基因的GO和IPA富集分析
对获得的差异表达基因进行功能注释和通路富集分析,共富集到13个显著的GO条目,33条显著的pathway通路。GO term中筛选到了肌肉发育相关和免疫反应相关等生物学进程(表4)。根据IPA分析筛选到RhoA信号、Cdc42信号、肝纤维化、上皮连接信号、肌动蛋白细胞骨架的信号等相关的pathway通路(表5)。Table 4
表4
表4差异基因富集的前10个GO分类
Table 4The 10 most GO categories of differential express gene enrichment
| GO术语 GO term | P值 P-value | 类型 Type | 基因个数 No.of gene |
|---|---|---|---|
| 防御反应 Defense response | 1.06E-09 | BP | 31 |
| 免疫效应的过程 Immune effector process | 0.000000121 | BP | 20 |
| 病毒过程的负调节 Negative regulation of viral process | 0.00000252 | BP | 9 |
| 干扰素-α反应 Response to interferon-alpha | 0.00151 | BP | 4 |
| 多细胞组织过程调节 Regulation of multicellular organismal process | 0.00803 | BP | 32 |
| 肌肉系统进程 Muscle system process | 0.0366 | BP | 10 |
| 淋巴细胞介导免疫 Lymphocyte mediated immunity | 0.0387 | BP | 8 |
| 细胞表面受体信号通路 Cell surface receptor signaling pathway | 0.0463 | BP | 12 |
新窗口打开
Table 5
表5
表5差异基因IPA富集前14条代谢信号通路
Table 5The 14 most pathway of differential express gene IPA enrichment
| 通路 Pathway | -ln(P值) -ln P-value | 分子 Molecules |
|---|---|---|
| 干扰素信号 Interferon Signaling | 5.56E+00 | OAS1,IRF9,IFIT1,IFIT3,MX1 |
| 模式识别受体在识别细菌和病毒方面的作用 Role of Pattern Recognition Receptors in Recognition of Bacteria and Viruses | 4.62E+00 | EIF2AK2,C1QC,OAS1,IFIH1,C1QB,OAS2,C1QA |
| 细胞-固体模式识别受体激活IRF Activation of IRF by Cytosolic Pattern Recognition Receptors | 4.38E+00 | DHX58,IRF9,IFIH1,ADAR,IFIT2 |
| 补体系统 Complement System | 4.10E+00 | C1QC,MASP1,C1QB,C1QA |
| Cdc42信号 Cdc42 Signaling | 3.20E+00 | MYL4,CDC42EP2,FCER1G,ARPC1B,APC2, |
| 无颗粒白细胞粘附和血球渗出 Agranulocyte Adhesion and Diapedesis | 2.93E+00 | MYL4,MMP11,HRH1,CXCL16,MYH6,MYL7 |
| 粒酶B信号 Granzyme B Signaling | 2.36E+00 | PRKDC,GZMB |
| 钙信号 Calcium Signaling | 2.29E+00 | GRIK1,MYL4,MYH6,TNNT1,MYL7 |
| 肝纤维化/肝星状细胞活化 Hepatic Fibrosis / Hepatic Stellate Cell Activation | 2.24E+00 | A2M,MYL4,MYH6,MYL7,IL10RA |
| 肌动蛋白细胞骨架的信号 Actin Cytoskeleton Signaling | 1.85E+00 | MYL4,MYH6,ARPC1B,APC2,MYL7 |
新窗口打开
2.9 差异表达基因qRT-PCR验证
为了验证RNA-seq测序得到数据的准确性,随机选取IFIT3、MX1、ISG12(A)、P2RY1、TNNT1、MYL7、XAF1、CEL、ATP1A2共9个基因进行qRT-PCR验证,以GAPDH作为内参基因,结果如图6所示。RNA-seq测序得到的各样品基因表达的FPKM值是由Reads比对数统计得来,qRT-PCR得到基因表达量是目的基因相对内参基因的相对表达量,这两种结果无法直接比较,且无法计算两种方法得到的表达量相关性。但ZBED6-KO组和ZBED6-WT组之间的差异倍数是具有可比性的,本研究将RNA-seq得到的FPKM值进行校正,即log2(fold_change)。因此根据两种方法得到的差异表达倍数来计算RNA-seq和qRT-PCR之间的相关性,判断两种方法的结果是否一致,结果如图6所示,qRT-PCR得到的9个基因的表达模式与RNA-seq得到的结果一致,进一步证实了RNA-seq测序的准确性。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6差异表达基因的 qRT-PCR 验证
qRT-PCR 中的误差线表示平均值的标准差(n=3)
-->Fig. 6qRT-PCR validation of differential expressed genes
The error line in qRT-PCR represents the standard deviation of the mean(n=3)
-->
3 讨论
ZBED6作为新的转录因子,自2009年被首次发现后,该基因就获得了高度关注[2, 3]。在小鼠、肉牛、绵羊等不同物种上对ZBED6的转录调控机制以及功能进行了不同程度的研究[2, 6-11],但在猪上的研究仍是空白,ZBED6影响心肌生长发育的分子机制也不是很清楚;近年来随着高通量测序和基因编辑技术CRISPR/ Cas9的发展及成熟,使得以转基因猪为模型研究ZBED6的分子功能成为可能[19,20,21]。前人研究显示ZBED6基因能够抑制IGF2的表达,
IGF2表达量的上调对心肌大小有重要的影响[3]。本研究利用qRT-PCR分析IGF2在心脏组织中的相对表达量,结果显示,ZBED6-KO组IGF2的表达量显著高于ZBED6-WT组,与小鼠、肉牛中的结果一致[2, 6-11, 22] ;表型特征鉴定结果显示ZBED6-KO猪的心脏比ZBED6-WT猪重,组织学分析结果显示ZBED6的敲除使家猪心肌肌纤维变粗,结缔组织减少,都说明了ZBED6的敲除在一定程度能够促进家猪心脏的生长发育。通过 RNA-seq 测序,在ZBED6-KO猪和ZBED6-WT猪心脏组织中得到184个差异表达基因,这些基因主要参与了肌肉系统进程、肌动蛋白细胞骨架、RhoA 通路及免疫反应等与肌肉发育相关的生物学过程和通路。
在肌肉发育相关途径中的上调基因主要有:CSRP3、LMCD1、ATP1A2、TNNT1,下调基因主要有:NPPA、P2RY1、HMCN2、KCNA5、PTGER3、MYL4、MYL7,说明ZBED6基因可能通过影响上述基因及其所在的通路调控心脏组织的发育和功能。
CSRP3是一个与心肌生长发育密切相关的基因,编码CRP3蛋白,表达与横纹肌及与肌源性分化相关组织,因此在心肌和骨骼肌组织中特异性表达[23,24,25]。CSRP3的表达与肌分化过程一致,是一个肌发生的正调节因子[23,24,25],并且其表达与肌分化过程一致,是一个肌发生的正调节因子[24]。细胞质CRP3 作为支架蛋白,与α肌动蛋白[26]、β-血影蛋白表达(t-cap)[23]及肌原纤维(Z-线)[27]相互作用,具备肌肉发育监管的结构特性。研究显示,CSRP3 缺陷小鼠显示扩张型心肌病(DCM)特征性症状的心衰[24]、增生性心肌炎及心脏功能紊乱的临床特征[24, 28]。本试验中CSRP3在ZBED6-KO猪心脏组织中的表达量是ZBED6-WT猪的1.96倍,推测ZBED6基因的敲除使家猪心脏组织中CSRP3 基因表达量明显升高,促进肌纤维和心肌的发育。
NPPA在肌肉系统进程及免疫反应等多个生物学过程和通路都有富集到,该基因在人上称为心房钠尿肽(ANP),在心脏中表达量最高,目前已经有研究显示,ANP在拮抗心脏肥大、减弱心肌收缩、利钠和利尿、血管舒张和重塑、脂类代谢等方面有重要作用[29,30]。ANP通过拮抗心脏成纤维细胞转化生长因子-H(TGF-h),进而抑制心肌纤维化和肥大。ANP通过与心肌细胞和血管平滑肌细胞膜上的相应受体结合,引起细胞内环一磷酸鸟苷(cGMP)水平上调,抑制外源性Ca2+内流和肌浆网Ca2+释放,使细胞内Ca2+减少导致血管扩张和减弱心肌收缩[31]。NPPA在ZBED6-WT猪心脏组织中表达量是ZBED6-KO猪的8.56倍,推测ZBED6基因的敲除导致家猪NPPA表达量明显降低,从而促进心脏组织心肌的发育及收缩。
RhoA作为信号分子开关调节一系列生物学功能,具有调控细胞骨架重排、诱导心肌肥厚、心肌细胞分化和血管平滑肌肥大等功能[32,33],其中MYL7、MYL4、CDC42EP2是RhoA通路中的关键基因。MYL7、MYL4都属于肌球蛋白(心肌收缩蛋白),在心脏发育和收缩功能中起着至关重要的作用,促进心肌细胞增殖分化[34]。Cdc42 是一个由CDC42EP2基因编码,与肾素相关的小G蛋白,在心脏、肝脏、肿瘤组织、胰腺中均有表达,Cdc42主要促进细胞的生长发育和增殖,通过控制肌动蛋白的骨架重组来调节细胞骨架[35,36]。在小鼠中发现,将其神经脊细胞Cdc42 特异性敲除时,可以引起细胞骨架的重构受阻,而且细胞的迁移也变得缓慢[37];在Cdc42 敲除小鼠中发现Cdc42 对肌动蛋白丝状伪足的形成以及迁移功能具有重要的调节作用,Cdc4在 AngiotensinⅡ诱导的心肌肥大模型中有促肥大的作用[38,39]。本试验中MYL7和MYL4在ZBED6-KO猪心脏组织中的表达量均显著低于ZBED6-WT猪,与前人研究以及ZBED6-KO猪心脏重高于ZBED6-WT猪的表型鉴定结果不一致;CDC42EP2在ZBED6-KO猪心脏组织中的表达量是ZBED6-WT猪的2.16倍,推测ZBED6基因的敲除导致家猪CDC42EP2表达量明显上调,CDC42EP2通过编码Cdc42 蛋白,从而促进家猪心脏的生长发育,与前人研究结果一致;MYL7、MYL4、CDC42EP2基因表达量的变化说明ZBED6基因敲除对心脏发育和功能的影响较为复杂,其调控机制需进一步研究。
差异基因富集分析显示,ZBED6基因敲除后影响了家猪免疫反应相关的生物学进程,富集到的主要基因有MYH6、OAS1和OAS2。2-5'寡腺苷合成酶OAS是细胞内的抗病毒蛋白,在哺乳动物先天性免疫系统中有重要作用[40,41]。研究显示,猪源OAS1和OAS2蛋白能抑制脑炎病毒在细胞中的增殖活性,而且能有效抑制子代病毒感染细胞,抑制子代病毒的增殖,降低子代病毒的数量,具有显著抑制病毒感染细胞的生物学特性[41,42]。本试验中OAS1、OAS2在ZBED6-KO猪心脏组织中的表达量分别是ZBED6-WT猪的2.21倍、2.29倍,推测ZBED6基因的敲除导致家猪OAS1、OAS2表达量上调,能够增强家猪抗病毒的能力。
按照功能将组成心脏的蛋白分为收缩蛋白和调节蛋白,其中肌球蛋白和肌动蛋白属于收缩蛋白,原肌球蛋白和肌钙蛋白属于调节蛋白[43]。MYH6是肌球蛋白重链,对心肌肥厚和心肌病有着重大影响[44]。有研究表明,在肥厚性心肌病和扩张性心肌病人体中MYH6的表达量下调1.74和2.88倍[44];本试验中,MYH6在ZBED6-KO猪心脏组织中的表达量均低于ZBED6-WT猪,这可能是导致ZBED6-KO猪心脏重量高于ZBED6-WT猪的重要原因。
4 结论
与ZBED6-WT猪相比,敲除ZBED6基因显著提高了家猪心脏重量,引发心脏组织发育的关键基因IGF2的表达量显著升高,心肌肌纤维变粗,结缔组织减少。RNA-Seq结果显示ZBED6基因敲除巴马小型猪和正常巴马小型猪共筛选到184个差异表达基因,其中上调基因114个,下调基因70个;差异表达基因的GO分析和IPA富集分析显示肌肉发育、免疫反应、RhoA信号和肌动蛋白细胞骨架的信号等相关的通路被显著富集到。因此,ZBED6基因可能通过上述生物学过程和通路影响了心脏组织的发育和功能。The authors have declared that no competing interests exist.
