0 引言
【研究意义】谷瘟病(致病菌Magnaporthe oryzae)是谷子生产中重要的流行性病害之一[1],严重时可造成大面积减产甚至绝收[2],种植谷子抗病品种是防治谷瘟病最经济、有效的措施。谷瘟病菌极易发生变异[3],因此鉴定各地区谷瘟病菌的无毒基因类型,了解谷瘟病菌无毒基因的分布及变异情况,可为谷子抗病基因的克隆指明方向。【前人研究进展】近年来在谷子抗瘟性鉴定方面开展了系列研究,而谷瘟菌的遗传变异研究鲜有报道。李志江等[4]利用采自谷子主产区的10个谷瘟病菌的单孢菌株对60份选自谷子核心种质资源的品种进行苗期接种鉴定,构建了谷瘟病菌生理小种的鉴别标准品种体系;SHARMA等[5]利用谷瘟病菌对155份谷子核心种质资源进行抗病鉴定,在温室接菌鉴定中,155个品种中有11个表现出对同一谷瘟病菌菌种具有相同的抗病能力;任世龙等[6]通过对64个谷瘟病菌的rDNA-IGS序列进行分析,发现谷瘟病菌的遗传分化非常明显,具有丰富的多样性,且具有寄主特异性。至今,谷瘟病菌无毒基因的相关研究尚未见报道,而稻瘟病菌无毒基因的相关研究已有大量报道。已有的研究发现,谷瘟病菌与谷子之间的互作关系与稻瘟病菌与水稻之间的互作相似,均符合Flor的“基因对基因”学说[7]。稻瘟病菌在侵染水稻过程中,水稻中的抗病基因与稻瘟病菌无毒基因编码产物相互作用,可引发寄主过敏性坏死反应(HR),从而抑制稻瘟病菌的进一步侵染,进而产生抗病作用[8,9]。病原菌在侵染寄主和寄主抵抗病原菌侵染的过程中具有强烈的相互选择,促使病原菌与寄主协同进化发展[10,11]。目前,多个稻瘟病菌的无毒基因被成功克隆,如Avr1-CO39[12]、Avr-pita[13]、ACE1[14]、AvrPiz-t[15]、Avr-pii、Avr-pia和Avr-pik[16]等。JIA等[17]通过酵母双杂交试验确定了Avr-pita176蛋白可直接结合到Pi-ta LRD区域,在植物细胞内启动Pi-ta介导的防御反应,并证实了“基因对基因”学说;IMAM等[18]将从印度东部的水稻种植区分离获得的63株稻瘟病菌用于该地区无毒基因的分布研究,发现AvrPiz-t和Avr-pik的分布水平为100%,Avr1-CO39的分布水平最低,仅为2%。【本研究切入点】谷瘟病菌与稻瘟病菌同属于有丝分裂孢子真菌中梨孢属(无性态为Pyricularia)的不同寄主分离物,稻瘟病菌中已克隆无毒基因可为谷瘟病菌无毒基因的克隆提供参考。本研究根据已克隆的7个稻瘟病菌无毒基因的序列设计特异性引物,对中国谷子主产区的76个谷瘟病菌菌株进行PCR检测,分析其含有的无毒基因的类型及变异情况。【拟解决的关键问题】鉴定不同地区谷瘟病菌所携带的无毒基因类型,确定无毒基因在菌株中的分布情况及其变异情况,为深入研究谷瘟病菌无毒基因变异机制打下基础,并为谷子抗病基因的克隆提供理论依据。1 材料与方法
1.1 材料
2014—2016年,从中国北方谷子产区采集具有典型谷瘟病症状的谷子病样,从中分离纯化谷瘟病菌,共分离获得谷瘟病菌单孢菌株76株(表1)。Table 1
表1
表176个谷瘟病菌供试菌株
Table 1Seventy-six strains of M. oryzae from foxtail millet
| 省(自治区、直辖市) Province (Autonomous region, municipality) | 采集地区 Sampling location | 菌株编号 Strain number | 省(自治区、直辖市) Province (Autonomous region, municipality) | 采集地区 Sampling location | 菌株编号 Strain number | |
|---|---|---|---|---|---|---|
| 河北 Hebei | 承德 Chengde | P1, P4 | 陕西 Shaanxi | 榆林 Yulin | P11, P48, P67 | |
| 邯郸 Handan | P5, P6, P7, P8 | 延安 Yan’an | P27, P37, P46, P47, P72 | |||
| 衡水Hengshui | P12 | 黑龙江 Heilongjiang | 肇东 Zhaodong | P6, P32, P33 | ||
| 保定 Baoding | P17, P61 | 双城 Shuangcheng | P10 | |||
| 唐山 Tangshan | P43 | 齐齐哈尔Qiqihaer | P75 | |||
| 张家口Zhangjiakou | P53 | 哈尔滨 Harbin | P30, P61 | |||
| 石家庄Shijiazhuang | P58 | 辽宁 Liaoning | 朝阳 Chaoyang | P18, P31, P54, P70, P71, P25 | ||
| 山西 Shanxi | 长治 Changzhi | P7, P13, P15, P23, P34, P36 | 吉林 Jilin | 四平 Siping | P21, P24 | |
| 大同 Datong | P14 | 吉林 Jilin | P39, P68 | |||
| 晋中 Jinzhong | P29, P55 | 公主岭Gongzhuling | P44 | |||
| 汾阳 Fenyang | P49 | 新疆 Xinjiang | 乌鲁木齐 Urumqi | P2, P62, P66 | ||
| 忻州 Xinzhou | P50 | 石河子 Shihezi | P45 | |||
| 河南 Henan | 安阳 Anyang | P20, P24, P69 | 内蒙古 Inner Mongolia | 赤峰Chifeng | P9, P52 | |
| 洛阳 Luoyang | P40, P57, P64, P65, P73 | 山东 Shandong | 临沂 Linyi | P3 | ||
| 郑州 Zhengzhou | P41, P56 | 济南 Ji’nan | P19, P26, P51, P63, P76 | |||
| 海南 Hainan | 三亚 Sanya | P59, P60 | 潍坊 Weifang | P28 | ||
| 北京 Beijing | 北京 Beijing | P38 | 淄博 Zibo | P35 |
新窗口打开
1.2 谷瘟病菌DNA的提取
将分离纯化的谷瘟病菌单孢菌株在PDA固体培养基上活化,挑取适量菌体转接到PDB液体培养基中,室温静止培养10 d,收集菌丝体,采用常规CTAB法提取谷瘟病菌的基因组DNA。1.3 引物设计
根据GenBank中7个已克隆的稻瘟病菌的无毒基因序列,利用Primer5软件设计无毒基因的引物,委托生工生物工程(上海)股份有限公司(上海生工)进行合成。引物序列见表2。Table 2
表2
表2用于扩增无毒基因的引物
Table 2Primers used for Avr-gene amplification
| 无毒基因 Avirulence gene | 引物序列 Primer sequence (5′-3′) | 预期片段大小 Expected fragment size (bp) | GenBank 登录号 GenBank accession number |
|---|---|---|---|
| ACE1 | F: CCCAGAGTTGGCGATGATGC | 611 | AJ704622.1 |
| R: ATGTGGCGGTGACAGAGGAC | |||
| Avr1-CO39 | F: CTCTGAACAACTAAGCAACA | 600 | AF463528.1 |
| R: CAACCTGGACTCTTATTGATC | |||
| Avr-piz-t | F: GATCAAATGAACACCAGGAA | 515 | EU837058.1 |
| R: CGATGAAGAATGGAAGAATG | |||
| Avr-pik | F: CTGCACACATCAACAGTCTT | 490 | AB498875.1 |
| R: CTACGTTGCCTTAACTGTGA | |||
| Avr-pia | F: TTATTGCTACCACCTTCCTC | 464 | AB498873.1 |
| R: GTAGTAACTGAGTTGGCGTT | |||
| Avr-pii | F: AATCCCCTTTTATTCC | 402 | AB498874.1 |
| R: GGCTTTTCCTATCCTTG | |||
| Avr-pita | F: GTTTCCGCCTTTATTGGTTTA | 1083 | AF207841.1 |
| R: TGACCGCGATTCCCTCCATT |
新窗口打开
1.4 PCR扩增及电泳检测
PCR反应体系(25 µL):2×Ex Taq Master Mix 12.5 µL,上下游引物各0.5 µL,模板DNA 2 µL(≤200 ng),ddH2O 9.5 µL,总体积25 µL。扩增程序为95℃预变性5 min;94℃ 30 s,51—58℃(根据不同引物而定)退火30 s,72℃延伸30—60 s,共35次循环;72℃ 10 min。PCR扩增产物用1.1%琼脂糖凝胶电泳检测。1.5 无毒基因的克隆测序及序列分析
使用凝胶回收试剂盒(美吉生物)进行PCR产物胶回收,回收产物与pMD19-T Vector 载体(宝生物工程有限公司)16℃过夜连接,通过热激法将连接产物转化到大肠杆菌DH5α感受态细胞中,挑取单克隆进行菌落PCR检测,并将阳性克隆送到上海生工进行测序。采用clustalX2.0软件对不同谷瘟病菌无毒基因全部测序序列进行核苷酸序列比对分析,挑选具有差异的无毒基因序列,通过在线(http://www.ebi.ac.uk/ Tools/st/)软件工具对核苷酸序列进行蛋白序列翻译,最后采用DNAman5.0软件对有差异的核苷酸序列和蛋白序列进行比对分析(文章序列比对图均采用DNAman5.0软件)。2 结果
2.1 谷瘟病菌无毒基因的分布
以谷瘟病菌76个单孢菌株的基因组DNA为模板,利用7个无毒基因的特异性引物进行PCR扩增,部分菌株的无毒基因扩增结果如图1所示。其中菌株P11和P34的无毒基因Avr1-CO39扩增片段较预期结果大490 bp,测序结果发现菌株P11和P34中的Avr1-CO39基因序列完全一致,均在启动子区插入了490 bp核苷酸,其中477 bp的插入序列与已知大小为477 bp的non-LTR retrotransposon:Mg-SINE(GenBank:AB607334.1)的相似度达99.16%(图2)。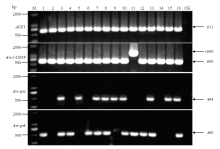 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1部分菌株的无毒基因的PCR扩增
M:Marker;CK:阴性对照(ddH2O)Negative control (ddH2O);1—16:病原菌菌株的DNA DNA samples of blast fungus strains
-->Fig. 1The PCR detection of Avr-genes in partial strains
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2谷瘟病菌P11插入核苷酸序列与non-LTR retrotransposon: Mg-SINE核苷酸序列比对分析
-->Fig. 2Sequence comparison analysis between inserted nucleotide sequence of P11 of M. oryzae and nucleotide sequence of non-LTR retrotransposon: Mg-SINE
-->
在76个谷瘟病菌中,无毒基因ACE1、Avr-pita、Avr1-CO39和AvrPiz-t的扩增率为100%,Avr-pik、Avr-pia和Avr-pii的扩增率分别为63.2%、42.1%和21.1%。7个无毒基因在不同地区谷瘟病菌中的扩增检测结果如表3所示。无毒基因ACE1、Avr-pita、Avr1-CO39和AvrPiz-t的扩增检出率为100%,不存在地区的差异性;无毒基因Avr-pik、Avr-pia和Avr-pii以不同频率在不同地区出现,除辽宁省Avr-pia的扩增检出率(50.0%)大于Avr-pik的扩增检出率(33.3%)之外,其余地区的无毒基因扩增检出率的高低符合Avr-pik>Avr-pia>Avr-pii。无毒基因Avr-pii在各个地区谷瘟病菌的扩增率均较低,扩增率最高的为河北省和辽宁省仅33.3%,最低为新疆自治区0,但在局部地区如河北承德Avr-pii的扩增率为100%。
Table 3
表3
表37个无毒基因在不同地区谷瘟病菌中的扩增检出率
Table 3The detection rates of 7 avirulent genes of M. oryzae from different regions
| 省(自治区、直辖市) Province (Autonomous region, municipality) | 菌株数 Number of strains | 无毒基因检出率Detection rate of avirulence gene (%) | ||||||
|---|---|---|---|---|---|---|---|---|
| ACE1 | Avr-pita | AvrPiz-t | Avr1-CO39 | Avr-pik | Avr-pia | Avr-pii | ||
| 河北Hebei | 12 | 100 | 100 | 100 | 100 | 66.7 | 41.7 | 33.3 |
| 山西Shanxi | 11 | 100 | 100 | 100 | 100 | 54.5 | 45.5 | 9.09 |
| 河南Henan | 10 | 100 | 100 | 100 | 100 | 70.0 | 30.0 | 10.0 |
| 陕西Shaanxi | 8 | 100 | 100 | 100 | 100 | 75.0 | 25.0 | 12.5 |
| 山东Shandong | 8 | 100 | 100 | 100 | 100 | 62.5 | 62.5 | 25.0 |
| 黑龙江Heilongjiang | 7 | 100 | 100 | 100 | 100 | 57.1 | 28.6 | 14.3 |
| 辽宁Liaoning | 6 | 100 | 100 | 100 | 100 | 33.3 | 50.0 | 33.3 |
| 吉林Jilin | 5 | 100 | 100 | 100 | 100 | 80.0 | 40.0 | 20.0 |
| 新疆Xinjiang | 4 | 100 | 100 | 100 | 100 | 50.0 | 25.0 | 0.00 |
| 内蒙古Inner Mongolia | 2 | 100 | 100 | 100 | 100 | 0 | 50.0 | 50.0 |
| 海南Hainan | 2 | 100 | 100 | 100 | 100 | 50.0 | 50.0 | 0 |
| 北京Beijing | 1 | 100 | 100 | 100 | 100 | 100 | 0 | 0 |
新窗口打开
2.2 谷瘟病菌群体单元型分析
基于不同无毒基因PCR扩增结果建立0—1数据库,7个无毒基因Avr-pita、AvrPiz-t、Avr-CO39、ACE1、Avr-pik、Avr-pia和Avr-pii的特异性指纹片段以a、b、c、d、e、f、g代替,将含有相同无毒基因带型的菌株归为一个单元型。结果显示76个供试菌株可以归为包含全部理论可能的8种不同的单元型(表4)。其中单元型AG2包含23个菌株,占供试菌株的30.2%,此类型菌株出现的频率最高,为优势单元型;单元型AG1和AG5,分别包含15和14个菌株,占供试菌株比例分别为19.7%和18.4%,说明供试谷瘟病菌菌株在群体遗传结构上层次较为丰富。Table 4
表4
表4谷瘟病菌的单元型
Table 4Haplotypes of M. oryzae strains
| 单元型类别 Type of haplotypes | 单元型 Haplotype | 菌株数 Number of strains | 所占百分比 Percentage (%) |
|---|---|---|---|
| AG1 | abcd | 15 | 19.7 |
| AG2 | abcde | 23 | 30.2 |
| AG3 | abcdf | 9 | 11.8 |
| AG4 | abcdg | 3 | 3.9 |
| AG5 | abcdef | 14 | 18.4 |
| AG6 | abcdeg | 3 | 3.9 |
| AG7 | abcdfg | 2 | 2.6 |
| AG8 | abcdefg | 7 | 9.2 |
新窗口打开
2.3 谷瘟病菌无毒基因的变异分析
2.3.1 无毒基因Avr-pia的变异分析 对全部32株谷瘟病菌的Avr-pia进行克隆测序分析发现,Avr-pia可划分为4种类型。类型一:Avr-pia-A与参考序列(AB498873.1)一致,以P41菌株为代表,包含10个菌株;类型二:Avr-pia-B以P5菌株为代表,包含20个菌株。以起始密码子的第一个碱基为+1位,上游为负,下游为正,该类型与参考序列相比均在-116、-109和-16 bp处分别存在C/T、G/T和C/A变异,但与参考序列的CDS区序列相同;类型三:Avr-pia-C型仅包含菌株P10,除包含Avr-pia-B型菌株的变异以外,在+150 bp处存在T/G变异,但为同义突变;类型四:Avr-pia-D仅包含菌株P18,除包含Avr-pia-B型菌株的变异以外,在+212 bp位点处存在C/T变异,导致该变异位点由编码苏氨酸变为编码异亮氨酸,该变异可能会使Avr-pia丧失无毒基因功能(图3)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3供试菌株的Avr-pia序列比对
A:Avr-pia碱基序列比对Comparison of nucleotide sequence of Avr-pia;B:Avr-pia氨基酸序列比对Comparison of amino acid sequence of Avr-pia
-->Fig. 3Sequence comparison of Avr-pia of tested strains
-->
2.3.2 无毒基因Avr-pii的变异分析 对全部16株谷瘟病菌的Avr-pii序列进行克隆测序分析发现,谷瘟病菌Avr-pii可以划分为3种类型(Avr-pii-A、Avr-pii-B和Avr-pii-C)。Avr-pii-A型共包含14个菌株,以菌株P4为代表,与参考序列(AB498874.1)一致;菌株P6在+139 bp处存在A/G变异,命名为Avr-pii-B型;菌株P3在+64 bp处存在A/G变异,命名为Avr-pii-C型,核苷酸的变异导致该位点由编码苏氨酸改为丙氨酸,Avr-pii-B型和Avr-pii-C型变异均为首次报道(图4)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4供试菌株的Avr-pii序列比对
A:Avr-pii碱基序列比对Comparison of nucleotide sequence of Avr-pii;B:Avr-pii氨基酸序列比对Comparison of amino acid sequence of Avr-pii
-->Fig. 4Sequence comparison of Avr-pii of tested strains
-->
2.3.3 无毒基因Avr-pita部分菌株变异分析 随机选取8株谷瘟病菌的Avr-pita进行测序分析,结果发现,与参考序列(AF207841.1)相比,Avr-pita序列变异丰富,主要变异形式包括单核苷酸的插入、缺失及多处位点的SNP。其中菌株P47与已报道的毒性菌株S06-13-1相似,在+31 bp处缺失T碱基,会使得Avr-pita失去无毒基因功能,导致与寄主不发生Pi-ta介导的抗病反应,代表菌株碱基序列比对如图5所示。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5供试菌株的Avr-pita 碱基序列比对
-->Fig. 5Nucleotide sequence of Avr-pita of tested strains
-->
3 讨论
近年来,谷瘟病在谷子主要产区普遍发生,河北、河南、山东等夏谷区发生尤为严重,生产上高感谷子品种的推广,加剧了谷瘟病的暴发。鉴定不同地区谷瘟病菌的无毒基因类型,可为将来克隆相应抗病基因,进而培育相关抗性基因的谷子品种提供参考。FARMAN等[19]对来自中国和日本等国的稻瘟病菌菌株Avr1-CO39区域基因组序列进行分析,发现大部分菌株的Avr1-CO39位点被单拷贝的RETRO5反转座子和一个重复元件REP1代替,基因组杂交表明RETRO5和REP广泛分布于稻瘟病菌中;TOSA等[20]对分离自水稻和马唐等寄主的梨孢菌进行Avr1-CO39序列分析,发现寄主为水稻的稻瘟病菌中的Avr1-CO39都不完整,而其他寄主的梨孢菌菌株具有完整的Avr1-CO39结构。与稻瘟病菌不同,本试验中所有谷瘟病菌中均检测出了Avr1-CO39,说明Avr1-CO39在谷瘟病菌进化中较为保守。Avr1-CO39的序列分析发现,来源地分别为陕西榆林和山西长治的谷瘟病菌菌株P11和P34具有相同的Avr1-CO39序列,均在Avr1-CO39的启动子区发生490 bp的基因插入,该插入序列与非长末端重复序列(非LTR)的反转录转座子Mg-SINE的相似度达99.16%。研究发现,非长末端重复序列反转录转座子在大多数真核生物的基因组存在,可产生内源性突变,成为潜在的基因毒性来源[21]。由此推测,菌株P11和P34可能是谷瘟病菌在与当地谷子品种的长期协同进化过程中为了躲避寄主谷子中抗性基因的识别而发生的变异,Avr1-CO39启动子区插入490 bp的片段后,该无毒基因是否有可能变为毒性基因,从而克服寄主的抗病性,还待进一步研究。
TAKAHASHI等[22]研究发现,毒性菌株S06-13-1中,由于Avr-pita的位点+31 bp处缺失了一个碱基T,导致寄主不发生Pi-ta介导的抗病反应。本试验发现,谷瘟菌株P47中的Avr-pita序列在+31 bp处缺失了一个碱基T,与毒性菌株S06-13-1中的序列相似,推测谷瘟菌株P47中的Avr-pita+31 bp处的突变可能会导致该无毒基因的功能丧失。ORBACH等[13]研究发现,无毒菌株的Avr-pita与亲本菌株O-137相比编码的蛋白有2—3个氨基酸不同,而毒性菌株的Avr-pita与菌株O-137相比编码的蛋白有7个氨基酸不同。本试验通过对8个谷瘟病菌的Avr-pita序列进行分析,发现谷瘟病菌的Avr-pita编码蛋白的氨基酸序列与菌株O-137均有3个以上的不同位点。谷瘟病菌的Avr-pita是否能够与谷子发生Pi-ta介导的抗病反应,还需进一步的试验验证。本试验发现Avr-pita的变异能力强,形式包括单核苷酸的缺失/点突变/插入等多种类型,这与前人研究结果相符[23,24,25]。利用谷瘟病菌Avr-pita丰富的变异研究谷瘟病菌的群体遗传结构可能是一种比较好的方法。
WOOLHOUSE等将病菌的无毒基因与寄主的抗病基因间的协同进化分为Arms race[26]和Red queen[26,27]两种模式,Arms race模式的基因进化多以碱基突变为主要形式,如Avr-pita[11]。Red queen模式进化方式则倾向于基因的增添和缺失,如Avr-pii和Avr-pia[16]。本试验中,Avr-pii和Avr-pia进化的主要方式为基因的增添和缺失,符合Red queen模式。但谷瘟病菌Avr-pii和Avr-pia进化的方式不仅有基因的增添和缺失,还含有序列内的核苷酸突变造成的氨基酸变异,如谷瘟病菌P18的Avr-pia在+212 bp位点处存在C/T变异,导致该变异位点由编码苏氨酸变为编码异亮氨酸。说明谷瘟病菌Avr-pii和Avr-pia的进化方式包含Arms race和Red queen两种模式。YOSHIDA等[16]的研究结果也表明,Avr-pik的进化方式不仅包括基因的增添和缺失,还包括基因内核苷酸序列的突变,笔者实验室对部分菌株的Avr-pik序列进行分析发现与YOSHIDA等[16]研究结果相符,相关结果待发表。
ACE1编码一个聚酮合酶和非核糖体肽合成酶的复合物,在水稻抗性基因识别真菌次生代谢产物的合成过程中触发[14]。余欢等[28]研究表明,只有牛筋草和狗尾草菌株中含有无毒基因ACE1,其他菌株中该基因已经缺失。本研究发现来源不同谷子产区的76个谷瘟病菌均能检测到ACE1,扩增检出率为100%,说明ACE1在谷瘟病菌进化中较为保守,并且没有丢失。
AvrPiz-t的序列不稳定,常发生变异,其变异机制主要为点突变导致的核苷酸替换和转座子Pot3的插入导致AvrPiz-t无毒功能的丧失。LI等[15]对比分析了9个毒性菌株及无毒菌株的AvrPiz-t序列,发现有毒菌株70-15中有转座子Pot3的插入和单个核苷酸的替换。本研究中电泳检测未检测到AvrPiz-t含有大片段的基因插入,测序分析发现AvrPiz-t序列存在变异,其变异方式主要是碱基的缺失、插入和点突变等。
无毒基因ACE1、Avr-pita、Avr1-CO39和AvrPiz-t在76个不同谷子产区的谷瘟病菌中的扩增率为100%,说明这些无毒基因在不同谷子产区均有分布,不存在地区的差异性。Avr-pia和Avr-pii的扩增率分别为42.1%和21.1%,但这两种无毒基因的CDS区相对保守,在局部谷子种植产区如河北承德地区Avr-pii的扩增率为100%。Avr-pik的进化方式不仅包括基因的增添和缺失,还包括基因内核苷酸序列的突变。
4 结论
谷瘟病菌AG2单元型为优势单元型,其次是单元型AG1和AG5。明确了不同地区谷瘟病菌所包含的无毒基因类型及变异方式,具体表现为谷瘟病菌中无毒基因ACE1、Avr-pita、Avr1-CO39和AvrPiz-t不存在地理来源差异,扩增率均为100%,变异方式主要为单核苷酸变异。而无毒基因Avr-pik、Avr-pia和Avr-pii在各地分布有差异。其中河南、陕西及吉林等地区无毒基因Avr-pik扩增率超过70%。山东省的无毒基因Avr-pia的扩增率最高为62.5%。变异方式主要为整个无毒基因的缺失,同样也包含核苷酸序列的单碱基变异。The authors have declared that no competing interests exist.
