0 引言
【研究意义】枯萎病(Fusarium wilt,FW),由半知菌亚门尖孢镰刀菌西瓜转化型(fon)寄生引起,是世界范围内西瓜生产中导致产量和品质降低最严重的一种真菌土传病害。目前,已报道的fon生理小种有0、1、2和3共4个,除了fon3,一些商业西瓜品种在一定程度上已经整合了生理小种fon0、fon1和fon2抗性[1]。但大部分品种不抗枯萎病,病菌能够在西瓜种植区很快扩散、蔓延并长期存活。因此,开发与枯萎病抗性紧密连锁的易于操作、成本低廉的分子标记,快速普及西瓜枯萎病分子标记辅助育种技术,缩短育种周期,快速提高西瓜品种的枯萎病抗性,符合中国重茬严重的西瓜产业发展需要。【前人研究进展】西瓜抗枯萎病分子标记研究起步较早,利用兼抗生理小种0号、1号和2号的野生西瓜种质PI196431-FR对枯萎病抗性基因进行了广泛的研究。XU等[2]利用野生种质材料PI196431-FR获得与生理小种1抗性基因相关的RAPD(random amplified polymorphic DNA)标记,并将其转化成了SCAR(sequence characteristic amplified region)标记[3]。LEVI等[4-5]先后构建了西瓜的两个遗传图谱,均包含许勇等[3]开发的枯萎病抗性标记。HAWKINS等[6]利用PI196431-FR和感枯萎病品种‘New Hampshire Midget’构建的遗传图谱鉴定到3个生理小种1号抗性和4个2号生理小种抗性相关的RAPD标记。丁群英等[7]也利用PI196431-FR获得与生理小种2抗性基因相关的RAPD分子标记。此外,LIN等[8]利用Sugar Baby和JSB筛选到枯萎病抗性相关的RAPD标记,并将其转化为SCAR标记用于分子标记辅助筛选。利用不同抗性水平的西瓜核心种质资源,羊杏平等[9]鉴定到6个与枯萎病生理小种1抗性显著关联的的SRAP(sequence-related amplified polymorphism)分子标记。随着基因组学和分子标记技术的快速发展,西瓜枯萎病研究取得实质性进展,开发的分子标记已应用于抗病育种工作。张屹等[10] 通过对高抗枯萎病生理小种1的栽培西瓜Calhoum Gary的研究发现,其抗性由单个显性基因Fon-1控制,与以往的研究结果一致[11],并利用Fon-1基因区间的SNP信息开发3个CAPS/dCAPS标记7716_fon、7419_fon和4451_fon。焦荻等[12]在此基因区间新开发出一个更为紧密连锁的dCAPS标记502124_fon。LAMBEL 等[1]的研究则认为枯萎病生理小种1抗性是一个典型的数量性状,并检测到一个西瓜枯萎病生理小种1抗性的主效QTL,命名为Fo-1.1,位于1号染色体遗传图谱的2.3—8.4 cM,Fon-1[10]就位于这个主效QTL的置信区间。此外,在1、3、4、9和10号染色体也检测到了效应较小的QTL[1]。【本研究切入点】以往检测到的枯萎病抗性连锁的分子标记多为RAPD,它是显性标记,无法区分纯合与杂合体的基因型,并且重复性差。将RAPD转化为SCAR,虽可以增加RAPD标记的稳定性和信息量,但由于技术原因,这些标记的实际应用可信度仍然偏低。随着西瓜全基因组测序的完成[13],利用重测序信息也检测到了与枯萎病抗性相关的基因和QTL,紧密连锁的分子标记为SNP或是基于SNP的CAPS/dCAPS标记。基于测序的SNP标记能直观地反映其DNA水平的多态性,但是测序成本限制了其直接应用。SNP衍生的 CAPS/dCAPS标记属于共显性标记,是结合PCR扩增与酶切反应的一种新型DNA标记,检测需要酶切,过程繁琐,周期较长,技术要求较严,成本较高。利用两个材料中的插入缺失(insertion/deletion)核苷酸位点而设计的分子标记称为InDel标记,具有变异稳定、多态性强、检测容易(同SSR标记)、成本低廉等优点,已经受到越来越多的关注。利用重测序技术开发的InDel标记在水稻[14-15]、小麦[16]、油菜[17]、黄瓜[18]等作物中已得到应用,但在西瓜中尚无报道。【拟解决的关键问题】本研究利用高抗枯萎病生理小种1的栽培品系‘ZXG01478’为研究材料,结合抗病鉴定结果和实验室已构建的高密度遗传连锁图谱[19]进行QTL定位,在1号连锁群鉴定了一个主效QTL(fon1),能够解释80.18%的表型变异。为了开发标记加密QTL区间,对两个亲本材料进行了22 x深度的重测序,检测两个亲本间的InDel遗传变异。根据主效QTL置信区间的InDel信息开发InDel标记实现对主效QTL(fon1)的精细定位。本研究结合QTL定位和亲本重测序开发与枯萎病抗性紧密连锁的易于检测通用的InDel分子标记,为西瓜枯萎病分子标记辅助育种提供技术支撑,同时加快该基因位点图位克隆的进程。1 材料与方法
1.1 试验材料
利用高抗枯萎病生理小种1的栽培西瓜品系‘ZXG01478’为母本,高感枯萎病生理小种1的栽培西瓜品系‘14CB11’为父本[19-21]杂交获得F1,进一步自交,获得93个单株的F2群体。父母本、F1和F2群体的各个单株在2013年秋季种植,自交留种并获得F3单瓜种子。供试的种质资源材料为国家西瓜甜瓜中期库(河南郑州)提供的130份包括地方品种、育成品种(系)和野生种质在内的西瓜材料[22],具有广泛的代表性。2014年利用父母本、F1和F3单瓜种子和130份西瓜资源材料进行枯萎病抗性鉴定,每个株系/品系50个单株,两个重复,反映亲本、F1、F2和西瓜资源材料的抗病性。所有材料于中国农业科学院郑州果树研究所基地玻璃温室内按常规方法种植。1.2 苗期人工接种抗枯萎病鉴定
西瓜枯萎病病菌为尖孢镰刀菌西瓜专化型(Fusarium oxysporum f. sp. niveurm)生理小种1。病菌接种参照吉加兵[23]的病麦粒拌土接种法进行。病情分级参照MARTYN等[24]的标准,以病株率作为抗性评价依据,即高抗(HR)病株率为0%—20%,抗病(R)病株率为21%—40%,中抗(MR)病株率为41%—60%,感病(S)病株率为61%—80%,高感病(HS)病株率为81%—100%。其中病株率<60%为抗病类型(包括HR、R和MR),病株率>61%为感病类型(包括S和HS)。1.3 QTL定位分析
利用SLAF-seq开发的SNP标记构建了高密度的遗传连锁图谱[19],结合抗病鉴定数据(病株率)进行西瓜枯萎病生理小种1的QTL定位。QTL扫描采用WinQTL cartographer 2.5软件(http://statgen.ncsu.edu/ qtlcart/WQTLCart.htm)的复合区间作图法(composite interval mapping,CIM)[25]。基于标准模型Model 6,余因子的选择采用前后回归相结合(forward-backward stepwise regression)的方法(Pin=0.05和Pout=0.05)。背景标记的数目、窗口大小、扫描间距和运行速度分别设为5、10 cM、5 cM和1 cM。QTL显著性的阈值用1 000次的排布测验(permutation test)[26]确定。采用P=0.05的显著性水平来确认QTL。1.4 QTL区域InDel标记的开发
对母本和父本材料利用Illumina HiSeqTM2500进行了22 x深度的重测序,参考基因组为全基因组测序的西瓜品种‘97103’[13]。InDel的检测主要使用GATK[27]软件工具包实现。利用自编的Perl程序提取插入缺失相应位置前后500 bp的序列,利用Primer 5.0软件[28]设计对应的InDel引物对。1.5 开发InDel标记的基因型鉴定和连锁分析
新鲜叶片的DNA提取采用改良的CTAB方法[29],InDel引物的PCR扩增参照笔者实验室的方法[30]进行。对PCR产物进行8%聚丙烯酰胺凝胶电泳、显影、染色和带型判读。加上InDel分子标记在F2群体的基因型鉴定结果和LG1上其他的SNP标记运用JoinMap 4.0软件(http://www.kyazma.nl/index.php/mc.JoinMap)构建遗传连锁图谱。软件参数设置如下:拟合度阈值≤5,最大重组率<0.4,最小LOD为2.0,作图函数选择Kossambi函数[31]。
2 结果
2.1 父母本、F1和F2群体的抗病鉴定
抗病母本‘ZXG01478’的苗期病株率为0,感病父本‘14CB11’的病株率为100%,F1苗期的病株率为21%,为抗病。F2群体的93个单株的病株率频率分布呈现明显的双峰分布(图1),以60%的病株率为界把群体分为抗病和感病两大类,72株抗病,20株感病,分离比基本符合3:1的分离比(χ2=0.52,P=0.47),表明西瓜枯萎病生理小种1抗性受一个主效QTL控制。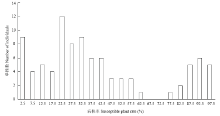 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1F2群体的病株率(%)频率分布图
-->Fig. 1Frequency distribution of susceptible plant rate (%) in F2 population
-->
2.2 QTL初定位
高密度的遗传连锁图谱[19]结合F2群体抗病鉴定表型进行了全基因组的QTL扫描。仅在LG1(1号连锁群)检测到一个效应很大的QTL,fon1(图2-左),其LOD峰值为26.05,解释80.18%的表型变异,为枯萎病抗性主效QTL。枯萎病抗性等位基因来自母本,加性效应为35.08%。置信区间为LG1的161.59—195.78 cM,对应的物理位置为1号染色体的193 333—2 775 577 bp。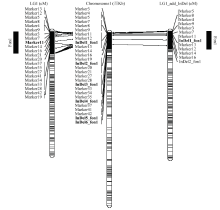 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2标记开发前后LG1(左和右)与物理图谱(中间)的比较分析左:LG1原始图谱,加粗的标记为QTL峰值标记。中间:1号染色体的物理图谱,加粗的标记为新开发的InDel标记。右:加上新开发的标记后LG1遗传图谱,加粗的标记为QTL峰值标记。
-->Fig. 2Integration of genetic maps (left and right) and physical map (middle)Left: Original linkage map for LG1. Marker in bold was located on the peak QTL region. Middle: Physical map for chromosome 1. Markers in bold were the new developed markers. Right: Re-constructed linkage map for LG1. Marker in bold was located on the peak QTL region.
-->
2.3 目标QTL区域InDel标记的开发
通过高通量的重测序,在父母本间共检测到45 065个InDels。在QTL的置信区间发现19个插入缺失片段大于20 bp的InDels,经引物设计与亲本筛选,获得理想引物12对,根据位于端点位置的插入缺失选取了6对InDel引物利用F2群体进行验证。引物相关信息见表1、表2。Table 1
表1
表1QTL区域开发的InDel的基本信息
Table 1Basic information of InDels on QTL region
| InDel名称 InDel name | 物理距离 Physical distance | InDel序列 Seq of InDel | InDel长度 Length of InDel |
|---|---|---|---|
| InDel1_fon1 | Chr1:464377 | TTTATTTTTTATTTTTTATTT | 21 |
| InDel2_fon1 | Chr1:2122292 | ATCTCCACCAGAGAATGCCCTCCCCCCCCACCCCC | 35 |
| InDel3_fon1 | Chr1:2269776 | GCACCAACATGCAAACAAACTCAAGAAGTTAGAATTCGAATTT | 43 |
| InDel4_fon1 | Chr1:2476380 | AGATTTGTCACAGACAAGGATATGGAAAGATA | 32 |
| InDel5_fon1 | Chr1:2650838 | GATCACGAGAGGTTTGTGCTCCTTCA | 26 |
| InDel6_fon1 | Chr1:2665461 | TACTATGAACTAATAATTTAAAATAATGGTTGTCAGAACTATTATG | 46 |
新窗口打开
Table 2
表2
表2开发标记的引物序列
Table 2Primer pairs for developed markers
| InDel名称 InDel name | 前引物 F_primer (5′ to 3′) | 后引物 R_primer (5′ to 3′) |
|---|---|---|
| InDel1_fon1 | TTCCAAAAGTGCAGATTTC | CACATGGGGATTGACTAAG |
| InDel2_fon1 | GAGCGCAAGGTAAGATCCAT | AAGAACTATACGTGGTCATT |
| InDel3_fon1 | CACAATTATAGCCTAGCCAAG | CGTATCAACTCGAAGGAAGAT |
| InDel4_fon1 | TAAGCATGGTGGAATGGAAG | CCAATAGTCCGTCATTTGTG |
| InDel5_fon1 | CAGAGGTCGTGTTCTTCAGG | TACTCAGTCAGTAATCCCAC |
| InDel6_fon1 | TATGTTTCCTTTCTTCCACCC | ACCTCCGACTACAACCACCTC |
新窗口打开
2.4 QTL区域的初步精细定位
首先挑选F2群体中的20个单株利用新开发的6个InDel标记进行基因型鉴定,检测到5个交换单株。如图3所示,F2株系,13QB135-010、13QB135-015和13QB135-020皆表现为抗枯萎病,将fon1定位于InDel2_fon1的上游;13QB135-009表现为高感枯萎病,将fon1定位于InDel2_fon1的上游;13QB135-018表现为高感枯萎病,将fon1定位于InDel3_fon1的上游。5个交换单株的基因型表型鉴定将fon1的区域锁定于InDel2_fon1的上游区域,因此,仅利用InDel1_fon1和InDel2_fon1对F2群体的全部93个株系进行基因型鉴定。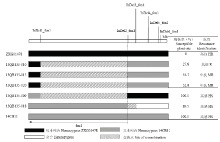 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3目标QTL区域的初步精细定位标记顺序为物理图谱的距离
-->Fig. 3Primary fine-mapping of target QTLThe markers in the order of the physical distance for reference watermelon line 97103
-->
2.5 连锁和QTL重新分析
利用JoinMap 4.0对LG1上的SNP标记和InDel1_ fon1、InDel2_fon1的遗传距离重新进行计算。加上两个新开发的标记后,LG1的遗传图谱和物理图谱的共线性(图4)与原始的图谱[19]基本没有变化,局部的共线性很好。利用重新分析的LG1和其他10 条连锁群重新进行了QTL扫描。结果同样在LG1检测到了效应很大的QTL(图2-右),其中新开发的一个分子标记InDel1_fon1出现在该QTL的峰值,并且LOD值为31.65,解释91.46%的表型变异。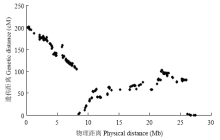 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4加上新开发的标记后遗传和物理图谱的共线性分析
-->Fig. 4Plot of genetic vs. physical distance for re-constructed LG1
-->
2.6 主效QTL fon1的精细定位
利用InDel1_fon1、InDel2_fon1及其中间和附近的8个SNP标记在F2群体中的基因型分析发现了9个交换单株。如图5所示,6个抗病交换单株中,13QB135-010和13QB135-044将fon1定位于Marker16的下游,13QB135-052和13QB135-069将fon1定位于Marker14和Marker11之间,13QB135-083将fon1定位于Marker16和Marker7之间,13QB135-105将fon1定位于Marker7的上游。3个感病交换单株中,13QB135-027和13QB135-009将fon1定位于Marker21的下游,13QB135-038将fon1定位于InDel2_fon1的下游。综合分析,将fon1的置信区间锁定于Marker14(Chr1:704 880 bp)和Marker11(Chr1:458 752 bp)之间,仅包含246 kb的物理距离。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5目标QTL区域的精细定位标记顺序为重新构建图谱的遗传距离
-->Fig. 5Fine-mapping of target QTLThe markers in the order of the genetic distance for re-constructed LG1
-->
2.7 开发InDel标记在在不同抗性品种中的验证分析
为验证所开发InDel标记的准确性和适用性,利用国家西瓜甜瓜中期库(河南郑州)提供的130份西瓜资源进行分析。田间抗性鉴定结果显示,共有36个抗病品种和94个感病品种。除了InDel1_fon1,还利用张屹等[10]开发的标记7716_fon对这些西瓜资源进行了基因型鉴定[22]。结果显示,标记InDel1_fon1与7716_fon在这些资源材料中基因型鉴定结果完全一致,与田间抗病表型性状的符合率达到70.8%,低于张屹等[10]利用164份西瓜育种材料的鉴定结果(98.7%),这可能与搜集到的西瓜资源遗传多样性及枯萎病抗性机理不同有关。部分资源分子鉴定和抗病鉴定结果见表3。Table 3
表3
表3部分西瓜资源的分子鉴定与抗病鉴定结果的比较
Table 3The comparison of molecular identification and resistance identification results in watermelon partial inbred lines
| 种质名称 Germplasm name | 种质类型 Germplasm type | 分子鉴定 Molecular identification | 病株率(%)/级别 susceptible plant rate/Classification | 种质名称 Germplasm name | 种质类型 Germplasm type | 分子鉴定 Molecular identification | 病株率(%)/级别 susceptible plant rate/Classification | |
|---|---|---|---|---|---|---|---|---|
| 白瓜籽 Baiguazi | 地方品种 Local variety | - | 100%/高感 HS | 久比例 Jiubili | 育成品种(系) Improved variety | + | 65%/感病 S | |
| 兰州黑皮Lanzhouheipi | 地方品种 Local variety | - | 94%/高感 HS | Dixielee | 育成品种(系)Improved variety | +/- | 31%/抗病 R | |
| 广州花皮Guangzhouhuapi | 地方品种 Local variety | - | 100%/高感 HS | All Sweet | 育成品种(系)Improved variety | +/- | 58%/中抗MR | |
| 三白瓜 Sanbaigua | 地方品种 Local variety | + | 92%/高感 HS | 96B41 | 育成品种(系)Improved variety | + | 32%/抗病 R | |
| 北瓜 Beigua | 地方品种 Local variety | - | 100%/高感 HS | 查理斯顿 Charleston | 育成品种(系)Improved variety | - | 67%/感病 S | |
| 抚州瓜 Fuzhougua | 地方品种 Local variety | + | 14%/高抗 HR | Moon and Star | 育成品种(系)Improved variety | - | 85%/高感 HS | |
| 阿克柯孜外Akekeziwai | 地方品种 Local variety | - | 100%/高感 HS | Tomato seed | 育成品种(系)Improved variety | - | 90%/高感 HS | |
| 阿拉克孜外Alakeziwai | 地方品种 Local variety | - | 100%/高感 HS | Sugar Baby | 育成品种(系)Improved variety | - | 93%/高感 HS | |
| 卡拉塔吾孜Kalatawuzi | 地方品种 Local variety | - | 100%/高感 HS | Black Stone | 育成品种(系)Improved variety | - | 94%/高感 HS | |
| 红瓜子 Hongguazi | 地方品种 Local variety | + | 100%/高感 HS | Black diamond | 育成品种(系)Improved variety | - | 100%/高感 HS | |
| 核桃纹 Hetaowen | 地方品种 Local variety | - | 100%/高感 HS | 墨西哥黑皮 Moxigeheipi | 育成品种(系)Improved variety | + | 95%/高感 HS | |
| 马铃瓜 Malinggua | 地方品种 Local variety | + | 100%/高感 HS | 红1号 Hong1 | 育成品种(系)Improved variety | + | 36%/抗病 R | |
| PI 537277 | 野生种质 Wild variety | - | 81%/高感 HS | 短蔓 Duanman | 育成品种(系)Improved variety | - | 64%/感病 S | |
| PI 494528 | 野生种质 Wild variety | + | 94%/高感 HS | 黄金 Huangjin | 育成品种(系)Improved variety | - | 95%/高感 HS | |
| 吉庆野西瓜 Jiqingyexigua | 野生种质 Wild variety | - | 84%/高感 HS | 黄小玉 Huangxiaoyu | 育成品种(系)Improved variety | - | 80%/感病 S | |
| Mpamophbiu | 育成品种(系) Improved variety | - | 95%/高感 HS | 香久山 Jiuxiangshan | 育成品种(系)Improved variety | - | 93%/高感 HS | |
| 火洲1号 Huozhou 1 | 育成品种(系) Improved variety | - | 100%/高感 HS | 斯拉夫拍卡 Silafupaika | 育成品种(系)Improved variety | - | 100%/高感 HS | |
| 桂引6号 Guiyin 6 | 育成品种(系)Improved variety | - | 70%/感病 S | 苏联2号 Sulian2 | 育成品种(系)Improved variety | - | 100%/高感 HS | |
| 华东26号 Huadong 26 | 育成品种(系)Improved variety | - | 100%/高感 HS | 苏蜜1号 Sumi1 | 育成品种(系)Improved variety | - | 95%/高感 HS | |
| 金瓜 Jingua | 育成品种(系) Improved variety | - | 62%/感病 S | 无杈早 Wuchazao | 育成品种(系)Improved variety | - | 100%/高感 HS | |
| 洛菲林 Luofeilin | 育成品种(系) Improved variety | - | 100%/高感 HS | 长蜜宝 Changmibao | 育成品种(系)Improved variety | - | 85%/高感 HS | |
| 克伦生 Kelunsheng | 育成品种(系) Improved variety | - | 33%/抗病 R | Sugarlee | 育成品种(系)Improved variety | + | 13%/高抗 HR | |
| Calhoun Gray | 育成品种(系) Improved variety | - | 35%/抗病 R | 黄皮小西瓜 Huangpixiaoxigua | 育成品种(系)Improved variety | - | 87%/高感 HS |
新窗口打开
3 讨论
利用新一代测序技术开发标记构建遗传连锁图谱,标记数量多,并覆盖基因组的大部分区域[19,32-33],为后续的重要性状基因定位研究提供了良好的平台。本研究仅仅利用包含93个单株的F2群体将枯萎病抗性主效QTL缩减至246 kb的物理区间内,材料的选取和试验方法的选择至关重要:(1)两个亲本抗性差异显著(母本高抗,父本高感),F1表现为抗病,F2群体的抗病和感病分离比符合3:1的分离比,表明在这个群体中西瓜枯萎病生理小种1抗性是典型的由单个显性抗性基因控制[34],只要标记密度足够大,较容易将目标QTL缩减至较小的范围,实现其精细定位。(2)利用简化基因组测序开发的全基因组的SNP标记构建高密度的遗传连锁图谱[19],并利用目标QTL区域的InDel信息加密QTL区间,两个亲本在目标QTL区域的主要遗传变异都考虑在内,开发高密度的标记加密目标QTL区间是可能的。随着西瓜基因组测序的完成[13],西瓜枯萎病抗性研究取得突破性进展。基于西瓜基因组重测序信息,张屹[10]、焦荻[12]等近年先后开发了与Fon-1紧密连锁的CAPS/dCAPS标记502124_fon、7716_fon、7419_fon和4451_fon,通过blast分析发现它们分别对应于1号染色体的502 124 bp、459 624 bp、458 344 bp和208 090 bp,其中502124_fon和7716_fon与Fon-1的连锁更为紧密,但Fon-1的具体位置、界限以及西瓜高抗枯萎病的分子机理并不十分清楚。本研究的精细定位分析将目标QTL区域锁定在1号染色体的458 752—704 880 bp的物理区间,结合张屹等[10]的研究,Fon-1位于7419_fon的下游,据此进一步将fon1的置信区间锁定在1号染色体的459 624—704 880 bp。在F2群体验证中,主效QTL紧密连锁的分子标记InDel1_fon1与7716_fon完全共分离,InDel1_fon1与502124_fon仅有两个抗枯萎病的单株基因型不同,且是纯合与杂合的区别。虽然无法利用7716_fon和502124_fon在F2群体中进一步缩减QTL区间,但至少可以证明新开发的标记InDel1_fon1与目标基因的连锁程度非常紧密,再加上InDel分子标记自身变异稳定、检测容易,不需要酶切等繁琐的步骤,成本低廉,选择效率高等优点,非常易于普及应用。
4 结论
QTL初定位在LG1检测到一个西瓜枯萎病生理小种1抗性主效QTL(fon1),验证了1号染色体上Fon-1的存在。利用全基因组的SNP标记(高密度遗传连锁图谱)和目标QTL区域的InDel标记将fon1置信区间缩减至246 kb的物理距离,实现了枯萎病抗性主效QTL的精细定位。并新开发了简便实用、高效低耗、紧密连锁的InDel标记(InDel1_fon1),可更好地用于西瓜抗枯萎病的分子辅助育种,同时加快了西瓜枯萎病抗性育种进程和抗性基因的克隆与功能验证。The authors have declared that no competing interests exist.
