 ,1, 任毅1, 时晓磊1, 王丽丽1, 张新忠2, 苏力坛·姑扎丽阿依1, 谢磊1, 耿洪伟
,1, 任毅1, 时晓磊1, 王丽丽1, 张新忠2, 苏力坛·姑扎丽阿依1, 谢磊1, 耿洪伟 ,1
,1Genome-Wide Association Analysis of Superoxide Dismutase (SOD) Activity in Wheat Grain
WANG JiQing ,1, REN Yi1, SHI XiaoLei1, WANG LiLi1, ZHANG XinZhong2, SULITAN· GuZhaLiAYi1, XIE Lei1, GENG HongWei
,1, REN Yi1, SHI XiaoLei1, WANG LiLi1, ZHANG XinZhong2, SULITAN· GuZhaLiAYi1, XIE Lei1, GENG HongWei ,1
,1通讯作者:
责任编辑: 李莉
收稿日期:2020-10-31接受日期:2020-12-28网络出版日期:2021-06-01
| 基金资助: |
Received:2020-10-31Accepted:2020-12-28Online:2021-06-01
作者简介 About authors
王继庆,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (2452KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
王继庆, 任毅, 时晓磊, 王丽丽, 张新忠, 苏力坛·姑扎丽阿依, 谢磊, 耿洪伟. 小麦籽粒超氧化物歧化酶(SOD)活性全基因组关联分析[J]. 中国农业科学, 2021, 54(11): 2249-2260 doi:10.3864/j.issn.0578-1752.2021.11.001
WANG JiQing, REN Yi, SHI XiaoLei, WANG LiLi, ZHANG XinZhong, SULITAN· GuZhaLiAYi, XIE Lei, GENG HongWei.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】小麦是全球重要粮食作物之一。随着人们生活水平的提高,优质已成为中国小麦主要育种目标[1]。超氧化物歧化酶(superoxide dismutase,SOD)对小麦面粉色泽及面制品营养品质有重要作用[2,3,4,5,6]。SOD能通过歧化反应过程中释放出的分子氧将面粉及面制品中类胡萝卜素等色素分子的共轭双键氧化为单键,从而对面粉颜色起到漂白作用[2,3,4]。面团流变学特性是小麦品质重要影响因素之一[7]。SOD通过影响面团中蛋白质的分子内或分子间巯基和二硫键交换反应,产生更多的二硫键,将肽链上的氨基酸残基聚拢起来,形成的网络结构中蛋白质分子的排列更有序,从而稳定蛋白质的构象,改善面团流变学特性[6,7,8]。此外,SOD也能通过歧化超氧化物阴离子自由基(O2-),生成的过氧化氢和分子氧,进一步将面粉中的膳食纤维组分降解为具有益生元功效的低聚糖等成分[9]。因此,挖掘小麦籽粒SOD活性基因,进而培育高SOD活性小麦品种,对改善面粉及面制品营养品质具有重要的意义。【前人研究进展】在植物中,SOD可分为Cu/Zn-SOD、Mn-SOD和Fe-SOD 3种类型[3]。小麦、玉米等主要谷物叶片的SOD活性研究表明,玉米具有最高的SOD活性,在2 500 U·g-1以上,分别是小麦、大豆和水稻的1.5、2和3倍[10]。BHARTI等[11]认为不同小麦品种(系)在最大分蘖期、开花期和籽粒灌浆期叶片SOD活性均存在显著性差异(P<0.05)。EYIDO?AN等[12]对小麦幼苗时期根冠组织进行了研究,也发现SOD活性在不同品种间相差1.5—2倍。刘家林等[13]在水稻全基因组水平上对SOD基因家族进行分析,在第3、4、5、6、7和8染色体上共发现9个SOD基因,包括6个Cu/Zn-SOD基因、2个Fe-SOD基因和1个Mn-SOD基因。赵艳等[14]将谷子中的8个SOD基因定位在第2、3、4、6、7和9染色体上,其中包括5个Cu/Zn-SOD基因和3个Mn-SOD基因。WU等[15]和BAEK等[16]利用中国春缺四体和双端体材料,将小麦籽粒Mn-SOD活性基因定位于小麦2A、2B和2D染色体长臂上,将Cu/Zn-SOD活性基因定位于7A、7B和7D染色体长臂上,其中,位于2D长臂上的Xfbb377—Xf'bcd410区段存在一个影响小麦种子中SOD含量的上位QTL位点[17]。JIANG等[18]从小麦全基因组中鉴定出26个SOD基因,分布于第2、4和7同源染色体上。KUMAR等[19]在小麦6D染色体上克隆到Mn-SOD基因。【本研究切入点】目前,对于小麦籽粒中脂肪氧化酶(lipoxygenase,LOX)和过氧化物酶(peroxidase,POD)等抗氧化酶类与小麦品质基因挖掘、克隆、功能标记开发等研究已较为深入,其中功能标记也已应用于育种实践[20,21],但对小麦籽粒SOD活性测定的研究以及对控制小麦籽粒SOD活性位点挖掘的研究仍鲜见报道。【拟解决的关键问题】本研究通过对212份小麦品种(系)的籽粒SOD活性进行测定,结合90K SNP芯片,进行该性状的全基因组关联分析,挖掘与SOD活性显著关联的位点,有利于进一步揭示小麦籽粒SOD活性的遗传机制,为小麦品质性状的分子标记辅助育种提供材料与方法。1 材料与方法
1.1 材料
供试小麦品种(系)共计212份,包括中国黄淮冬麦区93份,北部冬麦区35份,长江中下游冬麦区26份,西南冬麦区14份,44份来自法国、罗马尼亚和意大利等欧洲国家的品种(系)(电子附表1),上述材料均由新疆农业大学农学院小麦遗传育种课题组提供。供试材料分别于2016—2017年、2017—2018年和2018—2019年种植于新疆农业科学院玛纳斯试验站,随机区组设计,3次重复,单行种植,行长2 m,行距0.25 m。田间管理同当地一致,采用滴灌浇水,除冬灌水外,分别于冬小麦拔节前、孕穗期、开花期、灌浆前期和灌浆中期共滴灌5次。每年7月中旬采用人工单行混收,晾晒7 d后,为避免机械混杂,采用人工脱粒。Table 1
表1
表1不同环境中212份冬小麦籽粒SOD活性统计分析
Table 1
| 环境 Environment | 均值±标准差 Mean±SD | 变幅 Range | 变异系数 CV | 相关系数Correlation coefficient | 遗传力 h2 | ||
|---|---|---|---|---|---|---|---|
| 2016—2017 | 2017—2018 | 2018—2019 | |||||
| 2016—2017 | 1784.14±73.31 | 1348.71—2025.14 | 4.34 | 0.79 | |||
| 2017—2018 | 1798.26±97.28 | 1316.83—2016.18 | 5.23 | 0.60*** | |||
| 2018—2019 | 1774.25±78.14 | 1348.26—2021.86 | 4.85 | 0.64*** | 0.62*** | ||
| 均值Mean | 1781.28±73.40 | 1407.32—1988.93 | 4.72 | 0.86*** | 0.90*** | 0.87*** | |
新窗口打开|下载CSV
1.2 普通小麦籽粒SOD的提取
以磷酸缓冲溶液为提取液进行SOD粗酶液提取,首先称取10 g去杂去劣的小麦籽粒,利用筛孔为0.8 mm的旋风磨(瑞典LaboratoryMill 120)研磨成全麦粉,然后称取0.1 g全麦粉置于2 mL离心管中,加入1 mL 4℃预冷的0.05 mol·L-1磷酸缓冲溶液(pH 7.8),在混旋振荡器上振荡1 min,使缓冲液与全麦粉充分接触;最后将装有样品的离心管放于摇床上,冰浴摇晃2 h后,4℃ 10 000 r/min离心15 min,吸取上清液,即为SOD粗酶液。1.3 氮蓝四唑(NBT)光化还原法测定SOD活性
采用氮蓝四唑(NBT)光化还原法测定SOD活性[22]。SOD活性检测的底物为0.05 mol·L-1磷酸缓冲溶液150 μL、蒸馏水20 μL、130 mmol·L-1 Met溶液30 μL、750 μmol·L-1 NBT溶液30 μL、100 μmol·L-1 EDTA-Na2溶液30 μL和20 μmol·L-1核黄素溶液30 μL的混合液,混匀后立即使用,随后滴入10 μL的SOD粗酶液(设2支对照管,且对照管以缓冲液代替酶液)。将其中1支对照管置于暗处,其余均放于4 000 lx日光灯下,反应20 min。同一重复内材料的SOD活性测定使用同一批次配置的底物,以避免因底物浓度误差导致的试验误差。应用酶标仪(美国Synergy H1),在560 nm、25℃条件下测定OD值。已知SOD活性单位以抑制NBT光还原的50%为一个酶活性单位表示,SOD活性计算公式如下:SOD总活性(U·g-1)=(Ack-AE)×V/(Ack×0.5× W×Vt)
式中,SOD总活性以每克样品鲜质量的酶单位表示(U·g-1);Ack:照光对照管的吸光度;AE:样品管的吸光度;V:样品液总体积(mL);Vt:测定时样品用量(mL);W:样品鲜重(g)。
每个小麦品种(系)的SOD活性重复测定2次,如果2次测定结果的相差超过10%,则需进行重复测定。
1.4 表型数据的统计分析
采用Excel 2016和SPSS 21.0软件对SOD活性进行基本数据统计和分析,使用QTL IciMapping V4.1软件进行描述性统计分析和方差分析[23],计算广义遗传力公式为:h2=σg2/(σg2+σe2),其中,σg2为遗传方差,σe2为环境方差。1.5 群体结构与连锁不平衡分析
试验前期使用90K SNP芯片对212份冬小麦材料进行了基因分型,人工对分型结果进行质量控制,剔除数据缺失率>50%、杂合率>50%和最小等位基因频率(minor allele frequency,MAF)<0.05的SNP标记,共获得16 705个高质量SNP标记。应用Power Marker V3.25软件计算多态性信息量(polymorphic information content,PIC)。从筛选过的标记中,随机选取2 000个最小等位基因频率大于10%且在染色体上均匀分布的SNP标记,利用Structure 2.3.4软件进行群体结构分析。参数设置:Length of Burn-Period=10 000,MCMC Reps after Burn-in=100 000,选择Admixture Ancestry模型和Dependent Allele Frequencies模式,令K=2—12,每个K值重复运行5次,利用Tassel 5.0软件绘制群体结构分析图。以位点间的相关系数平方(r2)作为衡量多态性位点两两之间的连锁不平衡(linkage disequilibrium,LD)参数。利用Tassel 5.0软件计算r2,以第95百分位的r2值作为阈值估测LD衰减距离。关联分析中,超过LD衰减距离的2个位点则认为是2个不同的位点。1.6 全基因组关联分析
基于筛选到的高质量标记,利用Tassel 5.0软件中的Q+K混合线性模型进行全基因组关联分析,以P=1.0×10-3为阈值,判定SNP标记与目标性状关联的显著性[24]。利用R语言绘制关联分析结果的曼哈顿图和Q-Q图。2 结果
2.1 小麦籽粒SOD活性的表型变异
212份供试小麦品种(系)在不同环境下SOD活性平均值分别为1 784.14、1 798.26、1 774.25和1 781.28 U·g-1,年际间SOD活性最大差异为708.31 U·g-1,变异系数为4.34%—5.23%。不同环境之间的SOD活性呈极显著正相关,相关系数为0.60—0.90(P<0.001),遗传力为0.79(表1)。在不同环境下SOD活性均呈正态分布(图1),具有广泛的SOD活性。就本研究所采用的材料而言,不同麦区品种(系)的SOD活性表现为,北部冬麦区>国外品种>西南冬麦区>黄淮冬麦区>长江中下游冬麦区。北部冬麦区小麦品种(系)在不同环境下均表现出较高的SOD活性(表2)。图1
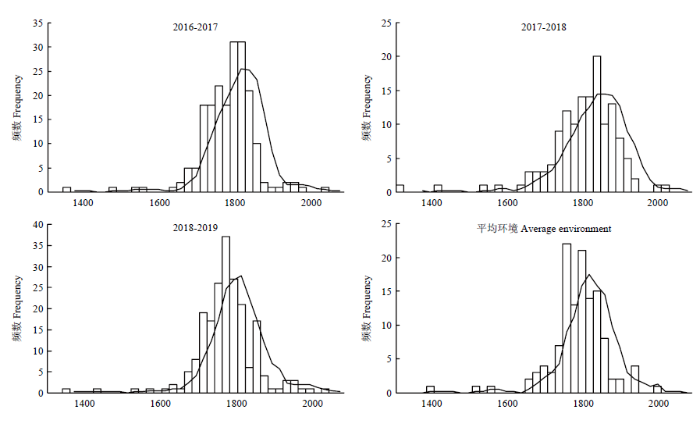 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1212份冬小麦在不同环境下的SOD活性分布频次
Fig. 1SOD activity distribution frequency of 212 winter wheat in different environments
Table 2
表2
表2不同麦区冬小麦籽粒SOD活性
Table 2
| 环境 Environment | 来源 Origin | ||||
|---|---|---|---|---|---|
| 北部冬麦区 Northern winter wheat region | 西南冬麦区 Southwest winter wheat region | 黄淮冬麦区 Huanghuai winter wheat region | 长江中下游冬麦区 Middle/lower reaches of the Yangtze River winter wheat region | 国外品种 Foreign varieties | |
| 2016—2017 | 1795.17 | 1782.41 | 1769.27 | 1733.88 | 1789.64 |
| 2017—2018 | 1804.36 | 1772.71 | 1790.38 | 1800.16 | 1800.91 |
| 2018—2019 | 1806.25 | 1799.82 | 1761.35 | 1747.94 | 1779.12 |
| 总计Total | 1801.74 | 1787.21 | 1771.19 | 1759.67 | 1788.26 |
新窗口打开|下载CSV
2.2 SNP多态性及分布
从小麦90K SNP芯片中筛选出具有多态性的SNP标记16 705个,分布在A、B和D基因组染色体上的SNP位点数目分别为7 153(42.82%)、7 346(43.97%)和2 206(13.21%),其中,B基因组的多态性最高,D基因组的多态性低于A和B 2个基因组(表3)。Table 3
表3
表3标记的分布、物理图谱长度及标记的多态性
Table 3
| 染色体 Chromosome | 标记数目 No. of markers | 长度 Length (Mb) | 标记密度 Density of marker | 遗传多样性 Genetic diversity | 多态信息含量 PIC |
|---|---|---|---|---|---|
| 1A | 1262 | 592.38 | 0.47 | 0.32 | 0.26 |
| 1B | 1293 | 688.60 | 0.53 | 0.34 | 0.27 |
| 1D | 573 | 495.14 | 0.86 | 0.31 | 0.26 |
| 2A | 1151 | 780.46 | 0.68 | 0.29 | 0.24 |
| 2B | 1293 | 799.62 | 0.62 | 0.32 | 0.26 |
| 2D | 481 | 650.94 | 1.35 | 0.31 | 0.25 |
| 3A | 894 | 749.46 | 0.84 | 0.31 | 0.26 |
| 3B | 1090 | 829.32 | 0.76 | 0.33 | 0.27 |
| 3D | 234 | 613.92 | 2.62 | 0.29 | 0.24 |
| 4A | 705 | 741.73 | 1.05 | 0.30 | 0.25 |
| 4B | 571 | 672.56 | 1.18 | 0.32 | 0.26 |
| 4D | 99 | 508.58 | 5.14 | 0.31 | 0.26 |
| 5A | 960 | 709.43 | 0.74 | 0.36 | 0.29 |
| 5B | 1157 | 712.82 | 0.62 | 0.35 | 0.28 |
| 5D | 240 | 563.41 | 2.35 | 0.31 | 0.25 |
| 6A | 1007 | 617.40 | 0.61 | 0.34 | 0.27 |
| 6B | 1081 | 720.82 | 0.67 | 0.31 | 0.26 |
| 6D | 319 | 472.61 | 1.48 | 0.31 | 0.25 |
| 7A | 1174 | 736.44 | 0.63 | 0.30 | 0.25 |
| 7B | 861 | 750.49 | 0.87 | 0.31 | 0.25 |
| 7D | 260 | 637.17 | 2.45 | 0.29 | 0.24 |
| A基因组 A genome | 7153 | 4927.29 | 0.69 | 0.32 | 0.26 |
| B基因组 B genome | 7346 | 5174.23 | 0.70 | 0.33 | 0.27 |
| D基因组 D genome | 2206 | 3941.77 | 1.79 | 0.31 | 0.24 |
| 总计Total | 16705 | 14043.30 | 0.84 | 0.32 | 0.26 |
新窗口打开|下载CSV
21条染色体中1B、2B染色体上的标记数目最多,均有1 293个,4D染色体上的标记数目最少,只有99个。3个染色体组的PIC表现为B(0.27)>A(0.26)>D(0.24)。全基因组的PIC平均值为0.26。
2.3 群体结构与连锁不平衡分析
利用Structure 2.3.4软件对每个可能的K值模拟运算,用K值与ΔK值做图(图2-A),在K=3处,ΔK值最大,曲线变化程度最大。由图2-B可以看出参试材料分为3个亚群,亚群1包含59份(27.83%)品种(系),主要来自国外(20份)和北京市(19份),亚群2包含93份(43.87%)品种(系),主要来自陕西省(14份)、四川省(10份)和江苏省(9份),亚群3包含60份(28.30%)品种(系),主要来自河南省(24份)。利用Tassel 5.0软件计算得到212份小麦品种(系)在基因组A、B、D和全基因组的LD衰减距离分别为5、8、9和7 Mb,依据全基因组的LD衰减距离,将在物理图谱上前后7 Mb区间内的位点认定为一个候选位点。图2
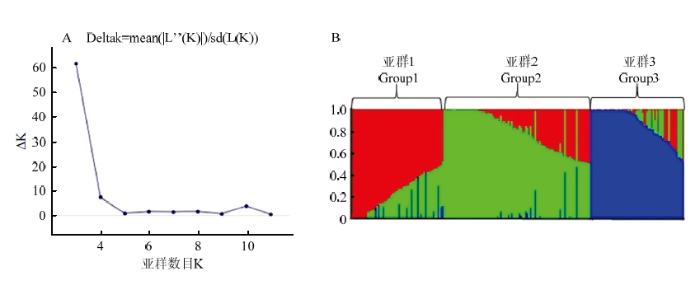 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2212份小麦品种(系)群体结构分析
A:群体的?K值;B:群体结构示意图
Fig.2Population structure analysis of 212 wheat varieties (lines)
A: Estimation of ?K value in population; B: Group structure diagram
2.4 标记-性状关联分析
利用Tassel 5.0软件将212份小麦品种(系)的SOD活性结合90K芯片的16 705个高质量SNP标记进行全基因组关联分析,采用Q+K混合线性模型,共检测到29个与SOD活性显著关联的位点(图3和表4),贡献率为5.37%—32.43%;2016—2017年检测到与SOD活性相关的16个显著关联位点,分别位于1B、2B、3B、3D、4B、4D、5A、5B、5D、6B、6D和7B等12条染色体上,贡献率为5.37%—16.62%;2017—2018年检测到与SOD活性相关的5个显著关联位点,分别位于1B、2A、6A和6B等4条染色体上,贡献率为6.27%—17.00%;2018—2019年检测到与SOD活性相关的11个显著关联位点,分别位于1B、2B、4B、5A、5B、6B和6D等7条染色体上,贡献率为6.21%—32.43%;平均环境下检测到与SOD活性相关的20个位点,分别位于1A、1B、2A、2B、2D、3D、4B、5A、5B、5D、6A、6B和6D等13条染色体上,贡献率为5.37%—32.43%。其中14个显著关联位点在2个及以上环境下均检测到,分布于1B、2A、2B、3D、4B、5A、5B、5D、6A、6B和6D等11条染色体上,贡献率为5.37%—32.43%。在3个环境下同时检测到9个显著关联位点,分布于1B、2B、4B、5A、5B、6B和6D染色体上,贡献率为6.21%—16.62%。图3
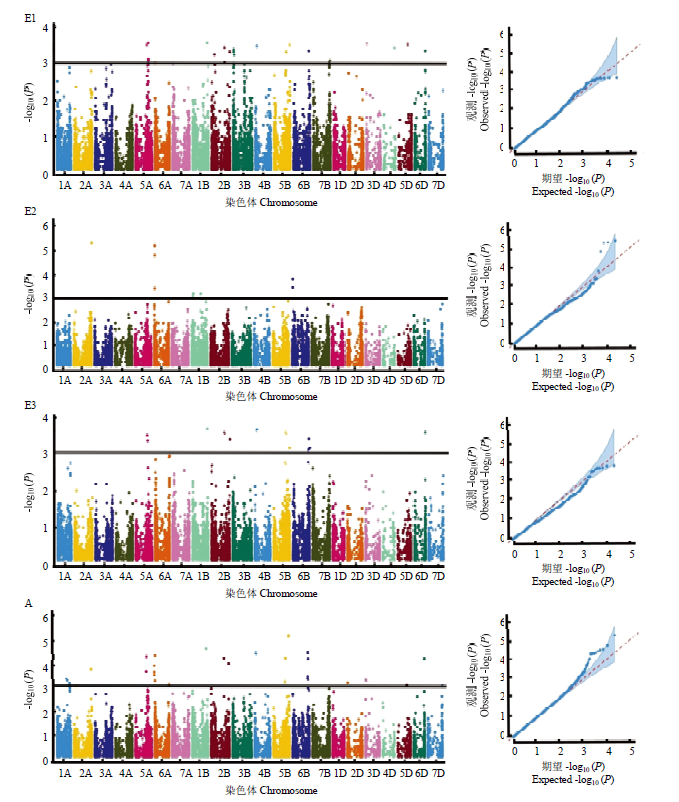 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3不同环境下SOD活性的曼哈顿图和Q-Q图
E1:2016—2017年环境点;E2:2017—2018年环境点;E3:2018—2019年环境点;A:平均环境
Fig. 3Manhattan plot and Q-Q plot of SOD activity in different environments
E1: 2016-2017 environmental point; E2: 2017-2018 environmental point; E3: 2018-2019 environmental point; A: Average environment
Table 4
表4
表4SNP-GWAS检测到的SOD活性显著相关的位点
Table 4
| 标记 Marker | 染色体 Chromosome | 物理位置 Position (bp) | 环境 Environment | P值 P value | 贡献率 R2 (%) |
|---|---|---|---|---|---|
| Ex_c12584_2014 | 1A | 435061794 | A | 5.77E-04 | 5.94 |
| BS00109991_51 | 1A | 536439240—536614333 | A | 7.90E-04—8.82E-04 | 7.02—7.07 |
| Ra_c5683_2584 | 1A | 551460924 | A | 9.44E-04 | 5.38 |
| Kukri_c8533_1398 | 1B | 49886407 | E2 | 9.60E-04 | 8.43 |
| Ra_c11303_359 | 1B | 379383435 | E2 | 9.78E-04 | 8.46 |
| BS00022411_51 | 1B | 629159210 | E1/E3/A | 2.88E-05—2.97E-04 | 8.93—11.10 |
| Tdurum_contig92425_1574 | 2A | 779977420 | E2/A | 6.80E-06—2.13E-04 | 8.52—16.45 |
| BS00038217_51 | 2B | 99961034 | E1 | 6.35E-04 | 8.66 |
| RFL_Contig1987_3440 | 2B | 555840884 | E1/E3/A | 7.00E-05—3.92E-04 | 8.83—11.28 |
| BS00026037_51 | 2B | 793151228 | E1/E3/A | 1.24E-04—5.25E-04 | 14.23—16.62 |
| BS00049876_51 | 2D | 12891888 | A | 7.40E-04 | 7.36 |
| Kukri_c40882_76 | 3B | 19250811—19390877 | E1 | 4.50E-04—6.44E-04 | 6.04—6.38 |
| CAP8_c9373_277 | 3D | 21005688 | E1/A | 3.13E-04—5.86E-04 | 5.81—6.81 |
| Tdurum_contig12116_297 | 4B | 60543887 | E1/E3/A | 4.52E-05—3.71E-04 | 8.40—10.97 |
| Excalibur_c29496_799 | 4D | 475027917 | E1 | 3.96E-04 | 7.68 |
| BS00022191_51 | 5A | 476402782 | E1/E3/A | 6.11E-05—3.21E-04 | 8.57—10.77 |
| BS00024602_51 | 5A | 499660243 | E3/A | 2.64E-04—4.51E-04 | 26.29—32.43 |
| Kukri_c14889_1086 | 5A | 540052370—540611794 | E1 | 3.04E-04—9.20E-04 | 6.22—9.17 |
| tplb0061l23_1365 | 5B | 506768500 | A | 6.90E-04 | 7.18 |
| BobWhite_rep_c62475_70 | 5B | 531199416 | E1/E3/A | 6.96E-05—5.30E-04 | 7.93—11.03 |
| RAC875_c33791_320 | 5B | 679804074 | E1/E3/A | 8.46E-06—7.05E-04 | 6.91—10.13 |
| RAC875_c49940_385 | 5D | 399290149 | E1/A | 3.21E-04—9.48E-04 | 5.37—6.69 |
| RAC875_c13610_1239 | 6A | 1497847—2402603 | E2/A | 8.61E-06—6.32E-04 | 6.29—17.00 |
| BS00037162_51 | 6A | 617689729 | A | 8.99E-04 | 7.93 |
| BS00082893_51 | 6B | 166226—169518 | E2 | 2.24E-04—5.29E-04 | 13.18—13.22 |
| Kukri_c49331_77 | 6B | 664266146—667884621 | E1/E3/A | 4.25E-05—7.81E-04 | 6.21—11.61 |
| Kukri_c338_109 | 6B | 708660893 | E3 | 7.10E-04 | 9.40% |
| Excalibur_c57840_227 | 6D | 456465650 | E1/E3/A | 7.43E-05—4.78E-04 | 8.21—10.83 |
| Kukri_c16814_103 | 7B | 700830992 | E1 | 9.32E-04 | 5.69 |
新窗口打开|下载CSV
2.5 小麦籽粒SOD活性候选基因预测
将与小麦籽粒SOD活性显著关联且稳定遗传的SNP标记序列在普通小麦中国春基因组数据库中进行检索,并在NCBI数据库中进行BLASTx序列比对[25],筛选到7个SOD基因和2个与SOD活性相关的候选基因(表5)。在这些候选基因的注释中,TraesCS5A01G263500编码锌指应激蛋白(Zinc finger stress protein);TraesCS6B01G707800LC注释为谷胱甘肽转移酶基因(glutathione transferase gene)。Table 5
表5
表5筛选获得候选基因信息
Table 5
| 位点 Marker | 染色体 Chr. | 物理位置 Position (bp) | 基因 Gene | 基因注释或编码蛋白 Gene annotation or coding protein |
|---|---|---|---|---|
| BS00026037_51 | Chr.2B | 758592689 | TraesCS2B01G567600 | 超氧化物歧化酶Superoxide dismutase |
| CAP8_c9373_277 | Chr.3D | 30983003 | TraesCS3D01G069900 | 铜、锌超氧化物歧化酶Superoxide dismutase [Cu-Zn] |
| 31432328 | TraesCS3D01G070200 | 铜、锌超氧化物歧化酶 Superoxide dismutase [Cu-Zn] | ||
| BS00022191_51 | Chr.5A | 476402782 | TraesCS5A01G263500 | 锌指应激蛋白Zinc finger stress protein |
| BobWhite_rep_c62475_70 | Chr.5B | 531620481 | TraesCS5B01G525700 | 铜、锌超氧化物歧化酶Superoxide dismutase [Cu-Zn] |
| 551408544 | TraesCS5B01G373700 | 铜、锌超氧化物歧化酶Superoxide dismutase [Cu-Zn] | ||
| RAC875_c13610_1239 | Chr.6A | 8971283 | TraesCS6A01G021400 | 超氧化物歧化酶Superoxide dismutase |
| Kukri_c49331_77 | Chr.6B | 664393248 | TraesCS6B01G707800 | 谷胱甘肽转移酶Glutathione transferase |
| Excalibur_c57840_227 | Chr.6D | 430047351 | TraesCS6D01G431500 | 铜、锌超氧化物歧化酶Superoxide dismutase [Cu-Zn] |
新窗口打开|下载CSV
3 讨论
3.1 SOD活性的分析
小麦面粉色泽和面制品营养品质是小麦品质遗传改良的重要内容。多酚氧化酶(polyphenol oxidase,PPO)、过氧化物酶(peroxidase,POD)、木聚糖酶(xylanase)和α-淀粉酶(α-amylase)等能够影响面粉色泽和面制品营养品质[21,26-28]。对PPO、POD、EC3.2.1.8和EC3.2.1.1等研究已较为深入,但是对超氧化物歧化酶SOD研究较少,籽粒中SOD活性变化规律及遗传机理未见报道。SOD活性属于数量性状,既能够稳定遗传给后代,同时又受环境影响[29]。蒲光兰等[30]研究表明,群体性状变异程度或变异幅度越大,对种质变异和创新贡献率就越高,遗传力的大小能反映了某一性状的差异。本研究表明,SOD活性在多个环境下的变幅为1 316.83—2 025.14 U·g-1,平均值为1 784.14、1 798.26、1 774.25和1 781.28 U·g-1。不同环境下的变异系数介于4.34%—5.23%,表明不同供试材料在不同环境下均表现出丰富的表型变异,但绝大部分供试材料的SOD活性较低,具有较强的遗传改良潜力。其广义遗传力为0.79,表明不同供试材料的SOD活性虽受环境因素的影响,但遗传因素是导致其表型变异的主要原因。另外SOD活性较高的京冬22和陕715可作为优良亲本进行育种工作,为籽粒SOD活性改良奠定基础。经过K-S检验,SOD活性在多个环境下均呈正态分布,且供试小麦品种(系)遗传背景来源广泛,具有较好的遗传多样性,适合进行全基因组关联分析。对不同麦区SOD活性比较发现,北部冬麦区>国外品种>西南冬麦区>黄淮冬麦区>长江中下游冬麦区,这可能是由于逆境胁迫会使SOD活性升高[31,32],北部冬麦区和国外品种所在区域纬度较西南冬麦区、黄淮冬麦区及长江中下游冬麦区纬度更高,高纬度地区比低纬度地区气候更加恶劣,长期寒冷和干旱胁迫导致北部冬麦区和国外品种(系)SOD活性更高。而西南冬麦区SOD活性高于黄淮冬麦区和长江中下游冬麦区的原因可能是西南冬麦区在20世纪已经开始着重于小麦品质改良研究,注重选育高品质的小麦作为主推品种[33]。因此,西南冬麦区品种(系)的平均SOD活性高于以上2个麦区。
3.2 SOD活性GWAS分析
全基因组关联分析具有研究时间短、检测精度高、高通量和低成本等特点[34]。STICH等[35]研究认为GWAS中使用Q+K混合模型法是目前降低假阳性关联的最好方法。本研究采用Q+K混合模型法对212份供试材料的SOD活性进行全基因组关联分析,研究结果表明,共检测到29个显著关联的SNP位点,分布在小麦的16条染色体上。鞠晓影等[36]利用DH群体在1B上检测到SOD活性QTL;卫宪云等[37]和赵新华等[38]利用不同的RIL群体分别在2D和7B上检测到SOD活性QTL;本研究同样在1B、2D和7B上发现了SOD活性显著关联位点。同时还在2A、2B、4B、4D和6D上发现7个显著关联位点,这与JIANG等[18]和KUMAR等[19]研究结果一致。同时,对2B上的标记BS00026037_51进行候选基因筛选,通过基因功能注释确定基因TraesCS2B01G567600为SOD基因(758.59 Mb),这与JIANG等[18]报道的基因TaSOD2.2(758.59 Mb)相同。与此同时,本研究还检测到3D、5A、5B、5D、6A和6B上多个稳定遗传的新位点。其中5A(BS00024602_51)贡献率最大,为26.29%—32.43%。在3个环境下同时检测5A(BS00022191_51)、5B(BobWhite_rep_c62475_70和RAC875_c33791_320)和6B(Kukri_c49331_77)位点,贡献率为6.21%—16.62%。对以上位点进行候选基因挖掘,筛选到2个SOD基因和2个与SOD活性相关的基因。在下一步研究中,依据5A上的位点贡献率最高,5B上存在多个稳点遗传的位点,可考虑优先选择5A和5B染色体作为热点区域进一步精细定位并开发相应的育种可用标记,剖析小麦籽粒SOD活性的遗传机理,为小麦品质改良工作提供基础。
3.3 候选基因预测
本研究利用中国春基因组数据库获取与SOD活性显著关联的SNP标记序列,并在NCBI数据库中进行BLASTx序列比对,进行候选基因筛选,根据基因功能注释信息,筛选到7个SOD基因和2个与SOD活性相关的候选基因。位于3D和5B染色体上各2个Cu/ Zn-SOD活性基因以及6D染色体上的1个Cu/Zn-SOD活性基因,2B和6A染色体上各1个SOD活性基因。位于5A染色体上的基因TraesCS5A01G263500编码锌指应激蛋白,能抑制细胞活性氧积累和丙二醛的形成,且能恢复线粒体膜电位平衡和抗氧化酶活性,在此过程中达到调节SOD活性的作用[39,40],位于6B染色体上的基因TraesCS6B01G707800注释为谷胱甘肽转移酶基因,该基因参与植物细胞抗氧化剂的再生过程,在大分子的生物合成、细胞信号传导和蛋白质间相互作用等方面具有重要的调节作用,是抗氧化系统的重要组成部分[41,42]。本研究筛选的SOD基因和与SOD活性相关基因编码不同蛋白,在植物代谢途径中直接或间接调控SOD活性。4 结论
获得29个与SOD活性显著关联的位点,其中14个位点被重复检测到。获得7个小麦籽粒SOD活性候选基因,2个与SOD活性相关候选基因,分别为编码锌指应激蛋白基因和谷胱甘肽转移酶基因,它们通过参与抗氧化剂再生、抑制活性氧积累并组成抗氧化系统来调节SOD活性。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[D].
[本文引用: 1]
[D].
[本文引用: 1]
DOI:10.2135/cropsci2013.10.0708URL [本文引用: 2]
DOI:10.1080/07352689409701914URL [本文引用: 3]
DOI:10.1016/S2095-3119(13)60573-0URL [本文引用: 2]
[本文引用: 1]
DOI:10.1094/CCHEM.1997.74.5.651URL [本文引用: 2]
[本文引用: 2]
[本文引用: 2]
DOI:10.1016/j.foodchem.2011.01.048URL [本文引用: 1]
[本文引用: 1]
DOI:10.1111/jipb.2006.48.issue-6URL [本文引用: 1]
DOI:10.1007/s12298-013-0201-7URL [本文引用: 1]
DOI:10.1007/s11738-003-0007-2URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1104/pp.120.2.513URL [本文引用: 1]
DOI:10.1139/g05-102URL [本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
DOI:10.7717/peerj.8062URL [本文引用: 3]
DOI:10.1016/j.ijbiomac.2020.06.026URL [本文引用: 2]
DOI:10.2135/cropsci2011.07.0365URL [本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/j.cj.2015.01.001URL [本文引用: 1]
DOI:10.1038/ng1702URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1186/1471-2164-10-1URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
