 ,上海交通大学农业与生物学院,上海 200240
,上海交通大学农业与生物学院,上海 200240Genome-Wide Identification of Cucumber ERF Gene Family and Expression Analysis in Female Bud Differentiation
PAN Jian, WEN HaiFan, HE HuanLe, LIAN HongLi, WANG Gang, PAN JunSong, CAI Run ,School of Agriculture and Biology, Shanghai Jiao Tong University, Shanghai 200240
,School of Agriculture and Biology, Shanghai Jiao Tong University, Shanghai 200240通讯作者:
责任编辑: 赵伶俐
收稿日期:2019-05-28接受日期:2019-10-29网络出版日期:2020-01-01
| 基金资助: |
Received:2019-05-28Accepted:2019-10-29Online:2020-01-01
作者简介 About authors
潘健,E-mail:nillice@sina.com。

摘要
关键词:
Abstract
Keywords:
PDF (5595KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
潘健, 温海帆, 何欢乐, 连红莉, 王刚, 潘俊松, 蔡润. 黄瓜ERF基因家族鉴定及其在雌花芽分化中的表达分析[J]. 中国农业科学, 2020, 53(1): 133-147 doi:10.3864/j.issn.0578-1752.2020.01.013
PAN Jian, WEN HaiFan, HE HuanLe, LIAN HongLi, WANG Gang, PAN JunSong, CAI Run.
0 引言
【研究意义】黄瓜(Cucumis sativus L.)是重要的园艺作物之一,其栽培面积仅次于番茄和洋葱[1],为世界第三大蔬菜。雌花子房是黄瓜经济价值的体现,其分化与发育决定了果实的产量和质量。黄瓜果实的形成经历了花芽性别分化和子房发育两个阶段,植物激素乙烯在这一过程中起至关重要的作用[2]。乙烯响应因子(ethylene response factor,ERF)家族是一类植物特异性转录因子家族,多个ERF家族成员在不同物种中参与到乙烯信号的响应与调控[3,4,5]。乙烯是影响黄瓜雌花分化与子房发育的重要因素,系统地对黄瓜基因组中ERF基因家族进行鉴定与分析,可以为深入探究黄瓜雌花分化与发育的分子机制提供数据基础。【前人研究进展】植物ERF基因家族成员数量众多,且在各种生物和非生物胁迫下发挥重要功能[6]。在一些茄科植物中,LeERF2/TERF2通过与GCC-box 结合,诱导乙烯的生物合成[3]。在香蕉(Musa acuminate)中,MaERF9通过结合MaACS1的启动子激活其转录,而 MaERF11 通过结合 MaACS1和MaACO1的启动子,抑制其转录[4]。类似的,苹果(Malus domestica)中MdERF3和MdERF2通过结合MdACS1启动子,分别激活和抑制其转录[5]。这些研究结果说明ERF在乙烯合成过程中起重要作用。ERF1是较早被克隆并进行功能研究的ERF之一[7]。后续研究中,ERF1被报道通过与茉莉酸、生长素等互作,参与植物防卫反应[8,9],也有报道显示ERF1通过调控下胚轴细胞壁增厚和伸长参与植物的光形态建成[10]。在黄瓜性别分化研究中,已报道CsERF110和CsERF31具有分别结合A基因(CsACS11)[11]和M基因(CsACS2)的启动子,激活其转录的功能[12]。【本研究切入点】在已有研究中,黄瓜ERF基因家族已基于9930_V1版本基因组进行过鉴定分析[13],但由于早期版本基因组拼接和注释的质量有限,且无法将基因定位于染色体,目前研究人员普遍使用的是9930_V2版本基因组[14]。所以,基于更通用版本基因组对ERF基因家族进行鉴定,将会提供更全面和准确的鉴定结果。【拟解决的关键问题】通过基于9930_V2版本基因组对ERF基因家族进行鉴定与表达分析,明确ERF在雌花分化与发育阶段的特异表达情况,为解释这一生物学过程的分子机制奠定理论基础。1 材料与方法
试验于2017年春季在上海交通大学农业与生物学院进行。1.1 黄瓜材料
本研究试验材料为华北类型黄瓜近等基因系:全雌株系419-19(FFMMAA基因型)和主茎强雄株系419-2(ffMMAA)。材料种植培养环境为28℃ 18 h昼,18℃ 6 h夜。顶芽选取四叶一心时期,不同时期雌花芽选取四叶一心时期顶芽内不同大小的雌花芽。所取材料采用3个独立生物学重复,液氮速冻后,置于-80℃超低温冰箱备用。1.2 黄瓜ERF基因家族成员鉴定
根据文献和TAIR数据库获得拟南芥(Arabidopsis thaliana)ERF基因家族的ID和氨基酸序列信息。黄瓜基因组数据信息均获取自CuGenDB(http:// cucurbitgenomics.org)。采用双向BLAST方法获取黄瓜基因组中ERF基因ID。双向BLAST方法简述如下:采用Geneious软件将拟南芥ERF基因家族成员比对至黄瓜9930_V2版本基因组,获取基因ID,再将所获取基因ID比对至Swiss-Port数据库,确认获取ID为ERF家族成员。通过Geneious软件进行序列多重比对,鉴定保守结构域。同时,通过NCBI网站的CDD工具预测ERF基因的AP2/ERF结构域,确定每个基因均包含该结构域。采用TBtools软件[15]绘制黄瓜ERF基因家族成员在染色体上的物理位置。1.3 黄瓜ERF基因家族进化、基因结构和GO富集分析
利用MEGA7软件对黄瓜ERF基因家族成员进行系统进化树分析,分析序列为氨基酸,采用Neighbor Joining(NJ)法进行建树,Bootstrap值设置为1 000。采用EMBOSS软件进行ERF基因家族成员的信号肽和跨膜域预测。通过MEME(http://meme-suite.org)网站工具进行motif预测分析。将Motif预测、CDD结构域鉴定以及进化树分析结果,通过TBtools软件整合并制图。VENN交集分析采用VENNY2.1[16],GO富集分析在葫芦科基因组网站(http:// cucurbitgenomics. org)进行。1.4 实时荧光定量PCR(qRT-PCR)表达检测
将植物材料研磨,采用康为世纪公司试剂盒RNA抽提、反转录获取cDNA。qRT-PCR试验采用罗氏公司FastStart Essential DNA Green Maste试剂,程序参见试剂盒说明书。以CsActin(Csa6M484600.1)作为内参,所用引物均查询自qPrimerDB数据库(https://biodb.swu.edu.cn/qprimerdb/)。采用3个生物学重复与3个技术重复。试验数据用Excel软件进行处理,采用2-ΔΔCT方法进行相对定量分析,显著性分析采用t-test公式计算。在表达趋势分析中,将不同时期花芽中表达的CsERF基因家族成员的qRT-PCR相对定量结果导入Omicshare趋势分析工具(www.omicshare.com/tools),采用t-test 检验两个时期之间表达差异大于2倍(P value<0.05)的基因,定义为表达存在显著差异的基因。1.5 酵母单杂交分析
在生工生物公司合成包含3个串联重复GCC-box的启动子DNA序列“TCCAAGGAGCCGCCGGG”(来源于CsERF39的启动子区,下划线为酶切位点),将合成序列通过诺唯赞ClonExpress II重组试剂盒与pLacZ载体融合,构建为pLacZ-3xGCC-box载体。采用TaKaRa的PrimerStar GXL DNA聚合酶克隆CsERF9和CsERF31的开放和阅读框,将其与pB42AD载体融合,构建为pB42AD-CsERF9和pB42AD- CsERF31载体。酵母单杂交分析中,采用EGY48a菌株,以空载体为负对照,在SD/-Trp/-Ura缺陷型培养基进行筛选培养,并在加有X-gal的SD/-Trp/-Ura缺陷型定性培养基上进行互作鉴定[17]。2 结果
2.1 黄瓜ERF基因家族成员的鉴定与相关序列信息
通过双向BLAST分析,以拟南芥AP2/EREBP基因家族为参考,对黄瓜基因组中ERF基因家族进行鉴定,共鉴定出138个具有AP2/ERF结构域的基因(图1),根据其在9930_V2基因组中的ID,分别命名为CsERF1—CsERF138。长度介于126—745个氨基酸。通过BLAST比对,将9930_V2基因组ERF家族成员比对到拟南芥TAIR10基因组中,获取对应基因名称(表1)。相比根据9930_V1版本所鉴定的103个ERF基因[13],本研究新鉴定出25个ERF家族成员,并将全部138个ERF定位于染色体。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1黄瓜ERF家族成员AP2/ERF保守结构域序列
Fig. 1The AP2/ERF conserved domain in ERF members of cucumber
Table 1
表1
表1CsERF基因家族相关信息
Table 1
| 名称 Name | 黄瓜基因组基因编号 ID in cucumber (9930_V2) | 注释到拟南芥成员 Annotated to Arabidopsis | 成员名称 Name in Arabidopsis | 所属亚族 Class | 氨基酸个数 Number of amino acids |
|---|---|---|---|---|---|
| CsERF1* | Csa1G000550 | AT5G61890 | ERF114 | 2 | 216 |
| CsERF2 | Csa1G042290 | AT1G68550 | ERF118 | 2 | 336 |
| CsERF3 | Csa1G075030 | AT1G28360 | ERF12 | 4 | 164 |
| CsERF4 | Csa1G075060 | AT5G44210 | ERF9 | 4 | 211 |
| CsERF5 | Csa1G269870 | AT1G50680 | - | 5 | 339 |
| CsERF6 | Csa1G275920 | AT1G16060 | ADAP | 6 | 363 |
| CsERF7 | Csa1G340430 | AT1G15360 | WIN1 | 7 | 223 |
| CsERF8 | Csa1G364460 | AT5G11590 | DREB3 | 9 | 250 |
| CsERF9 | Csa2G177210 | AT3G23240 | ERF1B | 1 | 228 |
| CsERF10 | Csa1G597730 | AT1G19210 | ERF017 | 9 | 203 |
| CsERF11* | Csa1G605670 | AT2G05990 | MOD1 | 5 | 745 |
| CsERF12* | Csa2G001000 | AT5G11590 | DREB3 | 9 | 252 |
| CsERF13 | Csa2G006270 | AT1G15360 | WIN1 | 7 | 246 |
| CsERF14* | Csa2G092800 | AT5G17430 | BBM | 6 | 650 |
| CsERF15 | Csa2G138780 | AT4G13620 | ERF062 | 10 | 400 |
| CsERF16 | Csa1G423190 | AT5G57390 | AIL5 | 6 | 466 |
| CsERF17 | Csa2G177220 | AT3G23230 | ERF098 | 1 | 126 |
| CsERF18 | Csa2G279250 | AT4G36920 | AP2 | 6 | 483 |
| CsERF19* | Csa2G279260 | AT2G23340 | ERF008 | 9 | 159 |
| CsERF20* | Csa2G297760 | AT2G33710 | ERF112 | 2 | 285 |
| CsERF21* | Csa2G307850 | AT1G78080 | RAP2-4 | 10 | 362 |
| CsERF22 | Csa2G349090 | AT1G12980 | ESR1 | 4 | 358 |
| CsERF23* | Csa2G354000 | AT4G37750 | ANT | 6 | 696 |
| CsERF24 | Csa2G356600 | AT1G46768 | RAP2-1 | 9 | 142 |
| CsERF25* | Csa2G363010 | AT1G75490 | DREB2D | 8 | 212 |
| CsERF26 | Csa2G374590 | AT5G11590 | DREB3 | 9 | 231 |
| CsERF27 | Csa2G382540 | AT5G25190 | ERF003 | 7 | 175 |
| CsERF28* | Csa2G382550 | AT5G25190 | ERF003 | 7 | 164 |
| CsERF29* | Csa2G401240 | AT1G22810 | ERF019 | 9 | 148 |
| CsERF30* | Csa3G017320 | AT4G17500 | ERF1A | 1 | 279 |
| CsERF31 | Csa3G135120 | AT3G23240 | ERF1B | 1 | 228 |
| CsERF32 | Csa3G018320 | AT4G17490 | ERF6 | 1 | 344 |
| CsERF33 | Csa3G019400 | AT2G44940 | ERF034 | 9 | 214 |
| CsERF34 | Csa3G042390 | AT5G52020 | ERF025 | 9 | 170 |
| CsERF35 | Csa3G073900 | AT3G20310 | ERF7 | 4 | 214 |
| CsERF36* | Csa3G114470 | AT5G65510 | AIL7 | 6 | 283 |
| CsERF37 | Csa3G116720 | AT2G44840 | ERF13 | 1 | 144 |
| CsERF38 | Csa3G116730 | AT2G44840 | ERF13 | 1 | 157 |
| CsERF39 | Csa3G389850 | AT3G23240 | ERF1B | 1 | 224 |
| CsERF40* | Csa3G120390 | AT4G27950 | CRF4 | 3 | 283 |
| CsERF41 | Csa3G124760 | AT5G19790 | RAP2-11 | 3 | 243 |
| CsERF42 | Csa3G133130 | AT1G68550 | ERF118 | 2 | 213 |
| CsERF43 | Csa3G135620 | AT3G23230 | ERF098 | 1 | 158 |
| CsERF44 | Csa3G135630 | AT3G23230 | ERF098 | 1 | 133 |
| CsERF45 | Csa3G152130 | AT5G18560 | ERF086 | 4 | 326 |
| CsERF46* | Csa3G164580 | AT3G16770 | RAP2-3 | 2 | 225 |
| CsERF47 | Csa3G180260 | AT4G25470 | DREB1C | 9 | 213 |
| CsERF48* | Csa3G207390 | AT4G34410 | ERF109 | 2 | 274 |
| CsERF49 | Csa3G357110 | AT4G27950 | CRF4 | 3 | 326 |
| CsERF50* | Csa3G118010 | AT2G44940 | ERF034 | 9 | 230 |
| CsERF51 | Csa3G630280 | AT4G13040 | - | 10 | 234 |
| CsERF52 | Csa3G646560 | AT3G14230 | RAP2-2 | 2 | 370 |
| CsERF53* | Csa3G652380 | AT1G72570 | AIL1 | 6 | 490 |
| CsERF54 | Csa3G736760 | AT2G28550 | RAP2-7 | 6 | 527 |
| CsERF55*# | Csa3G751430 | AT2G36450 | ERF024 | 9 | 183 |
| CsERF56* | Csa3G751440 | AT4G25490 | DREB1B | 9 | 204 |
| CsERF57 | Csa3G812170 | AT3G57600 | DREB2F | 8 | 333 |
| CsERF58* | Csa3G822440 | AT2G41710 | - | 6 | 432 |
| CsERF59 | Csa3G827310 | AT3G54320 | WRI1 | 6 | 337 |
| CsERF60 | Csa3G865380 | AT1G75490 | DREB2D | 8 | 184 |
| CsERF61 | Csa3G878210 | AT5G25390 | SHN3 | 7 | 194 |
| CsERF62 | Csa3G895680 | AT4G32800 | ERF043 | 9 | 227 |
| CsERF63 | Csa4G001970 | AT3G16770 | RAP2-3 | 2 | 272 |
| CsERF64 | Csa4G004960 | AT1G15360 | WIN1 | 7 | 190 |
| CsERF65 | Csa4G006210 | AT5G25190 | ERF003 | 7 | 180 |
| CsERF66 | Csa4G007070 | AT1G28160 | ERF087 | 4 | 227 |
| CsERF67 | Csa4G007650 | AT1G71450 | ERF021 | 9 | 179 |
| CsERF68 | Csa4G023020 | AT5G61600 | ERF104 | 1 | 194 |
| CsERF69 | Csa4G051360 | AT4G23750 | CRF2 | 3 | 319 |
| CsERF70 | Csa4G192030 | AT5G19790 | RAP2-11 | 3 | 225 |
| CsERF71 | Csa4G268100 | AT5G19790 | RAP2-11 | 3 | 211 |
| CsERF72* | Csa4G290800 | AT1G51190 | PLT2 | 6 | 615 |
| CsERF73 | Csa4G292470 | AT2G28550 | RAP2-7 | 6 | 456 |
| CsERF74 | Csa4G314390 | AT2G40340 | DREB2C | 8 | 372 |
| CsERF75 | Csa4G370550 | AT1G21910 | ERF012 | 9 | 232 |
| CsERF76 | Csa4G630010 | AT3G15210 | ERF4 | 4 | 193 |
| CsERF77 | Csa4G641590 | AT3G50260 | ERF011 | 9 | 149 |
| CsERF78* | Csa4G644740 | AT4G37750 | ANT | 6 | 573 |
| CsERF79 | Csa4G649620 | AT1G74930 | ERF018 | 9 | 186 |
| CsERF80 | Csa4G652640 | AT5G25190 | ERF003 | 7 | 203 |
| CsERF81 | Csa5G139630 | AT4G27950 | CRF4 | 3 | 268 |
| CsERF82 | Csa5G146300 | AT5G19790 | RAP2-11 | 3 | 210 |
| CsERF83 | Csa5G150420 | AT1G64380 | ERF061 | 10 | 304 |
| CsERF84 | Csa5G151530 | AT4G23750 | CRF2 | 3 | 318 |
| CsERF85 | Csa5G155560 | AT5G52020 | ERF025 | 9 | 252 |
| CsERF86* | Csa5G155570 | AT4G25470 | DREB1C | 9 | 225 |
| CsERF87 | Csa5G165850 | AT4G17500 | ERF1A | 1 | 250 |
| CsERF88 | Csa5G167110 | AT5G07580 | ERF106 | 1 | 160 |
| CsERF89* | Csa5G167120 | AT5G51190 | ERF105 | 1 | 235 |
| CsERF90 | Csa5G174570 | AT5G51990 | DREB1D | 9 | 200 |
| CsERF91 | Csa5G175970 | AT2G28550 | RAP2-7 | 6 | 441 |
| CsERF92 | Csa5G598600 | AT1G24590 | ESR2 | 4 | 341 |
| CsERF93 | Csa5G608380 | AT1G68840 | RAV2 | 5 | 344 |
| CsERF94 | Csa5G609620 | AT1G68550 | ERF118 | 2 | 309 |
| CsERF95* | Csa5G612310 | AT3G54320 | WRI1 | 6 | 439 |
| CsERF96* | Csa5G637750 | AT5G19790 | RAP2-11 | 3 | 395 |
| CsERF97 | Csa5G647260 | AT5G25810 | TINY | 9 | 163 |
| CsERF98 | Csa5G649870 | AT1G33760 | ERF022 | 9 | 163 |
| CsERF99 | Csa5G649890 | AT1G71450 | ERF021 | 9 | 192 |
| CsERF100* | Csa6G011730 | AT2G40220 | ABI4 | 8 | 305 |
| CsERF101 | Csa6G012810 | AT2G40340 | DREB2C | 8 | 410 |
| CsERF102 | Csa6G167230 | AT1G50640 | ERF3 | 4 | 219 |
| CsERF103* | Csa6G040610 | AT1G21910 | ERF012 | 9 | 201 |
| CsERF104 | Csa6G042450 | AT2G46310 | CRF5 | 3 | 243 |
| CsERF105* | Csa6G055940 | AT5G61890 | ERF114 | 2 | 404 |
| CsERF106 | Csa6G091830 | AT5G13330 | ERF113 | 2 | 191 |
| CsERF107 | Csa6G104640 | AT4G32800 | ERF043 | 9 | 213 |
| CsERF108* | Csa6G124180 | AT2G40340 | DREB2C | 8 | 179 |
| CsERF109 | Csa6G133770 | AT4G27950 | CRF4 | 3 | 281 |
| CsERF110* | Csa6G017030 | AT5G61890 | ERF114 | 2 | 310 |
| CsERF111* | Csa6G296960 | AT2G28550 | RAP2-7 | 6 | 497 |
| CsERF112 | Csa6G318160 | AT3G16770 | RAP2-3 | 2 | 231 |
| CsERF113 | Csa6G361330 | AT1G19210 | ERF017 | 9 | 213 |
| CsERF114 | Csa6G404260 | AT3G54320 | WRI1 | 6 | 366 |
| CsERF115 | Csa6G421660 | AT1G50640 | ERF3 | 4 | 214 |
| CsERF116#* | Csa6G450420 | AT2G36450 | ERF024 | 9 | 224 |
| CsERF117* | Csa6G486790 | AT5G61890 | ERF114 | 2 | 338 |
| CsERF118 | Csa6G490860 | AT4G34410 | ERF109 | 2 | 193 |
| CsERF119 | Csa6G491020 | AT4G36920 | AP2 | 6 | 537 |
| CsERF120* | Csa6G491030 | AT5G67190 | ERF010 | 9 | 173 |
| CsERF121* | Csa6G496390 | AT4G37750 | ANT | 6 | 629 |
| CsERF122 | Csa6G500550 | AT1G78080 | RAP2-4 | 10 | 271 |
| CsERF123 | Csa6G518040 | AT3G15210 | ERF4 | 4 | 246 |
| CsERF124 | Csa6G518290 | AT5G47220 | ERF2 | 1 | 177 |
| CsERF125 | Csa6G518300 | AT4G18450 | ERF091 | 1 | 277 |
| CsERF126 | Csa7G047400 | AT2G20880 | ERF053 | 10 | 397 |
| CsERF127 | Csa7G049230 | AT5G44210 | ERF9 | 4 | 209 |
| CsERF128* | Csa7G073700 | AT4G34410 | ERF109 | 2 | 256 |
| CsERF129 | Csa7G352440 | AT4G13040 | - | 10 | 231 |
| CsERF130 | Csa7G375820 | AT3G23240 | ERF1B | 1 | 231 |
| CsERF131 | Csa7G375830 | AT3G23230 | ERF098 | 1 | 148 |
| CsERF132* | Csa7G431330 | AT1G16060 | ADAP | 6 | 367 |
| CsERF133 | Csa7G432080 | AT5G25190 | ERF003 | 7 | 196 |
| CsERF134 | Csa7G432130 | AT1G80580 | ERF084 | 3 | 183 |
| CsERF135 | Csa7G447150 | AT5G11590 | DREB3 | 9 | 201 |
| CsERF136* | Csa7G448110 | AT1G53910 | RAP2-12 | 2 | 389 |
| CsERF137 | CsaUNG003730 | AT5G13910 | LEP | 4 | 228 |
| CsERF138* | CsaUNG031640 | AT1G13260 | RAV1 | 5 | 317 |
新窗口打开|下载CSV
通过EMSS软件对138个CsERF家族成员进行信号肽和跨膜域预测分析,结果显示有40个CsERF蛋白预测含有信号肽(表1中标注*),其中CsERF55和CsERF116预测含有跨膜结构域(表1中标注#)。
将138个CsERF成员根据物理位置定位到7条染色体上,结果如图2所示。其中,CsaUNG003730和CsaUNG031640未定位于7条染色体中,通过两基因比对至9930_V3版本基因组,发现其定位于5号染色体。同时,通过将138个CsERF家族成员的CDS序列与转录组拼接结果比较,发现Csa2G001000和Csa2G307850两个基因的转录本存在注释错误,依照转录组数据修正后的两基因用于后期生物信息学分析。
图2
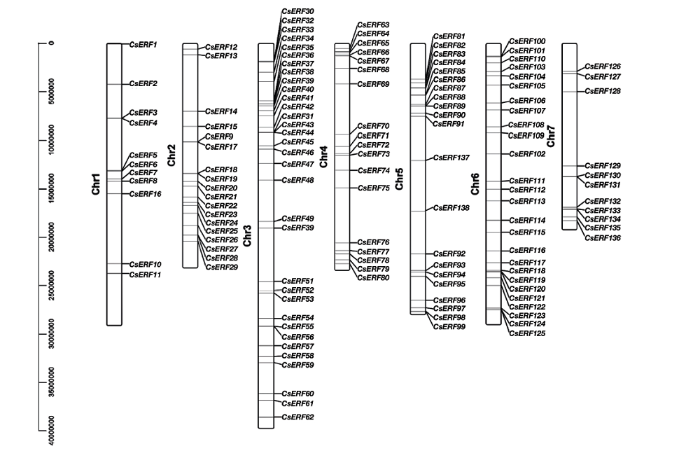 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2黄瓜ERF基因家族在染色体上的位置
Fig. 2The chromosome location of the ERF gene family in cucumber
2.2 黄瓜ERF基因家族进化分析
为了解CsERF家族成员的进化特性,对138个基因构建系统进化树(图3)。CsERF家族成员共分为10个亚家族(Class I—X),其中成员最多的为Class IX,包含37个基因。成员最少的是Class V,包含4个具有B3结构域的基因。图3
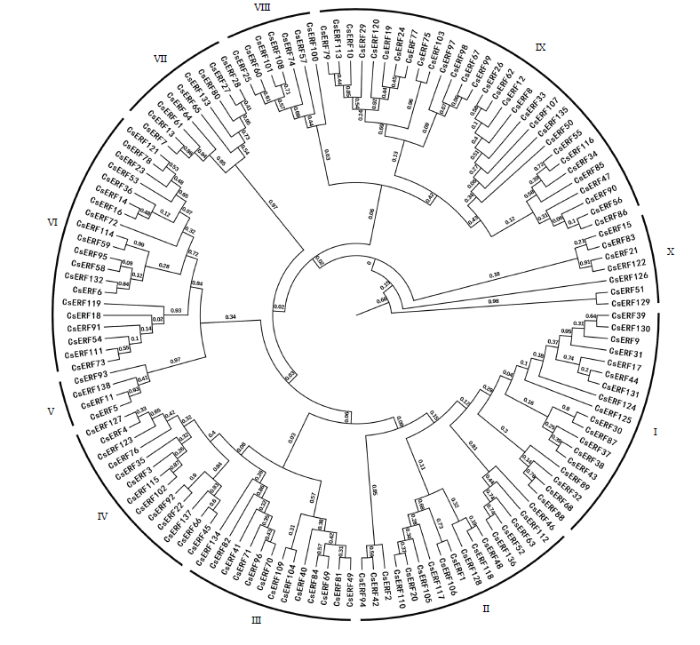 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3黄瓜ERF家族进化树(罗马数字I-X代
Fig. 3The phylogenetic tree of ERF family in cucumber (Roman numerals I-X indicate 10 sub-families)
2.3 黄瓜ERF基因家族基因结构分析
CsERF基因家族的结构分析结果如图4所示,共鉴定出7个motif,其中motif 1在所有家族中均出现,而在除了Class VI外的其他家族中,大部分成员均包含按顺序排列的motif 2-3-1-4。在Class VI中,大部分成员具有按相同顺序排列的motif 2-3/6-1-4-5-2-7- 1-4,说明这一结构域在该亚家族中较为保守。在Class IX中,具有一个特殊的motif 8与motif 4相邻。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4黄瓜ERF基因家族的基因结构分析
红色字体代表在FFMMAA基因型材料顶芽中高表达,蓝色字体代表在ffMMAA基因型材料顶芽中高表达,红色三角号代表雌花芽发育初期表达呈上调趋势,蓝色三角号代表雌花芽发育初期表达呈下调趋势
Fig. 4Gene structure analysis of ERF family members in cucumber
Genes in red indicate highly express in FFMMAA genotype line, genes in blue indicate highly express in ffMMAA genotype line, red and blue triangles indicate genes show an up- and down-regulated trend in female bud development, respectively
综合motif分析结果,虽然不同亚族中包含motif的种类以及排列顺序不尽相同,但在同一亚族中,均包含相同或相似的motif排序,这可能与该亚家族的分子功能相关。
结构域分析表明,所有CsERF家族成员均具有保守的AP2/ERF结构域,其中4个基因具有B3结构域。在CsERF家族成员中,多数亚家族成员无内含子结构,但Class II、Class VI和Class VII亚家族中的大部分成员,基因内具有多个内含子结构。其中Class VI亚家族较为特殊,其成员均包含2个AP2/ERF结构域,且多数内含子分布在AP2/ERF结构域中,推测该亚家族在进化过程中AP2/ERF结构域发生了复制,造成这一结构域被内含子间隔的特殊现象。
2.4 黄瓜ERF在不同性型材料中的特异表达分析
ERF基因家族在多种植物中被报道参与乙烯信号传导过程,在黄瓜花芽性别分化过程中,乙烯信号是决定雌花分化的关键。为鉴定黄瓜ERF家族成员与性别决定与雌花分化过程的关系,通过RT-qPCR方法对全雌株系419-19(FFMMAA)和419-2主茎强雄株系M12(ffMMAA)植株顶芽转录本进行检测。表达存在显著差异(P value<0.05)的基因如图5所示,CsERF9、CsERF30、CsERF31、CsERF37、CsERF87、CsERF98、CsERF105、CsERF106和CsERF110这9个基因在全雌株系中高表达(图5-A),CsERF10、CsERF48、CsERF68、CsERF69、CsERF71、CsERF76、CsERF81、CsERF89、CsERF127和CsERF128这10个基因在强雄株系中高表达(图5-B)。在ERF基因家族中,上述差异表达基因主要集中于Class I和Class II亚家族,且具有成簇分布的趋势,如CsERF9和CsERF31相邻并在全雌株系中高表达,CsERF30、CsERF87和CsERF37也在全雌株系中高表达。图5
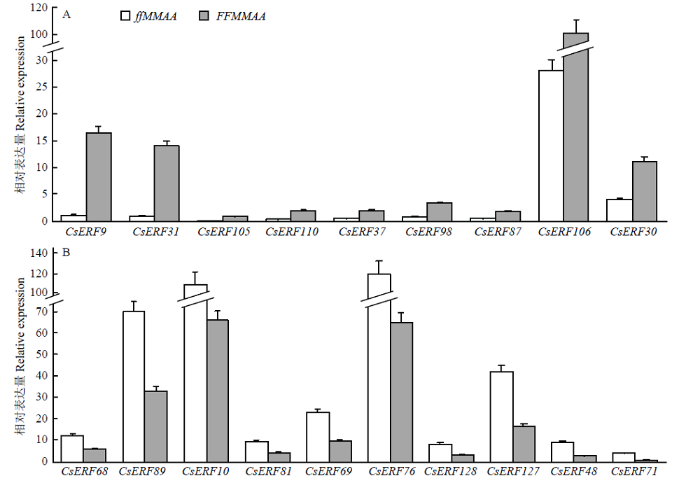 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5在FFMMAA(A)和ffMMAA(B)基因型材料顶芽中显著高表达的CsERF家族成员
Fig. 5The significant differential expression CsERF in FFMMAA (A) and ffMMAA (B) cucumber lines shoot apical (t test, P value<0.05)
2.5 黄瓜ERF基因家族成员在雌花发育初期的表达模式分析
有研究表明,乙烯信号在促进雌花分化的同时,对果实发育也存在调控作用,如CsSF1通过结合M基因(CsACS2)介导其泛素化,进而精细调控M基因的表达,控制果实的发育[18]。M基因的特异表达时期是雌花分化的1—10 mm时期[19],所以本研究分别选取这一时期2、5和10 mm的雌花芽,对ERF基因家族成员和部分ACO和ACS的表达变化趋势进行检测,其中5个ACO命名与前人一致[20]。将不同时期间存在显著表达变化(P<0.05)且差异大于2倍以上的基因,定义为表达存在显著差异的基因。结果如图6所示,共有61个ERF在雌花芽发育初期表达趋势发生变化,其中29个ERF在2—5 mm阶段表达上调,而5—10 mm阶段表达无显著变化,2个ERF在2—10 mm阶段均呈上升趋势。同时,25个ERF在2—5 mm阶段表达下调,而5—10 mm阶段表达无显著变化,5个ERF在2—10 mm阶段均呈下降趋势。此外,A和CsACO2在5—10 mm时期特异高表达,而M和CsACO1在2—5 mm阶段特异高表达。图6
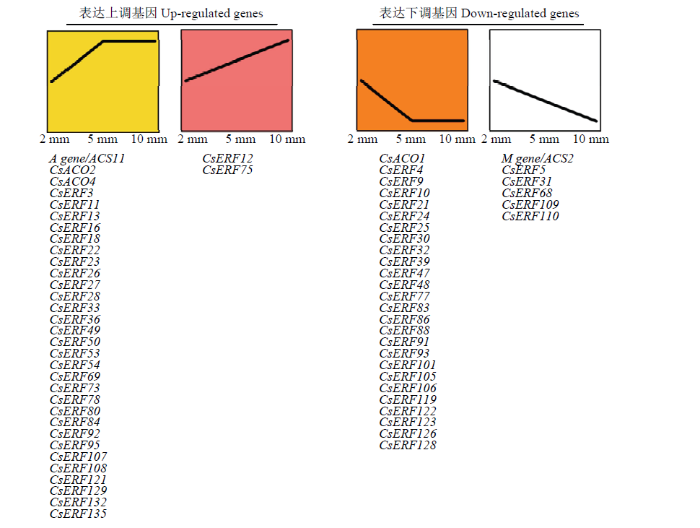 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6CsERF家族成员在雌花芽发育初期表达存在显著变化的成员
Fig. 6The significant differences in the expression of CsERF in early stages of female bud development
值得注意的是,ERF基因家族在雌花芽发育初期的表达趋势具有明显的结构特异性。Class I和Class II亚家族中,表达变化的ERF均呈现下降趋势。同时,Class VI和Class VII中,表达变化的ERF多数呈上升趋势。此外,一些在系统进化树中相邻的基因,也表现出相似的变化趋势,如Class VI中的CsERF16、CsERF36、CsERF53、CsERF23、CsERF78和CsERF121,以及Class IX中的CsERF33、CsERF107、CsERF135和CsERF50。这说明一些进化上趋于保守的基因家族成员具有相似的表达模式,在雌花芽发育初期所发挥的功能可能相似。
2.6 ERF基因家族成员对GCC-box元件具有结合作用
很多ERF转录因子通过与其下游应答元件GCC-box的相互作用来调控基因表达。通过对黄瓜基因组注释基因启动子区进行序列分析,共检测到882个基因启动子具有GCC-box(AGCCGCC)。为验证黄瓜ERF是否具有保守的结合GCC-box功能,选取拟南芥ERF1同源基因CsERF9和CsERF31,通过酵母单杂交系统验证其与GCC-box的结合作用,发现两基因均可以直接结合GCC-box(图7)。这说明部分黄瓜ERF家族成员具有保守结合GCC-box元件的功能,同时也为从转录组数据中高效筛选下游直接调控靶基因提供了分子依据。图7
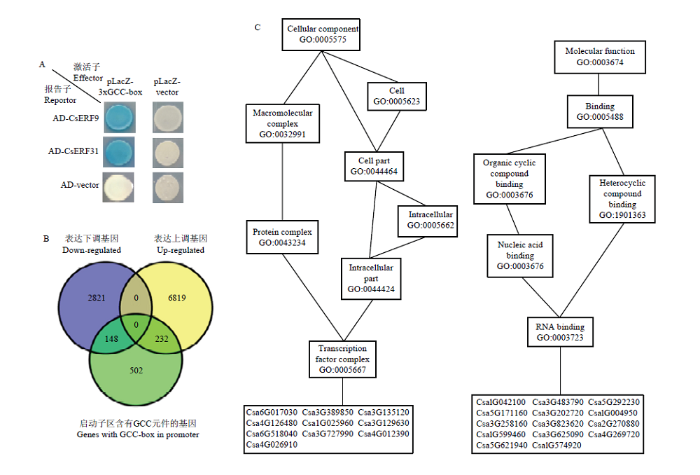 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7酵母单杂交系统验证CsERF31和CsERF9对GCC-box的互作(A),雌花芽发育初期表达成下降趋势的基因与启动子具有GCC-box的基因VENN分析(B),交集中表达下调(左)和上调(右)基因的GO富集分析(C)
Fig. 7Yeast one-hybrid assay of interaction between CsERF31, CsERF39 and GCC-box (A), the two sets of down-regulated and GCC-box in promoter genes analyzed using the VENN method (B), and GO enrichment analysis of down-regulated (left) and up-regulated (right) genes with GCC-box in promoters (C)
为进一步研究雌花芽发育初期受ERF转录因子调控的基因表达变化,从国家基因库生物大数据平台(CNGBdb)的CNP0000657项目中提取2、5和10 mm雌花芽转录组数据(CNS0110220、CNS0110221、CNS0110222、CNS0110223、CNS0110224、CNS0110225)进行表达趋势分析。共筛选出2 969个基因与CsERF9和CsERF31表达趋势相似,随着雌花芽分化表达呈下降趋势,同时筛选出7 051个基因的表达随雌花芽分化呈上升趋势。通过将这些基因与882个启动子具有GCC-box的基因进行VENN交集分析,发现有148个基因的表达随着雌花芽分化下调,而232个基因上调,且这些基因启动子均具有GCC-box(图7-B)。将上调和下调基因进行分子功能(molecular function)GO富集分析,发现上调基因中富集最显著的是GO: 0005667途径,共有10个基因富集其中,注释与转录复合体(transcription factor complex)相关。在下调基因中富集最显著的是GO:0003723途径,共有14个基因富集其中,注释与RNA结合(RNA binding)相关。
3 讨论
3.1 雌花芽分化2 mm阶段是器官发育的关键时期
自1994年在拟南芥中分离出第一个与花发育相关基因AP2[21]以来,已报道大量对AP2/ERF转录因子的基因家族分析,包括玉米[22]、粟[23]、葡萄[24]和橙[25]等。本研究主要关注黄瓜ERF基因家族在黄瓜雌花芽的分化与发育过程中的表达趋势变化,进而为深入研究这一重要生物学过程的分子机制提供数据基础。根据前人对黄瓜花芽性别分化时期的划分,6—7时期(Stage 6—7)是花芽性别分化时期[26]。在6—7时期,花芽长度小于1 mm,而在2 mm阶段,是花芽由性别分化向雌器官起始发育的转变期。2 mm阶段的雌花芽,子房已经起始发育,而在5—10 mm阶段,已发育出具有明显形态特征的子房,各雌器官均已分化完成。在ERF基因家族中,大部分基因的表达变化集中于雌花芽2—5 mm阶段,说明这一阶段可能发生了复杂的基因表达调控,使雌性器官得以正确的分化。3.2 Class I和Class II类基因可能在雌花的分化与发育过程中发挥功能
在黄瓜ERF基因家族的10个亚家族中,Class I和Class II成员的表达模式较为特殊。值得注意的是在雌花芽分化初期,Class I和Class II中所有表达发生变化的基因,均呈现下降趋势(图4中蓝色箭头标注基因)。表达量呈下降趋势暗示这些基因可能在花芽发育最初阶段发挥功能,随着子房发育,其表达被抑制而趋于稳定。此外,选取不同性别类型植株和不同雌花芽发育阶段进行表达检测,是将雌花的性别分化与雌性器官发育假定为两个生物学过程。因此,图4中“红色字体,蓝色三角号”基因(即在全雌株顶芽高表达,并随雌花芽发育表达下降)推测与性别分化更相关,“蓝色字体,红色三角号”基因(即在强雄株顶芽高表达,并随雌花芽发育表达上升)则可能与雌花芽发育更相关,“蓝色字体,蓝色三角号”基因(即在强雄株顶芽高表达,并随雌花芽发育表达下降)更有可能与雄花分化相关。值得注意的是,在所有具有表达变化的ERF中,未发现在全雌株系中高表达,且随着雌花芽分化表达上调的基因(即图4中“红色字体,红色三角号”)。据此推测ERF参与性别分化与雌花芽发育的过程受到严格调控,在不同性别花芽、不同花芽分化阶段发挥不同的功能。
3.3 部分黄瓜ERF的调控机制与模式植物相似
在黄瓜ERF基因家族中,CsERF9、CsERF31、CsERF39以及CsERF130在拟南芥中的直系同源基因均为ERF1B。拟南芥ERF1B在乙烯信号传导通路中,受上游EIN3/EIL1等基因调控,通过激活下游基因参与多种生物学过程[27]。在上述4个基因中,CsERF31已被报道具有正调控M基因(CsACS2)的功能,这说明其很可能通过乙烯信号传导途径参与花芽的性别分化过程。此外,CsERF105和CsERF110在拟南芥中的同源基因均为ERF114,且两基因具有与CsERF31和CsERF9相似的表达趋势,其中CsERF110已被报道具有正调控A基因(CsACS11)的功能[10],而CsERF31被报道可能参与M基因自反馈调控[28,29,30]。有报道显示,F(CsACS1G)、M(CsACS2)和A(CsACS11)的不同基因型材料,其乙烯释放速率存在显著差异[31],上述结果暗示,这类基因很可能通过乙烯信号传导途径介导黄瓜雌花的分化。在Class VI中,CsERF23、CsERF78和CsERF121在拟南芥中的同源基因均为ANT。ANT在拟南芥中调控花芽等侧生器官发育,其突变体在育性、子房发育和侧生器官分化方面均存在缺陷[32]。本研究中,CsERF23、CsERF78和CsERF121随雌花芽发育表达上调,这暗示其可能具有调控雌性器官发育的功能。CsERF69和CsERF84在拟南芥中的同源基因为CRF2。CRF2在拟南芥中受细胞分裂素诱导,转移至细胞核调控下游基因[33]。CsERF69和CsERF84的表达随雌花芽分化上调,推测其同样受到细胞分裂素等激素的调控,在子房发育过程中行使功能。
此外,拟南芥ERF1同源蛋白CsERF9和CsERF31具有结合乙烯响应元件GCC-box的功能,说明黄瓜ERF家族基因可能存在与模式植物相似的分子调控方式影响下游基因的表达。通过进一步的生物信息学分析,鉴定出大量在雌花芽发育初期表达变化的基因,对启动子具有GCC-box的基因进行GO聚类分析,发现富集最显著的是转录因子复合体和RNA结合两个途径。转录因子复合体和RNA结合蛋白均为调控RNA转录或转录后翻译的影响因子,其在雌花芽发育初期表达的集中变化,说明这一过程可能涉及转录激活调控、转录后调控以及翻译后修饰等一系列调控过程,近期报道的CsSF1介导M(CsACS2)蛋白泛素化调控子房发育也为这一过程提供了新的分子证据[18]。综上所述,推测ERF在雌花芽发育初期的表达变化,直接或间接影响了下游基因的表达,使雌花芽中各器官得以正确分化,进而完成果实的发育。
4 结论
本研究从黄瓜9930_V2基因组中鉴定出138个具有AP2/ERF结构域的ERF基因家族成员,可分为10个亚族。在花芽性别分化和雌花芽发育不同阶段中,ERF具有明显的表达变化,并且其表达变化与基因结构存在相关性。一些ERF可以结合保守元件GCC-box,推测其通过结合GCC-box来调控下游靶基因的表达。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[OL],
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
2007-2015, [OL].
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
