 ,山东农业大学园艺科学与工程学院/作物生物学国家重点实验室/山东果蔬优质高效生产协同创新中心,山东泰安 271018
,山东农业大学园艺科学与工程学院/作物生物学国家重点实验室/山东果蔬优质高效生产协同创新中心,山东泰安 271018Genome Identification of PpGRAS Family and Expression Pattern Analysis of Responding to UV-B in Peach
LI Chen, ZHAO XueHui, WANG QingJie, WANG XuXu, XIAO Wei, CHEN XiuDe, Fu XiLing, LI Ling, LI DongMei ,College of Horticulture Science and Engineering, Shandong Agricultural University/State Key Laboratory of Crop Biology/Shandong Collaborative Innovation Center for Fruit & Vegetable Production with High Quality and Efficiency, Taian 271018, Shandong
,College of Horticulture Science and Engineering, Shandong Agricultural University/State Key Laboratory of Crop Biology/Shandong Collaborative Innovation Center for Fruit & Vegetable Production with High Quality and Efficiency, Taian 271018, Shandong通讯作者:
责任编辑: 赵伶俐
收稿日期:2019-06-11接受日期:2019-08-3网络出版日期:2019-12-16
| 基金资助: |
Received:2019-06-11Accepted:2019-08-3Online:2019-12-16
作者简介 About authors
李晨,Tel:15650453756;E-mail:15650453756@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (7636KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李晨, 赵雪惠, 王庆杰, 王旭旭, 肖伟, 陈修德, 付喜玲, 李玲, 李冬梅. 桃GRAS家族全基因组鉴定与响应UV-B的表达模式分析[J]. 中国农业科学, 2019, 52(24): 4567-4581 doi:10.3864/j.issn.0578-1752.2019.24.011
LI Chen, ZHAO XueHui, WANG QingJie, WANG XuXu, XIAO Wei, CHEN XiuDe, Fu XiLing, LI Ling, LI DongMei.
0 引言
【研究意义】桃是中国北方的主栽果树之一,随着果树集约化栽培的推行,设施桃栽培得到了迅速发展。然而与露天栽培相比,设施栽培果树表现出果实甜度变低、风味偏淡、综合品质下降等问题,棚膜吸收、反射太阳光导致设施内UV-B弱化是重要的影响因素之一[1]。GRAS基因家族成员在植物生长发育的调节中发挥着关键作用,包括光信号转导、GA信号调节和腋芽分生组织形成等[2]。对桃GRAS基因家族进行生物信息学分析、组织特异性表达及响应UV-B处理的表达模式分析,有利于深入了解GRAS家族基因的潜在功能,并对桃响应UV-B光信号的基因挖掘具有重要意义。【前人研究进展】自20世纪80年代后期,人类活动造成了臭氧层空洞,而臭氧量的减少导致到达地表的UV-B辐射增强。UV-B是紫外线中的中波辐射(280—320 nm),约占太阳辐射总量的1.5%。其能量高,且能被核酸、蛋白质及脂类等物质吸收。研究表明UV-B具有损伤DNA、改变蛋白质结构;破坏光合作用;造成植株矮化、株型缩小,生长受抑制等负作用,对生态系统和动、植物生长产生深远影响[1,3-4]。事实上,UV-B对植物的影响效应具有双重性,高UV-B辐射对植物生长产生不良影响,低剂量的UV-B在基因表达、代谢活动以及生长发育等方面反而会起到正面的调节作用[5,6]。UV-B辐射是应激源还是调节器与其辐射强度和时间有关[7]。设施条件下的UV-B强度较露天弱,叶片中紫外吸收物质较露天少,补充UV-B后含量总体得到提升[8]。比较紫外线敏感品种‘旭日’和不敏感品种‘朝阳’叶片中的类黄酮含量发现,经较高强度UV-B辐射后,前者类黄酮含量先升高后降低,变化明显,后者则相对稳定,表明朝阳更能有效适应UV-B强度的增加[7]。光受体UV RESISTANCE LOCUS8(UVR8)被发现后,UV-B由环境胁迫因子变成了特殊的信号调控因子[1],其在光感受中具有独特的调节机制[9,10]。在UV-B处理后,UVR8能与下游多种转录因子相互作用,从而启动光形态发生[11]。长时间以较低UV-B剂量照射作物,并未发现ROS积累及其编码基因的表达,反而会启动UVR8信号通路,引起良性应激,但UV-B剂量超出作物承受范围时会引起破坏性应激[12]。更有研究发现大多数植物在UV-B辐射下表现出植株形态、生理生化和激素等各个方面的变化[1]。转录因子在植物生长发育过程中发挥尤为重要的作用。GRAS基因家族通常被认为是植物特异性转录调节因子家族[13],能够响应激素、光等多种信号来调节植物的生长发育。GRAS蛋白质由可变的N末端和高度保守的C末端组成,其被称为GRAS结构域。该结构域主要由两个亮氨酸七肽重复序列(LHRI和LHRII)组成的VHIID、PFYRE和SAW共3个基序组成[14,15]。在拟南芥中,将GRAS家族蛋白分为8个亚家族:DELLA、LS、SCR、SHR、PAT1、HAM、SCL9(LISCL)和SCL4/7[14]。而在水稻中[15]也认为可能会分为8个亚科,但包括一个新的亚家族SCL3,而不是SCL4/7。葡萄中GRAS家族蛋白序列也分成了8个亚家族,且其氨基酸序列主要为α-螺旋和随机卷曲,其三维结构相似[16]。拟南芥、水稻和其他植物种类的GRAS蛋白序列构建系统进化树分析发现,其具有12个独立的分支[17],这些研究表明该基因家族在植物中表现出多样化。其中,DELLA亚家族成员AtGAI参与了拟南芥中的赤霉酸信号传导[18],葡萄中VvRGL1/RGL2可能响应GA负调控参与无核果实的发育[19],AtLAS亚家族成员Ls在番茄的腋芽分生组织中起作用[20],AtPAT1亚家族成员AtPAT1、AtHAM维持了拟南芥顶端分生组织的属性与更新[17,21],PAT1分支中的PAT1和SCL13作为光敏色素信号通路的中间介质发挥作用[22],AtSCL3亚家族成员AtSCL3对促进拟南芥的根细胞伸长起作用[23]。MA等[24]证明在胡杨中过量表达PeSCL7(AtSCL4/7亚家族)增强了转基因拟南芥的抗旱性和耐盐性。【本研究切入点】目前在模式植物拟南芥中,对GRAS基因家族的结构、进化、相互作用机理以及抵御逆境等方面的研究较为深入。然而,在木本植物特别是果树中该家族基因的系统报道较少,桃GRAS基因家族成员响应UV-B光信号的功能有待进一步研究。【拟解决的关键问题】以设施栽培的‘中油5号’油桃的叶片、果实和韧皮部为研究材料,对GRAS家族中的48个成员进行生物信息学分析,旨在了解其基本理化性质、家族分类、保守结构域以及基因结构等;通过检测GRAS基因家族成员在UV-B处理后的叶片、果实和韧皮部中的表达水平,了解GRAS家族的基因性质,筛选该家族中可能响应UV-B光信号的重要基因。1 材料与方法
试验于2018年在山东农业大学园艺科学与工程学院进行。1.1 试验材料
试材为7年生设施油桃‘中油5号’(Prunus persica var. nectarina cv. Zhongyou5)。自开花至果实成熟期间,用1.44 kJ·m-2·d-1 UV-B照射处理植株(此剂量由前期试验筛选得出)。UV-B由专用LED UV-B灯(40 W,297 nm,南京Kazhi)提供,悬挂在树体上方1.5 m处,通过电子自动控制装置控制紫外灯的开/关时间,每天上午9:30—10:30补充1 h,阴雨天和下雪天停止照射。使用配备有297 nm探针的UV-B型单通道UV辐照器(北京师范大学光电仪器厂)测定灯下80—120 cm范围内的UV-B辐射剂量为1.44 kJ·m-2·d-1,对该范围内的功能叶(以树体外围一年生枝的生长点为起始点,倒数第6—8片叶)、果实和一年生枝韧皮部进行取样,以相同设施条件下未进行UV-B照射的‘中油5号’为对照,3次重复。液氮速冻后-80℃保存备用。1.2 数据来源与分析方法
1.2.1 桃GRAS的获取、理化性质预测与基因染色体定位分析从Plant TFDB(http://planttfdb.cbi.pku.edu. cn/)下载桃GRAS家族数据,利用SMART(http://smart. embl-heidelberg.de/)和NCBI-CDD工具进行蛋白结构预测,检测候选蛋白的结构域,去除不含GRAS结构域的序列,筛选候选基因。利用ProtParam tool(http://web.expasy.org/protparam/)对所有GRAS家族氨基酸序列进行分子量和等电点预测。根据染色体的位置依次命名,利用MCScanX(http://chibba.pgml.uga. edu/mcscan2/)[25]和Circos软件(http://circos.ca/)[26]绘制桃GRAS基因家族成员在染色体上的定位分布。2个或多个同源基因在染色体上的物理位置不超过100 kb即视为串联重复基因[27]。
1.2.2 系统进化树构建、结构域分析及基因结构分析
参照NAKANO等[28]的分类方法,将拟南芥与桃GRAS家族成员的蛋白序列用Clustal W进行多重序列比对后导入进化树分析软件MEGA 6.0,采用相邻连接法(neighbor-joining NJ;执行参数:Bootstrap method 1000,Poisson model,Pairwise deletion)构建系统进化树,对桃GRAS蛋白进行亚族分类。利用MEME(http://me-me-suite.org/index.html)对桃GRAS保守域结构进行分析。利用GSDS online(http://gsds.cbi. pku.edu.cn/)预测基因结构。
1.3 总RNA的提取和qRT-PCR
使用RNAprep Pure多糖多酚植物总RNA提取试剂盒(天根,Cat No.DP441)提取液氮冻样的RNA,利用HiScript ® II Q RT SuperMix for qPCR(+gDNA wiper)试剂盒(Vazyme公司,中国)合成cDNA第一链。采用实时荧光定量PCR的方法,用cDNA模板检测各基因表达水平,利用Primer 3.0设计荧光定量引物,引物序列见表1,内参为桃β-Actin,由生工生物工程(上海)股份有限公司合成。使用ChamQTM Universal SYBR® qPCR Master Mix(Vazyme)进行实时荧光定量PCR。qRT-PCR反应体系为:10.0 μL 2×ChamQ Universal SYBR qPCR Master Mix,上、下游引物分别为0.4 μL(10 μmol·L-1),1.0 μL模板,8.2 μL ddH2O。每个样品进行3次技术重复。反应条件为:95℃预变性30 s;95℃ 5 s,60℃ 30 s,40个循环,绘制溶解曲线,95℃ 15 s,60℃ 60 s,95℃ 15 s。用2-∆∆CT法对试验数据进行定量分析。
Table 1
表1
表148个PpGRAS的qRT-PCR引物序列
Table 1
| 引物名称 Locus name | 上游序列(5'-3') Forward primer (5'-3') | 下游序列(5'-3') Reverse primer (5'-3') |
|---|---|---|
| Prupe.1G119100 | CACCCTTCTTTGTCACACGG | CCCAGCCCTCACATTCCTAA |
| Prupe.1G119200 | AACAATTGTGGGGACTTCGC | ACCTGCCCTACAAACCCTAC |
| Prupe.1G199300 | TCCTCTCCGAGTTCAACCAC | CGAGCCTTATCCCGGAATCT |
| Prupe.1G237500 | ACTGGAGGAGCTCTGAGAGA | ATGACGTTCCTGATTTCGCG |
| Prupe.1G329600 | CTGAGTTCGCAATGAGGAGC | GGGCCTCAATAAAACGGGTC |
| Prupe.1G501500 | GCATGCGTTCGATCTGGATT | CAATGAGCTCGTGGAAGTCG |
| Prupe.2G028700 | CCATCCTCATCGGCTCAGAT | CTTTCTCGGCGAGCTCTTTC |
| Prupe.2G138400 | CACCACCTGACCAGTACCAT | TCCGTTTCTGGCAGGGTATT |
| Prupe.2G237300 | GAGGGAAAGGAAAGGGTGGA | TCCTTCCATCCCAACAGCAT |
| Prupe.2G299400 | TTAGCTTTGAAGTCCCGGGT | ATTCGTCGCTGATTTGTCCG |
| Prupe.3G040400 | GCGGATTAGTGCAGAGATGC | GACTCCTGATAGCCTCCGTC |
| Prupe.3G162500 | TCAAGGCAGCCAGTGGATTA | AATTGCCTCTCCTGGTCGAA |
| Prupe.3G187000 | TGGTGGACGAAGATGCAGAT | GAACCCCTCGTACGCTATCA |
| Prupe.3G201900 | GCATGCGAAGGAGAGGAAAG | TCCAAAATGAAGTGCACCGG |
| Prupe.3G222800 | CACCCTTCTTTGTCACACGG | CCCAGCCCTCACATTCCTAA |
| Prupe.3G249700 | ACGACTCTGAGCCACAAGAA | TTCTGCTTCTTTGAACCCGC |
| Prupe.3G309800 | GCAGCATTCTTCCCCGTATG | GCCAAGAAATGTGACACCGT |
| Prupe.4G007500 | AGGGGTAGGGGAAGCTAGAA | TGCTTCTCCCTTCTTCTGCA |
| Prupe.4G132100 | AGTGGTGGCTATTTCGGTGA | GCCTCCTCAACTCCCTTCTT |
| Prupe.4G200200 | TCTTCCACTTCCAAGCGCTA | CCCAGCCCTCACATACCTAG |
| Prupe.4G233000 | TGGCAGGCTAGGTATGTGAG | CTTTCCATCCCTGCAGCATC |
| Prupe.4G258200 | TCTTCCACTTCTCTGCCCTG | GCAAACCAGCCCTTGTACAA |
| Prupe.5G072300 | AGAGTTGTGAGGCCTGAGAC | CTTCCATCCTTGCAGCATCC |
| Prupe.5G091700 | TCTGAGCACAAAGAGGGGAG | GCCTTATCTGCCTGAGTTGC |
| Prupe.5G156100 | GCTGGCGATTGTTAACGGAT | ATCCTTTCTGAACCCTCGCA |
| Prupe.6G037000 | CCCTCTCTTCTTCAACCGGT | CAAAACCTGCCCTCCTCAAC |
| Prupe.6G073300 | TTGGTCAATCTTTGGAGCGC | GAACCCCTTCTCCCATGTCA |
| Prupe.6G073400 | GGCCACAAGTTCCTCCTTTG | CTACCGCCAAGTTGCTCATC |
| Prupe.6G073500 | GTTGACGGGCTTGACTTTGT | ACTAAACGTCACGGGGCTAA |
| Prupe.6G073600 | TTTTGCAAGGCCACAGACTC | ACCGTTTGAAGCCACATGAC |
| Prupe.6G073700 | GACATCGATGAGGAGGAGCT | TGTGGATCAAATGGAGGCCT |
| Prupe.6G073800 | TGGTCATGTCCGAGGTGTAC | CCATTGTTCTCCTCCACCCT |
| Prupe.6G073900 | CGATGAACCAAGGGATGCAG | TCGCCACAAACCCTCTGTAT |
| Prupe.6G074000 | TCGAAATCTCCGAGCTCTCC | ATTGGCGGTGAAGTGAGAGA |
| Prupe.6G220800 | GATTTGGCGACGCTGAGATT | GCTCCACCATATGCCTCTCT |
| Prupe.6G223200 | AGGAAGGAGGAAGGGGAGAT | TTGACTTGGTTATGGGGCCT |
| Prupe.6G257000 | AAAGATGGAGGACACTGGCA | GGTTGTCTCATTGTGCTGCA |
| Prupe.6G282800 | CTGGCTATCTCCTGGGATGG | TCAGGAGAGGCTAGCTGAGA |
| Prupe.6G300900 | GGAGGGTGTGGAGAGAGAAG | AGAGAGGCCTTTCGTTCCAA |
| Prupe.7G150900 | CGCTTTCTCTCTCACTCCCT | CAGAAAACCGGAACCCAGTG |
| Prupe.7G153700 | ACGAAGGGTCATCATCTGCA | TGGGAAATTTGCTCAAGGGC |
| Prupe.7G236100 | ACTCACTCGATGCTAGCCTC | CCAAAGTGTACCCATCGCAG |
| Prupe.8G016200 | CCCCTTCATCAAGTTTGCCC | ATGTTCCCACTCCGGTCATT |
| Prupe.8G054000 | TGAGGGAAATGGAGCAGAGG | GCCACACGCTGAAAAGAAGA |
| Prupe.8G220900 | TTGCATGCCACCAAGAAGTC | TCTTCTGCTTGCCTGCTACT |
| Prupe.8G266100 | TCTCATGCACCTCCGATCTC | ATATGGAATGGTCGGGCGAT |
| Prupe.I004400 | AAGACCAGTCACTTCTCCGG | CGAAACGCTGCTTCCGATTA |
| Prupe.I004500 | CAAGAGCTATTGGTTCGGGC | CCTGTTTCGATCAGCTCTGC |
| Actin | GTTATTCTTCATCGGCGTCTTCG | CTTCACCATTCCAGTTCCATTGTC |
新窗口打开|下载CSV
1.4 数据分析
采用Microsoft Excel 2003和SPSS 19.0软件进行数据处理和统计分析,包括单因素方差分析和差异显著性分析,应用GraphPad Prism 6.01软件绘图。2 结果
2.1 桃GRAS转录因子家族成员与染色体分布分析
从桃基因组序列中鉴定得到48个GRAS转录因子家族成员,根据基因在染色体上的位置,对所有PpGRAS转录因子进行系统编号。PpGRAS开放式阅读框长度在1 218—2 550 bp,蛋白长度在406—850 aa,平均长度590.52 aa,分子质量在45 828.1—78 010.1 Da,等电点在4.36—7.56,保守结构域分析结果表明,鉴定出的48个PpGRAS蛋白均具有GRAS特征结构域。桃GRAS转录因子的详细信息见表2。Table 2
表2
表2桃GRAS及其相关信息
Table 2
| 基因编号 Gene name | 基因登录号 Locus name | 染色体 Chromosome | 类别 Group | 定位 Locus | 开放阅读 框长度 ORF length (bp) | 蛋白长度 Protein length (aa) | 分子量 Molecular weight (Da) | 等电点 Isoelectric point |
|---|---|---|---|---|---|---|---|---|
| PpGRAS1 | Prupe.1G119100 | 1 | Ⅵ | 9323354 9325141 | 1788 | 596 | 66738.9 | 4.93 |
| PpGRAS2 | Prupe.1G119200 | 1 | Ⅴ | 9326642 9327967 | 1326 | 442 | 49126.9 | 6.94 |
| PpGRAS3 | Prupe.1G199300 | 1 | Ⅲ | 19270496 19271845 | 1350 | 450 | 50539.2 | 5.08 |
| PpGRAS4 | Prupe.1G237500 | 1 | Ⅴ | 25203094 25204641 | 1548 | 516 | 57213.2 | 6.51 |
| PpGRAS5 | Prupe.1G329600 | 1 | Ⅳ | 31282447 31283783 | 1311 | 437 | 48095.8 | 6.83 |
| PpGRAS6 | Prupe.1G501500 | 1 | Ⅶ | 41498576 41500096 | 1521 | 507 | 55337.3 | 5.20 |
| PpGRAS7 | Prupe.2G028700 | 2 | Ⅴ | 2987997 2990761 | 1386 | 462 | 52324.3 | 6.28 |
| PpGRAS8 | Prupe.2G138400 | 2 | Ⅷ | 19548826 19551964 | 2145 | 715 | 78010.1 | 5.40 |
| PpGRAS9 | Prupe.2G237300 | 2 | Ⅱ | 25838372 25841556 | 2007 | 669 | 75561.2 | 5.62 |
| PpGRAS10 | Prupe.2G299400 | 2 | Ⅰ | 28850212 28852416 | 1938 | 646 | 70121.6 | 7.19 |
| PpGRAS11 | Prupe.3G040400 | 3 | Ⅲ | 2895471 2897333 | 1647 | 549 | 61096.3 | 6.41 |
| PpGRAS12 | Prupe.3G162500 | 3 | Ⅳ | 18144529 18147307 | 1782 | 594 | 64551.4 | 4.91 |
| PpGRAS13 | Prupe.3G187000 | 3 | Ⅴ | 20103771 20105111 | 1341 | 447 | 48568 | 5.80 |
| PpGRAS14 | Prupe.3G201900 | 3 | Ⅰ | 21021917 21025350 | 1668 | 556 | 62383.7 | 6.96 |
| PpGRAS15 | Prupe.3G222800 | 3 | Ⅲ | 22307572 22309814 | 1590 | 530 | 58784.2 | 6.51 |
| PpGRAS16 | Prupe.3G249700 | 3 | Ⅵ | 23979126 23980343 | 1218 | 406 | 45828.1 | 7.56 |
| PpGRAS17 | Prupe.3G309800 | 3 | Ⅶ | 26939961 26942210 | 2127 | 709 | 78477.6 | 6.15 |
| PpGRAS18 | Prupe.4G007500 | 4 | Ⅰ | 396663 398565 | 1623 | 541 | 60455.4 | 6.24 |
| PpGRAS19 | Prupe.4G132100 | 4 | Ⅵ | 7314327 7315982 | 1599 | 533 | 59633.6 | 4.61 |
| PpGRAS20 | Prupe.4G200200 | 4 | Ⅰ | 12432037 12435877 | 1758 | 586 | 65220.3 | 5.84 |
| PpGRAS21 | Prupe.4G233000 | 4 | Ⅰ | 14976407 14979370 | 1713 | 571 | 63145.4 | 5.17 |
| PpGRAS22 | Prupe.4G258200 | 4 | Ⅵ | 18325403 18326818 | 1416 | 472 | 52583.6 | 6.00 |
| PpGRAS23 | Prupe.5G072300 | 5 | Ⅷ | 8700201 8703734 | 2268 | 756 | 82196.4 | 6.55 |
| PpGRAS24 | Prupe.5G091700 | 5 | Ⅴ | 10228192 10230468 | 2094 | 698 | 76771.2 | 5.37 |
| PpGRAS25 | Prupe.5G156100 | 5 | Ⅵ | 13931708 13933297 | 1590 | 530 | 58679 | 6.31 |
| PpGRAS26 | Prupe.6G037000 | 6 | Ⅰ | 2793373 2794605 | 1233 | 411 | 46253.6 | 6.94 |
| PpGRAS27 | Prupe.6G073300 | 6 | Ⅱ | 4999593 5001653 | 2061 | 687 | 77912 | 4.95 |
| PpGRAS28 | Prupe.6G073400 | 6 | Ⅱ | 5005442 5008392 | 2154 | 718 | 81656.6 | 6.58 |
| PpGRAS29 | Prupe.6G073500 | 6 | Ⅱ | 5010337 5012421 | 2085 | 695 | 78198.1 | 6.34 |
| PpGRAS30 | Prupe.6G073600 | 6 | Ⅱ | 5020790 5022844 | 2055 | 685 | 77016.8 | 6.09 |
| PpGRAS31 | Prupe.6G073700 | 6 | Ⅱ | 5025889 5029019 | 2076 | 692 | 78813.9 | 6.22 |
| PpGRAS32 | Prupe.6G073800 | 6 | Ⅱ | 5037609 5040687 | 2121 | 707 | 79850.6 | 5.92 |
| PpGRAS33 | Prupe.6G073900 | 6 | Ⅱ | 5044672 5047324 | 2355 | 785 | 88209.9 | 5.58 |
| PpGRAS34 | Prupe.6G074000 | 6 | Ⅱ | 5052682 5054723 | 1998 | 666 | 74774.8 | 6.41 |
| PpGRAS35 | Prupe.6G220800 | 6 | Ⅲ | 22642849 22644378 | 1530 | 510 | 57075.9 | 5.46 |
| PpGRAS36 | Prupe.6G223200 | 6 | Ⅰ | 22836944 22839388 | 1437 | 479 | 53107.6 | 5.58 |
| PpGRAS37 | Prupe.6G257000 | 6 | Ⅱ | 24964599 24968826 | 2253 | 751 | 84278.9 | 6.66 |
| PpGRAS38 | Prupe.6G282800 | 6 | Ⅲ | 26358992 26360827 | 1611 | 537 | 60502 | 4.36 |
| PpGRAS39 | Prupe.6G300900 | 6 | Ⅴ | 27305816 27309379 | 2550 | 850 | 91964.8 | 6.81 |
| PpGRAS40 | Prupe.7G150900 | 7 | Ⅷ | 16214663 16216336 | 1674 | 558 | 62194.2 | 6.20 |
| PpGRAS41 | Prupe.7G153700 | 7 | Ⅵ | 16381917 16385916 | 1806 | 602 | 65807.8 | 5.08 |
| PpGRAS42 | Prupe.7G236100 | 7 | Ⅳ | 20509687 20511389 | 1614 | 538 | 59160.2 | 5.81 |
| PpGRAS43 | Prupe.8G016200 | 8 | Ⅰ | 1519578 1522426 | 1641 | 547 | 60817.4 | 4.83 |
| PpGRAS44 | Prupe.8G054000 | 8 | Ⅳ | 6129612 6131018 | 1407 | 469 | 53325 | 7.17 |
| PpGRAS45 | Prupe.8G220900 | 8 | Ⅵ | 20048389 20050058 | 1539 | 513 | 57646.5 | 6.55 |
| PpGRAS46 | Prupe.8G266100 | 8 | Ⅶ | 22267137 22269341 | 1923 | 641 | 72100.3 | 5.58 |
| PpGRAS47 | Prupe.I004400 | U | Ⅱ | 740 3677 | 2121 | 707 | 79888.5 | 5.82 |
| PpGRAS48 | Prupe.I004500 | U | Ⅱ | 13496 16456 | 2037 | 679 | 76744.6 | 6.60 |
新窗口打开|下载CSV
依据PpGRAS的染色体位置信息,利用Circos软件绘制其染色体定位图。由图1可知,PpGRAS中有2个基因(PpGRAS47和PpGRAS48)无匹配的染色体定位信息。染色体定位分析发现,PpGRAS在桃的8条染色体上呈现不均匀分布,其中第6号染色体上分布最多,为14个;其次是第3号染色体,为7个;第5和7号染色体分布最少,均为3个。由于基因重复能显著促进基因家族的扩展和蛋白质功能的多样化,本研究分析了该基因家族的串联重复。结果显示7对共线GRAS基因(PpGRAS4/PpGRAS25;PpGRAS5/ PpGRAS12;PpGRAS8/ PpGRAS23;PpGRAS9/PpGRAS33;PpGRAS9/PpGRAS37;PpGRAS14/ PpGRAS18;PpGRAS28/ PpGRAS37)和10个串联GRAS基因(PpGRAS1/ PpGRAS2;PpGRAS27/PpGRAS28/PpGRAS29/PpGRAS30/ PpGRAS31/PpGRAS32/PpGRAS33/PpGRAS34),占PpGRAS总数的20.83%。共线基因是能通过复制在另一基因组以相同连续顺序存在的旁系同源基因,除PpGRAS8和PpGRAS23外,所有共线GRAS都在同一亚家族中。
图1
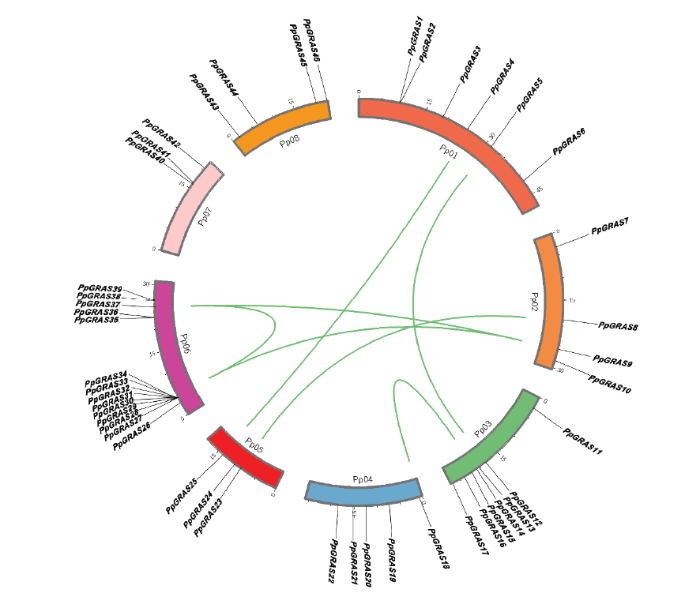 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1桃GRAS家族基因染色体定位及其共线性预测
Fig. 1Chromosome localization and collinear prediction of GRAS family genes in peach
2.2 桃GRAS蛋白序列与拟南芥系统进化树的对比分析
通过与拟南芥GRAS蛋白聚类分析,将桃中48个GRAS蛋白分为9个亚家族(图2),这些亚家族是根据早期研究分类[15]或他们中的一名成员在新发现的亚家族中命名,分别为PAT1、SHR、HAM、LISCL、SCR、LS、DELLA、SCL3、U。每个亚家族的GRAS成员可能具有相似或相关的功能。一般而言,大多数亚家族都拥有来自2个物种的GRAS成员(图2)。然而,在桃中新鉴定出来的U亚家族不包括任何拟南芥成员,表明拟南芥中谱系特异性基因缺失。LISCL是最大的子家族,包含12个成员;而在拟南芥中LISCL也是最大的子家族,包含7个成员;其次是SCL3亚家族,有9个成员。PpGRAS家族成员共形成8个旁系同源基因对,其中3对的自展值为100,而且Prupe.6G073900.1.p/Prupe.6G074000.1.p的自展值为99,U家族仅有的两个成员Prupe.3G309800.1.p/Prupe. 8G054000.1.p的自展值达87。此外,PpGRAS与AtGRAS形成了14个直系同源基因对,其中12对的自展值为100(图2)。图2
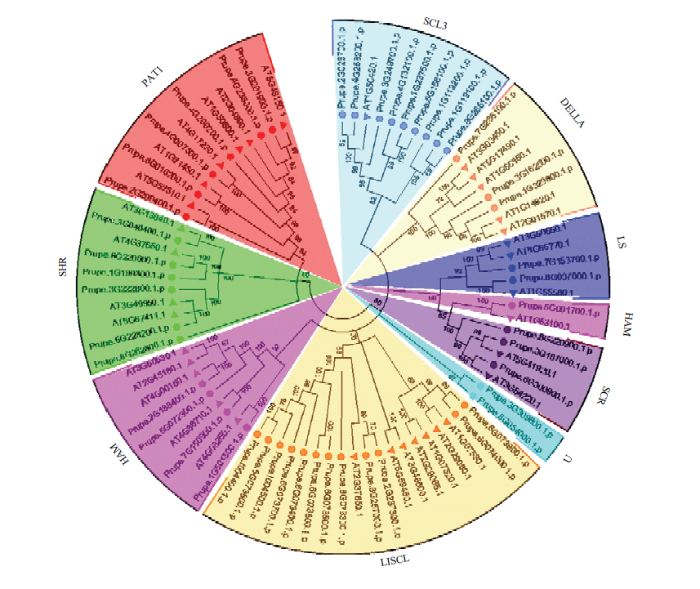 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2桃与拟南芥GRAS蛋白序列系统进化树比对
Fig. 2Phylogenetic tree comparison of peach and Arabidopsis GRAS protein sequences
2.3 桃GRAS家族保守元件和基因结构分析
基因结构多样性作为进化的可能基础,可以为多基因家族的进化提供有价值的信息[15,28]。基于每个预测基因的编码序列(CDS)和基因组DNA序列分析基因结构(图3)。桃中GRAS家族基因的内含子数量最多为1,其中40个基因没有内含子,其余8个基因(Prupe.2G138400.1.p、Prupe.8G220900.1.p、Prupe. 6G300900.1.p、Prupe.6G073900.1.p、Prupe. 3G040400.1.p、Prupe.8G016200.1.p、Prupe.4G233000.1.p、Prupe. 3G309800.1.p)仅有1个内含子。通常,来自桃中相同亚家族(SCL3、DELLA和LS)的GRAS具有相同的结构。图3
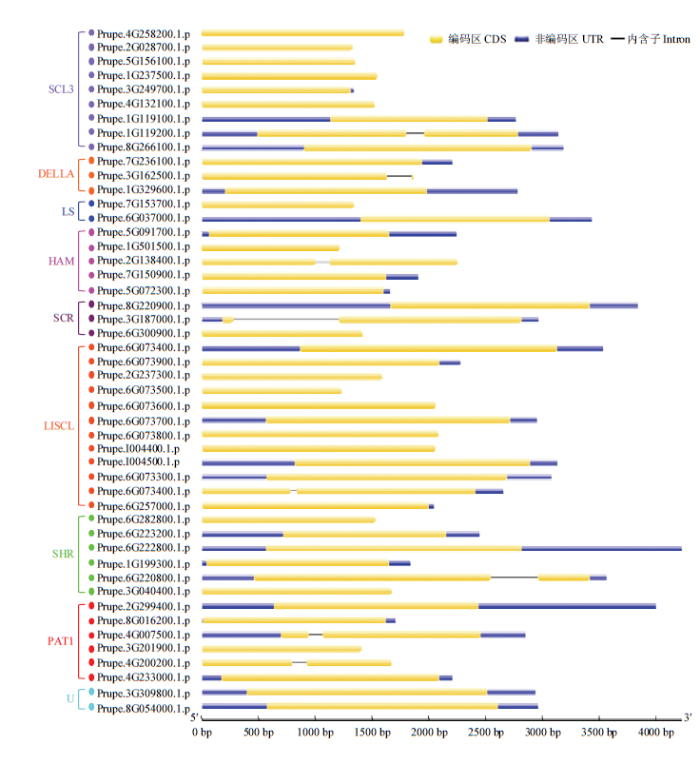 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3桃GRAS结构
Fig. 3Structures of GRAS genes in peach
为揭示桃中GRAS蛋白质的多样性,进一步确定桃GRAS家族成员间的关系,采用MEME预测桃GRAS家族基因中的元件。从候选的桃GRAS蛋白质中共鉴定出20个保守元件(图4),并列出了这些保守元件的结构域序列标识(即氨基酸组成)(图5)。本研究发现,所有的桃GRAS蛋白都含有Motif 4,表明其在GRAS的家族中高度保守;此外,高度保守的Motif 2同样存在于桃所有GRAS蛋白中。其中motif 2、11在SCL3亚家族成员的N端高度保守,而DELLA亚家族的3个成员的保守基序表现出较好的一致性,共有10个保守元件(motif 2、3、4、7、9、15、17、18、19和20),LS亚家族共有8个保守元件(motif 2、3、4、7、11、15、17和19),HAM亚家族中有5个共有保守元件(motif 2、4、5、9和19),SCR亚家族中共有7个保守元件(motif 2、3、4、5、7、9和17),LISCL家族中具有特异性保守元件Motif 13和在其N端高度保守的Motif 16,SHR亚家族中仅有3个共有保守元件(motif 4、5和17),PAT1亚家族中共有9个保守元件(motif 2、3、4、5、6、7、9、17和19),U亚家族共有7个保守元件(motif 2、3、4、5、9、17和19)。由此发现系统发育树中密切相关的GRAS含有相同的保守元件,而且有的亚家族中具备专有的保守元件,表明同一个亚家族的基因具备相似的功能。从桃GRAS的基因结构和保守元件位置分析,确定大多数成员在各个亚家族中是保守的。
图4
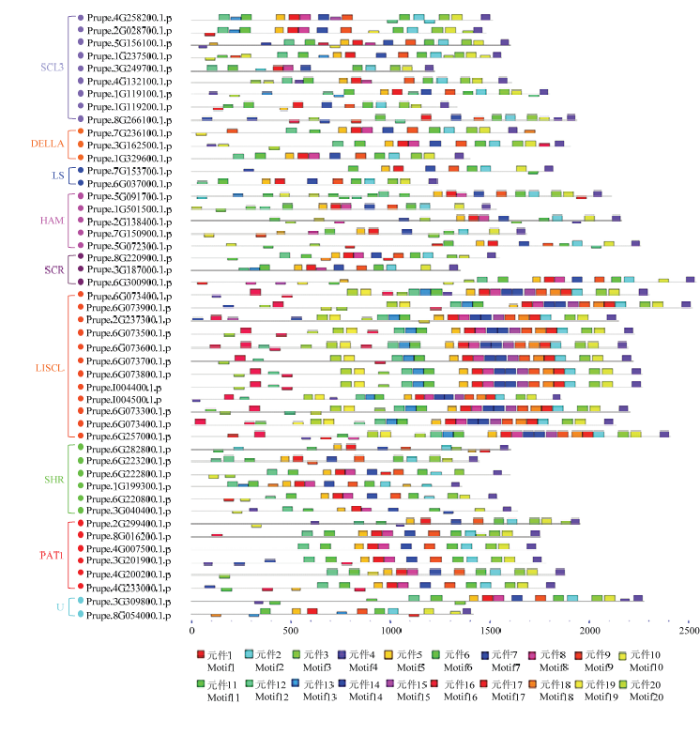 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4桃GRAS基因家族保守元件
Fig. 4Conserved motif of GRAS gene family in peach
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5桃GRAS结构域的序列标识
Fig. 5Sequence logos of GRAS domains in peach
2.4 桃GRAS家族基因的组织特异性表达分析及其对UV-B的响应
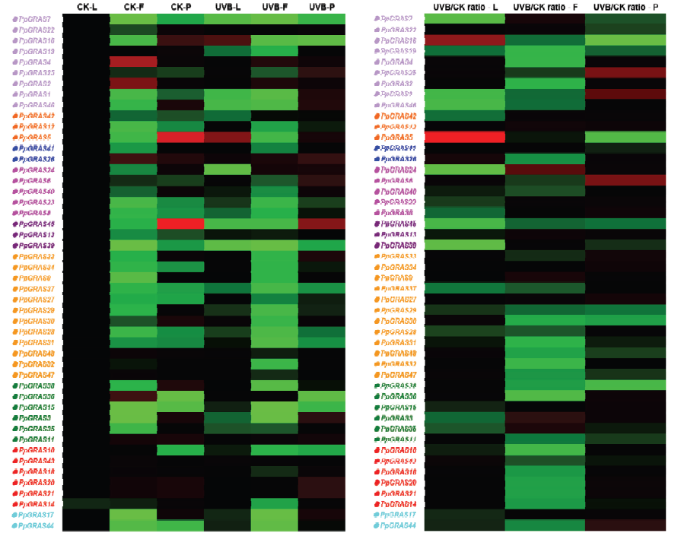
GRAS家族基因在叶片、果实和韧皮部中的组织特异性分析如图6-A所示。48个PpGRAS在叶片、果实和韧皮部3种组织中均有表达,表达模式存在一定的差异。33.33%的基因(16个)在叶片中的表达量高于其余两种组织。PpGRAS5、PpGRAS16和PpGRAS45在桃韧皮部中高度表达;PpGRAS2、PpGRAS4、PpGRAS26和PpGRAS36在果实中的表达最高。UV-B处理后48个基因在3个组织中也均有表达,其表达模式与CK不尽相同。其中,PpGRAS5和PpGRAS16在叶片中的表达量高于其他组织;PpGRAS6、PpGRAS26和PpGRAS45在桃韧皮部中高表达;而有25个基因在果实中的表达量低于其他组织。并且,有8个基因(PpGRAS2、PpGRAS4、PpGRAS11、PpGRAS13、PpGRAS22、PpGRAS43、PpGRAS47和PpGRAS48)在3个组织中的表达水平基本一致。图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6GRAS家族基因在桃中的组织特异性表达(A)及UV-B处理的基因表达量与CK基因表达量的比值(B)
L:叶片 Leaf;F:果实 Fruit;P:韧皮部 Phloem
Fig. 6Tissue expression pattern of the PpGRAS genes in peach (A); the ratio of the gene expression level of UV-B treatment to the gene expression level of CK (B)
响应UV-B信号的PpGRAS在不同组织内的表达量与CK的比值见图6-B。在叶片中,PpGRAS5和PpGRAS16在UV-B处理后上调表达,而31.25%的基因(15个)表达量下调。在果实中,PpGRAS13经UV-B处理后上调表达,而有9个基因(PpGRAS2、PpGRAS4、PpGRAS10、PpGRAS14、PpGRAS19、PpGRAS24、PpGRAS26、PpGRAS32和PpGRAS42)下调表达。在韧皮部中,UV-B处理后有29.17%的基因(14个)上调表达,而PpGRAS5和PpGRAS16经处理后表达下调。另外,有7个基因(PpGRAS11、PpGRAS15、PpGRAS17、PpGRAS41、PpGRAS43、PpGRAS47和PpGRAS48)经UV-B处理后在各个组织内均未表现出明显变化。
3 讨论
3.1 PpGRAS转录因子家族的鉴定和分析
GRAS是涉及植物生长和发育等各方面重要的转录因子。GRAS基因家族在植物中广泛分布,且家族成员的数量与基因组的大小无关[21]。植物体在生物进化过程中主要通过基因组的广泛复制和多样化,产生基因家族中的多成员[29,30]。在拟南芥中,GRAS家族发生了2次串联重复事件[2]。而且,GRAS基因家族的串联重复现象在番茄、水稻、白杨和辣椒等物种中均被检测到[2,21,31]。本研究鉴定出48个PpGRAS基因家族成员,低于水稻(57个)[15]、辣椒(58个)[21]和番茄(53个)[32]等作物,可能是由于桃基因组中没有大规模的片段复制,且相对保守等原因造成的。通过染色体定位分析发现,PpGRAS在桃基因组中广泛分布,且存在明显的串联重复现象,串联重复基因占20.833%(10/48),这表明串联重复在PpGRAS基因家族扩增和进化中起着重要作用。研究发现,水稻中的一些GRAS蛋白质并未找到合适的亚家族[2],本研究发现,在桃GRAS家族中同样有两个基因(PpGRAS17、PpGRAS44)在系统进化树分析中不属于已知的8个亚家族,而且其可靠性达87,将其划分为U亚家族,因而将桃中GRAS共划分为9个亚家族[15]。该U亚家族成员基因的表达有所差异,其中PpGRAS17在果实中表达较低,而PpGRAS44在果实和韧皮部中表达都较低;UV-B处理后PpGRAS17的表达基本不受影响,但PpGRAS44的表达在韧皮部中上调。而DELLA、LISCL和PAT1亚家族的基因结构具有明显的一致性,表明这些亚家族的基因具备更为相似的功能。而且还发现LISCL和PAT1亚家族的基因表达模式同样具有明显的特异性,主要在叶中表达。但是有些亚家族内基因的表达模式有所不同,可能参与了不同的生长发育过程。推测同一亚家族之间不同GRAS的表达差异可能与保守基序外的序列有关。此外,基因表达模式的研究可为PpGRAS家族基因的功能验证提供依据。GRAS基因家族的系统发育分析为进一步研究桃的功能基因组提供了理论基础。
3.2 PpGRAS响应UV-B处理的表达分析
GRAS蛋白在多种植物中被发现,功能呈多样性,并在不同品种、不同组织和不同生长阶段所发挥的作用不尽相同[2,32]。目前,模式植物拟南芥的GRAS基因家族中的多个亚家族成员的功能已被验证[15]。本研究发现,设施桃叶片、果实和韧皮部3个组织中均有多个GRAS响应UV-B处理,响应情况不尽相同。3.2.1 桃叶片中PpGRAS响应UV-B光信号的表达分析
DELLA蛋白作为负调控因子参与了GA信号的转导过程。当DELLA蛋白接收GA信号时,会迅速降解,植株表现正常的GA响应程序[23],而DELLA突变体植株却表现出GA不敏感表型[33]。本研究发现,PpGRAS5作为DELLA亚家族成员中AtRGL同源基因,在UV-B处理后,PpGRAS5在叶片中上调表达最为显著。有研究表明,在桃上进行UV-B处理后造成了叶片中内源赤霉素含量降低[34]。由此推测,UV-B处理后可能导致叶片中内源赤霉素含量降低,又经由GA信号调控了PpGRAS5,从而使其上调表达。
矮牵牛中PhHAM主要在侧生器官原基和茎原维管组织中表达,对维持茎尖分生组织活性发挥了重要作用。矮牵牛ham突变体植株的叶片数明显减少,茎尖分生组织丧失分化特性,阻止了器官的形成[35]。而本研究发现,桃HAM亚家族成员PpGRAS24经UV-B处理后在叶片中表达下调最为明显,推测其可能响应UV-B处理后对叶片的叶原基分化起着重要作用,具体功能尚需进一步验证。
3.2.2 桃果实中PpGRAS响应UV-B光信号的表达分析
远红光、红光和白光配合处理都降低了SCL21的转录水平,而且与SCL21相互作用的PAT1蛋白主要参与了phy A介导的光信号转导过程,在光信号转导中起着重要作用[22]。本研究发现,PpGRAS10作为PAT1亚家族中AtSCL8的同源基因,经UV-B处理后在果实中显著下调表达,表明PpGRAS基因家族成员在响应UV-B光信号中也可能发挥了重要作用。
PpGRAS13作为桃SCR亚家族的成员,在桃果实中经UV-B处理后上调表达最为显著。SHR和SCR蛋白的作用会导致细胞分裂过程的缺陷[36],相似结果也在水稻[37]和玉米[38]中发现;而且OsSCR不仅参与了根的发育调控,同时也参与了叶片保卫细胞的形成[39]。陈丽华等[40]研究发现,经GA处理后的‘无核白鸡心’葡萄的果形明显拉长,而且SCR在GA处理后呈现整体性的上调表达,SCR可能在响应GA处理后调控了葡萄果实细胞的分裂和分化。因而,推测PpGRAS13可能具有响应UV-B光信号调控桃果实纵、横径的功能。
3.2.3 桃韧皮部中PpGRAS响应UV-B光信号的表达分析
在拟南芥根部,SCL3能作为GAI和RGA的衰减子调控伸长区和分化区细胞的伸长,SCL3-DELLA互作调节的GA信号途径,能与SHR/SCR途径联合调控分裂区基本组织的分裂时间和程度[23]。相似的结论同样出现在毛竹中[39]。本研究发现,SCL3亚家族成员PpGRAS16在韧皮部中经UV-B处理后下调表达最为明显,推测PpGRAS16可能负响应UV-B光信号,减弱对桃韧皮部细胞分裂程度的抑制。
果实发育过程中,如果基因在幼果期的表达水平高于成熟期,则其可能在果实前期发育过程中发挥重要功能。黄伟[32]研究发现,番茄中Soly11g005610.1.1的表达在果实转色期有显著上升,其可能对启动果实成熟具有重要作用。而本研究发现,PpGRAS6作为Soly11g005610.1.1的同源基因,经UV-B处理后在韧皮部中上调表达最为明显,说明其可能在“源-库”间有机物的运输中起着重要作用。
4 结论
通过生物信息学分析预测出48个桃GRAS基因家族成员,分为9个亚家族,基因结构、染色体分布和保守元件分布等多种生信分析表明这些基因在进化过程中比较保守。采用qRT-PCR技术对桃GRAS基因家族成员进行组织特异性分析,发现48个基因在叶片、果实和韧皮部中均有表达,但表达变化不尽相同。本研究还探索了PpGRAS基因家族成员对UV-B的响应模式,发现多数PpGRAS能响应UV-B处理,表明PpGRAS基因家族可能在光响应方面发挥着多种潜在功能。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 4]
[本文引用: 4]
[本文引用: 5]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 7]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 4]
[本文引用: 2]
[本文引用: 2]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 3]
[D].
[本文引用: 3]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
