 ,, 闫立英
,, 闫立英 ,河北科技师范学院,河北秦皇岛 066004
,河北科技师范学院,河北秦皇岛 066004Inheritance and QTL Mapping for Parthenocarpy in Cucumber
NIU ZhiHong, SONG XiaoFei, LI XiaoLi, GUO XiaoYu, HE ShuQiang, HE LuanJingZhi, FENG ZhiHong, SUN ChengZhen ,, YAN LiYing
,, YAN LiYing ,Hebei Normal University of Science and Technology, Qinhuangdao 066004, Hebei
,Hebei Normal University of Science and Technology, Qinhuangdao 066004, Hebei通讯作者:
责任编辑: 赵伶俐
收稿日期:2019-01-21接受日期:2019-06-5网络出版日期:2020-01-01
| 基金资助: |
Received:2019-01-21Accepted:2019-06-5Online:2020-01-01
作者简介 About authors
牛志红,Tel:18332567462;E-mail:zhihongn@163.com。
宋晓飞,Tel:13613358355;E-mail:songxiaofei1979@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (5044KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
牛志红, 宋晓飞, 李晓丽, 郭晓雨, 何书强, 贺栾劲芝, 冯志红, 孙成振, 闫立英. 黄瓜单性结实性状遗传与QTL定位[J]. 中国农业科学, 2020, 53(1): 160-171 doi:10.3864/j.issn.0578-1752.2020.01.015
NIU ZhiHong, SONG XiaoFei, LI XiaoLi, GUO XiaoYu, HE ShuQiang, HE LuanJingZhi, FENG ZhiHong, SUN ChengZhen, YAN LiYing.
0 引言
【研究意义】黄瓜(Cucumis sativus L.)属于葫芦科甜瓜属一年生草本植物,是设施栽培高产高效的主要蔬菜作物之一。单性结实是指子房不经过授粉受精而发育成果实的现象[1],单性结实性状是设施黄瓜绿色标准化生产中直接与产量和品质密切相关的重要性状,强单性结实黄瓜品种在设施栽培的无虫媒条件下,子房无需植物生长调节剂等处理就能够正常膨大形成无籽果实,从而实现优质丰产节本。因此,深入解析黄瓜单性结实性遗传分析和QTL定位,对提高设施专用黄瓜品种育种效率具有重要意义。【前人研究进展】国内外****对单性结实性的遗传规律研究较多,但由于生长条件、试验设计及所选用材料的不同而研究结果不同。PIKE等[2]和DE PONTI等[3]认为单性结实性状属于质量遗传,但目前大多数****[4,5,6,7,8,9,10,11,12]认为黄瓜单性结实性具有数量性状的遗传特征,SUN等[7,8]认为黄瓜单性结实性由至少2个基因控制的数量性状;闫立英等[9,10,11,12]利用主基因+多基因混合遗传模型分析法研究表明黄瓜单性结实性是1—2对主效基因控制的数量性状。陈学好等[13]筛选出与黄瓜非单性结实基因连锁的ISSR分子标记,遗传连锁距离为18.9 cM。闫立英[14]发现1个与黄瓜非单性结实性紧密连锁的AFLP分子标记,遗传距离为9.7 cM。SUN等[15]采用区间作图和复合区间作图的方法,利用ALFP、SCAR等多种标记对美国加工型全雌黄瓜单性结实性进行图谱构建和QTL分析,两个种植地点共检测到10个QTL,主要分布在3个连锁群上。CALVIN等[16]对美国加工黄瓜进行单性结实QTL分析,共检测出7个位点,分布于2、4、5、6、7号染色体上,其中parth5.1、parth6.1和parth7.1三个位点与强单性结实品种有关。武喆等[17]发现7个与单性结实有关的QTL位点,分布于1、2、3、5、7染色体上,推测基因Csa2M035330.1和Csa2M070880.1与单性结实相关性更大。LI等[18]通过RNA-Seq检测出14个与单性结实相关的基因,涉及到生长素、细胞分裂素和赤霉素等信号传导及其交互作用。张婷等[19,20]发现12个与单性结实相关的候选基因,但都定位于2号染色体,推测Cs5NG4(Csa2M042770.1)是单性结实的主要候选基因。【本研究切入点】目前黄瓜单性结实QTL定位作图群体多为暂时性群体,无法验证QTL的稳定性,研究结果不一,而与单性结实性相关的基因较少。【拟解决的关键问题】本研究基于黄瓜强单性结实自交系‘6457’和弱单性结实自交系‘6426’构建的重组自交系(RIL)F2:8,利用SSR标记构建高质量分子标记连锁图谱,初步定位黄瓜单性结实性状关联位点,并结合QTL-Seq数据分析筛选出与黄瓜单性结实有关的关键位点和基因,为分子标记辅助育种提供有价值的参考。1 材料与方法
1.1 材料
以本校黄瓜课题组自主选育的雌雄同株类型的强单性结实自交系‘6457’为母本(P1),与多代选育的雌雄同株的弱单性结实自交系‘6426’(P2)配制F1,采用单子传代法构建了116个重组自交系(RIL)F2:8。1.2 表型鉴定方法
2016年春季、2017年春季、2018年春季,将所得116个RIL群体,每个株系定植10株,采用双高垄栽培,大行距80 cm,小行距50 cm,株距23 cm,田间常规管理。在植株开花结果期,参照李锡香等[21]、闫立英等[14]单性结实性状鉴定方法和标准进行单性结实表型鉴定。具体方法:每天下午逐株检查并对次日将开的雌花进行束花隔离,次日上午挂牌标记。注明雌花节位及开花日期,花后8—10 d调查坐果情况,计算单株单性结实坐果率(正常瓜数/标记雌花数×100%),用平均单系单性结实坐果率表示双亲和重组自交系单性结实性的强弱。1.3 遗传连锁图谱的构建
采用改良CTAB法[22]提取黄瓜基因组DNA。SSR引物序列来源于‘9930’和‘Gy14’黄瓜全基因组测序信息[23],从中选择均匀分布于7条染色体上的1 220对SSR标记,以亲本‘6457’和‘6426’为模板进行多态性筛选,获得多态性好、差异显著的SSR标记引物,对116个RIL群体进行PCR扩增,PCR产物用6%的聚丙烯酰胺凝胶电泳分离,采用银染法进行显色,条带统计,分别以1和0记录电泳谱带的有无,建立SSR数据库,与母本‘6457’带型一致的标记记为A,与父本‘6426’带型一致的标记记为B,模糊不清或者丢失的带记为‘-’,-1、-2、-3分别表示同一引物的不同多态性位点。利用SPSS进行数据处理,经χ2检测,在P≤0.05水平,去除偏分离标记引物,再利用JoinMap 4.1软件进行黄瓜连锁图谱的构建。1.4 不同年份黄瓜单性结实遗传性状分析和QTL定位
调查2016年春季、2017年春季以及2018年春季黄瓜单性结实性状表型数据,进行平均单系单性结实坐果率的次数分布分析。利用Map QTL 4.0软件,以区间作图法(IM)对黄瓜单性结实性状进行QTL分析。选用1 cM的步长,在α=0.01的水平上,利用Permutation检验法重复检验1 000次,将LOD阈值设定为2.5,确定QTL在染色体上的位置和数目。1.5 基于QTL-Seq的黄瓜单性结实相关基因定位
利用QTL-Seq技术[24]和原理,在RIL群体中挑选单性结实性状差异显著的强单性结实株系和弱单性结实株系各20株,分别构建强单性结实池(parthenocarpy,par)和弱单性结实池(non-parthenocarpy,np),利用Illumina HiSeqTM PE150测序仪对亲本池和混合池展开20×和10×覆盖度的全基因组重测序,获得大量的SNP标记。计算每个位点的SNP频率(SNP-index=该位点与参考序列不同的reads数/总reads数),计算△SNP-index,进行1 000次置换检验。选取95%的置信水平作为筛选的阈值,大于阈值的窗口作为候选区间。1.6 候选基因分析与预测
利用黄瓜基因组数据库和拟南芥数据库(https:// www.arabidopsis.org/)对定位到的黄瓜单性结实相关基因进行同源比对,分析各基因功能,筛选与黄瓜单性结实相关的参与激素调节、信号转导及功能代谢途径的基因。2 结果
2.1 黄瓜单性结实遗传分析
对2016年春季、2017年春季以及2018年春季的亲本及RIL群体单性结实性状表型鉴定结果进行相关分析,结果表明不同年份间相关系数均达极显著水平,2016年春季与2017年春季、2016年春季与2018年春季、2017年春季与2018年春季黄瓜重组自交系单性结实率相关系数分别为0.78**、0.77**、0.71**,均达极显著水平,说明不同年份间单性结实表型相对稳定。进一步进行次数分布分析(图1),2016年春季、2017年春季及2018年春季,‘6457’平均单系单性结实坐果率分别为97.95%、87.42%、77.15%,均表现为强单性结实,而‘6426’平均单系单性结实坐果率分别为17.91%、12.50%、11.88%,均表现为弱单性结实,双亲单性结实表型相对稳定且差异明显;RIL群体平均单系单性结实坐果率均为连续分布,符合数量遗传特征。图1
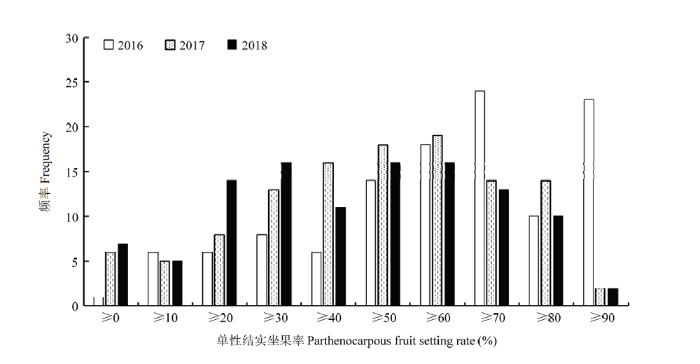 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图12016—2018年春季RIL群体平均单系单性结实坐果率次数分布图
Fig. 1Distribution of average parthenogenetic fruit setting percentage per line in the spring of 2016, 2017 and 2018
2.2 黄瓜连锁图谱的构建
以亲本‘6457’和‘6426’为模板,利用1 220对SSR引物进行筛选,共获得在双亲间存在多态性,条带清晰的引物组合169对,多态性为13.9%。169对多态性好的引物,经过χ2检测,在P≤0.05水平,87对标记表现出偏分离,去除偏分离标记引物,采用JoinMap 4.1软件,获得一张包含11个连锁群的遗传图谱,包含82个SSR标记,该图谱覆盖基因组555.0 cM,平均图距为6.8 cM。每个连锁群上标记数在2—23个,长度在3.3—120.4 cM。最大的连锁群含有23个标记,覆盖基因组120.4 cM,平均图距5.2 cM,最小的连锁群含有2个标记,覆盖基因组3.3 cM(图2)。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2黄瓜(‘6457’ב6426’)SSR遗传连锁图谱及单性结实性状QTL定位
Fig. 2SSR genetic linkage group and QTL mapping of parthenocarpy in the cucumber (‘6457’ב6426’)
2.3 不同年份黄瓜单性结实QTL定位
基于上述图谱,对2016年春季、2017年春季以及2018年春季3年数据检测,均得到1个与黄瓜单性结实性相关的QTL位点,位于3号染色体上的标记SSR19430和SSR15419之间,遗传距离6.6 cM。2016年春季的QTL对应的LOD值为2.95,贡献率为11%(图3-A);2017年春季的QTL对应的LOD值3.33,贡献率为12.5%(图3-B);2018年春季的QTL对应的LOD值1.54(超过单连锁群阈值),贡献率为6.3%(图3-C)。3年数据重复性好,QTL定位结果一致。图3
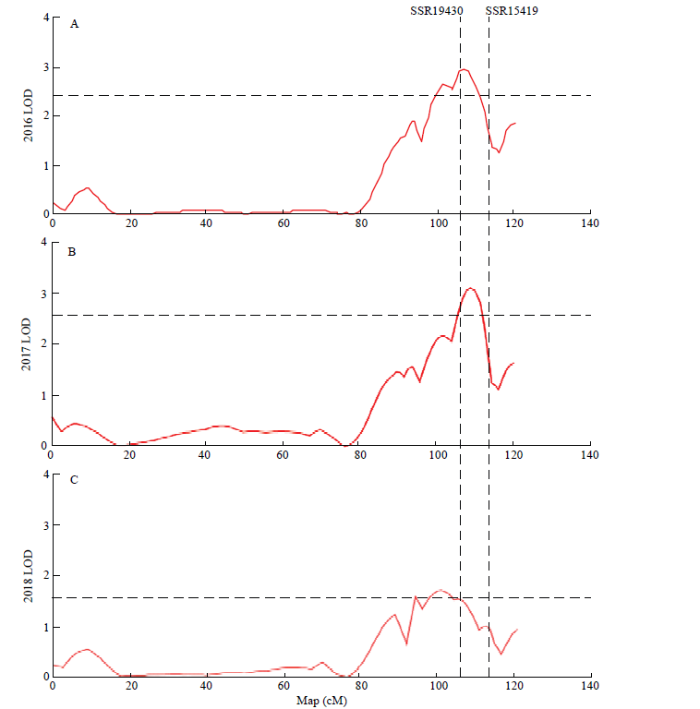 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图32016年春季(A)、2017年春季(B)以及2018年春季(C)黄瓜(‘6457’ב6426’)单性结实性状第3号染色体上QTL定位结果
Fig. 3QTL mapping on chromosome 3 of parthenocarpy in the cucumber (‘6457’ב6426’) on the spring of 2016 (A) , 2017 (B) and 2018 (C)
2.4 基于QTL-Seq技术的黄瓜单性结实QTL定位
2.4.1 混池测序数据质量分析对亲本P1、P2及20株强单性结实株系组成的混池(par)和20株弱单性结实株系组成的混池(np)开展全基因组重测序,共得到24.945 G原始数据,过滤后4个样本有效数据量在4 004.935—7 672.465 M,总数据量23.567 G。利用测序结果的QPhred数值(Phred Score,Q),计算碱基错误率,发现Q20≥93.99%、Q30≥87.01%,碱基错误率低于0.03%,测序质量高,GC含量在38.80%—39.84%之间(表1)。以黄瓜基因组作为参考基因组,参考基因组大小为197 271 687 bp,所有样本的比对率在87.06%—88.14%,对参考基因组(排除N区)的平均覆盖深度在12.40—21.61 X,1X覆盖度(至少有一个碱基的覆盖)在97.21%以上(表2)。
Table 1
表1
表1黄瓜(‘6457’ב6426’)单性结实性状QTL-Seq测序数据质量分析
Table 1
| 样品 Sample | 原始数据 Raw base (bp) | 有效数据 Clean base (bp) | 有效率 Effective rate (%) | 碱基错误率 Error rate (%) | Qphred值 Q20 (%) | Qphred值 Q30 (%) | GC含量 GC content (%) |
|---|---|---|---|---|---|---|---|
| P1 | 5376170100 | 5069987400 | 94.30 | 0.03 | 96.68 | 92.68 | 38.92 |
| np | 8447689500 | 7672464900 | 88.11 | 0.04 | 93.99 | 87.01 | 39.84 |
| P2 | 4140457200 | 4004934600 | 96.73 | 0.03 | 96.88 | 93.12 | 39.20 |
| par | 6980472300 | 6819972600 | 97.70 | 0.03 | 95.03 | 89.38 | 38.80 |
新窗口打开|下载CSV
Table 2
表2
表2黄瓜(‘6457’ב6426’)单性结实性状QTL-Seq测序深度及覆盖度分析
Table 2
| 样品类型 Sample | Reads比对数 Mapped reads | Reads总数 Total reads | 比对率 Mapping rate (%) | 样品平均覆盖深度 Average depth (X) | 1X数据覆盖比率 Coverage at least 1X (%) | 4X数据覆盖比率 Coverage at least 4X (%) |
|---|---|---|---|---|---|---|
| P1 | 29754479 | 33799916 | 88.03 | 15.27 | 98.12 | 92.06 |
| P2 | 23533219 | 26699564 | 88.14 | 12.40 | 97.21 | 86.53 |
| par | 39918316 | 45466484 | 87.80 | 20.24 | 98.65 | 95.70 |
| np | 44530059 | 51149766 | 87.06 | 21.61 | 98.70 | 95.57 |
新窗口打开|下载CSV
2.4.2 黄瓜单性结实QTL定位
以亲本P1作为参考亲本,分析计算两个子代在亲本间163 926个标记位点的SNP-index(即SNP的频率)。根据强单性结实池与弱单性结实池的SNP-index,计算两个池的SNP-index的差值,以染色体位置为横坐标,以1 Mb为单位窗口,1 kb为步长,绘制par池SNP-index(图4-A)、np池SNP-index(图4-B)和△SNP-index图(图4-C)。根据混池之间基因频率差异筛选,对强单性结实(par)与非单性结实池(np)间SNP和Indel统计显示:与参考基因组相比,两个子代池共有287 805个位点发生SNP突变,在外显子区域发现同义突变10 654个,非同义突变9 390个,缺失型终止密码子变异50个,获得性终止密码子变异162个。
图4
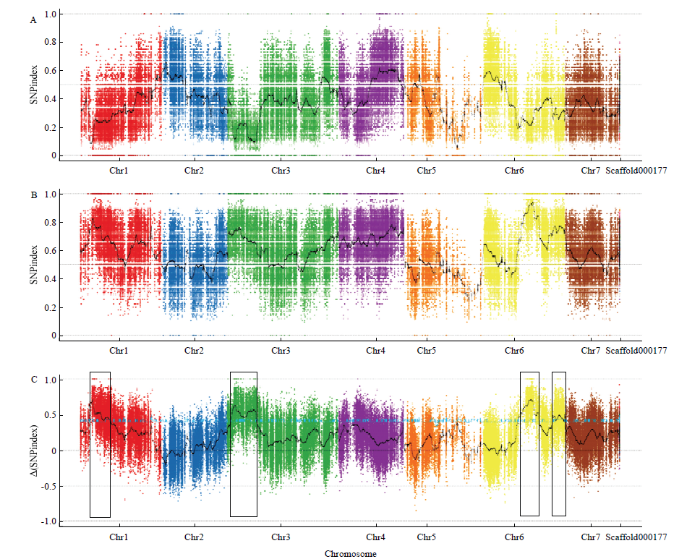 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4黄瓜(‘6457’ב6426’)单性结实SNP-index在染色体上的分布
A:par混池SNP-index在染色体上的分布;B:np混池SNP-index在染色体上的分布;C:△SNP-index在染色体上的分布,显著QTL在图中以空心框标注
Fig. 4Distribution of SNP-index on chromosomes for parthenocarpy in cucumber (‘6457’× ‘6426’)
A: The distribution of SNP-index on chromosomes parthenocarpy pools; B: The distribution of SNP-index on chromosomes in non-parthenocarpy pools; C: The distribution of Delta SNP-index on chromosomes, significant QTLs are marked with open boxes
在95%的置信水平下,以△SNP-index大于阈值的窗口作为候选区,共检测到4个QTL(图4),分别位于1号染色体(4.38—11.00 Mb)、3号染色(2.24—10.66 Mb)和6号染色体(15.67—17.93 Mb;26.33—27.49 Mb),其中3号染色体QTL-Seq检测到的QTL与前述Map QTL检测到的2016—2018年度间稳定的QTL基本重合,QTL-Seq结果包含Map QTL检测结果(图5)。
图5
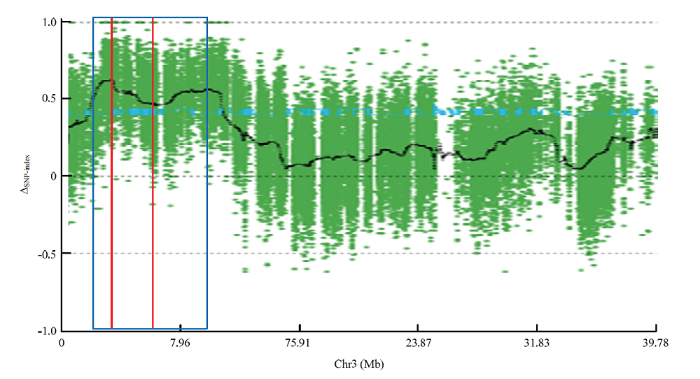 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5黄瓜(‘6457’ב6426’ )单性结实性状3号染色体上Map QTL(红框)与QTL-Seq (蓝框)位置比较
Fig. 5Overlaping of Map QTL (red box) and QTL-Seq (blue box) on chromosome 3 for parthenocarpy in cucumber (‘6457’× ‘6426’)
2.5 候选基因分析与预测
根据黄瓜基因组,上述4个QTL区间内共获得候选多态性标记位点129个,提取ANNOVAR的注释结果,优先挑选引起stop loss或者stop gain或非同义突变或可变剪接的位点所在的基因作为候选基因,共获得与黄瓜单性结实性状相关的候选基因19个,含有27个SNP位点(表3)。Table 3
表3
表3黄瓜单性结实性状相关候选基因和功能预测
Table 3
| 基因号 Gene ID | 变异类型 Variant | 位置 Position | 参考基因组基因型/该位点其他基因型Ref/Alt | 拟南芥库里功能注释 Functional annotation of Arabidopsis |
|---|---|---|---|---|
| Csa1G042220 | Nonsynonymous | 4159708 | T/C | 编码ASPG1,与抗旱相关Encodes ASPG1 (ASPARTIC PROTEASE IN GUARD CELL 1). Functions in drought avoidance through abscisic acid (ABA) signalling in guard cells |
| Csa1G049980 | Nonsynonymous | 5680530 | A/G | 肽酶C78,泛素折叠修饰特异性肽酶1/2 Peptidase C78, ubiquitin fold modifier-specific peptidase 1/2 |
| Csa6G367130 | Nonsynonymous | 16841983 | C/G | 靶圈套卷曲螺旋结构域蛋白 Target SNARE coiled-coil domain protein |
| Csa1G042830 | Upstream | 4532431 | A/G | 钙依赖性脂质结合(CALB结构域)家族蛋白 Calcium-dependent lipid-binding (CaLB domain) family protein |
| Csa1G045620 | Upstream | 5048161 | T/C | 编码钙依赖性蛋白激酶 Target SNARE coiled-coil domain protein |
| Csa1G045620 | Upstream | 5048182 | T/C | 编码钙依赖性蛋白激酶 Target SNARE coiled-coil domain protein |
| Csa1G045620 | Upstream | 5048193 | G/A | 编码钙依赖性蛋白激酶 Target SNARE coiled-coil domain protein |
| Csa1G045620 | Upstream | 5048246 | G/A | 编码钙依赖性蛋白激酶 Target SNARE coiled-coil domain protein |
| Csa3G002320 | Upstream | 239366 | G/C | RB1诱导的卷曲螺旋蛋白 RB1-inducible coiled-coil protein |
| Csa3G036410 | Upstream | 2537701 | C/T | 富含亮氨酸的重复蛋白激酶家族蛋白 Leucine-rich repeat protein kinase family protein |
| Csa3G043910 | Upstream | 3022472 | A/G | GRAS家族转录因子 GRAS family transcription factor |
| Csa3G043910 | Upstream | 3022485 | T/A | GRAS家族转录因子 GRAS family transcription factor |
| Csa3G045170 | Upstream | 3188752 | T/C | 跨膜蛋白 Transmembrane protein |
| Csa3G047740 | Upstream | 3294577 | C/T | 咖啡酸/5-羟阿魏酸O-甲基转移酶 A caffeic acid/5-hydroxyferulic acid O-methyltransferase |
| Csa3G047740 | Upstream | 3294578 | G/A | 咖啡酸/5-羟阿魏酸O-甲基转移酶 A caffeic acid/5-hydroxyferulic acid O-methyltransferase |
| Csa3G073810 | Upstream | 3855792 | G/A | 编码参与介导植物对发病机制的反应的蛋白质 Encodes a protein involved in mediating plant responses to pathogenesis |
| Csa3G144130 | Upstream | 9538626 | G/A | 编码与CBL相互作用蛋白激酶SNF1和SOS2具有相似性的丝氨酸/苏氨酸蛋白激酶 Encodes a serine/threonine protein kinase with similarities to CBL-interacting protein kinases, SNF1 and SOS2 |
| Csa3G144130 | Upstream | 9538714 | G/A | 编码与CBL相互作用蛋白激酶SNF1和SOS2具有相似性的丝氨酸/苏氨酸蛋白激酶 Encodes a serine/threonine protein kinase with similarities to CBL-interacting protein kinases, SNF1 and SOS2 |
| Csa6G324800,Csa6G324810 | Upstream | 15102611 | C/G | 编码与NAD相关的苹果酸脱氢酶活性的蛋白 Encodes a protein with NAD-dependent malate dehydrogenase activity, located in chloroplasts |
| Csa6G344310 | Upstream | 15681038 | A/G | 腺苷酸核苷酸水解酶类似结构域蛋白的激酶 Kinase with adenine nucleotide alpha hydrolases-like domain-containing protein |
| Csa6G344310 | Upstream | 15681043 | T/C | 腺苷酸核苷酸水解酶类似结构域蛋白的激酶 Kinase with adenine nucleotide alpha hydrolases-like domain-containing protein |
| Csa6G362930 | Upstream | 16350065 | A/C | 亚精胺合成酶 Encodes a spermine synthase |
| Csa6G375720 | Upstream | 16932936 | T/C | 抗病蛋白(CC-NBS-LRR类)家族 Disease resistance protein (CC-NBS-LRR class) family |
| Csa6G375720 | Upstream | 16932970 | A/G | 抗病蛋白(CC-NBS-LRR类)家族 Disease resistance protein (CC-NBS-LRR class) family |
| Csa6G396650 | Upstream | 17566966 | C/T | 编码一种调节细菌肽聚糖传感和对细菌感染免疫的赖氨酸基序蛋白 Encodes a lysin-motif protein mediating bacterial peptidoglycan sensing and immunity to bacterial infection |
| Csa6G513690 | Upstream | 26648579 | A/C | 编码参与苯丙氨酸生物合成的质体局部芳香族脱水酶 Encodes a plastid-localized arogenate dehydratase involved in phenylalanine biosynthesis |
新窗口打开|下载CSV
将单性结实QTL-Seq分析所得候选基因,利用BLAST软件将候选区域的19个基因与拟南芥库进行同源比对,发现19个候选基因编码区中有3个非同义突变,分别为Csa1G042220、Csa1G049980和Csa6G367130。
QTL-Seq与QTL均定位到了3号染色体,其候选基因的上游序列2 000 bp区间内检测到7个变异位点,分别位于:Csa3G002320(Unknown protein)、Csa3G036410(Receptor-like protein kinase,RLPK)、Csa3G043910(DELLA protein GAI,DPG)、Csa3G045170(Unknown protein)、Csa3G047740(O- Methyltransferase-like protein,OMLP)、Csa3G073810(Vacuolar protein sorting-associated protein VTA1-like protein,VTA1L)、Csa3G144130(CBL-interacting protein kinase 20,CBLIPK20)上游,编码蛋白激酶、跨膜蛋白和GRAS家族转录因子等(表3)。其中,Csa3G047740和Csa3G073810是3号染色体上年度可重复区间的两个基因,推测这两个基因是与单性结实性状相关的重要候选基因。Csa3G043910(DPG)与赤霉素介导的果实发育有关,因此,推测Csa3G043910(DPG)是黄瓜单性结实候选基因。
此外Csa6G362930基因编码亚精胺合成酶,而亚精胺是影响黄瓜单性结实和果实生长发育的重要多胺类激素,推测Csa6G362930也是与黄瓜单性结实性相关的候选基因。
3 讨论
3.1 关于单性结实的遗传分析
无论是前人采用经典遗传学研究方法[4,5,6,7,8],还是笔者课题组采用的主基因+多基因研究方法[9,10,11,12],研究均表明黄瓜单性结实性是由1—2对主效基因控制的数量性状。闫立英等[10,11]基于‘6457’ב6426’构建的多世代联合分析,发现分离世代均表现出明显的主基因+多基因的遗传特征。本试验对‘6457’ב6426’配组的RIL群体进行平均单系单性结实坐果率的次数分布分析,黄瓜单性结实性状符合数量性状遗传特征。3.2 关于单性结实QTL定位研究
研究认为[25,26],以初定位为目的,100—200个作图群体,每隔10 cM左右有一个标记就足够,若5—10 cM有一个标记,即使再增加标记也不会提高QTL检测功效。本试验采用116个RIL永久作图群体,构建了1张包含11个连锁群的遗传图谱,覆盖基因组555.0 cM,平均图距为6.8 cM,图谱已满足初定位要求。SUN等[15]采用区间作图和复合区间作图的方法,利用AFLP、SCAR和RAPD等标记对美国加工型全雌黄瓜单性结实性进行图谱构建和QTL分析,两个种植地点共检测到10个QTL,主要分布在3个连锁群上。与本研究相比,虽考虑到环境对单性结实的影响,但研究材料和标记手段不同,且SUN等[15]的研究中连锁群未能与黄瓜染色体对应,因此相比较下,本研究结果更有参考价值。武喆等[17]利用1 335对SSR引物和143对Indel引物对F2:3家系进行两季筛选,多态性为11.7%。本研究利用1 220对SSR引物对RIL群体进行两年筛选,多态性为13.9%。相同之处:分子标记多态性都偏低,这可能与两亲本都是栽培种有关;都考虑到环境对单性结实的影响,保证了试验结果的准确性。但试验材料和定位到单性结实位点不同,武喆等[17]所用为暂时性群体F2:3家系,并检测出7个与单性结实有关的QTL位点,分布于1、2、3、5、7号染色体上,认为Parth2-1是单性结实性主效QTL位点。本研究利用永久性作图群体RIL,且传代多年,定位到1、3、6号染色体上。虽与武喆等[17]定位到的主效QTL位点不同,但与其认为的Parth3-1(SSR17751-UW084149)和Parth3-2(SSR16667-UW085093)两个微效QTL单性结实位点相近,这也更加说明单性结实性状与3号染色体相关。此外,还定位到了6号染色体上的8个基因,为进一步定位黄瓜单性结实关键基因提供参考。
3.3 QTL-Seq与传统作图QTL定位结果的比较
本研究采用QTL-Seq方法检测到黄瓜单性结实性的4个QTL,分别位于1、3、6号染色体。采用Map QTL方法在3号染色体上(3.33—5.57 Mb)上检测到年度间稳定的QTL,且位于QTL-Seq在3号染色体上检测到(2.24—10.66 Mb)的QTL区间内,而Map QTL方法在1号和6号染色体上未能检测到QTL的原因可能是遗传连锁图谱标记密度较低所致[27,28,29]。3.4 关于单性结实候选基因预测
目前黄瓜单性结实基因研究较少,LI等[18]利用RNA-Seq发现14个与单性结实相关的基因,功能涉及生长素、细胞分裂素等。武喆等[17]推测出基因Csa2M035330.1和Csa2M070880.1与单性结实相关的可能性更大。张婷等[19,20]发现12个定位于2号染色体上的单性结实候选基因,推测Cs5NG4(Csa2M042770.1)是单性结实的重要候选基因。本研究利用Map QTL和QTL-Seq两种方法,将与单性结实性有关的QTL共同指向3号染色体,且QTL-Seq结果包含Map QTL结果,在QTL-Seq检测到的19个候选基因中,Csa3G047740和Csa3G073810位于Map QTL和QTL-Seq两种方法检测结果的重合区域,故推测这两个基因是与单性结实性状相关的主要关键基因。此外,3号染色体上的7个基因分别编码蛋白激酶、跨膜蛋白和GRAS家族转录因子等。其中DELLA蛋白属于GRAS家族,是GA信号传导途径的核心作用元件,且GA与植物花和果实发育有关[30,31,32],而Csa3G043910(DPG)是GAI的DELLA蛋白,因此推测其是单性结实的关键基因。
此外,多胺对番茄[33]、苹果[34,35,36]、柑橘[37]、李[38]等许多植物开花结果和果实发育具有明显的调节作用。亚精胺是影响黄瓜单性结实和果实生长发育的重要多胺类激素,陈学好等[39]和于杰等[40]发现未授粉子房施用外源亚精胺能显著提高子房内源亚精胺、精胺与腐胺水平,促进其单性结实和果实生长。黄瓜Csa6G362930编码亚精胺合成酶(亚精胺合成途径关键酶),虽然该基因不在3号染色体上,但推测其与黄瓜单性结实有关。与前人研究结果相比较,本研究推测到的与黄瓜单性结实相关的基因并无重合,为进一步解析黄瓜单性结实现象提供了参考。
4 结论
本研究以强单性结实自交系‘6457’与弱单性结实自交系‘6426’构建的RIL为试材,基于SSR标记构建出一张覆盖基因组555.0 cM,平均图距为6.8 cM的连锁图谱。传统QTL检测到年度间稳定遗传的1个QTL,位于3号染色体SSR19430—SSR15419标记之间,遗传距离6.6 cM。QTL-Seq发现4个与黄瓜单性结实性状相关的QTL,分别位于1、3、6号染色体上,QTL-Seq结果包含QTL初定位结果,推测Csa3G047740和Csa3G073810是与单性结实性状相关的重要关键基因;Csa3G043910和Csa6G362930也是与黄瓜单性结实性状相关的候选基因。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 3]
[本文引用: 3]
[本文引用: 3]
[本文引用: 3]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 4]
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 2]
[D].
[本文引用: 2]
[本文引用: 3]
[本文引用: 1]
[本文引用: 5]
[本文引用: 5]
[本文引用: 2]
[D].
[本文引用: 2]
[D].
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
