 ,1,2, 张成福3, 信金伟3
,1,2, 张成福3, 信金伟3Cloning and Identification of Long-Chain Non-Coding RNA Linc24063 and Its Correlation with the Expression Level of miRNAs in Yak
WANG Hui1,2, CHAI ZhiXin1,2, ZHU JiangJiang1,2, ZHONG JinCheng ,1,2, ZHANG ChengFu3, Xin JinWei3
,1,2, ZHANG ChengFu3, Xin JinWei3通讯作者:
责任编辑: 林鉴非
收稿日期:2018-11-5接受日期:2019-05-9网络出版日期:2019-07-16
| 基金资助: |
Received:2018-11-5Accepted:2019-05-9Online:2019-07-16
作者简介 About authors
王会,E-mail: wanghui892321@sina.cn。

摘要
关键词:
Abstract
Keywords:
PDF (984KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
王会, 柴志欣, 朱江江, 钟金城, 张成福, 信金伟. 牦牛Linc24063的克隆鉴定及其与miRNAs 表达水平的相关性分析[J]. 中国农业科学, 2019, 52(14): 2538-2547 doi:10.3864/j.issn.0578-1752.2019.14.012
WANG Hui, CHAI ZhiXin, ZHU JiangJiang, ZHONG JinCheng, ZHANG ChengFu, Xin JinWei.
0 引言
【研究意义】在机体内,lncRNA可通过miRNA发挥调控作用。一方面,lncRNA可作为miRNA吸附剂,与miRNA靶基因竞争性结合miRNA;另一方面,lncRNA可作为竞争性内源RNA(ceRNA),与具有相同miRNA结合元件的mRNA竞争miRNA,抑制翻译、调节可变剪切或调节mRNA降解[1]。牦牛主要分布于海拔3 000 m以上的青藏高原地区,为藏区人民提供肉、奶产品。牦牛奶具有高蛋白、高乳脂率的特点,研究lncRNA在牦牛乳腺组织中的具体功能及调控机制,对更好地利用牦牛遗传资源具有重要意义。【前人研究进展】在牦牛等哺乳动物中,由于早期配子遗传的精子和卵子的表观遗传修饰不同,导致接近1%的蛋白质编码基因表达具有亲本特异性,这种亲本依赖的表观遗传现象被定义为基因组印记[2,3]。迄今为止,在人类、小鼠、牛和绵羊等哺乳动物中分别鉴定出350个、236个、45个和18个印记基因(http://igc.otago.ac.nz),这些印记基因通常以簇的形式存在。Dlk1-Dio3印记域位于人染色体14q32、小鼠染色体12q、牛染色体 21q和绵羊染色体18q上,因其在胚胎发育[4]、癌症的发生[5]、细胞增殖分化[6]及机体各代谢调控通路[7]中发挥重要作用而备受关注,是目前研究较多的印记区域之一。Dlk1- Dio3印记域包括3个父源表达的编码蛋白基因(Dlk1、Dio3和 Rtl1/Peg11)与母系表达的长链非编码RNA(如lncRNA:Meg3、Meg8、Meg9)、miRNA和snoRNA[8]。研究表明,Dlk1-Dio3印记域在物种间高度保守,广泛参与到机体各生理及病理过程中。Dlk1-Dio3印记区域的激活与小鼠干细胞多能性水平相关[9];Dlk1参与调控多能干细胞[10]、肌细胞的增殖与分化[7];Dlk1启动子区域甲基化可增加非小细胞肺癌细胞的侵袭能力[11]。LncRNA是长度大于200 bp的一类非编码RNA,在胚胎发育[12]、细胞增殖分化[13]、精子发生[14]等多种生命过程中发挥重要的调控作用。Dlk1-Dio3印记域上有多个具备重要功能的lncRNA,如高脂饮食可增加小鼠Meg3表达量,Meg3通过调控FoxO1的表达增强肝脏胰岛素抵抗,干扰Met3可逆转小鼠由高脂饮食引起的血液甘油三酯含量的上调[14]。近年来,人们在不同物种Dlk1-Dio3印记域间发现并鉴定到了新lncRNA,如在鼠Meg8与Meg9、Meg9与Dio3之间分别发现并鉴定了2个lncRNA,即lncRNA B830012L 14Rik和AK044800,lncRNA B830012L 14Rik在小鼠大脑、肺脏、心脏和肝脏组织中广泛表达[15],而lncRNA AK044800主要在胚胎时期的前脑组织中表达[16];对牛Dlk1-Dio3印记域lncRNA鉴定发现,该区域包含Linc24061、Linc24063、 Linc24064三个lncRNAs,它们在在心脏、肾脏及肌肉组织中均广泛表达[17,18]。【本研究切入点】前人研究表明,Dlk1-Dio3印记域内的lncRNA,能参与机体多种生物学过程调控。但在牦牛上,未见此印记域内lncRNA的研究报道。【拟解决的关键问题】本研究旨在牦牛Dlk1- Dio3印记域Meg8与Meg9间鉴定牦牛Linc24063,通过生物信息学对其靶miRNA进行预测,结合组织表达谱分析其表达量在乳腺组织中与靶miRNAs的相关性,从而为牦牛Linc24063通过miRNA发挥功能的调控机制研究提供试验依据。1 材料与方法
1.1 试验材料
2018年4月,在西藏自治区昌都市类乌齐县,选取处于第一泌乳期、体重接近、健康的12头母牦牛,屠宰后采集卵巢、心脏、肝脏、肾脏、乳腺和大脑组织,DEPC反复冲洗后锡箔纸包装,迅速置于液氮保存,带回实验室备用。1.2 RNA提取及牦牛Linc24063扩增
各组织RNA提取参照Trizol(Invitrogen)试剂说明书。RNA经纯度和质量检测合格后备用。利用牛Dlk1-Dio3印记域Meg8与Meg9间的序列进行搜索并与牦牛基因组比对,鉴别出牛Linc24063(NCBI登录号:KU956000.1)序列与牦牛基因组序列高度保守,根据此序列利用Primer premier 5设计牦牛Linc24063基因5′ RACE(cDNA末端快速扩增)和3′ RACE引物。将牦牛各组织RNA等量混合后,利用SMARTer RACE cDNA Amplification Kit(Clontech, Palo Alto, CA, USA)按照操作说明书进行RACE试验,获取Linc24063的5′端和3′端。5′ RACE引物为:5′-TTAAACATTCCTAACATCTGCC TAC-3′,3′ RACE引物为5′-GGGACCCTGAGGCCCAGTCCATTC-3′。
1.3 牦牛Linc24063序列分析
利用NCBI BLAST对牦牛Linc24063序列在物种间的保守性及染色体上的定位进行分析;利用在线软件Coding Potential Calculator(http://cpc2.cbi.pku.edu.cn/)对Linc24063、已知lncRNA Meg9(登录号:NR_132275.2)和编码基因β-酪蛋白(CSN2,登录号:MH378280.1)的编码能力进行预测;利用miRanda和mireap软件分析与Linc24063相互作用的miRNAs;利用DAVID在线软件(https://david.ncifcrf.gov/)对与Linc24063相互作用的miRNAs的靶基因进行GO富集和KEGG信号通路分析。1.4 牦牛Linc24063原核表达
将牦牛Linc24063全长亚克隆至Pet-28a载体的EcoRⅠ和HindⅢ酶切位点上,获得Linc24063-28a原核表达质粒,转化至BL21感受态细胞,获得含重组质粒Linc24063-28a表达菌种,提取质粒用EcoRⅠ和HindⅢ酶切鉴定。CSN2-28a原核表达质粒为本实验室保存。Linc24063-28a和CSN2-28a诱导表达步骤参照文献[19]。取1 mL诱导后的菌液沉淀,用100 μL的1×SDS-PAGE Loading Buffer重悬沉淀,沸水中煮沸10 min,保证菌体完全裂解。10 000×g离心5 min后,取上清10 μL用于SDS-PAGE分析。凝胶用考马斯亮蓝R-250染色液染色,脱色液脱色,凝胶成像。
1.5 牦牛Linc24063组织表达分析
利用PrimeScript TM RT reagent Kit with gDNA Eraser(TaKaRa, 大连)试剂盒将各组织总RNA反转录为cDNA,Linc24063表达量的检测参照SYBR Premix Ex TaqTM试剂盒(Takara)说明书,内参基因选择核糖体蛋白S9(RPS9,登录号:XM_005899362.2)、广泛表达蛋白(UXT, 登录号:XM_014483477.1)[20];对于miRNA表达量的检测利用Stem-loop方法 [21],内参基因选择5S rRNA。利用Bio-Rad CFX96(伯乐,美国)定量仪进行牦牛Linc24063及miRNAs组织表达谱分析。扩增体系均为:2×SYBR? Premix Ex TaqTM Ⅱ10 μL,cDNA模板1 μL,上下游引物各1 μL,ddH2O 7 μL。qPCR条件为:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,39个循环。所用引物列于表1。Table 1
表1
表1实时荧光定量PCR引物
Table 1
| 基因名称 Gene name | 引物序列(5′-3′) Primer sequence (5′-3′) | 产物大小 Product length (bp) |
|---|---|---|
| Linc24063 | F:ACTGTGAACATAGATGGTGAGG | 141 |
| R:CACGGGGTCACAAAGAGTCA | ||
| RPS9 | F:GTGGTGAACATCCCGTCCTT | 130 |
| R:GCCACTGCACCTTGTAACAC | ||
| UXT | F:CGAGGCTTTCATCTCTGACG | 138 |
| R:TCCGAGTGATTAGCTTCCTGG | ||
| 18S rRNA | F:GTGGTGTTGAGGAAAGCAGACA | 79 |
| R:CGATCCCTGTCCTCACCTCATC | ||
| MiR-200a | RT:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACaacatcgt | |
| F:CTGGtaacactgtctggta | 70 | |
| MiR-24 | RT:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACctgttcct | |
| F:GGAtggctcagttcagc | 69 | |
| MiR-27a | RT:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACcggaactt | |
| F:GGCacttcacagtggct | 69 | |
| MiR-141 | RT:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACccatcttt | |
| F:GGCataacactgtctggt | 69 | |
| miRNA universal primer | GTGCAGGGTCCGAGGT |
新窗口打开|下载CSV
1.6 数据分析
基因表达量差异分析均使用2-△△ct法,对于Linc24063的定量选择UXT和RPS9的几何平均值进行分析。利用SPSS 19.0皮尔逊系数(双尾)反应Linc24063与miRNA的相关性。相关性以|R |≥0.8为极强相关,0.6≤|R |≤0.8为强相关,0.4≤|R |≤0.6为中度相关,|R |≤0.4为不相关。结果表示为平均值+标准误。*,P<0.05为差异显著,**,P<0.01为差异极显著。2 结果
2.1 长链非编码RNA Linc24063序列特征
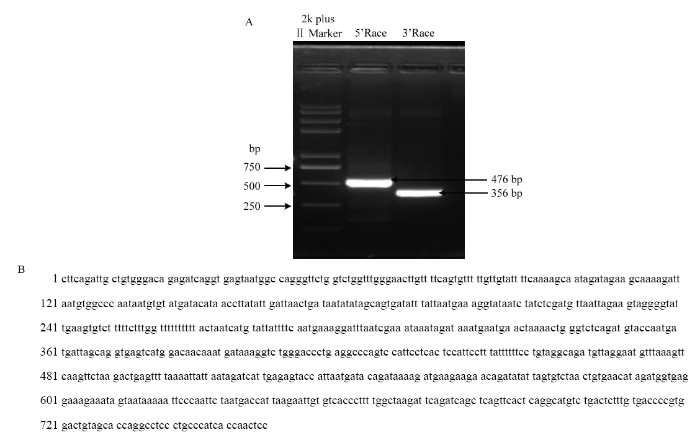
利用SMARTer RACE cDNA Amplification Kit 扩增牦牛长链非编码RNA Linc24063,结果表明,5′ RACE片段大小为476 bp,3′ RACE片段大小为356 bp(图1-A),测序分析表明Linc24063大小758 bp(图1-B),位于牦牛21号染色体的Dlk1-Dio3印记域。NCBI blast分析物种间保守性,结果表明牦牛Linc24063与牛、绵羊、山羊的序列相似性分别为98%、90%和90%,而与人和小鼠的序列相似性较低。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1Linc24063 5′ Race和3′ Race电泳图(A)及序列(B)
Fig. 1The 5′ Race and 3′ Race electrophoresis analysis (A) and the sequence (B) of Linc24063
2.2 长链非编码RNA Linc24063的鉴定
利用在线软件Coding Potential Calculator对长链非编码RNA Linc24063、已知lncRNA Meg9和编码基因CSN2的编码能力预测分析,结果表明:Linc24063与长链非编码RNA Meg9类似,编码潜能均较低,明显区别于编码基因CSN2,属于非编码RNA(表2)。Table 2
表2
表2Linc24063、Meg9和CSN2基因编码潜能分析
Table 2
| 基因 Gene | 编码/非编码 Coding/Noncoding | 编码潜能 Coding probability | Fickett 分 Fickett score | 等电点 Isoelectric point |
|---|---|---|---|---|
| Linc24063 | Noncoding | 0.01 | 0.35 | 9.52 |
| Meg9 | Noncoding | 0.17 | 0.36 | 7.90 |
| CSN2 | coding | 1.00 | 0.45 | 5.89 |
新窗口打开|下载CSV
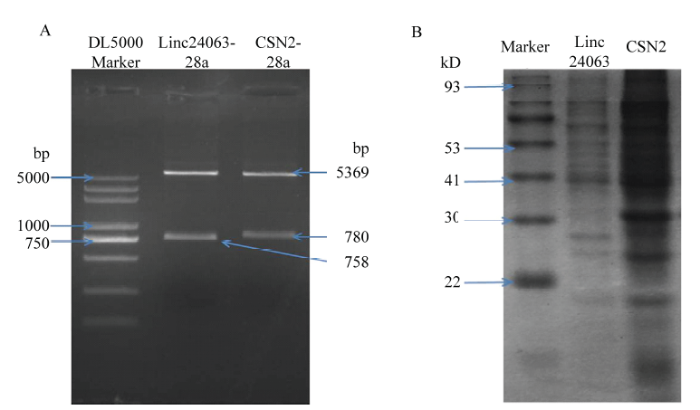
为验证Linc24063是否具备编码能力,本研究将Linc24063连接pET-28a原核表达载体,提取质粒进行EcoRⅠ和HindⅢ双酶切鉴定,由图2-A可知,Linc24063-28a和CSN2-28a分别检测到一条758 bp和780 bp大小的片段,进一步通过测序验证目的片段成功插入原核表达载体。利用原核表达系统在体外对Linc24063和编码基因CSN2进行翻译,结果表明:Linc24063在30 kD附近没有检测到蛋白表达,表明其不能有效的翻译蛋白,而在相同试验条件下,编码基因CSN2在30 kD附近有蛋白表达,表明CSN2能很好的表达蛋白(图2-B)。以上研究结果表明,牦牛Linc24063是一个真正的长链非编码lncRNA。
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2Linc24063的原核表达
(A)Linc24063与CSN2原核表达载体酶切鉴定;(B)Linc24063与CSN2原核表达
Fig. 2The prokaryotic expression of Linc24063
(A) Identification of prokaryotic expression vector of Linc24063 and CSN2 by restriction endonuclease. (B) The prokaryotic expression of Linc24063 and CSN2
2.3 长链非编码RNA Linc24063组织表达谱
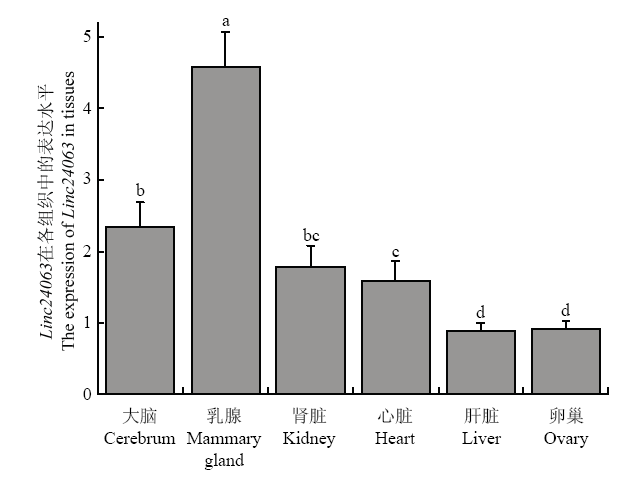
利用RT-qPCR技术,笔者研究了Linc24063在牦牛不同组织的表达谱。结果显示,Linc24063在乳腺表达量最高,显著高于其他组织(P<0.05),其次大脑和肾脏,在肝脏和卵巢中表达量较低(图3)。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3Linc24063在各组织中的表达量
小写字母a,b表示差异显著,P<0.05
Fig. 3The expression of Linc24063 in tissues
The lowercase a, b means significant difference, P<0.05
2.4 长链非编码RNA Linc24063生物信息学分析
下载miRbase 22数据库中普通牛(牦牛的近源物种)和绵羊所有的miRNAs,利用miRanda和mireap软件分析与Linc24063具有相互作用的miRNA,共发现21个物种间保守miRNAs(表3)。Table 3
表3
表3与Linc24063相互作用的保守miRNAs
Table 3
| Name | 成熟体序列(5′-3′)Mature sequence(5′-3′) |
|---|---|
| bta-miR-137 | UUAUUGCUUAAGAAUACGCGUAG |
| bta-miR-139 | UCUACAGUGCACGUGUCUCCAGU |
| bta-miR-141 | UAACACUGUCUGGUAAAGAUGG |
| bta-miR-154a | UAGGUUAUCCGUGUAGCCUUCG |
| bta-miR-200a | UAACACUGUCUGGUAACGAUGUU |
| bta-miR-221 | AGCUACAUUGUCUGCUGGGUUU |
| bta-miR-223 | UGUCAGUUUGUCAAAUACCCCA |
| bta-miR-24 | UGGCUCAGUUCAGCAGGAACAG |
| bta-miR-27a | AGGGCUUAGCUGCUUGUGAGCA |
| bta-miR-30b-5p | UGUAAACAUCCUACACUCAGCU |
| bta-miR-369-3p | AAUAAUACAUGGUUGAUCUUU |
| bta-miR-374a | UUAUAAUACAACCUGAUAAGUG |
| bta-miR-376b | AUCAUAGAGGAAAAUCCAUGUU |
| bta-miR-378b | ACUGGACUUGGAGUCAGAAGGC |
| bta-miR-378c | ACUGGACUUGGAGUCAGAAGU |
| bta-miR-455-3p | GCAGUCCAUGGGCAUAUACACU |
| bta-miR-487a | AAUCAUACAGGGACAUCCAGU |
| bta-miR-500 | UAAUCCUUGCUACCUGGGUGAGA |
| bta-miR-543 | AAACAUUCGCGGUGCACUUCUU |
| bta-miR-551a | GCGACCCAAUCUUGGUUUCCA |
| bta-miR-655 | AUAAUACAUGGUUAACCUCUCU |
新窗口打开|下载CSV
由2.3研究表明,Linc24063在乳腺的表达量最高,我们推测其在乳腺发育或乳合成过程中具有重要作用,由此结合前人在乳腺中miRNAs表达谱的研究[22,23],发现13个miRNAs可能在乳腺组织中与Linc24063具有相互作用。对这13个miRNAs的靶基因进行GO富集和KEGG信号通路分析,结果表明,这些miRNAs显著富集到RNA聚合酶II相关基因的转录等生物学过程中(表4)。KEGG信号通路分析表明,这些miRNAs显著参与到TGF-beta信号通路、PI3K-Akt信号通路、ErbB信号通路、MAPK信号通路等(表5)。
Table 4
表4
表4与Linc24063相互作用的miRNAs靶基因的前10条GO富集条目
Table 4
| GO条目 Go Term | 基因数目 Gene count | 富集倍数 Fold enrichment | P值 P value |
|---|---|---|---|
| Nucleus细胞核 | 201 | 1.63 | 7.98E-14 |
| Cytoplasm细胞质 | 171 | 1.28 | 2.06E-04 |
| Regulation of transcription from RNA polymerase II promoter RNA聚合酶II启动子转录的调控 | 116 | 1.98 | 4.76E-06 |
| Nucleoplasm 核质 | 83 | 1.48 | 2.68E-04 |
| Metal ion binding金属离子结合 | 71 | 1.42 | 0.00231 |
| Cytosol胞液 | 60 | 1.37 | 0.011998 |
| RNA polymerase II core promoter proximal region sequence-specific DNA binding DNA特异性结合RNA聚合酶II核心启动子近区序列 | 52 | 2.92 | 3.51E-08 |
| Transcription, DNA-templated DNA为模板的转录 | 42 | 1.57 | 0.004197 |
| Transcriptional activator activity, RNA polymerase II core promoter proximal region RNA聚合酶II核心启动子近区转录激活活性 | 33 | 3.05 | 2.15E-06 |
| Sequence-specific binding序列特异性结合 | |||
| Chromatin binding染色质结合 | 30 | 2.22 | 8.71E-05 |
新窗口打开|下载CSV
Table 5
表5
表5与Linc24063相互作用的miRNAs靶基因的信号通路
Table 5
| 信号通路条目 Pathway term | 基因数 Input number | P值 P-value | 校正后P值 Corrected P-value |
|---|---|---|---|
| TGF-beta signaling pathway TGF-β信号通路 | 16 | 1.46E-06 | 8.22E-05 |
| PI3K-Akt signaling pathway PI3K-Akt信号通路 | 28 | 0.000267 | 0.0063 |
| Axon guidance 轴突导向 | 18 | 0.000392 | 0.00865 |
| ErbB signaling pathway ErbB 信号通路 | 10 | 0.002087 | 0.03393 |
| Chronic myeloid leukemia慢性粒细胞白血病 | 9 | 0.002965 | 0.04313 |
| FoxO signaling pathway FoXO信号通路 | 13 | 0.003366 | 0.04738 |
| Pathways in cancer 癌症通路 | 27 | 0.003372 | 0.04783 |
| Insulin signaling pathway胰岛素信号 | 13 | 0.003495 | 0.04786 |
| Maturity onset diabetes of the young糖尿病 | 5 | 0.003572 | 0.04789 |
| MAPK signaling pathway MAPK信号通路 | 19 | 0.003611 | 0.04952 |
新窗口打开|下载CSV
2.5 长链非编码RNA Linc24063与miRNAs表达相关性分析
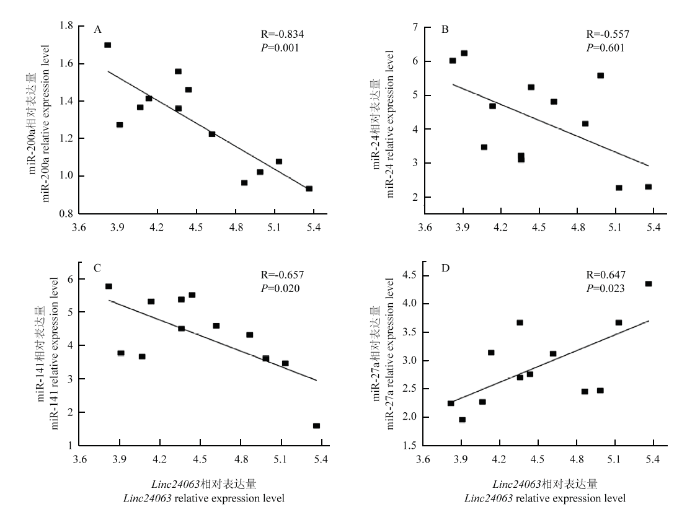
为探究Linc24063与miRNAs的功能相关性,本研究利用RT-qPCR在乳腺组织中检测Linc24063与miR-200a 、miR-24、miR-141及miR-27a的表达量,并进行了皮尔逊相关性分析(图4),结果表明:Linc24063在12头牦牛乳腺组织中的表达量与miR-200a(R=-0.834,P=0.001)和miR-141(R=-0.657,P=0.02)的表达量均显著负相关,与miR-27a的表达量显著正相关(R=0.647,P=0.023),而与miR-24无显著相关性(P>0.05)。以上研究结果表明,Linc24063在牦牛乳腺组织中可能通过调控miR-200a、miR-141和miR-27a基因的表达,从而参与到乳腺发育或乳成分合成及代谢的生理过程中。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图412头牦牛乳腺组织Linc24063与miRNAs表达量相关性分析
Fig. 4The correlation between expression levels of Linc24063 and miRNAs in 12 mammary gland of yaks
3 讨论
目前,关于lncRNA的研究不仅局限于模式动物及人上,在猪、牛、羊上也开展了大量研究,但是关于牦牛lncRNA的研究较少。本研究首次通过RACE技术获取并鉴定了牦牛Linc24063,位于牦牛21号染色体的Dlk1-Dio3印记域,在反刍动物牛、羊中具有较高的保守性(>90%),与前人对Dlk1-Dio3印记域的研究表明该印记域在物种间具有较强的保守性的结果一致[24]。组织表达谱分析结果表明,Linc24063在所检测的6个组织中均有表达,且在乳腺中表达量最高,其次是大脑和肾脏,表明Linc24063在这些组织中可能均具备生物学功能。生物信息学分析结果表明:共筛选到21个与Linc24063相互作用的物种间保守miRNAs 。结合前人研究对miRNAs在乳腺组织中的表达谱研究[22,23],笔者筛选了13个miRNAs可能在乳腺组织中与Linc24063具有相互作用。对这13个miRNAs的靶基因进行GO富集和KEGG信号通路分析表明:与Linc24063相互作用的miRNAs的靶基因富集到TGF-beta信号通路、PI3K-Akt信号通路、胰岛素信号通路及与神经发育、保护的轴突引导(Axon guidance)信号通路中。TGF-beta信号通路[25]、PI3K-Akt信号通路[26,27]、胰岛素信号通路是与乳腺发育、乳脂乳蛋白合成密切相关的信号通路,结合Linc24063在乳腺组织中表达量最高的特点,推测Linc24063可能在乳腺发育及乳脂乳蛋白合成等生物学过程中发挥重要作用。
为探索Linc24063可能的作用机制,本研究随机挑选了4个可能与其具有相互作用的miRNAs,在乳腺组织中对其表达量进行了相关性分析。结果表明,Linc24063与miR-200a、miR-141、miR-27a均显著相关。前人研究表明miR-200a在乳腺上皮细胞中通过调控β-酪蛋白和E-钙粘蛋白以维持上皮细胞的极性[28],同时在乳脂、乳蛋白合成信号通路中发挥重要功能[29];miR-141和 miR-27a可分别通过调控STAT5蛋白、PPARγ蛋白的表达,参与到乳蛋白、乳脂的合成中[30]。由此可以推测,Linc24063可能通过调控miR-200a、miR-141、miR-27a参与到牦牛乳蛋白及乳脂的生物合成中,但是其具体功能和作用机制还需进行研究。
4 结论
牦牛Dlk1-Dio3印记域存在一个在反刍动物中保守的长链非编码RNA Linc24063,全长758 bp。在乳腺及大脑组织中具有较高表达水平,生物信息学分析表明Linc24063可能参与到PI3K-Akt、胰岛素等与乳脂乳蛋白密切相关的信号通路中。在乳腺组织中Linc24063与乳脂乳蛋白合成相关的miRNAs,如miR-200a、miR-27a和miR-141具有显著的相关性。以上研究结果为Linc24063的功能研究提供了基础数据。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1016/j.cell.2011.07.014URL [本文引用: 1]
[本文引用: 1]
DOI:10.1016/S0065-2660(02)46006-3URL [本文引用: 1]
DOI:10.1093/hmg/ddy235URL [本文引用: 1]
DOI:10.1080/17476348.2017.1355241URL [本文引用: 1]
DOI:10.1007/s00018-014-1698-9URL [本文引用: 1]
DOI:10.1016/j.cmet.2018.02.022URL [本文引用: 2]
DOI:10.1101/gr.172701URL [本文引用: 1]
DOI:10.1074/jbc.M110.131995URL [本文引用: 1]
DOI:10.1038/ng.1110URL [本文引用: 1]
DOI:10.3892/ol.2017.6019URL [本文引用: 1]
DOI:10.11843/j.issn.0366-6964.2017.01.001URLMagsci [本文引用: 1]

<p>胚胎发育是一个复杂的过程,其发生受到了很多转录调控因子的调控。随着高通量深度测序技术的发展,研究发现非编码RNA(ncRNA)对动物胚胎发育、X染色体失活、性别调控、脑部发育都具有十分重要的调控作用。其中,miRNA主要通过与其靶向mRNA的3′UTR结合参与调控胚胎发育的相关基因; lncRNA通过转录干扰或是修饰染色质来调控相关基因从而介导哺乳动物X染色体失活和昆虫W染色体的剂量补偿;piRNA通过沉默转座子来维持生殖细胞DNA完整性,并参与生殖细胞形成及家蚕性别调控;circRNA可以作为miRNA的海绵调控动物脑部的发育。本文拟从miRNA、lncRNA、piRNA、circRNA等ncRNA角度阐述其在胚胎发育过程中的研究进展,为进一步研究ncRNA参与调控动物胚胎发育过程的作用机制提供参考。</p>
DOI:10.11843/j.issn.0366-6964.2017.01.001URLMagsci [本文引用: 1]

<p>胚胎发育是一个复杂的过程,其发生受到了很多转录调控因子的调控。随着高通量深度测序技术的发展,研究发现非编码RNA(ncRNA)对动物胚胎发育、X染色体失活、性别调控、脑部发育都具有十分重要的调控作用。其中,miRNA主要通过与其靶向mRNA的3′UTR结合参与调控胚胎发育的相关基因; lncRNA通过转录干扰或是修饰染色质来调控相关基因从而介导哺乳动物X染色体失活和昆虫W染色体的剂量补偿;piRNA通过沉默转座子来维持生殖细胞DNA完整性,并参与生殖细胞形成及家蚕性别调控;circRNA可以作为miRNA的海绵调控动物脑部的发育。本文拟从miRNA、lncRNA、piRNA、circRNA等ncRNA角度阐述其在胚胎发育过程中的研究进展,为进一步研究ncRNA参与调控动物胚胎发育过程的作用机制提供参考。</p>
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 2]
URL [本文引用: 2]
DOI:10.1016/j.bbrc.2015.11.048URL [本文引用: 1]
DOI:10.1007/s10735-011-9337-3URL [本文引用: 1]
DOI:10.1007/s10059-013-2275-zURL [本文引用: 1]
DOI:10.11843/j.issn.0366-6964.2016.09.013URLMagsci [本文引用: 1]

<p>旨在鉴别牛<em>Dlk</em>1-<em>Dio</em>3印记区域内的新印记lncRNAs。本研究选取位于<em>Meg</em>8与<em>Meg</em>9基因间的2组表达序列标签(EST),通过RT-PCR克隆序列并进行分析,结果发现其具有lncRNA的特征,命名为<em>Linc</em>24062和<em>Linc</em>24063。分析<em>Linc</em>24062和<em>Linc</em>24063在牛8个组织(心、肝、脾、肺、肾、骨骼肌、皮下脂肪和大脑)中的表达,发现2个lncRNAs在被检测的组织中均表达。利用基于单核苷酸多态性(SNP)位点的直接测序法,通过比较杂合子牛中SNP位点处基因组PCR扩增产物与RT-PCR扩增产物的测序峰图分析<em>Linc</em>24062和<em>Linc</em>24063的印记状态,发现<em>Linc</em>24062和<em>Linc</em>24063在牛被检测组织中均为单等位基因表达,说明<em>Linc</em>24062和<em>Linc</em>24063在牛中是印记的。</p>
DOI:10.11843/j.issn.0366-6964.2016.09.013URLMagsci [本文引用: 1]

<p>旨在鉴别牛<em>Dlk</em>1-<em>Dio</em>3印记区域内的新印记lncRNAs。本研究选取位于<em>Meg</em>8与<em>Meg</em>9基因间的2组表达序列标签(EST),通过RT-PCR克隆序列并进行分析,结果发现其具有lncRNA的特征,命名为<em>Linc</em>24062和<em>Linc</em>24063。分析<em>Linc</em>24062和<em>Linc</em>24063在牛8个组织(心、肝、脾、肺、肾、骨骼肌、皮下脂肪和大脑)中的表达,发现2个lncRNAs在被检测的组织中均表达。利用基于单核苷酸多态性(SNP)位点的直接测序法,通过比较杂合子牛中SNP位点处基因组PCR扩增产物与RT-PCR扩增产物的测序峰图分析<em>Linc</em>24062和<em>Linc</em>24063的印记状态,发现<em>Linc</em>24062和<em>Linc</em>24063在牛被检测组织中均为单等位基因表达,说明<em>Linc</em>24062和<em>Linc</em>24063在牛中是印记的。</p>
DOI:10.1007/s10709-016-9939-5URL [本文引用: 1]
DOI:10.1016/j.bbagrm.2016.05.003URL [本文引用: 1]
DOI:10.1002/jcp.v232.5URL [本文引用: 1]
DOI:10.1093/nar/gni178URL [本文引用: 2]
DOI:10.1186/1471-2164-13-731URL [本文引用: 2]
DOI:10.3168/jds.2016-11461URL [本文引用: 1]
DOI:10.1016/j.tig.2008.03.011URL [本文引用: 1]
DOI:10.1023/A:1009568532177URL [本文引用: 1]
DOI:10.1016/j.mce.2017.04.025URL [本文引用: 1]
DOI:10.1021/acs.jafc.7b05305URL [本文引用: 1]
DOI:10.1371/journal.pone.0065127URL [本文引用: 1]
URLMagsci [本文引用: 1]

<P><FONT face=Verdana>旨在构建pAdprimiR200a重组腺病毒,使其在奶山羊乳腺上皮细胞中稳定表达 miR200a,研究 miR200a 对乳脂合成相关基因 mRNA 表达的影响。以西农萨能羊 DNA 为模板扩增 primiR200a。采用 AdEasy 系统构建重组腺病毒载体 pAdprimiR200a,并于 HEK 293 细胞株中进行病毒的包装、扩繁。TCI50 法测定病毒滴度。qRTPCR 检测 miR200a 及 16 个乳脂合成相关基因 mRNA 表达水平。序列分析结果显示, primiR200a 包括 premiR200a 86 bp 和侧翼序列共 297 bp。酶切结果证实 pAdprimiR200a 构建成功。 AdprimiR200a 滴度达 8×109 PFU·mL-1。实时定量结果表明,AdprimiR200a (MOI 为 200)感染奶山羊乳腺上皮细胞 72 h,miR200a 的表达量较对照高出 2.4 倍。同时 miR200a 的过表达引起 10 个基因 mRNA 表达量下调,6 个基因 mRNA 表达量上调。其中脂肪酸从头合成相关基因 FASN、脂滴生成相关基因 TIP47 及脂肪酸运输相关基因 FABP4 降幅较大,分别下降了 0.47、0.89及 0.65 倍。而 TAG 生成相关基因 DGAT1 和脂解相关基因 HSL 较对照分别增加了 0.52和 1.49 倍。本研究获得的重组腺病毒 AdprimiR200a 能在山羊乳腺上皮细胞中稳定表达 miR200a,同时 miR200a 过表达影响乳脂合成相关基因 mRNA 表达水平。</FONT></P><P><FONT face=Verdana></FONT> </P>
URLMagsci [本文引用: 1]

<P><FONT face=Verdana>旨在构建pAdprimiR200a重组腺病毒,使其在奶山羊乳腺上皮细胞中稳定表达 miR200a,研究 miR200a 对乳脂合成相关基因 mRNA 表达的影响。以西农萨能羊 DNA 为模板扩增 primiR200a。采用 AdEasy 系统构建重组腺病毒载体 pAdprimiR200a,并于 HEK 293 细胞株中进行病毒的包装、扩繁。TCI50 法测定病毒滴度。qRTPCR 检测 miR200a 及 16 个乳脂合成相关基因 mRNA 表达水平。序列分析结果显示, primiR200a 包括 premiR200a 86 bp 和侧翼序列共 297 bp。酶切结果证实 pAdprimiR200a 构建成功。 AdprimiR200a 滴度达 8×109 PFU·mL-1。实时定量结果表明,AdprimiR200a (MOI 为 200)感染奶山羊乳腺上皮细胞 72 h,miR200a 的表达量较对照高出 2.4 倍。同时 miR200a 的过表达引起 10 个基因 mRNA 表达量下调,6 个基因 mRNA 表达量上调。其中脂肪酸从头合成相关基因 FASN、脂滴生成相关基因 TIP47 及脂肪酸运输相关基因 FABP4 降幅较大,分别下降了 0.47、0.89及 0.65 倍。而 TAG 生成相关基因 DGAT1 和脂解相关基因 HSL 较对照分别增加了 0.52和 1.49 倍。本研究获得的重组腺病毒 AdprimiR200a 能在山羊乳腺上皮细胞中稳定表达 miR200a,同时 miR200a 过表达影响乳脂合成相关基因 mRNA 表达水平。</FONT></P><P><FONT face=Verdana></FONT> </P>
DOI:10.1016/j.gene.2013.03.050URL
