 ,1, 陈亮
,1, 陈亮 ,1
,1Screening, Cloning and Functional Research of the Rare Allelic Variation of Caffeine Synthase Gene (TCS1g) in Tea Plants
LIU YuFei1,2, JIN JiQiang1, YAO MingZhe ,1, CHEN Liang
,1, CHEN Liang ,1
,1通讯作者:
编委: 赵伶俐
收稿日期:2018-12-4接受日期:2019-02-25网络出版日期:2019-05-16
| 基金资助: |
Received:2018-12-4Accepted:2019-02-25Online:2019-05-16
作者简介 About authors
刘玉飞,Tel:18737616921;E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (5997KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
刘玉飞, 金基强, 姚明哲, 陈亮. 茶树咖啡碱合成酶基因稀有等位变异TCS1g的筛选、克隆及功能[J]. 中国农业科学, 2019, 52(10): 1772-1783 doi:10.3864/j.issn.0578-1752.2019.10.010
LIU YuFei, JIN JiQiang, YAO MingZhe, CHEN Liang.
0 引言
【研究意义】咖啡碱(caffeine,Cf)是茶叶的重要功能成分之一,具有兴奋和刺激神经的作用[1,2,3,4],但是过量的摄入会使一些敏感人群出现失眠等现象[5,6,7]。因此,保持茶叶应有风味和营养价值的低咖啡碱茶和茶制品受到了特殊需求消费群体的青睐[8]。低咖啡碱茶树育种是实现茶叶低(无)咖啡碱最经济、安全和有效的方法[9]。从我国丰富多样的茶树种质中发掘咖啡碱合成酶稀有等位基因资源,探析其遗传机制,对低(无)咖啡碱茶树育种具有重要理论和实践指导作用。【前人研究进展】咖啡碱的生物合成是以黄嘌呤核苷(xanthosine,XR)为底物,通过三步甲基化、一步脱核苷酸化的核心途径来实现的[10,11,12](图1)。核心途径第一步(N-7)、第二步(N-3)和第三步(N-1)甲基化反应均由N-甲基转移酶类(N-methyltransferases,NMTs)催化完成,其甲基供体是S-腺苷-L-甲硫氨酸(S-adenosyl-L-methionine,SAM)[13]。茶树咖啡碱合成酶(tea caffeine synthase,TCS)属于NMTs,可以催化N-3和(或)N-1甲基化反应。KATO等[14]和YONEYAMA等[15]分别克隆到一个TCS(TCS1和TCS2)的cDNA全长,并通过原核表达发现TCS1具有催化N-3和N-1甲基化的活性,而TCS2不具有NMT活性。金基强等[16]克隆了6条茶树咖啡碱合成酶的基因组全长(TCS1—TCS6),其中TCS6为假基因,而TCS3、TCS4、TCS5在嫩叶中表达量很低。以上研究均表明TCS1是催化茶树咖啡碱生物合成后两步甲基化的关键酶。JIN等[17]发现TCS1在茶组(Sect. Thea)植物中具有多个等位变异,根据TCS15′上游调控区域的插入/缺失突变在茶组植物中鉴定到6个TCS1等位变异(TCS1a、TCS1b、TCS1c、TCS1d、TCS1e、TCS1f),并克隆了这6个等位变异的cDNA全长,其中TCS1a、TCS1b、TCS1c分别与NCBI上已登录的TCS1、ICS1(来源于滇缅茶,C. irrawadiensis)和PCS1(来源于毛叶茶C. ptilophylla)的序列一致,蛋白重组试验发现TCS1d、TCS1e、TCS1f与TCS1a(TCS1)都具有催化N-3和N-1甲基化的活性,而YONEYAMA等[15]研究发现ICS1(TCS1b)和PCS1(TCS1c)仅具有催化N-3甲基化反应的功能。TCS1具有多个等位变异是由茶树咖啡碱合成酶基因的独立进化和近期的快速演化机制导致[18,19,20,21]。也正是由于TCS1序列变异多样,导致了我国茶树资源嘌呤生物碱具有不同的分布模式:多数资源以咖啡碱为主(2.5%—4.5%)[16],少数资源以可可碱[22,23,24]或茶叶碱[25]为优势组分。【本研究切入点】TCS1是茶树咖啡碱合成的关键基因,具有丰富的等位变异,以往研究主要对茶种植物中含有的TCS1等位变异进行了筛选,但对茶树近源植物(多为野生茶树资源)筛选较少,因此有必要对更多的茶树近源植物进行鉴定,发掘新的TCS1稀有等位变异。【拟解决的关键问题】通过进一步筛选我国茶树资源,从而确定TCS1是否还存在新的稀有等位变异;克隆新的TCS1稀有等位变异的基因序列,研究其序列特征和蛋白活性,进一步解析茶树咖啡碱合成的机制,为低咖啡碱茶树育种寻找新的优异等位基因。图1
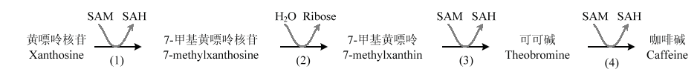 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1咖啡碱生物合成的核心途径
(1)7-甲基黄嘌呤核苷合成酶(黄嘌呤核苷N-甲基转移酶);(2)N-甲基核苷酶;(3)可可碱合成酶(单甲基黄嘌呤N-甲基转移酶):TCS1b(ICS1)、TCS1c(PCS1);(4)咖啡碱合成酶(二甲基黄嘌呤N-甲基转移酶);(3-4)双功能咖啡碱合成酶:TCS1a、TCS1d、TCS1e、TCS1f。SAM:S-腺苷-L-甲硫氨酸;SAH:S-腺苷-L-高半胱氨酸;Ribose:核糖。图片参考文献[10,11,12]
Fig. 1The major biosynthetic pathway of caffeine synthesis
(1) 7-methylxanthosine synthase (xanthosine N-methyltransferase); (2) N-methylnucleosidase; (3) theobromine synthase (monomethylxanthine N-methyltransferase; TS): ICS1 (TCS1b), PCS1 (TCS1c); (4) caffeine synthase (dimethylxanthine N-methyltransferase; CS); (3-4) dual-functional caffeine synthase: TCS1a, TCS1d, TCS1e, TCS1f. SAM, S-adenosyl-Lmethionine; SAH, S-adenosyl-L-homocysteine. The picture refers to references [10-12]
1 材料与方法
试验于2016—2018年在中国农业科学院茶叶研究所进行。1.1 试验材料
以国家种质杭州茶树圃采集的200份(多为地方品种和野生茶树资源)和云南采集的473份茶树资源(来源于临沧古茶树群体:野生、栽培大理茶C. taliensis,阿萨姆茶C. sinensis var. assamica及其中间过渡型茶树)为试验材料,取一芽二叶新梢,迅速液氮固样,并置于-80℃保存,待基因组DNA的提取;另采摘龙井43(LJ43,C. sinensis)、广东可可茶(CCT,C. ptilophylla)和龙陵17(LL17,C. taliensis)春季第一轮一芽二叶新梢,一部分迅速液氮固样,并置于-80℃保存,用于总RNA的提取;另一部分采用120℃热风干燥5 min,75℃烘干,作为生化成分测定的样品。1.2 TCS1稀有等位变异的筛选
利用DNAsecure新型植物基因组DNA提取试剂盒(DP320-03,天根生化科技)进行茶树基因组DNA的提取,提取完毕后所有DNA均稀释至50 ng·μL-1。利用特异引物TCS1P InDel F/R(表1),对提取的基因组DNA进行PCR扩增,扩增结束后在2.5%的琼脂糖凝胶上电泳检测。扩增区域为起始密码子ATG上游303 bp与第一外显子67 bp处。Table 1
表1
表1试验引物及其序列
Table 1
| 引物名称 Primer name | 引物序列Primer sequence (5′-3′) |
|---|---|
| TCS1P InDel F | TATGTCATGTTTCTATTATTT |
| TCS1P InDel R | TACTTTCTCCTTCTCCTCTGT |
| TCS1cDNAF | CACTGCTGTGGCAGCTGGC |
| TCS1cDNAR | CAACTTCTCATTTCTCCCAAC |
| TCS1gF | CGGGATCCATGGGGAAGGTGAACGAAG |
| TCS1gR | CGGAATTCCTATCCAACAATCTTGGAA |
| 18S-F | TCTCAACCATAAACGATGCCGACCAG |
| 18S-R | TTTCAGCCTTGCGACCATACTCCC |
| TCS1-CS-F | TTACTTTTCTGACGAGGCA |
| TCS1-CS-R | TGCGTTAAGAGCTTGAAGG |
| TCS1-TS-F | ATTACTTTTCTCCGACGAGGCG |
| TCS1-TS-R | TGCAGTAAGAGCTTGAAGAA |
| TCS1-Total-F | AGCTGGCCTCTTTGCYATAA |
| TCS1-Total-R | ACTAYTTTCTCCTTCTCCTC |
新窗口打开|下载CSV
1.3 生化成分测定
采用高效液相色谱法(high performance liquidchromatography,HPLC)测定生化样中生物碱含量,各样品均独立重复3次。待测样品的前处理参照GB/T 8313—2018[26],液相色谱测定条件参照金基强等[16]的测定方法。用SPSS17.0进行数据的统计分析。1.4 总RNA的提取、cDNA的合成和TCS1等位变异cDNA全长的克隆
茶树总RNA的提取采用RNA快速提取试剂盒(RN5301,北京艾德莱生物科技),并利用FastKing cDNA第一链合成试剂盒(Kr116-02,天根生化科技)进行第一链cDNA的合成。以反转录得到的cDNA为模板进行PCR扩增,获取目的基因。参考JIN等[17]研究设计PCR扩增的引物TCS1cDNAF/R(表1)。1.5 蛋白活性测定
表达载体pMAL-c5x和目标基因(添加酶切位点的引物TCS1gF/R见表1)同时进行双酶切,酶切成功后进行重组连接、鉴定,以构建蛋白活性测定的表达载体。将成功构建的表达载体和pMAL-c5x空载体分别转化至大肠杆菌表达菌株BL21(DE3) pLysS(CD701-03,全式金)的感受态细胞并进行阳性验证。阳性菌扩大培养到250 mL,然后利用IPTG(终浓度0.5 mmol?L-1)诱导蛋白表达:37℃,120 r/min,诱导4 h。蛋白纯化和蛋白活性检测参考JIN等[17]的方法进行,其中细胞破碎功率改为277 W。蛋白定量采用Bradford法进行(DQ101-01,全式金),每个样品均独立重复测定3次。
1.6 实时荧光定量PCR
以茶树18s作为内参基因(引物序列见表1)。其中TCS1-CS、TCS1-TS和TCS1-Total分别是研究表达蛋白同时具有可可碱和咖啡碱合成酶(caffeine synthase,CS)活性等位变异、表达蛋白仅具有可可碱合成酶(theobromine synthase,TS)活性等位变异和TCS1整体表达水平的引物。
Real-Time PCR反应使用定量试剂盒(KK4601,Kapa Biosystems),反应条件:95℃预变性1 min;95℃变性5 s,60℃退火延伸34 s,40个循环;60—95℃分析溶解曲线。应用ABI sequence detection system(Applied Biosystems,USA)进行检测,每个样品做3个生物学重复。
1.7 序列分析
利用DNAMAN软件包对序列进行拼接;EXPASY(2 结果
2.1 TCS1等位变异的筛选
利用特异引物TCS1P InDel F/R扩增673份茶树资源中TCS1等位变异的5′上游调控区域(目标片段长度为368 bp),从部分大理茶(C. taliensis)资源中扩增出一条620 bp左右的特异条带,通过测序验证,该条带大小为617 bp。序列比对(图2)显示该片段与已往鉴定的TCS1a、TCS1b、TCS1c、TCS1d、TCS1e、TCS1f序列差异明显,长度长95—347 bp,相似度在52.3%—94.6%,因此将具有该片段的TCS1稀有等位变异命名为TCS1g。此外,本研究还发现TCS1g、TCS1b、TCS1c的起始密码子“ATG”均比TCS1a和TCS1d、TCS1e、TCS1f延后15 bp(图2)。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2TCS1g与TCS1其他6个等位变异5′上游调控区域的多序列比对
Fig. 2Comparison of allelic variations in 5’-untranslated region of TCS1g and other six allelic variants of TCS1
2.2 TCS1gcDNA全长的克隆
通过TCS1等位变异的鉴定发现,具有TCS1g这一等位变异的茶树资源,同时还含有一个或多个其他类型的等位变异,为了更好地研究TCS1g的功能,本研究选择具有常规等位变异TCS1a和特异等位变异TCS1g的大理茶资源LL17为试验材料。从LL17总cDNA中获得了TCS1g的全长cDNA序列,编码区长度为1 098 bp,编码365个氨基酸,生物信息学分析结果显示TCS1g分子量为40.9 kD,理论等电点为5.1,酸性氨基酸(天冬氨酸和谷氨酸)有50个,碱性氨基酸(精氨酸和赖氨酸)有33个,其中含量最高的3种氨基酸是:亮氨酸(10.7%)、谷氨酸(8.8%)和丝氨酸(8.8%)。TCS1g与TCS1a—TCS1f的核酸序列相似度在94.1%—99.2%,与TCS1a—TCS1f的蛋白序列的相似度均大于90%,其中与具有TS活性的TCS1b、TCS1c相似度都大于96%(表2)。TCS1g与咖啡中现已克隆(NCBI登陆)的NMTs编码区序列的相似性在29.3%—39.8%,与可可中分离的具有可可碱合成酶功能的BCS1(AB096699)[15]的序列相似度为55.0%。利用茶树、可可和咖啡中NMT的cDNA序列进行系统发生树分析(图3),发现不同物种的NMT独立成簇,茶树中TCS1的不同等位变异聚为两类,Ⅰ类包括新克隆的TCS1g和表达蛋白仅具有可可碱合成酶活性的TCS1b、TCS1c,Ⅱ类包括TCS1a、TCS1d、TCS1e、TCS1f。
Table 2
表2
表2TCS1g与TCS1其他6个等位变异编码区核酸序列(矩阵的上半部分)和蛋白序列(矩阵的下半部分)的相似性(%)比较
Table 2
| TCS1a | TCS1b | TCS1c | TCS1d | TCS1e | TCS1f | TCS1g | |
|---|---|---|---|---|---|---|---|
| TCS1a | - | 94.6 | 94.1 | 97.4 | 96.4 | 97.8 | 95.3 |
| TCS1b | 91.0 | - | 99.2 | 94.5 | 95.4 | 95.3 | 98.9 |
| TCS1c | 89.9 | 98.6 | - | 94.1 | 94.7 | 94.7 | 98. 5 |
| TCS1d | 94.6 | 89.6 | 88.2 | - | 96.8 | 98.8 | 95.4 |
| TCS1e | 93.8 | 91.2 | 89.9 | 93.5 | - | 97.4 | 96.0 |
| TCS1f | 95.4 | 91.0 | 89.6 | 97.3 | 95.4 | - | 96.0 |
| TCS1g | 92.1 | 98.1 | 96.7 | 91.0 | 92.3 | 92.3 | - |
新窗口打开|下载CSV
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3山茶属(Camellia)、咖啡属(Coffea)和可可属(Theobroma)植物咖啡碱合成相关的N-甲基转移酶CDS序列的系统发育树
空心圆、空心正方形和空心三角分别标注山茶属、咖啡属和可可属的N-甲基转移酶基因。TCS1g加粗突显
Fig. 3Phylogenetic tree of N-methyltransferase CDS sequences (related to caffeine synthesis) from Camellia, Coffea and Theobroma
The hollow triangles, hollow circles and hollow squares mark N-methyltransferase genes of Camellia, Coffea, and Theobroma, respectively. TCS1g is highlighted by bold
对已经克隆的TCS1a—TCS1f和新克隆的TCS1g的氨基酸序列进行序列比对(图4),结果显示TCS1g与TCS1的其他等位变异一样,都具有NMT编码蛋白的3个SAM结合域A、B和C以及一个保守域YFFF[17,28-29]。另外,TCS1b、TCS1c、TCS1g相比于TCS1a、TCS1d、TCS1e、TCS1f还有13个氨基酸残基的变异(图4)。TCS1g蛋白质二级结构预测结果显示:47.4%为α-螺旋、13.2%为延伸链、4.9%为β-转角、34.5%为无规则卷曲;通过同源建模的方式对TCS1g进行了蛋白质三级结构模拟(图5),绿色和红色分别标注是TCS1g与甲基受体和甲基供体结合的氨基酸残基。
图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4TCS1g与TCS1其他6个等位变异的氨基酸序列比对
SAM的结合域A、B和C以及一个保守域YFFF用红框标出[15,28-29]。与底物特异性结合的关键位点用篮框标出[15,17,30]。TCS1b、TCS1c、TCS1g相比于TCS1a、TCS1d、TCS1e、TCS1f的变异位点用棕黄色三角标出
Fig. 4Comparison of amino acid sequences of TCS1g and other six allelic variants of TCS1
The proposed SAM-binding motifs (A, B’, and C) and conserved region that is nominated as ‘‘YFFF-region’’ are shown by red open boxes [15,28-29]. The amino acid residues indicated by a blue open box play a critical role in substrate recognition [15,17,30]. The TCS1b, TCS1c and TCS1 g variant sites compared to TCS1a, TCS1d, TCS1e and TCS1f are indicated by a brownish yellow triangle
图5
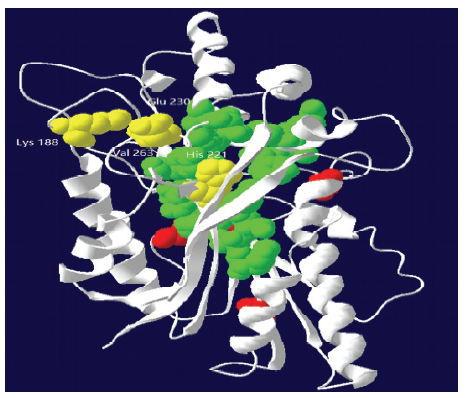 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5TCS1g编码蛋白的三级结构预测模型
黄色氨基酸残基为139—287区间TCS1b、TCS1c、TCS1g与TCS1a、TCS1d、TCS1e、TCS1f不同的氨基酸残基。绿色和红色分别标注是与甲基受体和甲基供体结合的氨基酸残基
Fig. 5Predicted tertiary structure homology-modeling of TCS1g gene
In the interval 139-287, amino acid residues different from TCS1b, TCS1c, TCS1g and TCS1a, TCS1d, TCS1e, TCS1f are indicated in yellow. Green and red are labeled as amino acid residues that bind to the methyl acceptor and the methyl donor, respectively
2.3 TCS1g编码蛋白活性的测定
在甲基供体SAM存在下,将重组酶制剂与底物7-甲基黄嘌呤或可可碱一起27℃孵育1 h后,通过HPLC检测发现,以7-甲基黄嘌呤为底物的反应有可可碱的生成,以可可碱为底物的反应中未检测到有咖啡碱生成,说明TCS1g与TCS1b、TCS1c功能相似,都只有TS活性,不具有CS活性。测定结果(表3)显示,TCS1g的TS活性为44.3 pkat/mg,约为对照TCS1c的2.5倍,但小于对照TCS1a的TS活性。Table 3
表3
表3TCS1g重组蛋白活性分析
Table 3
| 重组蛋白 Recombinant enzyme | 底物/甲基化位置 Substrate/Methylation position | Tb/7-mX (%) | |
|---|---|---|---|
| 7-mX/3-N (pkat/mg) | Tb/1-N (pkat/mg) | ||
| TCS1a | 111.9±5.0 | 27.1±0.1 | 24.2±1.1 |
| TCS1c | 17.9±0.9 | ND | 0 |
| TCS1g | 44.3±0.3 | ND | 0 |
新窗口打开|下载CSV
2.4 TCS1g的表达水平分析
实时荧光定量PCR结果(图6)显示,LL17(具有TCS1a和TCS1g两种等位变异)中TCS1整体表达水平比LJ43低58.0%,比CCT高1.8倍。为了明确TCS1g的表达水平,设计了引物TCS1-TS和TCS1-CS,来区分LL17中功能不同的TCS1a和TCS1g的表达水平,引物TCS1-TS用于检测表达蛋白仅具有TS活性的基因:TCS1c、TCS1g,TCS1-CS用于检测表达蛋白同时具有TS和CS活性的基因:TCS1a。以仅具有等位变异TCS1a的LJ43和仅具有TCS1c的CCT分别作为引物TCS1-TS和TCS1-CS的阴性对照,图6表明,使用TCS1-TS检测LJ43和TCS1-CS检测CCT时,表达水平均极低,说明设计的引物特异性高,能区分两种不同功能的等位变异。定量结果(图6)表明,LL17中TCS1g(TCS1-TS检测结果)有表达,但表达水平较低:只有对照CCT中TCS1c表达量的34.5%,另外LL17中TCS1a(TCS1-CS检测结果)的表达也较低,为对照LJ43中TCS1a的41.6%。图6
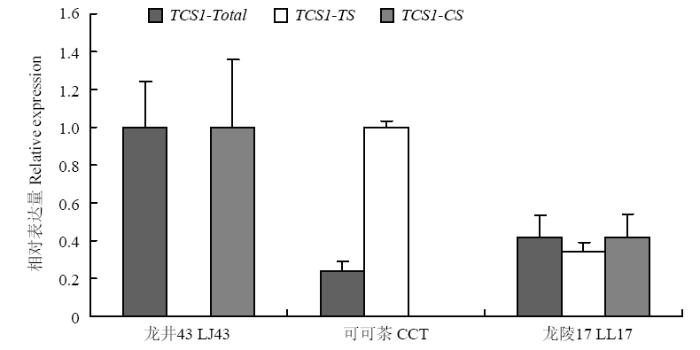 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6实时定量荧光PCR分析TCS1g表达水平
TCS1-CS,TCS1-TS和TCS1-Total分别表示表达蛋白同时具有可可碱和咖啡碱合成酶活性的等位变异、表达蛋白仅具有可可碱合成酶活性的等位变异和整体TCS1的表达水平
Fig. 6Real-time PCR was used to analyze TCS1g specific expression
TCS1-CS, TCS1-TS, and TCS1-Total are the expression levels of allelic variation encoding proteins with both theobromine synthase (TS) and caffeine synthase (CS) activities, only with TS activities, and total TCS1, respectively
2.5 具有TCS1g茶树资源LL17生物碱的含量
为了明确具有TCS1g等位变异的大理茶资源LL17(含有TCS1g和TCS1a两个等位变异)生物碱含量,本研究通过HPLC法进行了测定(表4),可可碱和咖啡碱的含量分别为5.4 mg·g-1和40.3 mg·g-1。虽然LL17具有可可碱合成酶基因TCS1g,但是其生物碱分布模式与仅具有可可碱合成酶基因TCS1c的CCT(高可可碱、无咖啡碱)不同,而是与仅具有TCS1a的LJ43(可可碱和咖啡碱的含量分别为1.8 mg·g-1和31.2 mg·g-1)相似。TCS1a编码的蛋白能将大部分可可碱转化生成咖啡碱,从而导致具有TCS1a 的茶树资源没有大量可可碱的积累,LL17具有TCS1a这一等位基因,所以LL17没能像CCT(只具有可可碱合成酶基因TCS1c)那样积累大量的可可碱。
Table 4
表4
表4含等位变异TCS1g的茶树资源LL17嘌呤生物碱的含量
Table 4
| 资源 Germplasm | 学名 Scientific name | 等位变异 TCS1 allelic variation | 可可碱 Theobromine (mg·g-1) | 咖啡碱 Caffeine (mg·g-1) |
|---|---|---|---|---|
| LJ43 | Camellia sinensis | TCS1a | 1.8±0.0 | 31.2±0.1 |
| CCT | C. ptilophylla | TCS1c | 56.3±0.2 | ND |
| LL17 | C. taliensis | TCS1a、TCS1g | 5.4±0.1 | 40.3±0.3 |
新窗口打开|下载CSV
3 讨论
3.1 TCS1具有丰富的等位变异
本研究通过对673份茶树资源进行筛选,鉴定到一个新的TCS1等位变异TCS1g。TCS1g与JIN等[17]克隆的TCS1a、TCS1b、TCS1c、TCS1d、TCS1e、TCS1f的氨基酸序列相似度均大于90%,核酸序列相似度均大于94%(表2),与茶树中其他NMT的相似度也都大于91%,而与咖啡中NMT家族的基因序列的相似度均低于40%,与可可中分离的具有可可碱合成酶功能的BCS1的序列相似度约55%。以NCBI中已登录的茶树、咖啡和可可中的NMT的cDNA序列构建进化树,结果表明不同物种的NMTs独立成簇,而不是不同属中相同功能的NMT聚在一起。新克隆的具有可可碱合成酶功能的TCS1g也同样与茶树中的NMT聚在一起,这一新的聚类分析与前人[18-21,32]的结果相符,表明不同植物中咖啡碱的生物合成是独立进化形成的。茶树NMT的独立和近期快速的进化机制[20]导致了TCS1具有丰富的等位变异,本研究TCS1g的发现也进一步表明了TCS1等位变异极其丰富。也正是由于TCS1序列变异多样,导致TCS1酶活性多样,从而形成了我国茶树资源嘌呤生物碱具有不同的分布模式。3.2 TCS1g编码蛋白无咖啡碱合成酶活性的重要位点
氨基酸序列比对结果显示,TCS1g的蛋白序列与TCS1b和TCS1c的相似性程度都大于96%,并且TCS1g、TCS1b、TCS1c明显聚为Ⅰ类(图3),即TS类[17](表达蛋白仅具有可可碱合成酶活性)。本研究原核表达蛋白的结果也表明TCS1g仅具有催化7-甲基黄嘌呤生成可可碱的功能,不具有催化可可碱生成咖啡碱的功能,这与聚类结果相符;另外,TCS1a、TCS1d、TCS1e、TCS1f属于Ⅱ类,即CS类(表达蛋白具有可可碱和咖啡碱合成酶活性)。TS类编码的氨基酸序列相比于CS类的TCS1a、TCS1d、TCS1e(氮端缺少一个苏氨酸残基)、TCS1f的氮端缺少一个“ELATA”序列;此外,TS类编码的氨基酸序列还有多个残基的变异。YONEYAMA等[15]通过构建杂交酶(TCS1a和TCS1c)发现决定TCS1蛋白底物结合特异性的区域是139—287这一段氨基酸残基,说明这一区域仅有的4个氨基酸残基(188、221、230和263位)的变异决定了TS类与CS类底物特异性。分析发现188位的赖氨酸(Lys)-精氨酸(Arg)、230位的谷氨酸(Glu)-天冬氨酸(Asp)和263位的缬氨酸(Val)-异亮氨酸(Ile)这3个位点的变异均是同性质氨基酸残基的变异,对蛋白的等电点和亲水性影响很小。另外,通过同源建模发现,这3个突变位点的氨基酸残基都位于蛋白的外侧,距离酶活性中心较远,因此,推测其对蛋白结构和功能影响不大。221位的组氨酸(His)突变为Arg导致TCS1g编码蛋白的等电点(5.05变为5.06)和亲水性(-0.119变为-0.123)都发生改变,图5显示221位氨基酸残基位于酶活性的中心,以往研究指出221位的His突变为Arg会改变酶与底物结合的位阻[17]。另外,YONEYAMA等[15]通过定点突变发现仅具有TS活性的TCS1c第221位氨基酸残基(对应于TCS1a的第225位氨基酸残基的位置)由His突变为Arg后,TCS1c不仅具有TS活性,还具有CS活性;李萌萌等[32]研究发现当第225位的Arg突变为His,TCS1a则不再具有CS活性。综上所述,茶树TCS1编码蛋白的第221位氨基酸残基(对应于TCS1a的第225位氨基酸残基的位置)的突变将直接决定其是否具有CS活性。本研究克隆得到的TCS1g编码蛋白的第221位氨基酸残基也为His(对应于TCS1a的第225位氨基酸残基的位置),并且仅具有TS活性,所以根据以上分析,可以推断第221位氨基酸残基是导致TCS1g仅具有TS活性(不具有CS活性)的关键位点,其他位点的变异可能对蛋白活性的高低有影响。
3.3 TCS1稀有等位变异在低咖啡碱茶树育种中的作用
利用特异引物TCS1P InDel F/R鉴定了673份茶树资源具有的TCS1等位变异,发现这些茶树资源中大多数都含有TCS1a这一等位变异,而TCS1b—TCS1f以及TCS1g类型的稀有等位变异只存在于个别的茶树资源中。常规茶树资源中的TCS1a表达蛋白能将茶树中的大部分可可碱转化生成咖啡碱,这也是多数茶树资源咖啡碱较高含量[17]的主要原因,所以在茶树资源中很难直接筛选得到低咖啡碱植株,并且也很难利用常规的茶树资源通过杂交育种的方式育成低(无)咖啡碱茶树品种。另外,有报道通过理化方法降低茶叶中咖啡碱的含量[33],但是这种方法生产的茶叶风味会发生变化,并且生产成本较高[34]。因此,利用特异的茶树资源作为亲本进行杂交育种是快速选育出综合性状优良、低咖啡碱含量茶树新品种的有效方法。笔者课题组前期开发了有效的InDel标记,可以对茶树资源具有的TCS1等位基因进行鉴定。JIN等[17]利用该标记发现TCS1具有多个等位变异(TCS1a、TCS1b、TCS1c、TCS1d、TCS1e、TCS1f),笔者通过进一步的鉴定又获得了一个新的等位变异TCS1g。其中只具有TS活性的等位变异(TCS1b、TCS1c、TCS1g)和具有低转录水平的等位变异(TCS1f)均是低咖啡碱茶树育种可利用的优异等位变异,具有这些有利等位变异的茶树资源是低咖啡碱茶树育种的优异茶树资源。本研究筛选到的多份茶树资源都可以作为以后低咖啡碱杂交育种的亲本。
4 结论
获得了一个新的茶树TCS1稀有等位变异TCS1g的全长cDNA,其编码蛋白的分子量为40.9 kD,理论等电点为5.1,具有催化7-甲基黄嘌呤生成可可碱的能力,未检测到催化可可碱生成咖啡碱的活性,TS活性大小为44.3 pkat/mg;TCS1g与TCS1b、TCS1c的序列相似度均大于96%,都属于TS类;推测第221位氨基酸残基是导致TCS1g仅具有TS活性(不具有CS活性)的关键性位点。(责任编辑 赵伶俐)
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
DOI:10.1037/h0040683URL [本文引用: 1]
DOI:10.1016/S0278-6915(02)00096-0URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1186/1749-799X-1-1 [本文引用: 1]
DOI:10.1016/j.annepidem.2004.12.011URL [本文引用: 1]
DOI:10.1007/s00198-006-0109-yURL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
DOI:10.1016/j.phytochem.2007.10.029URL [本文引用: 2]
DOI:10.1007/s10068-010-0041-yURL [本文引用: 2]
[本文引用: 1]
DOI:10.1038/35023072 [本文引用: 1]
[本文引用: 9]
[本文引用: 3]
[本文引用: 3]
DOI:10.1016/j.plaphy.2015.12.020URL [本文引用: 13]
DOI:10.1074/jbc.M009480200URL [本文引用: 2]
DOI:10.1146/annurev-arplant-042110-103814URL [本文引用: 1]
DOI:10.1016/j.molp.2017.04.002URL [本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1002/(ISSN)1522-2683URL [本文引用: 1]
DOI:10.1105/tpc.014548URL [本文引用: 3]
[本文引用: 3]
[D].
[本文引用: 2]
[D].
[本文引用: 2]
DOI:10.1021/acs.jafc.8b03433URL [本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
