 ,2, JonG.Hickford2,3
,2, JonG.Hickford2,3Polymorphisms of ADIPOQ Gene and Their Association with Growth and Carcass Traits in Sheep
AN QingMing1,2, ZHOU HuiTong2,3, WU ZhenYang1, LUO YuZhu ,2, Jon G. Hickford2,3
,2, Jon G. Hickford2,3通讯作者:
编委: 林鉴非
收稿日期:2018-11-5接受日期:2019-03-18网络出版日期:2019-05-16
| 基金资助: |
Received:2018-11-5Accepted:2019-03-18Online:2019-05-16
作者简介 About authors
安清明,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (548KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
安清明, 周辉通, 吴震洋, 罗玉柱, JonG.Hickford. 绵羊脂联素基因(ADIPOQ)多态性及其与生长及胴体性状关联性分析[J]. 中国农业科学, 2019, 52(10): 1807-1817 doi:10.3864/j.issn.0578-1752.2019.10.013
AN QingMing, ZHOU HuiTong, WU ZhenYang, LUO YuZhu, Jon G. Hickford.
0 引言
【研究意义】随着人们生活水平的逐步提高,绿色健康的饮食已成为日常生活的主题之一,而羊肉因其独特的营养风味越来越受到人们的偏爱。育种学家们也在根据市场需求逐渐把如何提高羊肉的产量和质量、满足人类追求绿色健康的饮食需求作为品种改良的主要参考因素,其中,从人类营养学角度来分析绵羊的胴体脂肪组成已逐渐成为一个研究热点。本研究通过检测影响绵羊生长、胴体性状的候选基因,评估该基因突变对不同性别绵羊生长性状、屠宰公羔的胴体性状的影响,以期丰富绵羊生长及胴体性状的分子遗传研究基础。【前人研究进展】脂联素,也称脂肪连接蛋白(adiponetin,ADIPOQ或Acrp30),是一种脂肪组织分泌的具有生物活性的脂肪因子,现阶段研究发现其主要与胰岛素耐受性调节、机体肥胖、心血管疾病和II类糖尿病等多种生理疾病相关[1,2]。脂联素由ADIPOQ基因编码,该基因最早在人类3号染色体上发现,总长约17kb,包含3个外显子和2个内含子(其中外显子1不参与蛋白编码),共编码247个氨基酸。编码的ADIPOQ蛋白分子量约为30kD,主要由信号肽、可变区、N-胶原蛋白三螺旋区和C-球状区组成[3,4]。研究发现,ADIPOQ蛋白主要通过与脂联素受体(ADIPOR-1和ADIPOR-2)蛋白结合,调节AMP激酶与PPAR配体的生物活性,从而间接调节脂肪酸氧化和糖类摄取[5],也可通过调节PPAR-α信号因子,从而刺激肌肉组织及肾脏组织的脂肪酸氧化而减少甘油三酯的分泌,进而改变生物体对胰岛素的敏感性,从而参与到胰岛素的分泌调节中[6,7]。2015年,孟宪然等[8]应用RNA-Seq技术对山羊的转录组进行高通量测序分析,结果表明ADIPOQ基因是研究家畜肉品质的重要候选基因。其它一些研究也表明,ADIPOQ基因对家畜的生产性状有一定影响,如ADIPOQ基因启动子区核苷酸c.-67G/A和c.-892C/T变异对猪胴体性状及肉质有显著影响[9],内含子g.1735A/G变异对猪肩部脂肪量有显著影响[10];在牛ADIPOQ基因启动子区中,核苷酸c.-176A/G变异对胴体性状有一定影响,c.-199C/T、c.-34G/A变异对牛眼肌面积和背膘厚度也具有一定影响[11,12]。【本研究切入点】近年来,关于羊ADIPOQ基因多态性变异也有报道[13],但相关变异对绵羊生长性状的性别差异和对公羔胴体性状的分子遗传机制研究却未见报道。【拟解决的关键问题】本研究以ADIPOQ基因作为不同性别罗姆尼羊生长性状、公羔胴体性状的候选基因,检测不同绵羊群体ADIPOQ基因相应区域的遗传变异,结合不同性别罗姆尼羊的生长性状及屠宰公羔的胴体性状,分析罗姆尼羊ADIPOQ基因变异对生长性状的性别差异和对公羔胴体性状的影响。1 材料与方法
研究于2015年4—8月在新西兰林肯大学动物基因标记实验室完成。1.1 试验样品表型
用于检测基因多态性的316份样本主要来自于新西兰南岛26个不同农场的8个绵羊品种(美利奴羊,萨福克羊,特克赛尔羊,考利代羊,罗姆尼羊,派伦代羊,丘陵陶赛特羊和杜泊羊),饲养过程严格按照不同品种分地域饲养,以保证品种间无基因交流。用于表型性状分析的试验样本均采自新西兰南岛单个牧场的17只优秀种公羊的子一代,共1 185只罗姆尼羊。羔羊在出生当天佩戴耳标,记录耳号、性别、出生日期、初生重和出生等级(单羔、双羔或三羔)。3周龄左右对羔羊进行断尾并测定断尾重,同时用FTA卡收集羔羊血样。3月龄断奶,记录并测定断奶重,再结合初生重计算所有羔羊断奶前生长速度。样本在后期的胴体生长性状测定中,由于不可抗拒因素(如羔羊死亡,耳标脱落等因素),部分羔羊缺乏相关表型性状数据,因此,用于结果分析的羔羊数量与最初血液采集总数量有一定偏差。屠宰公羔的热胴体重及V-GR在屠宰厂直接测定,其它胴体性状在屠宰公羔时采用澳大利亚肉品和畜牧业有限公司开发的视频图像分析系统(VIASCAN○?Sastek)技术直接估算相关胴体肌肉性状(肩部瘦肉量、腰部瘦肉量、后腿瘦肉量、总瘦肉量(肩部、腰部、后腿瘦肉量总和)、肩部、腰部及后腿瘦肉比例)。
1.2 试验方法
1.2.1 基因组DNA提取 羔羊用FTA卡(Whatman, Middlesex, UK)收集血液,父本公羊从颈静脉采血后滴于FTA卡,常温保存。采用“两步法”提取基因组DNA[14]。1.2.2 引物设计及PCR扩增 根据GenBank公布的绵羊ADIPOQ基因序列(GenBank Number:NC_019458.1),应用Primer 5.0在线软件设计特异性引物P1和P2(表1),分别用于扩增ADIPOQ基因Exon-1和Exon-2区序列,引物由Integrated DNA Technologies(Coralville, IA, USA)合成。
Table 1
表 1
表 1绵羊ADIPOQ基因的扩增引物信息
Table 1
| 引物名称 Primers | 引物序列(5′-3′) Sequences of primer | 扩增片段长度 Length of amplicons (bp) | 退火温度 Temperature (°C) |
|---|---|---|---|
| P1 | F:TTCCTGCTTCTGATCTTGACC | 388 bp | 58.0 °C |
| R:CAGCCTAGAAATTGAATCAGTC | |||
| P2 | F:ACAGCGTGGATCTGGGTTC | 390 bp | 62.0 °C |
| R:CACAATTCACTTTCGGCTGC |
新窗口打开|下载CSV
PCR反应体系:总体积20 μL,包括FTA卡1.2mm 血样圆片1个,10×PCR Buffer缓冲液2 μL,5×Q Buffer缓冲液2 μL,3 μmol·L-1 MgCl2溶液1.2 μL,150 μmol·L-1 dNTPs 1.2 μL,0.25 μmol·L-1上下游混合引物1 μL,5U·μL-1 Taq聚合酶0.1 μL和ddH2O加至20 μL。
PCR扩增程序:94?C预变性2 min,94?C变性30 s,58?C(P1引物)、62?C(P2引物)退火30 s,72?C延伸30 s,37个循环,延伸7 min,4?C保存。PCR产物用1%琼脂糖凝胶电泳检测。
1.2.3 SSCP检测 取琼脂糖检测过的PCR扩增产物2 μL于灭菌PCR离心管中,加入8 μL变性上样缓冲液(98%去离子甲酰胺、10 mmol·L-1 EDTA、0.025%二甲苯氰、0.025%溴酚蓝),经金属浴105?C热变性5 min后立即置于冰水混合物中冷却5 min,上样于14%非变性聚丙烯酰胺凝胶中(Acr:Bis=37.5:1),0.5×TBE缓冲液中电泳。电泳结束后进行银染显色,判断相应SSCP带型。P1和P2引物最佳SSCP检测条件见表2。
Table 2
表2
表2SSCP电泳条件
Table 2
| 扩增产物 Amplicons | 凝胶浓度 PAGE concentration(%) | 电泳条件 Electrophoresis condition |
|---|---|---|
| P1 | 14 | 300V,17.0?C,19h |
| P2 | 14 | 200V,32.5?C,19h |
新窗口打开|下载CSV
1.3 数据处理
应用MEGA5软件比对等位基因序列;Popgen32.0软件计算等位基因频率、有效等位基因数(Ne)、纯合度(Ho)、杂合度(He)、并进行χ2检验;PIC软件计算多态信息含量(PIC);SHEsis软件分析两个区域之间SNPs的连锁平衡性;本研究利用Minitab(Version 16,Minitab Inc.,Pennsylavania)一般线性模型评估特定等位基因的存在/缺失和基因型对绵羊生长速度、胴体生产性能的影响。对单性状存在(1)/缺失(0)分析模型中,等位基因/基因型、父本、初生重/出生等级为固定因素,遵从下列最小二乘法方差模型:
Yijknm = μ+Gi+Mk+Fm+Xn+eijknm
其中,Y为表型,μ为群体平均值,Gi为父本,Mk为初生重/出生等级(取决于哪个因素对结果更具影响力),Fm为等位基因/基因型,Xn为因素互作效应,eijknm为随机误差。
在单个等位基因存在/缺失分析模型中,仅对群体频率大于5%的等位基因进行关联性分析。同时需对结果P<0.2的等位基因进行互作校正。分析基因型对生产性状的影响时,仅对群体频率大于10%的基因型进行关联性分析。所有分析值均采用“平均值±标准误”表示,P<0.05为显著水平,P<0.2为有影响趋势,P>0.2为无影响。
2 结果
2.1 PCR扩增及产物SSCP检测
P1和P2引物扩增绵羊ADIPOQ基因Exon-1和Exon-2区域,扩增产物经琼脂糖电泳检测与目的片段预测长度一致,并无特异性条带。两对引物PCR扩增产物SSCP检测结果见图1和图2。Exon-1区检测到A1,B1,C1和D1等4种等位基因。Exon-2区检测到A2,B2,C2和D2等4种等位基因。图1
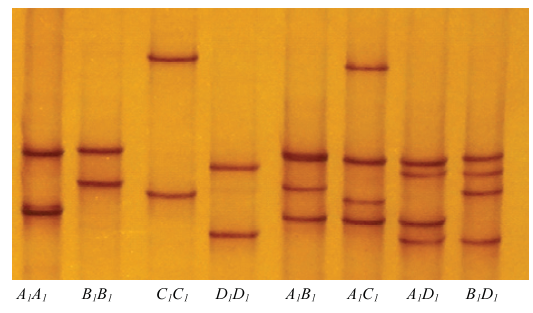 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1绵羊ADIPOQ基因Exon-1区SSCP 检测
Fig. 1SSCP for Exon-1 of ovine ADIPOQ gene
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2绵羊ADIPOQ基因Exon-2区SSCP 检测
Fig. 2SSCP for Exon-2 of ovine ADIPOQ gene
2.2 绵羊ADIPOQ基因等位基因序列比对
ADIPOQ基因起始密码子位于Exon-2区,因此Exon-1区序列不参与氨基酸编码。绵羊ADIPOQ基因不同区域序列比对结果见表3,Exon-1区共检测到7个SNPs突变,Exon-2区检测到6个SNPs突变,其中Exon-2区的c.46 T/C为非同义突变,导致第16位的中性络氨酸(Tyr)和碱性组氨酸(His)变异。Table 3
表3
表3绵羊ADIPOQ基因Exon-1区和Exon-2区突变位点
Table 3
| Exon-1 | Exon-2 | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 突变位点 Mutation sites | 等位基因 Allele | 突变位点 Mutation sites | 等位基因 Allele | ||||||
| A1 | B1 | C1 | D1 | A2 | B2 | C2 | D2 | ||
| c.-9831 | A | G | A | A | c.46# | A | G | A | A |
| c.-9791 | C | C | C | A | c.135 | C | C | C | T |
| c.-9790 | G | T | G | G | c.199+24 | G | T | G | G |
| c.-9644 | G | G | A | G | c.199+71 | G | G | A | G |
| c.-9640 | A | G | A | A | c.199+94 | A | G | A | A |
| c.-9632 | C | T | C | C | c.199+126 | C | T | C | C |
| c.-9631 | A | G | A | A | |||||
新窗口打开|下载CSV
2.3 绵羊ADIPOQ基因多态性分析
2.3.1 绵羊ADIPOQ基因群体遗传学分析 Exon-1区,等位基因A1和B1在所有品种中均为优势等位基因,整体频率分别为55.70%和42.07%,除了在考利代羊中,A1和B1在的频率分别为39.02%和57.32%。在Exon-1区的4个等位基因中,C1仅在罗姆尼羊中检测到,频率为4.93%,而D1在罗姆尼、美利奴、派伦代和考利代羊中检测到,其余品种中没有检测到该等位基因,但其频率极低,频率分别为1.47%、0.70%、3.66%和3.56%,结果见表4。Table 4
表4
表4ADIPOQ基因不同区域在各品种中的等位基因频率
Table 4
| 品种 (Breed) | 数量 Number | 外显子1区 Exon-1 | 外显子2 Exon-2 | ||||||
|---|---|---|---|---|---|---|---|---|---|
| A1 | B1 | C1 | D1 | A2 | B2 | C2 | D2 | ||
| 美利奴Merino | 68 | 50.00 | 48.53 | 0.00 | 1.47 | 55.15 | 5.15 | 9.55 | 30.15 |
| 罗姆尼Romney | 71 | 52.82 | 41.55 | 4.93 | 0.70 | 76.76 | 0.00 | 4.93 | 18.31 |
| 萨福克Suffolk | 42 | 53.57 | 46.43 | 0.00 | 0.00 | 75.00 | 1.19 | 0.00 | 23.81 |
| 特克塞尔羊Texel | 22 | 56.82 | 43.18 | 0.00 | 0.00 | 100.00 | 0.00 | 0.00 | 0.00 |
| 考利代羊Corriedale | 41 | 39.02 | 57.32 | 0.00 | 3.66 | 75.61 | 2.44 | 6.10 | 15.85 |
| 派伦代羊Perendale | 14 | 57.14 | 39.29 | 0.00 | 3.57 | 89.29 | 0.00 | 0.00 | 10.71 |
| 杜泊羊Dorper | 39 | 76.92 | 23.08 | 0.00 | 0.00 | 56.41 | 1.28 | 38.46 | 3.85 |
| 丘陵陶塞特Dorset down | 19 | 81.58 | 18.42 | 0.00 | 0.00 | 100.00 | 0.00 | 0.00 | 0.00 |
| 总数Overall | 316 | 55.70 | 42.09 | 1.11 | 1.10 | 72.78 | 1.74 | 8.70 | 16.78 |
新窗口打开|下载CSV
在Exon-2区,A2为优势等位基因且在所有品种中均有检测,基因频率为72.78%,但在特克赛尔和丘陵陶塞特羊中只检测到等位基因A2。等位基因B2是基因频率最低的,只有1.74%,且在罗姆尼、丘陵陶塞特、特克赛尔和派伦代羊中没有检测到。C2在除派伦代、特克塞尔、萨福克和丘陵陶塞特羊的其它品种中均能检测到,基因频率为8.70%。D2是仅次于A2的等位基因,基因频率为16.77%,但在特克塞尔和丘陵陶塞特羊中没有检测到该等位基因,结果见表4。
绵羊ADIPOQ基因区域的遗传特性及Hardy- Weinberg平衡检验结果见表5。在Exon-1区,杜泊羊和丘陵陶塞特羊有较高的纯合度;在Exon-2区,仅有派伦代羊有较高的纯和度。比较不同群体间的多态性信息含量(PIC),大部分品种在两段区域中均为中度多态(0.25<PIC<0.5),但美利奴羊在Exon-2区呈高度多态(PIC>0.5),特克塞尔、派伦代和丘陵陶塞特在Exon-2区呈低度多态(PIC<0.25)。在所检测的8个品种中,两段多态区域的检测等位基因数与有效等位基因数均不相同,均为观测等位基因数大于有效等位基因数。Hardy-Weinberg平衡检验结果表明,在所检测的两段区域中,大多数品种均处于平衡状态(P>0.05),只有罗姆尼羊在Exon-1区P<0.05、美利奴羊和杜泊羊在Exon-2区P<0.01,偏离了Hardy-Weinberg平衡。
Table 5
表5
表5不同绵羊ADIPOQ基因遗传特性及Hardy-Weinberg平衡检验
Table 5
| 品种 Breed | 外显子1 Exon-1 | 外显子2 Exon-2 | 卡方检验域 χ2 testing | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Na | Ne | Ho | He | PIC | Na | Ne | Ho | He | PIC | 外显子1 Exon-1 | 外显子2 Exon-2 | |
| 美利奴 Merino | 3.00 | 2.06 | 0.53 | 0.47 | 0.40 | 4.00 | 2.45 | 0.32 | 0.68 | 0.53 | P = 0.45 | P<0.01 |
| 罗姆尼 Romney | 4.00 | 2.02 | 0.61 | 0.39 | 0.45 | 3.00 | 1.60 | 0.59 | 0.41 | 0.33 | P<0.05 | P = 0.53 |
| 萨福克 Suffolk | 2.00 | 1.99 | 0.40 | 0.60 | 0.37 | 3.00 | 1.61 | 0.52 | 0.48 | 0.32 | P = 0.23 | P = 0.09 |
| 特克塞尔羊 Texel | 2.00 | 1.96 | 0.68 | 0.31 | 0.37 | 3.00 | 1.58 | 0.57 | 0.43 | 0.00 | P = 0.08 | P = 0.11 |
| 考利代羊 Corriedale | 3.00 | 2.07 | 0.41 | 0.59 | 0.42 | 4.00 | 1.66 | 0.61 | 0.39 | 0.36 | P = 0.13 | P = 0.12 |
| 派伦代羊 Perendale | 3.00 | 2.07 | 0.57 | 0.43 | 0.42 | 2.00 | 1.23 | 0.79 | 0.21 | 0.17 | P = 0.67 | P = 0.72 |
| 杜泊羊 Dorper | 2.00 | 1.55 | 0.64 | 0.36 | 0.29 | 4.00 | 2.14 | 0.46 | 0.54 | 0.44 | P = 0.99 | P<0.01 |
| 丘陵陶塞特 Dorset Down | 2.00 | 1.43 | 0.63 | 0.37 | 0.26 | 2.00 | 1.72 | 0.67 | 0.33 | 0.00 | P = 0.37 | P = 0.13 |
新窗口打开|下载CSV
2.3.2 绵羊ADIPOQ基因不同区域间连锁平衡分析 常用于衡量连锁不平衡的两个参数为D’和r2,其中区域内重组事件发生连锁不平衡的概率通过D’值反映,而连锁分析通过r2值反映。SLATKIN等[15]研究分析认为D’值大于0.8则表明位点间强连锁且不平衡,ARDLIE等[16]分析认为r2值大于0.33表明不同位点是紧密连锁且趋向于一个整体遗传。本研究中Exon-1区域中A1、B1的与Exon-2区域中的A2、D2最为普遍,基因频率最高,因此,将其做连锁不平衡分析发现,绵羊ADIPOQ基因不同变异区域之间D’为0.952,大于0.8,且r2值为0.365,大于0.33,因此这两段区域处于强连锁不平衡状态,表明这两段区域的变异为高度连锁,趋向于整体遗传。
2.3.3 绵羊ADIPOQ基因变异与生长及胴体性状相关性分析 在1 185只羔羊中,在Exon-1区域中检测到4个等位基因中等位基因C1和D1在新西兰罗姆尼羊中的频率小于5%,不能用于关联分析,因此,只对等位基因A1和B1进行关联分析。
对具有完整生长数据特征的550只公羔和537只母羔进行等位基因存在与缺失分析,结果表明,公羔中携带等位基因A1的群体具有较低的断尾重(P=0.019)、断奶重(P=0.017)和断奶前生长速度(P=0.025),等位基因B1与公羔生长性状无显著相关。在母羔中等位基因A1与生长性状无显著相关,但发现携带等位基因B1的群体具有较高的断尾重(P=0.019);模型多因素矫正以上结果同样发现具有显著相关(P<0.05),结果见表6。
Table 6
表6
表6ADIPOQ基因Exon-1区等位基因与罗姆尼公/母羔羊生长性状关联性分析
Table 6
| 生长性状 Growth traits | 等位 基因 Allele | 其它等位基因 Other Allele in model | 公羔 Male Lambs | 母羔 Female Lambs | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 存在 Present (1) | 数目 Number | 缺失 Absent (0) | 数目 Number | P值 P-value | 存在 Present (1) | 数目 Number | 缺失 Absent (0) | 数目 Number | P值 P-value | |||
| 初生重 Birth weight (kg) | A1 | None | 5.51±0.16 | 400 | 5.48±0.17 | 150 | 0.800 | 4.90±0.20 | 382 | 4.81±0.21 | 155 | 0.278 |
| B1 | None | 5.57±0.16 | 418 | 5.44±0.17 | 132 | 0.173 | 4.88±0.20 | 408 | 4.87±0.22 | 129 | 0.938 | |
| 断尾重 Tailing weight (kg) | A1 | None | 12.91±0.53 | 400 | 13.61±0.56 | 150 | 0.019 | 10.58±0.62 | 382 | 10.60±0.65 | 155 | 0.934 |
| B1 | None | 13.18±0.32 | 418 | 13.62±0.39 | 132 | 0.155 | 10.67±0.61 | 408 | 10.06±0.65 | 129 | 0.019 | |
| A1 | B1 | 12.91±0.53 | 400 | 13.75±0.56 | 150 | 0.010 | ||||||
| 断奶重 Weaning weight (kg) | A1 | None | 30.43±0.90 | 400 | 31.64±0.95 | 150 | 0.017 | 25.05±1.05 | 382 | 24.86±1.10 | 155 | 0.661 |
| B1 | None | 30.04±0.55 | 418 | 30.75±0.66 | 132 | 0.179 | 25.12±1.04 | 408 | 24.38±1.10 | 129 | 0.089 | |
| A1 | B1 | 30.52±0.91 | 400 | 31.85±0.96 | 150 | 0.010 | ||||||
| 断奶前生长速度 Pre-weaning growth rate (g·d-1) | A1 | None | 289.10±9.3 | 400 | 300.80±9.7 | 150 | 0.025 | 234.20±1.1 | 382 | 233.60±1.2 | 155 | 0.887 |
| B1 | None | 279.60±5.7 | 418 | 284.80±6.9 | 132 | 0.355 | 234.90±1.1 | 408 | 229.40±1.1 | 129 | 0.233 | |
新窗口打开|下载CSV
基因型与不同性别绵羊生长性状相关性分析表明,不同基因型与公羔的断尾重和断奶重有显著相关(P<0.05),携带B1B1基因型的群体较其它基因型群体有更高的断尾重和断奶重;但在母羔中未发现基因型与生长性状有显著相关,结果见表7。
Table 7
表7
表7ADIPOQ基因Exon-1区基因型与罗姆尼公/母羔羊生长性状关联性分析
Table 7
| 生长性状 Growth traits | 基因型 Genotype | 公羔 Male lambs | 母羔 Female lambs | ||||
|---|---|---|---|---|---|---|---|
| 平均值±标准误 Mean±SE | 数目 Number | P值 P-value | 平均值±标准误 Mean±SE | 数目 Number | P值 P-value | ||
| 初生重 Birth weight (kg) | A1A1 | 5.42±0.17 | 132 | 0.291 | 4.87±0.22 | 129 | 0.521 |
| A1B1 | 5.56±0.17 | 268 | 4.90±0.21 | 253 | |||
| B1B1 | 5.51±0.17 | 150 | 4.81±0.21 | 155 | |||
| 断尾重 Tailing weight (kg) | A1A1 | 13.17±0.56 | 132 | 0.026 | 10.06±0.65 | 129 | 0.056 |
| A1B1 | 12.76±0.54 | 268 | 10.70±0.62 | 253 | |||
| B1B1 | 13.54±0.56 | 150 | 10.57±0.64 | 155 | |||
| 断奶重 Weaning weight (kg) | A1A1 | 30.82±0.96 | 132 | 0.028 | 24.38±1.10 | 129 | 0.160 |
| A1B1 | 30.20±0.92 | 268 | 25.22±1.05 | 253 | |||
| B1B1 | 31.54±0.95 | 150 | 24.83±1.09 | 155 | |||
| 断奶前生长速度 Pre-weaning growth rate(g·d-1) | A1A1 | 291.90±9.9 | 132 | 0.055 | 229.40±11.7 | 129 | 0.445 |
| A1B1 | 287.30±9.5 | 268 | 235.40±11.1 | 253 | |||
| B1B1 | 300.00±9.8 | 150 | 233.30±11.6 | 155 | |||
新窗口打开|下载CSV
关联分析时将所有公羔和母羔置于同一混合群体模型中,将性别作为影响因素进行GLMs模型分析矫正,其它参数相同,分析结果表明混合群体模型中等位基因和基因型均对羔羊生长性状无显著影响,结果未列出。
对屠宰的456只公羔进行胴体性状相关性分析,结果表明等位基因A1与热胴体重、腰部瘦肉量、后腿瘦肉量和总瘦肉量均有显著相关(P<0.05),携带等位基因A1的群体具有较低的热胴体重、腰部瘦肉量、后腿瘦肉量和总瘦肉量;等位基因B1与肩部瘦肉比例、后腿瘦肉量和后腿瘦肉比例有显著相关(P<0.05);携带等位基因B1的群体具有较低的肩部瘦肉比例和较高的后腿瘦肉量、后腿瘦肉比例;多因素矫正以上结果同样具有显著相关(P<0.05)。其它胴体性状与这两个等位基因均没有显著相关,结果见表8。
Table 8
表8
表8ADIPOQ基因启动子变异体对罗姆尼羔羊胴体肌肉性状的影响
Table 8
| 性状 Traits | 等位基因 Allele | 其它等位基因 Other allele in model | 平均值±标准误 (Mean ± SE) | P值 P-value | |||
|---|---|---|---|---|---|---|---|
| 存在 Present | 数量 Number | 缺失 Absent | 数量 Number | ||||
| 热胴体重(kg) Hot weight (H-W) | A1 | None | 17.04 ± 0.29 | 331 | 17.34 ± 0.31 | 125 | 0.086 |
| B1 | None | 17.07 ± 0.29 | 341 | 17.32 ± 0.32 | 115 | 0.149 | |
| A1 | B1 | 17.07 ± 0.30 | 331 | 17.45 ± 0.32 | 125 | 0.036 | |
| B1 | A1 | 17.09 ± 0.30 | 341 | 17.43 ± 0.32 | 115 | 0.060 | |
| GR值(cm) GR value (V-GR) | A1 | None | 2.36 ± 0.48 | 331 | 2.19 ± 0.49 | 125 | 0.550 |
| B1 | None | 2.17 ± 0.47 | 341 | 2.54 ± 0.50 | 115 | 0.196 | |
| A1 | B1 | 2.39 ± 0.48 | 331 | 2.30 ± 0.51 | 125 | 0.752 | |
| B1 | A1 | 2.17 ± 0.18 | 341 | 2.52 ± 0.51 | 115 | 0.234 | |
| 后退瘦肉量(%) Leg yield | A1 | None | 20.69 ± 0.21 | 331 | 21.09 ± 0.21 | 125 | 0.001 |
| B1 | None | 20.93 ± 0.21 | 341 | 20.67 ± 0.22 | 115 | 0.039 | |
| A1 | B1 | 20.68 ± 0.21 | 331 | 21.04 ± 0.22 | 125 | 0.004 | |
| B1 | A1 | 20.95 ± 0.20 | 341 | 20.77 ± 0.22 | 115 | 0.164 | |
| 腰部瘦肉量(%) Loin yield | A1 | None | 14.16 ± 0.15 | 331 | 14.41 ± 0.16 | 125 | 0.006 |
| B1 | None | 14.28 ± 0.15 | 341 | 14.20 ± 0.16 | 115 | 0.359 | |
| A1 | B1 | 14.16 ± 0.15 | 331 | 14.40 ± 0.16 | 125 | 0.009 | |
| B1 | A1 | 14.29 ± 0.15 | 341 | 14.27 ± 0.16 | 115 | 0.755 | |
| 肩部瘦肉量(%) Shoulder yield | A1 | None | 16.93 ± 0.17 | 331 | 17.12 ± 0.18 | 125 | 0.067 |
| B1 | None | 16.99 ± 0.17 | 341 | 17.05 ± 0.18 | 115 | 0.550 | |
| A1 | B1 | 16.95 ± 0.17 | 331 | 17.16 ± 0.18 | 125 | 0.044 | |
| B1 | A1 | 16.70 ± 0.17 | 341 | 17.11 ± 0.18 | 115 | 0.301 | |
| 总瘦肉量(%) Total yield | A1 | None | 51.79 ± 0.43 | 331 | 52.63 ± 0.45 | 125 | 0.001 |
| B1 | None | 52.20 ± 0.43 | 341 | 51.92 ± 0.45 | 115 | 0.281 | |
| A1 | B1 | 51.79 ± 0.43 | 331 | 52.60 ± 0.45 | 125 | 0.002 | |
| B1 | A1 | 52.24 ± 0.43 | 341 | 52.14 ± 0.46 | 115 | 0.712 | |
| 后腿瘦肉比例(%) Proportion of leg yield | A1 | None | 39.94 ± 0.21 | 331 | 40.07 ± 0.22 | 125 | 0.290 |
| B1 | None | 40.09 ± 0.20 | 341 | 39.79 ± 0.22 | 115 | 0.017 | |
| A1 | B1 | 39.92 ± 0.21 | 331 | 39.98 ± 0.22 | 125 | 0.589 | |
| B1 | A1 | 40.09 ± 0.21 | 341 | 39.81 ± 0.22 | 115 | 0.027 | |
| 腰部瘦肉比例(%) Proportion of loin yield | A1 | None | 27.34 ± 0.17 | 331 | 27.37 ± 0.18 | 125 | 0.733 |
| B1 | None | 27.35 ± 0.17 | 341 | 27.35 ± 0.18 | 115 | 0.932 | |
| A1 | B1 | 27.34 ± 0.17 | 331 | 27.37 ± 0.18 | 125 | 0.742 | |
| B1 | A1 | 27.36 ± 0.17 | 341 | 27.36 ± 0.18 | 115 | 0.992 | |
| 肩部瘦肉比例(%) Proportion of shoulder yield | A1 | None | 32.72 ± 0.22 | 331 | 32.55 ± 0.23 | 125 | 0.209 |
| B1 | None | 32.55 ± 0.22 | 341 | 32.86 ± 0.23 | 115 | 0.020 | |
| A1 | B1 | 32.74 ± 0.22 | 331 | 32.64 ± 0.23 | 125 | 0.447 | |
| B1 | A1 | 32.55 ± 0.22 | 341 | 32.83 ± 0.23 | 115 | 0.036 | |
新窗口打开|下载CSV
基因型与胴体性状相关分析表明,基因型与热胴体重、腰部瘦肉量、后腿瘦肉量和总瘦肉量均有显著相关(P<0.05),与其它胴体性状无显著相关(P>0.05);其中携带基因型B1B1的个体均有较高的热胴体重、腰部瘦肉量、后腿瘦肉量和总瘦肉量,结果见表9。
Table 9
表9
表9ADIPOQ基因启动子基因型对罗姆尼羔羊胴体肌肉性状的影响
Table 9
| 性状 Traits | 基因型 Genotype | 数量 Number | 平均值±标准误 Mean ± SE | P值 P-value |
|---|---|---|---|---|
| 热胴体重(kg) Hot weight (H-W) | A1A1 | 115 | 17.24 ± 0.32 | 0.039 |
| A1B1 | 216 | 16.89 ± 0.31 | ||
| B1B1 | 125 | 17.27 ± 0.31 | ||
| GR值(cm) GR value (V-GR) | A1A1 | 115 | 2.56 ± 0.51 | 0.412 |
| A1B1 | 216 | 2.21 ± 0.49 | ||
| B1B1 | 125 | 2.12 ± 0.50 | ||
| 后腿瘦肉量(%) Leg yield | A1A1 | 115 | 20.59 ± 0.22 | 0.002 |
| A1B1 | 216 | 20.77 ± 0.21 | ||
| B1B1 | 125 | 21.13 ± 0.21 | ||
| 腰部瘦肉量(%) Loin yield | A1A1 | 115 | 14.14 ± 0.16 | 0.021 |
| A1B1 | 216 | 14.17 ± 0.15 | ||
| B1B1 | 125 | 14.41 ± 0.16 | ||
| 肩部瘦肉量(%) Shoulder yield | A1A1 | 115 | 17.00 ± 0.18 | 0.109 |
| A1B1 | 216 | 16.89 ± 0.18 | ||
| B1B1 | 125 | 17.10 ± 0.18 | ||
| 总瘦肉量(%) Total yield | A1A1 | 115 | 51.74 ± 0.45 | 0.005 |
| A1B1 | 216 | 51.84 ± 0.44 | ||
| B1B1 | 125 | 52.65 ± 0.45 | ||
| 后腿瘦肉比例(%) Proportion of leg yield | A1A1 | 115 | 39.78 ± 0.22 | 0.050 |
| A1B1 | 216 | 40.06 ± 0.21 | ||
| B1B1 | 125 | 40.13 ± 0.22 | ||
| 腰部瘦肉比例(%) Proportion of loin yield) | A1A1 | 115 | 27.34 ± 0.18 | 0.944 |
| A1B1 | 216 | 27.34 ± 0.17 | ||
| B1B1 | 125 | 27.37 ± 0.18 | ||
| 肩部瘦肉比例(%) (Proportion of shoulder yield | A1A1 | 115 | 32.88 ± 0.23 | 0.050 |
| A1B1 | 216 | 32.60 ± 0.23 | ||
| B1B1 | 125 | 32.50 ± 0.23 |
新窗口打开|下载CSV
3 讨论
本研究所检测的绵羊包括肉用、毛用和肉毛兼用的8种来自新西兰不同农场的商业品种绵羊,共有13个核苷酸变异位点被发现,该结果表明绵羊ADIPOQ基因具有丰富的多态性。已有研究发现在人类[17,18]、猪[19]、牛[11]及山羊[20]中也发现该基因具有一定的核苷酸变异,进一步验证了绵羊ADIPOQ基因的遗传多样性。在发现的13个突变位点中,Exon-2区的c.46 T/C突变为非同义突变,导致第16位的中性络氨酸(Tyr)与碱性组氨酸(His)的变化。该核苷酸突变恰位于ADIPOQ蛋白信号肽区域且临近于信号肽与易变区的分界处,推测该氨基酸的突变可能导致ADIPOQ蛋白空间结构的改变,从而影响信号肽的相关功能,诸如可能影响ADIPOQ蛋白的转录后修饰信号位点的结合、信号肽工作的水准及进程。已有研究表明,在人类编码区相近位置,rs2241766 G/T的变异导致氨基酸变异与多个人群ADIPOQ基因表达水平具有关联性[21,22],因此本研究发现的氨基酸突变值得进一步研究其具体生理功能,如其是否对绵羊生长、胴体组成及脂肪含量存在影响。本研究所检测的等位基因中只有等位基因A1和B1较为普遍,等位基因C1和D1的数量较少,这可能由于本研究所选的罗姆尼羊均为商品羊,经过了牧民长期的自然选择,含有等位基因C1和D1的群体因为某些生产性能不符合当地的需求已被筛选淘汰。分析等位基因A1和B1对绵羊生长性状的影响发现,其对绵羊的生长性状存在性别差异,等位基因A1的存在/缺失对公羔的断尾重、断奶重和断奶前生长速度均有显著影响,而对母羔却无影响;而等位基因B1的存在/缺失却只对母羔的断尾重有显著影响,对公羔却无影响。这表明ADIPOQ基因变异对绵羊生长性能的影响存在性别差异。相关研究表明,母牛胎盘中ADIPOQ蛋白的表达水平与牛犊的初生重具有显著影响[23],在非洲裔美国人群体中也发现ADIPOQ基因变异对表达水平和肥胖存在性别差异,且该变异仅与非洲裔美国人群体中的妇女肥胖有显著关联[24]。目前亦有其它研究发现一些与肌肉生长调控相关基因中也存在一定的性别差异,REISZ-PORSZASZ等[25]发现MSTN基因在雄性小鼠中可能对其肌肉生长具有抑制作用,HAN等[26]发现MSTN基因变异在绵羊中存在性别比例差异。LEE等[27]发现WFIKKN2基因变异与小鼠的不同生长时期的肌肉比重存在性别关联性差异,而WANG等[28]发现WFIKKN2基因变异与绵羊生长性状存在性别关联性差异。这与本研究结果类似,同时在实际生产中一般都是屠宰优质公羔肉用,选留优质母羔用于繁殖,因此有必要进一步加大群体数量,研究该变异对生长性状性别差异的有效机制,进一步证明本研究结果,最终用于指导提高屠宰公羔的生产效益。
ADIPOQ基因变异对罗姆尼羊的胴体性状有一定影响。本试验结果表明,等位基因A1与腰部瘦肉量、后腿瘦肉量和总瘦肉量有一定相关,存在等位基因A1的群体较缺失的群体平均低(0.40±0.19)%,(0.25±0.17)%和(0.84±0.16)%,这意味着对一个胴体重为18kg的羔羊来说,缺失较存在的个体相当于增加了151.2g左右的瘦肉,尽管看起来增加的量并不是很多,但对于养羊产业及现代居民对羊肉需求的营养水平来讲,这意味着更好的市场、更高的利润,尤其是腰部瘦肉量的提高,更具有价值,因为腰部瘦肉对羔羊胴体来讲是价值最高的部位。而等位基因B1与肩部瘦肉比例和后腿瘦肉比例有一定关联性。进一步基因型分析结果还表明,不同基因型与热胴体重、腰部瘦肉量、后腿瘦肉量和总瘦肉量也相关,且本研究中发现的标记位点均与改善绵羊的瘦肉产量相关,而现代居民对肉制品的需求正在转变,越来越倾向于追求健康的饮食,过高脂肪含量的肉制品为消费者所不选择,缘由为过于肥胖的肉制品会导致肥胖、糖尿病或心脏类疾病[29]。本研究中的发现结果在绵羊上少有报道,但在其它动物上已有相关报道。如SHIN等[12]发现ADIPOQ基因突变与牛的背最长肌、脂肪厚度和眼肌面积等性状相关;CIESLAK等[9]发现ADIPOQ基因突变与猪的眼肌面积和腰部肌肉纤维长度等性状相关;OWECKI等[31]发现ADIPOQ基因突变可引起猪体内脂肪连接蛋白水平的改变。这进一步证明了本研究的结果,因此本研究中的相应等位基因和基因型可作为选育提高绵羊胴体性状的分子标记。
4 结论
本研究在8个绵羊群体的ADIPOQ基因中的2个不同区域共发现13个突变位点,其中Exon-2区的c.46 T/C突变为非同义突变,且这2个区域变异为高度连锁。罗姆尼羊Exon-1区等位基因与基因型均可影响绵羊生长性状和胴体性状,淘汰携带等位基因A1的个体和选留存在等位基因B1的个体、或选留B1B1基因型的个体,均可有效改善绵羊后代群体的部分生长性状和胴体性状。(责任编辑 林鉴非)
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
DOI:10.1007/s00109-005-0011-7URL [本文引用: 1]
DOI:10.1074/jbc.270.45.26746URL [本文引用: 1]
DOI:10.2337/diabetes.52.2.550URL [本文引用: 1]
DOI:10.1038/nature01705 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
DOI:10.5713/ajas.2006.779URL [本文引用: 1]
DOI:10.1111/age.2006.37.issue-6URL [本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/j.ab.2006.03.042URL [本文引用: 1]
DOI:10.1038/nrg2361 [本文引用: 1]
DOI:10.1038/nrg777 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s10528-011-9428-6URL [本文引用: 1]
DOI:10.1371/journal.pone.0112480URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.5713/ajas.2010.90392URL [本文引用: 1]
DOI:10.1073/pnas.1309907110URL [本文引用: 1]
DOI:10.1038/srep12347URL [本文引用: 1]
[本文引用: 1]
