 , 李慧慧
, 李慧慧
Accurate Identification of Varieties by Nucleotide Polymorphisms and Establishment of Scannable Variety IDs for Soybean Germplasm
WEIZhong-Yan , LIHui-Hui
, LIHui-Hui
通讯作者:
收稿日期:2017-06-11
接受日期:2017-11-21
网络出版日期:2018-03-12
版权声明:2018作物学报编辑部作物学报编辑部
基金资助:
作者简介:
-->
展开
摘要
关键词:
Abstract
Keywords:
-->0
PDF (3201KB)元数据多维度评价相关文章收藏文章
本文引用格式导出EndNoteRisBibtex收藏本文-->
大豆(Glycine max L.)是豆科(Fabaceae)大豆属(Glycine)一年生草本作物。大豆起源于中国已得到国际****普遍认可[1,2], 我国大豆种质资源丰富, 保存的数量居全世界之首[3]。大豆也是我国重要的经济作物之一, 在全国都有广泛种植[4], 到目前为止, 已育成和推广大豆品种超过1800个[5]。随着大豆种质资源和品种的交换, 种子市场中“同物异名”和“套牌”、“冒牌”现象日趋严重, 这不仅给市场管理和消费者带来困扰, 更会给品种的选育、经营及种植等相关人员和单位带来经济损失。因此, 种质资源和品种身份证的构建具有重要意义。
作物品种资源种子身份证的构建已从形态标记向高通量分子鉴定技术发展。传统的鉴定方法主要是形态标记、生理标记和生化指标, 随着品种资源数目的增加, 仅仅基于这些特性已经难以对现有品种准确鉴定[6]。因此急需开发一套准确可靠、简单易行的快速鉴定体系, 为作物新品种审定及种质材料评价提供科学依据。20世纪以来, DNA分析技术开始被大量应用于植物学研究, 开发出RFLP、RAPD、AFLP、SSR以及SNP、SRAP等一系列标记[7]。分子标记不但能够节省常规田间调查和收集整理数据的时间, 而且具有不受环境影响、鉴别品种准确和变异极丰富等优点[8], 利用分子标记构建DNA指纹图谱进行品种真伪性已成为发展趋势。鉴于方法的稳定和有效性, 国际植物品种权保护联盟(UPOV)在BMT测试指南草案中已将构建DNA指纹数据库的标记方法确定为SSR和SNP[9]。较SSR标记相比, SNP具有针对性强、变异来源丰富、潜在数量巨大等特点。Jung等[10]首次在辣椒中分析438对COSII引物, 选出40个可以鉴别79个热带商业品种和17个甜椒品种的SNP标记。Shirasawa 等[11]研究发现, 利用8个SNP标记可以区分43个水稻品种。王大莉[12]用72个SNP标记构建了22个香菇主栽品种的DNA指纹图谱。利用SNP分子标记鉴定作物的指纹图谱, 对作物品种资源管理与品种保护利用及评价无疑将起到积极的作用。
迄今为止, 受制于SNP标记自动化筛选及检测成本高等问题, SNP标记在大豆种质资源的鉴定方面鲜有报道, 利用与表型性状相关的功能性SNP鉴定资源, 且最终以条形码的形式表现的研究更未见报道。本研究利用与主要农艺性状密切相关的功能基因SNP标记, 精准鉴定不同生态区的大豆种质, 分析其DNA指纹图谱, 同时结合大豆品种属性, 构建其分子身份证, 以期为大豆遗传研究、分子辅助育种和品种鉴定提供依据。
1 材料与方法
1.1 供试材料
北方种质208份, 于2008年至2010年连续3年种植于北方地区3个不同的地点(黑龙江、吉林、内蒙古); 黄淮种质245份, 于2008年至2010年连续3年种植于黄淮地区3个不同的地点(山东、河南、河北); 南方种质146份, 于2008年至2010年连续3年种植于南方地区3个不同的地点(湖北、江西、广西)(图1)。并对其茸毛色、结荚习性、百粒重、熟期、胞囊线虫抗性等表型进行多年多点鉴定。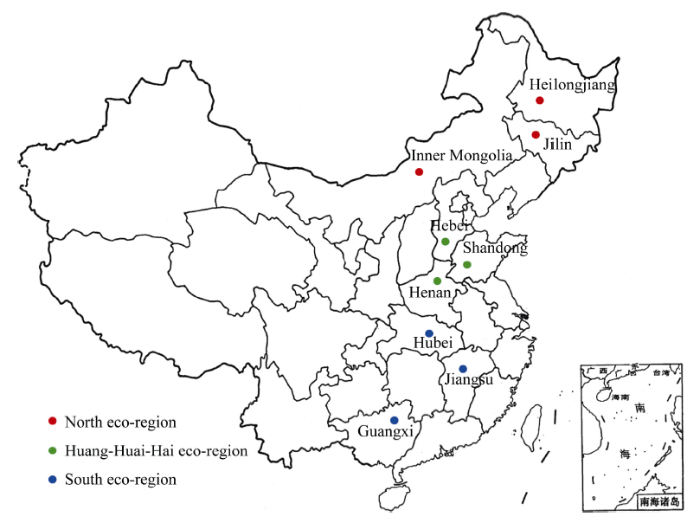 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1大豆精准鉴定种质的种植分布图
-->Fig. 1Planting locations of the three sets of soybean germplasm
-->
1.2 DNA提取和纯化
萌发后取每份大豆种质5个单株的幼叶混样, 使用Fermentas试剂盒提取基因组DNA。其后加2 µL稀释20倍的RNase A, 放到37°C培养箱中15 min, 去除DNA中的RNA, 于-80°C保存待送样。1.3 SNP标记鉴定
选用的23个SNP位点中, 3个来自大豆结荚习性相关基因Glyma19g37890; 2个来自大豆茸毛色相关基因Glyma06g21920; 7个来自大豆熟期相关基因Glyma06g23026, Glyma10g36600, Glyma19g41210和Glyma20g22160; 4个来自大豆SCN抗性相关基因Glyma18g02681和Glyma08g11350; 6个来自大豆SMV抗性相关基因Glyma02g13600、Glyma02g 13380、Glyma13g26000和Glyma14g204500; 1个来自大豆百粒重相关基因Glyma13g22850 (表1)。采用Infmium芯片技术(Bioyong Technologies Inc.), 鉴定全基因组或特定SNP位点。Table 1
表1
表1SNP标记多样性分析
Table 1Analysis of SNP markers diversity
| 性状 Trait | 基因 Gene | SNP位点 SNP locus | 染色体 Chr. | 物理位置 Physical location | 纯合等位 基因比例 Homozygous allele ratio (%) | 变异类型 Mutation type | 基因型 Genotype | 遗传多样性指数 Genetic diversity index |
|---|---|---|---|---|---|---|---|---|
| 结荚习性 Stem growth habit | Glyma19g37890 | AR2 | 19 | 44981190 | 89.60 | 1TV | G/T | 0.334 |
| AR4 | 19 | 44980194 | 95.14 | 1TV | G/A | 0.194 | ||
| AR5 | 19 | 44980087 | 89.95 | 2TS | A/T | 0.351 | ||
| 茸毛色 Pubescence color | Glyma06g21920 | AR7 | 6 | 18540661 | 87.25 | 3D | A/- | 0.382 |
| TT8 | 6 | 18540852 | 57.56 | 3D | C/- | 0.701 | ||
| 熟期 Maturity | Glyma06g23026 | TT10 | 6 | 20007177 | 99.16 | 3D | A/- | 0.049 |
| Glyma10g36600 | TT11 | 10 | 44732850 | 62.39 | 1TV | A/T | 0.696 | |
| Glyma19g41210 | TT12 | 19 | 47516339 | 99.83 | 2TS | G/A | 0.012 | |
| TT13 | 19 | 47513779 | 99.16 | 3D | -/T | 0.048 | ||
| Glyma20g22160 | TT14 | 20 | 32089731 | 100.00 | 4ND | G/- | 0 | |
| PRO15 | 20 | 32091662 | 99.33 | 3D | A/- | 0.040 | ||
| PRO16 | 20 | 32091044 | 100.00 | 4ND | T/- | 0 | ||
| 胞囊线虫 SCN | Glyma18g02681 | PRO19 | 18 | 1712103 | 55.54 | 2TS | T/C | 0.722 |
| Glyma08g11350 | AT21 | 8 | 8280937 | 90.12 | 2TS | T/C | 0.360 | |
| AT22 | 8 | 8281297 | 88.57 | 1TV | A/T | 0.337 | ||
| AT23 | 8 | 8281564 | 94.59 | 2TS | A/G | 0.376 | ||
| 花叶病毒病 SMV | Glyma02g13600 | AT26 | 2 | 11929770 | 99.50 | 2TS | A/G | 0.210 |
| SER27 | 2 | 11930414 | 65.28 | 1TV | G/T | 0.032 | ||
| Glyma02g13380 | SER32 | 2 | 11693604 | 88.01 | 1TV | C/G | 0.665 | |
| Glyma13g26000 | SER36 | 13 | 29227216 | 90.12 | 1TV | G/C | 0.439 | |
| Glyma14g38500 | SER43 | 14 | 47631542 | 57.94 | 1TV | T/A | 0.708 | |
| SER46 | 14 | 47633021 | 100.00 | 4ND | G/C | 0 | ||
| 百粒重 100-seed weight | Glyma13g22850 | SER64 | 13 | 27548370 | 80.57 | 1TV | A/C | 0.514 |
新窗口打开
1.4 SNP多样性计算
利用PowerMaker 3.25软件[13]计算SNP标记在精准鉴定种质中的分布频率及遗传相似系数; 参照Poole[14]介绍的Shannon-Weaver公式, 多样性指数H = -∑Pi ln Pi, 式中Pi为每个位点的等位变异频率。1.5 SNP鉴别能力模拟
利用14个SNP在599份大豆种质中的等位变异频率, 通过R程序模拟分析(http://www.r-project. org/)。共设置8个模拟群体, 群体大小分别为50、100、500、1000、1500、2000、2200和2500。在20个SNP中(除去3个没有碱基差异的标记: TT14, PRO16, SER46)随机选取14个SNP对上述8个群体进行鉴别分析, 该过程一共模拟1000次。最后利用每个群体1000次模拟结果的平均值比较分析。1.6 数据统计分析
利用Flapjack软件[15]分析单倍型; 利用MEGA5.1软件对供试大豆品种进行聚类分析, 并确定材料的遗传结构; 利用条码在线生成器(http:// barcode.tec-it.com/barcode-generator.aspx)对品种身份证进行条码转换。2 结果与分析
2.1 SNP标记多样性分析
本研究23个SNP标记共来自于13个基因, 分别与结荚习性、茸毛色、熟期、百粒重、大豆胞囊线虫病与花叶病毒病抗性相关(表1), 用这些标记分析599份种质, 每个SNP标记其遗传多样性指数变异范围为0~0.722, 平均值为0.312。在23个SNP标记中, 9个发生颠换, 变异范围为0.50%~36.93%; 5个发生缺失, 变异范围为0.67%~99.16%; 6个为转换, 变异范围为0.17%~43.79%。绝大多数位点都有2~3种基因型, 只有3个位点仅有一种基因型, 分别是TT14、PRO16和SER46。利用599份种质的23个SNP鉴定数据分析表明, 精准鉴定种质间的平均遗传相似性系数为0.7923, 以“中豆24”与“茶色豆”遗传差异最大, 为0.0435, 而“Magnolid”与“Nova”遗传差异最小, 为0.9783。根据中国大豆3个生态区划分(表2), 北方生态区(吉林、黑龙江和内蒙古3个省、区) 208个品种间的平均遗传相似系数为0.8295; 黄淮海生态区(包括山东、河南与河北3个省) 245个品种间的平均遗传相似系数为0.7941; 南方生态区(包括湖北、江西和广西3个省、区) 146个品种间的平均遗传相似系数为0.7982。表明品种间遗传差异以北方生态区最小, 南方生态区次之, 黄淮海生态区最大。
Table 2
表2
表2大豆3个主要生态区种质资源的遗传相似性
Table 2Genetic similarity of germplasm resources among three main eco-regions
| 种植区 Planting area | 品种数 No. of varieties | 遗传相似系数 Genetic similarity coefficient | ||
|---|---|---|---|---|
| 最大值 Max. | 最小值 Min. | 平均 Average | ||
| 北方生态区 North eco-region | 208 | 0.9783 | 0.0435 | 0.8295 |
| 黄淮海生态区 Huang-Huai-Hai eco-region | 245 | 0.9783 | 0.3261 | 0.7941 |
| 南方生态区 South eco-region | 146 | 0.9783 | 0.3478 | 0.7982 |
新窗口打开
2.2 GlySNP14的构建
通过对23个SNP位点的等位变异分析, 发现两类特殊的SNP, 一类是2个具有特异等位变异的SNP位点(TT12和AT22), 仅用该位点即可区分“半野生”和“黄豆<2>”, 也可将这2份种质与其他种质区分开来。另一类是3个没有碱基差异的SNP位点, 包括TT14、PRO16和SER46, 这3个位点不能区分任何一种参试的种质, 不适宜用于身份证构建(表1)。具有多态性的20个SNP对599份种质进行单倍型分析, 共形成225个单倍型(图2-A), 其中142个为特异单倍型(图2B), 即20个SNP可以将这142份种质完全区分。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2基于SNP标记的单倍型分析
A: 599份大豆品种的单倍型分析; B: 特异单倍型分析。
-->Fig. 2Analysis of haplotype based on SNP markers
A: Analysis of haplotype for 599 soybean varieties; B: Analysis of specific haplotype.
-->
在去除上述两类特殊等位变异的基础上, 进一步对18个多态性SNP进行组合分析, 采用随机组合和按SNP多样性指数由高到低逐一组合两种方法, 选出鉴别品种能力最佳标记组合GlySNP14。如图3所示, 选用多样性指数高的SNP组合的鉴别种质效率明显优于随机组合, 选用多样性指数最高的2个SNP标记PRO19和SER43进行组合, 可以鉴别出1份种质, 平均每个标记鉴别0.5份种质, 而随机选用AR2和AR5进行组合, 不能鉴别出任何种质; 在上述基础上, 增加TT8与PRO19和SER43进行组合, 可以鉴别出3份种质, 平均每个SNP鉴别效率为0.5, 而利用随机选取的AR7与AR2和AR5进行组合, 仅能鉴别出1份种质, 平均每个SNP鉴别效率为0.33。当多样性指数高的前12个SNP标记(AR2、AR5、AR7、TT8、TT11、PRO19、AT21、AT23和SER32、36、43、64)进行组合时, 可以鉴别98份大豆种质, 每个SNP的平均鉴别效率最高, 达到8.17, 即该12个SNP标记为最佳组合。利用上述12个SNP标记与具有特异等位变异的2个SNP标记(TT12和AT22)相结合组成GlySNP14, 在599份大豆种质中共形成176个单倍型, 其中100个为特异单倍型。即利用GlySNP14可以将100份大豆种质完全区分, 同时也可以将该100份大豆种质从其他种质种中区分出来。
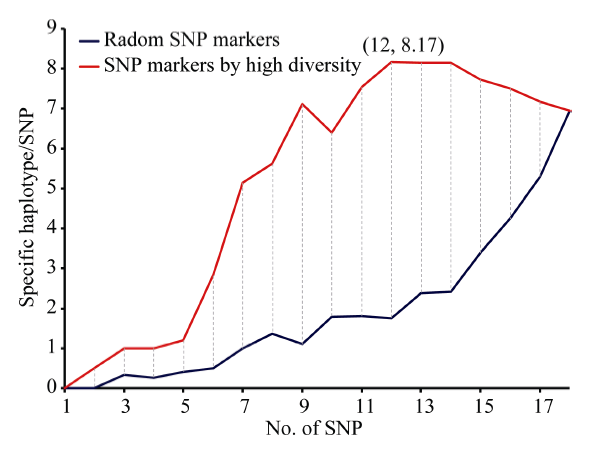 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3SNP标记组合分析
-->Fig. 3Analysis of SNP markers combination
-->
2.3 GlySNP14的聚类分析
GlySNP14可以将100份大豆种质完全区分开(图4), 共分为8组, A与F组分别以地方和选育品种为主, 各占56.25%和76.02%; B组的3个品种分别是1个美国春大豆和2个地方品种; C、G和H组均由国外和选育品种组成; E组10个品种中3个为美国春大豆, 7个为黄淮夏大豆; 最后D组为混合组, 20个品种中40%为东北春大豆, 国外品种和南方春大豆各占30%。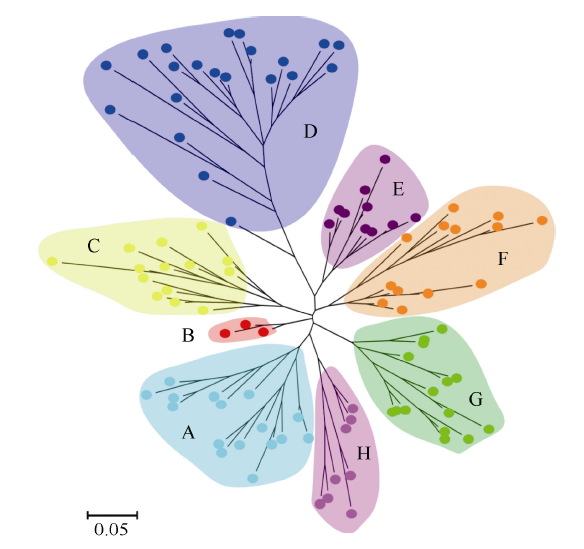 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4基于SNP标记的100份大豆品种聚类分析图
-->Fig. 4Dendrogram of 100 soybean varieties based on SNP markers
-->
2.4 GlySNP14鉴别能力的模拟分析
SNP大多只有两种碱基形式, 被视为二等位标记[12]。虽然14个SNP理论上有214种组合方式, 但在现实种质或群体材料中形成的组合方式要比理论上低很多。利用每个SNP在599份种质中形成的等位变异频率, 对选出的14个SNP的品种鉴别能力的模拟分析表明, 这14个SNP最多可鉴别750个不同种质。为了验证GlySNP14的鉴别效率, 从20个SNP中(除去3个没有碱基差异的标记TT14、PRO16和SER46)随机选出14个SNP进行鉴别能力模拟分析, 该过程模拟1000次。结果如图5所示, 随机选择的任意14个SNP组合, 最多只能鉴别361个品种。在8个不同大小的模拟群体中, GlySNP14的鉴别能力均强于随机选择的SNP标记。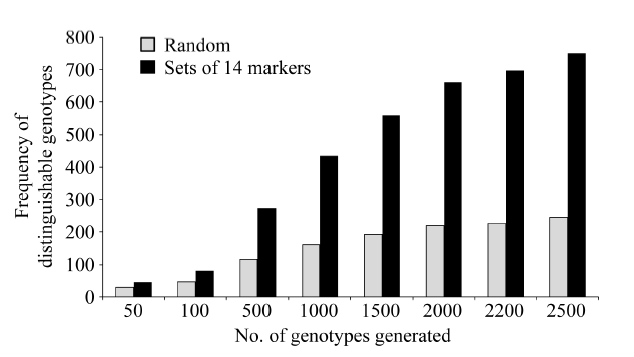 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5随机选择的14个SNP与GlySNP鉴别能力的模拟分析
-->Fig. 5Simulating analysis of variety identification capability of the GlySNP and random sets of 14 SNPs
-->
2.5 SNP指纹图谱构建
为构建身份证, 对大豆种质的SNP数据进行数字化编码。在14个SNP标记中共有10种基因型, 分别为AA、GG、CC、TT、DELDEL、TA、CT、CG、GA和C.DEL, 用数字1~5对碱基A、G、C、T和DEL编码。例如, 地方品种“茶色豆”的14个SNP标记基因型分别为GG、AA、AA、CC、TT、GG、TT、CC、TT、GG、CC、GG、TT和AA, 转换成28位的指纹编码为2211113344224433442233224411, 其中第1~4位的“2211”表示前2个SNP位点AR2和AR5基因型为GG和AA, 第5~8位的“1133”表示位列第3、第4位的SNP位点基因型为AA与CC, 其余20位的编码以此类推。2.6 大豆种质商品码构建
除SNP指纹编码外, 进一步对大豆品种的商品信息编码。基本商品信息分为3个部分: (1)作物及品种属性代码。利用7位数字标识作物及品种属性, 其中1~6位表示大豆种属(大豆属于大田作物中的豆科作物); 第7位表示大豆栽培、野生类型, 即“1”表示栽培大豆, “2”表示野生大豆。(2)区域代码。用于表示大豆品种的来源地区, 以各省市的行政区划代码组成, 即河北为13、山东为37、黑龙江为23等, 国外品种以00表示。(3)品种类别代码。用“1、2”表示大豆的地方与选育两种类型。例如, 大豆品种“青仁黑豆”的商品码为“0102011221”, 其中前7位“0102011”为作物及品种属性代码, 其中第1、第2位的“01”表示大田作物; 第3、第4位的“02”表示豆科; 第5、第6位的“01”表示大豆; 第7位的“1”表示栽培豆。第8、第9位的“22”是行政区划代码, 表示该品种来源地为吉林省。第10位“1”表示该品种为地方品种。2.7 大豆种质身份证构建
大豆种质身份证由上述的商品码与指纹码构成, 总数为38位。以“北丰14”为例, 其身份证号为01020112322211115541223444111133224141, 其作物属性为大田作物-豆科-大豆-栽培种(01-02-01-1); 品种来源为黑龙江选育品种(23-2); 品种DNA指纹为2211115541223444111133224141, 表示其14个SNP分子标记基因型分别为GG、AA、AA、DELDEL、TA、GG、CT、TT、AA、AA、CC、GG、TA和TA。依据此方法完成了其他大豆种质身份证的构建。利用条码在线生成软件分别生成大豆品种身份证的一维码与二维码。利用条码在线生成软件分别生成100份大豆品种身份证的一维码与二维码。“北丰14”的身份证条码如图6-B, 其余99份品种的部分身份证条码见表3。将本研究中大豆品种身份证以一维码或二维码的形式来标示相应的种子, 就可以对种子的相关信息快速识别和规范管理, 也对相应品种的知识产权提供了保护和科学依据。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6“北丰14”品种身份证及条形码示意图
A: “北丰14”品种身份证构成; B: “北丰14”品种身份证条形码。
-->Fig. 6Schematic diagram of “Beifeng14” variety ID
A: Composition of “Beifeng 14” variety ID; B: Bar code of “Beifeng 14” variety ID.
-->
Table 3
表3
表3部分大豆品种的身份证条码信息
Table 3ID information of some soybean varieties
| 品种 Variety | 统一编号 Number | 类别 Category | 品种身份证条形码 Bar code of variety ID | 二维码 QR code |
|---|---|---|---|---|
| 青仁黑豆 Qingrenheidou | ZDD18049 | 地方种 Landrace |  |  |
| 小粒秣食豆 Xiaolimoshidou | ZDD17767 | 地方种 Landrace |  |  |
| 花黑虎 Huaheihu | ZDD18558 | 地方种 Landrace |  |  |
| 滑绿豆 Hualvdou | ZDD10129 | 地方种 Landrace |  |  |
| 青棵圆豆Qingkeyuandou | ZDD08633 | 地方种 Landrace |  |  |
| 铁角黄 Tiejiaohuang | ZDD20188 | 选育种 Bred variety |  |  |
| 吉林30 Jilin 30 | ZDD23704 | 地方种 Landrace |  |  |
| 锦豆33 Jindou 33 | ZDD00745 | 选育种 Bred variety |  |  |
| 冀豆17 Jidou 17 | ZDD24685 | 地方种 Landrace |  |  |
| 晋豆31 Jindou 31 | ZDD24705 | 选育种 Bred variety |  |  |
| 科新4号 Kexin 4 | ZDD23866 | 选育种 Bred variety |  |  |
| L64-1061 | WDD00247 | 选育种 Bred variety |  |  |
| L66-707 | WDD00230 | 选育种 Bred variety |  |  |
| L72-1140 | WDD00242 | 美国春 American Spring |  |  |
| Nattosan | WDD01547 | 美国春 American Spring |  |  |
| Wilkin | WDD00504 | 美国春 American Spring |  |  |
| Mustang | WDD01992 | 美国春 American Spring |  |  |
| Newton | WDD01583 | 美国春 American Spring |  |  |
| Peking | WDD00467 | 美国春 American Spring |  |  |
新窗口打开
3 讨论
3.1 品种DNA指纹图谱的不同表现形式
利用分子标记构建DNA指纹图谱, 已逐渐向快速高效监测方向发展, 并趋向于数字化。赵洪锟等[16] 构建吉林大豆骨干亲本及主推品种RAPD标记指纹图谱, 采用的是分子标记在琼脂糖凝胶电泳中的迁移率大小及条带的有无。随后, 从定性描述转为二位码, 将分子标记条带的有无转化为“0”和“1”编码成数字指纹[17]。但上述二位码大多属于单纯的遗传变异编码, 并未与品种信息相结合。陆徐忠等[18]利用SSR标记鉴定水稻品种, 并将SSR标记信息与商品信息相结合构建了供试水稻品种身份证, 并最终以条形码的形式表现。该方法构建的品种身份证表示形式简便, 易于监测。但前人所使用的SSR标记多为随机标记, 无法与表型性状相联系。相比之下, SNP具有针对性强, 变异来源丰富, 且潜在数量巨大等特点。然而, 利用SNP标记, 尤其是像本研究利用功能标记构建作物身份证, 并最终以条形码的形式表现的研究尚未见报道。
本研究构建的大豆种质资源身份证, 不同于遗传上单纯的分子指纹图谱。该身份证由38个数字组成, 包括品种属性码和分子指纹码, 其中品种属性码由作物属性、品种来源、品种类别3个部分组成, 最终将品种身份证编码转换为条码形式(一维码和二维码)。利用该方法构建的大豆品种身份证可以应用在多种途径上, 例如市场上商品种子的管理, 将品种身份证条形码或二维码标识于种子包装上, 可以方便种子信息的获得与防伪。同时本试验构建身份证用的分子标记是主要农艺性状功能基因SNP标记, 包括结荚习性、茸毛色、熟期、百粒重、胞囊线虫病与花叶病毒病抗性。上述农艺性状均与大豆品质和农业生产及农民利益息息相关, 有望用于进一步丰富品种分子身份证信息。
3.2 SNP标记与SSR标记指纹图谱的比较
指纹图谱构建所用标记的选择向快速、成本低和高通量检测方向发展。国际植物品种权保护联盟(UPOV)确定构建DNA指纹数据库的标记包括SSR和SNP[9]。SSR具有多态性高的优点, 但相比之下, SNP标记在基因组中更为丰富。Jones等[19]同时利用88个SSR标记和187个SNP标记对58个玉米自交系和2个杂交种进行基因型鉴定分析表明, SSR标记的平均等位变异数目是SNP标记的2.5倍, 但平均杂合率比SNP高0.5倍; 相比之下, SNP的可重复性(98.7%)显著高于SSR标记(91.7%)。张昆鹏等[20]比较9个油菜品种的SSR指纹图谱和SNP指纹图谱发现, 基于SNP标记计算的遗传系数更精确, 结果更可靠。这表明SNP标记的高重复性和准确性能够保证不同条件下检测结果的正确性和可比性, 这对于种质和品种的鉴定、品种权的保护与管理都是非常重要的技术支撑。大豆身份证构建的相关研究中, SSR标记是鉴别品种的常用方法。但相比之下, SNP在大豆中分布更为广泛。到目前为止, SNP在栽培大豆中的总数达到4~5×106个[21], 野生大豆PAN-GENOME中存在3.63~4.72×106个[23], 几乎每1 kbp就可发现4个SNP。虽然与SSR相比, SNP等位变异类型有限, 但这一不足可以通过提高SNP数量来克服; 由于SNP大多只有两种碱基形式, 被视为二等位标记, 具有简化基因分型方法及后续数字编码的优势。与SSR标记比, SNP是核苷酸自身变异, 不需要读取分子量大小, 采用测序方法直接获得等位变异基因型, 遗传稳定性高, 更适合于数字化建库。
最少SNP标记的选择是降低构建指纹图谱成本的核心。与SSR的等位基因数多相比, SNP需要的数量比较多。Yoon等[6]比较了23个SNP核心标记与Song等[22]开发的13个SSR标记鉴定大豆种质的效率, 结果表明, 平均每个SSR位点的等位变异数为7.80, 平均多样性为0.73, 可以将101个品种完全鉴别; 每个SNP位点的平均等位变异数为2, 平均多样性为0.45, 可鉴别132份供试品种; 而对23个SNP位点鉴别能力模拟分析结果表明, 最多能鉴别2200份种质。相比之下, 本研究选用的14个标记模拟分析中最多可以鉴别750份种质, 但GlySNP14标记组合在实际的599份大豆种质中鉴别出100份种质。这可能是由于选用的标记为重要性状相关标记, 有些标记为功能基因标记, 多态性相对较低有关, 但这些标记在鉴别品种的同时, 具有提供基因型数据的优点。这为今后作物种质SNP 指纹图谱库的构建提供了有益的启示。
4 结论
从23个功能基因相关SNP中挑选出14个, 精准鉴定了大豆种质品种。GlySNP14在599份大豆品种中鉴定出100份种质并对其构建了品种身份证, 该身份证由品种的属性信息与分子指纹信息两部分组成, 并最终以条码形式(一维码和二维码)表示, 为大豆种质资源管理与保护利用提供了便利。The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | . |
| [2] | . |
| [3] | . . |
| [4] | . . |
| [5] | . . |
| [6] | . |
| [7] | . . |
| [8] | . . |
| [9] | . . |
| [10] | . |
| [11] | . |
| [12] | . |
| [13] | . |
| [14] | |
| [15] | . |
| [16] | . . |
| [17] | . . |
| [18] | . . |
| [19] | |
| [20] | . |
| [21] | . |
| [22] | . |
| [23] | . |
