 , 盖红梅
, 盖红梅
Genetic Contribution of Lumai 14 to Qingnong 2 Revealed by SSR and SNP Markers
LIYu-Gang , GEHong-Mei
, GEHong-Mei
通讯作者:
收稿日期:2017-04-10
接受日期:2017-11-21
网络出版日期:2018-02-12
版权声明:2018作物学报编辑部作物学报编辑部
基金资助:
作者简介:
-->
展开
摘要
关键词:
Abstract
Keywords:
-->0
PDF (1897KB)元数据多维度评价相关文章收藏文章
本文引用格式导出EndNoteRisBibtex收藏本文-->
小麦是我国的重要口粮作物, 其品种改良对产量提高具有重要贡献。在小麦品种改良过程中, 育种家每年配制数百或上千个杂交组合, 但往往依赖于少数几个亲本材料, 这些能衍生出多个品种的材料被称为骨干亲本[1]。我国年推广面积超过66.7万公顷的小麦品种中, 多数含有骨干亲本血统, 老一代骨干亲本往往是新一代骨干亲本的奠基者[2], 例如小偃6号、矮孟牛、周8425B、阿勃、胜利麦等在小麦品种改良中发挥了重要作用, 是著名的骨干亲本[3,4,5,6,7]。鲁麦14因其适应性广、稳产、抗病性等优点, 成为20世纪90年代初期黄淮冬麦区推广面积最大的品种[1], 累计推广783.5万公顷[8]。鲁麦14不仅是主栽品种, 还是育种亲本, 衍生出了济麦22、泰农18等40多个品种[9,10,11], 已成为山东省乃至黄淮冬麦区的新一代骨干亲本。青农2号(山东省青丰种子有限公司选育)是鲁麦14的衍生品种之一, 2010年通过山东省审定(鲁农审2010070号), 2012年通过安徽省认定。该品种高产、稳产, 抗病性好, 2016年安徽赤霉病大暴发, 田间自然发病状态下, 其赤霉病穗率低于10% (张家法, 私人交流)。
作物进化经历了自然选择和人工选择, 人工选择主要包括早期驯化和近100多年的品种改良。驯化使小麦的遗传多样性大幅降低[12], 驯化选择常发生在少数关键基因或位点, 对基因的选择几乎一步到位[13]。张学勇等[13]在单个基因水平, 综述了作物主要育种性状, 发现虽然作物育种史短暂, 但其对基因组影响强烈, 是一些重要代谢途径不断优化的过程。在全基因组方面, Lai等[14]和Zhou等[15]通过重测序分别研究了玉米品种Zheng 58和水稻品种黄华占的基因组组成和育种过程中的重组事件。虽然利用SSR标记对小麦基因组开展了很多研究[6-7,16-18], 但是SSR标记密度低, 无法满足当前高通量基因组研究需要。SNP标记是生物基因组中普遍存在的单核苷酸多态性, 具有密度高、代表性强、遗传稳定性好和自动化程度高等优点[19], 为第3代分子标记。随着分子生物学的发展, 小麦测序工作取得突破性进展[20,21,22], 在大量测序数据积累的基础上, 芯片技术得到快速发展, 先后开发出小麦Illumina iSelect 9k [23]、Illumina iSelect 90k [24]、Affymetrix Axiom 660k (http://wheat.pw.usda.gov/ggpages/topics/Wheat 660_SNP_array_developed_by_CAAS.pdf)等芯片。芯片技术已成为分析小麦遗传多样性[25,26]、数量性状遗传[27,28,29,30]、关联分析[19,31-32]等研究的重要手段, 大大推动了小麦基因组研究的发展。
本研究采用SSR标记和小麦90k芯片SNP标记比较分析了鲁麦14衍生品种青农2号的遗传组成, 结合农艺性状解析亲本鲁麦14和烟农15对青农2号的遗传贡献, 为分子标记辅助育种提供依据。
1 材料与方法
1.1 青农2号选育过程及其亲本
青农2号系谱为鲁麦14/烟农15//矮秆麦, 1995年配制鲁麦14/烟农15杂交组合, 2000年对杂交后代的优异株系进行鉴定试验, 954(5)-4品系表现优良, 但植株偏高; 将该株系与组合内矮秆麦杂交和系统选育, 2004—2005和2005—2006连续两年田间鉴定和品比试验, 获得表型性状优良的954(7)-8高代品系; 2006—2009年参加山东省小麦品种审定试验, 2010年通过审定, 并定名为青农2号。本研究以青农2号及其亲本鲁麦14、烟农15为试验材料。1.2 基因组DNA提取
2013年10月12日, 将供试材料播种在青岛市农业科学研究院城阳试验基地, 2014年4月10日, 在田间剪取2片幼叶冷冻保存, 并标记取样单株。用DNA提取试剂盒(天根公司)提取供试材料的基因组DNA, 并用NanoDrop ND-1000分光测光仪(Thermo Fisher, USA)检测DNA样品浓度和质量。1.3 分子标记分析
1.3.1 SSR标记 选取覆盖小麦全基因组的326对SSR引物(见附表1), 由北京奥科生物技术有限公司合成。用这些引物检测出350个SSR位点, 其中A、B和D 基因组分别有106、92和133个, 另外19个位点无染色体定位。PCR体系和程序、聚丙烯酰胺凝胶检测及SSR标记条带的读取方法, 详见盖红梅等[9]的描述。Table 1
表1
表1性状调查的年份和地点
Table 1Years and locations for trait investigation
| 年份 Year | 地点 Location | 环境数 Number of environments |
|---|---|---|
| 2013-2014 | 山东青岛城阳, 山东泰安肥城, 河南新乡 | 3 |
| 2014-2015 | 山东青岛城阳1), 山东青岛平度, 山东泰安肥城, 河南新乡 | 4 |
| 2015-2016 | 山东青岛城阳1), 山东青岛平度, 山东泰安肥城 | 3 |
新窗口打开
1.3.2 90k芯片 由北京博奥晶典生物技术有限公司完成iSelect Illumina 90k芯片分析, 共获得81 587个SNP位点。采用Genome studio v2011.1软件, 对*.bmp文件和*.idat文件进行数据校正和SNP分型[32] (王玉泉, 个人交流)。校正后, 获得有效SNP位点26 026个, 亲本鲁麦14和烟农15间的差异位点6564个, 染色体定位的位点有5924个[24]。
1.4 亲本遗传贡献及青农2号的遗传组成图谱绘制
统计亲本间差异位点, 当青农2号的分型与鲁麦14一致时计为鲁麦14的贡献位点, 当与烟农15一致时计为烟农15的贡献位点。某一亲本对青农2号的遗传贡献率为该亲本的贡献位点占亲本间差异位点的比例。采用python语言编程(https://www.python.org/)绘制青农2号遗传组成图谱。每条染色体由4根柱形图组成, 从左至右前3根分别代表鲁麦14、烟农15和青农2号基因型, 第4根表示SNP位点在整合遗传图谱中的染色体位置, 即每一条黑色的刻度线代表对应SNP位点的cM, 大段空白表示对应数十甚至数百个SNP位点位于同一染色体位置, 详细染色体位置见附表2。遗传组成图谱的主要色系为红色、蓝色、浅黄色和灰色, 分别代表鲁麦14特有等位变异, 烟农15特有等位变异, 亲本共有等位变异, 非亲本等位变异。
1.5 农艺性状调查及统计分析
自2013年10月至2016年6月连续3个小麦生长季, 在山东省3个试点(青岛城阳、青岛平度和泰安肥城)及河南新乡进行了10点次的农艺性状调查(表1), 每品种2行, 行长2 m, 行距25 cm。其中, 2014—2015和2015—2016年度在青岛城阳进行了雨养和灌溉对比研究, 用于比较供试材料农艺性状在2种水分供应条件下的差异。且每个环境下均调查株高、抽穗期、开花期、旗叶长、旗叶宽、穗长、穗下节间长、穗叶距、抽穗度、小穗数、最大小穗粒数、穗粒数、千粒重、粒长、粒宽和粒厚共16个性状。性状数据按雨养(2014—2015和2015—2016年度, 青岛城阳雨养)、灌溉(2014—2015和2015—2016年度, 青岛城阳灌溉)和雨养+灌溉(10点次所有环境)分别统计。利用SAS软件进行最优无偏估计(BLUP), 获得不同环境条件下亲本和子代的农艺性状最优估计值。2 结果与分析
2.1 青农2号亲本的系谱追踪及基因组差异分析
系谱追踪发现鲁麦14遗传基础丰富, 含有我国蚰包麦、美国包打300炮、智利欧柔等丰产亲本, 日本农林10号、小罂粟等抗旱、矮秆亲本, 罗马尼亚的洛夫林13、意大利维尔、智利如罗、L277/4等著名抗源血统(图1-A); 而烟农15来自单交组合蚰选57/郑引4号(图1-B), 前者是当地高产品种, 后者是意大利高产抗病品种, 是黄淮冬麦区的重要育种亲本。虽然鲁麦14和烟农15遗传背景有较大差异, 但均含蚰包麦血统, 鲁麦14含1/4血统, 烟农15含1/2血统。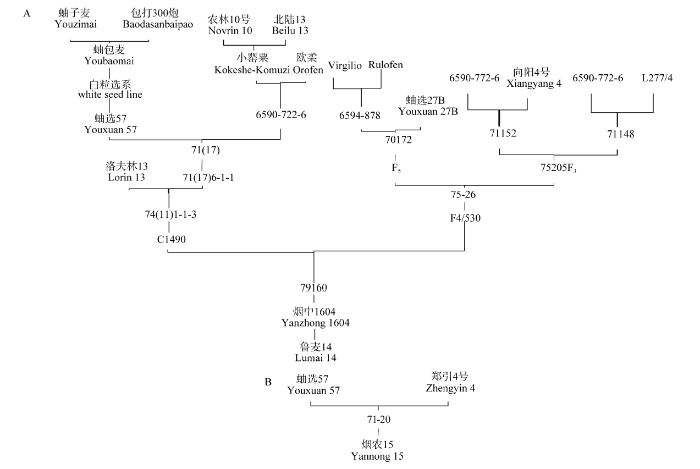 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1青农2号亲本鲁麦14(A)和烟农15(B)的系谱
该系谱根据方正等[
-->Fig. 1Pedigree of Lumai 14 (A) and Yannong 15 (B)
The pedigree was summarized from Fang et al. [
-->
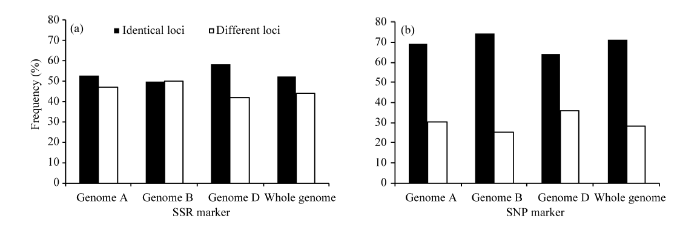
350个SSR和26 026个SNP标记的全基因组扫描结果揭示出亲本间遗传信息有所差异, 其中331个SSR和20 802个SNP可定位在小麦不同染色体上; SSR多态性位点146个, 占44.11% (146/331), SNP多态性位点5924个, 占28.48% (5924/20 802)。从不同基因组分子标记分布看, SSR在A、D基因组的亲本相同位点多于差异位点, 在B基因组两者异同位点相当(图2-a); SNP在A、B、D基因组上均表现两亲本相同位点远多于差异位点(图2-b)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2鲁麦14和烟农15 SSR (a)和SNP位点(b)的异同比例和染色体组分布
-->Fig. 2Percentage and genomic distribution of identical and different SSR (a) and SNP loci (b) between Lumai 14 and Yannong 15
-->
2.2 鲁麦14和烟农15对青农2号的遗传贡献
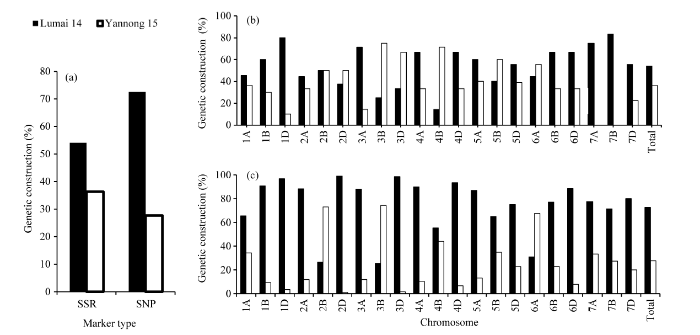
经全基因组扫描发现鲁麦14对青农2号的遗传贡献大于烟农15 (图3和表2), 鲁麦14和烟农15传递至青农2号的SSR位点分别占54.11%和36.30%,而传递的SNP位点分别占72.55%和26.98% (图3-a), 两种标记间存在较大差距。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3用SSR和SNP标记分析鲁麦14和烟农15对青农2号的贡献
(a) 全基因组水平; (b) SSR标记在不同染色体的分布; (c) SNP标记在不同染色体的分布。
-->Fig. 3Genetic contribution of Lumai 14 and Yannong 15 to Qingnong 2 revealed by SSR and SNP markers
(a) Whole genome level; (b) Chromosomal distribution of SSRs; (c) Chromosomal distribution of SNPs.
-->
Table 2
表2
表2鲁麦14和烟农15在21条染色体对青农2号的遗传贡献
Table 2Genetic contribution of Lumai 14 and Yannong 15 on Qingnong 2 with SSR and SNP at 21 chromosomes
| 染色体 Chr. | SSR | SNP | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 差异位点 Differential loci | 鲁麦14 Lumai 14 | 贡献率 Contribution (%) | 烟农15 Yannong 15 | 贡献率 Contribution (%) | 差异位点 Differential loci | 鲁麦14 Lumai 14 | 贡献率 Contribution (%) | 烟农15 Yannong 15 | 贡献率 Contribution (%) | |||||||||||||||||
| 1A | 11 | 5 | 45.45 | 4 | 36.36 | 660 | 433 | 65.61 | 227 | 34.39 | ||||||||||||||||
| 1B | 10 | 6 | 60.00 | 3 | 30.00 | 685 | 620 | 90.51 | 65 | 9.49 | ||||||||||||||||
| 1D | 10 | 8 | 80.00 | 1 | 10.00 | 145 | 140 | 96.55 | 5 | 3.45 | ||||||||||||||||
| 2A | 9 | 4 | 44.44 | 3 | 33.33 | 270 | 238 | 88.15 | 32 | 11.85 | ||||||||||||||||
| 2B | 6 | 3 | 50.00 | 3 | 50.00 | 302 | 80 | 26.49 | 220 | 72.85 | ||||||||||||||||
| 2D | 8 | 3 | 37.50 | 4 | 50.00 | 325 | 321 | 98.77 | 3 | 0.92 | ||||||||||||||||
| 染色体 Chr. | SSR | SNP | ||||||||||||||||||||||||
| 差异位点 Differential loci | 鲁麦14 Lumai 14 | 贡献率 Contribution (%) | 烟农15 Yannong 15 | 贡献率 Contribution (%) | 差异位点 Differential loci | 鲁麦14 Lumai 14 | 贡献率 Contribution (%) | 烟农15 Yannong 15 | 贡献率 Contribution (%) | |||||||||||||||||
| 3A | 7 | 5 | 71.43 | 1 | 14.29 | 362 | 318 | 87.85 | 43 | 11.88 | ||||||||||||||||
| 3B | 4 | 1 | 25.00 | 3 | 75.00 | 202 | 51 | 25.25 | 150 | 74.26 | ||||||||||||||||
| 3D | 3 | 1 | 33.33 | 2 | 66.67 | 66 | 65 | 98.48 | 1 | 1.52 | ||||||||||||||||
| 4A | 3 | 2 | 66.67 | 1 | 33.33 | 173 | 155 | 89.60 | 18 | 10.40 | ||||||||||||||||
| 4B | 7 | 1 | 14.29 | 5 | 71.43 | 134 | 73 | 54.48 | 58 | 43.28 | ||||||||||||||||
| 4D | 3 | 2 | 66.67 | 1 | 33.33 | 15 | 14 | 93.33 | 1 | 6.67 | ||||||||||||||||
| 5A | 5 | 3 | 60.00 | 2 | 40.00 | 319 | 277 | 86.83 | 42 | 13.17 | ||||||||||||||||
| 5B | 5 | 2 | 40.00 | 3 | 60.00 | 233 | 151 | 64.81 | 81 | 34.76 | ||||||||||||||||
| 5D | 18 | 10 | 55.56 | 7 | 38.89 | 44 | 33 | 75.00 | 10 | 22.73 | ||||||||||||||||
| 6A | 9 | 4 | 44.44 | 5 | 55.56 | 390 | 121 | 31.03 | 263 | 67.44 | ||||||||||||||||
| 6B | 6 | 4 | 66.67 | 2 | 33.33 | 511 | 393 | 76.91 | 117 | 22.90 | ||||||||||||||||
| 6D | 3 | 2 | 66.67 | 1 | 33.33 | 77 | 68 | 88.31 | 6 | 7.79 | ||||||||||||||||
| 7A | 4 | 3 | 75.00 | 0 | 0.00 | 369 | 286 | 77.51 | 83 | 22.49 | ||||||||||||||||
| 7B | 6 | 5 | 83.33 | 0 | 0.00 | 607 | 433 | 71.33 | 166 | 27.35 | ||||||||||||||||
| 7D | 9 | 5 | 55.56 | 2 | 22.22 | 35 | 28 | 80.00 | 7 | 20.00 | ||||||||||||||||
| Total | 146 | 79 | 54.11 | 53 | 36.30 | 5924 | 4298 | 72.55 | 1598 | 26.98 | ||||||||||||||||
新窗口打开
在染色体水平, 鲁麦14对青农2号的遗传贡献, SSR位点比例最低为4B的14.29%, 最高为7B的83.33%; SNP位点比例最低为3B的25.25%, 最高为2D的98.77%。SSR和SNP标记的遗传贡献在多数染色体水平趋势一致, 如在3B、6A染色体均表现为鲁麦14的遗传贡献小于烟农15, 在1D、3A、4A、4D、5A、6B、6D、7A、7B、7D染色体均表现为鲁麦14贡献大幅超过烟农15 (图3-b), 但2A, 2B, 2D, 3D, 4B, 5B等染色体两种标记结果不一致。以SSR标记检测显示, 鲁麦14遗传贡献大于50%的染色体有1B、1D、3A、4A、4D、5A、5D、6B、6D、7A、7B和7D, 而以SNP标记检测显示除2B、3B、6A外的其他18条染色体上鲁麦14的遗传贡献均超过50%。
2.3 亲本遗传信息在青农2号染色体上的分布
亲本遗传信息主要以染色体大片段形式传递到子代, 如1A、1B、2B、3A、4A、5A、6A、6B、6D、7A、7B等染色体。有的染色体大片段是由位于同一遗传距离的数十甚至数百个SNP位点构成, 如1A、1B、3D、5A等的大片段; 有的染色体大片段在整合遗传图谱中有较多重组, 但在青农2号组合中表现为保守遗传, 如2D、6D、7A、7B等的大片段(图4)。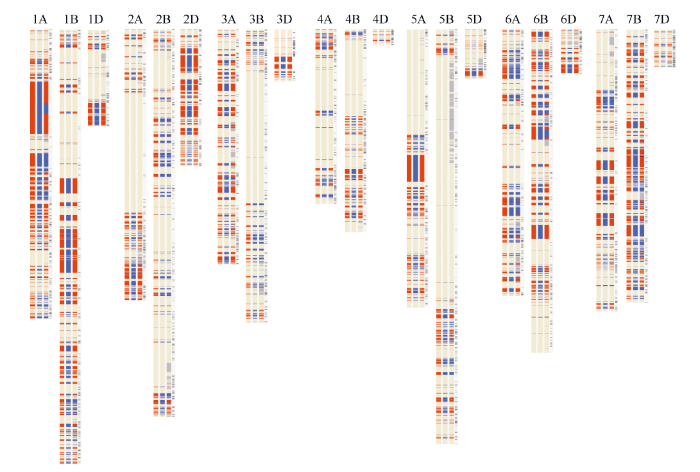 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4青农2号的遗传组成图谱
每组图谱前3根表示染色体, 自左向右依次代表鲁麦14、烟农15和青农2号, 第4根表示SNP在整合遗传图中的染色体定位。红色、蓝色、浅黄色、灰色分别表示鲁麦14特有、烟农15特有、共有、非亲本等位变异。
-->Fig. 4Genetic composition map of Qingnong 2
The first three columns (from left to right) in each group indicate chromosomes of Lumai 14, Yannong 15, and Qingnong 2, and the fourth column shows the chromosomal locations of SNP in the integrated genetic map. Red, blue, light yellow, and gray indicate alleles that Lumai 14 unique, Yannong 15 unique, shared with them and not from the two parents.
-->
青农2号的1A、1B、1D、2A、2D、3A、3D、4A、4D、5A、6B、6D、7A、7B、7D染色体上遗传信息绝大部分来自鲁麦14, 在2B、3B、4B、5B、6A染色体上则分布着较多烟农15的遗传位点。此外, 在5B和7A染色体上有较大片段的非亲本来源信息(图4)。
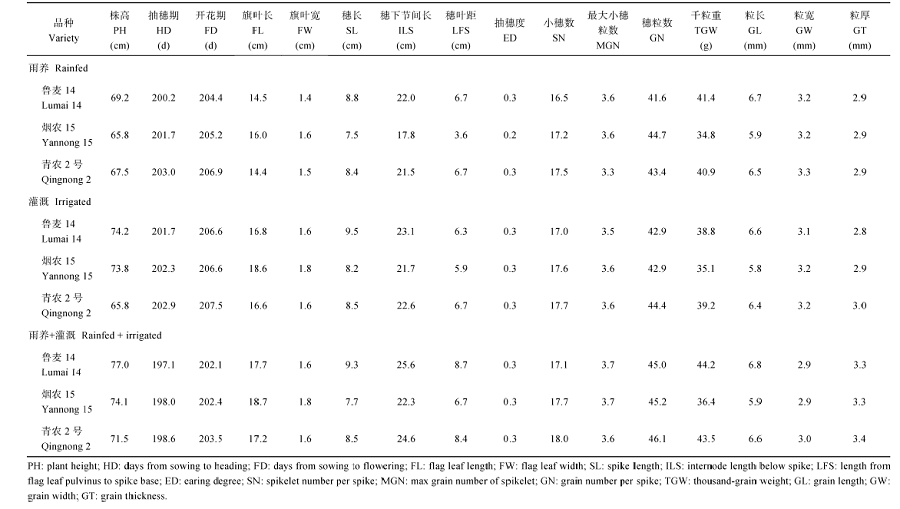 |
2.4 青农2号及亲本的农艺性状表现
农艺性状最优无偏估计值(BLUP)表明, 青农2号的株高在雨养条件下, 介于亲本之间, 但在灌溉条件和雨养+灌溉条件下, 均低于低值亲本烟农15。青农2号的抽穗期、开花期长于高值亲本烟农15。青农2号的旗叶长和宽跟低值亲本鲁麦14接近。穗长介于亲本之间, 穗下节间长、穗叶距均与高值亲本鲁麦14接近, 低值亲本烟农15的穗叶距在雨养和灌溉条件下差异明显, 而青农2号和鲁麦14均变化较小。青农2号的有效小穗数和穗粒数更接近于高值亲本烟农15。青农2号的千粒重、粒长性状接近于高值亲本鲁麦14, 其粒宽和粒厚在不同水分条件下略有差异, 但均为高值水平。3 讨论
3.1 SSR标记和SNP标记在基因组研究的应用比较
SSR标记[6-7,16-18]和SNP标记[19, 25-32]分别作为第2代和第3代分子标记, 对作物基因组研究的发展有重要作用。在玉米[34]、葡萄[35]和鼠尾芥[36]上的研究均表明, SSR标记的遗传多样性高于SNP标记, 在揭示遗传分化方面有较高相关性。张利莎等[37]认为EST-SSR和SNP标记对麦芽纯度的检测结果有效且可靠, 但在检测灵敏度、效率和成本方面, SNP标记较EST-SSR标记更具优势。近年来, 随着高通量测序、高密度芯片技术的发展, SNP检测效率快速提升, 成本大幅降低, 在今后的群体遗传学研究中将成为主流[38]。本研究表明, SSR标记在鲁麦14和烟农15间多态性比例高于SNP标记。在揭示亲本遗传贡献方面(图3), 两种标记揭示的规律基本一致, 但贡献率存在较大区别。SSR标记主要位于重复序列区域, SNP标记可能遍布基因组的更多区域, 包括基因编码区等, 虽然两者位于染色体的不同区域, 但其反映的遗传贡献信息却基本一致。这可能与作物育种中亲本染色体大片段甚至整条染色体的遗传传递有关。在品种遗传组成和亲本遗传贡献方面, 采用基因组覆盖度高、检测通量水平高的SNP标记开展研究, 可以达到从基因组水平对育种更好的理解。
3.2 品种改良对基因组的影响
作物品种改良是人类有目的、有计划地通过人工杂交、诱变, 促进基因重组交换、定向改良作物的过程[13]。株型、穗形、高产、优质等质量或数量性状成为育种家的选择目标。这种人工选择对基因组的影响是什么?对青农2号的研究结果表明, 子代基因组大部分遗传信息来自鲁麦14 (SSR标记: 54.11%; SNP标记: 72.55%), 来自另一亲本烟农15的遗传信息相对较少。理论上系谱地位相等的亲本应对子代的贡献一致, 但在本研究中出现“偏向性选择”现象, 而且在染色体水平也存在这种现象, 比如在2B、3B、6A染色体青农2号的遗传信息主要偏向于烟农15, 在1A、1B、1D、2A、2D、3A、3D、4A、4D、5A、6D、7A、7B、7D等染色体主要偏向于鲁麦14。“偏向性选择”现象在作物育种中比较普遍, Lai等[14]发现玉米品种Zheng 58获得父辈亲本478和Inbred X的遗传信息分别为43%和57%, 而获得祖辈亲本8112和5003的遗传信息分别为31%和12%; 邹少奎等[18]对周麦23的研究表明, 亲本周麦13的遗传贡献(63.04%)远高于另一亲本新麦9号(36.96%)。对表型性状目的性很强的定向改良, 应为导致子代遗传信息偏向某一亲本的主要原因。此外, 图4显示青农2号亲本间的重组事件在基因组中分布不均匀。如1A染色体可见多处亲本间重组事件, 但在Wang等[24]的整合遗传图谱中同一遗传距离处, 鲁麦14和烟农15也几乎不交换或仅有少数重组; 在1B染色体上, 绝大部分为鲁麦14遗传信息, 仅在端部有极少数重组; 1D、2D、3D、4D等几乎整条染色体从鲁麦14传递至青农2号; 2B、6A染色体绝大部分为烟农15遗传信息, 仅有少数重组事件; 重组较多的有3B、4B、5B、6B、7B等B基因组染色体。此外, 还发现即使是重组较多的染色体, 也多表现为较大染色体区段间的重组。表明青农2号的杂交和育种选择, 并非亲本基因组均匀且大范围的重组和交换, 而是较大染色体片段间的重组甚至整条染色体的替换。而通过百万数量级SNP完成的Zheng 58的遗传组成图谱, 更加清晰地体现了染色体大片段遗传传递的现象[14]。表明作物育种选择存在很强的选择牵连效应[16], 许多不良性状的连锁遗传难以打破, 同时也反映了优良遗传背景的选择对作物亲本选配具有重要意义。
4 结论
两种分子标记揭示的亲本对青农2号的遗传贡献趋势一致, 但亲本间SSR标记的多态性比例高于SNP标记。鲁麦14对青农2号的遗传贡献大于烟农15, 偏离1∶1理论值, 表明育种选择存在较强的偏向性, 两亲本向子代的遗传信息传递以染色体大片段重组和整合为主要方式。致谢: 感谢中国农业科学院作物科学研究所郝晨阳博士、河南科技学院王玉泉博士在小麦90k芯片分析过程中给予的帮助; 感谢中国农业科学院作物科学研究所张学勇研究员课题组和泰安市农业科学研究院王瑞霞博士在河南新乡和山东泰安供试材料农艺性状采集过程中给予的帮助。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | . . |
| [3] | . . |
| [4] | . . |
| [5] | . . |
| [6] | . |
| [7] | . . |
| [8] | . . |
| [9] | . . |
| [10] | . . |
| [11] | . . |
| [12] | . |
| [13] | . . |
| [14] | . |
| [15] | . |
| [16] | . . |
| [17] | . . |
| [18] | . |
| [19] | . |
| [20] | [J]. |
| [21] | . |
| [22] | . |
| [23] | . |
| [24] | , Morten Lillemo, Mather D, Appels R, Dolferus R, Brown-Guedira G, Koral A, Akhunova A R, Feuillet C, Salse J, Morgante M, Pozniak C, Luo M C, Dvorak J, Morell M, Dubcovsky J, Ganal M, Tuberosa R, Lawley C, Mikoulitch L, Cavanagh C, Edwards K J, Hayden M, Akhunov E. Characterization of polyploid wheat genomic diversity using a high-density 90,000 single nucleotide polymorphism array. |
| [25] | . |
| [26] | . |
| [27] | . . |
| [28] | . |
| [29] | . . |
| [30] | . . |
| [31] | . |
| [32] | . |
| [33] | . . |
| [34] | . |
| [35] | . |
| [36] | . |
| [37] | . . |
| [38] | . |
