山西省农业科学院棉花研究所, 山西运城 044000;
Cloning and Expression Analysis of Galactosyltransferase Gene GhGalT1 Promoter in Cotton
QINLi-Xia, LIJing, ZHANGHuan-Yang, LISheng, ZHUMeng-Jie, JIAOGai-Li, WUShen-Jie通讯作者:
收稿日期:2017-04-11
接受日期:2017-09-10
网络出版日期:2018-02-12
版权声明:2018作物学报编辑部作物学报编辑部
基金资助:
作者简介:
-->
展开
摘要
关键词:
Abstract
Keywords:
-->0
PDF (2741KB)元数据多维度评价相关文章收藏文章
本文引用格式导出EndNoteRisBibtex收藏本文-->
糖基转移酶(glycosyltransferase, GT, EC 2.4.x.y)广泛存在于植物体内, 负责催化小分子化合物(激素、次级代谢物等)的糖基化, 将活性糖基从供体转移到受体(小分子化合物), 从而改变这些化合物的生物化学性质[1,2]。按照序列同源性及催化底物特性及糖苷键立体化学性质将GTs分为94个不同的家族 (http://www.cazy.org/fam/acc_GT.html)[3]。越来越多的证据显示GTs在调节植物激素和植物抗逆反应中发挥重要作用。
在拟南芥中过量表达UGT84B1导致转基因植株表现出生长素缺陷表型, 植株矮化, 多分枝, 叶片皱缩, 根系失去向地性生长[4,5]。UGT74E2在体外能糖基化生长素IBA, UGT74E2过量表达转基因拟南芥植株表现出矮化、多分枝, 抗盐、抗旱性增强[6]。在烟草中过量表达玉米素合成相关糖基转移酶ZOG1, 导致转基因比非转基因材料需多添加10倍的玉米素才能诱导愈伤组织形成芽[7]。拟南芥UGT76C1、UGT76C2、UGT85A1、UGT73C1和UGT73C5参与调控植物对细胞分裂素的响应[8,9]。UGT73C5和UGT73C6催化油菜素内酯的糖基化, 在拟南芥中过量表达UGT73C5植株表现出油菜素内酯缺陷表型, 同时伴随着高水平的油菜素内酯糖苷物的积累, 而在突变体株系中则未检测到油菜素内酯糖苷物[10,11,12]。从拟南芥分离出的叶片伤口处检测到茉莉酸糖苷的富集, 表明GTs在激素调节和病原菌防御之间也起重要作用[13]。UGT73B1、UGT73B2、UGT73B3和UGT71C1功能缺失突变体增强了植物对氧化胁迫的抗性[14,15,16]。在拟南芥中过量表达水杨酸合成糖基转移酶AtSGTl导致植株体内水杨酸及其糖苷水平降低, 进而增加对半活体营养侵染型病原菌假单胞菌的敏感性[17]。虽然以上研究已表明糖基转移酶参与植物耐逆过程, 但对其具体作用机理尚不清楚, 糖基转移酶参与植物耐逆过程的分子机制有待深入探索。
非生物胁迫(干旱、土壤盐渍化、低温冷害等)是限制农业生产、威胁作物产量和品质的重要因素, 而传统育种方法并不能精准有效地提高植物耐受逆境能力。因此, 通过植物基因工程技术结合传统育种手段, 使抗逆基因在目标作物中特异表达, 以提高作物对逆境胁迫的抗性, 逐渐成为作物育种中的一个重要手段。启动子作为启动目标基因表达的关键调控元件, 主要有组成型启动子、组织特异型启动子和诱导型启动子[18]。虽然组成型启动子在启动外源基因表达中具有高效、广谱和稳定性强等优点, 但是由于高效、持续表达目标基因容易造成一些负面影响(如植株生长异常等), 而诱导型启动子是在特定条件下启动目标基因的表达, 很大程度上缓解了由于大量异源蛋白的高表达对植物生长造成的伤害[19,20]。
棉花的生长和发育过程经常受到高盐、干旱等非生物胁迫因素的影响, 导致产量降低。到目前为止, 在棉花中参与调节激素平衡及抗逆胁迫方面的糖基转移酶尚未见报道。我们之前从棉花cDNA文库中分离了一个棉花糖基转移酶GhGalT1基因[21], 为研究其在植物逆境应答中的功能, 本研究分离克隆了GhGalT1基因起始密码子ATG上游539 bp的启动子序列。通过PlantCARE软件分析发现其包含许多与逆境、激素等相关的顺式作用元件, 推测该启动子受干旱、热、脱水、激素等胁迫诱导表达, 为此我们构建了含有该启动子的重组表达载体pBI121-pGhGalT1-GUS, 并通过浸花法转化拟南芥对其功能进行了验证, 以期丰富植物诱导型启动子种类, 为植物抗逆遗传育种改良提供理论依据。
1 材料与方法
1.1 植物材料
利用本实验室保存的陆地棉(Gossypium hirsutum L.)品种Coker 312克隆GhGalT1基因的启动子, 拟南芥(Arabidopsis thaliana, 哥伦比亚型)用于启动子重组质粒的转化与功能验证。1.2 棉花基因组DNA的提取
选取饱满的Coker 312棉花种子, 以70%乙醇处理1 min, 10%过氧化氢(H2O2)处理1.5 h, 无菌水冲洗3~4次, 随后加入适量无菌水浸泡至种子根尖露白。去掉种皮, 置1/2 MS培养基上萌发, 28ºC培养5~7 d后, 选取生长健壮的幼苗移栽至土壤中, 待幼苗长出2片真叶时, 取2 g左右幼嫩叶片利用CTAB法提取基因组DNA[22]。1.3 GhGalT1基因启动子的克隆及顺式作用元件分析
以GhGalT1编码区序列为探针, Blast搜索中国农业科学院棉花研究所网站的陆地棉(Gossypium hirsutum)基因组序列数据库(http://cgp.genomics.org. cn/page/species/blast.jsp), 将该基因上游2 kb左右序列作为目标区域。利用在线顺式作用元件分析工具PLACE (http://www.dna.affrc.go.jp/PLACE/signalup.htm)和 PlantCARE (http://bioinformatics.psb.ugent.be/webtools/
plantcare/html/)分析启动子序列, 预测顺式作用元件。
以Coker 312的基因组DNA为模板, 使用PrimeSTAR HS DNA Polymerase高保真酶, PCR扩增GhGalT1基因的启动子目的片段, 命名为pGhGalT1, 琼脂糖凝胶电泳检测扩增条带大小。扩增体系为5×Prime STAR 缓冲液 (Mg2+ plus) 4 μL,dNTPs (各2.5 mmol L-1) 4 μL, 模板DNA (500 ng μL-1) 1 μL, 引物pGhGalT1-F (10 μmol L-1) 1 μL, pGhGalT1-R (10 μmol L-1) 1 μL (表1), PrimeSTAR HS DNA聚合酶 (2.5 U μL-1) 0.2 μL, 用ddH2O补至20 μL。反应程序为98ºC 10 s, 55ºC 15 s, 72ºC 1 min, 30个循环; 72ºC 10 min。将扩增得到的序列连接至pBluescript(pSK)载体上测序验证, 与棉花基因组数据库序列比对。
Table 1
表1
表1本试验中所用引物序列
Table 1Primers used in this study
| 引物名称 Primer name | 上游引物序列 Forward primer sequence (5′-3′) | 下游引物序列 Reverse primer sequence (5′-3′) |
|---|---|---|
| pGhGalT1 | GGGGTCGACTAACAACGGTGAAGAAAGAA | GGTGGATCCAACTGTGAATTTGAAGAGAAG |
| GUS | ATGTTACGTCCTGTAGAAACC | TCATTGTTTGCCTCCCTGCTGC |
| NPTII | ATGATTGAACAAGATGGATTGC | TCAGAAGAACTCGTCAAGAAGGC |
| AtActin2 | GAAATCACAGCACTTGCACC | AAGCCTTTGATCTTGAGAGC |
| GUS-RT | ATGTTCTGCGACGCTCAC | CCCGGCTAACGTATCTGT |
新窗口打开
1.4 GhGalT1启动子载体构建、拟南芥转化及阳性植株检测
用Sal I和BamH I分别酶切植物表达载体pBI101和扩增的GhGalT1基因启动子目的片段pGhGalT1后, 进行连接, 获得重组质粒pBI101- pGhGalT1-GUS (图1), 转化农杆菌GV3101菌株。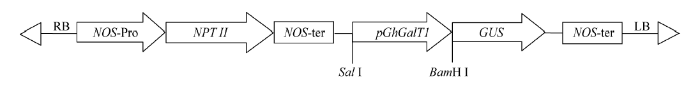 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1pBI101-pGhGalT1-GUS表达载体构建示意图
-->Fig. 1Map of construction of the pBI101-pGhGalT1-GUS expression vector
-->
采用农杆菌介导的浸花法转化拟南芥[23]。经卡那霉素抗性筛选后获得T1代转基因拟南芥。取T1代转基因植株幼苗叶片, 利用SDS法提取基因组DNA, 并以此为模板通过PCR扩增检测阳性植株, 扩增引物为pGhGalT1-F和pGhGalT1-R、GUS-F和GUS-R和抗性标记基因NPTII-F和NPTII-R (表1), 产物大小分别为539、1812和795 bp。选取PCR检测呈阳性的单株收获种子并加代, 再经卡那霉素抗性筛选及PCR检测得到T3代纯合转基因阳性株系。
1.5 GUS组织化学染色
取野生型和T3代转基因拟南芥植株种子, 经10% NaClO溶液表面消毒后, 分别点播于MS培养基上, 4ºC春化处理2 d, 于恒温光照培养箱(16 h光照/8 h黑暗, 22~24ºC)培养, 分别取生长5、10和15 d幼苗、茎、花和角果、成熟叶片等组织进行GUS组织化学染色[24]。选取长势一致的幼苗浸入GUS染液(含10 mmol L-1 EDTA, 100 mmol L-1 NaH2PO4·H2O, 0.5% Triton X-100, 1 mmol L-1 K3Fe(CN)6, 1 mmol L-1 K4Fe(CN)6·3H2O, 0.1% X-Gluc)中, 于37ºC染色6~8 h, 除去染液。再用70%乙醇室温脱色3~5 h, 其间更换70%乙醇2~3次, 直至各组织的色素完全退去。取样品在Leica MZ16f体视显微镜下观察, 照相。
取适量生长10 d的转基因拟南芥幼苗分别移至含150 mmol L-1 NaCl、300 mmol L-1甘露醇、100 μmol L-1 6-BA、50 μmol L-1茉莉酸甲酯(MeJA)和500 nmol L-1油菜素内酯(BL)的MS液体培养基中处理6 h后, 进行GUS染色分析, 本试验重复3次。
1.6 GUS酶活测定
GUS活性的荧光检测方法参照Jefferson [25]。将生长10 d幼苗分别用MS液体和添加有150 mmol L-1 NaCl、300 mmol L-1甘露醇、100 μmol L-1 6-BA、50 μmol L-1 MeJA或500 nmol L-1 BL的MS液体培养基诱导6 h。取0.1 g样品于预冷的研钵中, 加0.5 mL预冷的研磨缓冲液(100 mmol L-1 K3PO4, pH 7.8, 1 mmol L-1 EDTA, 1% Triton X-100), 迅速研磨成匀浆后转入预冷的1.5 mL离心管中, 2130×g, 4ºC离心10 min, 上清液即为GUS蛋白粗提取液。以4-MUG为作用底物进行酶促反应后, 反应产物在365 nm的激发光下测定455 nm的荧光强度(HITACHI F-2500), 考马斯亮蓝法测定蛋白质含量。1.7 GUS基因的表达分析
取适量生长10 d携有启动子序列的拟南芥幼苗分别移至含150 mmol L-1 NaCl、300 mmol L-1甘露醇、100 μmol L-1 6-BA、50 μmol L-1 MeJA或500 nmol L-1 BL的MS液体培养基中处理6 h后收取幼苗材料。用RNA-Solv Reagent (Omega)提取幼苗总RNA, 经DNase (RQ1 RNase-Free DNase, Promega)消化后, 用逆转录酶(M-MLV Reverse Transcriptase, Promega)合成cDNA。应用实时荧光定量RT-PCR技术(MJ Research, Opticon 2), 以cDNA为模板, 用GUS基因特异性引物GUS-RTF和GUS-RTR (表1)和Real-time PCR Master Mix (TOYOBO)进行PCR反应。以拟南芥的Actin2作为内参基因, 目标基因每一个循环的扩增都被SYBR-Green荧光检测, 具体步骤及分析参照Li等[26]的方法, 重复3次, 统计分析试验结果。2 结果与分析
2.1 GhGalT1启动子的克隆及顺式作用元件分析
靠近GhGalT1编码区开放阅读框5′上游539 bp (图2)区域序列具有TATA-box、CAAT-box等真核生物启动子基本调控元件, 此外还包含多种逆境响应功能元件, 如干旱诱导的MYB结合元件MBS、脱水响应元件MYCCONSE、热应答元件HSE、防御与胁迫应答元件TC-rich repeats、MeJA响应元件CGTCA-motif、调控生物节律元件以及多种光响应相关元件等(图3和表2)。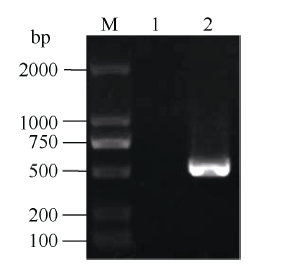 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2棉花GhGalT1基因启动子的克隆
M: DNA marker DL2000; 1: 阴性对照; 2: pGhGalT1目的片段。
-->Fig. 2Amplified fragment of GhGalT1 gene promoter in cotton
M: DNA marker DL2000; 1: negative control; 2: GhGalT1 promoter sequence.
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3GhGalT1启动子序列及预测的顺式作用元件
-->Fig. 3Sequence and predicted cis-acting elements of GhGalT1 promoter
-->
Table 2
表2
表2GhGalT1启动子主要顺式作用元件及预测的功能
Table 2The cis-acting elements and predicted functions in the GhGalT1 promoter sequence
| 顺式作用元件 cis-acting element | 核心序列 Core sequence | 位置 Location (bp) | 功能 Function |
|---|---|---|---|
| 3-AF binding site | CACTATCTAAC | -472 to -462 | 光响应元件 Light responsive element |
| ATCT-motif | AATCTAATCT | -401 to -392 | 光响应元件 Part of a conserved DNA module involved in light responsiveness |
| Box 4 | ATTAAT | -505 to -499 | 光响应元件 Part of a conserved DNA module involved in light responsiveness |
| Box 1 | TTTCAAA | -387 to -381 | 光响应元件Light responsive element |
| GAG-motif | AGAGAGT | -246 to -240 | 光响应元件 Part of a light responsive element |
| MYCCONSE | CACCTG | -298 to -293 | 脱水响应 Dehydration responsiveness |
| CGTCA-motif | CGTCA | -175 to -171 | 茉莉酸甲酯响应元件 MeJA responsive element |
| MBS | CAACTG | -141 to -136 | 干旱诱导的MYB 结合位点 MYB binding site involved in drought-inducibility |
| TC-rich repeats | ATTTTCTTCA | -460 to -451 | 防御及胁迫响应元件 Cis-acting element involved in defense and stress responsiveness |
| HSE | AAAAAATTTC | -249 to -259 | 热胁迫响应元件 Heat stress responsiveness |
| Sp 1 | CC(G/A)CCC | -152 to -147 | 光响应元件Light responsive element |
| Skn-1 motif | GTCAT | -520 to -524, -325 to -321, -206 to -202 | 胚乳特异表达 Endosperm specific expression |
| TCCC-motif | TCTCCCT | -156 to -148 | 光响应元件 Light responsive element |
| Circadian | CAANNNNATC | -364 to -355 | 调控生物节律 Cis-acting regulatory element involved in circadian control |
新窗口打开
2.2 GhGalT1启动子载体构建及拟南芥转化与阳性植株的鉴定
为了研究GhGalT1启动子的活性及其表达模式, 将该启动子扩增产物pGhGalT1 (图2)及植物表达载体pBI101分别用Sal I和Bam HI双酶切后连接, 经PCR扩增和酶切检测验证获得正确的重组质粒(图4-A和B)。通过农杆菌介导法转化拟南芥, 获得转基因植株。经卡那霉素抗性筛选和PCR检测呈阳性的单株收获种子并加代(图5, 图6-A~C), 获得T3代纯合转基因阳性株系(图6-D)。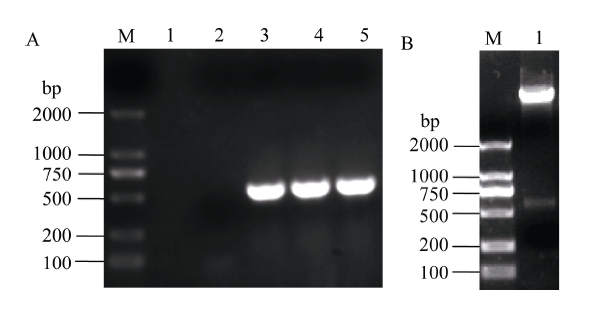 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4 GhGalT1启动子载体的构建
M: DNA marker DL2000; A: pBI101-pGhGalT1-GUS质粒的PCR检测; 1和2: 阴性对照; 3: 阳性对照; 4~5: pBI101-pGhGalT1-GUS质粒; B: 重组质粒pBI101-pGhGalT1-GUS经Sal I和BamH I双酶切电泳.
-->Fig. 4Vector construction of GhGalT1 promoter
M: DNA marker DL2000; A: electrophoregram of pBI101-pGhGalT1-GUS plasmid detected by PCR; 1 and 2: negative control; 3: positive control; 4-5: pBI101-pGhGalT1-GUS plasmid; B: electrophoregram of pBI101-pGhGalT1-GUS plasmid digested by Sal I and BamH I.
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5T1代转基因拟南芥阳性植株的卡那霉素抗性筛选
箭头所指部位为具有卡那霉素抗性的绿色小苗。
-->Fig. 5Kanamycin resistance screening of T1 generation transgenic positive plants
Arrowheads indicate seedlings with kanamycin resistance.
-->
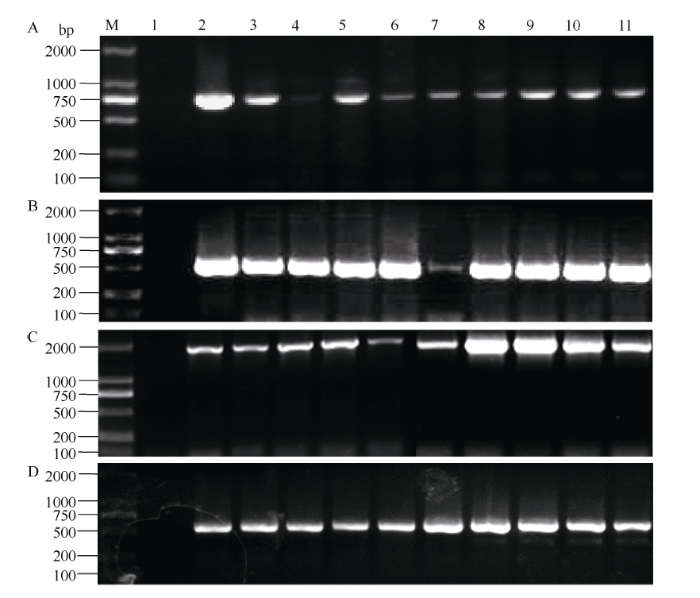 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6转基因拟南芥阳性植株的PCR检测
A~C: T1代转基因拟南芥阳性植株的PCR检测; D: T3代转基因拟南芥阳性植株的PCR检测; A: 用抗性标记基因NPTII引物扩增的产物; B和D: 用GhGalT1启动子pGhGalT1引物扩增的产物; C: 用GUS基因引物扩增的产物; 1: 野生型对照; 2: 阳性质粒对照; 3~11: 不同株系T1和T3代转基因植株。
-->Fig. 6Positive detection in transgenic Arabidopsis lines by PCR
A-C: electrophoregram of positive detection by PCR in some T1 generation of transgenic Arabidopsis with NPTII primers (A), pGhGalT1 primers (B) and GUS primers (C), respectively; D: electrophoregram of positive detection by PCR in T3 generation of transgenic Arabidopsis with pGhGalT1 primers; 1: wild type; 2: positive control; 3-11: different lines from T1 and T3 generations of transgenic plants.
-->
2.3 GUS组织化学染色
图7表明GUS信号主要在生长5、10和15 d转基因植株幼苗的主根及侧根的根尖表达, 在10 d以后的子叶及莲座叶边缘也有GUS表达, 而在茎、花、角果等部位未检测到GUS信号, 表明靠近GhGalT1编码区开放阅读框上游的539 bp的GhGalT1启动子区域可以驱动GUS基因表达, 是启动子的活性区域。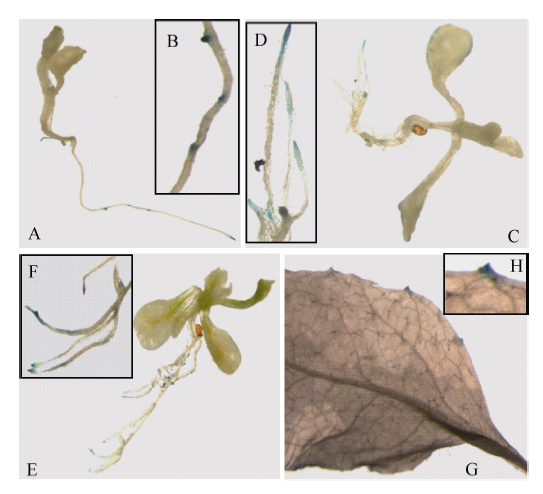 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7GhGalT1启动子转基因拟南芥的GUS活性检测
A~B: 5 d幼苗, B为A的局部放大; C~D: 10 d幼苗, D为C的局部放大; E~F: 15 d小苗, F为E的局部放大; G~H: 成熟叶片, H为G的局部放大。
-->Fig. 7Histochemical assay of GUS activity in pGhGalT1:GUS transgenic Arabidopsis
A-B: five-day-old seedlings; B: magnification of root regions of five-day-old seedlings; C-D: ten-day-old seedlings; D: magnification of root tip regions of ten-day-old seedlings; E-F: 15-day-old seedlings; F: magnification of root tip regions of 15-day-old seedlings; G-H: mature leaf; H: magnification of mature leaf regions of G.
-->
2.4 不同胁迫条件下的GUS染色及定量分析
Genevestigator网站(https://genevestigator.com/ gv/)分析显示, 与GhGalT1基因同源的拟南芥AtGalTs基因受盐、干旱、激素(6-BA、MeJA、BL等)的诱导。将生长10 d的幼苗移至分别添加150 mmol L-1 NaCl、300 mmol L-1甘露醇、100 μmol L-1 6-BA、50 μmol L-1 MeJA和500 nmol L-1 BL的MS液体培养基中处理6 h, 然后进行GUS染色分析。图8显示, 与无胁迫对照相比, 经NaCl、甘露醇和MeJA处理后, 转基因株系根中GUS信号明显减弱; 用BL处理后, 根中GUS信号显著增强; 而在6-BA处理的小苗根中GUS信号完全消失, 子叶和莲座叶中的GUS信号却明显增强。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图8盐、干旱和激素处理GhGalT1启动子转基因拟南芥的GUS活性检测
生长10 d幼苗在MS液体培养基(A~C)和分别添加150 mmol L-1 NaCl (D~E)、300 mmol L-1甘露醇(F~G)、100 μmol L-1 6-BA (H~J)、50 μmol L-1 MeJA (K~L)和500 nmol L-1 BL (M~N)的MS液体培养基处理后的GUS染色结果; 其中B和C为A的局部放大、E为D的局部放大、G为F的局部放大、I和J为H的局部放大、L为K的局部放大、N为M的局部放大。
-->Fig. 8Histochemical assay of GUS activity of GhGalT1 promoter in transgenic Arabidopsis under stresses
A-C: ten-day-old seedlings in mock treatment; D-N: ten-day-old seedlings treated with 150 mmol L-1 NaCl (D-E), 300 mmol L-1 mannitol (F-G), 100 μmol L-1 6-BA (H-J), 50 μmol L-1 MeJA (K-L) or 500 nmol L-1 BL (M-N). B, C: magnification of root tip and cotyledon in ten-day-old seedlings in mock treatment; E: magnification of root tip in ten-day-old seedlings under 150 mmol L-1 NaCl treatment; G: magnification of root tip in ten-day-old seedlings under 300 mmol L-1 mannitol treatment; I, J: magnification of root tip and cotyledon in ten-day-old seedling with 100 μmol L-1 6-BA treatment; L: magnification of root tip in ten-day-old seedlings with 50 μmol L-1 MeJA treatment; N: magnification of root tip in ten-day-old seedlings under 500 nmol L-1 BL treatment.
-->
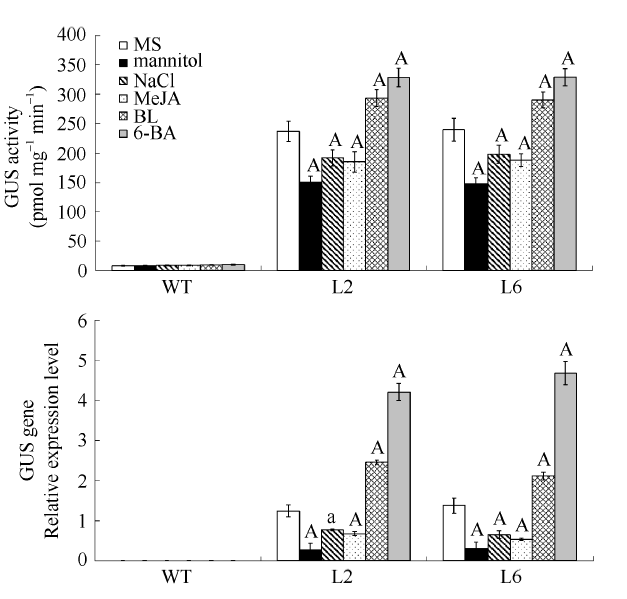
进一步定量确定启动子的表达活性, 对盐、干旱、激素(6-BA、MeJA、BL)处理过的10 d幼苗进行GUS酶活测定, 同时提取总RNA后, 对GUS基因进行实时荧光定量PCR分析其表达量。结果经NaCl、甘露醇和MeJA处理后, GUS活性及GUS基因表达量显著降低, 6-BA、BL处理后GUS活性及GUS基因表达量显著升高(图9), 表明GhGalT1基因的启动子受盐、渗透胁迫和激素(6-BA、MeJA、BL等)的诱导。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图9胁迫条件下转基因拟南芥GUS定量分析
mannitol: 甘露醇; NaCl: 氯化钠; MeJA: 茉莉酸甲酯; BL: 油菜素内酯; 6-BA: 6-苄氨基嘌呤。误差棒表示±标准差, n > 50。柱子上大、小写字母表示胁迫处理与对照在0.01和0.05概率水平的差异显著(t检验)。WT: 野生型; L2和L6: pGhGalT1:GUS转基因拟南芥株系2和株系6。
-->Fig. 9Quantification of GUS activity and GUS gene in transgenic Arabidopsis under different stresses
NaCl: sodium chloride; MeJA: methyl jasmonate; BL: brassinolide; 6-BA: 6-Benzylaminopurine. Mean values and SE (bar) were shown from three independent experiments (n > 50). The capitals and lowercase indicate significant difference between stress treatment and the control at the 0.01 and 0.05 probability levels, respectively (t-test). WT: wild type; L2 and L6: pGhGalT1:GUS transgenic lines 2 and 6.
-->
3 讨论
真核生物基因表达受染色体水平上的基因活化调节、转录水平、转录后水平和翻译水平的调控, 外源基因在受体内的表达受到诸多因素的影响, 如插入位置、拷贝数、启动子类型等, 但很大程度上依赖于外源基因的启动子[27]。目前, 研究启动子的方法主要有生物信息学分析及试验分析两种。利用生物信息学分析启动子序列, 预测其中的顺式作用元件种类、数目及位置, 可以为进一步确定启动子的功能奠定基础。试验分析主要有瞬时表达、5′端片段缺失分析[28]、凝胶阻滞[29]、酵母单杂交[30]等方法。本研究采用生物信息学分析和启动子融合GUS基因瞬时表达相结合的方法, 研究GhGalT1基因启动子的重要顺式作用元件和表达特性。通过生物信息学分析发现, 在GhGalT1启动子区域存在多种重要的非生物胁迫应答元件, 例如响应干旱、防御与胁迫等逆境的元件。Genevestigator网站(https://genevestigator.com/gv/)分析也显示与GhGalT1基因同源的拟南芥AtGalTs基因受盐、干旱、激素(6-BA、MeJA、BL等)的诱导。GUS组织化学染色表明邻近GhGalT1基因开放阅读框5′端上游的539 bp启动子区段是其活性核心区域; 同时生物信息学分析结果也显示该活性区域中包含了响应干旱和渗透胁迫的顺式作用元件。我们对转539 bp片段启动子拟南芥进行胁迫处理, 结果发现在NaCl、甘露醇和MeJA处理后, 转基因株系中GUS信号、GUS酶活性及GUS表达量明显降低。在BL处理后, 其活性及表达量显著增加, 而在6-BA处理后, GUS信号由根尖转移至子叶及莲座叶边缘部位且GUS活性及表达量显著升高。推测GhGalT1基因的启动子响应盐、干旱和激素胁迫应答, 可能是通过结合干旱诱导型MYB结合元件MBS、热应答元件HSE、防御与胁迫应答元件TC-rich repeats、MeJA响应元件CGTCA-motif来调节的。
利用诱导性启动子在特定条件下驱动相应基因的高效表达, 增强植物对不良环境条件的抵抗性, 近年来逐渐成为研究热点。目前已有一些关于植物诱导型启动子的研究报道, 如马铃薯损伤诱导型启动子Wun1[31]、柑橘冷诱导型启动子pPtcor8[32]、马铃薯光诱导型启动子Sgt1p[33]、拟南芥冷诱导型启动子CBF3[34]、拟南芥逆境诱导型启动子rd29A[35]、棉花受多逆境诱导表达的GhWRKY64基因启动子[36]、小麦干旱和渗透胁迫诱导型启动子TaPP2AbB″-α[37]等。这些能够同时受多种因素诱导表达的启动子被称为多元调控启动子, 目前能够广泛应用于转基因研究的多元调控启动子仍然比较少, 而本研究所克隆的棉花GhGalT1启动子受干旱和高盐、激素诱导表达, 属于多逆境诱导型启动子, 为了将该启动子更精确地用于棉花遗传改良, 后续的工作将对该启动子进行缺失片段分析, 确定对盐、干旱及不同激素响应的顺式作用元件及功能区, 有望进一步应用于植物遗传育种改良研究。
4 结论
从陆地棉品种Coker 312基因组DNA中克隆了GhGalT1基因的启动子pGhGalT1, 长度为539 bp, 该区域包含响应干旱、热、脱水、防御与胁迫应答的顺式作用元件MBS、HSE、MYCCONSE、TC-rich repeats和CGTCA-motif。NaCl、甘露醇或MeJA处理后, 转基因株系中GUS活性显著降低, BL或6-BA处理后, GUS活性显著升高, 验证了启动子活性区的功能。本研究结果为选用启动子改良作物提供了依据。The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | . |
| [2] | . |
| [3] | . |
| [4] | . |
| [5] | . |
| [6] | . |
| [7] | . |
| [8] | . |
| [9] | . |
| [10] | . |
| [11] | . |
| [12] | . |
| [13] | . |
| [14] | . |
| [15] | . |
| [16] | . |
| [17] | |
| [18] | . . |
| [19] | . |
| [20] | . |
| [21] | |
| [22] | . |
| [23] | . |
| [24] | . |
| [25] | . |
| [26] | . |
| [27] | . |
| [28] | . |
| [29] | . |
| [30] | . |
| [31] | . |
| [32] | . . |
| [33] | . . |
| [34] | . . |
| [35] | . . |
| [36] | . . |
| [37] | . . |
