
新疆农业大学农学院 / 新疆农业大学农业生物技术重点实验室, 新疆乌鲁木齐 830052;
Establishment of CRISPR/Cas9 Genome Editing System Based on GbU6 Promoters in Cotton (Gossypium barbadense L.)
LIJi-Yang, LEIJian-Feng, DAIPei-Hong, YAORui, QUYan-Ying, CHENQuan-Jia, LIYue, LIUXiao-Dong
通讯作者:
收稿日期:2017-05-21
接受日期:2017-11-21
网络出版日期:2018-02-12
版权声明:2018作物学报编辑部作物学报编辑部
基金资助:
展开
摘要
关键词:
Abstract
Keywords:
-->0
PDF (4512KB)元数据多维度评价相关文章收藏文章
本文引用格式导出EndNoteRisBibtex收藏本文-->
棉花是我国重要的经济作物和纺织工业原料。随着我国科技水平的飞速发展, 棉花种植业经历了近20年的蓬勃成长, 棉花的总产量及单位面积产量得到了成倍的增长, 目前已进入世界棉花高产国行列[1]。尽管棉花产业飞速发展, 但棉花生产中依然面临诸多急需解决的问题, 如干旱和盐胁迫是影响我国作物生产最主要的非生物胁迫因子[2,3]; 枯、黄萎病是世界级的植物病害, 对棉花生产造成严重损失; 造成生产的棉花纤维品质较低, 生产成本居高不下[4,5], 植棉效益日益降低, 严重影响了我国棉花的生产安全[6]。要从根本上解决这些问题就需要培育出抗病抗逆优质高产高效的棉花新种质, 而创制出这些新种质最终需要棉花生物学作为理论基础, 需要棉花功能基因组学来解析农艺性状调控的基因网络[7,8,9]。近几年新的基因组编辑技术的诞生和发展, 为挖掘棉花农艺性状关键调控功能基因提供了强大的技术工具, 不仅如此, 基因组编辑技术还有助于用来创造更多优异的育种材料[10]。
CRISPR/Cas9 (clustered regularly interspaced short palindromic repeats/CRISPR associated nuclease 9, Cas9)基因组编辑技术是2013年在国际上兴起的一种可以针对基因组靶向修饰的技术[11]。该技术起源于细菌长期进化过程中产生的一套免疫机制[12], 包括2个主要部分即小片段单链向导RNA (single guide RNA, sgRNA)和具有靶向识别sgRNA的核酸酶Cas9。通过带有靶向序列的sgRNA引导Cas9核酸酶在基因组特异性位点(PAM)附近切割, 最终由细胞启动基因组同源重组修复(HR)或非同源末端连接(NHEJ)导致基因组序列可逆或不可逆突变[13,14]。由于基因组编辑过程必须依赖内切核酸酶对基因组的切割, 因此, 理论上, 当基因组双链断裂修复时, 在其修复位点可以进行基因组定向插入、缺失、置换等类型的核苷酸修饰[14,15], 由于sgRNA可以根据基因组不同位点单独设计靶向序列而无需考虑Cas9核酸酶的特异性, 因此相比锌指核酸酶(zinc-finger nuclease, ZFN)和类转录激活效应物核酸酶(transcription activator-like effector nuclease, TALEN)等基因组编辑技术, CRISPR/Cas9基因组编辑技术操作更加简便, 成本更低, 编辑效率更高, 基因编辑更加精准, 脱靶效应更低[15,16,17], 目前已在多种植物中被成功应用[18,19,20,21,22], 在作物功能基因组学研究中展现出巨大的应用前景[9, 22-24]。CRISPR/Cas9基因组编辑技术体系中U6或U3启动子是驱动sgRNA转录的重要元件, 最近利用拟南芥U6启动子在棉花(Gossypium hirsutum Linn, 陆地棉)中已成功建立了CRISPR/Cas9基因组编辑技术体系[25,26]。然而利用海岛棉内源U6启动子在海岛棉中建立CRISPR/Cas9基因组编辑技术体系的研究还未见报道。
在拟南芥中, I型香叶酰基转移酶AtGGB蛋白通过对底物蛋白异戊二烯化的修饰[27,28], 调控了气孔的关闭, 在拟南芥抗旱反应中起负调控作用, ggb突变体的失水率明显低于野生型, 其抗旱性高于对照[29]。在棉花二倍体基因组中有一个单拷贝的与AtGGB高度同源的基因(蛋白同源性为59%)。本研究利用棉花内源的U6启动子[30], 在棉花中成功地建立了CRISPR/Cas9基因组编辑技术体系, 并验证了棉花基于GGB和ERA1基因在该技术体系中靶位点的有效性, 为后续研究棉花GGB基因的抗旱功能、创制新的棉花抗旱种质资源和进行棉花功能基因组学研究奠定了重要的技术基础。
1 材料与方法
1.1 供试材料
原生质体制备所用的供试材料为新海16胚性愈伤组织, 由新疆农业大学农业生物技术重点实验室提供, 由本实验室自行扩繁获得, 载体GbU6-4P:: GUS-sgRNA和GbU6-5P::GUS-sgRNA由本实验室保存, 两种不同密码子优化的Cas9基因(Cas9-I和Cas9-II)分别由中国科学院上海生命科学研究院冯争艳和中国农业科学院生物技术研究所孟志刚惠赠, 限制性内切酶和DNA聚合酶均为NEB和Thermo公司产品, 纤维素酶和离析酶由IUZMI株式会社代理提供, 其余试剂及药品均由国内生物公司代销, 引物合成及测序由上海杰李生物科技有限公司完成。1.2 载体构建
以新海16基因组DNA为模板, 克隆GbGGB和GbERA1基因部分序列, 用于寻找合适的靶序列。靶序列的设计基于以下原则: (1)在四倍体棉花新海16中A组和D组的序列完全一样; (2)具有合适的限制性内切酶位点; (3) GC含量在40%~60%之间。将经过DNA复性反应后的靶序列分别连接于以Bbs I酶切获得的GbU6-4P::GUS-sgRNA和GbU6-5P:: GUS-sgRNA线性化载体, 以不含靶序列的GbU6-4P-sgRNA和GbU6-5P-sgRNA载体作为阴性对照。反应体系为20 µL, 包括10 µmol L-1的上下游靶序列(表1)各10 µL, 复性程序为95~20°C, 每5 s降低0.5°C。经测序验证获得上述GbU6-4P (5P)::GGB-sgRNA和GbU6-4P(5P)::ERA1-sgRNA质粒正确后, 再将其以酶切连接方式(以Kpn I、Hind III酶切Cas9-I载体, 以Kpn I、Xba I酶切Cas9-II载体)分别连接在2种不同密码子优化的Cas9基因表达载体上(图1), 分别命名为GbU6-5P(4P)::GGB- sgRNA-Cas9I、GbU6-5P(4P)::ERA1-sgRNA-Cas9I和GbU6-5P(4P)::GGB-sgRNA-Cas9II、GbU6-5P(4P):: ERA1-sgRNA-Cas9II。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1海岛棉基因组编辑载体结构示意图
A: 含Cas9I编辑载体; B: 含Cas9II编辑载体。
-->Fig. 1Sketch map of genome editing structure of cotton (Gossypium barbadense L.)
A: editing vector containing Cas9I; B: editing vector containing Cas9II.
-->
Table 1
表1
表1本研究使用的引物信息
Table 1Primers used in this study
| 引物名称 Primer name | 序列 Sequence (5'-3') | 引物名称 Primer name | 序列 Sequence (5'-3') |
|---|---|---|---|
| GbU6-4PERA1-sg1F | GATTGTTCGCAGAATGCATGACGG | GbU6-5PGGB-sg2F | AAGTGCTCTGTCGAAGTACTGAAG |
| GbU6-5PERA1-sg1F | AAGTGTTCGCAGAATGCATGACGG | GbU6GGB-sg2R | AAACCTTCAGTACTTCGACAGAGC |
| GbU6ERA1-sg1R | AAACCCGTCATGCATTCTGCGAAC | Test GGB-sgF | AAGTGGAAAGAGAATGGCGAC |
| GbU6-4PERA1-sg2F | GATTGTCTTTCGCAGAATGCATGA | Test GGB-sgR | AGCTAATTCGCTATTGCAATCAATC |
| GbU6-5PERA1-sg2F | AAGTGTCTTTCGCAGAATGCATGA | Test ERA1-sgF | GTATCCTCTTACCGCCTTTC |
| GbU6ERA1-sg2R | AAACTCATGCATTCTGCGAAAGAC | Test ERA1-sgF | CATACTTACTGAGATGGCTGT |
| GbU6-4PGGB-sg1F | GATTGTGTCGAAGTACTGAAGCGG | FJ-1F | GTAAAACGACGGCCAG |
| GbU6-5PGGB-sg1F | AAGTGTGTCGAAGTACTGAAGCGG | FJ-1R | CAGGAAACAGCTATGAC |
| GbU6GGB-sg1R | AAACCCGCTTCAGTACTTCGACAC | FJ-2F | TAAACTGAAGGCGGGAAACG |
| GbU6-4PGGB-sg2F | GATTGCTCTGTCGAAGTACTGAAG | FJ-2F | CGGTTCTGTCAGTTCCAAACG |
新窗口打开
1.3 富集高浓度PCR片段
将构建的编辑载体酶切检测正确后, 进行80~200不等倍数稀释, 利用富集引物(FJ, 表1), 采用超保真pfu fast fly (北京全式金)扩增编辑载体的核心系统区域, 参照说明书推荐体系, PCR 产物加入等体积的酚、氯仿、异戊醇(1∶1∶1), 剧烈混匀后, 10 000×g离心3 min; 取上清液至新离心管中, 加入1/10上清液体积的醋酸钠(3 mol L-1, pH 5.2)和2.5倍上清液体积的预冷(-20°C)的无水乙醇, 混匀于-20°C放置10 min后, 于4°C下10 000×g离心5 min; 弃上清液, 加入1 mL 75%酒精润洗后, 10 000×g离心2 min; 吸尽上清液, 室温放置 5 min待酒精挥发完后, 加双蒸水溶解并调整浓度至0.5~1.0 µg µL-1, -20°C保存备用。1.4 原生质体的制备与转化
选择新海16扩繁培养约15 d的胚性愈伤组织作为原生质体制备材料, 采用纤维素酶和离析酶双酶体系制备原生质体, 以改良的CPW (甘露醇浓度增至0.5 mol L-1, 额外添加0.1% BSA)溶液[1]作为酶解反应的母液, 20 mL酶解体系包括12 mL超纯水、CPW大量母液(10×) 2 mL、CPW微量(1000×) 20 µL、0.5 mol L-1甘露醇、10 mmol L-1 MES、2%纤维素酶、1%离析酶, 55°C孵育10 min, 待溶液恢复至室温, 加入0.1% BSA, 再定容至20 mL, 使用前用0.22 µm滤膜抽滤制成酶解工作液, 取10 mL上述工作液转移至70 mm无菌培养皿, 将颗粒大小约3 mm的棉花胚性愈伤组织均匀地铺在酶解液表面, 轻微晃动平皿, 使胚性愈伤组织与酶解液充分接触, 于28°C, 50 r min-1避光酶解反应14 h以上。取10 µg富集的PCR产物, 方法参照拟南芥原生质体转化方法[31]直接转化棉花原生质体, 过程略有改进, 悬浮原生质体不使用MMG溶液, 采取振荡过夜孵育, 于28°C, 50 r min-1避光过夜孵育转化后的原生质体。1.5 内源靶基因编辑效应的检测
过夜孵育转化后的原生质体经镜检后, 采用12 000 r min-1离心1 min收集沉淀, 弃去上清液后, 用于基因组DNA提取, 方法参照Trans Easypure plant Genomic DNA Kit操作要求, 将提取的DNA经过浓度检测后, 取等质量提取的DNA, 采用guide RNA序列上选择的检测用限制性内切酶(GbERA1采用Nsi I、GbGGB采用Sca I酶切)进行过夜酶切和不酶切2种处理, 分别以2种处理的原生质体DNA为模板, 进行PCR检测, PCR反应体系参照NEB公司Phusion超保真DNA聚合酶推荐反应体系, 扩增产物进行琼脂糖凝胶电泳分析。1.6 编辑靶序列的克隆测序
采用EasyPure Quick Gel Extraction Kit (北京全式金)回收上述PCR扩增产物,参照试剂盒说明书方法, 取4 μL产物于25°C, 5 min与pEASY-Blunt Zero Cloning (北京全式金)载体连接, 连接产物转化大肠杆菌Trans T1, 37°C过夜培养, 以M13测序引物进行菌落PCR鉴定, 挑取阳性单克隆于液体LB培养基中过夜活化, 用于测序,采用DNAstar和DNAMAN软件比对分析测序序列。1.7 靶基因内不同区域突变频率的计算
从转化未酶切PCR产物的Trans T1过夜培养的平皿中, 随机挑选100个长势均一的单菌落测序, 通过DNAstar软件比对测序结果, 统计Test检测区域与guide RNA结合的靶序列区30 bp及其前后各3个30 bp区域碱基突变个数并计算相应区域碱基突变频率, 利用制图软件绘制Test区域碱基突变频率折线图。2 结果与讨论
2.1 编辑载体构建
采用酶切方式检测上述构建的载体, 酶切结果符合预期设计, 证明连接GbU6-5P、GbGGB和GbERA1不同靶序列、以及含有不同密码子优化的Cas9的编辑载体均构建成功(图2)。基于GbU6-4P的编辑载体鉴定结果图略。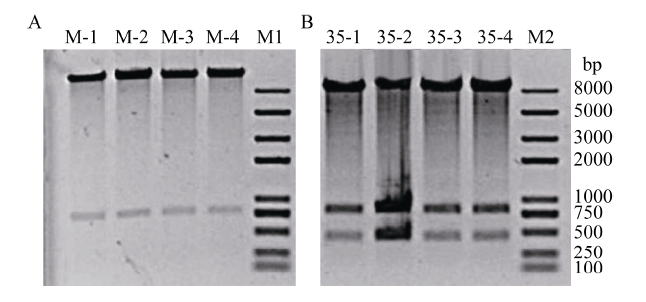 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2基因编辑载体的酶切鉴定
M1, M2: 2K plus DNA marker. A: M-1 to M-4 were GbU6-5P::GGB-sgRNA1-Cas9IIGbU6-5P::GGB-sgRNA2-Cas9II, GbU6-5P::ERA1-sgRNA1-Cas9II, and GbU6-5P:: ERA1-sgRNA2-Cas9II, respectively. B: 35-1 to 35-4 were GbU6-5P::GGB-sgRNA1-Cas9I, GbU6-5P::GGB-SgRNA2-Cas9I, GbU6-5P::ERA1-sgRNA1-Cas9I, and GbU6-5P:: ERA1-sgRNA2-Cas9I.
-->Fig. 2Restriction enzyme digestion identification of gene editing vector
-->
2.2 原生质体制备与转化
体系优化获得的原生质体在显微镜下呈圆球状, 形态饱满, 符合去壁细胞形态, 经计算其数量可达1×106个 mL-1以上, 细胞活性染色剂抽样镜检检测显示, 仅极少原生质体为破碎或失活状态, 证明使用该体系制备的原生质体可以满足本研究的后续试验要求。经转化并过夜孵育后的原生质体, 未出现细胞成簇聚集现象, 细胞均匀悬浮于转化液中, 经细胞活性和荧光检测, 细胞大部分呈现正常活性状态, 说明该体系可以用于棉花原生质体的转化(图3)。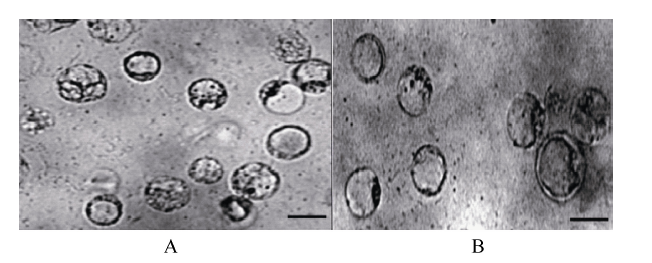 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3棉花原生质体转化前后形态图
A: 棉花原生质体酶解过夜镜检结果(×40); B: 转化GbU6-5-GGB-sgRNA1-Cas9I载体核心片段的棉花原生质体过夜孵育后镜检结果(×40)。Bar=100 μm。
-->Fig. 3Morphological changes of cotton protoplasts before and after transformation
A: result of cotton protoplasts overnight microscopy test (×40); B: results of microscopic test overnight examination of cotton prtoplasts transformed into the core fragments of GbU6-5-GGB-sgRNA 1-Cas9 I vector (×40). Bar=100 μm.
-->
2.3 CRISPR/Cas9基因组编辑效应检测
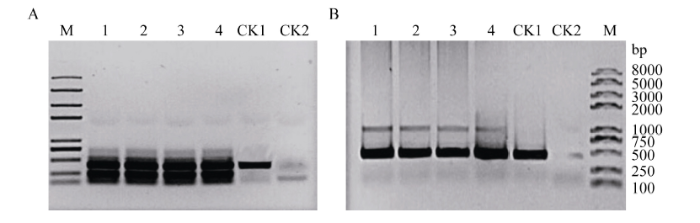
使用GbU6-5P启动子来驱动GbERA1和GbGGB各2条guide RNA的转录, 同时再与两种Cas9基因组合构建基因编辑载体。对这8种基因编辑载体转化获得的原生质体基因组DNA, 进行酶切/PCR检测(检测原理见图4)后发现, 酶切前的样品和对照都能扩增出预期大小的条带, 而且亮度基本相同。而酶切后对照几乎没有扩增产物, 但样品却扩增出与酶切前样品同样大小的条带, 表明转化基因编辑载体的原生质体内, 目标基因靶序列酶切位点处出现突变, 导致限制性内切酶不能切割DNA, 维持了模板DNA的完整性, 所以扩增出预期大小的条带, 而对照未发现明显的突变现象(图5)。对酶切/PCR片段克隆测序表明, 在GGB基因靶位点上, 位于PAM位点上游限制性内切酶位点区域碱基组成均出现不同类型的突变(图6~图8), 其中以碱基替换为主(包括转换和颠换), 仅在少数突变位点检测到碱基缺失现象(图8)。与此同时对于ERA1基因靶位点和使用GbU6-4启动子的样品, 结果也基本相似(结果图略)。为了排除PCR扩增导致上述的碱基突变, 对于GbGGB-sgRNA2靶位点, 我们对酶切前原生质体基因组DNA进行同样的PCR扩增, 其扩增产物进行克隆测序。在100个阳性克隆测序结果中, 分别对在靶序列30个碱基区域内和两侧相邻各3个30 bp区域内碱基的突变频率进行分析, 结果显示靶序列30个碱基区域内的碱基突变频率远远高于两侧相邻各3个30 bp区域内碱基的突变频率(图9)。表明靶序列区碱基的突变确实是CRISPR/Cas9系统基因编辑的结果。上述结果证明基于海岛棉GbU6-4和GbU6-5启动子的CRISPR/Cas9基因编辑载体系统均能在海岛棉中实现靶向基因编辑的功能。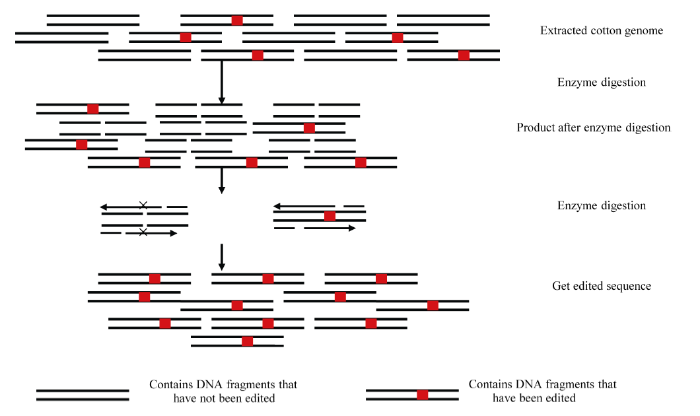 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4酶切/PCR检测CRISPR/Cas9基因组编辑效应原理示意图
红色区域为被编辑突变的酶切位点。
-->Fig. 4Schematic diagram of digestion /PCR for editing effect of CRISPR/Cas9
The red region is the enzyme-digested site that has been edited.
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5酶切/PCR检测CRISPR/Cas9基因组编辑效应
M: 2000 plus DNA marker。A: 1、2分别为转化GbU6-5P::ERA1-sgRNA1-Cas9I基因组酶切前、后PCR扩增结果, 3、4分别为转化GbU6-5P::GGB-sgRNA1-Cas9I基因组酶切前、后PCR扩增结果。B: 1、2分别为转化GbU6-5P::ERA1-sgRNA2-Cas9I基因组酶切前、后PCR扩增结果, 3、4分别为转化GbU6-5P::GGB-sgRNA2-Cas9I基因组酶切前、后PCR扩增结果, CK1、CK2为转化阴性对照载体基因组酶切前后PCR扩增结果。
-->Fig. 5Digestion /PCR for editing effect of CRISPR/Cas9
M: 2000 plus DNA marker. A: 1, 2: PCR amplification of GbU6-5P:: ERA1-sgRNA1-Cas9 I genome before and after enzyme digestion; 3, 4: PCR amplification of GbU6-5P:: GGB-sgRNA1-Cas9 I genome before and after enzyme digestion. B: 1, 2: PCR amplification of GbU6-5P:: ERA1-sgRNA2-Cas9 I genome before and after enzyme digestion; 3, 4: PCR amplification of GbU6-5P::GGB-sgRNA2-Cas9 I genome before and after enzyme digestion; CK1, CK2: PCR amplification of negative control genome before and after enzyme digestion.
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6GbGGB-sgRNA2靶位点编辑效应(碱基置换型)测序检测结果
-->Fig. 6Sequencing of GbGGB-sgRNA2 target site that were edited (type of base substitution)
-->
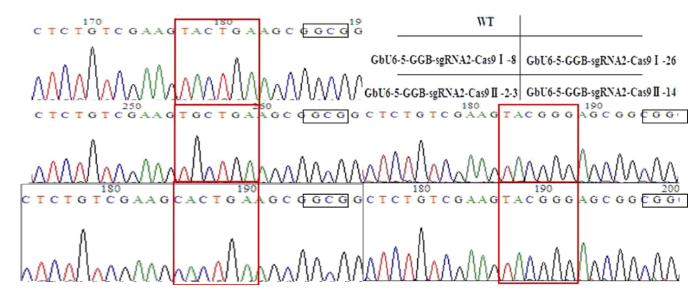 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7GbGGB-sgRNA2靶位点编辑效应(碱基置换型)测序峰图
红色方框指示碱基突变的区域; 黑色方框指示PAM位点区域。
-->Fig. 7Sequencing peaks figure of GbGGB-sgRNA2 target site that were edited (type of base substitution)
The red box indicates the region is base pair were changed; the black box indicates the PAM site area.
-->
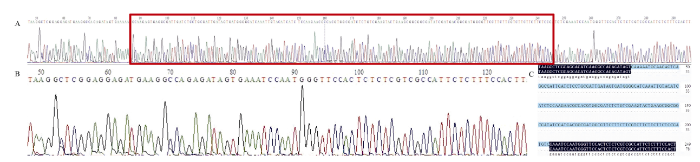 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图8GbGGB-sgRNA2靶位点编辑效应(碱基缺失型)测序峰图
红色方框指示突变序列(B)碱基相比对照(A)测序峰图发生碱基缺失区域;C图显示碱基缺失区域。
-->Fig. 8Sequencing peaks figure of GbGGB-sgRNA2 target site that were edited (type of base deletion)
The red box indicates the region of deletion sequence(B) compared with the control (A); C indicates the base deletion region.
-->
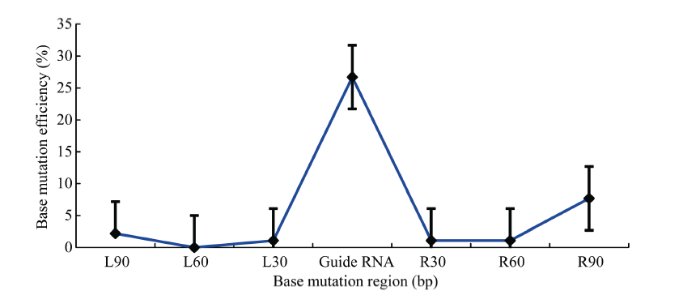 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图9GbGGB-sgRNA2靶位点区域(guide RNA)及其两侧位点的突变效率分析
L30、L60、L90分别表示guide DNA靶序列左侧30、60、90 bp区域; R30、R60、R90分别表示guide DNA靶序列右侧30、60、90 bp区域。
-->Fig. 9Mutation efficiency of GbGGB-sgRNA2 target site (guide RNA) and its two flanking region
L30, L60, and L90 indicate the 30, 60, and 90 bp region on the left of guide DNA respectively; R30, R60, and R90 indicate the 30, 60, and 90 bp region on the right of guide DNA, respectively.
-->
3 讨论
3.1 原生质体制备与转化
CRISPR/Cas9介导的基因组编辑技术在植物中的研究已逐渐趋于成熟, 目前棉花的CRISPR/Cas9基因组编辑技术刚刚在陆地棉中成功建立[25,26], 但在海岛棉中还未见报道。与陆地棉相比, 海岛棉存在体细胞胚胎发生困难、转化周期长、效率低等问题, 因此采用瞬时转化法来快速验证构建的海岛棉CRISPR/Cas9基因组编辑载体的有效性就成为唯一的方法。与模式植物不同, 棉花原生质体制备与转化受材料来源、酶解体系、反应条件、转化方式等因素显著影响。本研究在前期实验基础上, 尝试海岛棉多种组织, 但均未获得符合要求的原生质体, 而利用再分化形成的胚性愈伤组织最终获得了满足试验要求的原生质体(图3)。在转化过程中的孵育阶段, 采用缓慢振荡孵育方式, 可以避免原生质体在PEG的作用下出现成簇聚集现象而影响转化成功率。3.2 CRISPR/Cas9基因组编辑效应检测
CRISPR/Cas9基因组编辑突变位点检测一般采用酶切和PCR相结合的方法[25]。以先PCR后酶切方式进行编辑效率检测时, 虽然可以保证酶切反应底物的浓度, 提高检出率, 但由于PCR过程中存在的偏扩增现象, 可能造成突变产物扩增过程中被遗漏。另外由于基因组发生编辑后序列在修复过程中碱基的突变可能造成DNA链不稳定, 导致聚合酶延伸时滑落, 造成突变序列难以指数性扩增, 最终造成产物累积量过少而无法准确检测编辑的效应。因此采用先酶切后PCR方式编辑效应检测是一种有效可靠的方法。U6启动子作为CRISPR/Cas9基因组编辑系统的重要元件之一, 目前已经在棉花中克隆得到, 并且验证具有转录活性[30]。然而基于棉花U6启动子的CRISPR/Cas9基因编辑载体系统能否在棉花中实现基因定点编辑的效应, 目前并不清楚。我们采用先酶切后PCR的方式检测了基于海岛棉GbU6-4和GbU6-5启动子的CRISPR/Cas9基因编辑载体系统的编辑效应, 成功地在海岛棉原生质体中检测到靶序列的突变现象(图5)。通过克隆测序分析显示, 突变的类型主要以碱基替换为主, 少数为碱基缺失, 这与陆地棉中的结果[25]相似, 试验中并未检测到碱基的插入。为了验证单碱基的改变是否是PCR扩增过程中随机产生的突变, 我们对扩增克隆的整个PCR片段序列进行了分析, 结果显示靶位点碱基的突变频率要远远高于其两侧其他位点的突变频率。通过克隆测序, 我们没有检测到对照样品中存在突变序列, 然而对照样品基因组DNA酶切后PCR产物依然还有微量的条带(图5), 原因可能是棉花属多酚高糖植物, 基因组在提取过程中残留的多糖和酚类物质在酶切反应时都可能抑制限制性内切酶活性, 导致靶序列模板酶切不彻底, 再经PCR扩增时, 就产生了微量的条带。
在验证CRISPR/Cas9基因编辑载体系统的编辑效应时, 我们使用了两种不同密码子优化的Cas9基因, 结果显示这2种Cas9基因都能有效实现对靶位点双链DNA的切割和编辑, 这也暗示着棉花蛋白质合成体系中可能没有明显的密码子使用偏好性。
4 结论
采用高浓度CRISPR/Cas9核心DNA片段瞬时转化棉花原生质体, 成功检测到海岛棉内源基因的突变。证明了基于棉花内源U6启动子的CRISPR/ Cas9基因组编辑技术体系在海岛棉中的有效性, 表明该体系可以用于定向创制棉花的突变体, 为棉花功能基因组学研究提供了重要的技术基础。The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | . . |
| [3] | . . |
| [4] | . . |
| [5] | . . |
| [6] | . . |
| [7] | . . |
| [8] | . . |
| [9] | . |
| [10] | . |
| [11] | . |
| [12] | . |
| [13] | . |
| [14] | . |
| [15] | . |
| [16] | . |
| [17] | . |
| [18] | . |
| [19] | . |
| [20] | . |
| [21] | . |
| [22] | |
| [23] | . |
| [24] | . |
| [25] | . |
| [26] | . |
| [27] | . |
| [28] | . |
| [29] | . . |
| [30] | . . |
| [31] | . |
