
西南大学玉米研究所 / 南方山地农业教育部工程研究中心, 重庆400715
Cloning and Imprinting Characterization Analyses of Paternally Expressed Gene ZmVIL1 in Maize Endosperm
LIUChao-Xian
通讯作者:
收稿日期:2017-07-11
接受日期:2017-11-21
网络出版日期:2018-03-12
版权声明:2018作物学报编辑部作物学报编辑部
基金资助:
展开
摘要
关键词:
Abstract
Keywords:
-->0
PDF (4444KB)元数据多维度评价相关文章收藏文章
本文引用格式导出EndNoteRisBibtex收藏本文-->
基因组印记是一种表观遗传学现象, 在动植物中普遍存在。印记基因在DNA序列上没有发生任何变化, 等位基因的表达水平因其亲本来源的不同(父本或者母本)而表现出较大的差异[1], 即来自某一亲本的等位基因表达, 而来自另一亲本的等位基因不表达或者表达量极低。根据印记基因的亲本来源可将印记基因分为父本印记(来自父本的等位基因表现印记)和母本印记(来自母本的等位基因表现印记), 根据印记基因的印记特性可分为等位基因特异性印记(某些等位基因表现印记)和基因特异性印记(所有等位基因表现印记), 根据印记基因的表达特性可分为二元印记(2个等位基因中只有一个表达, 而另一个则完全沉默)和差异印记(2个等位基因都表达, 其中一个表达量较高, 一个较低)及持续性印记(在一个较长的时间段均表现印记)和瞬时印记(只在其中一个时间点或者极短的时间段表现印记)[2]。
印记基因首先是在植物中发现的。R1是在20世纪70年代初发现的第一个印记基因, 该基因调控玉米籽粒花色苷的合成。当R1父本遗传时, 玉米籽粒呈斑驳状着色; 然而当R1母本遗传时, 则整个玉米籽粒着色[3]。20世纪80年代, 对小鼠进行细胞核移植时证实了哺乳动物中也存在基因印记现象。核移植实验结果表明双亲贡献的基因组遗传信息并不完全相同, 只有同时具有来源于双亲染色体的胚胎才能存活下来; 仅拥有父本基因组或母本基因组的二倍体胚胎均不能完成发育过程[4,5]。随后在哺乳动物中鉴定出了越来越多的印记基因, 且其功能得到了阐释[6]。研究结果发现哺乳动物中印记基因功能通常比较保守, 在胚胎生长发育中具有重要作用。现已阐明印记基因调控哺乳动物胎盘发育、胎儿发育、胎儿后天行为及代谢等过程, 并且印记基因甲基化状态的改变可能会导致疾病[7,8]。
相对哺乳动物基因组印记的研究, 植物基因组印记研究较滞后。目前在拟南芥、水稻、玉米等植物中均鉴定出了大量的印记基因[9,10,11]。玉米是异花授粉植物, 籽粒较大且胚乳持续存在, 因此在基因组印记研究中具有独特优势。玉米中除了R1印记基因外, 在随后研究中陆陆续续鉴定出了几个印记基因, 其中包括父本印记基因ZAG2[12]、Meg1[13]、nrp1[14]、dzr1[15]、Mez1[16]、mee1[17]、fie1、fie2[18]等。相对于数量较多的父本印记基因, 母本印记基因数量较少[19,20]。然而在21世纪初, 随着转录组测序技术的发展, 玉米印记基因的数量急剧增加。Waters等[11]对B73、Mo17正反交授粉后14 d的胚乳和胚进行转录组深度测序, 共鉴定出了100个印记基因, 其中包括54个父本印记基因、46个母本印记基因。Zhang等[21]同样利用转录组测序技术, 以B73、Mo17为材料, 对正反交授粉后10 d的胚乳和胚的基因表达谱进行分析, 鉴定出了179个编码蛋白的印记基因, 其中111个是母本印记基因, 68个是父本印记基因; 此外还鉴定出了38个印记的长链非编码RNA。ZmVIL1 (GRMZM2G339820)就是其中被鉴定的印记基因之一。ZmVIL1是拟南芥VIL1的同源基因, 然而该基因的全长、基因的印记特性及时空表达模式尚不清楚。本研究通过快速扩增cDNA末端技术(RACE, rapid amplification cDNA ends)获得了该基因的cDNA全长; 研究了该印记基因在几个优良杂交种中的印记模式及对该基因的时空表达模式进行分析, 从而为研究该基因在玉米籽粒发育中的功能及其参与的基因表达调控网络奠定了基础。
1 材料与方法
1.1 材料、菌株和试剂
玉米自交系B73、Mo17、黄C、178、郑58、昌7-2、PH6WC、PH4CV、大肠杆菌(E. coli) DH5α均由本实验室保存。植物总RNA提取试剂TRIzol、质粒提取试剂盒(TIANprep Rapid Mini Plasmid Kit)、胶回收试剂盒(TIANgel Midi Purification Kit)购于天根生化科技(北京)有限公司; RNA反转录试剂盒(RevertAid RT Reverse Transcription Kit)和限制性内切酶购于Fermeantas公司; pEASY-T1载体、HiFi DNA 聚合酶购于北京全式金生物技术有限公司; RACE试剂盒购自Clontech公司。1.2 不同杂交组合授粉后不同阶段胚及胚乳的分离
将玉米自交系B73、Mo17、黄C、178、郑58、昌7-2、PH6WC、PH4CV于2013年种植于西南大学重庆歇马基地。玉米散粉期, 分别对B73和Mo17、郑58和昌7-2、黄C和178、PH6WC和PH4CV做正反交, 剥离郑58和昌7-2、黄C和178、PH6WC和PH4CV正反交授粉后14 d胚乳及B73和Mo17正反交授粉后14 d胚; 每隔2 d一次剥离B73和Mo17正反交组合授粉后10~28 d胚乳, 于液氮中速冻, -80ºC储藏用于RNA提取。1.3 快速扩增cDNA末端
1.3.1 5′RACE模板cDNA的制备 吸取2.75 μL授粉后14 d的B73胚乳RNA (523 mg L-1)及1 μL 5°-CDS Primer A (12 μmol L-1)至0.2 mL无RNA酶离心管中, 混匀, 短暂离心, 收集溶液于管底。将离心管置PCR仪上, 72ºC 3 min, 42ºC 2 min。14 000 × g, 离心10 s收集溶液于管底。加入1 μL SMART II A Oligonucleotide (12 μmol L-1), 混匀并短暂离心, 收集溶液于管底。然后依次加入各反应组分, 混匀, 短暂离心, 收集反应液于管底。然后再加入5 × First- Strand buffer 2.0 μL、DTT (20 mmol L-1) 1.0 μL、dNTP Mix (10 mmol L-1) 1.0 μL、RNase Inhibitor (4 ×107 U L-1) 0.25 μL、SMARTScribe Reverse Transcriptase (1 × 108 U L-1)1.0 μL。将上述反应液混匀, 短暂离心, 在PCR反应仪中42ºC 90 min, 然后70ºC 10 min。最后加入100 μL pH 8.0的TE缓冲液稀释, -20ºC保存。1.3.2 3° RACE模板cDNA的制备 吸取3.75 μL授粉后14 d的B73胚乳RNA (480 mg L-1)及1 μL 3°-CDS Primer A (12 μmol L-1)至0.2 mL无RNA酶离心管中, 混匀, 短暂离心, 收集溶液于管底。将离心管置PCR仪上, 72ºC 3 min, 42ºC 2 min。14 000×g, 离心10 s收集溶液于管底。然后再加入5×First-Strand buffer 2.0 μL、DTT (20 mmol L-1) 1.0 μL、dNTP Mix (10 mmol L-1) 1.0 μL、RNase Inhibitor (4×107 U L-1) 0.25 μL、SMARTScribe Reverse Transcriptase (1×108 U L-1) 1.0 μL。将上述反应液混匀, 短暂离心, 在PCR反应仪中42ºC 90 min, 然后70ºC 10 min。最后加入100 μL pH 8.0的TE缓冲液稀释, -20ºC保存。
1.4 CAPS (Cleaved Amplified Polymorphic Sequences)标记的开发
设计ZmVIL1基因特异性引物, 对B73、Mo17、黄C、178、郑58、昌7-2、PH6WC和PH4CV cDNA进行扩增。将PCR产物连接到pEASY-T1载体, 转化DH5α感受态细胞。挑选阳性克隆送华大基因公司测序。然后用SNP2CAPS软件[22]对B73和Mo17、郑58和昌7-2、黄C和178、PH6WC和PH4CV之间的测序结果进行分析, 开发CAPS标记。1.5 ZmVIL1的组织特异性表达分析
根据ZmVIL1在玉米染色体的物理位置, 在qTeller (http://www.qteller.com/qteller4/)下载ZmVIL1在根、顶端分生组织、叶、叶舌、叶鞘、雄穗、雌穗、花药、花粉、花丝、胚珠、授粉后5 d和10 d籽粒、授粉后25 d胚和胚乳中的表达丰度RPKM值(每百万reads中来自某基因每千碱基长度的reads数), 分析该基因在各组织中的表达。1.6 系统进化树的构建
拟南芥VIN3 (AT5G57380)、VIL1 (AT3G24440)、VIL2 (AT4G30200)、VIL3 (AT2G18880)和VIL4 (AT2G18870)蛋白序列下载于TAIR数据库(http:// www.arabidopsis.org/index.jsp)。根据蛋白序列号从NCBI网站下载(https://www. ncbi.nlm.nih.gov/)水稻OsVIL1、OsVIL2、OsVIL3和OsVIL4[23]。将目标序列导入MEGA6[24], 以默认参数利用CLUSTALW对导入序列比对, 随后以NJ算法1000次重复构建系统发育树[25]。
2 结果与分析
2.1 ZmVIL1 基因cDNA的克隆
2.1.1 ZmVIL1 cDNA 5°和3° RACE 根据Gramene (http://ensembl.gramene.org/)预测的ZmVIL1基因CDS序列, 分别设计了5° RACE和3° RACE巢式引物(表1), 利用B73授粉后14 d籽粒胚乳RNA反转录合成的5° RACE和3° RACE 全长 cDNA作为模板进行PCR扩增。如图1-A所示, 分别用ZmVIL1-5° race和ZmVIL1-3° race引物对ZmVIL1基因的5°和3°全长cDNA模板扩增后, 电泳结果显示非特异性扩增条带较多。因此分别用5°和3° RACE巢式引物ZmVIL1-5° race-nest-1和ZmVIL1-3° race-nest-1以第1轮RACE 产物稀释50倍后, 吸取1 μL作为模板进行第2轮RACE。电泳结果显示ZmVIL1基因第2轮5° RACE PCR产物非特异性扩增条带仍然较多; 3° RACE虽然主带清晰, 但是PCR产物浓度较低(图1-B)。因此又通过第3轮RACE巢式引物ZmVIL1-5° race-nest-2和ZmVIL1- 3° race-nest-2, 以第2轮RACE产物同样稀释50倍后吸取1 μL PCR产物作为模板进行扩增。结果显示5° RACE产物有清晰的主带, 但基因非特异性扩增依然存在(图1-C)。根据预测的ZmVIL1基因CDS序列及RACE引物的位置, 推测第3轮5° RACE产物大于700 bp, 因此参照分子量标准回收700 bp以上的条带; 第3轮3° RACE产物主带清晰, 因此回收500 bp左右主带。并分别将回收的5° RACE和3° RACE条带连接到克隆载体上进行DNA测序。Table 1
表1
表1ZmVIL1 5° RACE、3° RACE、CDS扩增及印记特性分析引物序列
Table 1Primer sequences for 5° RACE, 3° RACE, CDS amplification and imprinting characterization analyses of ZmVIL1
| 引物名称 Primer name | 正向引物序列 Forward primer sequence (5°-3°) | 反向引物序列 Reverse primer sequence (5°-3°) | 内切酶 Restriction enzyme |
|---|---|---|---|
| ZmVIL1-5° race | GCCCATCGCATTCCAACTGCCTTACTAC | ||
| ZmVIL1-5° race-nest-1 | GCGTTTAGGTCGGGGACAGAAGATGAG | ||
| ZmVIL1-5° race-nest-2 | GGTCAGGAGAACTCAACCACTCGTCAGC | ||
| ZmVIL1-3° race | CTCGGCAAGTAAGCAAGGTGAATAGCGT | ||
| ZmVIL1-3° race-nest-1 | GCAACTGGATGGGGATTATGAGCACTG | ||
| ZmVIL1-3° race-nest-2 | AGGTAGTAAGGCAGTTGGAATGCGATGG | ||
| ZmVIL1-CDS | ATGCCCAAAACACCTCCTGT | CTCAGCGAACCACAACAACAAA | |
| ZmVIL1-imprinting | ATTCAAAGAGCTGACGAGTGGT | TCATAATCCCCATCCAGTTGCC | Hph I |
新窗口打开
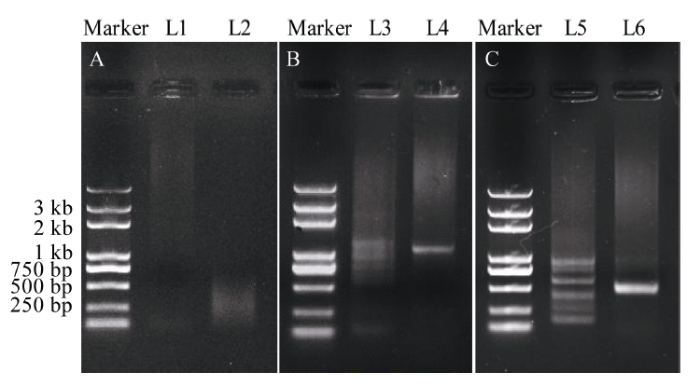 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1ZmVIL1基因5° RACE和3° RACE
A、B、C: ZmVIL1第1轮、第2轮和第3轮RACE产物电泳。L1、L3、L5: ZmVIL1 cDNA 5° RACE产物电泳; L2、L4、L6: ZmVIL1 cDNA 3° RACE产物电泳; Marker: D2000 plus。
-->Fig. 15° RACE and 3° RACE for ZmVIL1 cloning
A, B, C: the gel electrophoresis for the first, second and third round RACE PCR products. L1, L3, L5: the gel electrophoresis for ZmVIL1 cDNA 5° RACE; L2, L4, L6: the gel electrophoresis for ZmVIL1 cDNA 3°RACE; Marker: D2000 plus.
-->
2.1.2 ZmVIL1基因全长的获得及预测编码蛋白
以获得的ZmVIL1基因5° RACE和3° RACE序列为模板设计引物ZmVIL1-CDS (表1)以B73授粉后14 d胚乳cDNA为模板进行扩增, 连接到克隆载体上, 挑选阳性克隆送测序。将获得的测序序列与Gramene预测的ZmVIL1基因cDNA序列比对, 然后选择ZmVIL1基因5° RACE、3° RACE的正确序列和ZmVIL1-CDS扩增序列拼接, 从而获得ZmVIL1基因cDNA全长。序列分析结果显示该基因cDNA全长2201 bp, CDS长1797 bp; 编码蛋白由598个氨基酸组成(GenBank ID: MF437288)。ZmVIL1与拟南芥同源蛋白VIN3[26]和VIL1[27]序列比对表明, ZmVIL1和其他蛋白一样都具有保守的锌指蛋白和纤连蛋白结构域(图2)。
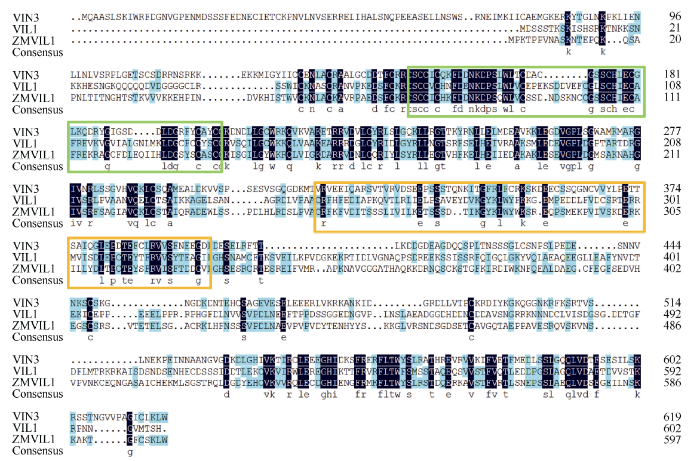 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2ZmVIL1与拟南芥VIN3、VIL1蛋白序列比对淡绿色和橙色方框内分别为保守的锌指蛋白和纤连蛋白结构域。
-->Fig. 2Protein sequences alignment for ZmVIL1 in maize, and VIN3, VIL1 in Arabidopsis
-->
The boxes colored with light green and orange respectively represent the conserved domains of zinc finger protein and fibronectin protein.
2.2 ZmVIL1印记特性鉴定的CAPS标记开发
CAPS标记是鉴定印记基因印记特性的重要工具, 该标记成功开发与否依赖于DNA序列之间是否有丰富的单核苷酸多态性。在已获得B73 ZmVIL1全长cDNA序列的基础上, 利用ZmVIL1-CDS对Mo17授粉后14 d胚乳cDNA进行扩增, 并对PCR产物克隆测序, 获得了Mo17 ZmVIL1等位基因完整的CDS序列(GenBank ID: MF573938)。比对B73和Mo17 ZmVIL1 CDS序列, 发现在CDS序列中的3个位点, 即第1380、第1450和第1530碱基有单核苷酸多态性(图3), 并且内切酶Hph I能识别B73 ZmVIL1第1380和第1450碱基周围DNA序列, 然而在Mo17 ZmVIL1等位基因CDS序列中没有Hph I的识别位点。因此设计ZmVIL1-imprinting引物(表1)用于扩增包括1380和1450单核苷酸多态性位点的DNA片段, 然后用内切酶Hph I对扩增产物进行酶切, 从而区别B73和Mo17的ZmVIL1等位基因。利用相同的方法, 用ZmVIL1-imprinting对黄C、178、郑58、昌7-2、PH6WC和PH4CV授粉后14 d胚乳cDNA进行扩增测序, 发现黄C、昌7-2和PH6WC中ZmVIL1等位基因扩增片段同样存在与B73一样的Hph I识别位点, 而在178、郑58和PH4CV扩增片段中没有该酶的识别位点。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3B73和Mo17中ZmVIL1 CDS序列单核苷酸多态性位点箭头所示位置为B73和Mo17 ZmVIL1 CDS在第1380、第1450和第1530碱基位置的单核苷酸多态性位点。
-->Fig. 3SNPs in ZmVIL1 CDS of B73 and Mo17The arrows indicate the SNPs at the 1380th, 1450th, and 1530th base pairs in ZmVIL1 CDS of B73 and Mo17.
-->
2.3 ZmVIL1在B73和Mo17正反交授粉后14 d胚乳及胚中的印记特性分析
分离B73、Mo17、B73×Mo17 (B×M)、Mo17×B73 (M×B)授粉后14 d的胚乳及胚, 提取RNA反转录合成cDNA第1链。用ZmVIL1-imprinting分别扩增B73、Mo17、B×M、M×B授粉后14 d的胚和胚乳cDNA, 用Hph I对PCR产物酶切之后进行电泳检测。结果显示ZmVIL1在B×M、M×B胚乳中, 来自母本的等位基因完全不表达, 而来自父本的等位基因表达(图4-A), 因此ZmVIL1在胚乳中表现为母本印记; 然而在胚中来自父本和母本的ZmVIL1等位基因均正常表达(图4-B), 因此ZmVIL1在胚中并不表现印记现象。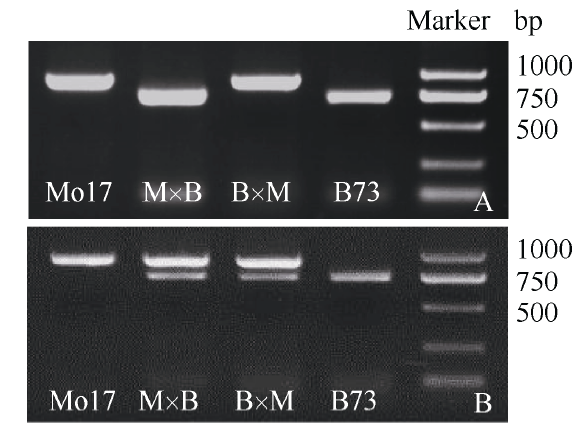 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4ZmVIL1在B73、Mo17正反交授粉后14 d胚乳及胚中的印记特性分析
A: 授粉后14 d胚乳中ZmVIL1的印记特性分析; B: 授粉后14 d胚中ZmVIL1的印记特性分析; Marker: D2000 plus。
-->Fig. 4Imprinting characterization analysis of ZmVIL1 in 14 DAP endosperm and embryo of B73 and Mo17 reciprocal hybrids
A: imprinting characterization analysis of ZmVIL1 in 14 DAP endosperm; B: imprinting characterization analysis of ZmVIL1 in 14 DAP embryo. Marker: D2000 plus.
-->
2.4 ZmVIL1 在黄C和178、郑58和昌7-2、PH6WC和PH4CV正反交授粉后14 d胚乳中的印记特性分析
分离黄C (HC)、178、黄C×178 (HC×178)、178×黄C (178×HC)、PH6WC、PH4CV、PH6WC × PH4CV、PH4CV × PH6WC、昌7-2 (C)、郑58 (Z)、昌7-2×郑58 (C×Z)、郑58×昌7-2 (Z×C)授粉后14 d的胚乳RNA反转录成cDNA, 用ZmVIL1-imprinting对各自交系及正反交组合cDNA进行扩增, 扩增产物用Hph I酶切。结果表明在所有杂交组合中, 来自母本的所有ZmVIL1等位基因表现印记(图5), 这与ZmVIL1在B × M和M×B 14 d胚乳中的印记状态是一致的。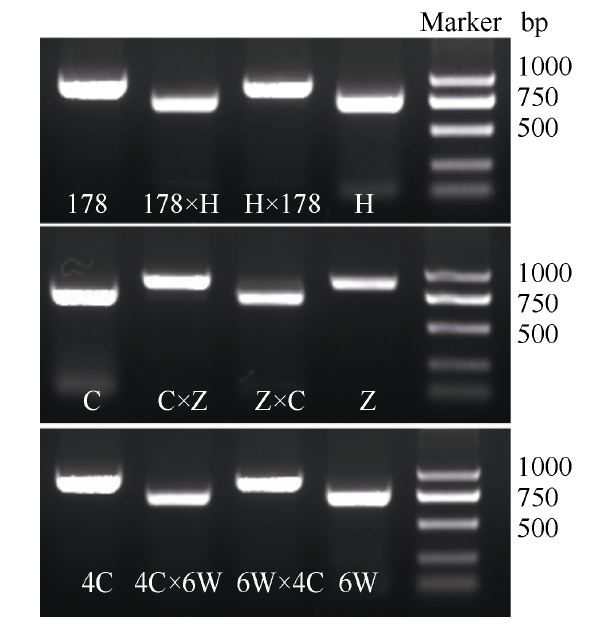 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5ZmVIL1在黄C和178、郑58和昌7-2及PH4CV和PH6WC正交反授粉后14 d胚乳中印记特性分析
H: 黄C; C: 昌7-2; Z: 郑58; 4C: PH4CV; 6W: PH6WC; Marker: D2000 plus。
-->Fig. 5Imprinting characterization analysis of ZmVIL1 in 14 DAP endosperm of Huang C and 178, Zheng 58 and Chang 7-2, PH6WC and PH4CV reciprocal hybrids
H: Huang C; C: Chang 7-2; Z: Zheng 58; 4C: PH4CV; 6W: PH6WC; Marker: D2000 plus.
-->
2.5 ZmVIL1在授粉后10~28 d B73×Mo17、Mo17×B73胚乳中印记特性分析
为了阐明ZmVIL1在授粉后10~28 d胚乳中的印记情况, 我们又分离了B73×Mo17和Mo17×B73授粉后的10、12、16、18、20、22、24、26、28 d胚乳。同样用ZmVIL1-imprinting特异性引物扩增胚乳cDNA, 用Hph I对PCR产物酶切消化后进行电泳。结果显示在B73×Mo17和Mo17×B73 12~28 d胚乳中, 来自母本等位基因ZmVIL1完全沉默, 仅来自父本的等位基因表达, 因此母本等位基因ZmVIL1在12~28 d胚乳中处于持续印记状态(图6)。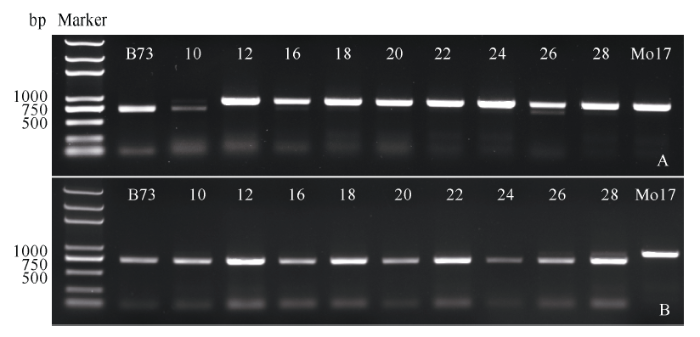 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6ZmVIL1在B73、Mo17正反交授粉后10~28 d胚乳中印记特性分析
A: ZmVIL1在B73×Mo17授粉后10~28 d胚乳中的印记特性分析; B: ZmVIL1在Mo17×B73授粉后10~28 d胚乳中的印记特性分析; Marker: D2000 plus。
-->Fig. 6Imprinting characterization analysis of ZmVIL1 in 10-28 DAP endosperm of B73 and Mo17 reciprocal hybrids
A: imprinting characterization analysis of ZmVIL1 in 10-28 DAP endosperm of B73×Mo17; B: imprinting characterization analysis of ZmVIL1 in 10-28 DAP endosperm of Mo17×B73; Marker: D2000 plus.
-->
2.6 ZmVIL1的组织特性表达分析
转录组测序技术为研究基因的时空表达模式提供了极大的便利。利用Davidson等[28]、Li等[29]、Wang等[30]、Jia等[31]转组录测序结果中ZmVIL1的表达丰度对该基因的时空表达特性进行分析。从ZmVIL1基因的整体表达情况来看, 该基因在玉米生殖生长阶段表达量较高, 其中在雌穗、花丝、雄穗和玉米授粉后10 d籽粒中表达量最高, 其次是在玉米胚珠、授粉后25 d的胚和胚乳中表达较高。在营养器官中, 根和顶端分生组织的表达量较高, 在玉米叶、叶舌和叶鞘中表达较低(图7)。由此揭示ZmVIL1不但在玉米籽粒发育中具有重要作用, 而且在玉米雄穗和雌穗的发育中也具有重要作用。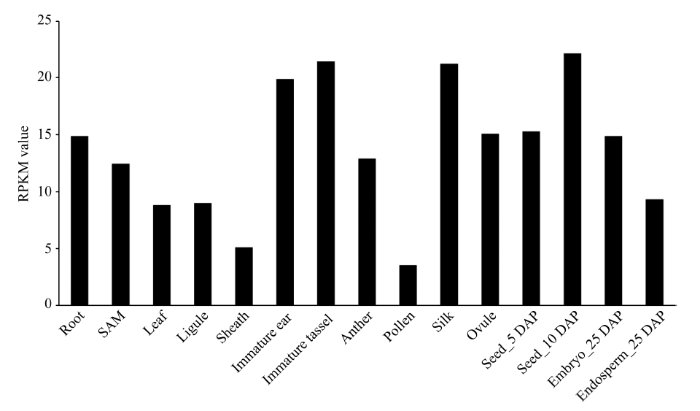 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7ZmVIL1在玉米不同组织中的表达
Root: 根; SAM (shoot apical meristem): 顶端分生组织; Leaf: 叶; Ligule: 叶舌; Sheath: 叶鞘; Immature ear: 雌穗; Immature tassel: 雄穗; Anther: 花药; Pollen: 花粉; Silk: 花丝; Ovule: 胚珠; Seed_5 DAP: 授粉后5 d籽粒; Seed_10 DAP: 授粉后10 d籽粒; Embryo_25 DAP: 授粉后25 d胚; Endosperm_25 DAP: 授粉后25 d胚乳。
-->Fig. 7Expression pattern of ZmVIL1 in different maize tissues
-->
3 讨论
玉米是我国第一大粮食作物, 玉米生产在我国粮食安全体系中具有重要作用。胚乳是玉米经济性状的重要表现, 胚乳的发育对种子的大小具有显著的影响[32]。胚乳发育分子生物学研究对于了解玉米籽粒发育的遗传及调控具有重要意义。印记基因主要存在于开花植物胚乳中[33]。ZmVIL1是一个印记基因[11], 我们利用RACE克隆了ZmVIL1基因cDNA的全长, 预测编码蛋白由598个氨基酸组成, 具有典型的锌指蛋白和纤连蛋白保守结构域, 这与拟南芥VIN3、VIL1蛋白保守结构功能域是一致的, 由此可以推测ZmVIL1在功能上可能具有保守性。印记基因的表达具有亲源效应[34], 其等位基因因亲本来源的不同致使其表达呈现一定的差异, 不同的印记基因有不同的印记特性。例如, ZAG2在胚和胚乳中均表现印记, 虽然ZAG2在B × M和178 × HC杂交组合中表现差异印记, 而在其他一些杂交组合表现二元印记, 但是ZAG2在所有杂交组合中均表现为父本特异性印记[12]; 不同于ZAG2, ZmVIL1只在胚乳中印记, 并且在所有的杂交组合中均表现母本基因特异性的二元印记; 与ZAG2相同的是, 其在授粉后12~28 d胚乳中表现持续印记。印记基因在种子发育过程中具有重要作用, Berger和Chaudhury[35]指出增加额外的父本基因组会使胚乳变大, 反之增加额外的母本基因组导致胚乳变小。Haig和Westoby[36]也认为印记基因在调控营养从母体到后代的转运中具有重要作用; 父本印记基因倾向于抑制营养的分配, 而母本印记基因则促进营养的转运。Meg1是第一个在植物中被发现的控制营养物质吸收及分配的印记基因。Costa等[13]研究表明玉米印记基因Meg1在籽粒发育过程中控制营养物质的分配与吸收, 在胚乳营养物质转运及细胞分化过程中是必不可少的; Meg1基因被敲除后, 籽粒中果糖、葡萄糖含量显著降低, 籽粒变小; 反之提高Meg1的表达量, 玉米籽粒则显著增大。结合Davidson等[28]对籽粒转录组测序结果, 在授粉后10 d籽粒中ZmVIL1表达量在所有组织中最高, 由此可推测ZmVIL1可能在该时期的玉米籽粒发育中具有重要功能。
同时我们也注意到ZmVIL1在玉米花序中也具有很高的表达水平。ZmVIL1是拟南芥VIN3的同源基因。VIN3是VIN3/VERNALIZATION LIKE基因家族中的一员[27,37], 这个基因家族成员还包括VIL1-4。VIN3、VIL1和VIL2在拟南芥中是重要的促花基因; VIL1和VIN3共同通过组蛋白修饰抑制FLC和FLM基因的表达, 从而促进拟南芥开花[27,37]。VIL2在无光周期诱导条件下抑制FLC同源基因MAF5从而促进开花[38]。此外, 在水稻中也鉴定出4个OsVIL基因(OsVIL1-4)[23]。OsVIL2功能的缺失导致无论在长日照或者短日照条件下水稻开花的延迟[39]。玉米是不需要经低温春化的短日照作物, ZmVIL1与拟南芥、水稻同源基因编码蛋白的分子系统进化分析显示(图8), ZmVIL1与水稻OsVIL3、OsVIL1和拟南芥VIL1聚为一个分支, 因此可以推测ZmVIL1在玉米花发育过程中也具有重要作用, 但由于在植物基因组长期的进化过程中, ZmVIL1与VIL1在功能上可能产生了分化。至于ZmVIL1在玉米花发育中是否具有重要作用及扮演何种角色需要进一步的研究。
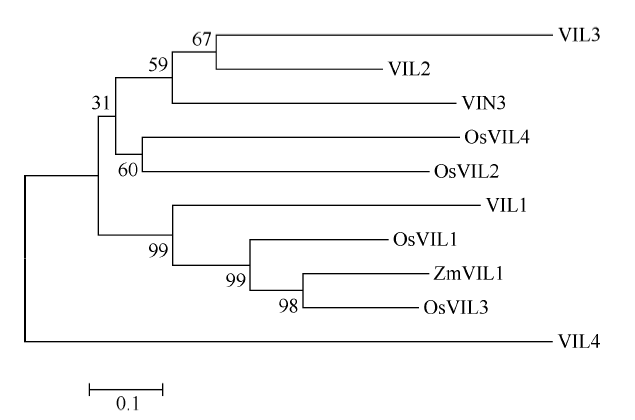 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图8ZmVIL1与拟南芥、水稻中VIN3蛋白家族分子进化 关系分析
-->Fig. 8Molecular evolution relationship analysis of ZmVIL1 with VIN3 protein family in Arabidopsis and rice
-->
4 结论
玉米胚乳印记基因ZmVIL1 cDNA全长2201 bp, CDS长1797 bp, 预测编码蛋白由598个氨基酸组成。在B73和Mo17正反交授粉后14 d的胚乳中来自母本的等位基因完全沉默, 而在胚中2个等位基因正常表达, ZmVIL1是一个父本表达的二元胚乳印记基因。在B73和Mo17正反交授粉后12~28 d胚乳中母本等位基因ZmVIL1处于持续印记状态。在郑58和昌7-2、黄C和178及PH6WC和PH4CV正反交授粉后14 d胚乳中, 均只有父本等位基因表达, 由此可知ZmVIL1又是一个基因特异性的印记基因。ZmVIL1在玉米籽粒、雄穗和雌穗中均有较高的表达量, 推测ZmVIL1在玉米生殖生长过程中具有重要功能。The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | . |
| [2] | . |
| [3] | . |
| [4] | . |
| [5] | . |
| [6] | . |
| [7] | . |
| [8] | . |
| [9] | . |
| [10] | . |
| [11] | . |
| [12] | . |
| [13] | . |
| [14] | . |
| [15] | . |
| [16] | . |
| [17] | . |
| [18] | . |
| [19] | |
| [20] | . |
| [21] | . |
| [22] | . |
| [23] | . |
| [24] | . |
| [25] | . |
| [26] | . |
| [27] | . |
| [28] | . |
| [29] | . |
| [30] | . |
| [31] | . |
| [32] | . |
| [33] | . |
| [34] | . |
| [35] | . |
| [36] | . |
| [37] | . |
| [38] | . |
| [39] | . |
