 ,1, 叶俊华
,1, 叶俊华 ,2, 程朝平1, 魏兴华2, 叶新福
,2, 程朝平1, 魏兴华2, 叶新福 ,1,*, 杨窑龙
,1,*, 杨窑龙 ,2,*
,2,*Xian-geng identification by SNP markers in Oryza sativa L.
ZHENG Xiang-Hua ,1, YE Jun-Hua
,1, YE Jun-Hua ,2, CHENG Chao-Ping1, WEI Xing-Hua2, YE Xin-Fu
,2, CHENG Chao-Ping1, WEI Xing-Hua2, YE Xin-Fu ,1,*, YANG Yao-Long
,1,*, YANG Yao-Long ,2,*
,2,*通讯作者: *杨窑龙, E-mail:yangxiao182@126.com;叶新福, E-mail:yexinfu@126.com
收稿日期:2020-12-1接受日期:2021-04-26网络出版日期:2022-06-16
| 基金资助: |
Received:2020-12-1Accepted:2021-04-26Published online:2022-06-16
| Fund supported: |
作者简介 About authors
郑向华,E-mail:zxhua57@126.com;
叶俊华,E-mail:yejunhua1994@qq.com

摘要
亚洲栽培稻(Oryza sativa L.)分为籼、粳2个亚种, 随着杂交水稻的发展、种间杂种优势的利用, 籼粳之间的界限变得越来越模糊。本研究利用3000份水稻种质资源信息, 通过计算约2000万个单核苷酸多态性(single nucleotide polymorphism, SNP)位点的SNP-index值, 进行籼粳特异SNP位点筛选, 最终得到4084个籼粳特异SNP位点(4k-SNP); 同时确定以籼粳指数作为水稻品种籼粳鉴定的指标。研究进一步采用大规模简单随机取样等统计分析方法对籼粳特异位点进行数据降维处理, 将4k-SNP精简至40个SNP位点(40-SNP), 用于水稻籼粳鉴定。为了验证40-SNP的籼粳鉴定效果, 本研究一方面利用水稻生产上推广的82份选育品种, 对40-SNP籼粳鉴定结果与4k-SNP鉴定结果进行比较, 结果发现40-SNP与4k-SNP得出的粳型指数非常接近, 相关系数为0.99; 另一方面利用全球6类型(indica、aus、rayada、aromatic、tropical japonica、temperate japonica)水稻品种共49份材料, 对40-SNP籼粳鉴定结果与4k-SNP及程氏指数法籼粳鉴定结果进行比较, 发现40-SNP与4k-SNP及程氏指数法籼粳鉴定结果的相关系数分别在0.98和0.86以上。这些结果证实了40-SNP对水稻品种籼粳鉴定的有效性及准确性。另外发现40-SNP对水稻6种亚群类型也有很好鉴别效果, 其中indica的粳型指数 < 0.20, aus的粳型指数在0.20~0.40, rayada和aromatic的粳型指数在0.60~0.85之间, tropical japonica的粳型指数 > 0.90, temperate japonica粳型指数最高, 基本为1.00。本研究为研究水稻籼粳分化、杂种优势利用以及水稻种子管理条例制定等方面提供了数据支撑及理论基础。
关键词:
Abstract
Asian cultivated rice (Oryza sativa L.) is divided into two subspecies of xian and geng. With the development of hybrid rice and utilization of interspecific heterosis, the boundaries between xian and geng are becoming more and more vague. In this study, based on the SNP-index value of 20 million single nucleotide polymorphism (SNP) loci from 3000 rice germplasm resources, we captured 4084 xian-geng specific SNP loci named as 4k-SNP and used the xian-geng index as an indicator for xian-geng identification. Furthermore, the 4k-SNP was reduced to 40-SNP (40 SNP loci) for indica/japonica identification by using the statistical analysis methods such as large-scale simple random sampling based on the dimensionality reduction algorithms. To verify the effectiveness of 40-SNP on xian-geng identification, 82 bred varieties were used in this study to compare the results of 40-SNP xian-geng identification and 4k-SNP identification. The result showed that the geng index obtained from 40-SNP and 4k-SNP were very close, and the correlation coefficient was 0.99. Moreover, a total of 49 varieties, belonging to six subgroups (indica, aus, rayada, aromatic, tropical japonica, and temperate japonica), were used to compare the xian-geng identification results of 40-SNP with those of 4k-SNP and Cheng’s index. And the correlation coefficients of xian-geng identifications between 40-SNP and 4k-SNP as well as between 40-SNP and Cheng’s index were above 0.98 and 0.86, respectively. These results verified the validity and accuracy of 40-SNP on xian-geng identification in Oryza sativa L. In addition, 40-SNP also had a good distinguishability for the six subgroups in rice, and the xian-geng index of indica, aus, rayada, aromatic, tropical japonica, and, temperate japonica was less than 0.20, 0.20-0.40, 0.60-0.85, more than 0.90, and 1.00, respectively. This study provides the data and theoretical basis for the differentiation of xian-geng and the utilization of heterosis and, formulation of seed management regulations.
Keywords:
PDF (1614KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
郑向华, 叶俊华, 程朝平, 魏兴华, 叶新福, 杨窑龙. 利用SNP标记进行水稻品种籼粳鉴定. 作物学报, 2021, 48(2): 342-352 DOI:10.3724/SP.J.1006.2022.02085
ZHENG Xiang-Hua, YE Jun-Hua, CHENG Chao-Ping, WEI Xing-Hua, YE Xin-Fu, YANG Yao-Long.
经过半个多世纪众多****的研究, 运用形态学、生理学、生态学、血清学、杂种亲和性、同工酶及分子标记技术, 最终确认籼稻和粳稻是普通栽培稻分化最为深刻和最为主要的2个方向。一般而言, 在形态上, 籼稻耐湿耐热、茎秆柔软、分蘖力强、叶片较阔、叶色淡绿、粒形细长、颖毛短少、易落粒、米质不黏等, 适于在热带和亚热带生长; 粳稻则耐旱耐寒、茎秆坚硬、分蘖力弱、叶片较狭、叶色浓绿、粒形短圆、颖毛长密、不易落粒、米质黏性, 适于在温带和热带、亚热带的高海拔地区生长。在抗逆、抗病性上, 籼稻一般较耐热、不耐肥, 对稻瘟病抵抗性较强; 粳稻耐寒性较强、耐肥, 稻瘟病抗性较弱。籼稻和粳稻在地理分布上也有明显不同, 籼稻比较适宜在低纬度、低海拔的湿热地区种植, 在我国主要分布于华南热带和淮河以南的亚热带低地, 粳稻比较适宜在高纬度或低纬度的高海拔地区生长, 在我国主要分布于华东太湖流域和华北、西北、东北等温度较低的地区以及南部热带、亚热带的高地[1]。
随着水稻杂交育种以及水稻功能基因组学的不断发展, 大量水稻杂种不育基因的克隆与利用, 使得籼粳亚种间不亲合性得到有效解决, 从而促进了籼粳杂交育种的发展, 使籼粳亚种间强大的杂种优势得到有效利用[2,3,4]。然而所有这些研究的基础是正确地判定籼、粳亚种属性。籼、粳亚种鉴定对水稻种质资源的利用有着至关重要的作用。Cheng等[5]建立了包括稃毛、酚反应、1~2穗节长、抽穗期壳色、叶毛以及籽粒长宽比的6个性状, 即程氏指数法; 陈跃进等[6]对指标进行调整, 去掉了籽粒长宽比, 增加了KClO3抗性指标。另外也有****运用生理生化指标来鉴定籼粳, 徐正进等[7]利用水稻节的大小维管束数目来判定籼粳, 籼稻穗颈节的大小维管束数目比约为1.0, 而粳稻约为0.5。籼稻倒2节与穗颈节的大维管束数目比约为1.5, 而粳稻约为2.5。另外, 同工酶标记也被用于籼粳鉴定[8,9]。近年来, 由于分子标记的发展, 利用分子标记的方法鉴定籼粳已经成为最主流的方法, 其中利用简单序列重复(simple sequence repeats, SSR)[10,11,12]和插入缺失(insertion-deletion, InDel)标记最为方便和快捷。Lu等[13]利用日本晴和9311基因组信息筛选到34个籼粳特异InDel标记用于籼粳鉴定, 并采用籼粳指数作为判定籼粳的重要指标。该方法操作方便简单, 重复性好, 是一个比较实用的籼粳鉴定方法。
随着测序技术的发展及测序成本的降低, 单核苷酸多态性(single nucleotide polymorphism, SNP)已经逐渐取代SSR和InDel标记, 成为最主要的分子标记, 在基因克隆[14]、遗传结构判定[15]、全基因组关联分析[16,17]等方面得到了有效利用。SNP有其独特的优点: 分布广泛, 基因组密度高; 较SSR相比, 具有更高的遗传稳定性; 一般SNP具有2个等位类型, 多态性较低, 但更加方便与计算机技术相结合, 适用于高效自动化分析。因此, 本研究基于基因组丰富的SNP信息, 借助大数据方法筛选出籼粳特异的SNP位点, 进一步利用统计分析方法精简至适宜的SNP数目, 最终确定一套用于鉴定籼、粳亚种的SNP集。并且利用生产上推广的选育品种以及全球部分水稻资源对已构建的SNP集进行籼、粳鉴定成效进行验证, 从而证实这套SNP集在籼、粳亚种鉴定过程中具有良好的应用前景。
1 材料与方法
1.1 供试材料
选取生产上推广的82份选育品种, 包括64个籼稻(含常规稻与杂交稻)、13个粳稻以及5个籼粳杂交稻品种, 所用试验材料由中国水稻研究所国家作物种质中期库提供(附表1); 同时选取根据Wang等[12]和王彩红等[18]通过SSR分子标记已明确类型的49份亚种栽培稻品种, 该部分试验材料由国际水稻研究所国际水稻资源中心(International Rice Germplasm Center, IRGC)提供(附表2)。所有试验材料均种植于中国水稻研究所杭州市富阳试验基地。单本移栽, 每份材料设4行, 每行6株, 种植间距为20 cm × 20 cm。田间正常水肥管理, 待水稻成熟后, 每个株系收取中间单株的种子, 干燥后保存, 备用。
1.2 试验方法
1.2.1 DNA提取及全基因组测序 所有试验材料的种子, 浸种2 d, 催芽1 d后, 置于铺有一层滤纸的培养皿中, 放到光照培养箱中进行发芽成苗。当幼苗长至一叶一心期, 取幼叶至液氮条件下研磨成粉, 约0.2 g粉末样品用于DNA提取。采用CTAB法[19]进行水稻叶片基因组DNA提取, 并将提取的DNA溶解于灭菌双蒸水中, 于-20ºC环境下保存备用。通过Qubit 4.0以及琼脂凝胶电泳的方法分别检测基因组DNA的浓度与纯度, 以确保其质量满足DNA文库构建的要求, 即无污染、无降解, 总量大于1.0 µg, 浓度大于20 ng µL-1。用超声波DNA打断仪(Covaris S220, 美国)将基因组DNA处理成350 bp左右的DNA片段, 利用NEB Next Ultra DNA Library Prep试剂盒(Illumina, 美国)进行基因组DNA文库构建, 构建过程中利用安捷伦2100高灵敏DNA反应试剂盒(安捷伦科技有限公司, Waldbronn, Germany)以及Qubit 4.0进行文库质量控制。本研究构建了82份选育品种以及49份全球水稻品种的基因组DNA文库。构建好的文库采用PE150双末端测序法在Illumina NovaSeq 6000平台进行测序, 基因组覆盖度为25倍。
1.2.2 测序数据质量控制与数据处理 分析测序原始数据碱基错误率, 并对测序原始数据(raw reads)进行过滤, 去除接头、低质量的reads等, 获得clean reads。利用BWA[20]及PICARD等生物信息学工具, 经mem参数将clean reads与参考基因组序列(日本晴序列, IRGSP-1.0)进行比对与定位。比对分析后, 利用GATK[21]和SAMtools进行变异提取。利用Plink以及VCFtools工具在整个基因组的变异信息中提取目标SNP基因型信息。利用ANNOVAR软件包进行SNP基因功能注释[22]。
1.2.3 籼粳特异SNP鉴定 ΔSNP-index被用来筛选籼、粳特异标记(图1-A), 本研究中SNP-index定义为: SNP-index = 1-Nref/Nall。其中, Nref是指在一个群体中, 某一SNP位点与参照基因组(粳稻品种: 日本晴)具有相同的等位型的品种数目; Nall是指在一个群体中, 某一SNP位点上所有的具有确定基因型的品种数目; ΔSNP-index = SNP-indexx - SNP- indexg。其中, SNP-indexx和SNP-indexg分别为籼稻群体与粳稻群体的SNP-index。ΔSNP-index越接近1, 说明该SNP位点在籼、粳亚种中特异性越高, 该位点即为籼粳特异位点。采用籼粳指数作为水稻籼粳鉴定的指标, 其中籼粳指数分为籼型指数与粳型指数。其中, 籼型指数是指籼型等位变异个数与所有等位变异个数的比值; 粳型指数是指粳型等位变异个数与所有等位变异个数的比值。
1.2.4 统计分析 简单随机取样方法由于数据量大, 采用perl程序编写小脚本, 利用计算机运算进行取样(重复数为1000次), 同时计算每一组SNP的籼粳指数。t-检测、方差分析等统计运算主要是利用Microsoft Excel 2010以及SAS 8.0 (SAS, Inc., Cary, North Carolina, USA)软件计算。
2 结果与分析
2.1 籼粳特异标记获取原则
利用3000份水稻品种重测序数据信息(分别对籼稻群体和粳稻群体中的每个SNP位点的SNP-index进行计算, 并计算每个SNP位点的ΔSNP-index值, 挑选出ΔSNP-index大于0.7的SNP标记, 约50万个。依据筛选条件: (1) 缺失率小于0.05; (2) 位于非转座子中; (3) 连锁不平衡系数r2 < 0.1; 最终得到4084个籼粳特异SNP, 并将其命名为4k-SNP (图1-B)。
图1
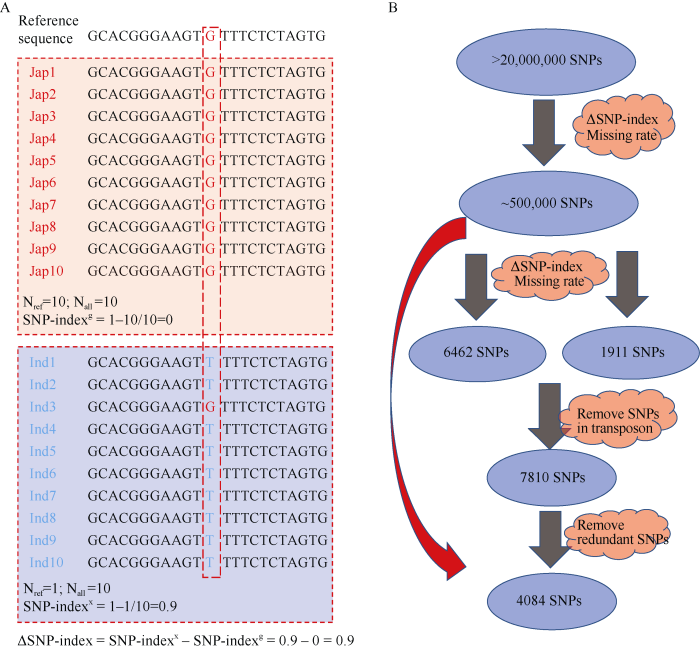 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1籼粳特异标记获取
A: ΔSNP-index计算方法; B: 籼粳特异标记获取流程图。
Fig. 1Acquisition of special xian-geng markers
A: the calculation method of ΔSNP-index; B: the flow chart of specific xian-geng markers acquisition.
2.2 最小SNP数目确定
用于鉴定籼粳成分的籼粳特异标记越多, 其鉴定结果越准确, 但籼粳特异标记越多成本越高、速度越慢。如何在保证精确度的前提下尽量减少标记数目则是关键所在。从82份选育品种中选取9份作为确定最小SNP数目的材料, 包括3份粳稻常规稻(楚粳27、秀水03、南粳46)、3份籼稻(扬两优6号、丰两优1号、荆两优10号)以及3份籼粳杂交稻(甬优9号、甬优12、甬优1512)。经重测序后, 获得此9份选育品种的4k-SNP基因型, 并计算其籼型指数或粳型指数(后续中均只计算粳型指数)。结果发现, 3个粳稻材料的粳型指数均大于0.9, 3个籼稻杂交稻的粳型指数处在0.1左右, 而3个籼粳杂交稻的粳型指数在0.5左右(表1)。这些数据在一定程度上说明4k-SNP可以较为准确地区分水稻品种籼、粳属性。
Table 1
表1
表19份水稻选育品种的粳型指数与最小SNP数目
Table 1
| 名称 Variety name | 类型 Type | 粳型指数 Geng index | 最小SNP数 Minimum number of SNPs |
|---|---|---|---|
| 楚粳27 Chujing 27 | 粳稻 Japonica | 0.933 | 35 |
| 秀水03 Xiushui 03 | 粳稻 Japonica | 0.967 | 40 |
| 南粳46 Nanjing 46 | 粳稻 Japonica | 0.979 | 35 |
| 扬两优6号 Yangliangyou 6 | 籼稻 Indica | 0.052 | 35 |
| 丰两优1号 Fengliangyou 1 | 籼稻 Indica | 0.088 | 40 |
| 荆两优10号 Jingliangyou 10 | 籼稻 Indica | 0.050 | 45 |
| 甬优12 Yongyou 12 | 籼粳交 Indica-japonica hybridization | 0.518 | 40 |
| 甬优1512 Yongyou 1512 | 籼粳交 Indica-japonica hybridization | 0.466 | 35 |
| 甬优9号 Yongyou 9 | 籼粳交 Indica-japonica hybridization | 0.514 | 40 |
新窗口打开|下载CSV
将4k-SNP进行基因分型得到的粳型指数作为期望值。从4k-SNP中随机选取不同数目的SNP集(从5个SNPs到1000个SNP, 每个SNP集简单随机取样重复1000次), 分别计算不同SNP集每次取样的粳型指数, 得到随机值, 继而对随机值与期望值进行比较分析。结果发现随机值围绕着期望值上下波动, 并且随着标记数的增加, 这种波动幅度逐渐变小(图2-A和附图1)。对每个标记数的随机值进行标准差计算, 结果发现, 当标记数很少的时候, 标准差比较大, 达到0.077。随着标记数的增多, 标准差明显下降, 下降的趋势则随着标记数的增多而变得平缓(图2-B)。就整体而言, 标准差一直处于下降趋势。
图2
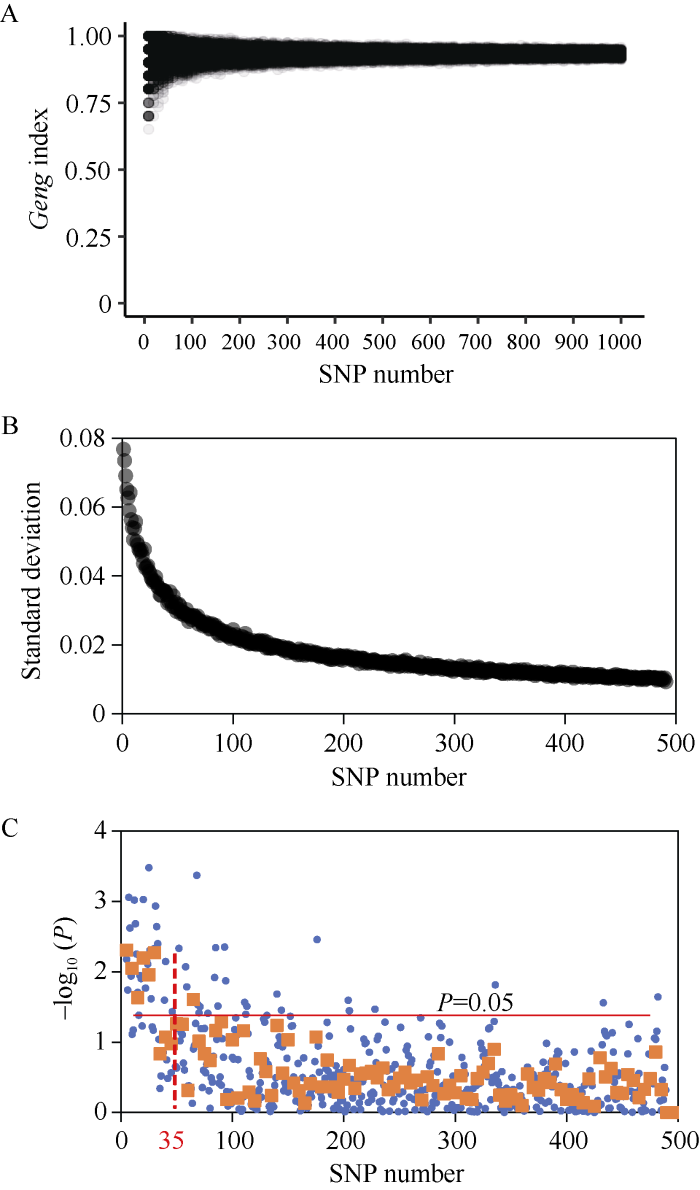 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2最少SNP标记数目确定
A: 粳型指数与SNP数目的散点图; B: 粳型指数的标准差与SNP数目的曲线图; C: 利用统计分析确定最小标记数目。P值计算是通过对每5个SNP数目与相邻5个SNP数目粳型指数的标准差进行t检测而得出; 红色实线是指P = 0.05的阈值; 蓝色圆点是指-log10(P)的值; 红色方块是指每5个SNP数目-log10(P)的平均值。
Fig. 2Determination of minimum number of SNPs
A: scatter chart of geng index of different SNPs with 1000 repetitions; B: curve chart of standard deviation of geng index of different SNPs; C: statistical analysis for determining minimum SNPs. P-value was calculated from the t-test between the standard deviation of every five continuous SNP numbers and that of next five continuous SNP numbers. Red line indicates the threshold of P-value (P = 0.05), blue point indicates -log10(P) value, and red square indicates the average -log10(P) value of each five SNP numbers.
然而, 如何在准确度与标记数之间找到平衡点?由SNP数目和标准差做出的曲线图(图2-B)可以看出, 标准差的下降先陡后缓, 达到平台期的SNP数目可作为最少SNP数目。本研究采用滑窗计算来鉴定最小SNP数目, 以任意10个SNP数目为窗口, 以1个SNP数目为步长做滑窗计算。对窗口内前5个SNP数目粳型指数的标准差与后5个SNP数目的粳型指数标准差进行差异显著性比较, 若标准差无显著性差异, 则此时对应的SNP数目为最小SNP数目, 且为随机SNP标记所需要的最小SNP数目(图2-C)。我们通过对上述9份品种的最小SNP数目进行分析, 以P=0.05为阈值发现, 当前后相邻5个SNP数目的粳型指数标准差无显著差异时, 最小随机SNP数目位于35~45之间(表1), 最终确定籼、粳亚种鉴定所需的最小SNP标记的数目为40 (40-SNP)。
2.3 SNP位点的确定
40-SNP位点的确定主要考虑以下4个条件: (1) SNP分布; (2) 缺失率; (3) ΔSNP-index值; (4) 可能的功能位点。最终从4k-SNP中选取40个SNP用于水稻籼粳鉴定(表2)。对于SNP分布, 我们发现每条染色体选取SNP数目为2~4个, 染色体分布比较平均(附图2-A); 另外, 40-SNP也比较均匀地分布于水稻基因组上, 能够一定程度上体现整个基因组上的籼粳成分信息(附图2-B)。40个SNP在3000份水稻品种中的缺失率均小于5%, 说明在大部分水稻品种中均包含40-SNP位点信息。就ΔSNP-index值而言, 40-SNP的ΔSNP-index值也比较高, 位于0.7~0.8之间, 说明这些位点籼粳特异程度也较高(表2)。对40-SNP位点进行基因注释, 结果显示, 有19个SNP位于外显子中, 其中18个SNP为非同义突变, 改变了基因编码的蛋白序列, 1个为同义突变。另外, 3个SNP位于内含子, 7个SNP位于基因间隔区, 其余SNP位于基因上下游及非翻译区等(附表3和附图3)。Table 2
表2
表240个籼粳特异SNP位点的具体信息
Table 2
| 编号 ID | 染色体 Chr. | 位置 Position | 粳型SNP Geng SNP | 籼型SNP Xian SNP | 杂合型SNP Hybrid SNP | ΔSNP-index | SNP位置 SNP position | 突变类型 Mutation type |
|---|---|---|---|---|---|---|---|---|
| IG1 | 1 | 12,556,378 | A | T | A/T | 0.757 | Exonic | 非同义突变 Non-synonymous SNV |
| IG2 | 1 | 17,768,964 | C | A | C/A | 0.715 | Downstream | |
| IG3 | 1 | 22,381,235 | T | G | T/G | 0.709 | UTR3 | |
| IG4 | 1 | 28,913,384 | C | T | C/T | 0.746 | Exonic | 非同义突变 Non-synonymous SNV |
| IG5 | 2 | 12,079,928 | A | G | A/G | 0.752 | Exonic | 非同义突变 Non-synonymous SNV |
| IG6 | 2 | 20,225,356 | T | G | T/G | 0.749 | Upstream | |
| IG7 | 2 | 29,516,322 | C | G | C/G | 0.747 | Exonic | 非同义突变 Non-synonymous SNV |
| IG8 | 2 | 33,038,088 | C | A | C/A | 0.755 | Exonic | 非同义突变 Non-synonymous SNV |
| IG9 | 3 | 2,936,058 | G | A | G/A | 0.737 | Intronic | |
| IG10 | 3 | 3,461,333 | T | C | T/C | 0.730 | Intergenic | |
| IG11 | 3 | 4,109,581 | C | T | C/T | 0.745 | Exonic | 非同义突变 Non-synonymous SNV |
| IG12 | 3 | 32,716,455 | A | C | A/C | 0.793 | Upstream | |
| IG13 | 4 | 12,290,320 | A | G | A/G | 0.745 | Exonic | 非同义突变 Non-synonymous SNV |
| IG14 | 4 | 20,612,989 | C | T | C/T | 0.745 | Intergenic | |
| IG15 | 4 | 24,973,823 | T | C | T/C | 0.717 | Exonic | 非同义突变 Non-synonymous SNV |
| IG16 | 5 | 21,148,417 | A | C | A/C | 0.727 | Upstream | |
| IG17 | 5 | 25,953,822 | C | G | C/G | 0.788 | UTR3 | |
| IG18 | 6 | 12,025,192 | A | G | A/G | 0.787 | Downstream | |
| IG19 | 6 | 20,502,262 | G | A | G/A | 0.769 | Exonic | 非同义突变 Non-synonymous SNV |
| IG20 | 6 | 31,049,929 | C | T | C/T | 0.702 | Exonic | 非同义突变 Non-synonymous SNV |
| IG21 | 7 | 8,962,741 | C | A | C/A | 0.736 | Intergenic | |
| IG22 | 7 | 14,923,259 | A | G | A/G | 0.778 | Exonic | 同义突变 Synonymous SNV |
| IG23 | 7 | 17,,136,369 | T | C | T/C | 0.722 | Exonic | 非同义突变 Non-synonymous SNV |
| IG24 | 7 | 28,782,595 | C | T | C/T | 0.740 | Intronic | |
| IG25 | 8 | 14,427,488 | C | A | C/A | 0.751 | Intergenic | |
| IG26 | 8 | 18,825,519 | A | G | A/G | 0.726 | Exonic | 非同义突变 Non-synonymous SNV |
| IG27 | 8 | 21,801,474 | G | A | G/A | 0.791 | Upstream | |
| IG28 | 8 | 28,268,656 | T | C | T/C | 0.749 | Exonic | 非同义突变 Non-synonymous SNV |
| IG29 | 9 | 3,099,717 | T | C | T/C | 0.794 | Exonic | 非同义突变 Non-synonymous SNV |
| IG30 | 9 | 4,396,038 | C | T | C/T | 0.732 | Intergenic | |
| IG31 | 9 | 14,812,651 | T | C | T/C | 0.756 | Exonic | 非同义突变 Non-synonymous SNV |
| IG32 | 9 | 16,775,838 | G | A | G/A | 0.743 | Intergenic | |
| IG33 | 10 | 14,820,587 | T | C | T/C | 0.782 | Downstream | |
| IG34 | 10 | 20,325,921 | G | A | G/A | 0.796 | Downstream | |
| IG35 | 10 | 21,759,092 | G | A | G/A | 0.723 | Exonic | 非同义突变 Non-synonymous SNV |
| IG36 | 11 | 808,969 | A | G | A/G | 0.761 | Intergenic | |
| IG37 | 11 | 5,533,977 | G | A | G/A | 0.737 | Exonic | 非同义突变 Non-synonymous SNV |
| IG38 | 12 | 18,352,099 | C | G | C/G | 0.746 | Exonic | 非同义突变 Non-synonymous SNV |
| IG39 | 12 | 24,494,788 | A | C | A/C | 0.736 | Intronic | |
| IG40 | 12 | 27,263,526 | G | T | G/T | 0.750 | Upstream |
新窗口打开|下载CSV
2.4 利用40-SNP进行籼粳鉴定验证
为了验证40-SNP的可靠性, 分别计算82份选育品种在4k-SNP与40-SNP条件下的粳型指数。发现每个品种在4k-SNP与40-SNP条件下的粳型指数均很接近。相关分析结果表明(图3), 40-SNP条件下得到的粳型指数与4k-SNP条件下的粳型指数成极显著正相关(P < 0.001), 相关系数高达0.99, 说明40-SNP计算得出的粳型指数基本可以替代4k-SNP的粳型指数。从而也证实了用40-SNP进行籼、粳亚种鉴定的真实性和有效性。图3
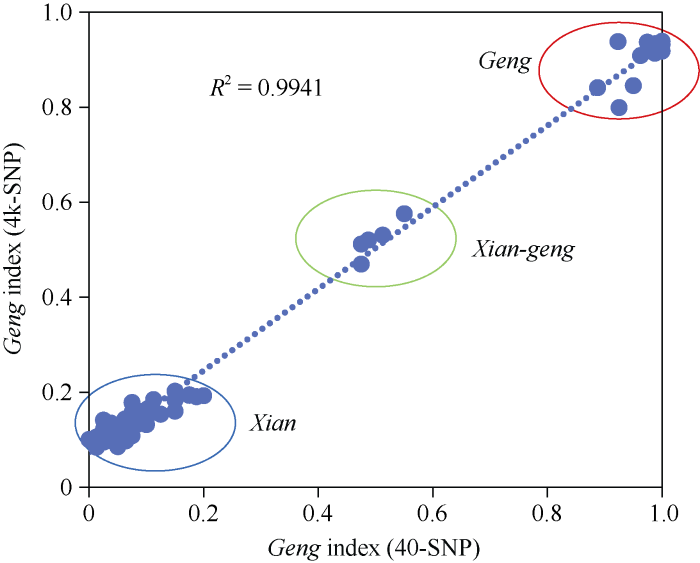 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3利用82份水稻选育品种进行40-SNP与4k-SNP的粳型指数比较
Fig. 3Comparison of geng index between 40-SNP and 4k-SNP in 82 bred rice varieties
众所周知, 程氏指数法是早期进行籼、粳亚种鉴定常用的方法, 该方法利用6性状的表型特征来判别水稻籼、粳属性, 操作简单有效。前期在亚洲栽培稻程氏指数与SSR标记分类的比较分析中[18], 我们获得了用于本研究的49份全球水稻资源的程氏指数数据。在本研究中, 分别用4k-SNP和40-SNP计算49份品种的粳型指数, 发现二者相关系数高达0.98 (P < 0.001) (图4-A)。同时我们将40-SNP鉴定的粳型指数与程氏指数进行相关性分析, 结果显示二者呈极显著正相关(P < 0.001), 相关系数达0.86 (图4-B)。说明利用基因型数据进行籼粳鉴定与表型数据进行鉴定结果相似, 也从另一方面证实了40-SNP的可靠性。
图4
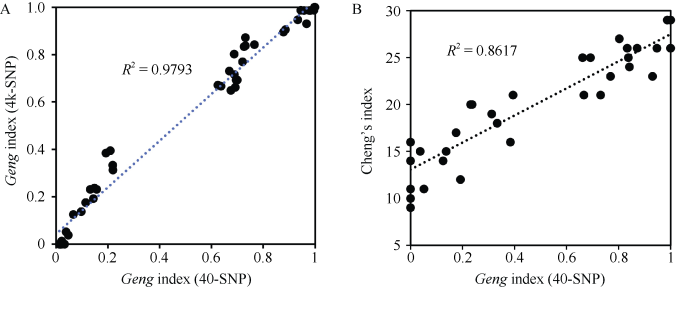 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4利用49份全球水稻品种验证40-SNP鉴定籼粳的准确性
A: 40-SNP与4k-SNP计算得出的粳型指数相关性分析; B: 40-SNP的粳型指数与程氏指数相关性分析。
Fig. 4Accuracy of xian-geng identification for 40-SNP in 49 global rice varieties
A: correlation analysis of geng index between 40-SNP and 4k-SNP; B: correlation analysis between geng index calculated by 40-SNP and Cheng’s index.
2.5 40-SNP可以进行水稻亚群鉴定
本研究选用的49份全球水稻资源, 包含6种水稻亚群类型(indica、aus、rayada、aromatic、tropical japonica、temperate japonica)。对各品种的粳型指数值归类研究发现, 6种水稻亚群的粳型指数具有一定的差异性和规律性。其中, 温带粳稻(temperate japonica)类型的粳型指数最高, 基本为1.00; 紧接着的是热带粳稻(tropical japonica)类型, 其粳型指数基本在0.90以上; 再接着的是aromatic和rayada类型, 其粳型指数在0.60~0.85之间; 之后是aus类型, 其粳型指数在0.20~0.40, 而最后indica类型的粳型指数处在0.20以内(图5)。方差分析结果显示6类水稻品种的粳型指数存在显著差异, 且遗传距离越远的品种, 差异也就越明显(表3)。图5
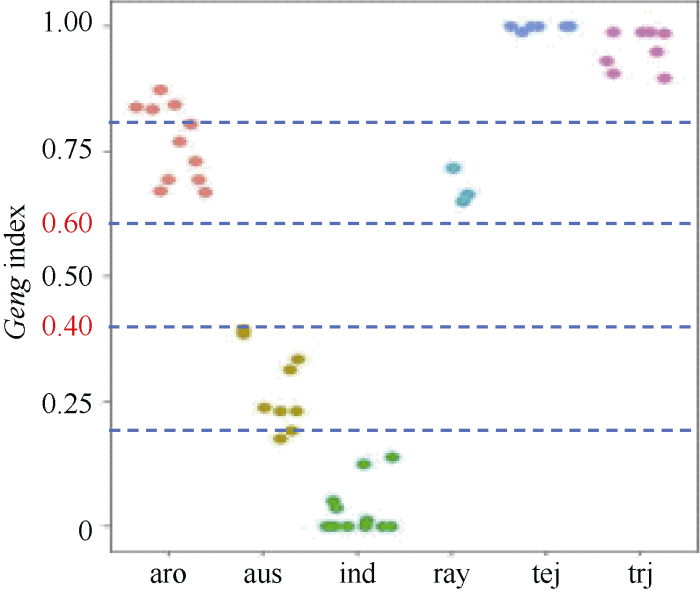 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5水稻6类型粳型指数分布
Fig. 5Geng index distribution of six types of rice varieties
aro: aromatic; aus: aus; ind: indica; ray: rayada; tej: temperate japonica; trj: tropical japonica.
40-SNP的籼粳热图(图6)可以发现, 从温带粳稻(temperate japonica)至indica类型, 粳型指数越来越小的类型, 其粳稻成分越来越少, 反之则籼稻成分越来越多。从而推测, 6种水稻亚群类型品种的差异很大程度上是由籼、粳成分的不同所造成的。因而可以借助40-SNP, 计算水稻品种的籼粳指数, 从而鉴定品种类型。
图6
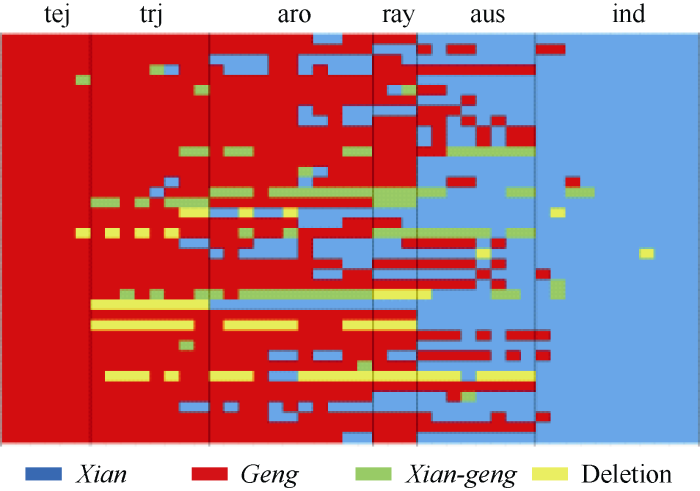 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图649份全球水稻品种籼粳成分热图
Fig. 6Heat map of xian-geng components of 49 rice varieties
Table 3
表3
表3水稻六类型粳型指数方差分析
Table 3
| 类型 Type | 平均值±标准差 Mean ± SD | 多重比较 Multiple comparison (LSD, P=0.05) | 多重比较 Multiple compariso n (LSD, P=0.01) | F值 F-value | P值 P-value |
|---|---|---|---|---|---|
| tej | 0.998 ± 0.005 | a | A | 2.43 | < 0.001 |
| trj | 0.953 ± 0.039 | b | A | ||
| aro | 0.765 ± 0.077 | c | B | ||
| ray | 0.676 ± 0.036 | c | B | ||
| aus | 0.277 ± 0.082 | d | C | ||
| ind | 0.030 ± 0.050 | e | D |
新窗口打开|下载CSV
3 讨论
早在20世纪初, 科学家们就已经开始籼、粳亚种鉴定的研究, 主要是从学术角度填补水稻分类学上的空白[23,24,25,26,27,28]。随着现代育种进程的发展以及亚种内与亚种间杂交的不断利用, 籼粳之间的界限变得越来越模糊, 在新的发展时代, 对籼粳鉴定提出了新的要求。利用分子标记的方法鉴定水稻籼粳是一种快捷高效的方法, 它可以消除水稻季节性与生命周期的影响, 具有重大应用前景。除学术研究需求外, 鉴定籼、粳亚种类型还存在以下一系列政策性的需求: (1) 稻谷收购与储存的需要。目前, 稻谷的收购价在籼稻、粳稻之间存有差异, 市场上一般采用观察粒形来区分籼稻和粳稻, 但这并不准确; (2) 稻米进口配额的需要。稻米进口过程中, 籼稻、粳稻都有各自的配额; 一般来说, 籼稻配额不够, 会产生用籼稻冒用粳稻的情况, 因此也需要能够正确判别籼粳的技术支持; (3) 品种管理的需要。籼稻与粳稻进行区试的要求不一样, 粳稻要求相对籼稻较低; 对于越来越多的籼粳杂交后代, 对其进行分类显得尤为重要。因此, 迫切需要相应的高效水稻品种籼粳鉴定方法。籼粳鉴定最经典的方法是程氏指数法[1,5]。本研究40-SNP鉴定的结果与程氏指数法鉴定的结果接近, 但与程氏指数法相比, 本研究的鉴定方法不需要种植水稻, 不受季节性影响, 只需要少量的水稻任意部位的组织进行DNA提取即可, 方便快捷; 结合高效的SNP分型技术, 如KASP、芯片定制、多重PCR等方法, 能够实现水稻籼粳鉴定的规模化。分子标记法鉴定水稻品种籼粳特性是随着分子标记的发展而不断变化, 从一开始的RFLP、RAPD标记, 到后来的SSR标记以及InDel标记。其中应用最为广泛的分子标记法是Lu等[13]利用日本晴与9311基因组差异开发的34对InDel标记。本研究所用的SNP分子标记与其具有很大的区别: 首先, 标记来源不同, 前者InDel标记是从2个品种的基因组差异中筛选出来的, 虽然经过中性检验, 但由于品种数不多, 也很难真实代表籼、粳2个亚种。本研究SNP标记是从上千份籼、粳水稻品种中, 利用SNP-index筛选出来的, 能够较好地代表籼、粳亚种之间的特异标记。其次, 本研究对鉴定籼粳的标记数也经过统计分析, 对水稻籼粳特性鉴定更具有说服力。最后, 在标记鉴定能力的验证方面, 前者InDel标记并没有进行有效的验证, 本研究中最后筛选出来的40-SNP, 与程氏指数、4k-SNP鉴定结果进行比较, 并且验证所用的品种均为生产上应用的选育品种, 更能贴近水稻生产实际, 具有很大的应用前景。同时, 对挑选的40-SNP进行基因功能注释发现, 有19个位点位于基因中, 并且改变了基因编码的氨基酸, 其中, IG3位点, 其位于1号染色体22,381,235位置, 与SaM基因有关, 影响水稻籼粳亚种杂种不育, 是籼粳分化的关键位点[29,30]; IG35位点, 位于10号染色体21,759,092位置, 位于OsNRT1.1B基因内, 前人研究表明, 该位点的变异就是影响水稻籼粳对氮肥利用效率不同的主要原因[31,32]。除此之外, 本研究还发现利用40-SNP在一定程度上可以区分全球6类水稻资源。标准差在一定程度上可以反应数据的变异程度, 标准差越小则变异程度越低。在本研究中, 每个SNP数目重复1000次取样后, 随着SNP数目的增多, 标准差越来越小, 而且会一直变小。因此, 判断标准差何时到达平台期是很困难的。一般可以通过肉眼观察曲线图, 近似判断拐点的位置, 但这种方法得出的最小标记数目会因人而异, 具有盲目性, 结果难以统一。而本研究利用相邻的5个SNP数目之间进行标准差的显著性比较的方法来判断拐点位置, 该方法是经过统计验证, 具有科学性和可行性。另外, 每个SNP数目重复1000次取样, 也能够更好的反映真实情况。籼粳指数是判断水稻品种籼粳特性的重要指标, 而水稻籼粳分化过程中, 不仅仅只会出现籼粳分明的情况, 水稻品种也会出现籼粳中间型, 而且种间杂种优势的利用, 会出现越来越多的中间类型。前人研究将水稻分为籼、偏籼、中间型、偏粳以及粳5个类型。本研究中利用40-SNP对生产上的选育品种进行籼粳指数分析时发现, 生产上的品种籼粳指数比较集中, 基本处在籼粳指数前20%与后20%, 而甬优系列为代表的典型籼粳交品种籼粳指数都处于0.5左右。另外, 在全球6类水稻资源中, 温带粳稻、热带粳稻、aromatic以及rayada类型均属于粳亚种, aus和indica类型则属于籼亚种。所有粳亚种的粳型指数均分布在0.6~1.0之间, 而籼亚种的粳型指数则分布在0~0.4之间, 没有品种粳型指数位于0.4~0.6之间。综合本研究结果, 考虑将水稻品种籼粳特性分为粳稻、籼粳以及中间型, 具体的籼粳指数范围如表4所示。Table 4
表4
表4利用籼粳指数进行籼粳类型鉴定
Table 4
| 籼型指数 Xian index | 粳型指数 Geng index | 籼粳类型鉴定 Xian and geng subspecies identification |
|---|---|---|
| ≥ 0.6 | ≤ 0.4 | 籼稻 Indica |
| 0.4-0.6 | 0.4-0.6 | 中间型 Intermediate type |
| ≤ 0.4 | ≥ 0.6 | 粳稻 Japonica |
新窗口打开|下载CSV
4 结论
本研究利用3000份水稻品种详细信息, 采用SNP-index的方法筛选籼粳特异SNP; 通过一系列条件, 最终筛选到可用于鉴定水稻籼粳特性的40-SNP标记信息。通过40-SNP与4k-SNP计算得出的粳型指数比较, 并结合程氏指数法及已知类型品种对其进行验证, 保证40-SNP的真实性与可靠性。本研究结果为水稻亚种间杂种优势利用, 水稻籼粳分化以及水稻种子管理条例制定等方面提供了数据支撑及理论基础, 具有重要的应用价值与意义。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
