 ,, 李文才, 孙琦, 于彦丽, 赵勐, 鲁守平, 李艳娇, 孟昭东
,, 李文才, 孙琦, 于彦丽, 赵勐, 鲁守平, 李艳娇, 孟昭东 ,*山东省农业科学院玉米研究所/小麦玉米国家工程实验室/农业部黄淮海北部玉米生物学与遗传育种重点实验室, 山东济南 250100
,*山东省农业科学院玉米研究所/小麦玉米国家工程实验室/农业部黄淮海北部玉米生物学与遗传育种重点实验室, 山东济南 250100A study of expression pattern of auxin response factor family genes in maize (Zea mays L.)
LI Wen-Lan ,, LI Wen-Cai, SUN Qi, YU Yan-Li, ZHAO Meng, LU Shou-Ping, LI Yan-Jiao, MENG Zhao-Dong
,, LI Wen-Cai, SUN Qi, YU Yan-Li, ZHAO Meng, LU Shou-Ping, LI Yan-Jiao, MENG Zhao-Dong ,*Maize Research Institute, Shandong Academy of Agricultural Sciences/National Engineering Laboratory of Wheat and Maize/Key Laboratory of Biology and Genetic Improvement of Maize in Northern Yellow-Huai Rivers Plain, Ministry of Agriculture, Jinan 250100, Shandong, China
,*Maize Research Institute, Shandong Academy of Agricultural Sciences/National Engineering Laboratory of Wheat and Maize/Key Laboratory of Biology and Genetic Improvement of Maize in Northern Yellow-Huai Rivers Plain, Ministry of Agriculture, Jinan 250100, Shandong, China通讯作者:
收稿日期:2020-07-7接受日期:2020-11-13网络出版日期:2021-06-12
| 基金资助: |
Received:2020-07-7Accepted:2020-11-13Online:2021-06-12
| Fund supported: |
作者简介 About authors
E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (2120KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李文兰, 李文才, 孙琦, 于彦丽, 赵勐, 鲁守平, 李艳娇, 孟昭东. 玉米生长素响应因子家族基因的表达模式分析[J]. 作物学报, 2021, 47(6): 1138-1148. doi:10.3724/SP.J.1006.2021.03043
LI Wen-Lan, LI Wen-Cai, SUN Qi, YU Yan-Li, ZHAO Meng, LU Shou-Ping, LI Yan-Jiao, MENG Zhao-Dong.
生长素响应因子(auxin response factor, ARF)是植物所特有的一类转录因子[1], 通过特异性地结合生长素响应元件(AuxREs)调节下游靶基因的转录, 参与诸多植物生长发育过程的调控, 例如向性运动、顶端优势、干旱胁迫响应等。研究ARF基因的表达模式对于全面了解玉米ARF基因家族的生物学功能具有重要意义。
生长素信号转导过程涉及早期响应基因(AUX/IAA、GH3和SAUR基因家族等)和与AuxREs相互作用的ARF家族基因[2]。ARF蛋白结合到生长素调控基因启动子区的生长素响应元件(5'-TGTCTC-3')上, 抑制或激活这些基因的转录[3], 从而影响植物的生长发育。拟南芥、水稻、二穗短柄草、鹰嘴豆和丹参等多种植物中的ARF基因已有相关研究[4,5,6,7,8,9]。拟南芥中第一个被发现的ARF转录因子是ARF1, 是以人工合成的生长素响应元件为诱饵, 通过活性很高的酵母单杂系统克隆得到的[3]。ARF1蛋白包括665个氨基酸, N端包含1个DNA结合结构域; 中间的一段区域富含脯氨酸, 丝氨酸和苏氨酸含量也较高[3], 与其他富含脯氨酸结构域的转录抑制子和转录激活子序列类似[4], 该区域可能是转录抑制或转录激活结构域; C端与Aux/IAA基因编码蛋白的结构域III和IV有类似结构[3,10]。拟南芥中有22个基因编码ARF蛋白[11], 其中MP/ARF5参与胚胎发生与维管形成[12]; NPH4/ARF7参与植物向光性和向重力性[13]; ARF19控制叶扩展和侧根生长, 并与NPH4/ARF7表现出功能冗余性[14,15]; ARF6和ARF8参与花成熟调控, 且呈现功能冗余性[16]。此外, 马铃薯ARF6参与顶端休眠, 调控与顶端分生组织连接维管束的发育[17]; 水稻OsARF1受生长素诱导, 参与胚芽鞘的向性运动[18]; 番茄DR12也是ARF家族基因, 参与种子发育、幼苗生长和果实成熟[19]。
玉米有35个ARF基因[20], 基因结构和进化关系等已有相关分析, 但其表达模式还有待深入研究。关于ARF基因在玉米不同组织器官中的表达模式以及在冷、热、盐和渗透胁迫下的表达模式还未见详细报道。Liu等[20]通过全基因组序列分析, 找到了35个玉米ARF基因, 并对基因结构和进化关系等进行了分析。本研究拟通过分析ARF基因在玉米不同组织器官中的表达模式以及在冷、热、盐和渗透胁迫下的表达模式, 探究ARF基因在玉米生长发育和逆境胁迫响应过程中的作用, 为全面解析玉米ARF基因的生物学功能提供有用信息。
1 材料与方法
1.1 试验材料与生长条件
试验所用的玉米材料是B73自交系。分别取材播种后14 d (V3时期)的初级根、第2片叶、整个幼苗植株、播种后25 d (V5时期)的中部茎秆和播种后34 d (V7时期)的整个幼嫩雄花序及雌花序, 用于实时定量PCR分析。逆境胁迫试验所用的材料在光照培养箱中进行长日照培养(16 h光照/8 h黑暗), 生长14 d。冷胁迫处理是将幼苗置于4℃培养箱中进行处理, 热胁迫处理是将幼苗置于40℃培养箱中进行处理, 盐胁迫处理是利用250 mmol L-1的NaCl对幼苗进行处理, 渗透胁迫处理是利用15% PEG-6000对幼苗进行处理。每种胁迫处理均在处理0、1、2、4和8 h后, 收集对照植株和处理植株的第2片叶用于实时定量PCR分析。1.2 RNA提取和实时定量PCR分析
利用天根的多糖多酚总RNA提取试剂盒提取各样品的总RNA, 按照反转录试剂盒说明书完成cDNA的合成和纯化。实时定量PCR分析利用Bio-Rad CFX96实时定量检测系统(Bio-Rad, Hercules, CA, USA), 引物序列见表1, 18S rRNA基因作为内参基因, 根据2-ΔΔCT计算基因相对表达量。对于胁迫处理下玉米幼苗的qRT-PCR分析, 将各处理0 h各基因表达量设为1, 并以此对照计算其在其他时间点的相对表达量。实验数据通过SPSS18.0软件进行差异性分析。Table 1
表1
表1实时定量PCR的引物序列
Table 1
| 基因名 Gene name | 基因号 Gene accession | 引物名称 Primer name | 引物序列 Primer sequence (5°-3°) |
|---|---|---|---|
| ARF1 | Zm00001 | arf1-s | CGTGTATATGTATCCTTCC |
| d030803 | arf1-a | ATTGTCTTCTGAGTACCA | |
| ARF2 | Zm00001 | arf2-s | AAACGACTCTGGGTATGT |
| d031064 | arf2-a | CTGAAGGACTTGTGTCTG | |
| ARF3 | Zm00001 | arf3-s | AACGGCATCTCTAACTAC |
| d031522 | arf3-a | ACGAATCTATGGAATTGAAC | |
| ARF4 | Zm00001 | arf4-s | AGTTCCGATGGCAGTGTT |
| d032683 | arf4-a | CGAGGAACCGATGCAGAT | |
| ARF5 | Zm00001 | arf5-s | CGCAACAACAACAGGCATG |
| d001879 | arf5-a | GGGCTAAAAAGGGACTGGTT | |
| ARF6 | Zm00001 | arf6-s | AATCTCAGCAGCAGTTAA |
| d001945 | arf6-a | CTTGGGACTCTTGGTTTA | |
| ARF7 | Zm00001 | arf7-s | CTAGTGACGCCCTGTACC |
| d003601 | arf7-a | AGTAATAGACACGCTCGC | |
| ARF8 | Zm00001 | arf8-s | GTAGTTGAAGTGGATAATTGTT |
| d041056 | arf8-a | TCTGGAGAAGGCTGATTA | |
| ARF9 | Zm00001 | arf9-s | TCTGAACCTCTGGTATCC |
| d041056 | arf9-a | AACAATTATCCACTTCAACTAC | |
| ARF10 | Zm00001 | arf10-s | TGAACTCGAAATCAGCTGC |
| d042267 | arf10-a | AACTCCAACCTCCACTTGC | |
| ARF11 | Zm00001d | arf11-s | ATGTTACAGGGAATGGGAATG |
| 043431 | arf11-a | CAACAGTTTGAGGAGCAGACG | |
| ARF12 | Zm00001d | arf12-s | ATGGTAGACTTGATAGGA |
| 043922 | arf12-a | CATATTCACAGTTCCAGTA | |
| ARF13 | Zm00001d | arf13-s | CAAGGCAATCACAATCTG |
| 049295 | arf13-a | CTGTCTGTTCATCCCAAA | |
| ARF14 | Zm00001d | arf14-s | AATGACCGTTCTACTCCAATCA |
| 050781 | arf14-a | CTATCTCAATGCCAAACAATCT | |
| ARF15 | Zm00001d | arf15-s | ATGAGGTCTTCGCCAGGAT |
| 051172 | arf15-a | GGACTGTGTCAGCGTCTTG | |
| ARF16 | Zm00001d | arf16-s | AGAACATTGCTGATAGAT |
| 053819 | arf16-a | TTGTGTATGACCTTGAAT | |
| ARF17 | Zm00001d | arf17-s | CCGTATATCCAAGGGTTTTG |
| 014013 | arf17-a | ATGTGGGGTCTCTTTATGTCA | |
| ARF18 | Zm00001d | arf18-s | GCAGCAGATGGGGAAGCA |
| 014377 | arf18-a | AACTCGACCGAACCGACG | |
| ARF19 | Zm00001d | arf19-s | AGAGGACGGCGGCAAGAT |
| 014507 | arf19-a | TGCTCGCCCTCGGGTAGT | |
| ARF20 | Zm00001d | arf20-s | CCACCAATGAAGCAAGAA |
| 015243 | arf20-a | GATAGACAACATCTGACACAT | |
| ARF21 | Zm00001d | arf21-s | AACAGAACAGCATTCAGT |
| 014690 | arf21-a | TGATTCAGTGGAAGAGATG | |
| ARF22 | Zm00001d | arf22-s | GCTTTCCGCCAGCCTCA |
| 016838 | arf22-a | CCGGTGTCACCACCGATG | |
| 基因名 Gene name | 基因号 Gene accession | 引物名称 Primer name | 引物序列 Primer sequence (5°-3°) |
| ARF23 | Zm00001d | arf23-s | CTGAGAGGACGGTGAGCAA |
| 000358 | arf23-a | CGCGACAGCCGAGAGGT | |
| ARF24 | Zm00001d | arf24-s | GCCACTTTCAAGTCAGATT |
| 036593 | arf24-a | TTGGATTGTGCTCTCAGA | |
| ARF25 | Zm00001d | arf25-s | CCCTCTTCTGTTCTTATGTTT |
| 038698 | arf25-a | TACTTCTTCACGGTTGGT | |
| ARF26 | Zm00001d | arf26-s | GCATTCGCCCTCTTCTGTT |
| 038698 | arf26-a | AGGCTCGCTTCCATTTACA | |
| ARF27 | Zm00001d | arf27-s | TTCCATCTCAATCCTCAT |
| 039006 | arf27-a | TCTATCCTCTTTCTTATTCAC | |
| ARF28 | GRMZM2 | arf28-s | TTATGGTTCCAATACAAGAA |
| G075715 | arf28-a | CTCATTCCTATTCCTTAGC | |
| ARF29 | Zm00001d | arf29-s | TTCAAGATCAGGGTTCAG |
| 011953 | arf29-a | GATTCAACCGTCAGAGAA | |
| ARF30 | Zm00001d | arf30-s | CAAGTTCTTCAACATCAG |
| 045026 | arf30-a | ATCCTGTATTATGGTTCAA | |
| ARF31 | Zm00001d | arf31-s | ACTCGCTGGGAAGAGGGCT |
| 023659 | arf31-a | CCTTTTGTCTGCTTCACCAC | |
| ARF32 | Zm00001d | arf32-s | AGCTGGTGCGGGGCAAC |
| 025871 | arf32-a | CCTGCAAGGCCTCAATGAC | |
| ARF33 | Zm00001d | arf33-s | AGTTGAATGCTCTTGGTA |
| 026540 | arf33-a | GTGAATCTGTGCTTCTTG | |
| ARF34 | Zm00001d | arf34-s | ATGCTGGGTTGTTTGGTT |
| 026590 | arf34-a | GCGGCTAGAAAGTGGAAT | |
| ARF35 | Zm00001d | arf35-s | ATGATATTGGAGCAGATG |
| 026687 | arf35-a | AAGAGCATTATGGTGTTC | |
| 18S rRNA | 18S-s | AAACGGCTACCACATCCAAG | |
| 18S-a | CCTCCAATGGATCCTCGTTA |
新窗口打开|下载CSV
1.3 利用转录组数据对ARF基因进行表达分析
利用从MaizeGDB数据库获得的玉米ARF基因的ID号在Genevestigator网站(http://www.genevestigator. com/gv/)搜索ARF基因在不同组织器官中的转录表达数据, 然后利用Genevestigator网站的系统软件对转录组表达数据进行分析, 获得ARF基因在不同组织器官中的表达模式热点分布图[21]。1.4 启动子区顺式作用元件分析
利用Gramene网站(http://ensembl.gramene.org/ Zea_mays/Info/Index)搜索确定玉米ARF家族基因起始密码子上游1500 bp的启动子区序列, 利用PlantCare数据库(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)的顺式作用元件分析工具, 对玉米ARF家族基因启动子区序列进行顺式作用元件分析[22]。2 结果与分析
2.1 玉米ARF家族基因在不同组织器官中的表达分析
为探究生长素响应因子ARF家族成员在玉米中的表达模式, 我们利用实时定量PCR方法对ARF基因在6种不同组织器官(根、茎、叶、幼苗、幼嫩雄花序和幼嫩雌花序)中的表达量进行了检测(图1)。ARF10、ARF16和ARF34在玉米中组成型表达, ARF1在茎、幼苗和雄花序中高表达, ARF28在叶和雌花序中高表达, 而剩余的30个基因均在雄花序和雌花序中优势表达。从整体空间表达上看, 各基因的表达量高低呈现生殖器官>营养器官。玉米花序发育涉及小穗对原基、小穗原基、花器官原基等各类原基发育起始过程, 在这些原基起始部位都存在生长素积累和转运[23]。作为生长素响应因子, ARF基因在雌花序和雄花序中高水平表达, 暗示ARF基因参与花序发育调控过程。图1
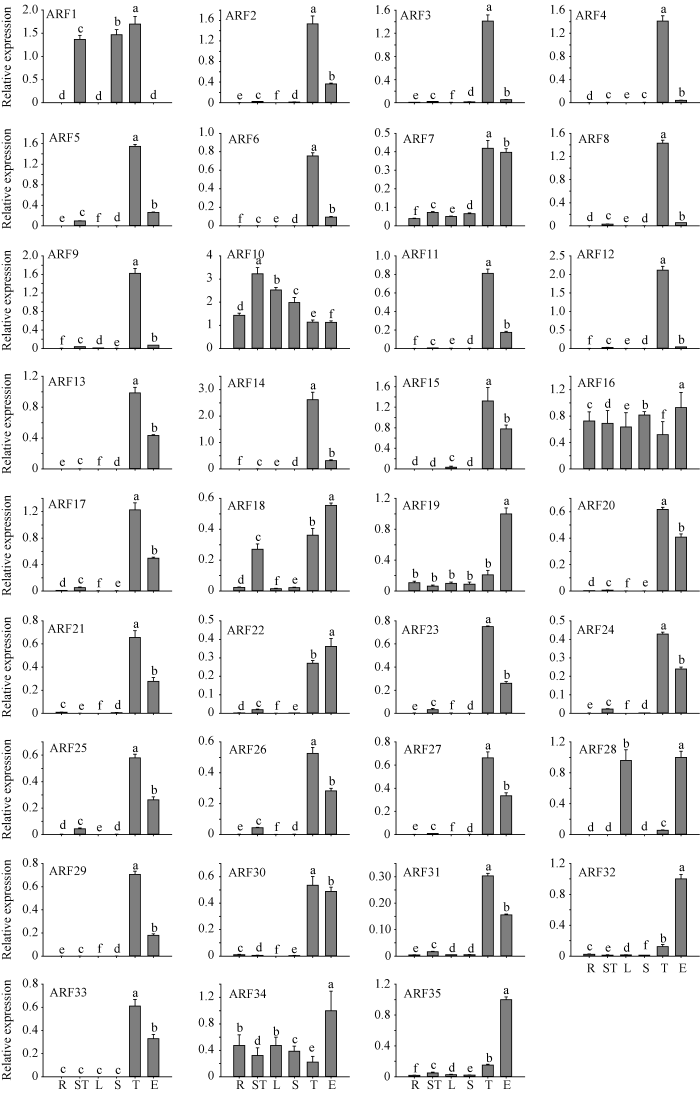 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1玉米ARF家族基因在不同组织器官中的表达水平
R: 根; ST: 茎; L: 叶; S: 幼苗; T: 雄花序; E: 雌花序。图柱上的不同字母代表Duncan’s multiple range tests的显著差异(P < 0.05)。
Fig. 1Expression patterns of ARF genes in different tissues and organs
R: root; ST: stem; L: leaf; S: seedling; T: tassel; E: ear. Values marked with different letters indicate significant differences at P < 0.05 by Duncan’s multiple range tests.
2.2 转录组数据库中玉米ARF家族基因在不同组织器官中的表达分析
为进一步了解玉米ARF家族基因的表达模式, 我们利用Genevestigator数据库中的转录组数据对ARF家族基因在更多不同组织器官(41个组织器官)中的表达模式进行了分析, 共获得31个玉米ARF基因(未获得ARF9、ARF19、ARF26和ARF28这4个基因的表达信息)的表达信息, 结果如图2所示。除了花粉, ARF10、ARF16和ARF34在所检测的其余组织器官中均有表达, 这与实时定量PCR结果基本吻合。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2利用转录组数据获得的玉米ARF家族基因在不同组织器官中的表达水平
图中每个基因在所有检测组织器官中的表达量设为100%, 颜色的深浅代表表达百分率。
Fig. 2Expression profile of ARF genes in different tissues of maize by Genevestigator analysis
The relative expression of each gene in all detected tissues and organs was set at 100%, and the shade of color represents the percentage of relative expression levels.
ARF1和ARF2在花药中优势表达, 这与定量结果显示其在雄花序中优势表达是一致的。此外, 除了ARF1和ARF2, 其余29个玉米ARF基因在雌花序发育(pistil, ovary, ear, spikelet)和种子发育过程(embryo, endosperm, embryo sac, embryo-surrounding region, basal endosperm transfer layer)中均有较高的表达量。这与实时定量PCR结果显示ARF基因在生殖器官发育过程中优势表达也是一致的。然而, 与定量结果显示ARF基因在茎中低表达(除了ARF1和ARF10)相反, 有29个ARF基因(除ARF2和ARF3)在茎中具有较高的表达水平。这可能是由于取材时期和试验材料不同所造成的结果差异。结合转录组数据和实时定量PCR结果, 我们可以推测ARF基因在玉米生长发育和生殖器官发育过程中均发挥着重要的作用。
2.3 玉米ARF家族基因的顺式作用元件分析
为更好了解玉米ARF家族基因在逆境胁迫响应过程中的作用, 我们对玉米ARF家族基因起始密码子上游1500 bp的启动子区进行顺式作用元件分析(表2), 除转录和光响应相关必需顺式作用元件, 在玉米ARF家族基因的启动子区找到大量顺式作用元件, 根据功能可分为: 植物激素响应相关顺式作用元件(Element A)、生长发育相关顺式作用元件(Element B)和逆境胁迫相关顺式作用元件(Element C)。Element A中的脱落酸响应元件(ABRE)分布于除ARF15和ARF19之外的每一个ARF基因启动子区, 茉莉酸甲酯响应元件(CGTCA-motif, TGACG-motif, MeJA-responsive element)分布在除ARF14、ARF19、ARF27、ARF34和ARF35之外的30个ARF基因启动子区, 赤霉素响应元件P-box (gibberellin-responsive element)和GARE-motif (gibberellin-responsive element)分别位于ARF1、ARF2、ARF3、ARF4、ARF13、ARF18、ARF19、ARF24、ARF25、ARF26和ARF3、ARF5、ARF8、ARF9、ARF22、ARF24、ARF27、ARF28、ARF33、ARF34的启动子区, 生长素响应元件AuxRR-core和TGA-element分别位于ARF1、ARF16、ARF23、ARF29和ARF1、ARF5、ARF6、ARF7、ARF11、ARF14、ARF15、ARF17、ARF20、ARF23、ARF33和ARF34的启动子区。Xing等[24]分析了31个ARF基因启动子区的生长素响应元件, 与本文结果基本吻合。Element B中的分生组织相关响应元件(CAT-box)位于ARF2、ARF3、ARF5、ARF6、ARF7、ARF11、ARF12、ARF16、ARF19、ARF22、ARF23、ARF25、ARF26、ARF28、ARF29、ARF32、ARF33和ARF34的启动子区, 种子特异调控响应元件(RY-element)分布在ARF6、ARF17、ARF27、ARF32和ARF33的启动子区, 生物钟相关响应元件(circadian)只分布在ARF30和ARF31的启动子区, 胚乳表达相关元件(GCN4_motif)只分布在ARF4和ARF32的启动子区。Element C中的低温响应元件(low temperature- responsive element, LTR)位于ARF3、ARF4、ARF5、ARF7、ARF8、ARF9、ARF15、ARF19、ARF20、ARF23、ARF25、ARF26、ARF29、ARF31和ARF34的启动子区, 干旱胁迫响应元件(MYB binding site involved in drought-inducibility, MBS)位于ARF1、ARF4、ARF6、ARF7、ARF8、ARF9、ARF12、ARF15、ARF17、ARF18、ARF19、ARF21、ARF23、ARF24、ARF28、ARF30和ARF35的启动子区, 防御胁迫响应元件(TC-rich repeats)位于ARF8、ARF9、ARF13、ARF25、ARF26和ARF30的启动子区, 水杨酸响应元件(TCA-element)位于ARF3、ARF5、ARF11、ARF14、ARF17、ARF19、ARF20、ARF23、ARF24、ARF28和ARF32的启动子区。其中, 植物激素响应相关顺式作用元件最多, 每个ARF基因都含有2~15个; 生长发育相关顺式作用元件分布在23个ARF基因的启动子区, 主要包括RY-element、GCN4-motif和circadian等; 逆境胁迫相关顺式作用元件主要分布在29个ARF基因的启动子区, 主要包括MBS、LTR和TC-rich repeats等。每个ARF基因的启动子区都包含多个顺式作用元件, 表明玉米ARF基因在植物激素响应、生长发育和逆境胁迫响应等方面均发挥重要作用。Table 2
表2
表2玉米ARF家族基因启动子区的顺式作用元件
Table 2
| 基因 Gene | 元件A Element A | 元件B Element B | 元件C Element C |
|---|---|---|---|
| ARF1 | ABRE1, AuxRR-core1, CGTCA-motif2, P-box2, TGA-element2, TGACG-motif2 | MBS1 | |
| ARF2 | ABRE3, CGTCA-motif3, P-box1, TGACG-motif3 | CAT-box2 | |
| ARF3 | ABRE3, CGTCA-motif4, GARE-motif1, P-box1, TGACG-motif4 | CAT-box2 | TCA-element2, LTR1 |
| ARF4 | ABRE1, CGTCA-motif1, P-box1, TGACG-motif1 | GCN4_motif1 | LTR1, MBS1 |
| ARF5 | ABRE1, CGTCA-motif1, GARE-motif1, TGA-element1, TGACG-motif1 | CAT-box3 | TCA-element1, LTR2 |
| ARF6 | ABRE2, CGTCA-motif1, TGA-element1, TGACG-motif1 | CAT-box2, RY-element1 | MBS1 |
| ARF7 | ABRE2, CGTCA-motif1, TGA-element1, TGACG-motif1 | CAT-box1 | LTR3, MBS1 |
| ARF8 | ABRE2, CGTCA-motif3, GARE-motif1, TGACG-motif3 | LTR1, MBS1, TC-rich repeats1 | |
| ARF9 | ABRE2, CGTCA-motif3, GARE-motif1, TGACG-motif3 | LTR1, MBS1, TC-rich repeats1 | |
| ARF10 | ABRE1, CGTCA-motif5, TGACG-motif5 | ||
| ARF11 | ABRE2, CGTCA-motif2, TGA-element1, TGACG-motif2 | CAT-box2 | TCA-element1 |
| ARF12 | ABRE1, CGTCA-motif1, TGACG-motif1 | CAT-box1 | MBS2 |
| ARF13 | ABRE2, CGTCA-motif2, P-box1, TGACG-motif2 | TC-rich repeats1 | |
| ARF14 | ABRE2, TGA-element1 | TCA-element2 | |
| ARF15 | CGTCA-motif3, TGA-element1, TGACG-motif3 | LTR1, MBS1 | |
| ARF16 | ABRE1, AuxRR-core1, CGTCA-motif2, TGACG-motif2 | CAT-box3 | |
| ARF17 | ABRE1, CGTCA-motif4, TGA-element1, TGACG-motif4 | RY-element1 | MBS1, TCA-element1 |
| ARF18 | ABRE2, CGTCA-motif1, P-box1, TGACG-motif1 | MBS1 | |
| ARF19 | P-box1 | CAT-box1 | LTR1, MBS1, TCA-element1 |
| ARF20 | ABRE3, CGTCA-motif1, TGA-element1, TGACG-motif1 | LTR1, TCA-element1 | |
| ARF21 | ABRE7, CGTCA-motif2, TGACG-motif2 | MBS1 | |
| ARF22 | ABRE6, CGTCA-motif2, GARE-motif1, TGACG-motif2 | CAT-box1 | |
| ARF23 | ABRE4, AuxRR-core1, CGTCA-motif1, TGA-element2, TGACG-motif1 | CAT-box1 | LTR1, MBS2, TCA-element1 |
| ARF24 | ABRE3, CGTCA-motif2, GARE-motif1, P-box2, TGACG-motif2 | MBS1, TCA-element1 | |
| ARF25 | ABRE1, CGTCA-motif1, P-box1, TGACG-motif1 | CAT-box3 | LTR1, TC-rich repeats1 |
| ARF26 | ABRE1, CGTCA-motif1, P-box1, TGACG-motif1 | CAT-box3 | LTR1, TC-rich repeats1 |
| ARF27 | ABRE4, GARE-motif1 | RY-element1 | |
| ARF28 | ABRE1, CGTCA-motif2, GARE-motif1, TGACG-motif2 | CAT-box2 | MBS2, TCA-element2 |
| ARF29 | ABRE3, AuxRR-core1, CGTCA-motif1, TGACG-motif1 | CAT-box1 | LTR1 |
| ARF30 | ABRE6, CGTCA-motif4, TGACG-motif4 | circadian1 | MBS2, TC-rich repeats1 |
| ARF31 | ABRE1, CGTCA-motif2, TGACG-motif2 | circadian1 | LTR2 |
| ARF32 | ABRE5, CGTCA-motif1, TGACG-motif1 | CAT-box1, GCN4_motif1, RY-element3 | TCA-element1 |
| ARF33 | ABRE4, CGTCA-motif3, GARE-motif1, TGA-element1, TGACG-motif3 | CAT-box4, RY-element1 | |
| ARF34 | ABRE1, GARE-motif1, TGA-element1 | CAT-box2 | LTR1 |
| ARF35 | ABRE3 | MBS1 |
新窗口打开|下载CSV
2.4 玉米ARF家族基因在冷、热、盐和渗透胁迫条件下的表达分析
顺式作用元件分析显示有29个玉米ARF基因启动子区包含逆境胁迫相关顺式作用元件。为进一步研究玉米ARF家族基因在逆境胁迫响应过程中的作用, 我们选取了9个在叶片中表达量较高的ARF基因(ARF7、ARF10、ARF15、ARF16、ARF18、ARF19、ARF32、ARF34和ARF35), 利用实时定量荧光PCR方法检测玉米ARF家族基因在冷、热、盐及渗透胁迫下的表达量变化(图3)。在冷胁迫处理下, 所有被检测ARF基因的表达模式都是类似的, 在处理1 h时表达量迅速上升, 随后降到较低水平, 处理4 h后又开始上升。在热胁迫处理下, ARF16基因1 h时表达量达到峰值, 是未处理时的25倍, 随后又逐渐下降, 而其余8个ARF基因的表达量则是随着时间逐渐上升。在盐胁迫处理下, 多数ARF基因的表达量变化不大(除了ARF10在处理4 h时表达量达到峰值随后又开始下降外), 比较有趣的一点是, ARF35的表达量在处理后迅速下降并维持在较低水平, 暗示该基因可能是通过与其他ARF基因相反的途径参与盐胁迫调控过程。在渗透胁迫处理下, ARF15、ARF18和ARF19的表达量是先上升后下降, 在处理8 h时表达量达到峰值; ARF10、ARF32、ARF34和ARF35的表达模式是相似的, 处理1 h时表达量达到峰值, 随后下降并维持在较低水平; ARF7和ARF16的表达量变化不大。这些结果表明, ARF基因涉及参与多种逆境胁迫响应过程。图3
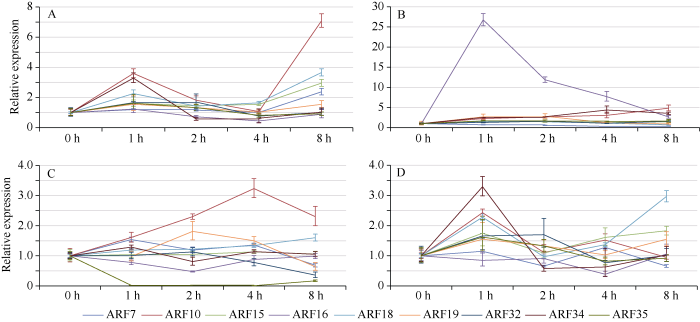 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3玉米ARF家族基因在不同逆境胁迫下的表达水平
A: 冷胁迫处理; B: 热胁迫处理; C: NaCl胁迫处理; D: 渗透胁迫处理。
Fig. 3Transcriptional patterns of ARF genes under different stress treatments
A: cold stress treatment; B: hot stress treatment; C: NaCl stress treatment; D: osmotic stress treatment.
3 讨论
作为重要的植物激素, 生长素几乎参与了植物生长发育的每个阶段。而ARF基因作为生长素响应因子, 通过特异性地结合生长素响应元件调节下游靶基因的转录, 参与诸多植物生长发育过程[2]。根据标记基因表达情况可将ARF家族分为3类: 一类(Clade A)具有转录激活功能, 一类(Clade B)具有转录抑制功能, 一类(Clade C)既不具有转录激活功能也不具有转录抑制功能[25]。这3类ARF基因的功能分类与进化关系是一致的[26]。结合不同数据库分析得到的转录组数据和实时定量PCR结果可以看出, Clade C主要在幼嫩雄花序中优势表达, 而Clade A和Clade B则主要在幼嫩花序中高水平表达, 有的在幼嫩雌花序中优势表达, 有的在雄花序中优势表达。不管ARF基因通过转录激活(Clade A)还是转录抑制(Clade B), 又或者通过别的调控途径(Clade C), 都会通过参与调控生长素相关基因影响玉米花序发育过程。ARF基因参与了诸多生物学过程。拟南芥中的arf2突变体花期、叶衰亡和花脱落时间推迟[27], 推测与之同源的玉米ARF10和ARF29也参与花发育和叶片发育, 而这2个基因都在幼嫩花序中优势表达, 也从侧面印证这一推测; AtARF5参与胚模式建成和维管组织形成[12,28-29], 而作为同源基因的玉米ARF6在幼嫩花序和中柱中高表达, ARF33在幼嫩花序和果皮中高表达, 这也暗示这2个基因可能参与胚发育和微管形成; AtARF6调控花成熟[16,30], 同源基因玉米ARF16和ARF18在幼嫩花序和胚中高表达, 可能参与花和果穗形成过程; AtARF8是果实起始发育的负调控因子[16,30], 黄瓜AtARF8的同源基因Csa019264和Csa019265在单性结实和非单性结实品种的表达差异大[31], 而玉米AtARF8的同源基因ARF5和ARF34在幼嫩花序和胚中高表达, 推测参与果穗的发育和形态建成。然而玉米ARF基因众多, 通过DAP-seq测序技术获得的顺式作用元件分析结果显示, ARF基因在Clade A、Clade B和Clade C组间的结合位点特异性和靶基因等差异不大[32], 暗示基因功能冗余现象严重, 这也是至今还未发现有表型的玉米arf突变体的原因[25], 开展功能研究还需新的思路和方法。
随着玉米基因组测序完成, 高通量测序技术被广泛应用于基因功能研究。通过分析数据库收录的测序数据, 可以获得不同部位组织器官在不同发育时期、不同环境条件下的表达模式。Liu等[20]通过EST数据库预测ARF基因主要在雄穗和果穗中表达, Gallavotti等[26]通过数据库数据分析ARF基因在胚、幼嫩花序和成熟花序中优势表达, 而本文结合实时定量PCR方法和现有数据库更新的转录组数据分析了玉米ARF基因在不同组织器官的表达模式, 发现ARF基因主要在雄花序和雌花序中优势表达, 充分说明ARF基因在花序发育过程中发挥重要功能。
ARF基因在植物的生长发育过程中发挥了重要作用, 也参与了许多逆境胁迫响应过程。拟南芥ARF5参与维管形成, 而干旱条件下植株的维管合成能力加强, 暗示ARF5通过参与维管合成而影响植株的抗旱性[6]; ARF10和ARF16通过控制根冠的发育来影响植株的抗旱性[33]; 大豆ARF基因的表达量在水分胁迫下发生明显变化, 暗示ARF基因参与了大豆水分胁迫响应[34]。玉米中的ARF基因在冷、热、盐和渗透胁迫处理下, 表达量均发生了明显变化, 这暗示ARF基因参与了玉米多种胁迫响应过程。
4 结论
玉米ARF基因家族的ARF10、ARF16和ARF34呈组成型表达, 其余32个ARF基因均在幼嫩雄花序和雌花序中高表达, 参与了玉米花序发育过程; 玉米ARF基因家族在冷、热、盐和渗透胁迫处理下表达量发生明显变化, 暗示玉米ARF基因家族参与了多种逆境胁迫响应过程。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 4]
[本文引用: 2]
[本文引用: 1]
URLPMID:28883480 [本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
URLPMID:15960621 [本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:10318972 [本文引用: 2]
[本文引用: 2]
URLPMID:29145642 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:17021043 [本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1038/s41467-018-06977-6URLPMID:30375394 [本文引用: 1]

AUXIN RESPONSE FACTORS (ARFs) are plant-specific transcription factors (TFs) that couple perception of the hormone auxin to gene expression programs essential to all land plants. As with many large TF families, a key question is whether individual members determine developmental specificity by binding distinct target genes. We use DAP-seq to generate genome-wide in vitro TF:DNA interaction maps for fourteen maize ARFs from the evolutionarily conserved A and B clades. Comparative analysis reveal a high degree of binding site overlap for ARFs of the same clade, but largely distinct clade A and B binding. Many sites are however co-occupied by ARFs from both clades, suggesting transcriptional coordination for many genes. Among these, we investigate known QTLs and use machine learning to predict the impact of cis-regulatory variation. Overall, large-scale comparative analysis of ARF binding suggests that auxin response specificity may be determined by factors other than individual ARF binding site selection.
[本文引用: 1]
[本文引用: 1]
URLPMID:23810914 [本文引用: 1]
