 ,, 方启迪, 丁元昊, 池华斌, 张献龙, 闵玲
,, 方启迪, 丁元昊, 池华斌, 张献龙, 闵玲 ,*华中农业大学植物科学技术学院/作物遗传改良国家重点实验室, 湖北武汉 430070
,*华中农业大学植物科学技术学院/作物遗传改良国家重点实验室, 湖北武汉 430070GhMADS7 positively regulates petal development in cotton
MA Huan-Huan ,, FANG Qi-Di, DING Yuan-Hao, CHI Hua-Bin, ZHANG Xian-Long, MIN Ling
,, FANG Qi-Di, DING Yuan-Hao, CHI Hua-Bin, ZHANG Xian-Long, MIN Ling ,*National Key Laboratory of Crop Genetic Improvement / College of Plant Science and Technology, Huazhong Agricultural University, Wuhan 430070, Hubei, China
,*National Key Laboratory of Crop Genetic Improvement / College of Plant Science and Technology, Huazhong Agricultural University, Wuhan 430070, Hubei, China通讯作者:
收稿日期:2020-06-28接受日期:2020-11-13网络出版日期:2021-05-12
| 基金资助: |
Received:2020-06-28Accepted:2020-11-13Online:2021-05-12
| Fund supported: |
作者简介 About authors
E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (13027KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
马欢欢, 方启迪, 丁元昊, 池华斌, 张献龙, 闵玲. 棉花GhMADS7基因正调控棉花花瓣发育[J]. 作物学报, 2021, 47(5): 814-826. doi:10.3724/SP.J.1006.2021.04140
MA Huan-Huan, FANG Qi-Di, DING Yuan-Hao, CHI Hua-Bin, ZHANG Xian-Long, MIN Ling.
在1991年, Coen和Meyerowitz[1]根据拟南芥的花发育机制提出了经典的ABC模型, 后又衍生到ABCDE模型[2]。在ABC模型中, Coen将拟南芥的花器官从外至内分成萼片(Sepal)、花瓣(Petal)、雄蕊(Stamen)和心皮(Carpal) 4轮, 其中A类基因单独调控第1轮(萼片)的发育; A+B共同参与第2轮(花瓣)的发育; 第3轮(雄蕊)的发育由B+C共同调控; 而C类基因单独参与第4轮(心皮)的发育。而A类基因与C类基因具有相互拮抗的作用[3]。当突变AP1基因时, C类基因没有出现异位表达, 但当AP2基因突变或缺失时, C类基因功能便会从第3、4轮延伸到第1、2轮花器官, 因此说明A、C类基因相互拮抗主要是通过AP2来实现的[3]。相较于经典的ABC模型, 四因子模型似乎为更多研究者所认同。四因子模型就是将花器官发育的4轮分别用4个基因编码的蛋白所组成的四聚体表示, 第1轮的萼片发育由2个SEP蛋白和2个AP1蛋白所组成的四聚体表示; 花瓣则由1个AP1蛋白和1个SEP蛋白以及2个B类基因PI和AP3的蛋白来表示; 第3轮的雄蕊由AP3、PI、AG、SEP 4种蛋白的四聚体来表示; 心皮则由2个AG蛋白和2个SEP蛋白来表示。SEP蛋白是ABCDE模型中涉及所有花器官发育的E类基因的蛋白, 而其他蛋白则是由每一轮所对应的调控基因的蛋白组成。虽然四因子蛋白模型能更适用于所有物种, 但经典的ABC模型仍然是人们研究植物花器官发育的重要参考依据。
MADS-box基因是在植物花器官发育和信号传导中起着重要作用的一类转录因子, 其广泛存在于动植物以及真菌中。依据种类划分, 研究者将MADS-box基因分成I型和II型两大类[3]。其中MADS-box I型基因就是ARG80/SRF类群, 这类基因主要存在于动物和真菌中, 植物中也有少量此类群基因; 另外MADS-box II型基因主要是植物中的MIKC和动物、酵母中的MEF2。也就是说植物中的MADS-box基因绝大部分是属于MADS-box II型基因类群[4]。I型MADS-box通常只含有一个高度保守的MADS (M)结构域而没有其他结构, 并且I型一般只有1~2个外显子来编码MADS结构域[5], 所以I型MADS-box基因又称为M型MADS。I型基因又可进一步分为Mα、Mβ、Mγ、Mδ这4个亚族。相较于I型MADS-box基因, II型基因一般含有7个外显子和6个内含子[6], 编码较复杂的结构域。除了有典型的结构域之外, 还有Intervening (I)区、Keratin-like (K)区和C-terminal (C)区, 因此II型MADS-box基因也称为MIKC型MADS-box基因[7]。而根据I区的序列的不同, 植物中的II型基因又可分为MIKC*和MIKCC 2种类型[8]。
AG (AGAMOUS)基因是MIKCC型MADS-box基因[8], AG基因主要参与调控雄蕊和雌蕊的发育、控制花分生组织终止、调控胚珠发育和抑制A类基因[2,9-11]。在樱花中, AG基因功能的丧失会形成重瓣的花型, 推测极有可能影响了AG基因的原位表达[12]。梅花的AG突变体则会引起花瓣颜色的改变而不改变花型[13]。OsMADS3是水稻AG基因的同源基因, 由于其异位表达导致浆片被同源转化成雄蕊[14]; 番茄AG基因TAG1, 其反义RNA的转基因植物出现雄蕊变成花瓣的现象, 有义RNA的转基因植物中观察到互补表型, 即第1轮萼片被转化为成熟的果皮叶, 而雄蕊代替了第2轮花瓣[15]。因此, 在植物中C类的AG基因通常参与雄蕊和心皮的发育, 但也会影响花器官形态和果实发育。
棉花作为我国重要的经济作物, 除了作为重要的天然纺织材料外, 也是我国国民经济的重要组成部分。在棉花生长过程中, 棉花花器官的发育直接影响着棉花的产量和纤维品质。因此, 本研究选取MADS-box基因家族中对花器官的发育具有调控作用的AG亚族基因进行探究。首先通过同源序列比对获得了棉花中与拟南芥AG具有较高同源性的GhMADS7基因。通过棉花遗传转化, 获得了GhMADS7的转基因干涉材料, 在干涉材料中棉花花瓣发育受阻。组织切片发现, GhMADS7干涉材料花瓣维管束皱缩, 表达分析表明, GhMADS7干涉材料中调控花器官发育的A和B类基因的表达上调, 而参与花瓣发育的基因多属于A和B类基因, 因此我们推测GhMADS7可能通过抑制A和B类基因正调控棉花花瓣发育。
1 材料与方法
1.1 材料
转基因受体材料为陆地棉(Gossypium hirsutum L.) ‘YZ1’, 由华中农业大学作物遗传改良国家重点实验室棉花组提供。1.2 载体和菌株
TA克隆载体为pGEM-Teasy (Promega公司), pDONER221为BP反应入门载体, 购于Invitrogen公司, 用于GhMADS7遗传转化的RNA干涉载体基于Hellsgate4载体构建。用于载体转化的大肠杆菌为TOP10, 根瘤农杆菌为GV3101。1.3 主要酶类和试剂
RNA提取试剂盒购于Sigma公司, BP重组酶购于Invitrogen, 限制性内切酶购于NEB公司, DNA抽提、质粒提取以及PCR纯化试剂盒购于北京天根生物科技有限公司, 反转录酶M-MLV Reverse Transcriptase购于Promega公司, qRT-PCR试剂购于ABI公司。载体构建过程中所用的抗生素有氨苄青霉素、庆大霉素、卡那霉素、利福平等, 均购自Sigma公司。1.4 MADS-box基因分析
本研究中陆地棉基因序列来源于南京农业大学的数据库(1.5 GhMADS7基因的获得及序列分析
将拟南芥的AtAG蛋白序列置于由南京农业大学提交的陆地棉TM-1数据库(1.6 GhMADS7基因RNAi载体构建与遗传转化
通过BP重组反应构建GhMADS7基因RNAi抑制表达载体[16]。设计GhMADS7基因含有aatB接头的引物, 以‘YZ1’花药的cDNA为模板进行扩增, 反应体系包含cDNA模板2 μL、10× Easy Taq buffer 2 μL、dNTP 0.3 μL、Easy Taq 0.2 μL、正反向引物各0.25 μL、ddH2O 15 μL。TA克隆反应体系包含2× Rapid Ligation buffer 2.5 μL、T4 DNA Ligase 0.5 μL、pGEM-TEasy 1 μL、PCR产物1 μL。获得克隆后送华大基因测序验证。以阳性克隆的质粒为模板, 用带aatB接头的引物, 在高保真酶的作用下再次进行扩增, 进行BP反应4℃过夜, 反应体系包含BP Clonase TMII 酶混合物0.5 μL、PCR产物2 μL、pDONER221质粒1 μL、TE buffer 1.5 μL。反应产物转化TOP10感受态, 挑单克隆检测阳性, 对阳性单克隆提取质粒, 分别用Xhol I和Xbal I两种限制性核酸内切酶进行酶切检测, 酶切检测体系包含Cutsmart buffer 2 μL、Xhol I 0.5μL、Xbal I 0.5 μL、重组质粒12 μL、ddH2O 5 μL。将酶切检测正确的质粒转化GV3101保存备用。棉花遗传转化体系以陆地棉‘YZ1’下胚轴为转化受体, 遗传转化及植物再生过程具体操作参照文献[17]。1.7 转基因材料的分子鉴定
1.7.1 转基因植株阳性鉴定 取适量的植株幼嫩叶片, 利用改良的CTAB法抽提棉花总DNA[17], 将得到的DNA稀释成100 ng µL-1后作为模板, 以NPTII引物进行PCR扩增, PCR产物进行凝胶电泳, 根据有无条带及条带大小判断植株是否为转基因阳性植株。1.7.2 转基因植株拷贝数鉴定 取适量转基因阳性植株的幼嫩叶片, 采用植物基因组DNA抽提试剂盒(TIANGEN, China)提取基因组DNA。利用Southern杂交检测转基因植株拷贝数, 具体操作步骤见文献[17]。
1.8 RNA提取及qRT-PCR分析
通过游标卡尺量取‘YZ1’花蕾长度, 将其划分为5个发育时期, 即F<9 mm、F9~14 mm、F14~19 mm、F>19 mm和F0 (开花当天的花蕾), 将同一发育时期的花瓣、花药、柱头和胚珠进行组织分离, 立即放入液氮中, 保存于-80℃, 用于RNA提取及基因组织表达分析。分别取适量分离出的YZ1和转基因阳性植株的早期花蕾用于转基因植株基因表达量及A和B类基因表达量检测。所获得的样品采用改良的异硫氰酸胍法提取总RNA[18], 微量分光光度计(NanoDrop2000, USA)检测RNA浓度, 后电泳检测RNA质量。每份材料使用3 µg RNA进行反转录, 即取3 µg RNA于0.5 mL无RNA酶离心管中, 加入1 µL oligo dT, 用DEPC水补充体积至15 µL, 并简单离心。将上述混合体系置PCR仪中, 设置程序为70℃ 10 min, 之后冰上冷却10 min。向混合物中加入: 5 µL 5×MLV buffer、1 µL RNasin、1 µL M-MLV RTase、1.25 µL 10 mmol L-1 dNTP和1.75 µL DEPC水。充分混合后, 于PCR仪中42℃反应60 min, 然后70℃反应7 min, 反应结束后将产物放置于20℃保存。
反转的cDNA样品稀释100倍作为表达量检测的模板。以持家基因GhUBQ7 (Gh_A11G011460)为内参, 通过qRT-PCR检测目的基因的表达量, 反应体系包含7 µL稀释的cDNA模板、7 µL SYBR Green Master Mix Reagent (Bio-Rad公司)、正反引物各0.5 µL。qRT-PCR所用仪器为ABI-Prism 7500 (Applied Biosystems, USA), 反应程序为95℃ 60 s; 95℃ 15 s, 60℃ 40 s, 40个循环。
1.9 棉花花器官石蜡切片
通过对田间材料的表型考察发现, 在常温条件下, 转基因阳性植株花蕾在5~6 mm和7~8 mm长度左右出现表型。对出现表型之前的各个时期的花蕾按1 mm长度为单位进行划分, 后经固定、脱水、透明、浸蜡、包埋、修块、切片、脱蜡、染色、封片等步骤后可置于显微镜(Zeiss HAL 100)下观察, 其具体操作过程参照文献[18,19]。Table 1
表1
表1本试验所用引物及用途
Table 1
| 引物名称 Primer name | 引物序列 Primer sequence (5°-3°) | 用途 Purpose |
|---|---|---|
| ubiquitin7-F | GAAGGCATTCCACCTGACCAAC | 实时荧光定量PCR |
| ubiquitin7-R | CTTGACCTTCTTCTTCTTGTGCTTG | qRT-PCR |
| GhAMDS7-S | TCATAGTTCAATTCCTTGCCAGC | 基因扩增 |
| GhAMDS7-A | CTTGAATAGATGGAGGTGGAAGAGAG | Gene amplification |
| OGhMADS7-F | CCTTGCCAGCTTTGAACCAT | 基因扩增 |
| OGhMADS7-R | ATGGAGGTGGAAGAGAGAAGCTAC | Gene amplification |
| qGhMADS7-F | AATGATTACTCCAACCAAGACCAA | 实时荧光定量PCR |
| qGhMADS7-R | TTGGTAGAACATTAGAGGCTGCTG | qRT-PCR |
| bpGhMADS7-F | GGGGACAAGTTTGTACAAAAAAGCAGGCTGGCCTTGCCAGCTTTGAACCAT | BP反应 |
| bpGhMADS7-R | GGGGACCACTTTGTACAAGAAAGCTGGGTCATGGAGGTGGAAGAGAGAAGCTAC | BP reaction |
| NPTII-F | TTGTCACTGAAGCGGGAAGG | 探针 |
| NPTII-R | CGATACCGTAAAGCACGAGGAA | Probe |
新窗口打开|下载CSV
2 结果与分析
2.1 MIKCC型MADS-box基因家族进化树分析
利用拟南芥中的39条MIKCC型MADS-box基因序列, 于棉花数据库中进行Blast, 获得76条棉花的同源序列。后与单子叶模式植物水稻和双子叶模式植物拟南芥的蛋白序列共同利用MEGA6.1软件对其进行Construct/Test Neighbor-Joining Tree分析, 其中红色字体为陆地棉中MIKCC型MADS-box基因。MIKCC型基因可分为SPE、AGL6、AP1/FUL、AGL12、AG、AGL15、AGL17、SVP、PI/AP3、BS、TM3、SOC和FLC共13个亚族[20]。本研究中共在陆地棉中获得11个亚族, 不包含FLC和TM3 2个亚族(图1-A)。本研究重点关注AG亚族, 其是一类参与花器官生长发育的转录因子(紫色区域标识), 在陆地棉中共筛选出10个MIKCC型MADS-box的AG亚族基因, 分别是GhMADS2、GhMADS3、GhMADS4、GhMADS5、GhMADS7、GhMADS10、GhMADS76、GhMADS98、GhMADS99、GhMADS100 (图1-A)。对陆地棉AG亚族与拟南芥AtAG基因单独绘画进化树发现, GhMADS7和GhMADS98与拟南芥中的AtAG基因亲缘关系较近(图1-B), 故选取GhMADS7和GhMADS98进行后续分析。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1陆地棉MIKCC型MADS-box基因家族系统进化树
A: 陆地棉MIKCC型MADS-box基因家族进化树; B: 陆地棉MIKCC型MADS-box基因家族AG亚族进化树。
Fig. 1Phylogenetic tree analysis of MIKCC MADS-box gene family in Gossypium hirsutum
A: phylogenetic tree of MIKCC MADS-box gene family in Gossypium hirsutum; B: phylogenetic tree of AG subfamily of MIKCC MADS-box gene family in Gossypium hirsutum.
2.2 GhMADS7和GhMADS98基因序列及表达模式分析
通过对GhMADS7和GhMADS98的CDS序列分析发现, GhMADS7/98的CDS序列全长均包含705个碱基, 编码234个氨基酸的蛋白。将GhMADS7和GhMADS98蛋白序列于DNAMAN软件中进行序列比对分析发现, GhMADS7和GhMADS98基因的蛋白序列具有99%的相似度(图2-A), 后将GhMADS7和GhMADS98于NCBI网站中进行BLASP以对其保守结构域进行分析发现, GhMADS7和GhMADS98具有MADS-box基因家族代表性的MADS结构域以及区分I型和II型MADS-box基因的K-box结构域(图2-A, B), 说明GhMADS7和GhMADS98确为II型MADS-box基因成员。本研究在陆地棉各组织表达谱数据库[21]中获得的GhMADS7和GhMADS98的表达模式信息表明, 同属于AG亚族的GhMADS7和GhMADS98两者有着完全一样的表达模式, 即在陆地棉的柱头、胚珠和10 DPA种子中相对表达量较高, 其中在胚珠中表达最高(图2-C)。另外, 这2个基因的ID号显示GhMADS7 (Gh_D04G0341)和GhMADS98 (Gh_A05G3267)分别位于陆地棉的A和D亚基因组。结合蛋白序列、进化分析和表达分析结果表明, GhMADS7和GhMADS98极有可能为陆地棉中位于A和D亚基因组的同源基因, 因此我们选取了GhMADS7进行进一步试验。图2
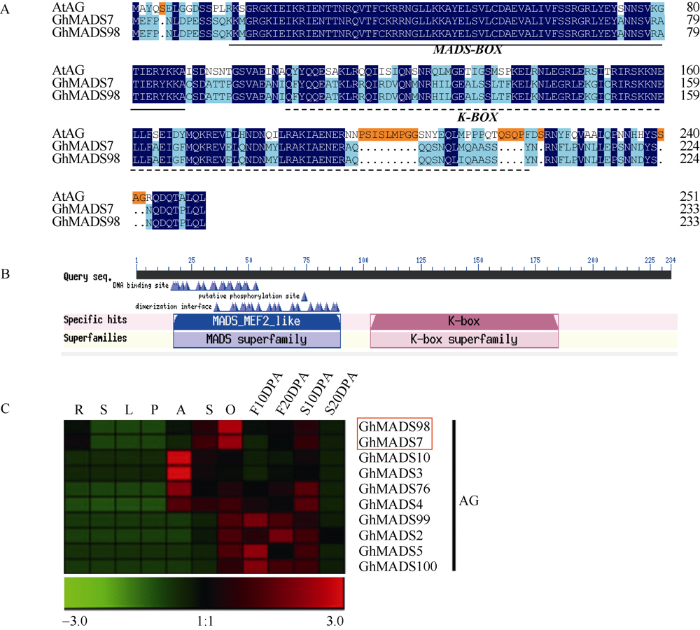 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2GhMADS7和GhMADS98基因序列及表达模式分析
A: GhMADS7基因及其同源基因蛋白序列分析; B: GhMADS7基因结构域分析; C: GhMADS7和GhMADS98基因表达模式分析。R: 根; S: 茎; L: 叶; P: 花瓣; A: 花药; S: 柱头; O: 胚珠; F10DPA/20DPA: 10 DPA和20 DPA纤维; S10DPA/20DPA: 10 DPA和20 DPA种子。
Fig. 2Sequence analysis and tissue expression pattern analysis of GhMADS7 and GhMADS98 in Gossypium hirsutum
A: alignment of the protein sequences of GhMADS7 and its homologous genes; B: the domain sequences analysis of GhMADS7; C: expression pattern analysis of GhMADS7 and GhMADS98 in Gossypium hirsutum. R: root; S: stem; L: leaf; P: petal; A: anther; S: stigma; O: ovule; F10 DPA/20 DPA: the fiber of 10 DPA/20 DPA; S10 DPA/20 DPA: the seed of 10 DPA/20 DPA.
2.3 GhMADS7基因表达模式分析
GhMADS7基因属于MIKCC型MADS-box基因家族AG亚族, 在植物中可能与花器官发育相关, 因此我们将陆地棉品种‘YZ1’的花蕾依据长度分成F<9 mm、F9~14 mm、F14~19 mm、F>19 mm和F0 (开花当天花蕾) 5个发育时期。并且将同一时期的花瓣(Petal)、花药(Anther)、柱头(Stigma)和胚珠(Ovule)进行组织分离, 以检测GhMADS7基因在陆地棉‘YZ1’不同发育时期的主要花器官中的表达变化。组织表达模式结果显示, GhMADS7基因在‘YZ1’开花当天花蕾的花瓣中呈现高量表达(图3-A); 在花药中, GhMADS7主要是在早中期(即在F<9 mm和F9~14 mm时期)花蕾中高量表达, 后期花药中几乎不表达(图3-B); 在柱头中, GhMADS7的表达量在花蕾长度为F9~14 mm和F0开花当天2个时期具有较高量表达(图3-C); GhMADS7的表达量除了在花蕾发育早期(即F<9 mm时期)的胚珠中未检测到表达外, 在其他时期的胚珠中表达量较高且不同时期间没有明显差异(图3-D)。因此, 我们推测GhMADS7基因在调控棉花花器官发育过程中对不同组织的调控作用具有时期差异性。图3
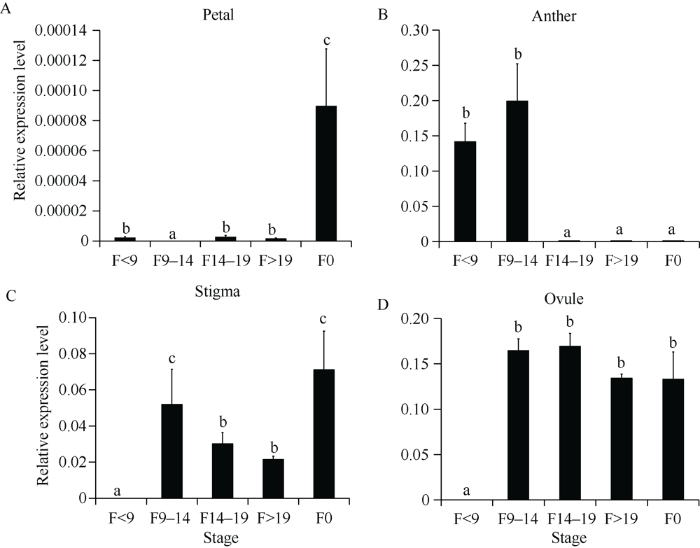 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3GhMADS7基因组织表达模式分析
F<9: 花蕾长度在9 mm以下; F9-14: 花蕾长度为9~14 mm; F14-19: 花蕾长度为14~19 mm; F>19: 花蕾长度在19 mm以上; F0: 开花当日花蕾。误差线为3次重复的误差。通过ANOVA方法分析A, B, C, D图的差异显著性, 不同的字母代表不同的差异显著性水平。
Fig. 3Expression pattern analysis of GhMADS7 in Gossypium hirsutum
F<9: the length of flower bud is less than 9 mm; F9-14: the length of flower bud is 9-14 mm; F14-19: the length of flower bud is 14-19 mm; F>19: the length of flower bud is above 19 mm; F0: the flower bud on the day of flowering. The error bars represent deviations of three replicates. Significant differences in A, B, C, D were found by one-way ANOVA (P<0.05). Values with different letters are significantly different at P<0.05.
2.4 降低GhMADS7的表达导致花瓣发育延缓
为进一步研究GhMADS7在陆地棉中的生物学功能, 本研究对GhMADS7 3° UTR 252 bp进行了干涉载体的构建, 并遗传转化陆地棉‘YZ1’。利用qRT-PCR检测发现, 5个干涉株系iGhMADS7-3、iGhMADS7-17、iGhMADS7-20、iGhMADS27、iGhMADS32相较于野生型‘YZ1’而言, 表达量均具有明显的下调, 其中iGhMADS7-27和iGhMADS7-32的表达水平的下调最明显(图4-A)。本研究选取表达量检测符合要求的转基因株系幼嫩的叶片进行DNA提取, 并进行基因插入拷贝数检测, Southern杂交检测的结果显示, 选取的5个转基因株系除iGhMADS7-3之外均为单拷贝(图4-B)。后续选取单拷贝且表达量低的iGhMADS7-27进行试验。图4
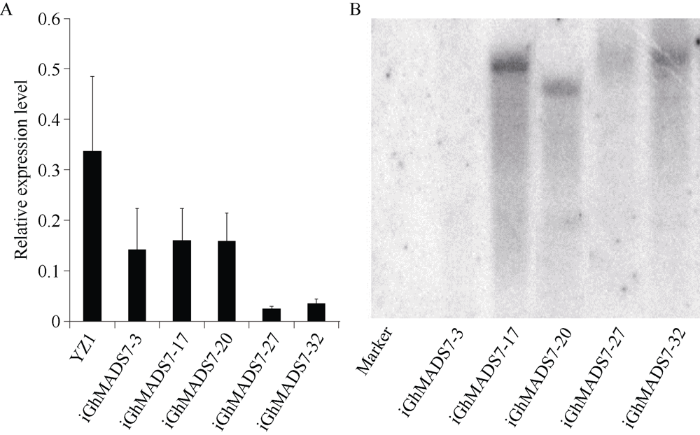 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4GhMADS7转基因株系表达量检测及T0代T-DNA插入拷贝数分析
A: GhMADS7转基因株系表达量检测; B: T0代转基因株系T-DNA插入拷贝数分析; YZ1: 野生型植株; iGhMADS7-3, iGhMADS7-17, iGhMADS7-20, iGhMADS7-27, iGhMADS7-32代表GhMADS7的5个干涉转基因株系。误差线表示3次技术重复的误差。
Fig. 4Relative expression level of GhMADS7 RNAi in different transgenic lines and copy number of T-DNA in T0 transgenic lines
A: relative expression level of GhMADS7 RNAi in different transgenic lines; B: copy number of T-DNA in T0 transgenic lines; YZ1: wild plant; iGhMADS7-3, iGhMADS7-17, iGhMADS7-20, iGhMADS7-27, iGhMADS7-32 represent five transgenic lines of GhMADS7 RNAi. The error bars represent deviations of three technical replicates.
我们将花蕾按以1 mm为界限进行长度划分, 选取了1~2 mm、2~3 mm、3~4 mm、4~5 mm、5~6 mm、6~7 mm、7~8 mm共7个时期进行花蕾纵切面观察。与野生型‘YZ1’相比, iGhMADS7-27出现花瓣发育延缓的表型最开始出现在花蕾长度为5~6 mm时期, 并随着花蕾的生长表型越来越明显(图5)。
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5GhMADS7-RNAi转基因植株表型考察
1~2 mm、2~3 mm、3~4 mm、4~5 mm、5~6 mm、6~7 mm、7~8 mm代表花蕾的长度; Se: 萼片; Pe: 花瓣; Sti: 柱头; Sta: 雄蕊; Ca: 心皮; Ov: 胚珠。图WT和iGhMADS7-27的标尺均为500 µm。
Fig. 5Phenotypic investigation of GhMADS7-RNAi transgenic plants
1-2 mm, 2-3 mm, 3-4 mm, 4-5 mm, 5-6 mm, 6-7 mm, and 7-8 mm represent the length of flower buds; Se: sepal; Pe: petal; Sti: stigma; Sta: stamen; Ca: carpel; Ov: ovule. Bar=500 µm (in WT and iGhMADS7-27).
为了解抑制GhMADS7转基因植株与野生型‘YZ1’植株的花瓣在细胞水平的变化情况, 本研究选取野生型‘YZ1’和iGhMADS7-27的转基因植株的花蕾进行石蜡切片观察发现, ‘YZ1’从2~3 mm花蕾开始花瓣维管束形成区细胞排列整齐有序, 一直到花蕾长度为5~6 mm时, 野生型‘YZ1’花瓣的维管束雏形形成, 并在花蕾长度为6~7 mm和7~8 mm时期花瓣维管束逐渐膨大生长; 而抑制GhMADS7的转基因株系iGhMADS7-27的花瓣维管束从2~3 mm时期至7~8 mm时期始终呈现排列紧凑皱缩的现象。但在相同长度的野生型和iGhMADS7-27花蕾中花瓣薄壁组织细胞层数保持一致(图6)。在花瓣中, 维管束的作用主要是输送养分和提供支撑, 因而推测抑制GhMADS7转基因植株iGhMADS7-27的维管束的皱缩可能会影响花瓣的正常生长发育, 从而导致干涉GhMADS7植株的花瓣发育在花蕾长度为6~7 mm和7~8 mm时出现明显延缓生长的表型。
图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6石蜡切片比较GhMADS7-RNAi转基因植株和野生型‘YZ1’的花瓣构造
iGhMADS7: 抑制GhMADS7转基因植株; YZ1: 野生型植株。(2~3) mm~(7~8) mm代表花蕾的发育长度。El: 上表皮细胞; Hl: 下表皮细胞; Pt: 薄壁组织; Vb: 维管束。8、10、12代表薄壁组织的细胞层数。图WT和iGhMADS7-27的标尺均为20 µm。
Fig. 6Comparison of petal structure between GhMADS7-RNAi transgenic plants and wild type YZ1 by paraffin section
iGhMADS7: suppression of GhMADS7 transgenic plants; YZ1: wild type plants. 2-3 mm to 7-8 mm represents the length of the bud. E1: epithelium cells; H1: hypodermic cells; Pt: thin-walled tissue; Vb: vascular bundle. 8, 10, 12 represent the cell layer number of the thin-walled tissue. Bar = 20 µm (in WT and iGhMADS7-27).
2.5 干涉GhMADS7转基因植株激活A、B类基因表达
在拟南芥的花器官发育形态的ABCDE模型中, 不同的花器官发育进程由不同类型基因调控[1]。而属于C类基因的GhMADS7基因, 若参照拟南芥的花器官发育调控机理, 应参与雄蕊和心皮的生长发育, 在GhMADS7-RNAi的转基因材料中却出现花瓣发育延缓的表型。花瓣的发育大多是由A、B类基因共同参与调控, 因此本研究在GhMADS7-RNAi的转基因材料中检测了陆地棉中的A类和B类基因的表达。结果表明, 在抑制GhMADS7转基因植株中, A类基因中的GhMADS41、GhMADS42、GhMADS43、GhMADS72、GhMADS73、GhMADS74和GhMADS89在出现明显表型之前的<5 mm的花蕾中有明显的上调, 且上调的趋势在花蕾长度为6~7 mm时最为明显(图7-A)。同时, B类基因中的GhMADS12、GhMADS51、GhMADS52、GhMADS53、GhMADS59和GhMADS87的表达量同样在6~7 mm时期呈明显上调趋势(图7-B)。说明在抑制GhMADS7转基因植株的花器官生长发育过程中, 当花蕾长度为6~7 mm时, 控制花瓣发育的A、B类基因发生明显的上调表达, 我们推测或许是由于抑制GhMADS7基因导致了花瓣发育延缓, 从而激活了调控花瓣发育的A、B类基因的表达。图7
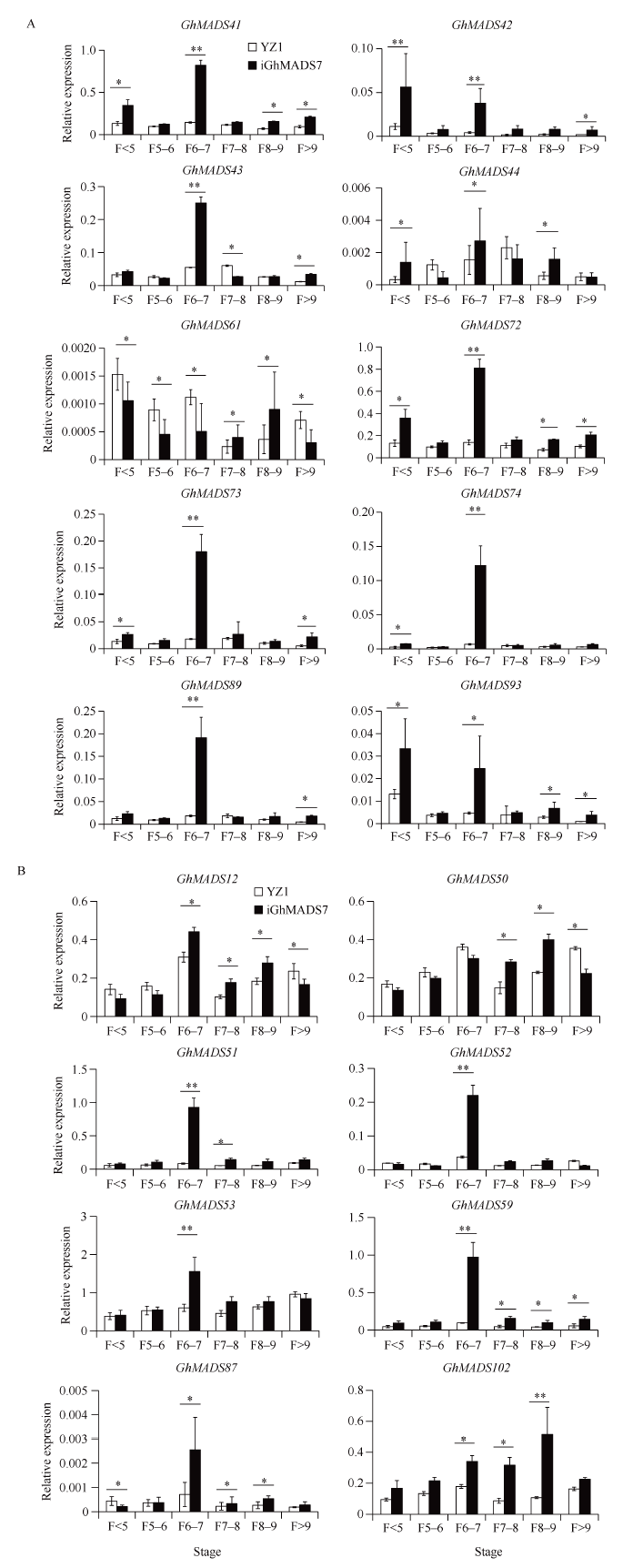 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7GhMADS7-RNAi转基因植株中A、B类基因表达量检测
A: A类基因在GhMADS7-RNAi转基因植株中表达量水平; B: B类基因在GhMADS7-RNAi转基因植株中表达量水平。YZ1: 野生型植株; iGhMADS7: 植株GhMADS7转基因植株。F<5: 花蕾长度在5 mm以下; F5-6: 花蕾长度为5~6 mm; F6-7: 花蕾长度为6~7 mm; F7-8: 花蕾长度为7~8 mm; F8-9: 花蕾长度在8~9 mm; F>9: 花蕾长度在9 mm以上。误差线表示3次重复的误差。*, **分别表示在0.05和0.01水平显著差异。
Fig. 7Relative expression levels of A and B type genes in GhMADS7 transgenic plants
A: the expression levels of A genes in GhMADS7-RNAi transgenic plants; B: the expression levels of B genes in GhMADS7-RNAi transgenic plants. YZ1: wild type plant; iGhMADS7: suppression of GhMADS7 transgenic plant. F<5: the length of flower bud is less than 5 mm; F5-6: the length of flower bud is 5-6 mm; F6-7: the length of flower bud is 6-7 mm; F7-8: the length of flower bud is 7-8 mm; F8-9: the length of flower bud is 8-9 mm; F>9: the length of flower bud is above 9 mm. The error bars represent deviations of three replicates. *, ** mean significant differences at the 0.05 and 0.01 probability levels.
3 讨论
MADS-box基因家族作为一类重要的转录调控因子, 通过与其他蛋白因子相结合参与植物的生长发育过程。GhMADS7的序列分析表明, GhMADS7具有MADS-box基因家族代表性的MADS结构域和高度保守的K结构域, 属于MIKCC型的MADS家族基因(图2)。GhMADS7与拟南芥AtAG基因同源性较高, AtAG在拟南芥中参与雄蕊和心皮的发育, AtAG功能的丧失会导致雄蕊和心皮转变成萼片和花瓣[22]。超表达茶树CsAG会导致出现花瓣缺失, 增厚的萼片上出现柱头和胚珠的现象[23]。在低温引起的月季花朵重瓣化研究中发现RhAG基因在月季重瓣中具有重要作用[24]。在紫薇重瓣花研究中发现, 与单瓣大花紫薇相比, 重瓣大花紫薇中C类基因LsAG1和LsAG2在花发育前期出现明显的上调表达[25]。山茶花AG类同源基因CjAG1基因主要对内轮器官起调控作用并能引起花瓣向雄蕊, 萼片向心皮演变[26]。因此我们推测GhMADS7可能同样参与棉花雄蕊和心皮的发育。通过观察GhMADS7干涉植株发现, 其出现花瓣发育延缓的表型, 且进一步分析表明, 花瓣出现发育延缓的时期是在花蕾长度为5~6 mm (图5), 并随着花蕾的生长表型越来越明显。在石蜡切片中发现干涉系植株花瓣的表皮细胞和薄壁组织中未发现与对照组有明显差异的现象, 但维管束从2~3 mm开始就表现为明显的收缩, 不能正常延伸(图6)。在夏槿中, 转化嵌合了AG阻遏物(AGSRDX)载体的植株会形成多余的维管束[27]。说明GhMADS7基因确实可能在花瓣的生长发育过程中起作用, 且可能与花瓣中的维管束的形成有关。花瓣的发育大多是由A、B类基因共同参与调控。我们通过检测A、B类基因的表达量发现, 在花蕾长度为6~7 mm时, 花瓣中的A、B类基因表达量有明显的上调(图7)。这种现象可能是由于抑制GhMADS7基因导致了花瓣发育延缓, 从而激活了调控花瓣发育的A、B类基因的表达。
前人从陆地棉中克隆出与拟南芥AG蛋白具有62%同源性的GhMADS3 (AY083173), 研究发现, 在烟草中异位表达GhMADS3会导致烟草萼片转变成心皮, 花瓣转变成雄蕊, 表明GhMADS3在花发育中具有与拟南芥AtAG相似的作用[28]。通过在NCBI网站上进行BLASTP发现, 其研究的基因与本研究中进化分析所用的GhMADS3 (Gh_A10G2220)有98%的相似度, 且本研究中GhMADS3与AtAG同源性为67%, 高于GhMADS7与拟南芥AG基因的同源性(64%, 图8), GhMADS3与GhMADS7同为C类基因, 猜测GhMADS3与AtAG的功能更为相似。在研究水稻C类基因OsMADS3和OsMADS58时发现, 分别干涉这2个基因会出现完全不同的花器官发育表型, 表明由AG调控的功能已经被划分为旁系基因OsMADS3 和OsMADS58, 研究还发现, OSMADS3和OSMADS58共同调节雄蕊的特征, 但OSMADS3发挥着更关键性的作用[29]。水稻中一种B类基因OsMADS16可能与C类基因OSMADS3和OsMADS58共同作用以决定花器官的形成, 但OSMADS3 和OSMADS58与OsMADS16互不影响表达[30]。在毛茛科C类MADS-box基因研究中发现了花瓣起源于雄蕊的有利证据, 并且推测C类基因在花瓣的形成中也发挥了部分作用[31]。C类基因的后续复制导致了旁系同源基因亚功能化同时又保持了部分功能冗余[32]。因此我们推测虽然GhMADS7与拟南芥AG基因具有64%的同源性, 但其可能正调控花瓣发育过程, 具体调控机制有待进一步研究。
图8
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8GhMADS7、GhMADS3与AtAG蛋白序列比对
A: GhMADS7与ATAG蛋白序列比对; B: GhMADS3与ATAG蛋白序列比对。
Fig. 8Alignment of the protein sequences of GhMADS7, GhMADS3, and AtAG
A: alignment of the protein sequences of GhMADS7 and ATAG; B: alignment of the protein sequences of GhMADS3 and AtAG.
4 结论
本研究克隆了1个AG亚家族MIKCC型的MADS-box家族基因GhMADS7, 其在花瓣、花药、柱头、胚珠等花器官中都有表达, 且在‘YZ1’开花当天花蕾的花瓣中呈现高量表达; 以‘YZ1’为遗传背景对GhMADS7基因进行RNAi干涉后发现, 其花蕾在5~6 mm时期开始出现花瓣发育延缓的表型; 且石蜡切片显示转基因株系iGhMADS7-27的花瓣维管束从2~3 mm时期至7~8 mm时期始终呈现排列紧凑皱缩的现象; 通过探究GhMADS7-RNAi转基因株系中A、B类基因的表达量发现, 在花蕾长度为6~7 mm时A、B类基因表达量明显上调。对比前人研究结果, GhMADS3在烟草中的异位表达后出现的表型与拟南芥AtAG在花发育过程中的功能相似。推测GhMADS3在花发育中担当C类基因的功能, 而本研究结果证实GhMADS7确与花瓣发育有关, 其调控机制还需进一步研究。Table S1
附表1
附表1用于进化树分析的78条MADS蛋白序列基因号
Table S1
| 基因号 ID | 基因名称 Name | 基因号 ID | 基因名称 Name | 基因号 ID | 基因名称 Name |
|---|---|---|---|---|---|
| Gh_D04G1849 | GhMADS1 | Gh_A08G1148 | GhMADS45 | Gh_D03G1493 | GhMADS75 |
| Gh_D05G2375 | GhMADS2 | Gh_A07G1339 | GhMADS46 | Gh_D10G0308 | GhMADS76 |
| Gh_A10G2220 | GhMADS3 | Gh_D08G1430 | GhMADS47 | Gh_D13G1226 | GhMADS77 |
| Gh_A10G2221 | GhMADS4 | Gh_A09G2157 | GhMADS48 | Gh_D11G0882 | GhMADS78 |
| Gh_D05G2596 | GhMADS5 | Gh_A04G1265 | GhMADS49 | Gh_D12G1000 | GhMADS79 |
| Gh_D04G0341 | GhMADS7 | Gh_A01G1608 | GhMADS50 | Gh_A04G1491 | GhMADS80 |
| Gh_A12G0910 | GhMADS9 | Gh_D03G0105 | GhMADS51 | Gh_D11G0883 | GhMADS81 |
| Gh_D10G0309 | GhMDS10 | Gh_D05G2452 | GhMADS52 | Gh_D12G1027 | GhMADS82 |
| Gh_D04G1892 | GhMADS11 | Gh_A05G2191 | GhMADS53 | Gh_D06G0245 | GhMADS84 |
| Gh_A02G0736 | GhMADS12 | Gh_D06G0267 | GhMADS54 | Gh_D11G3150 | GhMADS85 |
| Gh_A11G0754 | GhMADS13 | Gh_D02G2012 | GhMADS55 | Gh_D11G0082 | GhMADS86 |
| Gh_D12G2226 | GhMADS14 | Gh_A04G0934 | GhMADS57 | Gh_D02G0779 | GhMADS87 |
| Gh_A11G0077 | GhMADS15 | Gh_A12G0936 | GhMADS58 | Gh_A03G1085 | GhMADS88 |
| Gh_D02G1502 | GhMADS16 | Gh_A12G0570 | GhMADS59 | Gh_A07G0605 | GhMADS89 |
| Gh_D07G1814 | GhMADS17 | Gh_A11G0755 | GhMADS60 | Gh_A03G2004 | GhMADS90 |
| Gh_D13G0877 | GhMADS20 | Gh_D07G0780 | GhMADS61 | Gh_A12G2048 | GhMADS91 |
| Gh_D04G1451 | GhMADS27 | Gh_A07G1615 | GhMADS62 | Gh_A04G1264 | GhMADS93 |
| Gh_A13G0524 | GhMADS28 | Gh_A06G0244 | GhMADS64 | Gh_A13G0981 | GhMADS94 |
| Gh_D11G0400 | GhMADS36 | Gh_D13G0605 | GhMADS66 | Gh_A03G1563 | GhMADS95 |
| Gh_A06G1875 | GhMADS37 | Gh_A08G1275 | GhMADS67 | Gh_A12G0150 | GhMADS96 |
| Gh_D11G0534 | GhMADS38 | Gh_D12G0778 | GhMADS68 | Gh_A13G0425 | GhMADS97 |
| Gh_A12G0775 | GhMADS40 | Gh_D12G0163 | GhMADS70 | Gh_A05G3267 | GhMADS98 |
| Gh_A13G0751 | GhMADS41 | Gh_D09G2362 | GhMADS71 | Gh_A05G2136 | GhMADS99 |
| Gh_D04G1891 | GhMADS42 | Gh_D13G0878 | GhMADS72 | Gh_A05G2334 | GhMADS100 |
| Gh_A03G0634 | GhMADS43 | Gh_D07G0671 | GhMADS73 | Gh_Sca004768G07 | GhMADS101 |
| Gh_D02G1311 | GhMADS44 | Gh_D03G0922 | GhMADS74 | Gh_Sca007246G01 | GhMADS102 |
新窗口打开|下载CSV
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 2]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
