 ,1,**, 施龙建
,1,**, 施龙建 ,1,2,**, 田红丽1, 易红梅1, 杨扬1, 葛建镕1, 范亚明1, 任洁1, 王璐1, 陆大雷2, 赵久然
,1,2,**, 田红丽1, 易红梅1, 杨扬1, 葛建镕1, 范亚明1, 任洁1, 王璐1, 陆大雷2, 赵久然 ,1,*, 王凤格
,1,*, 王凤格 ,1,*
,1,*Identification of SNP core primer and establishment of high throughput detection scheme for purity identification in maize hybrids
WANG Rui ,1,**, SHI Long-Jian
,1,**, SHI Long-Jian ,1,2,**, TIAN Hong-Li1, YI Hong-Mei1, YANG Yang1, GE Jian-Rong1, FAN Ya-Ming1, REN Jie1, WANG Lu1, LU Da-Lei2, ZHAO Jiu-Ran
,1,2,**, TIAN Hong-Li1, YI Hong-Mei1, YANG Yang1, GE Jian-Rong1, FAN Ya-Ming1, REN Jie1, WANG Lu1, LU Da-Lei2, ZHAO Jiu-Ran ,1,*, WANG Feng-Ge
,1,*, WANG Feng-Ge ,1,*
,1,*通讯作者:
收稿日期:2020-06-14接受日期:2020-10-14网络出版日期:2021-04-12
| 基金资助: |
Received:2020-06-14Accepted:2020-10-14Online:2021-04-12
| Fund supported: |
作者简介 About authors
王蕊, E-mail:
施龙建, E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (1156KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
王蕊, 施龙建, 田红丽, 易红梅, 杨扬, 葛建镕, 范亚明, 任洁, 王璐, 陆大雷, 赵久然, 王凤格. 玉米杂交种纯度鉴定SNP核心引物的确定及高通量检测方案的建立[J]. 作物学报, 2021, 47(4): 770-779. doi:10.3724/SP.J.1006.2021.03031
WANG Rui, SHI Long-Jian, TIAN Hong-Li, YI Hong-Mei, YANG Yang, GE Jian-Rong, FAN Ya-Ming, REN Jie, WANG Lu, LU Da-Lei, ZHAO Jiu-Ran, WANG Feng-Ge.
玉米(Zea mays L.)是世界上最早且最充分利用杂种优势进行生产的作物, 国内目前90%以上的玉米生产用种是单交种[1]。杂交种的纯度高低是影响田间产量、产品品质以及农民收益的关键因素之一[2]。因此, 玉米杂交种纯度鉴定是把控玉米种子质量的重要手段, 尤其是对于杂交种中自交株的检测, 在企业质量自控、政府市场监测等多个环节提供重要依据。当前常用于玉米杂交种纯度鉴定的方法为田间小区种植鉴定法[3]、盐溶蛋白法[4]以及SSR (simple sequence repeats)标记法[5,6]等。但以上3种方法都存在一些不足, 田间小区种植鉴定周期长且结果易受环境和人员判断影响; 盐溶蛋白法在某些品种上找不到双亲差异或无法区分自交苗, 不能鉴定所有品种; SSR分子标记等鉴定法无法满足样本高通量的检测需求, 尤其是制种到市场销售期间短期大量品种纯度鉴定的需求[7]。因此探索出一套高效准确且适用于市场上绝大多数的玉米杂交种纯度鉴定方案具有重要意义。
近年来, 随着分子标记的快速发展, 第三代分子标记, 即SNP (single nucleotide polymorphism)标记, 因其数量多、集成高、可微型化及自动化程度高等优势, 广泛应用于种质资源研究、品种身份鉴定、分子辅助育种(marker-assisted selection, MAS)等[8,9,10,11]领域。SNP检测方法众多, 包括水解探针法、高密度基因芯片以及竞争性等位基因特异性PCR法(kompetitive allele-specific PCR, KASP)等[12,13,14]。其中KASP平台由于具有高效、低成本的特性, 目前已开发大麦[15]、棉花[16]等作物的纯度鉴定标记。本研究依据建成的国家审定玉米品种指纹数据[17], 选择60个SNP位点作为候选位点设计合成KASP引物, 以不同地区代表性品种为材料, 结合快速DNA提取法、高通量检测平台以及数据自动分析等筛选位点分型效果, 确定一套适用于玉米杂交种纯度鉴定的SNP核心引物组合, 形成基于KASP技术的玉米品种纯度鉴定检测体系, 并建立玉米纯度鉴定高通量检测方案。
1 材料与方法
1.1 供试材料
供试材料包括12组玉米杂交种及其亲本组成的三联体样品(表1), 用于测试KASP引物设计是否成功, 是否符合亲子关系以及快提法DNA的分型效果; 335份1984—2013年国家审定玉米杂交种标准样品[17], 用于构建SNP指纹数据并筛选候选位点; 市场购买玉米杂交种京科968种子若干及本实验室收集母本京724、父本京92, 用于纯度测试。Table 1
表1
表1玉米三联体样品信息表
Table 1
| 编号 No. | 杂交种 Hybrid | 母本 Maternal parent | 父本 Paternal parent |
|---|---|---|---|
| 1 | 先玉335 Xianyu 335 | PH6WC | PH4CV |
| 2 | 郑单958 Zhengdan 958 | 郑58 Zheng 58 | 昌7-2 Chang 7-2 |
| 3 | 农大108 Nongda 108 | X178 | 黄C Huang C |
| 4 | 鲁单981 Ludan 981 | 齐319 Qi 319 | Lx 9801 |
| 5 | 沈单16 Shendan 16 | K12 | 沈137 Shen 137 |
| 6 | 东单60 Dongdan 60 | A801 | 丹598 Dan 598 |
| 7 | 丹玉39 Danyu 39 | C8605-2 | 丹598 Dan 598 |
| 8 | 中单2号 Zhongdan 2 | Mo17 | 自330 Zi 330 |
| 9 | 掖单13 Yedan 13 | 掖478 Ye 478 | 丹340 Dan 340 |
| 10 | SC704 | B73 | Mo17 |
| 11 | 京科糯2000 Jingkenuo 2000 | 京糯6 Jingnuo 6 | 白糯6 Bainuo 6 |
| 12 | 农华101 Nonghua 101 | NH60 | S121 |
新窗口打开|下载CSV
1.2 DNA提取
采用改良CTAB法混株提取建库样品及参照样品DNA, 并用1%琼脂糖凝胶电泳及Nanodrop 2000检测DNA浓度及质量, 统一稀释到20 ng μL-1备用, 每份样品2个重复。纯度检测采用改良DNA快提法(K-Mate DNA提取试剂盒, LGC Genomic)单株提取。1.3 SNP纯度候选位点及引物设计
根据335份国家审定玉米杂交种标准样品的SNP指纹数据[17]统计384基础位点的双亲互补率、最小等位基因频率(minimum allele frequency, MAF)、多态性信息含量(polymorphic information content, PIC)等参数, 挑选出在染色体上分布均匀, 多态性好的60个位点作为纯度候选位点, 并设计合成KASP引物(表2) (LGC Genomic, 英国), 在2条正向引物序列的5′端加上FAM或HEX荧光基团的接头序列[18], 并按照上游引物12 μmol L-1, 下游引物30 μmol L-1混合制备成引物工作液备用。Table 2
表2
表2玉米杂交种纯度鉴定候选引物表
Table 2
| 引物编号 Primer ID | 引物名称 Primer name | 等位变异 Allelic variation | 引物序列 Primer sequence (5'-3') |
|---|---|---|---|
| MSNP01 | MG002 | A/G | F1: GGAACTGTGCGTAGTTCCTGAGA F2: GAACTGTGCGTAGTTCCTGAGG R: TGATCGAGGAGGACCCCTGCAA |
| MSNP02 | MG013 | A/G | F1: CCGGAGCTGGCTTCTGAATCAA F2: CGGAGCTGGCTTCTGAATCAG R: ACATCCCCATCCTGGGGAGGAA |
| MSNP03 | MG016 | T/C | F1: GAGCGTCAACGACAAAGCCAAGT F2: AGCGTCAACGACAAAGCCAAGC R: CCTGTTAGGTTGTAAGAATTGAGCCTTAT |
| MSNP04 | MG035 | A/G | F1: GGAGATGCTCTACAAGTTTGTCA F2: GGAGATGCTCTACAAGTTTGTCG R: CCTTCGAGGAGGCCAAGGACTT |
| MSNP05 | MG046 | A/G | F1: CCATGGTTTTAAGGAACTATCGAAAGA F2: CATGGTTTTAAGGAACTATCGAAAGG R: ATCAATGTACTCCCATAAGCAGCAACTTT |
| MSNP06 | MG049 | C/T | F1: CCGTATCATTTAGTGCATCAGAACC F2: CCGTATCATTTAGTGCATCAGAACT R: GAGAAATGCAATGCATCAGATCCGGAT |
| MSNP07 | MG055 | A/G | F1: CATCGAATGGCTGATCATGTTGCAT F2: ATCGAATGGCTGATCATGTTGCAC R: CCATTCCCTGTATGACAGACACGAT |
| MSNP08 | MG059 | C/T | F1: GCCCCTGCATTGTTTGCAGC F2: CTGCCCCTGCATTGTTTGCAGT R: GCCACTGCAAATCCAAAGAATCCGTA |
| MSNP09 | MG064 | G/T | F1: GACTAAACACACTCACTTTCCTGC F2: CTGACTAAACACACTCACTTTCCTGA R: AATCGGTGATGAGTGAGTGTCTAACTAAA |
| MSNP10 | MG066 | A/C | F1: AGGATCACAATCCATCTGCTGCAAA F2: GATCACAATCCATCTGCTGCAAC R: CCTGCAGTTGCTACTGATAGTTCTCAA |
| MSNP11 | MG067 | A/G | F1: ATGTTTCTCAGGACGGTAATAGTGAT F2: GTTTCTCAGGACGGTAATAGTGAC R: CCCATCCATTCCACATATTCGGCAA |
| MSNP12 | MG072 | A/C | F1: GAGTTGTGCTCTGATCCACCCT F2: AGTTGTGCTCTGATCCACCCG R: TATTGCGGCATTAAACAAGGGAAAGGAAA |
| MSNP13 | MG088 | C/T | F1: CGGCCTCCATGCTTGATGATG F2: CCGGCCTCCATGCTTGATGATA R: GTCGGTCGAGTCAAAATTCATTTTGGAT |
| MSNP14 | MG092 | C/T | F1: ACGTCTGACTTGCACGAAACGG F2: GACGTCTGACTTGCACGAAACGA R: CTCCGGAGATGATCTCCCGTAATTT |
| MSNP15 | MG094 | G/T | F1: ATTCGAACTGCTGCCTTGACTAATC F2: ATTCGAACTGCTGCCTTGACTAATA R: GCAGCAGTGACTATCCTTCTGAAGAA |
| MSNP16 | MG101 | A/C | F1: CAATTCCTCCCCTGCAATTTCAACAA F2: AATTCCTCCCCTGCAATTTCAACAC R: CAGCCTGGTCGTTGCTTCTGTAATT |
| MSNP17 | MG106 | C/T | F1: CCAATCAAGGCGGCAACATACC F2: ACCAATCAAGGCGGCAACATACT R: GCGTTCATGTTCATGGAAGGCCAAA |
| MSNP18 | MG111 | A/G | F1: ATTCTGAATGTAAAACTTAACATGCTGCTA F2: CTGAATGTAAAACTTAACATGCTGCTG R: AAGTCCTTCCAACTTTCAGCATAAGCAAA |
| MSNP19 | MG134 | A/C | F1: GGTTACACGACCAAATGAGTACCAT F2: GTTACACGACCAAATGAGTACCAG R: TAGGCAGAGCAGCCATTGACAAGTA |
| MSNP20 | MG135 | G/T | F1: CGCATCCTAATAACATAATTACTCACG F2: ACGCATCCTAATAACATAATTACTCACT R: GCGAAACGGGGTGTTAGATAGAGTT |
| 引物编号 Primer ID | 引物名称 Primer name | 等位变异 Allelic variation | 引物序列 Primer sequence (5'-3') |
| MSNP21 | MG143 | G/T | F1: TATCACTTGTGGATCTATATCTGTG F2: GCTTATCACTTGTGGATCTATATCTGTT R: TGAACCCAAAGCCTCGGTGTTCTTT |
| MSNP22 | MG148 | G/T | F1: AGCTTAGCAGAGCTGCATCTG F2: CTAGCTTAGCAGAGCTGCATCTT R: CCCACGTCACCTAGATAAGCCAAAA |
| MSNP23 | MG155 | A/G | F1: ATACAGTGAAACAGCTTGCACTGGA F2: CAGTGAAACAGCTTGCACTGGG R: TTAATTTTTGGAAGAGCTTGCGTTGGGAA |
| MSNP24 | MG157 | A/G | F1: AGTTACCTGTCATCGATCTCTGGAT F2: ACCTGTCATCGATCTCTGGAC R: AAAGCCCTCTGACAATGCTCCAGTA |
| MSNP25 | MG175 | C/T | F1: GATATTTCTGCAACTAAACATGGCAAG F2: GGATATTTCTGCAACTAAACATGGCAAA R: ATACTGGGGTTGTGGGGATAGGATT |
| MSNP26 | MG181 | A/G | F1: CCTCTGTAAGCGCAGTACTGGT F2: CTCTGTAAGCGCAGTACTGGC R: AAATTGCTATGCAAACAGGTTCTGGAGTA |
| MSNP27 | MG186 | C/T | F1: GAGCTAGTAAATATTGTTGTTGTTCCTC F2: AGAGCTAGTAAATATTGTTGTTGTTCCTT R: CGCCGACGGGACGACGGAT |
| MSNP28 | MG191 | A/G | F1: CATAAACAGTAGGTTTATCGCTGACATAA F2: AAACAGTAGGTTTATCGCTGACATAG R: GTGATAACCGATGCAAAATGCTGCTTAAT |
| MSNP29 | MG195 | C/T | F1: CCAAAGGATAGCACATCTTGGTG F2: GTCCAAAGGATAGCACATCTTGGTA R: TGTCAACCGCATCCTGGCAGATAAT |
| MSNP30 | MG207 | A/G | F1: ACTTCTCCATCCTCTTCCAACATATTA F2: CTTCTCCATCCTCTTCCAACATATTG R: AGCTGTCCACCATCAGTACTGGAAT |
| MSNP31 | MG215 | T/C | F1: AGATGGCATTGTGATCTGTGCACA F2: GATGGCATTGTGATCTGTGCACG R: AGCCGAAGGATTGATCCTCCTCAT |
| MSNP32 | MG216 | G/T | F1: GACGACGACTCCATCGTGACC F2: GACGACGACTCCATCGTGACA R: TCAACCCATGGCTGCTCACATGTAA |
| MSNP33 | MG221 | G/T | F1: GGCATTCTGATTTGACAGCCCAC F2: GGCATTCTGATTTGACAGCCCAA R: TCCTGATTCTGTACTTGATTGGACCAAA |
| MSNP34 | MG230 | C/T | F1: GCAGCTGAGAAACAATTGCAAAGTG F2: GCAGCTGAGAAACAATTGCAAAGTA R: GTACTCTCAGATGGTTTTGTGACATCAA |
| MSNP35 | MG236 | C/T | F1: GTGCTCGAACGAATCGACCAG F2: CGTGCTCGAACGAATCGACCAA R: CATCCATGGCGAAGCTCATGAACAA |
| MSNP36 | MG240 | G/T | F1: GAAACATGAATGCCCTAAATCCTTCG F2: AGAAACATGAATGCCCTAAATCCTTCT R: ATTATGTTCACCAAGTATCCAGATGGCAT |
| MSNP37 | MG262 | G/T | F1: GTAGCGTGTCTCTACGCTCTG F2: ATGTAGCGTGTCTCTACGCTCTT R: CAGCGCGTTACGACGAACTCCAA |
| MSNP38 | MG270 | A/C | F1: GGTTCCATGGCTACCTGACAAGT F2: GTTCCATGGCTACCTGACAAGG R: TAGGAGCTAGCCAAGAGCCTACTA |
| MSNP39 | MG271 | A/G | F1: CAGCGACCTCAAGAAGTTGAAGTAA F2: AGCGACCTCAAGAAGTTGAAGTAG R: GTACGACATGCAGTTTGACATCAAGTAT |
| MSNP40 | MG277 | A/G | F1: GAAGCTACTATTAGCAATGATCTATATGAT F2: AAGCTACTATTAGCAATGATCTATATGAC R: ACAGGATTGATAAACATTACCTGCAGGAA |
| MSNP41 | MG278 | A/G | F1: GATCGTTGTCTTCACAAATGAAGAATAGT F2: CGTTGTCTTCACAAATGAAGAATAGC R: GCGAGATATTGAAAGCTAGTGGTGCTA |
| 引物编号 Primer ID. | 引物名称 Primer name | 等位变异 Allelic variation | 引物序列 Primer sequence (5'-3') |
| MSNP42 | MG279 | A/G | F1: AACGTATGAGATGAACTCACCAGAAA F2: ACGTATGAGATGAACTCACCAGAAG R: CTCCGCCGCTGGTGGAGCTA |
| MSNP43 | MG288 | A/C | F1: GAACTAACTGAGTGTTAAAGGAGCTTAT F2: AACTAACTGAGTGTTAAAGGAGCTTAG R: CCTTGACACAACCGCTCTCCTTAAA |
| MSNP44 | MG292 | A/G | F1: AACCATTCCCTTCATACTTCTTCTCT F2: ACCATTCCCTTCATACTTCTTCTCC R: GGGAGTATCTTTTAGGAAGATGTACAGAT |
| MSNP45 | MG293 | A/C | F1: GACCTGAAATGCTTGGCGAGTCA F2: ACCTGAAATGCTTGGCGAGTCC R: GCAGGAGCCTTAGCGTGGCTAT |
| MSNP46 | MG298 | A/G | F1: CAATCCAAAGCAGAAAGAAGTTGTTCT F2: AATCCAAAGCAGAAAGAAGTTGTTCC R: CCAAAACAGTGAAGTGACCGCCAT |
| MSNP47 | MG304 | C/T | F1: CAAAGTGGTGTAAATGGATGGATCG F2: CAAAGTGGTGTAAATGGATGGATCA R: TTGGACACTCCAGGGGATCCTATA |
| MSNP48 | MG311 | C/T | F1: CCTCAGATCTCATCTATGCTGCC F2: CCTCAGATCTCATCTATGCTGCT R: CGTTTCCACATTTTCTGAAGGTTTCACAA |
| MSNP49 | MG328 | A/G | F1: GCCTCACACATCCATATACGTAGAA F2: CCTCACACATCCATATACGTAGAG R: CTTCCATGCATCGCCCTATGGATAT |
| MSNP50 | MG331 | C/T | F1: AACACTCATGTCTGCTCCAGGG F2: AAACACTCATGTCTGCTCCAGGA R: TCGATGTTTTCGATCCCAAGTTCAACATT |
| MSNP51 | MG334 | G/T | F1: CCACTTCTGCTCGTATGATCTTC F2: GCCACTTCTGCTCGTATGATCTTA R: TCCCGTAATCATCTGCTCGTCTGTA |
| MSNP52 | MG347 | A/G | F1: AACACGAGCTGGTTGATGGATTAGT F2: CACGAGCTGGTTGATGGATTAGC R: GCCTCTGGTACGTTAGTTTGCAGTT |
| MSNP53 | MG349 | A/G | F1: TACTGACCGAGCGATGCTGCT F2: CTGACCGAGCGATGCTGCC R: CGCTGATGGTCACAGAAACATCGTT |
| MSNP54 | MG353 | A/G | F1: ATACCCTCTCCACCAGTTGTTGAT F2: ACCCTCTCCACCAGTTGTTGAC R: TCGCAGGGAGGCGTCGTTCAA |
| MSNP55 | MG356 | A/C | F1: AAAAGTGCAGTTCCTTGCTGTTCATTT F2: AAGTGCAGTTCCTTGCTGTTCATTG R: CCCAATGAGCAAAAAGAATAGCACCAAA |
| MSNP56 | MG361 | C/T | F1: GAAGCAATCCTTCCGGAGGAATG F2: GAAGCAATCCTTCCGGAGGAATA R: GAATGTGCAGATTGGATTTGAGGGATAAA |
| MSNP57 | MG364 | A/G | F1: TGTTCCGAATAGCAAGTGATCTCTTT F2: GTTCCGAATAGCAAGTGATCTCTTC R: GGGAAACCTGCAGAATGCTGTTGAT |
| MSNP58 | MG369 | A/G | F1: GGTTGACATGAGACTTGCAGAGA F2: GGTTGACATGAGACTTGCAGAGG R: TCGGGAAGCCATACTTCACATGCAT |
| MSNP59 | MG371 | G/T | F1: CAAGTGCGCAGCAAGCCAAAAG F2: AAACAAGTGCGCAGCAAGCCAAAAT R: CCGTTCTTAAGCGCTCCATCCTTTT |
| MSNP60 | MG382 | C/T | F1: CACGAAGCTCTCGCGCTCTTC F2: CACGAAGCTCTCGCGCTCTTT R: GGCATGGAGCCCCTATCCTTGAT |
新窗口打开|下载CSV
1.4 PCR体系与程序
反应体系: 1.5 μL DNA (烘干), 0.5 μL 2×KASP PCR预混液(LGC Genomic, 1536格式, 标准含量ROX, 英国), 0.486 μL去离子水, 0.014 μL引物工作液。反应程序: 61~55℃梯度循环程序: 第1步: 94℃ 15 min, 94℃ 20 s, 第2步: 61~55℃10个循环, 每循环递减0.6℃, 第3步: 94℃ 20 s, 55℃ 60 s, 26个循环, 在高通量水浴锅PCR仪(Hydrocycler 64, LGC Genomic)上进行扩增反应。1.5 荧光检测及基因型数据采集
利用Pherastar荧光扫描仪采集原始荧光信号, 利用Kraken (V16.3.16.16288)软件进行基因分型, 将分型结果导入SNP-DNA数据库管理系统(V1.0.0, 软件登记号: 2018SR043088, 北京市农林科学院玉米研究中心研发)分析待测样品纯度。1.6 数据分析
使用SNP比对统计工具软件(V1.0, 软件登记号: 2018SR026743, 北京市农林科学院玉米研究中心研发)计算引物双亲互补率、MAF和PIC等参数。引物双亲互补率为该引物具有双亲互补基因型的品种数与总品种数的百分比, 样品双亲互补基因型比率为该样品上具有双亲互补基因型的引物数与总引物数的百分比。待测样品纯度计算为正常个体数目(待测样品总数减去非典型个体数目)与待测样品总数的百分比。2 结果与分析
2.1 适于玉米杂交种纯度鉴定核心引物的筛选与确定
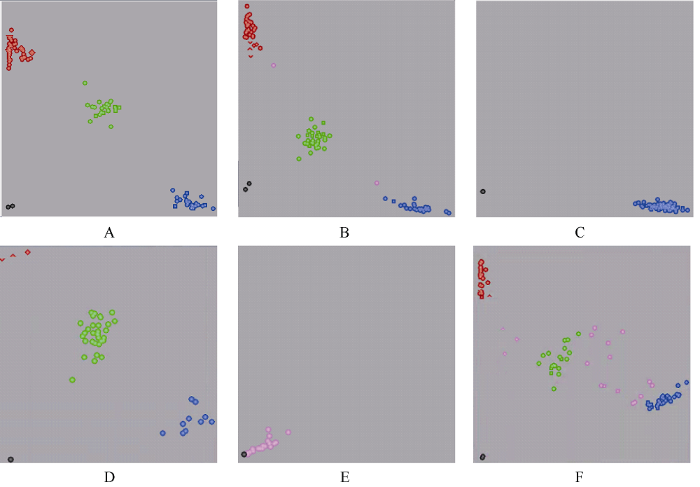
基于玉米384个SNP基础位点, 根据位点双亲互补区分能力、试验分型效果、染色体均匀分布等条件筛选确定1套适于玉米杂交种纯度鉴定的核心引物组合。具体筛选过程为: (1) 确定候选位点组合: 基于335份国审品种基础位点的指纹数据计算位点双亲互补率、多态性等参数, 基础位点双亲互补率介于24.2%~65.6%之间, MAF值介于0.32~0.50之间, PIC值介于0.34~0.38之间。候选位点的筛选条件为位点双亲互补率高于40%, MAF≥0.33, PIC≥0.35, 从而优选60个候选位点。(2) KASP引物评估: 将候选位点设计合成KASP引物, 使用12组玉米杂交种及其亲本三联体材料进行测试。其中57个引物都能分成紧密的三群, 分型结果与预期结果一致, 符合孟德尔遗传定律, 并且试验重复结果一致(图1-A); 3个引物不能正常分型或与预期结果不一致或扩增失败(图1-C, F), 从芯片位点转化为KASP引物的成功率为95%。(3) 快速DNA提取法评估: 为缩短DNA制备时间, 提高纯度鉴定效率, 进一步使用快提法对CTAB法分型表现好的位点进行测试, 选择CTAB法和快提法均能清晰的分成3个紧密的分型, 且与预期分型结果一致的位点(图1-D), 共有48个位点进入下一步的位点筛选; 9个引物使用CTAB法能够正常分型, 但使用快提法无法正常分型(图1-B, E), 不再进行纯度核心引物筛选。(4) 确定纯度鉴定核心引物: 综合位点区分能力及染色体均匀分布等, 筛选出20个KASP引物作为纯度鉴定核心引物(表3)。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1测试引物分型效果图
A, B, C, F为CTAB法分型效果; D, E为快提法分型效果。A, D为引物MG279, 在CTAB法和快提法均具有清晰、紧密的分型效果; B, E为引物MG304, 该位点使用CTAB法能够正常分型, 但快提法无法正常分型; C为引物MG262, KASP引物分型效果为单态; F为引物MG067, 分型效果较散且数据缺失率较高。图中每1个点代
Fig. 1Genotyping of tested primers
A, B, C, and F are the genotyping of CTAB method; D and E are the genotyping of fast extraction method. A and D are primer MG279, which have clear and close genotyping effect both by CTAB method and rapid extraction method; B and E are primer MG304, which has normal genotyping by CTAB method, but abnormal genotyping by rapid extraction method; C is primer MG262, of which KASP primer genotyping effect is monomorphic; F is primer MG067, of which KASP primer genotyping effect is wide and has high missing data rate. Each point represents the result of one reaction well, of which blue and red points represent two homozygous genotype, green points represent parental complementary genotype, pink points represent unused genotype and black points represent non-template control.
Table 3
表3
表3玉米杂交种纯度鉴定SNP核心引物信息表
Table 3
| 引物编号 Primer ID | 引物名称 Primer name | 染色体 Chromosome | 染色体位置 Chromosome position | 双亲互补率 Parental complementary rate (%) | 最小等位 基因频率 MAF | 多态信息 含量 PIC |
|---|---|---|---|---|---|---|
| MSNP01 | MG002 | 1 | 8974336 | 51.1 | 0.50 | 0.38 |
| MSNP06 | MG049 | 1 | 289408285 | 58.0 | 0.46 | 0.37 |
| MSNP09 | MG064 | 2 | 109563976 | 55.8 | 0.34 | 0.35 |
| MSNP12 | MG072 | 2 | 186339948 | 53.6 | 0.47 | 0.37 |
| MSNP14 | MG092 | 3 | 27775731 | 62.3 | 0.47 | 0.37 |
| MSNP16 | MG101 | 3 | 118083714 | 65.6 | 0.49 | 0.38 |
| MSNP20 | MG135 | 4 | 20077010 | 51.6 | 0.39 | 0.36 |
| MSNP22 | MG148 | 4 | 128874466 | 46.9 | 0.33 | 0.35 |
| MSNP25 | MG175 | 5 | 13402375 | 53.6 | 0.45 | 0.37 |
| MSNP30 | MG207 | 5 | 204879476 | 53.7 | 0.39 | 0.36 |
| MSNP31 | MG215 | 6 | 39822979 | 62.4 | 0.50 | 0.38 |
| MSNP36 | MG240 | 6 | 141476300 | 51.1 | 0.43 | 0.37 |
| MSNP38 | MG270 | 7 | 126677146 | 47.5 | 0.33 | 0.35 |
| MSNP42 | MG279 | 7 | 155821714 | 50.3 | 0.42 | 0.37 |
| MSNP45 | MG293 | 8 | 20978845 | 51.4 | 0.45 | 0.37 |
| MSNP48 | MG311 | 8 | 118299376 | 56.5 | 0.36 | 0.35 |
| MSNP52 | MG347 | 9 | 104695670 | 60.4 | 0.42 | 0.37 |
| MSNP53 | MG349 | 9 | 127197714 | 61.5 | 0.50 | 0.38 |
| MSNP56 | MG361 | 10 | 39960289 | 57.6 | 0.48 | 0.38 |
| MSNP58 | MG369 | 10 | 109396730 | 53.3 | 0.50 | 0.38 |
新窗口打开|下载CSV
2.2 适于玉米杂交种纯度鉴定核心引物的特征分析
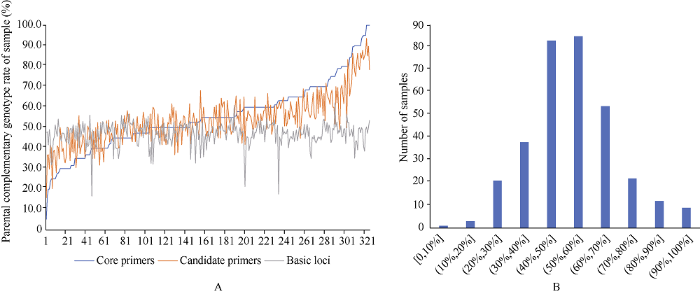
由于在进行纯度引物筛选时, 我们更加关注双亲互补引物, 因此在纯度鉴定核心引物筛选过程中双亲互补率是重要指标之一。筛选的20个核心引物的双亲互补率介于46.9%~65.6%之间, 平均双亲互补率为55.2%, 相比较于基础位点和候选位点的平均双亲互补率46.4%和53.8%, 核心引物的平均双亲互补率有所提高。83.6%的样品双亲互补基因型比率在核心引物上高于基础位点(图2-A)。核心引物MAF值介于0.33~0.50之间, 平均值为0.43, PIC值介于0.35~0.38之间, 平均值为0.37, 与基础位点相比均有所提高。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2样品双亲互补基因型信息统计图
A为样品双亲互补基因型比率比较图, 横坐标为按照核心引物的样品双亲互补基因型比率从低到高的样品排序, 纵坐标为样品双亲互补基因型比率; B为样品双亲互补基因型比率柱状统计图, 柱状图表示样品双亲互补基因型比率每增加10%区间的样品数。
Fig. 2Statistical chart of the parental complementary genotype information of samples
A is the comparison chart of parental complementary genotype rate of sample between three loci set, of which the abscissa is the parental complementary genotype rate of sample sorted from low to high according to the core primes and the ordinate is the parental complementary genotype rate of sample; B is the histogram of the parental complementary genotype rate of sample, of which the histogram shows the number of samples for each 10% rise in parental complementary.
在335份杂交种样品中, 99.7%样品的双亲互补基因型比率超过10%, 多个双亲互补引物可供筛选用于纯度鉴定(图2-B)。综合各项参数说明, 20个纯度鉴定核心引物均明显优于基础位点和候选位点, 也证明这20个核心引物能够有效应用于目前国内市场绝大部分玉米杂交种的纯度鉴定。
2.3 玉米SNP核心引物纯度鉴定实例及高通量检测方案的建立
使用SNP纯度鉴定核心引物对待测样品京科968进行纯度鉴定, 通过SNP-DNA指纹数据库查询在20个核心引物中有15个显示为双亲互补型, 从中随机选择MG072、MG135、MG175和MG215等4个引物进行纯度鉴定。试验放置的母本和父本自交系分型均与预期一致, 空白对照未检测到明显增加的荧光信号, 说明各引物扩增正常, 本次试验结果可信(图3)。在检测的110个个体中, 综合4个引物的分型结果判定, 共检出1个自交苗和2个异型株, 纯度为97.3%。根据国家标准规定, 玉米单交种的纯度不低于96.0%, 试验结果表明该供试样品纯度符合标准。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3待测样品京科968纯度鉴定分型图(快速提取法, 引物MG072)
图中每1个点代
Fig. 3Genotyping of purity identification of Jingke 968 (fast extraction method, primer MG072)
Each point represents the result of single seed, of which blue and red points represent two homozygous genotype, green points represent parental complementary genotype, black points represent non-template control and triangle points represent parental inbred lines samples.
在实际纯度检测过程中, 纯度待测样品情况是比较复杂的, 如是否提供样品信息、是否为批量样品、是否存在样品一致性问题等, 因此纯度检测试验设计不能使用固定模式。基于玉米纯度鉴定核心引物对批量样品纯度检测, 建立高通量纯度检测方案。当样品信息已知, 可通过玉米SNP-DNA指纹数据库查询双亲互补引物, 优先选择纯度核心引物进行检测。当样品信息未知, 可以选择纯度核心引物快速建立批量样品指纹, 并找到双亲互补引物进行检测。其中双亲互补引物用于快速准确鉴定自交苗, 对于遗传不稳定的位点, 由于其仍处于分离状态, 不应计入样品纯度统计。在试验流程上, 通过快速DNA提取法、高通量扩增、自动化扫描读取数据和数据库管理系统批量计算样品纯度等环节的有机结合, 共同实现批量样品高通量纯度检测。
3 讨论
3.1 KASP技术应用于玉米纯度鉴定的优势
玉米杂交种纯度鉴定应达到快捷、准确、高通量和低成本的要求。本研究在检测方法中选择适于高通量样品检测平台KASP技术结合快速DNA提取法, 在技术方案上具有以下优点。第一, 试验步骤快速简便, 易实现高通量自动化。KASP技术是目前应用SNP检测技术中试验步骤最简单的方法之一, 仅需要DNA提取、PCR扩增和数据采集3个环节。由于纯度样品需要对每个单株样品进行检测, 常规的DNA提取方法会造成时间和试验成本增加, 使用快速DNA提取法是纯度快速检测的前提。整体试验操作结合自动化工作站、高通量水浴和孔板荧光扫描仪等仪器能大幅提高检测效率, 目前最高配置的SNPLine平台每轮试验检测的最高通量超过8万个数据点, 如使用Arraytape平台可进一步提高检测通量。数据采集与统计利用软件自动化分析, 进一步提高数据采集的效率, 也减少人工读取数据误差的可能。当纯度检测样品量超过一定范围, 水浴PCR系统具有明显优势。假设有100份样品集中检测纯度, 每个样品检测100个单株, KASP平台使用4个位点检测, 总数据点为4万个, 仅需要一轮扩增即可完成, 扩增后进行全部荧光扫描时间约1 h; 使用荧光毛细管平台检测, 需选择适合的引物组合多重扩增并使用384 PCR仪扩增, 使用96通道毛细管电泳仪扫描, 需要25块PCR板和100块电泳板, 每块电泳板按照30 min扫描完成, 需要50 h即可完成电泳检测。与荧光毛细管平台相比, KASP平台通过使用高通量仪器大大缩短了扩增和检测时间; 通过自动化数据分型读取, 减少了数据人工分析时间。
第二, 试验对于DNA质量容忍度高, 取样部位限制少。DNA质量是影响筛选标记的是否好用的重要因素, 标记分型结果进一步影响自动化采集数据的效率。使用快速DNA提取法尤其考验测试位点对DNA质量的容忍度, 主要原因包括DNA拷贝数不如常规方法多, 上清液中含有杂质或PCR抑制剂, 获得的DNA链有可能存在降解断裂等[19,20,21]。在本研究测试的位点中, 有84.2%的位点在CTAB法和粗提法中均能够具有清晰稳定的3个基因型, 说明KASP技术大部分引物对于核酸质量及数量要求很低。原因主要是KASP上下游引物被设计在紧贴着SNP变异位点的几个碱基对范围内, 整体扩增产物为100 bp左右, 而且KASP引物在上游引物的5'端连接通用尾巴序列, 在扩增中与体系中的互补序列结合增加荧光基团, 在扩增循环中形成“完美扩增”。因此, 当DNA降解断裂等情况发生时, 与长目标片段相比, 这种设计使目标片段正常扩增的可能性增加。“完美扩增”使得模板DNA加入的拷贝数很少, 也可实现扩增。使用本文测试的快速DNA提取方法, 对于待测样品的取样部位要求限制少, 已经测试的部位, 包括玉米籽粒、幼苗及田间叶片等, 均取得了良好、稳定的分型效果。在实际应用中可以在种子生产和田间鉴定等多个场景中进行纯度检测。
第三, 检测仪器兼容性高, 检测成本低。使用KASP技术不仅可以选择高通量检测仪器, 还可以跟据常规检测样品通量和实验室已有仪器灵活选择适宜的仪器。扩增及荧光扫描仪器均选择广泛, 扩增仪器不仅可以使用水浴PCR仪, 还可以使用金属浴PCR仪, 也可以使用平板PCR仪等。荧光扫描既可以使用实时荧光定量PCR仪终点法读数, 也可以使用能够检测FAM和HEX/VIC荧光通道的酶标仪等。同时此类仪器维护成本也较低, 与电泳设备相比, 免去冲洗、换胶等周期性维护工作。KASP体系单个反应的检测成本低于1元, 具体成本取决于所选择的反应体系、检测样品量和检测试剂耗材等。当检测样本量增加时, 通过减少反应体积, 增加单位面积的检测数据量, 使检测成本大幅下降[11]。
3.2 兼容多平台的SNP位点的转化和应用
基于SNP标记对于亲本与子代差异的可识别性、不受环境影响特性以及试验结果可靠性。尽管玉米基因组中存在大量SNP位点, 但可供选择具有多态性、稳定和兼容多平台的SNP位点仍然只占少数[22], 因此位点筛选中各个评价指标非常重要, 决定了所选标记是否具有大规模应用的潜力。随着生物技术和生物信息学技术的不断发展, 挖掘转化SNP变异位点的方法包括从公共数据库中挖掘已有SNP位点并进行验证, 在不同平台间转化已有SNP位点以及通过重测序技术挖掘新的SNP位点等[16,18]。本文采用将已有商业化芯片SNP位点转化为KASP引物的策略, 并在KASP平台进一步筛选评估获得纯度核心引物。其中, 多态性和双亲互补率参数、是否为单拷贝和是否紧密分型等已通过芯片平台验证, 开发KASP标记主要考虑引物双亲互补率、是否与预期结果一致以及试验分型效果等信息。这种策略在第二阶段筛选过程中避免了稀有变异、多拷贝、单拷贝和偏分离等问题, 位点转化成功率较高, 多平台间的结果可以有效相互验证, 保证了标记分型结果的准确性。基于SNP多平台兼容性的特点, 我们可以更加灵活的制定检测方案, 将具有不同特点的平台结合使用—使用位点高通量的芯片平台构建样品SNP指纹数据库, 通过转化等位基因命名建立共享样品指纹数据库, 并获得其纯度核心引物指纹图谱。在数据库中查询确定双亲互补引物进行纯度鉴定, 利用KASP平台建立样本高通量的快速检测方案。其中, 纯度SNP核心引物应包含于真实性SNP核心引物, 既具有真实性核心引物的高区分能力, 同时可以保证建库指纹中包含了纯度核心引物, 可以同步采集纯度核心引物指纹数据。通过有机结合多个检测平台, 发挥不同技术平台的优势, 建立合理的检测方案。
3.3 农作物品种纯度检测技术发展展望
农作物品种纯度对于农作物生产具有重要的意义, 纯度检验主要包括2种类型和对象, 生产企业对于品种纯度摸底能够及时掌握不同批次种子纯度, 利于企业质量内控; 而政府抽检能监控市场流通种子质量, 利于政府宏观指导。因此, 纯度检测业务具有多元性和兼容性的特点, 在长期状态下应有多种方案并存, 可根据实际情况选择具体检测方案。从方案选择来看, 传统的田间鉴定能够有效观察品种在不同生长时期的表型变化; 蛋白检测具有检测流程简便、成本低特点; 分子标记检测能够有效识别自交苗, 不受生产季节的影响, 区分能力更高[23]。从分子标记类型来看, 二态性标记由于易实现数据采集和整合, 能够大幅提高检测效率、降低检测成本[24], 而多态性标记双亲互补率较高, 更容易获得双亲互补引物。进而引申到检测平台的选择上, 以荧光颜色为基础的二态性检测平台具有大幅增加样品检测能力的可能性, 因此更适合批量样品; 以长度多态为基础的电泳检测平台可以将引物组合成多重反应也能提高检测效率, 降低检测成本。随着检测技术的不断发展, 未来准确、稳定、简便和快捷的纯度鉴定方案将不断涌现并广泛服务于种业, 为政府监管和企业提供了更多纯度鉴定方案的选择。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 3]
[本文引用: 3]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
