 ,*, 许海霞
,*, 许海霞 ,*河南农业大学 / 省部共建小麦玉米作物学国家重点实验室 / 河南省粮食作物协同创新中心, 河南郑州450046
,*河南农业大学 / 省部共建小麦玉米作物学国家重点实验室 / 河南省粮食作物协同创新中心, 河南郑州450046Genome-wide association study for main agronomic traits in common wheat
ZHAI Jun-Peng, LI Hai-Xia, BI Hui-Hui, ZHOU Si-Yuan, LUO Xiao-Yan, CHEN Shu-Lin, CHENG Xi-Yong ,*, XU Hai-Xia
,*, XU Hai-Xia ,*Henan Agricultural University / National Key Laboratory of Wheat and Maize Crop Science / Collaborative Innovation Center of Henan Grain Crops, Zhengzhou 450046, Henan, China
,*Henan Agricultural University / National Key Laboratory of Wheat and Maize Crop Science / Collaborative Innovation Center of Henan Grain Crops, Zhengzhou 450046, Henan, China通讯作者:
第一联系人:
收稿日期:2019-01-6接受日期:2019-04-15网络出版日期:2019-05-14
| 基金资助: |
Received:2019-01-6Accepted:2019-04-15Online:2019-05-14
| Fund supported: |

摘要
关键词:
Abstract
Keywords:
PDF (1319KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
翟俊鹏, 李海霞, 毕惠惠, 周思远, 罗肖艳, 陈树林, 程西永, 许海霞. 普通小麦主要农艺性状的全基因组关联分析[J]. 作物学报, 2019, 45(10): 1488-1502. doi:10.3724/SP.J.1006.2019.91002
ZHAI Jun-Peng, LI Hai-Xia, BI Hui-Hui, ZHOU Si-Yuan, LUO Xiao-Yan, CHEN Shu-Lin, CHENG Xi-Yong, XU Hai-Xia.
小麦是中国主要粮食作物之一, 其产量占全国粮食总产量的22%左右, 小麦生产对中国粮食安全具有重要意义。小麦产量构成因子包括穗数、穗粒数和千粒重[1,2], 此外, 株高[3]、穗下节长和穗长[4]、粒宽[5]、可育小穗数[6]等其他农艺性状对小麦产量形成也有重要作用。育种工作者经过长期的品种改良, 培育出了一大批广适多抗的高产品种[7], 但传统育种方法盲目性大, 效率较低, 导致品种同质化严重[8]。分子标记辅助选择(marker-assisted selection, MAS)有助于提高选择效率和可预测性, 进而加速育种进程[9]。
小麦多数性状是多基因控制的数量性状[10,11], 受基因和环境的共同调控。QTL定位和全基因组关联分析(genome wide association study, GWAS)是研究数量性状的主要方法, Ramya等[12]利用复合区间作图法, 检测到25个与小麦籽粒性状有关的QTL, 单个位点的表型贡献率为4.15%~15.53%。Ren等[13]在两种灌溉条件下对小麦株高性状进行研究, 共发现17个QTL, 其中12个在灌溉条件下被检测到, 3个在干旱条件下被检测到, 仅有2个在2种环境中被重复检测到, 说明基因的表达受环境条件的影响较大。张坤普等[14]在5D染色体上Xwmc215和Xgdm63区间发现与穗粒数、总小穗数、小穗着生密度和可育小穗数相关的QTL, 遗传效应值分别为11.67%、13.83%、12.26%和10.22%。GWAS亦称关联作图(association mapping), 是以连锁不平衡(linkage disequilibrium, LD)为基础, 以自然群体为研究对象, 用于鉴定基因和定位基因的分析方法, 现已广泛应用于小麦[15]、玉米[16]、大豆[17]、花生[18]、水稻[19]等农作物。传统的QTL定位精度较低, 只能将目标基因定位在5~20 cM之间, 相较于双亲连锁分析, 全基因组关联分析考虑了更加复杂的遗传背景[20], 在解析小麦数量性状遗传机制方面的应用越来越广泛[21]。朱玉磊等[22]利用181对SSR标记与264份小麦材料进行穗发芽抗性的 GWAS 研究, 发现8个重复关联的标记。Wang等[23]对105份小麦材料进行GWAS分析, 共检测到6个与穗数相关联的位点, 分布在1D(1)、3B(2)、4A(1)和4B(2)染色体上, 表型贡献率为12.18%~16.01%。Mwadzingeni等[24]以93个普通小麦为研究对象, 在干旱胁迫和非胁迫的环境条件下对部分农艺性状进行GWAS分析, 检测到20个与穗长相关联的标记, 9个与穗粒数显著关联的标记。Liu等[25]运用GLM和MLM两种模型对小麦23个农艺性状进行GWAS, 发现了25个与重要农艺性状相关的候选基因。
目前利用SSR标记对小麦农艺性状进行关联分析的研究较多, 与SSR标记相比, SNP标记在基因组中分布更广, 数量更多, 定位精度更高, 是目前最具潜力的分子标记[26]。本研究以150份小麦品种(系)构成的自然群体为材料, 利用均匀分布于小麦21条染色体上的35,542个SNP标记, 对穗下节长、穗粒数、千粒重、株高、穗长、可育小穗数、抽穗期、每平方米穗数、粒宽共9个农艺性状进行全基因组关联分析, 筛选优势等位变异, 发掘相关候选基因, 为基因克隆和分子标记辅助选择提供理论依据。
1 材料与方法
1.1 试验材料与表型鉴定
选取150份小麦品种(系)作为自然群体(表1), 其中含黄淮南片国家小麦区域试验品种(系) 35份, 河南省小麦区域试验品种(系) 13份, 河南省小麦联合体品种比较试验品种(系) 30份, 本课题组选育出的综合性状表现优异的高代品系72份, 该群体一定程度上反映了黄淮南片麦区高产育种目标。2016—2017年度将参试材料分别种植于河南鹿邑华冠种业选种基地(E1: 17-鹿邑)、河南农业大学郑州科教园区(E2: 17-郑州); 2017—2018年度分别种植于河南农业大学原阳科教园区(E3: 18-原阳)、河南农业大学郑州科教园区(E4: 18-郑州)。采用随机区组设计, 每份材料种植6行, 行长2 m, 行距20 cm, 2次重复, 基本苗18~22万株 hm-2, 机器条播。在试验田四周设置保护行, 试验田土壤肥力、灌溉等田间管理基本一致。Table 1
表1
表1供试小麦材料
Table 1
| 类别Type | 品种(系) Variety (line) |
|---|---|
| 国家黄淮南片区域试验 Regional trail of wheat varieties in the south of Yellow and Huai Valley | 中麦170 Zhongmai 170, 周麦32 Zhoumai 32, 新麦32 Xinmai 32, 锦绣21 Jinxiu 21, 商麦167 Shangmai167, 许科168 Xuke168, 中新16 Zhongxin 16, 洛麦26 Luomai 26, 豫丰11 Yufeng 11, 中金13 Zhongjin 13, 郑育麦16 Zhengyumai 16, 郑麦618 Zhengmai 618, 龙科1221 Longke 1221, 淮核12013 Huaihe 12013, 涡麦66 Womai 66, 郑麦369 Zhengmai 369, 俊达109 Junda109, 众麦7号 Zhongmai 7, 新科麦169 Xinkemai 169, 周麦36 Zhoumai 36, 中育1211 Zhongyu 1211, 先麦12 Xianmai 12, 濮麦6311 Pumai 6311, 瑞华1426 Ruihua 1426, 高麦6号 Gaomai 6, 皖科06725 Wanke 06725, 光泰68 Guangtai 68, 轮选66 Lunxuan 66, 西农511 Xinong 511, 濉1216 Sui 1216, 豫农186 Yunong 186, 华成863 Huacheng 863, 鑫农518 Xinnong 518, 藁优5766 Gaoyou 5766, 瑞华055 Ruihua 055 |
| 河南省区域试验 Regional trial of wheat varieties in Henan Province | 藁麦5218 Gaomai 5218, 辉麦5号 Huimai 5, 西农364 Xinong 364, 中麦255 Zhongmai 255, 漯麦163 Luomai 163, 怀川11 Huaichuan 11, 创星8号 Chuangxing 8, 郑麦158 Zhengmai 158, 中麦578 Zhongmai 578, 丰德存21 Fengdecun 21, 周麦18 Zhoumai 18, 豫农804 Yunong 804 |
| 小麦联合体品种比较 试验 Comparative trail of wheat varieties in Henan Province | 华冠001 Huaguan 001, 河科大163 Hekeda 163, 华冠002 Huaguan 002, 平麦19 Pingmai 19, 华冠003 Huaguan 003, 平麦20 Pingmai 20, 华冠004 Huaguan 004, 豫农216 Yunong 216, 华冠005 Huaguan 005, 许优958 Xuyou 958, 滑昌麦68 Huachangmai 68, 滑昌麦66 Huachangmai 66, 滑昌麦26 Huachangmai 26, 豫农605 Yunong 605, 岭育麦216 Lingyumai 216, 豫农606 Yunong 606, 尚农4号 Shangnong 4, 豫农607 Yunong 607, 许麦8号 Xumai 8, 豫农805 Yunong 805, 许麦9号Xumai 9, 豫农806 Yunong 806, 许麦168 Xumai 168, 许麦169 Xumai 169, 农科668 Nongke 668, 豫农519 Yunong 519, 尚农3号 Shangnong 3, 河科大LYZB Hekeda LYZB, 河科大522 Hekeda 522, 豫农517 Yunong 517 |
| 高代品系 Breeding lines | 豫农225 Yunong 225, 豫农243 Yunong 243, 豫农7847 Yunong 7847, 豫农8981 Yunong 8981, 豫农227 Yunong 227, 豫农246 Yunong 246, 豫农955 Yunong 955, 豫农9005 Yunong 9005, 豫农231, Yunong 231, 豫农248 Yunong 248, 豫农7961 Yunong 7961, 豫农9053 Yunong 9053, 豫农233 Yunong 233, 豫农256 Yunong 256, 豫农8819 Yunong 8819, 豫农9068 Yunong 9068, 豫农235 Yunong 235, 豫农268 Yunong 268, 豫农8858 Yunong 8858, 豫农9071 Yunong 9071, 豫农236 Yunong 236, 豫农282 Yunong 282, 豫农8861 Yunong 8861, 豫农9092 Yunong 9092, 豫农238 Yunong 238, 豫农285 Yunong 285, 豫农8909 Yunong 8909, 豫农9107 Yunong 9107, 豫农240 Yunong 240, 豫农7838 Yunong 7838, 豫农8954 Yunong 8954, 豫农9110 Yunong 9110, 豫农9305 Yunong 9305, 豫农8051 Yunong 8051, 豫农8138 Yunong 8138, 豫农8636 Yunong 8636, 豫农9308 Yunong 9308, 豫农8054 Yunong 8054, 豫农8141 Yunong 8141, 豫农8639 Yunong 8639, 豫农9311 Yunong 9311, 豫农8057 Yunong 8057, 豫农8339 Yunong 8339, 豫农8681 Yunong 8681, 豫农9334 Yunong 9334, 豫农8069 Yunong 8069, 豫农8552 Yunong 8552, 豫农8684 Yunong 8684, 豫农9338 Yunong 9338, 豫农8072 Yunong 8072, 豫农8570 Yunong 8570, 豫农8774 Yunong 8774, 豫农8042 Yunong 8042, 豫农8075 Yunong 8075, 豫农8588 Yunong 8588, 豫农8792 Yunong 8792, 豫农8045 Yunong 8045, 豫农8129 Yunong 8129, 豫农8600 Yunong 8600, 豫农8804 Yunong 8804, 豫农8048 Yunong 8048, 豫农8135 Yunong 8135, 豫农8621 Yunong 8621, 豫农8813 Yunong 8813, 豫农9113 Yunong 9113, 豫农9119 Yunong 9119, 豫农9125 Yunong 9125, 豫农9116, Yunong 9116, 豫农9122 Yunong 9112, 豫农9132 Yunong 9132, 豫农223 Yunong 223, 豫农9266 Yunong 9266, 豫农9269 Yunong 9269 |
新窗口打开|下载CSV
试验小区中50%以上个体抽出1/3穗(不含芒)时定为抽穗期, 灌浆后期从每个小区随机选取10株测量穗下节长、株高、穗长和可育小穗数, 蜡熟后期调查每平方米穗数, 每个材料取30穗脱粒, 调查穗粒数和千粒重, 采用Perten SKCS 4100单粒谷物特性分析仪测定粒宽。
1.2 表型数据的统计分析
利用SPSS 22.0软件对表型数据进行正态性检验, 利用QTL IciMapping软件进行描述性统计分析和方差分析[27], 广义遗传力H2=σ2g/[σ2g+(σ2gL/L)+ (σ2e/Lr)], 其中σ2g为基因型方差, σ2e为残差方差, σ2gL为基因与环境互作方差, L为地点数, r为重复数。1.3 DNA提取及SNP分型
小麦生长至三叶期时取10株幼苗, 将叶片混合后利用改良的CTAB法提取DNA[28], 用1.0%琼脂糖凝胶电泳检验DNA质量和浓度。利用博奥晶典生物技术有限公司与中国科学院遗传与发育生物学研究所联合开发的小麦基因组35K SNP芯片对参试材料进行基因分型, 共检测到覆盖小麦全基因组的35,143个SNP标记, Allen等[29]经适应性评估发现, 这套芯片非常适合于六倍体小麦的基因分型。采用Tassel 5.0软件对标记信息去冗余, 剔除最小等位基因频率(minor allele frequency, MAF)小于5 %和缺失率(miss rate)大于25%的SNP, 最终得到9552个多态性高、稳定性好的优质SNP标记用于后续关联分析。多态性信息量(polymorphism information content): PIC = 1-∑P2ij (其中Pij表示第i位点的第j个等位变异出现的频率)[30]。1.4 群体结构与主成分分析
利用Structure 2.3.4进行群体结构分析, 将最佳群体数目K设置为1~12, 每个K值运算5次, 开始时的不作数迭代(length of burnin period)设置为10,000, 不作数迭代后的MCMC (number of MCMC reps after burnin)设置为100,000, 依据Evanno等[31]的方法选取最大ΔK对应的K值作为最佳亚群数目。利用Tassel 5.0 软件对参试材料进行基于SNP芯片数据的主成分分析。1.5 连锁不平衡分析
利用Tassel 5.0软件进行连锁不平衡分析, 设置LD衰减窗口大小为100, 即计算每个标记前后100对标记之间的连锁不平衡程度, 当标记间P≤0.001时, 认为存在显著的LD[32]。1.6 关联分析与候选基因筛选
利用GLM (general linear model)一般线性模型(Q、PCA)和MLM (mixed linear model)混合线性模型(K、PCA+K、Q+K)对各性状进行模型修正, 采用RStudio 1.1.453 (https://www.rstudio.com/)软件包lme4中lmer函数[33]计算每个性状的最佳线性无偏预测值(best linear unbiased prediction, BLUP), 去除环境效应, 依据 BLUP值的关联结果作出5种关联模型的Quantile-Quantile图(Q-Q Plot), 考虑到实际-lg (P)值与预期-lg (P)值的偏差, 以实际-lg (P)值最接近-lg (P)期望值的模型作为性状的最优模型[34], 基于最优模型分别对各性状在4个环境中的表型值和BLUP值进行GWAS, 以BLUP值的关联结果进行各性状的曼哈顿图(Manhattan Plot)作图。由RStudio 1.1.453软件完成Q-Q Plot、Manhattan Plot作图。
以LD距离作为候选基因的预测区间, 将多个环境中稳定出现的SNP标记的延伸序列在小麦TGACv1.0数据库(http://plants.ensembl.org/hmmer/ index.html)和NCBI网站(https://www.ncbi.nlm.nih. gov/)进行Blast比对, 对候选位点进行功能注释。
2 结果与分析
2.1 农艺性状表型数据的统计分析
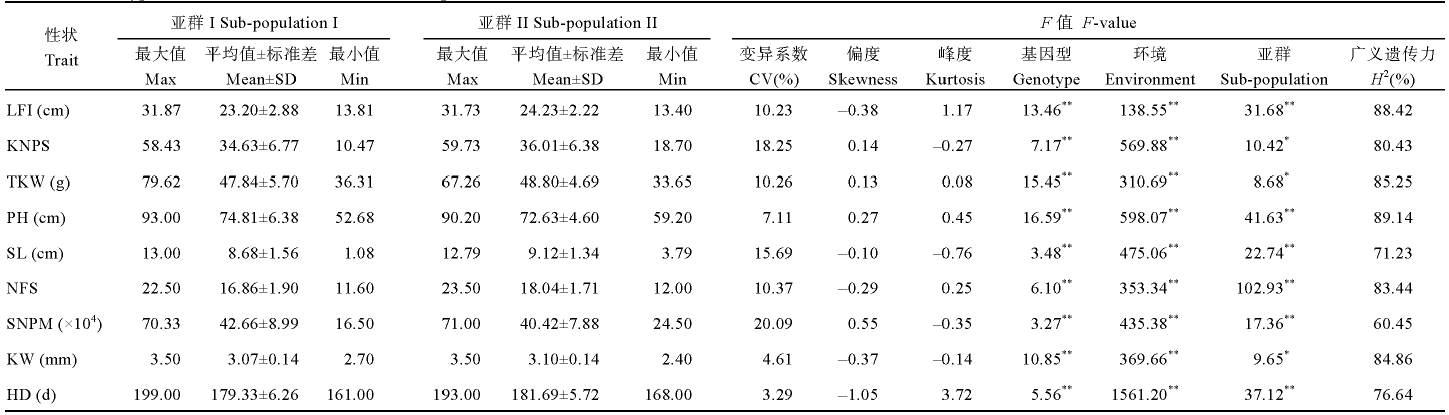
从表2可以看出, 9个农艺性状的广义遗传力均高于60%, 说明这些性状的表型主要受基因调控, 受环境影响较小。方差分析显示所有性状在基因型间和亚群间均存在显著或极显著差异, 表明研究的性状在该群体中具有丰富的遗传变异, 但不同性状的变异程度具有明显差异, 其中每平方米穗数(20.09%)和穗粒数(18.25%)的变异系数较大, 变异程度较高; 抽穗期(3.29%)和粒宽(4.61%)的变异系数较小, 变异程度较低。正态性检验结果表明, 除抽穗期外, 其他性状的峰度和偏度均接近0, 基本符合正态分布, 说明目标性状大部分是多基因控制的数量性状。Table 2
表2
表2小麦农艺性状表型变异及方差分析
Table 2
 |
新窗口打开|下载CSV
2.2 SNP标记分析
全基因组分析中PIC的变异范围为0.0950~ 0.5000, 平均值为0.3215, 其中3B染色体最高(0.3729), 4D染色体最低(0.2590), 3个染色体组中PIC表现为A (0.3317)>B (0.3240)>D (0.3100) (表3)。95.38% (9111/9552)的SNP标记具有物理位置信息, A、B、D染色体组分别检测到2747、3708和2656个SNP标记, 分别占总标记数的28.76%、38.82%和27.80%。全基因组最小等位基因频率 MAF 平均值为0.2329, 变异范围为0.0500~0.5000, 49.11%标记的MAF大于0.2, 16.25% 标记的MAF大于0.4, 物理图谱长度为14,541.4 Mb, 标记密度为1.70 Mb/marker。Table 3
表3
表3SNP标记信息
Table 3
| 染色体 Chr. | 标记数目 No. of markers | 物理长度Physical distance (Mb) | 最小等位基因频率 MAF | 多态性信息量 PIC | 标记密度 Density of marker (Mb marker-1) | ||
|---|---|---|---|---|---|---|---|
| Mean | Range | Mean | Range | ||||
| 1A | 450 | 593.3 | 0.2583 | 0.0500-0.5000 | 0.3404 | 0.0950-0.5000 | 1.32 |
| 1B | 717 | 689.0 | 0.1852 | 0.0526-0.4932 | 0.2843 | 0.0997-0.4999 | 0.96 |
| 1D | 635 | 495.2 | 0.1999 | 0.0504-0.5000 | 0.2955 | 0.0956-0.5000 | 0.78 |
| 2A | 517 | 780.7 | 0.2429 | 0.0500-0.5000 | 0.3275 | 0.0950-0.5000 | 1.51 |
| 2B | 635 | 801.2 | 0.2597 | 0.0500-0.5000 | 0.3450 | 0.0950-0.5000 | 1.26 |
| 2D | 557 | 651.4 | 0.226 | 0.0507-0.5000 | 0.3120 | 0.0963-0.5000 | 1.17 |
| 3A | 318 | 749.5 | 0.2472 | 0.0504-0.4966 | 0.3377 | 0.0958-0.5000 | 2.36 |
| 3B | 565 | 829.3 | 0.2870 | 0.0504-0.5000 | 0.3729 | 0.0956-0.5000 | 1.47 |
| 3D | 339 | 614.0 | 0.2583 | 0.0504-0.5000 | 0.3448 | 0.0956-0.5000 | 1.81 |
| 4A | 283 | 744.5 | 0.2114 | 0.0537-0.4966 | 0.3024 | 0.1016-0.5000 | 2.63 |
| 4B | 316 | 673.2 | 0.1716 | 0.0500-0.4966 | 0.2663 | 0.0950-0.5000 | 2.13 |
| 4D | 132 | 509.3 | 0.1676 | 0.0537-0.4823 | 0.2590 | 0.1016-0.4994 | 3.86 |
| 5A | 451 | 708.2 | 0.2338 | 0.0500-0.5000 | 0.3239 | 0.0950-0.5000 | 1.57 |
| 5B | 545 | 712.9 | 0.2384 | 0.0500-0.5000 | 0.3206 | 0.0950-0.5000 | 1.31 |
| 5D | 393 | 565.9 | 0.2269 | 0.0500-0.4966 | 0.3083 | 0.0950-0.5000 | 1.44 |
| 6A | 310 | 617.7 | 0.2449 | 0.0509-0.5000 | 0.3340 | 0.0965-0.5000 | 1.99 |
| 6B | 523 | 720.5 | 0.2265 | 0.0507-0.5000 | 0.3172 | 0.0963-0.5000 | 1.38 |
| 6D | 286 | 473.4 | 0.2041 | 0.0504-0.4932 | 0.2950 | 0.0956-0.4999 | 1.66 |
| 7A | 418 | 736.5 | 0.2696 | 0.0522-0.5000 | 0.3561 | 0.0989-0.5000 | 1.76 |
| 7B | 407 | 750.1 | 0.2737 | 0.0504-0.4966 | 0.3616 | 0.0958-0.5000 | 1.84 |
| 7D | 314 | 636.8 | 0.2649 | 0.0500-0.5000 | 0.3556 | 0.0950-0.5000 | 2.03 |
| Unmap | 441 | 488.8 | 0.2262 | 0.0507-0.5000 | 0.3133 | 0.0963-0.5000 | 1.11 |
| A | 2747 | 4930.4 | 0.2440 | 0.0500-0.5000 | 0.3317 | 0.0950-0.5000 | 1.79 |
| B | 3708 | 5176.2 | 0.2346 | 0.0500-0.5000 | 0.3240 | 0.0950-0.5000 | 1.40 |
| D | 2656 | 3946.0 | 0.2211 | 0.0500-0.5000 | 0.3100 | 0.0950-0.5000 | 1.49 |
| Whole | 9552 | 14541.4 | 0.2329 | 0.0500-0.5000 | 0.3215 | 0.0950-0.5000 | 1.70 |
新窗口打开|下载CSV
2.3 群体结构分析和主成分分析
利用Structure 2.3.4软件对群体遗传结构进行分析, 当K=2时, ΔK值最大(图1-A), 由图1-B可知, 参试材料被划分为2个亚群(sub-population), 其中亚群I (Sub-Pop I)包含39份材料, 亚群II (Sub-Pop II)包含111份材料; 基于基因型的PCA分析显示整个群体可分为2个群组, 分类结果与Structure 结果高度吻合(图1-C)。方差分析发现, 亚群I中品种(系)的穗下节长、穗粒数、千粒重、穗长、可育小穗数、粒宽和抽穗期均显著或极显著低于亚群II, 而株高和每平方米穗数极显著高于亚群II (图1-D和表2), 说明2个亚群在表型性状和遗传背景上均有较大差异。图1
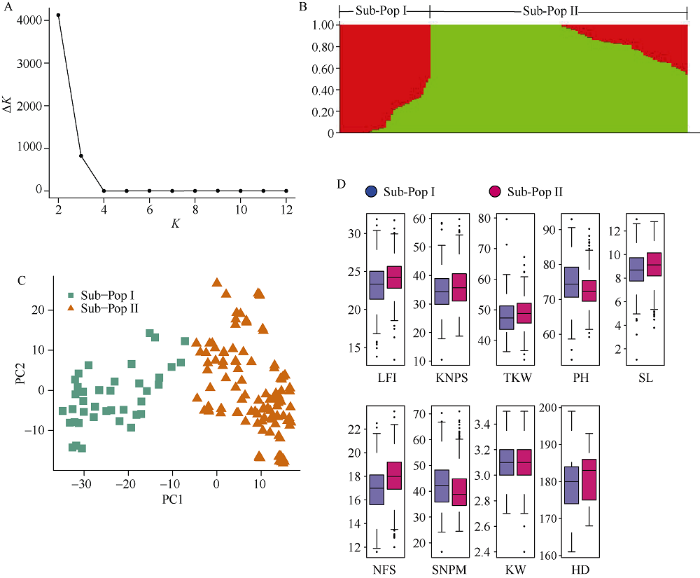 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1群体结构、主成分分析和亚群表型
1-A: ΔK折线图; 1-B: 群体结构; 1-C: PCA分析; 1-D: 亚群表型; 各性状详细名称见
Fig. 1Structure, PCA, and Sub-population phenotype
1-A: line chart of ΔK; 1-B: structure plot; 1-C: analysis of PCA; 1-D: sub-population phenotype. Name of each trait is given in
2.4 连锁不平衡分析
考虑连锁群(linkage group)间连锁不平衡背景的影响, 对连锁群间的r2值进行平方根转换, 以大于此分布95%的参数值为阈值, 用来截取同一连锁群内LD的衰减距离[35], A基因组、B基因组、D基因组和全基因组的r2阈值分别为0.2846、0.3162、0.2763和0.2817, 从连锁不平衡的衰减图可以看出, A基因组、B基因组、D基因组和全基因组的LD距离分别为4.7、8、11和6 Mb (图2)。依据全基因组的LD衰减距离, 将多环境中稳定出现的SNP标记在物理图谱上前后6 Mb的区间认定为一个候选位点, 推测可能有控制相关性状的QTL存在。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2连锁不平衡衰减图
Fig. 2Linkage disequilibrium plot
2.5 最优模型选择与全基因组关联分析
以Q-Q plot中实际-lg (P)值最接近-lg (P)期望值为判断标准, 确定各性状的最优关联模型, 从图3可以看出, 穗粒数、株高、可育小穗数和粒宽的最优关联模型是Q+K模型, 千粒重和抽穗期的最优关联模型为PCA+K模型, 穗下节长、穗长和每平方穗数的最优关联模型为K模型。图3
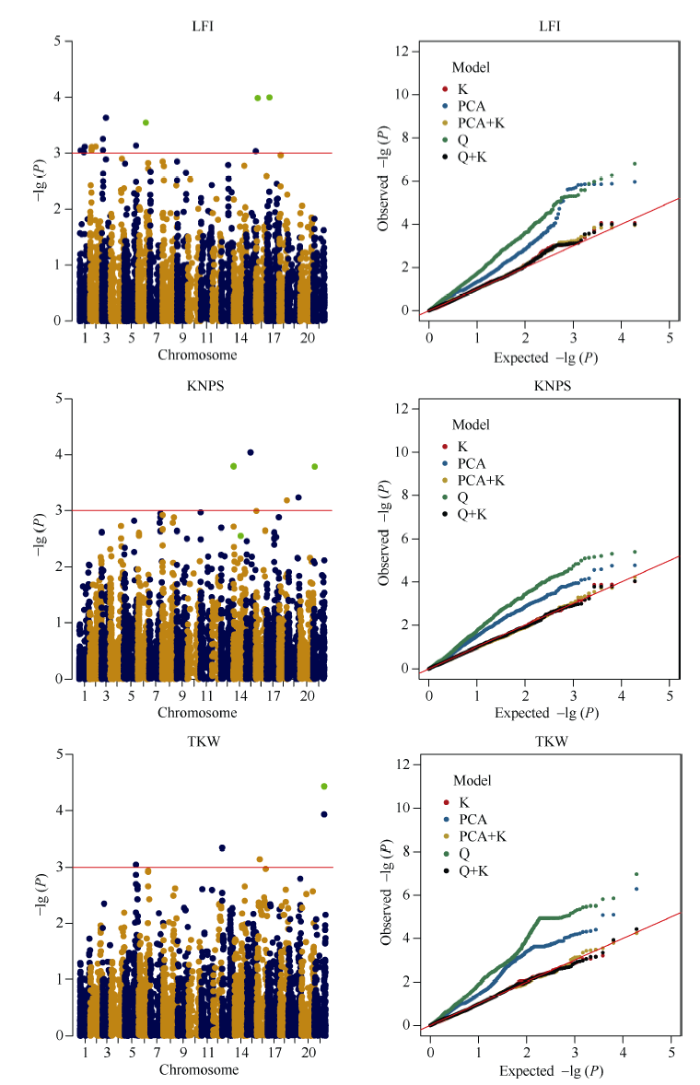 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3各性状的曼哈顿图和Q-Q图(部分列出)
各性状详细名称见
Fig. 3Manhattan plot and Q-Q plot of each trait (part of the plots)
Name of each trait is given in
针对不同性状选用其最优关联模型进行GWAS分析, 共检测到652个显著SNP位点(P≤0.001), 其中21个SNP在2个或2个以上的环境中被检测到, 属于稳定的SNP标记(表4)。检测到与穗下节长稳定关联的标记4个, 分别位于2D(1)、6A(1)和6B(2)染色体上, 其中AX-94532508、AX-94976370和AX-94918690在3个环境中被重复检测到, AX- 95108722在2个环境中被重复检测到, 可解释7.68%~10.51%的表型变异。检测到与穗粒数稳定关联的标记 4个, 位于5B(3)和7D(1)染色体上, 均在2个环境中被重复检测到, R2范围为7.67%~10.97%。7D染色体上的AX-95152265和1A染色体上的AX-95094324分别与千粒重和株高稳定关联, 均能在3个环境中稳定遗传, 分别解释9.16%~10.84%、8.18%~12.41%的表型变异。检测到与穗长稳定关联的标记2个, 位于2D(1)和3A(1)染色体上, 均在2个环境中被重复检测到, R2范围为9.89%~14.27%。检测到与可育小穗数相关联的稳定标记2个, 位于5B染色体上, 均在3个环境中被重复检测到, R2范围为7.69%~11.50%。检测到与抽穗期相关联的稳定标记10个, 分别位于1B(4)、2A(3)、5A(1)和7D(1)染色体上, AX-95197628、AX-95073002、AX-94430515、AX-94690816、AX-94402434和AX-95629896在2个环境中被重复检测到, AX-94748775、AX-94629503、AX-94446915和AX-94639471在3个环境中被重复检测到, 可解释8.07%~18.79%的表型变异, 其中AX-95629896在物理图谱上没有位置信息。没有检测到与每平方米穗数和粒宽稳定关联的SNP。
Table 4
表4
表4小麦农艺性状稳定关联的位点信息
Table 4
| 性状 Trait | 标记 SNP | 染色体 Chr. | 物理位置 Position | P-value | R2 (%) | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| E1 | E2 | E3 | E4 | E1 | E2 | E3 | E4 | |||||||||||||||||||
| LFI | AX-95108722 | 2D | 441569084 | 7.84E-04 | 9.49E-04 | 7.94 | 7.68 | |||||||||||||||||||
| AX-94532508 | 6A | 99932394 | 8.44E-04 | 1.35E-04 | 3.40E-04 | 7.92 | 10.51 | 9.24 | ||||||||||||||||||
| 性状 Trait | 标记 SNP | 染色体 Chr. | 物理位置 Position | P-value | R2 (%) | |||||||||||||||||||||
| E1 | E2 | E3 | E4 | E1 | E2 | E3 | E4 | |||||||||||||||||||
| AX-94976370 | 6B | 157777682 | 4.85E-04 | 8.44E-04 | 7.02E-04 | 8.60 | 7.86 | 8.09 | ||||||||||||||||||
| AX-94918690 | 6B | 157777813 | 4.85E-04 | 8.44E-04 | 7.02E-04 | 8.60 | 7.86 | 8.09 | ||||||||||||||||||
| KNPS | AX-94385515 | 5B | 10444933 | 2.53E-04 | 3.75E-04 | 9.58 | 9.18 | |||||||||||||||||||
| AX-94459753 | 5B | 10449146 | 2.55E-04 | 3.59E-04 | 9.55 | 9.08 | ||||||||||||||||||||
| AX-94799632 | 5B | 418015877 | 2.39E-04 | 9.94E-04 | 9.64 | 7.67 | ||||||||||||||||||||
| AX-94547362 | 7D | 81887538 | 1.78E-04 | 9.38E-05 | 10.06 | 10.97 | ||||||||||||||||||||
| TKW | AX-95152265 | 7D | 556123927 | 3.73E-04 | 1.08E-04 | 3.39E-04 | 9.17 | 10.84 | 9.16 | |||||||||||||||||
| PH | AX-95094324 | 1A | 519471561 | 8.88E-05 | 5.87E-05 | 8.53E-04 | 11.92 | 12.41 | 8.18 | |||||||||||||||||
| SL | AX-95108722 | 2D | 441569084 | 7.79E-06 | 2.18E-05 | 14.27 | 12.94 | |||||||||||||||||||
| AX-94542697 | 3A | 11350680 | 1.54E-04 | 6.10E-04 | 11.55 | 9.89 | ||||||||||||||||||||
| NFS | AX-94385515 | 5B | 10444933 | 5.60E-05 | 9.30E-04 | 7.39E-05 | 11.50 | 7.70 | 11.13 | |||||||||||||||||
| AX-94459753 | 5B | 10449146 | 5.38E-05 | 9.13E-04 | 7.46E-05 | 11.35 | 7.69 | 11.11 | ||||||||||||||||||
| HD | AX-95197628 | 1B | 652298582 | 3.34E-05 | 3.43E-04 | 12.40 | 9.13 | |||||||||||||||||||
| AX-95073002 | 1B | 652457758 | 4.36E-05 | 5.32E-04 | 12.13 | 8.62 | ||||||||||||||||||||
| AX-94748775 | 1B | 652468844 | 7.50E-04 | 3.77E-04 | 3.06E-04 | 8.40 | 9.19 | 9.50 | ||||||||||||||||||
| AX-94629503 | 1B | 652575175 | 7.24E-04 | 3.65E-04 | 3.01E-04 | 8.07 | 9.01 | 9.31 | ||||||||||||||||||
| AX-94446915 | 2A | 44094660 | 5.03E-05 | 1.15E-04 | 5.01E-07 | 11.82 | 10.63 | 18.79 | ||||||||||||||||||
| AX-94430515 | 2A | 45085276 | 3.41E-04 | 3.00E-05 | 9.10 | 12.61 | ||||||||||||||||||||
| AX-94639471 | 2A | 52130032 | 5.61E-04 | 1.92E-04 | 5.13E-04 | 8.65 | 10.03 | 8.69 | ||||||||||||||||||
| AX-94690816 | 5A | 620175970 | 9.08E-04 | 2.02E-05 | 9.33 | 17.06 | ||||||||||||||||||||
| AX-94402434 | 7D | 415430669 | 6.40E-04 | 1.38E-04 | 9.38 | 10.82 | ||||||||||||||||||||
| AX-95629896 | Unknown | Unknown | 9.19E-04 | 5.79E-04 | 8.35 | 8.74 | ||||||||||||||||||||
新窗口打开|下载CSV
21个稳定关联的位点中, 位于5B上的AX- 94385515和AX-94459753同时与可育小穗数和穗粒数相关联, 位于2D上的AX-95108722同时与穗长和穗下节长相关联, 说明这3个位点为一因多效位点。
2.6 稳定遗传的SNP效应分析
对稳定出现的标记进行单标记效应分析, 共发现14个优异等位变异(表5), 其中AX-95108722-G可使穗下节长度降低1.94 cm, 在群体中出现的频率占75.84%; AX-94385515-C、AX-94459753-C和AX-94547362-A可使穗粒数增加5粒左右, 在群体中出现的频率均在93%以上; AX-94385515-C和AX-94459753-C均可使可育小穗数增加1.25个, 在群体中所占比例为93.24%和93.29%。Table 5
表5
表5稳定关联的SNP及其等位变异的表型效应
Table 5
| 性状 Trait | 标记 SNP | 染色体 Chr. | 物理位置 Position | 等位变异 Allele | 表型差值Difference | 材料数 No. of varieties |
|---|---|---|---|---|---|---|
| LFI | AX-95108722 | 2D | 441569084 | T | 1.94 | 36 |
| G | 0 | 113 | ||||
| AX-94532508 | 6A | 99932394 | C | 2.79 | 133 | |
| T | 0 | 13 | ||||
| AX-94976370 | 6B | 157777682 | C | 2.58 | 133 | |
| T | 0 | 16 | ||||
| AX-94918690 | 6B | 157777813 | G | 2.58 | 133 | |
| A | 0 | 16 | ||||
| KNPS | AX-94385515 | 5B | 10444933 | C | 4.78 | 138 |
| T | 0 | 10 | ||||
| AX-94459753 | 5B | 10449146 | C | 4.78 | 139 | |
| T | 0 | 10 | ||||
| AX-94799632 | 5B | 418015877 | C | 3.22 | 17 | |
| T | 0 | 132 | ||||
| AX-94547362 | 7D | 81887538 | A | 5 | 139 | |
| G | 0 | 10 | ||||
| TKW | AX-95152265 | 7D | 556123927 | C | 4.91 | 12 |
| A | 0 | 135 | ||||
| PH | AX-95094324 | 1A | 519471561 | A | 3.42 | 96 |
| C | 0 | 46 | ||||
| SL | AX-95108722 | 2D | 441569084 | T | 0.63 | 36 |
| G | 0 | 113 | ||||
| AX-94542697 | 3A | 11350680 | C | 0.69 | 17 | |
| G | 0 | 104 | ||||
| NFS | AX-94385515 | 5B | 10444933 | C | 1.25 | 138 |
| T | 0 | 10 | ||||
| AX-94459753 | 5B | 10449146 | C | 1.25 | 139 | |
| T | 0 | 10 | ||||
| HD | AX-95197628 | 1B | 652298582 | T | 3.69 | 139 |
| C | 0 | 10 | ||||
| AX-95073002 | 1B | 652457758 | A | 3.71 | 136 | |
| G | 0 | 9 | ||||
| AX-94748775 | 1B | 652468844 | G | 2.64 | 131 | |
| A | 0 | 17 | ||||
| AX-94629503 | 1B | 652575175 | C | 2.65 | 131 | |
| A | 0 | 18 | ||||
| AX-94446915 | 2A | 44094660 | A | 3.34 | 139 | |
| G | 0 | 10 | ||||
| AX-94430515 | 2A | 45085276 | A | 1.88 | 37 | |
| C | 0 | 111 | ||||
| AX-94639471 | 2A | 52130032 | G | 3.19 | 136 | |
| A | 0 | 9 | ||||
| AX-94690816 | 5A | 620175970 | A | 3.95 | 7 | |
| G | 0 | 110 | ||||
| 性状 Trait | 标记 SNP | 染色体 Chr. | 物理位置 Position | 等位变异 Allele | 表型差值Difference | 材料数 No. of varieties |
| AX-94402434 | 7D | 415430669 | G | 2.22 | 129 | |
| A | 0 | 15 | ||||
| AX-95629896 | Unknown | Unknown | G | 2.88 | 135 | |
| A | 0 | 9 |
新窗口打开|下载CSV
检测到8个优异等位变异在供试群体中所占比例较低, 其中AX-94532508-T、AX-94976370-T和AX-94918690-A 分别使穗下节长度降低2.79、2.58和2.58 cm, 在群体中所占比例分别为8.90%、10.74%和10.74%; AX-95108722-T和AX-94542697-C 分别使穗长增加0.63 cm和0.69 cm, 在群体中占比24.16%和14.05%; 另外AX-94799632-C、AX-95152265-C、AX-95094324-C分别增加穗粒数3.22个、提高千粒重4.91 g、降低株高3.42 cm, 在群体中出现的频率分别为11.41%、8.16%和32.39%。
检测到与抽穗期相关联的等位变异10个, 其中AX-95197628-T、AX-95073002-A、AX-94748775-G、AX-94629503-C、AX-94446915-A、AX-94639471-G、AX-94402434-G和AX-95629896-G分别延长抽穗期3.69、3.71、2.64、2.65、3.34、3.19、2.22和2.88 d, 在群体中占比分别为93.29%、93.79%、88.51%、87.92%、93.29%、93.79%、89.58%和93.75%。AX-94430515-C和AX-94690816-G分别使抽穗期提前1.88 d和3.95 d, 在群体中出现的频率为75.00%和94.02%。
2.7 候选基因分析
利用21个稳定表达的SNP的延伸序列在 TGACv1.0数据库和NCBI网站中Blast, 共发现10个候选位点, 14个候选基因, 其中7个候选基因有功能注释(表6)。其中TraesCS5B02G010800编码7TM区域的钙依赖通道蛋白(Calcium-dependent channel), TraesCS5B02G237200编码NB-ARC结构域的植物抗性蛋白, TraesCS7D02G129700编码MATE蛋白, TraesCS7D02G436800编码植物生长素响应因子(Auxin response factor), TraesCS1B02G426300编码Rab GTPase 11家族蛋白, TraesCS2A02G092200编码植物损伤诱导蛋白WI12, TraesCS5A02G438800编码一种谷氨酰胺转移酶II类蛋白(Glutamine amidotransferases class-II (GATase), 另外7个候选基因的功能未知。Table 6
表6
表6小麦农艺性状候选基因及其功能注释
Table 6
| 性状 Trait | 候选位点 Candidate locus | 标记 SNP | 物理位置 Chr. | Position | 基因 Gene | 基因注释或编码蛋白 Gene annotation or coding protein |
|---|---|---|---|---|---|
| KNPS | 5B|4444933..16449146 | AX-94385515 | 5B|10444933 | TraesCS5B02G010800 | Calcium-dependent channel, 7TM region, putative phosphate |
| AX-94459753 | 5B|10449146 | TraesCS5B02G010800 | Calcium-dependent channel, 7TM region, putative phosphate | ||
| 5B|412015877..424015877 | AX-94799632 | 5B|418015877 | TraesCS5B02G237200 | NB-ARC domain | |
| 7D|75887538..87887538 | AX-94547362 | 7D|81887538 | TraesCS7D02G129700 | MATE_eukaryotic | |
| TKW | 7D|550123927..562123927 | AX-95152265 | 7D|556123927 | TraesCS7D02G436800 | Auxin response factor |
| SL | 3A|5350680..17350680 | AX-94542697 | 3A|11350680 | TraesCS3A02G017800 | Unknown |
| KNPS/NFS | 5B|4444933..16449146 | AX-94385515 | 5B|10444933 | TraesCS5B02G010800 | Calcium-dependent channel, 7TM region, putative phosphate |
| AX-94459753 | 5B|10449146 | TraesCS5B02G010800 | Calcium-dependent channel, 7TM region, putative phosphate | ||
| HD | 1B|646298582..658575175 | AX-95197628 | 1B|652298582 | TraesCS1B02G426000 | Unknown |
| AX-95073002 | 1B|652457758 | TraesCS1B02G426300 | Rab GTPase family 11 (Rab11)-like includes Rab11a, Rab11b, and Rab25 | ||
| AX-94748775 | 1B|652468844 | TraesCS1B02G426600 | Unknown | ||
| AX-94629503 | 1B|652575175 | TraesCS1B02G427100 | Unknown | ||
| 2A|38094660..51085276 | AX-94446915 | 2A|44094660 | TraesCS2A02G091700 | Unknown | |
| AX-94430515 | 2A|45085276 | TraesCS2A02G092200 | Wound-induced protein WI12 | ||
| 2A|46130032..58130032 | AX-94639471 | 2A|52130032 | TraesCS2A02G099200 | Unknown | |
| 5A|614175970..626175970 | AX-94690816 | 5A|620175970 | TraesCS5A02G438800 | Glutamine amidotransferases class-II (GATase). | |
| 7D|409430669..421430669 | AX-94402434 | 7D|415430669 | TraesCS7D02G325100 | Unknown |
新窗口打开|下载CSV
3 讨论
3.1 模型修正与关联分析
Yu等[36]提出的将亲缘关系作为协变量纳入分析的混合线性模型被证明是一种有效的关联模型, 即使是在较小的群体中也能表现出显著的关联能力, 与一般线性模型相比, 减少了假阳性的概率, 但是盲目使用 Q+K 模型可能会导致群体结构的过度修正, 出现假阴性的错误[23,36], 故有必要针对不同的性状进行不同模型的比较。本研究中穗粒数、株高、可育小穗数和粒宽适用Q+K模型, 千粒重和抽穗期适用 PCA+K模型, 穗下节长、穗长和每平方穗数适用 K 模型。3.2 小麦农艺性状的GWAS研究
小麦基因组重复序列多, 基因组庞大, 即使厘摩范围内也包含上百万个碱基对[37], 随着高通量测序技术的发展, 高密度SNP分型芯片为小麦复杂农艺性状的研究提供了更多的变异信息。本研究利用35K SNP芯片对小麦9个主要农艺性状进行GWAS分析, 检测到4个与穗下节长稳定关联的SNP, 位于2D(1)、6A(1)和6B(2)染色体上。Turuspekov等[21]和Jaiswal等[38]分别在2D和6B染色体上发现了与穗下节长显著关联的位点, 而位于6A染色体上的AX-94532508未见报道, 可能是一个新的位点。Jaiswal等[38]在5B染色体长臂上发现与穗粒数关联的位点(114 cM), 与本研究检测到的位点AX- 94799632距离较近。Shi等[39]在7D染色体上发现4个与穗粒数关联的位点(D_GDS7LZN02H6ID8_55、CAP7_c1383_548、Kukri_c35508_426、D_GA8KES 401AVKPJ_56), 其中CAP7_c1383_548与本研究发现的位点AX-94547362在物理图谱上仅距离3 Mb。Zanke等[40]在7D染色体上发现2个与千粒重关联的位点, 其中Kukri_c15768_68与本研究发现的AX-95152265距离5.9 Mb。在1A染色体长臂上检测到1个与株高稳定关联的SNP。Sukumaran等[20,41]和Jaiswal等[38]分别在相似位置检测到与株高关联的位点(116~117 cM, 126 cM)。检测到与穗长稳定关联的2个SNP分别位于2D和3A上。Mwadzingeni等[24]在相同染色体上也检测到穗长的关联位点。检测到2个与可育小穗数稳定关联的SNP, 位于5B上。Turuspekov等[21]在5B染色体上也检测到与可育小穗数关联的位点, 但与本研究发现的位点距离较远。检测到10个与抽穗期稳定关联的SNP, 其中9个具有物理位置信息, 位于1B(4)、2A(3)、5A(1)和7D(1)染色体上。Ain等[42]、Sukumaran等[20]、Mwadzingeni等[24]、Jaiswal等[38]和Zanke等[43]分别在1B、5A和7D染色体上也检测到与抽穗期关联的位点, 其中Sukumaran等[20]发现的位点Kukri_ c9411_676与本研究发现的AX-94690816距离较近。已知控制小麦光周期的基因Ppd-A1位于2AS上, 本研究也在2A染色体短臂上发现3个与抽穗期稳定关联的SNP, 其中AX-94446915与Ppd-A1相距7.16 Mb, 可能与Ppd-A1有一定联系。3.3 等位变异及其应用
本研究共发现14个优异等位变异, 其中6个在供试群体中所占比例相对较高, 8个在供试群体中所占比例较低, 例如增加穗粒数的等位变异AX- 94799632-C, 提高千粒重的等位变异AX-95152265- C, 这些等位变异效应较大, 可能与不利基因紧密连锁, 也可能是新的优异等位变异, 育种工作者可以结合分子标记在早期世代选择中加以利用。发现10个与抽穗期相关的等位变异, 其中8个延长了抽穗期, 2个推迟了抽穗期。与其他性状不同, 抽穗期等位变异的应用受地区环境的影响, 在适宜春性和弱春性品种种植的地区, 注重对抽穗较早的材料保留选择, 而在适宜冬性和半冬性品种种植的地区, 应优先选择抽穗较晚的材料。对于春季冻害较重的地区, 抽穗过早的品种更易受到春季冻害的影响, 而春季冻害较轻的地区, 抽穗早的品种在后期籽粒灌浆方面更占优势[44]。
3.4 候选基因分析
对21个稳定表达的SNP的延伸序列在 TGACv1.0数据库和NCBI网站中Blast, 其中7个有功能注释, 位于5B染色体上穗粒数和可育小穗数的候选基因 TraesCS5B02G010800 编码一种7TM区域的钙依赖通道蛋白(calcium-dependent channel)。Pei等[45]发现植物中Ca2+通道的激活会介导脱落酸的合成, 小麦籽粒灌浆后期, 干物质增多, 结合水含量上升, 自由水含量减少, 代谢减弱, 脱落酸开始增多, 推测 TraesCS5B02G010800可能参与植物中脱落酸的合成, 从而影响籽粒的结实和成熟。位于5B染色体上穗粒数的候选基因 TraesCS5B02 G237200编码NB-ARC结构域的植物抗性蛋白, Van等[46]发现NB-ARC结构域由NB、ARC1和ARC2三个结构域组成, 其结合状态可调节植物抗性蛋白的活性, 在植物中参与病原体的识别和先天免疫反应的后续激活, 位于7D染色体上穗粒数的候选基因TraesCS7D02G129700编码一种MATE蛋白, 基因注释显示MATE是植物中多基因家族, 其与植物抗病性和渗透胁迫下铁离子的稳定有关, 2个候选基因与植物的抗性和免疫反应有关, 推测其通过提高小麦抵御胁迫的能力来增加穗粒数。位于7D染色体上千粒重的候选基因 TraesCS7D02G436800编码植物生长素响应因子(Auxin response factor), 推测其通过调节植物中生长素的响应机制来影响粒重。位于1B染色体上抽穗期的候选基因TraesCS1B02 G426300编码Rab GTPase 11家族蛋白, 参与膜转运调节。一些RAB11成员在植物特有的发育过程中发挥作用, 包括细胞分裂和囊泡运输[47,48], 但大多数的RAB11成员的分子和生理功能仍不清楚。Asaoka等[49]在拟南芥中对 RAB11 亚群进行了亚细胞定位和动力学研究, 发现其家族成员RABA1在拟南芥中介导跨高尔基体和质膜之间的运输, 在盐胁迫耐受性上起关键作用。位于2A染色体上抽穗期的候选基因 TraesCS2A02G092200, 编码植物损伤诱导蛋白WI12, 基因注释显示它优先在细胞壁中积累, 研究表明它对植物细胞壁有增强作用[50]。位于5A染色体上抽穗期的候选基因 TraesCS5A02G438800 编码一种谷氨酰胺转移酶II类蛋白[Glutamine amidotransferases class-II (GATase)], 苏延萍等[51]通过拟南芥突变体试验和转录本验证, 发现编码此类蛋白的基因GATL12在拟南芥叶绿体发育中发挥重要作用, 推测 TraesCS5A02G438800 通过参与小麦中叶绿体的发育来调节植株的光感反应, 进而影响小麦的抽穗期。4 结论
共检测到21个与小麦主要农艺性状稳定关联的SNP标记, 有3个SNP与多个性状相关联, 8个优势等位变异在供试材料中所占比例较低。筛选出10个候选位点, 包含14个可能与小麦农艺性状相关的候选基因, 其中3个可能在植物抵御生物与非生物胁迫过程中起作用, 2个可能与植物激素的合成和响应有关, 1个可能与植物细胞壁的增强有关, 1个可能参与叶绿体的发育, 另外7个功能未知。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
