 , 叶兴国
, 叶兴国
* 通讯作者(Corresponding authors): 佘茂云, E-mail: ahxiaoshe@126.com; 叶兴国, E-mail: yexingguo@caas.cn;** 同等贡献(Contributed equally to this work) 收稿日期:2016-03-25 接受日期:2016-09-18网络出版日期:2016-09-28基金:
摘要
关键词:小麦; TaVIP1; 烟草; 农杆菌转化
Cloning, Molecular Characterization, and Functional Analysis of Wheat TaVIP1 Genes
ZHAO Pei
 , YE Xing-Guo
, YE Xing-Guo
Fund:
Abstract
Keyword:Wheat; TaVIP1; Tobacco; Agrobacterium-mediated transformation
Show Figures
Show Figures









相比传统杂交育种, 农杆菌介导的基因工程育种具有育种目标明确、育种周期短、成本低等优势。转基因技术已逐步成为玉米、大豆等作物的重要育种手段之一, 然而在小麦上极低的转化效率限制了该技术的应用, 亟待鉴定、挖掘与小麦转化效率相关的基因或蛋白, 利用基因工程手段提高小麦转化效率, 促进小麦转基因技术开展[1, 2, 3, 4, 5]。
在农杆菌介导的植物遗传转化过程中, 由农杆菌Ti质粒编码的毒性基因(virulence, vir)发挥了重要作用。迄今为止, 已发现至少有20种Vir蛋白参与转移DNA (transferred DNA, T-DNA)的形成和宿主基因组整合过程[6]。其中, VirE2是一种单链DNA结合蛋白, 通过与T-DNA单链非共价结合, 保护后者免于细胞内核酸酶降解, 同时引导其进入宿主基因组[7]。VIP1 (VirE2-interacting protein 1)是一种与VirE2蛋白特异互作的植物蛋白, 在VirE2与importinα 蛋白互作中起桥梁作用, 同时利用自身的核定位信号引导VirE2的核输入, 在酵母和动物中不存在其同系物[8, 9]。Tzfira等[10]以VirE2作为诱饵蛋白, 利用酵母双杂交的方法首次在拟南芥中筛选到与VirE2互作的VIP1蛋白, 序列分析发现AtVIP1含有亮氨酸拉链(basic-zipper protein, bZIP)结构域, 具有核定位(nuclear localization signal, NLS)功能。Djamei等[11]进一步研究证实, 拟南芥AtVIP1蛋白的第79位丝氨酸残基磷酸化与否决定了AtVIP1的核定位功能。Lacroix等[12]在植物细胞活体内发现了DNA-VirE2-VIP1复合物的存在, 证实了VIP1引导VirE2进入细胞核并与染色体上的核小体互作, 同时, 此复合物进一步与其他毒性蛋白(如VirF)等互作, 引导VirE2-VIP1降解并释放T-DNA。
Tzfira等[10, 13]研究VIP1在农杆菌介导的烟草转化中的作用时发现, 不论是瞬时转染还是稳定转化, 表达AtVIP1反义cDNA的烟草植株都表现出对农杆菌侵染的抗性, 而过表达拟南芥AtVIP1烟草植株, 则表现出较高的农杆菌转化效率。此外, Li等[14]研究发现, 部分突变的AtVIP1仅降低转基因拟南芥稳定转化效率, 但对瞬时侵染没有影响。
目前, 关于VIP1基因的研究主要集中在拟南芥和烟草等几个模式植物中, 在小麦中还没有该基因的研究报道。本研究根据拟南芥AtVIP1编码序列, 利用in silico技术克隆小麦TaVIP1基因家族, 通过对该基因家族的分子特性及在烟草转化中的功能初步分析, 为解析农杆菌介导的小麦低转化效率奠定理论基础。
1 材料与方法1.1 植物材料及过表达和亚细胞定位载体普通小麦(AABBDD, 2n = 42)品系CB037由中国农业科学院作物科学研究所陈孝研究员选育, 本课题组保存。普通小麦品系Luivo、植物表达载体pZP211和农杆菌菌株C58C1由美国内布拉斯加大学Tom Clemente教授提供。烟草品系NC89由中国农业科学院生物技术研究所裴新梧研究员提供。小麦二倍体野生材料PI10474 (Triticum monococcum, Tm)、PI330486 (Aegilops longissima, Ael)和TD38 (Aegilops tauschii, Aet), 四倍体小麦Langdon (AABB), Langdon与中国春D组染色体整套代换系材料1D(1A)、2D(2A)、3D(3A)、4D(4A)、5D(5A)、6D(6A)、7D(7A)和1D(1B)、2D(2B)、3D(3B)、4D(4B)、5D(5B)、6D(6B)、7D(7B) [15], 以及亚细胞定位载体p16318-GFP均由本课题组保存, 过表达载体pWMB006由本课题组构建。
1.2 基因组DNA、总RNA提取和cDNA合成取饱满无虫蛀的小麦种子, 经70%乙醇表面消毒1 min和20%次氯酸钠溶液灭菌15 min, 无菌水冲洗4次后, 置无菌水浸润的滤纸上, 室温暗培养至萌芽。随后移入营养钵, 20 d后取嫩叶用于RNA提取。采用RNAprep Pure Plant Kit (Qiagen, Germany), 参照说明书提取总RNA, 并用DNase I (Sigma D5025)清除残留的痕量基因组DNA, 然后按照PrimeScript 1st Strand cDNA Synthesis Kit (TaKaRa, 大连)操作说明, 以Oligo dT为引物合成cDNA第一链。同时, 利用CTAB法[16]提取普通小麦CB037和Luivo的基因组DNA, 经RNaseA处理并纯化后, 于-20℃保存备用。
1.3 小麦TaVIP1基因家族序列检索及克隆 以拟南芥AtVIP1的氨基酸序列(GenBank登录号为NM_103495)为种子序列, 采用tblastn比对方法, 在NCBI (http://www.ncbi.nlm.nih.gov/)及IWGSC (International Wheat Genome Sequencing Consortium)数据库(http://www.wheatgenome.org/)检索小麦全基因组shotgun数据, 参数设置为默认。根据得到的候选全长小麦VIP1基因序列, 利用Primer Premier 5.0软件(PREMIER BiosoftInternational, USA)设计全长引物(F: 5′ -CGCCATGGACCCTCGCTTC-3′ ; R: 5′ -AGG TTGCCCAGAGACTCCTCACAT-3′ )。分别以小麦gDNA和cDNA作为模板, 利用KOD高保真聚合酶(KOD+ Neo, TOYOBO, Japan)进行PCR扩增。扩增程序为95℃预变性5 min; 94℃变性45 s, 60℃退火1 min, 68℃延伸3 min, 35个循环; 68℃延伸10 min。扩增产物连入Trans-T simple载体(北京全式金生物技术有限公司), 并测序确认。
1.4 目标基因Southern blot检测将纯化的小麦gDNA, 分别用限制性内切酶EcoR V、Sac I和Dra I完全酶切。以TaVIP1基因cDNA全长作为探针, 利用地高辛标记Southern blotting试剂盒(Roche), 参照说明书进行Southern blot检测, 分析TaVIP1基因在普通小麦中的拷贝数。
1.5 目标基因染色体定位根据小麦VIP1基因序列设计特异扩增引物, 其中A基因组(F: 5′ -GAGGACTCACCATTTTGTTATC A-3′ ; R: 5′ -CACATTAGTTCCTCTCCTGCCA-3′ )、B基因组(F: 5′ -GGGCAGGGAGACAGTAACAGA-3′ ; R: 5′ -ACATGGTCGACCCTCAGATG-3′ )和D基因组特异引物(F: 5′ -ATTATGTTGAAAGCTTCCTTGCT ACA-3′ ; R: 5′ -CACATTAGTTCCTCTCCTGCTG-3′ )的扩增片段长度分别为727、514和618 bp; 同时以四倍体小麦Langdon及其与中国春D组染色体14个代换系为模板进行PCR扩增。按TOYOBO产品说明, 采用step down步骤, 以提高扩增特异性。扩增产物经1%琼脂糖凝胶电泳后照相, 记录VIP1基因染色体定位结果。利用A或B基因组特异引物扩增没有特异带出现时, 说明目标基因所在的染色体被D组的部分同源染色体代换, 即VIP1基因位于被代换的染色体上; 利用D基因组特异引物扩增出特异带时, 说明目标基因所在的D组染色体代换了Langdon中A组或B组的部分同源染色体, 即VIP1等位基因位于该D染色体上。
1.6 亚细胞定位利用报告基因GFP (green fluorescent protein)对目的基因进行亚细胞定位。根据目的基因cDNA序列设计基因融合表达引物(TaVIP1-GFP-F: 5′ -TATCT CTAGAGGATCCATGGACCCTCGCTTC-3′ ; TaVIP1- GFP-R: 5′ -TGCTCACCATGGATCCCATGAAATCCA TGGAGTCG-3′ ; 下画线为酶切位点), 利用同源重组(TaKaRa, 大连)将目的基因片段连接到p16318- GFP载体上, 命名为p16318-TaVIP1-GFP。采用Bio-Rad PDS1000/He基因枪(Bio-Rad, USA)无菌转化洋葱表皮细胞, 黑暗培养12~16 h, 以激光共聚焦显微镜(Zeiss LSM 700, Germany)观察绿色荧光信号, 检测波长为488 nm。
1.7 植物VIP1家族基因检索和聚类分析 根据拟南芥VIP1基因序列, 采用Blastp方法在NCBI (http://www.ncbi.nlm.nih.gov/)及Phytozomev11 (http://www.phytozome.net/)数据库中检索植物VIP1基因, 由于部分物种测序不完整, 本研究利用in silico技术进行全长序列拼接, 并利用softberry (http://www.softberry.com/)网站的FGENESH/ FGENESH+对序列编码区进行预测。
采用在线ClustalW2 (http://www.ebi.ac.uk/Tools/ clustalw2/index.html)对检索到的氨基酸序列进行同源比对, 经手动调整后, 利用Mega 6.0软件包中Neighbor-Joining法构建系统发育树, 对构建的进化树进行bootstraps检验, 设bootstrap参数为1000。
1.8 TaVIP1家族蛋白生化特性分析采用BioEdit软件(http://www.mbio.ncsu.edu/ bioedit/bioedit.html)手工编辑比对TaVIP1家族蛋白功能域, 利用WebLogo (http://weblogo.threeplusone. com/create.cgi)分析残基保守性。蛋白质bZIP结构域及核定位信号预测软件分别为2ZIP (http://www. expasy.org/)和PSORTII (http://www.genscript.com/ cgi-bin/tools/ psort2.pl)。
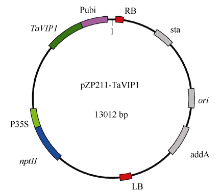
1.9 过表达载体的构建和烟草转化根据TaVIP1基因的ORF序列设计特异引物TaVIP1-BamHIF (5′ -AAAGGATCCATGGACCCTCG CTTC-3′ )和TaVIP1-SpeIR (5′ -AAAACTAGTACTC CTCACATGAAATCC-3′ ), 分别在上下游引物中引入BamH I和Spe I酶切位点, 将TaVIP1基因构建到pZP211载体上, 构建重组质粒pZP211-TaVIP1 (图1), 通过三亲杂交法将其转入农杆菌C58C1。利用携带pZP211-TaVIP1载体的农杆菌侵染预培养的烟草叶盘, 经卡那霉素筛选, 获得转基因植株。利用CTAB法从转基因烟草植株中提取基因组DNA, 利用引物TaVIP1F (5′ -CGCCATGGACCCTCGCTTC-3′ )和TaVIP1R (5′ -AGGTTGCCCAGAGACTCCTCACAT- 3′ )对转基因植株进行PCR检测。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 重组表达载体pZP211-TaVIP1结构图Fig. 1 Structure of expression vector pZP211-TaVIP1 |
2 结果与分析2.1 小麦TaVIP1基因克隆及拷贝数分析 以拟南芥AtVIP1的氨基酸编码序列(NM_ 103495)为种子序列, 采用in silico技术搜索NCBI中小麦的EST, 得到一条与之同源性较高的小麦候选EST, 登录号为AK335929, 利用NCBI中的ORF Finder程序确定起始和终止密码子的位置, 并根据其侧翼序列设计扩增全长引物, 成功从小麦品系CB037的cDNA中扩增出一条约1000 bp的片段。测序结果表明, 小麦TaVIP1基因cDNA全长987 bp, 编码328个氨基酸, 与烟草NtVIP1和拟南芥AtVIP1蛋白质序列相似性只有45%和51%。该小麦TaVIP1基因已提交NCBI数据库, 登录号为KF752431。
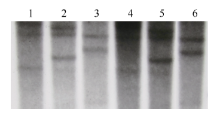
鉴于小麦属异源六倍体作物, 其基因组由3个异源二倍体构成, 理论上每个基因在小麦上应存在3个同源基因。据此, 本研究分别用限制性内切酶EcoR V、Sac I和Dra I对小麦品系CB037和Luivo的gDNA进行完全酶切, 以地高辛标记的克隆的TaVIP1基因全长序列作为探针进行Southern blot分析。结果表明, 2个小麦品系的gDNA经EcoR V和Sac I酶切后均出现清晰的3条杂交带(图2), 因此认为TaVIP1基因在普通小麦中有3个拷贝, 推测分别位于A、B、D染色体组上, 每个染色体组存在1个拷贝; 而2个小麦品系gDNA经Dra I酶切后只有2个杂交条带(图2), 推测与Dra I酶切位点有关, 存在大小相似的重叠片段。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 普通小麦基因组中TaVIP1基因Southern blot分析 1~3: 品系Luivo基因组DNA分别被EcoR V、Sac I和Dra I酶切; 4~6: 品系CB037基因组DNA分别被EcoR V、Sac I和Dra I酶切。Fig. 2 Southern blotting assay of TaVIP1 in the genome of common wheat 1-3: genomic DNA of line Luivo was digested by EcoR V, Sac I, and Dra I, respectively; 4-6: genomic DNA of line CB037 was digested by EcoR V, Sac I, and Dra I, respectively. |
2.2 小麦TaVIP1基因的染色体定位分析 Southern blot杂交已明确在小麦中存在3个TaVIP1同源基因, 进一步在IWGSC数据库, 利用Blastn在线比对工具检索到小麦3个TaVIP1高度相似序列(E值为0), 分别位于染色体4AL、4BS和4DS上, 序列识别号依次为TGACv1_scaffold_289134、TGACv1_scaffold_328709和TGACv1_scaffold_361639。利用FGENESH/FGENESH+对这3个高度相似序列编码区进行预测, 发现3个同源序列均包含4个外显子和3个内含子(图3), 与拟南芥及烟草VIP1 (NP_001312899)在基因结构上高度一致。进一步分析发现, 本研究所克隆的小麦TaVIP1基因中的一个同源基因KF752431位于4BS染色体上。序列分析表明, 普通小麦中3个TaVIP1同源基因编码蛋白相似性较高, 其中位于4AL与4DS染色体上的2个TaVIP1同源基因的相似性达99.1%, 而与4BS染色体上的TaVIP1同源基因的相似性为96.6%, 暗示4AL与4DS染色体上的TaVIP1基因可能存在功能重叠, 为此, 后续关于TaVIP1基因初步功能研究采用4BS上的同源基因。为了深入分析VIP1基因在小麦簇中的结构, 以小麦二倍体野生种为材料, 分别扩增到来源于AA、SS和DD组的VIP1基因, 3种扩增产物的序列与普通小麦相应基因组同源基因的序列没有差异, 而且其编码蛋白的大小也与普通小麦同源基因的相应蛋白一致。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 小麦簇TaVIP1基因序列结构粗竖线表示染色体定位用引物。Vertical lines show the primers for chromosome location assay.Fig. 3 Sequencing components of TaVIP1 gene in Triticeae |
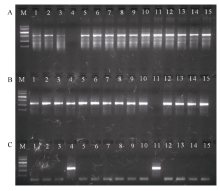
利用小麦3个TaVIP1同源基因特异引物, 对四倍体小麦Langdon及其与中国春D基因组14个代换系1D(1A)、2D(2A)、3D(3A)、4D(4A)、5D(5A)、5D(5A)、7D(7A)和1D(1B)、2D(2B)、3D(3B)、4D(4B)、5D(5B)、6D(6B)、7D(7B)进行PCR扩增。发现利用A、B基因组上TaVIP1基因的特异引物扩增时, 除4D(4A)和4D(4B)代换系外的所有材料均扩增出特异带, 说明A、B基因组中的TaVIP1等位基因分别位于4A和4B染色体上(图4-A, B); 利用D基因组TaVIP1基因的特异引物扩增时, 只有4D(4A)和4D(4B)代换系扩增出了特异带, 其他材料均没有扩增出特异带, 说明D基因组中的TaVIP1等位基因位于4D染色体上(图4-C)。根据PCR检测获得的染色体定位结果与序列数据库预测结果完全一致, 证明普通小麦中3个TaVIP1等位基因分别位于染色体4AL、4BS和4DS染色体上。
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 普通小麦中3个TaVIP1基因基于PCR扩增结果的染色体定位 A: 利用A基因组上TaVIP1同源基因特异引物的扩增结果; B: 利用B基因组上TaVIP1同源基因特异引物的扩增结果; C: 利用D基因组上TaVIP1同源基因特异引物的扩增结果。 M: DNA marker V, 自上而下分子量依次是2000、1500、1000、700、400和200 bp; 1~7: 四倍体小麦Langdon代换系1D(1A)、2D(2A)、3D(3A)、4D(4A)、5D(5A)、6D(6A)和7D(7A); 8~14: 四倍体小麦Langdon代换系 1D(1B)、2D(2B)、3D(3B)、4D(4B)、5D(5B)、6D(6B)和7D(7B); 15: Langdon。Fig. 4 Chromosome location of three homologous TaVIP1 genes in common wheat based on PCR amplification A: PCR amplification using the specific primers to the homologous TaVIP1 gene on A genome; B: PCR amplification using the specific primers to the homologous TaVIP1 gene on B genome; C: PCR amplification using the specific primers to the homologous TaVIP1 gene on D genome. M: DNA marker V. Bands from up to down are 2000, 1500, 1000, 700, 400, and 200 bp; 1-7: Langdon durum disomic substitution lines 1D(1A), 2D(2A), 3D(3A), 4D(4A), 5D(5A), 6D(6A), and 7D(7A), respectively; 8-15: Langdon durum disomic substitution lines 1D(1B), 2D(2B), 3D(3B), 4D(4B), 5D(5B), 6D(6B), and 7D(7B), respectively. |
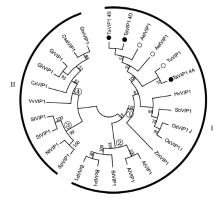
2.3 植物VIP1系统进化树构建 为了解析植物VIP1基因系统发生极其进化上的相关性, 从Phytozome v11及NCBI蛋白数据库中检索到部分植物VIP1蛋白全长序列, 结合本研究中克隆的小麦簇TaVIP1蛋白序列, 运用MEGA6.0软件进行比对并构建系统进化发育树。可以清晰看出, 进化树明显分成2个一级分支(罗马数字表示)和4 个二级分支(阿拉伯数字表示), 第I级分支包括单子叶和双子叶植物, 而第II分支主要包括双子叶植物。其中在第I级分支中, 单、双子叶植物来源的VIP1又明显形成2个独立的二级分支, 第①级分支主要包括小麦族植物, 而第②级分支则包括十字花科植物, 如拟南芥, 白菜等。茄科植物主要分布在第③级分支, 其余植物的VIP1分布在第④级分支中(图5)。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 植物VIP1蛋白序列系统发育树 罗马数字表示一级分支, 阿拉伯数字表示二级分支; 实心和空心圆分别表示本研究所克隆到的来源于六倍体小麦和二倍体小麦的TaVIP1。Fig. 5 Phylogenetic analysis of plant VIP1 protein sequences The primary and secondary branches are indicated by roman and arabic numbers, respectively. Filled and hollow circles represent the TaVIP1 proteins isolated from hexaploid and diploid wheat species, respectively. BoVIP1: Brassica oleracea, XP_013586120.1; BrVIP1: Brassica rapa, XP_009145063.1; BnVIP1: Brassica napus, XP_013706434.1; GhVIP1: Gossypium hirsutum, XP_016693775.1; CsVIP1: Citrus sinensis, XP_006472967.1; VvVIP1: Vitis vinifera, XP_010665015.1; AtVIP1: Arabidopsis thaliana, NP_564486.1; CsaVIP1: Cucumis sativus, XP_004141014.1; GrVIP1: Gossypium raimondii, XP_012446799.1; AlVIP1: Arabidopsis lyrata, XP_002891267.1; SpVIP1: Solanum pennellii, XP_015071489.1; GmVIP1: Glycine max, KRH70316.1; StVIP1: S. tuberosum, XP_006363906.1; NtVIP1: Nicotiana tabacum, NP_001312899.1; SlVIP1: S. lycopersicum, XP_004242016.1; TuVIP1: T. urartu; HvVIP1: Hordeum vulgare, BAK03813.1; ZmVIP1: Zea mays, XP_008668106.1; SbVIP1: Sorghum bicolor, XP_002441871.1; OsVIP1_J: Oryza sativa japonicaGroup, XP_015618202.1; OsVIP1_I: O. sativa indicaGroup, EAY82358.1; AetVIP1: Aegilops tauschii; AelVIP1: Ae. longissima; TaVIP1_4A: T. aestivum, chromosome 4A; TaVIP1_4B: T. aestivum, chromosome 4B; TaVIP1_4D: T. aestivum, chromosome 4D. |
2.4 植物VIP1蛋白功能保守域分析为了解析植物VIP1蛋白家族bZIP1结构及其核定位信号序列, 利用WebLogo在线分析软件, 对经2ZIP和PSORTII的预测结果进行核心区域及上下游10~15个氨基酸残基保守性分析。结果表明, 核定位信号核心氨基酸残基在所分析的植物VIP1蛋白中十分保守(图6-A); 同时, bZIP结构域中亮氨酸残基位置保守性高, 每隔6个氨基酸残基就存在一个亮氨酸(图6-B)。
图6
Fig. 6
| Figure OptionViewDownloadNew Window | |
 | 图6 植物VIP1蛋白核定位及bZIP结构域 A: 横线表示核定位结构域, 下箭头指示保守的极性氨基酸, 如赖氨酸(K)、精氨酸(R)、谷氨酸(E)。B: 横线表示bZIP结构域, 数字1~6指示保守的亮氨酸(L)残基。Fig. 6 Nuclear localization signal (NLS) and bZIP motifs of plant VIP1 proteins A: nuclear localization signal domain. Overbar shows the NLS motif and polar amino acid positions are marked with arrows, including lysine (K), arginine (R), and glutamic acid (E). B: overbar shows the bZIP domain and conserved leucine (L) residues are numbered (1-6). |
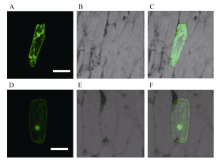
2.5 小麦TaVIP1的亚细胞定位为了明确小麦TaVIP1蛋白亚细胞定位, 根据TaVIP1基因序列设计in-fusion引物, 通过PCR扩增获得两端携带载体序列的TaVIP1基因的cDNA序列(去掉终止子), 利用同源重组的方法连接到载体p16318-GFP上, 使其与绿色荧光蛋白N端融合形成TaVIP1-GFP融合蛋白。将构建正确的融合蛋白表达载体通过基因枪轰击进入洋葱表皮细胞, 使融合蛋白瞬时表达, 以空载体p16318-GFP质粒作为对照。结果表明, 在细胞膜、细胞质和细胞核皆有GFP表达(图7-D、E和F), 而对照质粒的GFP则在整个细胞中随机分布(图7-A、B和C)。
图7
Fig. 7
| Figure OptionViewDownloadNew Window | |
 | 图7 融合蛋白TaVIP1-GFP的亚细胞定位 A~C: 转化TaVIP1-GFP融合蛋白的洋葱表皮细胞; D~F: 转化GFP的洋葱表皮细胞。A和D: 绿色荧光; B和E: 白光; C和F: 叠加。标尺=10 μ m。Fig. 7 Plasma membrane localization of TaVIP1-GFP fusion protein A-C: onion epidermal cells expressing TaVIP1-GFP fusion protein; D-F: onion epidermal cells expressing GFP protein only. A and D: green fluorescence images; B and E: bright-field images; C and F: overlap of fluorescence and bright-field images. Scale bar = 10 μ m. |
2.6 小麦TaVIP1基因初步功能研究 以烟草品系NC89幼嫩叶片为材料, 分别用携带pZP211-TaVIP1载体和pZP211空载体的农杆菌C58C1侵染其叶盘, 获得抗性再生植株。移栽到花盆, 待再生植株生长健壮时, 取少量叶片提取gDNA, 利用TaVIP1基因全长引物检测转基因阳性植株。重复3次TaVIP1基因转化烟草试验, 共侵染叶盘数47个, 但以PCR仅检测到1株TaVIP1呈阳性的转基因烟草植株, 转化率仅为2.1%, 而转化pZP211空载体对照组的转化率为35.0% (表1)。可见, 将TaVIP1基因在烟草中过表达, 对提高烟草的转化效率具有负面作用, 因此认为, 小麦TaVIP1基因可能抑制了T-DNA在植物细胞中的转运和向染色体上的整合。
表1
Table 1
表1(Table 1)
| 表1 TaVIP1基因过表达载体转化烟草的转化率 Table 1 Transformation efficiency of the vector overexpressing TaVIP1 in tobacco |
3 讨论农杆菌介导的植物遗传转化是一系列农杆菌蛋白和植物蛋白相互作用的结果[7, 17]。植物VIP1蛋白参与了T-DNA的核输入和向植物基因组上的整合, 在农杆菌的转化过程中发挥着重要作用[8, 10, 13]。鉴于小麦极低的转化效率, 本研究试图通过分析小麦TaVIP1基因的克隆和分子特性, 来解释农杆菌介导转化小麦效率较低的原因。
研究表明, 农杆菌转化拟南芥和烟草均具有较高的遗传转化效率, 而转化小麦的效率一直比较低[3]。 结合拟南芥VIP1与其遗传转化效率相关的报道[18], 我们比对分析了小麦TaVIP1与拟南芥AtVIP1及烟草VIP1基因的序列特点, 发现它们在基因结构上一致, 均由4个外显子组成(图3), 但其编码蛋白的相似性分别仅为45%和51%, 仅bZIP结构域十分保守。同时, 利用转基因的方法将小麦TaVIP1基因转入烟草, 对转化效率进行的初步分析表明, 在烟草中过表达小麦TaVIP1基因降低了烟草对农杆菌侵染的敏感性, 稳定转化率显著低于对照(表1)。推测原因可能由于过表达的TaVIP1基因导致其编码二聚化程度加剧, 干扰其介导的VirE2细胞核定位功能的发挥, 最终影响转化效率[14, 19]。Tzfira等[10, 13]研究发现, 在烟草中表达AtVIP1反义cDNA后, 受体烟草植株都表现出对农杆菌侵染的抗性, 相反, 过表达AtVIP1的烟草植株其农杆菌转化效率显著提高。最近, Shi等[19]探讨了拟南芥vip1-1突变体对农杆菌的敏感性, 发现无论瞬时转化还是稳定转化vip1-1突变体的转化率都与野生型相同。本研究结果与其类似。不同植物物种VIP1基因呈现转化作用的不一致性, 可能是因为植物种类间存在着巨大差异, 当它们用于农杆菌介导转化植物组织时, 因物种类型和植株发育时期存在很大差异, 一些基因的差异表达导致农杆菌转化效率在某些物种上明显提高, 但却在另一些物种上不表现提高效应。事实上, 我们通过小麦受体植株生理状态调节等策略, 农杆菌转化小麦部分基因型的效率已达20%左右(未发表), 证明这些基因型中TaVIP1基因可能与转化效率没有关系, 或在适宜的生理状态中TaVIP1基因表达处于有利T-DNA转化的最佳水平, 促进了转化效率的提高, 具体原因有待进一步探讨。
另外, 小麦TaVIP1基因与拟南芥AtVIP1基因在序列上存在较大差异, 在农杆菌转化过程中发挥作用的途径和位置可能不同。亚细胞定位研究表明, 小麦TaVIP1蛋白定位于细胞质和细胞核, 其中以细胞核定位为主, 表现为细胞核中荧光物质浓度较高(图7-F), 这与Tzfira等[10, 13]的报道一致。此外, 本研究发现有部分TaVIP1蛋白定位于细胞膜中, 这是现有文献中尚未报道的结果, 可能的原因是过表达TaVIP1基因引起其表达产物量异常丰富, 容易形成聚合物无法通过细胞膜, 同时, 由于亚细胞定位是以瞬时基因表达研究, 不可避免存在质粒滞留细胞间隙的现象, 从而造成定位在细胞膜的误判。在拟南芥中, 缺少氨基末端1~80个氨基酸的AtVIP1- GUS融合蛋白只定位于细胞核中[10]; 而全长AtVIP1-YFP融合蛋白定位于细胞核和细胞质中, 其中, 突变后模拟磷酸化状态的AtVIP1蛋白更趋于定位在细胞核中, 而突变后模拟非磷酸化状态的AtVIP1蛋白则定位于细胞质和细胞核中[11]。Shi等[19]发现, 突变后模拟磷酸化的AtVIP1蛋白和突变后模拟非磷酸化的AtVIP1蛋白都定位于细胞质和细胞核中, 但并没有发现磷酸化的AtVIP1更趋向于定位在细胞核的结果。不同物种的VIP1蛋白的亚细胞定位也有所不同, 主要原因是基因的序列差异, 但也可能受到启动子类型、植物材料和外界环境的影响。
农杆菌对植物的侵染和植物免疫是两个拮抗的过程, 推测VIP1调控植物免疫信号的转导过程与其协助农杆菌侵染植物的过程相互影响, 最后在不同植物中表现出不同的作用。对此, 史勇[20]提出了一个关于VirE2蛋白在农杆菌介导转化植物过程中作用的新模型, 认为T-DNA链的细胞核定位主要受农杆菌VirD2蛋白的影响。VirE2的主要作用是影响T-DNA链的形态使其更易通过核孔[17], VirE2在植物细胞中与VIP1互作调节植物防御基因的转录, VirE2与VIP1的互作使后者逗留在细胞质中。由于VIP1在一些植物中的表达量很低, 降低了VIP1激活植物防御基因的表达水平, 从而提高了转化效率, 这也部分说明过表达TaVIP1基因反而降低了烟草对农杆菌侵染的敏感性。
VIP1蛋白不仅参与农杆菌介导的植物转化, 其作为转录因子, 还调控许多下游基因的表达, 参与植物免疫信号转导路径[8]。本研究克隆的TaVIP1基因在农杆菌介导的小麦转化过程中并没有起到提高转化率的作用, 但VIP1具有保守的bZIP结构域, 属于bZIP转录因子家族的I亚族, 可通过与保守的VIP1应答原件VRE结合, 可激活许多防御相关基因的转录, 引起植物对生物胁迫和非生物胁迫的抵御反应[8, 21]。业已证明, VIP1被促分裂原蛋白激酶MPK3 (Mitogen-activated protein kinase 3)磷酸化, 进而迁移到细胞核, 激活防卫基因的表达[11]。在OsVIP1表达被抑制的RNAi水稻植株中, PBZ1基因的表达量相应下降, 而PBZ1基因属于PR10基因家族, 在植物抵御病原菌侵害时表达量上升[22]。AtVIP1还可通过调控ABA途径中的CYP707A1/3基因的表达, 参与拟南芥渗透势信号的转导途径[23]。另外, 抗逆相关基因硫氧还蛋白Trxh8和转录因子MYB44基因的启动子区域都含有特异的VRE区域, 是VIP1蛋白的靶基因, 参与响应假单胞杆菌等多种病菌侵染、损伤和氧化等逆境胁迫[11, 24]。通过对本研究克隆的TaVIP1基因的蛋白质结构域分析, 发现TaVIP1也具有保守的bZIP结构域, 属于bZIP转录因子家族的I亚族[8]。所以, 我们认为TaVIP1基因在提高小麦等植物的抗病和抗逆性方面具有潜在利用价值, 有待进一步鉴定。
4 结论克隆了小麦TaVIP1基因, cDNA全长987 bp, 编码328个氨基酸。证明TaVIP1在普通小麦中存在3个同源基因, 分别位于4AL、4BS和4DS染色体。TaVIP1蛋白分布于细胞膜、细胞质和细胞核上, 过表达小麦TaVIP1基因不能提高烟草转化率。小麦VIP1基因与拟南芥及烟草VIP1基因在序列、结构、亚细胞定位及功能等方面有显著差异, 可能小麦VIP1并非参与外源T-DNA在小麦细胞内的转运和向基因组上的整合, 这可能是小麦农杆菌转化效率较低的原因之一。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] | |
| [24] |
