

1. 西南大学 园艺园林学院 南方山地园艺学教育部重点实验室,重庆 400715;
2. 重庆市蔬菜学重点实验室,重庆 400715
收稿日期:2020-06-22;接收日期:2020-09-03;网络出版时间:2020-10-04
基金项目:重庆市技术创新与应用发展专项(No. cstc2019jscx-gksbX0159),西南大学石柱创新基金(No. SZ201902) 资助
摘要:随着全球气候的不断变化,植物常遭受热胁迫、干旱胁迫、冷胁迫、盐碱胁迫等多种非生物胁迫。植物热激转录因子(Heat shock transcription factors,HSFs) 作为植物体内广泛存在的一类转录因子,能够响应多种非生物胁迫。文中就HSFs的结构、信号调控机制以及其在主要植物拟南芥、番茄、水稻和大豆中的研究进行回顾和总结,以期为进一步阐明HSFs在逆境调控网络中的作用提供参考。
关键词:热激转录因子逆境转录因子调控机制
Heat shock transcription factor family in plants: a review
Nan Zhang1,2*, Yinghong Wang1,2*, Zhimin Wang1,2, Zhenyu Yue1,2, and Yi Niu1,2


1. Key Laboratory of Horticulture Science for Southern Mountainous Regions, Ministry of Education, College of Horticulture and Landscape Architecture, Southwest University, Chongqing 400715, China;
2. Chongqing Key Laboratory of Olericulture, Chongqing 400715, China
Received: June 22, 2020; Accepted: September 3, 2020; Published: October 4, 2020
Supported by: Chongqing Special Key Project of Technology Innovation and Application Development, China (No. cstc2019jscx-gksbX0159), Shizhu Innovation Fund Project of Southwest University (No. SZ201902)
Corresponding author: Yi Niu. Tel/Fax: +86-23-68250731; E-mail: niuy2001134@163.com.
*These authors contributed equally to this study.
Abstract: With the constant change of global climate, plants are often affected by multiple abiotic stresses such as heat stress, drought stress, cold stress and saline-alkali stress. Heat shock transcription factors (HSFs) are a class of transcription factors widely existing in plants to respond to a variety of abiotic stresses. In this article, we review and summarize the structure, signal regulation mechanism of HSFs and some research in plants like Arabidopsis thaliana, tomato, rice and soybean, to provide reference for further elucidating the role of HSFs in the stress regulation network.
Keywords: heat shock transcription factorsstresstranscription factorsregulatory mechanisms
随着全球温室效应的不断加剧,全球平均气温逐年升高,气候变化引起的非生物胁迫对作物生产构成了很大的威胁。根据联合国政府间气候变化专门委员会(Intergovernmental Panel on Climate Change,IPCC) 提供的数据表明,如果人类不改变现在的生活方式,不采取明确行动抑制温室气体排放,那么未来全球平均气温将有50%的可能会上升4 ℃。持续上涨的温度已导致粮食产量急剧下降,如小麦和玉米每10年分别减产1.9%和1.2%。当植物周围环境温度迅速升高10–15 ℃时,就会对植物造成威胁,形成热胁迫[1]。研究表明,热胁迫对大田作物的株高、根长、生物量生产和粮食品质都会产生显著的负面影响[2-3]。不仅如此,热胁迫还会影响植物的光合作用、对矿质元素的吸收、花药的开裂和授粉,甚至严重影响种子的萌发,导致收获的种子产量低、发芽率低[4]。
为了生存,植物形成了复杂且高效的调控网络以抵御和适应外界的胁迫,其中转录因子调控起着关键的作用。在发生热胁迫后,植物通过调节热激蛋白使自身可以耐受一定的热水平,也会通过调节脱落酸(Abscisic acid,ABA)、赤霉素(Gibberellin,GA)、茉莉酸(Jasmonic acid,JA)、吲哚乙酸(Indole-3-acetic acid,IAA) 等植物激素的分泌来增强抗热性[4],而这些过程都与热激转录因子密切相关。
植物热激转录因子是植物中研究最广泛的转录因子家族之一。自1990年Scharf等通过对番茄的细胞培养物进行热激处理,发现了与热激响应元件结合的转录因子HSFs后,关于HSFs的研究开始在多种植物中开展[5]。目前,已经在一些植物中鉴定出了许多HSFs转录因子家族成员,如小麦中含有56个,番茄中含有26个,水稻中含有26个,大豆中含有52个,拟南芥中含有21个等[6-8]。同时,HSFs转录因子作为胁迫响应基因表达信号转导链的终端组成成分,介导响应热或其他胁迫条件的基因激活[9]。现已研究发现,HSFs参与了包括高温胁迫、盐胁迫、干旱胁迫和氧化胁迫等多种逆境响应[10-12]。本文就近些年来植物HSFs转录因子的结构特征、信号调控机制以及其在不同物种、不同非生物胁迫中的抗逆功能进行回顾和总结,以期为深入了解HSFs转录因子响应非生物胁迫的分子机制提供一定的参考。
1 HSFs转录因子的结构特征HSFs是植物响应逆境信号的重要元件,主要包含5大结构域,即DNA结合结构域(DNA binding domain,DBD)、寡聚化结构域(Oligomerization domain,OD)、核定位信号(Nuclear localization signal,NLS)、核输出信号(Nuclear export signal,NES) 和C端转录激活结构域(C-transcriptional activation domain,CTAD)。
1.1 DNA结合结构域高度结构化的DNA结合结构域位于所有HSFs的N端附近,包含一个三螺旋束和一个小的四股反平行的β片,形成一个疏水核心。这种最核心最保守的区域结构是螺旋-转角-螺旋(Helix- Turn-Helix,HTH) 基序的变体,具有分解代谢激活蛋白的特征,也可以精确定位并识别热激启动子(HSE) 序列(5′-AGAAnnTTCT-3′),激活或抑制抗逆相关基因的转录[13-14]。与来自酵母和动物的HSFs转录因子相比,植物HSFs转录因子缺少一个由11个氨基酸残基组成的嵌入到β3和β4之间的环,从而改变了β-链之间的紧密联系[14-15]。
1.2 寡聚化结构域OD与DBD相邻,它包含2个疏水7肽重复区域HR-A和HR-B。HR-A/B最先是Crick提出的由3链组成的螺旋型螺旋卷曲结构和α-螺旋结构区,包含了大量的疏水氨基酸残基。HR-A/B区有2个类型,即Ⅰ型,在植物的HSFsB1和其他非植物的HSFs中都有这种特征结构;Ⅱ型,只存在于植物热激转录因子A1和A2型中,这些七聚氨基酸重复序列中间嵌入21个氨基酸残基,可产生2个重叠的HR基序[16]。同时,HR-A/B可通过可变长度的柔性接头(15–80氨基酸残基) 与DNA结合结构域连接[17]。当植株受到外界高温刺激后,植株体内的HSFs通过由HR-A/B构成的卷曲螺旋结构形成同源或异源三聚体,以促进与热激蛋白(Heat shock protein,HSP) 启动子的HSE元件结合,并诱导HSP的表达[18]。而OD的缺失显著影响HSFs单体间的聚合作用及其与目标基因的结合能力,因而OD是HSFs的重要组成部分。根据OD的独特性,HSFs基因家族可分为A、B和C三族,其中A族和C族的成员在HR-A和HR-B间分别插入21个和7个氨基酸,而B族两者间无插入序列[6, 19]。
1.3 核定位信号和核输出信号HSFs的核定位信号是蛋白输入核内所必需的结构,它与HR-A/B的C端相邻,由一簇富集精氨酸和赖氨酸的碱性氨基酸残基组成[20]。细胞核输出信号位于HSFs的C端,富含亮氨酸,可帮助HSFs从细胞核输出到细胞质。大多数植物HSFs有核定位信号和核输出信号两种结构域,在植物热激响应过程中调控核输出和核输入的平衡,两者的协同作用决定了HSFs在细胞内的分布[19]。
1.4 C端转录激活结构域CTAD是HSFs序列上最不保守的区域,含有短肽AHA (Aromatics,hydrophobic and acidic amino acid residues) 基序,且含有大量的芳香族氨基酸、疏水性氨基酸和酸性氨基酸残基。不同于其他结构域,CTAD是热激转录因子中保守性比较低的序列,但也参与了HSFs的诱导激活和表达,且AHA基序介导A类HSFs,使其具有转录激活因子的功能。一般认为B类和C类HSFs因为缺少此基序而不具备转录激活因子的功能(图 1)[16, 21]。但已有研究发现,B类HSFs具有转录抑制活性,如GmHSFB2b可直接与启动子结合来抑制GmNAC2的表达[22]。而TaHsfC2a是一种转录激活因子,调节小麦中TaHSP70d和TaGalSyn基因的表达[23]。
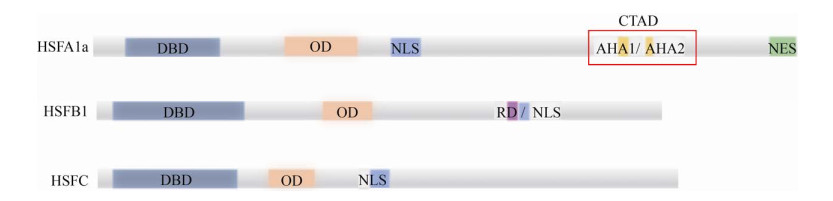 |
| 图 1 HSFs蛋白的基本结构[17] Fig. 1 The basic structures of the HSFs protein[17]. DBD: DNA binding domain; OD: oligomerization domain; NLS: nuclear localization signal; NES: nuclear export signal; CTAD: C-transcriptional activation domain; AHA: aromatics, hydrophobic and acidic amino acid residues; RD: tetrapeptid motif -LFGV- as core of repressor domain. |
| 图选项 |
2 HSFs在主要作物上的研究进展与动物和微生物相比,植物中HSFs的多样性使得对它们的研究变得复杂。然而,随着研究的深入,序列和表达谱比较显示了它们在胁迫和发育中的不同和重叠功能。一般认为,A类HSFs是热胁迫中的主要调节因子,可诱导抗逆基因表达。而B类HSFs正常条件下可抑制耐热基因的转录表达且在植物耐热性防御方面发挥重要的作用。关于C类HSFs的研究报道较少,但其也参与了逆境响应[24]。为了全面地了解植物中的HSFs家族成员,下面分别就其中几种植物热激转录因子的研究概况进行介绍。
2.1 番茄番茄是最早进行热激转录因子研究的植物,研究发现番茄中至少含有26个HSFs家族成员,其中17个属于A类HSFs,8个属于B类,1个属于C类[25]。在番茄热激反应过程中,Mishra发现与野生型番茄(WT) 相比,SlHsfA1过表达的植株(OE) 和SlHsfA1基因转录后沉默形成的植株(CS)在正常生长条件下的主要发育参数相似。然而,在45 ℃的环境温度下暴露1 h,CS植株死亡,而WT和OE植株在其发育过程中没有受到明显的影响。因此,尽管番茄SlHsfs的A类亚家族有17个成员,但SlHsfA1在诱导耐热性方面具有独特的主调节功能[26]。SlHsfA1在番茄中有4个成员(SlHsfA1a、SlHsfA1b、SlHsfA1c和SlHsfA1e),其中只有SlHsfA1a具有主调节功能,这可能是由于DNA结合结构域的差异造成的。研究发现,SlHsfA1a的DNA结合结构域中包含一个精氨酸残基,其位于β3-β4片层中,且在SlHsfA1蛋白中高度保守,而在SlHsfA1c中相同位置则是一个亮氨酸,在SlHsfA1e中则为半胱氨酸。虽然SlHsfA1b在此位置上也为精氨酸,但是包含SlHsfA1b-DBD的SlHsfA1a突变体与SlHsfA1a具有相似活性,而包含SlHsfA1c或SlHsfA1e-DBD的SlHsfA1a突变体活性降低[27]。
SlHsfA2在番茄不同的非生物胁迫反应和耐受性方面起着重要的调控作用[28]。SlHsfA2位于SlHsfA1的下游,可促进HS (Heat shock) 基因的表达,同时也是HSFs在高温下积累的主要因子之一[26, 29]。SlHsfA1和SlHsfA2协同激活基因时,AHA和OD都是必不可少的,而一个完整的DBD只需要两个HSFs中的一个即可。通过GUS染色、免疫共沉淀和HsfA2的核保留实验表明,SlHsfA1和SlHsfA2之间的相互作用取决于其OD的特殊性。尽管OD的序列和基本域结构非常相似,但在A类HSFs的HR-A/B区显然存在微妙但起决定性差异的序列,导致了这对相互作用的HSFs的形成[17, 30]。除此之外,SlHsfA2是耐热型番茄基因型发育中花药热应激系统的重要成员,热胁迫后,SlHsfA2被强烈激活,使其在花药中的表达比在其他器官中更高。通过qRT-PCR、原位RNA杂交、免疫印迹等实验发现,SlHsfA2和另一重要成员Hsp17-CⅡ在花药发育期间被精细调节,并且在短期和长期热胁迫条件下被进一步诱导,直至产生成熟的干花粉[31]。进一步研究发现,SlHsfA2抑制其在减数分裂和小孢子形成阶段受到胁迫的花粉的活力和发芽率,但对其更高阶段没有影响。一般来说,花粉减数细胞和小孢子诱导强热激反应的能力较低,所以其对HS的敏感性较强,但这种敏感性在非胁迫条件下通过发育调控的SlHsfA2和几个SlHsfA1a介导的HS应答基因也可以得到部分缓解[10]。
与SlHsfA1a一样,SlHsfA3也定位在细胞核和细胞质上[27, 32],且SlHsfA3不受外源ABA的影响。因为与对照相比,ABA处理后,SlHsfA3的含量并未显著变化[32]。SlHsfA4b和SlHsfA5对调控SlHsfA1/A2形成的网络起作用,SlHsfA4b替代SlHsfA2中的OD消除了杂合蛋白与SlHsfA1的相互作用,而带有SlHsfA4b-OD的SlHsfA1杂合蛋白又易受SlHsfA5抑制[30]。
番茄SlHsfB1是一种转录抑制因子,可以抑制HS诱导的基因,但也可以作为SlHsfA1a的共激活因子[33-34]。在正常条件下,番茄SlHsfB1蛋白可通过26S蛋白酶体快速降解[35]。在热胁迫下,由于HS诱导的其他HSFs具有更高的活性,SlHsfB1的抑制作用就极大增强了热激蛋白的诱导,与野生型相比,其耐热性增加。此外还发现,SlHsfB1的过表达刺激了SlHsfB1的共激活功能,因此诱导了HS相关蛋白在非应激条件下的积累,改变了各种生物过程中涉及的蛋白质的丰度,包括一些假定的管家功能,如同源的HSPs、翻译延伸因子和蛋白酶体相关蛋白等。同时,SlHsfB1的过表达和抑制都会导致幼苗的耐热性增强,并激活幼叶中的光系统保护机制。此外,Sotirios等还发现SlHsfB1对胁迫诱导的HSFs的启动子起抑制作用[34]。
2.2 拟南芥不同于番茄,在拟南芥中没有发现AtHsfA1a作为主调控因子调控植物应激反应,但其依旧是重要的转录激活物[36-38]。Qian等通过研究拟南芥过表达AtHsfA1植株发现,AtHsfA1a表达水平升高与胁迫响应早期诱导的Hsp18.2和Hsp70表达水平升高相关。通过对转基因植株进行不同的胁迫处理,之后进行抗HsfA1a抗体的甲醛交联和免疫沉淀研究发现,未经胁迫处理或山梨糖醇处理的样品中的免疫沉淀DNA仅产生弱条带,而受热激、低/高pH变化和H2O2胁迫的样品条带清晰。这一结果表明,AtHsfA1a的转录能力受到外源胁迫诱导[39]。而且,AtHsfA1a可以直接感知胁迫并被激活,这一过程依赖于其氧化还原状态[40]。但是与转基因拟南芥中AtHsfA1b的过表达导致HSFs的抑制不同,当AtHsfA1a在转基因植物中过表达时,似乎不能充分抑制HSFs的活性[39]。同时,研究发现拟南芥的4个AtHsfA1亚类基因(AtHsfA1a/b/d/e) 控制了65%以上的热胁迫诱导基因[41]。单敲除拟南芥中AtHsfA1a、AtHsfA1b、AtHsfA1d或AtHsfA1e的突变体在热激响应和拟南芥的长期耐热水平上没有明显的缺陷,但不同的三重突变体AtHsfA1a/b/d、AtHsfA1b/d/e、AtHsfA1a/b/e和AtHsfA1a/d/e以及四重突变体AtHsfA1a/b/d/e,均显示出对抗逆性的不同特异性。三重AtHsfA1a/b/d和四重AtHsfA1a/b/d/e突变体即使在中等高温下也无法适应,同时还发现,AtHsfA1b/d/e三重突变体对盐胁迫敏感,四重AtHsfA1a/b/d/e突变体会出现种子发育缺陷[42-43]。
AtHsfA2作为HSFs信号网络的关键组成部分,参与了细胞对各种环境的应激反应[44],且可以部分取代AtHsfA1在生长发育中的作用[45]。Liu采用Western blotting检测转基因株系(A2QK) 中AtHsfA2、AtHsp101、AtHsp90、AtHsa32和sHsp-CI的水平发现,在正常条件下,A2QK-7、A2QK-10和A2-Wt分别产生了高水平的AtHsfA2蛋白,而A2QK-9和A2QK-12则分别产生了无法检测到的和低水平的AtHsfA2蛋白,且植株中AtHsfA2的蛋白水平与AtHsfA2的转录水平一致。在非胁迫条件下,转基因品系中AtHsfA2的水平与AtHsp101、AtHsa32和sHsp-CI的水平相关,但与Hsp90不相关,表明在没有AtHsfA1的情况下,AtHsfA2可以激活某些Hsp的表达[45]。研究还发现,AtHsfA2转基因株系过表达后,其基础和获得的耐热性显著增强,而当AtHsfA2中插入T-DNA形成拟南芥突变体后,其基础和获得性耐热性降低,同时发现,盐、渗透和缺氧胁迫诱导下,过表达株系对这些胁迫也表现出较强的耐受性[46-47]。除此之外,AtHsfA2在细胞增殖中也起重要作用[46]。
而关于其他A类转录因子在拟南芥中的作用,研究发现UV-B、冷、干旱、臭氧、病原体等胁迫可以诱导AtHSFA4a的表达,当AtHSFA4a过表达后,与对照相比生长减少20%–30%。然而,当处于盐、缺氧或氧化胁迫时,过表达AtHSFA4a的植株则比对照表现更好[8, 48]。进一步研究发现,AtHSFA6b是由盐、渗透和ABA诱导,而不是由HS诱导[49]。
拟南芥AtHsfB1和AtHsfB2b作为热诱导HSFs表达的抑制因子,调节拟南芥获得耐热性。在hsfb1-hsfb2b双突变体植株中,大量热诱导基因在正常条件下(23 ℃) 的表达增强,而在42 ℃时,突变体植株表现出比野生型略高的耐热性,类似于Pro35S:HSFA2植株。此外,在延长热胁迫的条件下,热诱导型HSFs基因在hsfb1-hsfb2b中的表达始终高于野生型。这些结果表明,AtHsfB1和AtHsfB2b在非热胁迫条件下和热胁迫衰减阶段抑制了一般的热冲击响应。另一方面,AtHsfB1和AtHsfB2b似乎是热胁迫条件下诱导热激蛋白基因表达所必需的基因,同时它也是获得耐热性所必需的基因[50]。
2.3 水稻水稻作为单子叶植物的模式植物,对其热激转录因子家族也有一些研究。根据Rice Annotation数据库可知,OsHsf基因家族中有25个已确定的基因,其中13个基因属于A类,8个基因属于B类,其余4个属于C类。而在RGA数据库中搜索时,OsHsf的一个基因为LOC_Os06g226100,其缺少DNA结合结构域,但又确实存在寡聚域,认为它可能是一个变体类型,也视为了HSFs。因此,水稻中发现有26个编码OsHsf家族的基因[51-52]。
Liu等在热处理过程中发现OsHsfA2a在热激转录因子中的转录水平最高,且大多数OsHsfAs表达对盐、聚乙二醇(PEG) 和冷处理的反应主要发生在胁迫响应的后期,如OsHsfA4b、OsHsfA5和5个OsHsfA2s在热、盐和PEG胁迫下表达上调[7]。Wang等在热和H2O2共同处理下发现,A类Hsfs的表达高于B类和C类Hsfs,而OsHsfA2a、OsHsfA2c、OsHsfA2e、OsHsfA4a、OsHSFB2b和OsSHFC1b的转录上调仅在HS处理下检测到,说明水稻存在一个不依赖于H2O2的HS反应途径[53]。而在过表达OsHsfA2e的转基因拟南芥中,某些与胁迫相关的基因高度表达,包括几类热激蛋白,如HSP17.7-CⅡ、HSP26.5-P、HSP70等,且其耐热表型在子叶、莲座叶、花序、茎和种子中均被观察到[54]。对过表达OsHsfA7的转基因水稻进行形态观察和胁迫耐受性测定发现,转基因植株表现出较少、较短的侧根和根毛。而在盐处理下,过表达OsHsfA7的水稻表现出损害症状减轻和存活率较高的现象,且叶片电导率(REC) 和丙二醛(MDA) 含量均低于野生型植物。同时发现,在干旱和复水处理后,转基因水稻幼苗恢复正常生长,但野生型植物无法挽救。这些研究表明,OsHsfA7基因的过表达可以提高水稻幼苗对盐和干旱胁迫的耐受性。观察转基因植株中9个OsHSP基因的表达(OsHsp80.2、OsHsp74.8、OsHsp50.2、OsHsp71.1、OsHsp58.7、OsHsp23.7、OsHsp26.7、OsHsp24.1、OsHsp17.0) 发现,与WT相比,只有OsHsp24.1产生较高的表达。所以,OsHsp24.1可能是OsHsfA7的潜在靶基因,并参与了转基因水稻对高盐分或干旱胁迫的响应[55]。
Xiang等研究表明,水稻OsHsfB2b在热胁迫、盐胁迫、脱落酸和聚乙二醇的诱导下强烈表达,但是在受到冷胁迫时几乎没有受到影响。在干旱胁迫下,与野生型相比,过表达OsHsfB2b的转基因水稻REC和MDA含量增加,脯氨酸(Pro) 含量下降,而在OsHsfB2b-RNAi转基因水稻中,REC和MDA含量降低,Pro含量增加[56]。
OsHsfC1b主要定位于细胞核中,但在细胞溶质中也有少量存在。Romy等发现,盐、甘露醇和ABA诱导了OsHsfC1b的表达,但H2O2却没有。值得注意的是,在根系上,ABA诱导比盐诱导高2倍,在ABA处理30 min和3 h后,其含量分别达到了43倍和33倍。同时发现,OsHsfC1b缺失会导致盐和渗透胁迫耐受性降低,对ABA的敏感性增加以及与信号和离子平衡有关的盐响应基因的表达失调。此外,在盐胁迫下,sHSP基因在OsHsfC1b的敲除系植株中表现出增强的表达。与非胁迫条件下的对照植株相比,OsHsfC1b的敲除系植株生长受到阻碍[57]。
2.4 大豆大豆中含有52个HSFs家族成员,且在胁迫条件下,各HSFs的转录水平存在较大差异,其中Al和A2亚类基因的转录水平异常高。此外,每个基因在不同器官和不同发育阶段的表达水平也存在差异[58]。热胁迫诱导后,包括GmHsf12、GmHsf28、GmHsf34、GmHsf35和GmHsf47在内的5个GmHSFs显著上调。在低温胁迫下,GmHsf12和GmHsf28基因的RNA表达微弱增加,GmHsf35显著上调,GmHsf34和GmHsf47不变。在高盐胁迫下,GmHsf12的RNA表达上调显著,而其他基因直到胁迫后24 h才发生变化。在干旱胁迫下,GmHsf12、GmHsf34、GmHsf35和GmHsf47的转录水平显著上调,而GmHsf28的转录水平变化不大[59]。GmHSFA1属热激转录因子的A1类,为组成型基因。魏崃等发现,过表达的GmHSFA1可促进大豆中HSP70、HSP22、HSP23的转录表达,且GmHSFA1过表达后的植物抗干旱,耐热性能较好[60]。Li等发现GmHsf-34基因的过表达提高了拟南芥对干旱和高温胁迫的耐受性[58]。研究还发现,大多数GmHsf定位在细胞核中,如GmHsf-04、GmHsf-33、GmHsf-34、GmHsf35和 GmHsf47。而GmHsf28既定位在细胞核,又定位在细胞质[58-59]。通过观察大豆转基因毛状根体系和稳定的转基因大豆中过表达GmHsfB2b发现,GmHsfB2b均可以提高大豆的耐盐性。进一步研究表明GmHSFB2b可以直接激活黄酮类化合物合成途径,同时解除另一个转录因子GmNAC2的抑制作用,从而促进黄酮类化合物的合成,降低体内ROS的积累以提高大豆的耐盐性[22]。这些发现将有助于对大豆GmHsf在植物胁迫响应中的功能进行特性分析。
2.5 其他作物在玉米中,ZmHsf05过表达后的拟南芥幼苗增加了植株的基础和获得耐热性。在热胁迫后,与野生型幼苗相比,ZmHsf05过表达株系的存活率和叶绿素含量升高,且HSPs的表达也出现上调,表明ZmHsf05在植物的基础和获得耐热性中起重要作用[61]。MeHsf3通过调节木薯水杨酸的积累和信号传导来调节植物的免疫应答[62]。小麦TaHsfA2bs可能是参与花药发育的候选基因[63],而TaHSFA6在小麦各个器官中组成性表达,并在热激响应下表达上调,同时发现,抗凋亡基因(TaGAAP) 和Rubisco活化酶大型同工酶基因(TaRCA-L) 为TaHSFA6f的靶基因。也就是说,TaHsfA6f的过度表达上调了HSP和其他类型的热应激保护基因的表达,包括高尔基体抗凋亡蛋白和Rubisco活化酶[64]。Liu等进一步研究发现,TaHsfA2-1可以取代AtHsfA2的功能,提高植物的基础耐热性和获得性耐热性[65]。FaHsfC1b过表达提高了植株的存活率、叶绿素含量和光化学效率,同时在热胁迫下,会发生电解质渗漏,H2O2和O2含量降低的现象。通过qRT-PCR显示,过表达FaHsfC1b的转基因植物中热保护蛋白基因包括HSPs、AtGalSyn1、AtRof1和AtHSA32以及ABA合成基因(NCED3)的表达水平均显著上调。因此,认为FaHsfC1b可用作低温季节植物物种的遗传修饰候选基因,用于提高植株的耐热性[11]。在魔芋中发现,AaHSFA2a和AaHSFA2c蛋白均定位在细胞核中,且AaHsfA2a和AaHsfA2c基因在白魔芋叶中的表达量明显高于根和球茎,均在热处理1 h时达到表达顶峰[66]。
3 HSFs的信号转导途径HSFs作为参与逆境响应的主要调控因子,参与包括逆境信号的感知、蛋白质激酶在内的多种信号转导途径。目前研究较多的HSFs信号转导是钙信号转导途径、ROS信号转导途径和ABA信号转导途径。
3.1 HSFs的钙信号转导途径钙离子信号是植物生长发育和环境响应的核心调控因子,且钙信号作为热激信号转导途径中的主要上游组分,可直接或间接地调控HSFs的转录活性。在拟南芥中,热激处理后,AtCaM3、AtCaM7和Hsp18.2基因的表达增加,且AtCaM3表达的上调时间早于AtCaM7或Hsp18.2[67]。Liu等进一步研究发现,CaM结合蛋白激酶AtCBK3可以通过磷酸化AtHSFA1对热激反应起积极的调节作用。观察AtCBK3过表达系和AtCBK3空白系发现,HSFs与HSE的结合活性、Hsp基因mRNA水平和HSP合成在AtCBK3过表达系中上调,而在AtCBK3空白系中下调[68]。在小麦中,37 ℃热激处理期间,对植株进行CaCl2处理后,小麦Hsp26和Hsp70基因表达上调,相反,由于乙二醇双(2-氨基乙基醚)四乙酸(Ethylenebis(oxyethylenenitrilo) tetraacetic acid,EGTA)、氯丙嗪(Chlorpromazine,CPZ)、三氟拉嗪(Trifluoperazine,TFP)或N-(6-氨基己基)-5-氨-1-萘磺酰胺盐酸盐(N-(6- aminohexyl)-5-chloro-1-naphthalenesulfonamide,W7)处理,其含量下降[69]。Cao等研究发现,CaM3是百合Ca2+-CaM HS信号通路的关键成分。当在HS条件下,对百合进行CaCl2处理后,其电解质的泄漏显著减少,存活率升高。相反,EGTA处理加重了电解质渗漏,降低了存活率[70]。这些研究表明,Ca2+/CaM可能参与了HSF-HSP途径,并提高了植株的耐热性。
3.2 HSFs的ROS信号转导途径活性氧(Reactive oxygen species,ROS) 在多种应激中起到第二信使的作用,但植物组织中过量的ROS生成可直接引起氧化损伤,最终损害细胞的正常功能。在植物遭受非生物胁迫期间,ROS对转录因子的作用通常取决于一个或几个半胱氨酸残基的氧化[8],它可以直接影响HSFs寡聚化,也可以通过MAPK途径导致HSFs的进一步活化(图 2)。反之,HSFs会影响ROS基因的表达,也可通过刺激HSP伴侣蛋白的表达来影响ROS基因的表达[71]。Yu等以野生型番茄植株和CRISPR/Cas9介导的SlMAPK3突变体为材料,发现SlMAPK3突变体中SlHsp70、SlHsp90、SlHsp100和SlHsfA1a、SlHsfA2、SlHsfA3的转录水平显著高于野生植株,且SlMAPK3突变体的耐热性增强与抑制ROS生成和激活抗氧化酶有关,通过降低ROS的积累来减轻热胁迫下的氧化损伤[72]。AtHSFA7b可以诱导清除ROS的相关基因,使其活性提高并减少ROS的积累,最终提高了拟南芥的耐盐性[73]。
 |
| 图 2 HSFs信号转导途径的调控网络[8, 12, 71] Fig. 2 Regulatory network of HSFs signal transduction pathways[8, 12, 71]. |
| 图选项 |
3.3 HSFs的ABA信号转导途径HSFs在干旱和盐胁迫信号中起作用。干旱和盐胁迫会诱导ABA的积累及其与受体PYR/PYL/RCAR的结合,导致PP2Cs失活,SnRK2s活性增强并且磷酸化目标蛋白[8]。且SnRK2s结合脱落酸应答元件(ABRE) 后,可诱导ABA依赖型相关效应基因的表达,进而引起植株的生理生化变化。Sung等发现AtHsfA6a转录水平是由ABF/AREB蛋白与AtHsfA6a启动子直接结合引起的[74]。而Huang等进一步研究发现,AtHsfA6b基因也受ABA信号的调控,且其表达位于ABA感知的下游。在非生物胁迫和ABA处理下,AtHsfA6b的表达比AtHsfA6a更早且更强,而AtHsfA6b突变体也会影响AtHsfA6a表达[49]。除拟南芥AtHsfA6基因外,小麦的TaHsfA6f在ABA信号通路也存在一定的作用。TaHsfA6f转基因植株在种子萌发和幼苗生长过程中对ABA敏感。在正常生长条件下,转基因植株的ABA含量显著高于野生植株[75]。同时,在水稻中也发现OsHSFA3对ABA有反应,用1 μmol/L的ABA处理10 d生幼苗发现,与野生型相比,用ABA处理的所有转基因系的根长都显著缩短[76]。
4 展望随着分子生物学和生物信息学的快速发展,许多植物的基因组测序已经完成,HSFs转录因子作为植物抵抗非生物胁迫调控网络的关键因子之一,是目前的研究热点。HSFs转录因子的作用机制主要是和下游基因的HSE元件结合,启动基因表达。目前HSFs调控植物逆境的研究主要集中在A类HSFs上,而B类和C类的研究报道相对较少。同时,研究内容主要以热胁迫为主,对调控其他胁迫响应机制的了解仍很缺乏。已有研究表明HSFs由多基因家族成员编码,具有功能多样性,在不同植物组织、不同逆境条件下存在差异表达。同时,多个HSFs基因可协同调控植物的非生物胁迫,其精准调控逆境响应的网络依旧是研究的难点,迫切需要了解HSF基因在各种胁迫反应中的分子调控机制。因此,HSFs调控植物逆境响应仍有很大探索空间,值得深入系统地研究。
参考文献
| [1] | Lipiec J, Doussan C, Nosalewicz A, et al. Effect of drought and heat stresses on plant growth and yield: a review. Int Agrophys, 2013, 27(4): 463-477. DOI:10.2478/intag-2013-0017 |
| [2] | Nayyar H, Kaur R, Kaur S, et al. γ-aminobutyric acid (GABA) imparts partial protection from heat stress injury to rice seedlings by improving leaf turgor and upregulating osmoprotectants and antioxidants. J Plant Growth Regul, 2014, 33(2): 408-419. DOI:10.1007/s00344-013-9389-6 |
| [3] | Bahuguna RN, Solis CA, Shi WJ, et al. Post-flowering night respiration and altered sink activity account for high night temperature-induced grain yield and quality loss in rice (Oryza sativa L.). Physiol Plant, 2017, 159(1): 59-73. DOI:10.1111/ppl.12485 |
| [4] | Ali S, Rizwan M, Arif MS, et al. Approaches in enhancing thermotolerance in plants: an updated review. J Plant Growth Regul, 2020, 39(1): 456-480. DOI:10.1007/s00344-019-09994-x |
| [5] | Scharf KD, Rose S, Zott W, et al. Three tomato genes code for heat stress transcription factors with a region of remarkable homology to the DNA-binding domain of the yeast HSF. EMBO J, 1990, 9(13): 4495-4501. DOI:10.1002/j.1460-2075.1990.tb07900.x |
| [6] | Nover L, Bharti K, D?ring P, et al. Arabidopsis and the heat stress transcription factor world: how many heat stress transcription factors do we need?. Cell Stress Chaperone, 2011, 6(3): 177-189. |
| [7] | Liu AL, Zou J, Zhang XW, et al. Expression profiles of class A rice heat shock transcription factor genes under abiotic stresses. J Plant Biol, 2010, 53(2): 142-149. DOI:10.1007/s12374-010-9099-6 |
| [8] | Jacob P, Hirt H, Bendahmane A. The heat-shock protein/chaperone network and multiple stress resistance. Plant Biotechnol J, 2017, 15(4): 405-414. DOI:10.1111/pbi.12659 |
| [9] | Lin YX, Jiang HY, Chu ZX, et al. Genome-wide identification, classification and analysis of heat shock transcription factor family in maize. BMC Genomics, 2011, 12: 76. DOI:10.1186/1471-2164-12-76 |
| [10] | Fragkostefanakis S, Mesihovic A, Simm S, et al. HsfA2 controls the activity of developmentally and stress-regulated heat stress protection mechanisms in tomato male reproductive tissues. Plant Physiol, 2016, 170(4): 2461-2477. DOI:10.1104/pp.15.01913 |
| [11] | Zhuang LL, Cao W, Wang J, et al. Characterization and functional analysis of FaHsfC1b from Festuca arundinacea conferring heat tolerance in Arabidopsis. Int J Mol Sci, 2018, 19(9): 2702. DOI:10.3390/ijms19092702 |
| [12] | Guo M, Liu JH, Ma X, et al. The plant heat stress transcription factors (HSFs): structure, regulation, and function in response to abiotic stresses. Front Plant Sci, 2016, 7: 114. |
| [13] | Harrison CJ, Bohm AA, Nelson HC. Crystal structure of the DNA binding domain of the heat shock transcription factor. Science, 1994, 263(5144): 224-227. DOI:10.1126/science.8284672 |
| [14] | Nover L, Scharf KD, Gagliardi D, et al. The Hsf world: classification and properties of plant heat stress transcription factors. Cell Stress Chaperones, 1996, 1(4): 215-223. DOI:10.1379/1466-1268(1996)001<0215:THWCAP>2.3.CO;2 |
| [15] | Schultheiss J, Kunert O, Gase U, et al. Solution structure of the DNA-binding domain of the tomato heat-stress transcription factor HSF24. Eur J Biochem, 1996, 236(3): 911-921. DOI:10.1111/j.1432-1033.1996.00911.x |
| [16] | 封雷, 胡海涛. 热激转录因子在植物防御反应中的作用. 安徽农业科学, 2011, 39(4): 1931-1934, 1950. Feng L, Hu HT. Heat shock transcription factors on defense responses in plant. J Anhui Agric Sci, 2011, 39(4): 1931-1934, 1950 (in Chinese). DOI:10.3969/j.issn.0517-6611.2011.04.017 |
| [17] | Scharf KD, Berberich T, Ebersberger I, et al. The plant heat stress transcription factor (Hsf) family: Structure, function and evolution. Biochim Biophys Acta, 2012, 1819(2): 104-119. DOI:10.1016/j.bbagrm.2011.10.002 |
| [18] | 李丽, 刘双清, 杨远航, 等. 热激转录因子在植物抗非生物胁迫中的功能研究进展. 生物技术进展, 2018, 8(3): 214-220. Li L, Liu SQ, Yang YH, et al. Progress on the function of heat shock transcription factors in plant abiotic stress tolerance. Curr Biotechnol, 2018, 8(3): 214-220 (in Chinese). |
| [19] | 田尉婧, 殷学仁, 李鲜, 等. 热激转录因子调控植物逆境响应研究进展. 园艺学报, 2017, 44(1): 179-192. Tian WJ, Yin XR, Li X, et al. Regulation of stress responses by heat stress transcription factors (Hsfs) in plants. Acta Horticult Sin, 2017, 44(1): 179-192 (in Chinese). |
| [20] | 宫本贺. 百合热激转录因子L1HSFA1及其下游热激蛋白L1HSP70响应热胁迫的机制解析[D]. 北京: 中国农业大学, 2014. Gong BH. Mechanism analysis of response to heat stress of L1HSFA1 and its downstream L1HSP70 from lily (Lilium longiflorum)[D]. Beijing: China Agricultural University, 2014 (in Chinese). |
| [21] | D?ring P, Treuter E, Kistner C, et al. The role of AHA motifs in the activator function of tomato heat stress transcription factors HsfA1 and HsfA2. Plant Cell, 2000, 12(2): 265-278. DOI:10.1105/tpc.12.2.265 |
| [22] | Bian XH, Li W, Niu CF, et al. A class B heat shock factor selected for during soybean domestication contributes to salt tolerance by promoting flavonoid biosynthesis. New Phytol, 2020, 225(1): 268-283. DOI:10.1111/nph.16104 |
| [23] | Hu XJ, Chen DD, Mclntyre CL, et al. Heat shock factor C2a serves as a proactive mechanism for heat protection in developing grains in wheat via an ABA-mediated regulatory pathway. Plant Cell Environ, 2018, 41(1): 79-98. DOI:10.1111/pce.12957 |
| [24] | 焦淑珍, 姚文孔, 张宁波, 等. 园艺植物热激转录因子研究进展. 果树学报, 2020, 37(3): 419-430. Jiao SZ, Yao WK, Zhang NB, et al. Research progress of heat stress transcription factors(Hsfs) in horticultural plants. J Fruit Sci, 2020, 37(3): 419-430 (in Chinese). |
| [25] | Yang XD, Zhu WM, Zhang H, et al. Heat shock factors in tomatoes: genome-wide identification, phylogenetic analysis and expression profiling under development and heat stress. PeerJ, 2016, 4: e1961. DOI:10.7717/peerj.1961 |
| [26] | Mishra SK, Tripp J, Winkelhaus S, et al. In the complex family of heat stress transcription factors, HsfA1 has a unique role as master regulator of thermotolerance in tomato. Genes Dev, 2002, 16(12): 1555-1557. DOI:10.1101/gad.228802 |
| [27] | El-Shershaby A, Ullrich S, Simm S, et al. Functional diversification of tomato HsfA1 factors is based on DNA binding domain properties. Gene, 2019, 714: 143985. DOI:10.1016/j.gene.2019.143985 |
| [28] | Fragkostefanakis S, Simm S, Paul P, et al. Chaperone network composition in Solanum lycopersicum explored by transcriptome profiling and microarray meta-analysis. Plant Cell Environ, 2015, 38(4): 693-709. DOI:10.1111/pce.12426 |
| [29] | Hahn A, Bublak D, Schleiff E, et al. Crosstalk between Hsp90 and Hsp70 chaperones and heat stress transcription factors in tomato. Plant Cell, 2011, 23(2): 741-755. DOI:10.1105/tpc.110.076018 |
| [30] | Chan-Schaminet KY, Baniwal SK, Bublak D, et al. Specific interaction between tomato HsfA1 and HsfA2 creates hetero-oligomeric superactivator complexes for synergistic activation of heat stress gene expression. J Biol Chem, 2009, 284(31): 20848-20857. DOI:10.1074/jbc.M109.007336 |
| [31] | Giorno F, Wolters-Arts M, Grillo S, et al. Developmental and heat stress-regulated expression of HsfA2 and small heat shock proteins in tomato anthers. J Exp Bot, 2010, 61(2): 453-462. DOI:10.1093/jxb/erp316 |
| [32] | Li ZJ, Zhang LL, Wang AX, et al. Ectopic overexpression of SlHsfA3, a heat stress transcription factor from tomato, confers increased thermotolerance and salt hypersensitivity in germination in transgenic Arabidopsis. PLoS ONE, 2013, 8(1): e54880. DOI:10.1371/journal.pone.0054880 |
| [33] | Bharti K, Von Koskull-D?ring P, Bharti S, et al. Tomato heat stress transcription factor HsfB1 represents a novel type of general transcription coactivator with a histone-like motif interacting with the plant CREB binding protein ortholog HAC1. Plant Cell, 2004, 16(6): 1521-1535. DOI:10.1105/tpc.019927 |
| [34] | Fragkostefanakis S, Simm S, El-Shershaby A, et al. The repressor and co-activator HsfB1 regulates the major heat stress transcription factors in tomato. Plant Cell Environ, 2019, 42(3): 874-890. DOI:10.1111/pce.13434 |
| [35] | R?th S, Mirus O, Bublak D, et al. DNA-binding and repressor function are prerequisites for the turnover of the tomato heat stress transcription factor HsfB1. Plant J, 2017, 89(1): 31-44. DOI:10.1111/tpj.13317 |
| [36] | Lee JH, Hübel A, Sch?ffl F. Derepression of the activity of genetically engineered heat shock factor causes constitutive synthesis of heat shock proteins and increased thermotolerance in transgenic Arabidopsis. Plant J, 1995, 8(4): 603-612. DOI:10.1046/j.1365-313X.1995.8040603.x |
| [37] | Pr?ndl R, Hinderhofer K, Eggers-Schumacher G, et al. HSF3, a new heat shock factor from Arabidopsis thaliana, derepresses the heat shock response and confers thermotolerance when overexpressed in transgenic plants. Mol Gen Genet, 1998, 258(3): 269-278. DOI:10.1007/s004380050731 |
| [38] | Lohmann C, Eggers-Schumacher G, Wunderlich M, et al. Two different heat shock transcription factors regulate immediate early expression of stress genes in Arabidopsis. Mol Gen Genet, 2004, 271(1): 11-21. DOI:10.1007/s00438-003-0954-8 |
| [39] | Qian J, Chen J, Liu YF, et al. Overexpression of Arabidopsis HsfA1a enhances diverse stress tolerance by promoting stress-induced Hsp expression. Genet Mol Res, 2014, 13(1): 1233-1243. DOI:10.4238/2014.February.27.8 |
| [40] | Liu YF, Zhang CX, Chen J, et al. Arabidopsis heat shock factor HsfA1a directly senses heat stress, pH changes, and hydrogen peroxide via the engagement of redox state. Plant Physiol Biochem, 2013, 64: 92-98. DOI:10.1016/j.plaphy.2012.12.013 |
| [41] | Liu HC, Liao HT, Charng YY. The role of class A1 heat shock factors (HSFA1s) in response to heat and other stresses in Arabidopsis. Plant Cell Environ, 2011, 34(5): 738-751. DOI:10.1111/j.1365-3040.2011.02278.x |
| [42] | Liu HC, Charng YY. Acquired thermotolerance independent of heat shock factor A1(HsfA1), the master regulator of the heat stress response. Plant Signal Behavior, 2012, 7(5): 547-550. DOI:10.4161/psb.19803 |
| [43] | Yoshida T, Ohama N, Nakajima J, et al. Arabidopsis HsfA1 transcription factors function as the main positive regulators in heat shock-responsive gene expression. Mol Genet Genomics, 2011, 286(5/6): 321-332. |
| [44] | Nishizawa-Yokoi A, Nosaka R, Hayashi H, et al. HsfA1d and HsfA1e involved in the transcriptional regulation of HsfA2 function as key regulators for the Hsf signaling network in response to environmental stress. Plant Cell Physiol, 2011, 52(5): 933-945. DOI:10.1093/pcp/pcr045 |
| [45] | Liu HC, Charng YY. Common and distinct functions of Arabidopsis class A1 and A2 heat shock factors in diverse abiotic stress responses and development. Plant Physiol, 2013, 163(1): 276-290. DOI:10.1104/pp.113.221168 |
| [46] | Ogawa D, Yamaguchi K, Nishiuchi T. High-level overexpression of the Arabidopsis HsfA2 gene confers not only increased themotolerance but also salt/osmotic stress tolerance and enhanced callus growth. J Exp Bot, 2007, 58(12): 3373-3383. DOI:10.1093/jxb/erm184 |
| [47] | Banti V, Mafessoni F, Loreti E, et al. The heat-inducible transcription factor HsfA2 enhances anoxia tolerance in Arabidopsis. Plant Physiol, 2010, 152(3): 1471-1483. DOI:10.1104/pp.109.149815 |
| [48] | Pérez-Salamó I, Papdi C, Rigó G, et al. The heat shock factor A4A confers salt tolerance and is regulated by oxidative stress and the mitogen-activated protein kinases MPK3 and MPK6. Plant Physiol, 2014, 165(1): 319-334. DOI:10.1104/pp.114.237891 |
| [49] | Huang YC, Niu CY, Yang CR, et al. The heat stress factor HSFA6b connects ABA signaling and ABA-mediated heat responses. Plant Physiol, 2016, 172(2): 1182-1199. |
| [50] | Ikeda M, Mitsuda N, Ohme-Takagi M. Arabidopsis HsfB1 and HsfB2b act as repressors of the expression of heat-inducible Hsfs but positively regulate the acquired thermotolerance. Plant Physiol, 2011, 157(3): 1243-1254. DOI:10.1104/pp.111.179036 |
| [51] | Mittal D, Chakrabarti S, Sarkar A, et al. Heat shock factor gene family in rice: Genomic organization and transcript expression profiling in response to high temperature, low temperature and oxidative stresses. Plant Physiol Biochem, 2009, 47(9): 785-795. DOI:10.1016/j.plaphy.2009.05.003 |
| [52] | Guo JK, Wu J, Ji Q, et al. Genome-wide analysis of heat shock transcription factor families in rice and Arabidopsis. J Genet Genom, 2008, 35(2): 105-118. DOI:10.1016/S1673-8527(08)60016-8 |
| [53] | Wang C, Zhang Q, Shou HX. Identification and expression analysis of OsHsfs in rice. Journal of Zhejiang University Science B, 2009, 10(4): 291-300. DOI:10.1631/jzus.B0820190 |
| [54] | Yokotani N, Ichikawa T, Kondou Y, et al. Expression of rice heat stress transcription factor OsHsfA2e enhances tolerance to environmental stresses in transgenic Arabidopsis. Planta, 2008, 227(5): 957-967. DOI:10.1007/s00425-007-0670-4 |
| [55] | Liu AL, Zou J, Liu CF, et al. Over-expression of OsHsfA7 enhanced salt and drought tolerance in transgenic rice. BMB Rep, 2013, 46(1): 31-36. DOI:10.5483/BMBRep.2013.46.1.090 |
| [56] | Xiang JH, Ran J, Zou J, et al. Heat shock factor OsHsfB2b negatively regulates drought and salt tolerance in rice. Plant Cell Rep, 2013, 32(11): 1795-1806. DOI:10.1007/s00299-013-1492-4 |
| [57] | Schmidt R, Schippers JHM, Welker A, et al. Transcription factor OsHsfC1b regulates salt tolerance and development in Oryza sativa ssp. japonica. AoB Plants, 2012, 2012: pls011. |
| [58] | Li PS, Yu TF, He GH, et al. Genome-wide analysis of the Hsf family in soybean and functional identification of GmHsf-34 involvement in drought and heat stresses. BMC Genomics, 2014, 15(1): 1009. DOI:10.1186/1471-2164-15-1009 |
| [59] | Chung E, Kim KM, Lee JH. Genome-Wide analysis and molecular characterization of heat shock transcription factor family in Glycine max. J Genet Genom, 2013, 40(3): 127-135. DOI:10.1016/j.jgg.2012.12.002 |
| [60] | 魏崃, 吴广锡, 唐晓飞, 等. 过表达GmHSFA1大豆在干旱条件下对高温的响应. 大豆科学, 2016, 35(2): 257-261. Wei L, Wu GX, Tang XF, et al. Soybean responses to high temperatures under drought stress in the presence of an over-expressed GmHSFA1 gene. Soybean Sci, 2016, 35(2): 257-261 (in Chinese). |
| [61] | Li GL, Zhang HN, Shao HB, et al. ZmHsf05, a new heat shock transcription factor from Zea mays L. improves thermotolerance in Arabidopsis thaliana and rescues thermotolerance defects of the athsfa2 mutant. Plant Sci, 2019, 283(375): 384. |
| [62] | Wei YX, Liu GY, Chang YL, et al. Heat shock transcription factor 3 regulates plant immune response through modulation of salicylic acid accumulation and signalling in cassava. Mol Plant Pathol, 2018, 19(10): 2209-2220. DOI:10.1111/mpp.12691 |
| [63] | Ye JL, Yang XT, Hu G, et al. Genome-wide investigation of heat shock transcription factor family in wheat (Triticum aestivum L.) and possible roles in anther development. Int J Mol Sci, 2020, 21(2): 608. DOI:10.3390/ijms21020608 |
| [64] | Xue GP, Drenth J, McIntyre CL. TaHsfA6f is a transcriptional activator that regulates a suite of heat stress protection genes in wheat (Triticum aestivum L.) including previously unknown Hsf targets. J Exp Bot, 2015, 66(3): 1025-1039. DOI:10.1093/jxb/eru462 |
| [65] | Liu ZH, Li GL, Zhang HN, et al. TaHsfA2-1, a new gene for thermotolerance in wheat seedlings: Characterization and functional roles. J Plant Physiol, 2020, 246-247: 153135. DOI:10.1016/j.jplph.2020.153135 |
| [66] | 张蓓, 胡文霞, 靳燚, 等. 白魔芋热激转录因子AaHSFA2a和AaHSFA2c的分离、亚细胞定位及表达分析. 分子植物育种, 2018, 16(24): 8005-8012. Zhang B, Hu WX, Jin Y, et al. Isolation, subcellular localization and expression analysis of heat shock transcription factors AaHSFA2a and AaHSFA2c in Amorphophallus albus. Mol Plant Breed, 2018, 16(24): 8005-8012 (in Chinese). |
| [67] | Liu HT, Sun DY, Zhou RG. Ca2+ and AtCaM3 are involved in the expression of heat shock protein gene in Arabidopsis. Plant Cell Environ, 2005, 28(10): 1276-1284. DOI:10.1111/j.1365-3040.2005.01365.x |
| [68] | Liu HT, Gao F, Li GL, et al. The calmodulin-binding protein kinase 3 is part of heat-shock signal transduction in Arabidopsis thaliana. Plant J, 2008, 55(5): 760-773. DOI:10.1111/j.1365-313X.2008.03544.x |
| [69] | Liu HT, Li B, Shang ZL, et al. Calmodulin is involved in heat shock signal transduction in wheat. Plant Physiol, 2003, 132(3): 1186-1195. DOI:10.1104/pp.102.018564 |
| [70] | Cao X, Yi J, Wu Z, et al. Involvement of Ca2+ and CaM3 in regulation of thermotolerance in Lily (Lilium longiflorum). Plant Mol Biol Rep, 2013, 31(6): 1293-1304. DOI:10.1007/s11105-013-0587-y |
| [71] | Driedonks N, Xu JM, Peters JL, et al. Multi-level interactions between heat shock factors, heat shock proteins, and the redox system regulate acclimation to heat. Front Plant Sci, 2015, 6: 999. |
| [72] | Yu WQ, Wang L, Zhao RR, et al. Knockout of SlMAPK3 enhances tolerance to heat stress involving ROS homeostasis in tomato plants. BMC Plant Biol, 2019, 19: 354. DOI:10.1186/s12870-019-1939-z |
| [73] | Zang DD, Wang JX, Zhang X, et al. Arabidopsis heat shock transcription factor HSFA7b positively mediates salt stress tolerance by binding to an E-box-like motif to regulate gene expression. J Exp Bot, 2019, 70(19): 5355-5374. DOI:10.1093/jxb/erz261 |
| [74] | Hwang SM, Kim DW, Woo MS, et al. Functional characterization of Arabidopsis HsfA6a as a heat-shock transcription factor under high salinity and dehydration conditions. Plant Cell Environ, 2014, 37(5): 1202-1222. DOI:10.1111/pce.12228 |
| [75] | Bi HH, Zhao Y, Li HH, et al. Wheat heat shock factor TaHsfA6f increases ABA levels and enhances tolerance to multiple abiotic stresses in transgenic plants. Int J Mol Sci, 2020, 21(9): 3121. DOI:10.3390/ijms21093121 |
| [76] | Zhu MD, Zhang M, Gao DJ, et al. Rice OsHSFA3 gene improves drought tolerance by modulating polyamine biosynthesis depending on abscisic acid and ROS levels. Int J Mol Sci, 2020, 21(5): 1857. DOI:10.3390/ijms21051857 |
